Доклады Российской академии наук. Науки о жизни, 2020, T. 493, № 1, стр. 353-358
ИСПОЛЬЗОВАНИЕ ГЕНЕТИЧЕСКИ КОДИРУЕМЫХ ФЛУОРЕСЦЕНТНЫХ АПТАМЕРОВ ДЛЯ ВИЗУАЛИЗАЦИИ МАЛОЙ РНК MYCOBACTERIUM TUBERCULOSIS MTS1338 В ИНФИЦИРОВАННЫХ МАКРОФАГАХ
О. С. Быченко 1, *, Ю. В. Скворцова 1, А. С. Григоров 1, Т. Л. Ажикина 1
1 Федеральное государственное бюджетное учреждение науки Институт биоорганической химии
им. академиков М.М. Шемякина и Ю.А. Овчинникова Российской академии наук
Москва, Россия
* E-mail: bychenko.oksana@gmail.com
Поступила в редакцию 19.03.2020
После доработки 16.04.2020
Принята к публикации 16.04.2020
Аннотация
На примере малой РНК Mycobacterium tuberculosis MTS1338 впервые визуализирована субклеточная локализация малой некодирующей бактериальной РНК в инфицированных макрофагах. Исследуемая малая РНК была экспрессирована в микобактериях в виде модуля с РНК-аптамером, способным связывать флуорофор и активировать его флуоресценцию. Созданным микобактериальным трансформантом инфицирована клеточная линия макрофагов. Обработка инфицированных макрофагов флуорофором DFHBI-1T позволила детектировать флуоресценцию РНК модуля MTS1338-аптамер как непосредственно в микобактериях, так и в цитоплазме инфицированных макрофагов. Данная система важна в исследованиях по выявлению роли малых РНК M. tuberculosis в патогенезе туберкулеза, процессов их секреции, поиска эукариотических мишеней, а также молекулярных путей, на которые могут оказывать воздействие эти малые РНК.
Малые некодирующие РНК бактерий модулируют широкий спектр физиологических ответов и регулируют ключевые этапы жизнедеятельности у целого ряда возбудителей заболеваний [1, 2]. В последнее время обсуждается идея о том, что малые РНК внутриклеточных патогенных бактерий могут не только адаптировать собственный транскриптом под меняющиеся условия, но также взаимодействовать с транскриптомом инфицированного организма, вмешиваясь тем самым в процессы антибактериальной защиты. Выявление и исследование молекулярных цепей адаптации патогенных бактерий к иммунной защите хозяина при персистировании в макрофагах представляет собой важную научную проблему.
В данной работе исследуется малая некодирующая РНК Mycobacterium tuberculosis MTS1338, которая специфична только для микобактерий туберкулезного комплекса, и уровень ее экспрессии in vivo чрезвычайно высок и составляет приблизительно 10–1 относительно уровня экспрессии 16S rRNA [3]. Ранее было показано, что MTS1338 вносит существенный вклад во взаимодействие M. tuberculosis с макрофагами, адаптируя бактериальный транскриптом к агрессивной внутримакрофагальной среде. Изменения в метаболизме бактерий, вызываемые гиперэкспрессией этой малой РНК, приводят к модуляции иммунного ответа [4].
Существуют примеры того, как некодирующие бактериальные РНК внутриклеточных патогенов могут секретироваться в цитоплазму инфицированных клеток и модулировать иммунный ответ во время инфекции [5, 6]. Предполагается, что малая РНК M. tuberculosis MTS1338 также может секретироваться и влиять на защитные клетки иммунной системы. Для изучения этого процесса востребована система визуализации малых РНК бактерий с помощью флуоресцентной микроскопии.
Общий подход для визуализации исследуемой РНК в клетке – экспрессия этой РНК совместно с РНК-аптамером, способным связывать небольшую молекулу-флуорофор и активировать ее флуоресценцию (так называемая модульная РНК) [7]. Добавляя флуорофор в среду инкубирования клеток, экспрессирующих модульную РНК, мы имеем возможность определить клеточную локализацию малой РНК. Изначально методика была основана на использовании в качестве аптамера РНК, связывающую из раствора зеленый флуоресцентный белок (GFP) [8], однако для этой и других подобных методик требуется ко-экспрессия флуоресцентных белков в клетке, что может привести к неспецифической флуоресценции белков, не связавшихся с РНК [7]. В альтернативной стратегии используются последовательности РНК, которые благодаря своей особой третичной структуре способны связывать из раствора непосредственно саму молекулу-флуорофор и активировать ее флуоресценцию [9]. Как было показано Paige с соавторами [10], структурный аналог GFP, флуорофор DFHBI, (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-1,2-dimethyl-1H-imidazol-5(4H)-one), не вызывал неспецифическую флуоресценцию при его активации различными компонентами клеток, такими как клеточная РНК или ДНК. Среди последовательностей РНК-аптамеров хорошо зарекомендовала себя т.н. Broccoli, а также ее производные. Broccoli достаточно короткая (49 п.н.), обладает стабильной вторичной структурой, формирование которой мало зависит от биохимического состава среды, а также имеет яркую зеленую флуоресценцию при взаимодействии с модифицированным аналогом DFHBI, DFHBI-1T [11]. Была продемонстрирована возможность применения производных Broccoli для детекции некодирующих РНК в различных организмах [12, 13].
В серии ранее опубликованных работ было выяснено, что использование различных вариантов аптамера Broccoli может приводить к разной интенсивности флуоресценции и изменять правильный фолдинг исследуемой РНК [11–13]. С целью подбора оптимальных по размеру и яркости конструкций в данной работе использовались различные по длине аптамеры. Были сконструированы модульные РНК MTS1338 с Broccoli, 2xdBroccoli, F30-Broccoli, F30-2xdBroccoli, обладающие разными размерами и яркостью флуоресценции. C помощью сервера RNAfold (http://rna.-tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi) для всех сконструированных РНК была показана высокая вероятность образования правильной вторичной структуры, содержащей два функциональных домена – MTS1338 и аптамер (рис. 1А). Структура модульных РНК и используемые для их получения олигонуклеотидные праймеры приведены в табл. 1.
Рис. 1.
(а) Вторичная структура РНК модулей MTS1338_Broccoli, MTS1338_2xdBroccoli, MTS1338_F30-Broccoli, MTS1338_F30-2xdBroccoli согласно серверу RNAfold. Часть модуля, соответствующая MTS1338, выделена прямоугольником. (б) Флуоресценция модульных РНК in vitro. Модульные РНК (100 нг) фракционировали в 6% полиакриламидном геле, инкубировали гель в растворе флуорофора (10 мкМ DFHBI-1Т, 40 мМ HEPES pH 7.4, 100 мМ KCl, 1 мМ MgCl2) и анализировали с помощью флуоресцентного визуализатора ZOE Fluorescent Cell Imager (Bio-Rad) с использованием зеленого канала прибора.

Таблица 1.
Нуклеотидные последовательности генетических конструкций и олигонуклеотиды, использованные для их получения
| Название | Последовательность |
|---|---|
| rrnB-промотор | CTAGAGTGACCGCGTCTGACCAGGGAAAATAGCCCTCTGACCTGGGGATTTGACTCCCAGTTTCCAAGGACGTAACTTA |
| MTS1338 | ACCGGGGAAACCCGGTGATCTGCCCGAAGTGCTGGGCGATTGAGCGGGTATGTACACCCGGTTTGACCTACCGTCCCAAGACGGGGCTACCGCCTTCGGGCAGATCCTCATCCTGTT |
| term-терминатор | GCTTCCCCGCGAAAGCGGGGTTTTTTTTTTTAGCT |
| Broccoli | GAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTC |
| 2xdBroccoli | GAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTC |
| F30-Broccoli | TTGCCATGTGTATGTGGGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCCCACATACTCTGATGATCCTTCGGGATCATTCATGGCAA |
| F30-2xdBroccoli | TTGCCATGTGTATGTGGGAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTCCCACATACTCTGATGATCCAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTGGATCATTCATGGCAA |
| rrnB-MTS- 1338-F30-2xdBroccoli-term | CTAGAGTGACCGCGTCTGACCAGGGAAAATAGCCCTCTGACCTGGGGATTTGACTCCCAGTTTCCAAGGACGTAACTTAACCGGGGAAACCCGGTGATCTGCCCGAAGTGCTGGGCGATTGAGCGGGTATGTACACCCGGTTTGACCTACCGTCCCAAGACGGGGCTACCGCCTTCGGGCAGATCCTCATCCTGTTTTGCCATGTGTATGTGGGAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTCCCACATACTCTGATGATCCAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTGGATCATTCATGGCAAGCTTCCCCGCGAAAGCGGGGTTTTTTTTTTTAGCT |
| rrnB-F30-2xdBroccoli-term | CTAGAGTGACCGCGTCTGACCAGGGAAAATAGCCCTCTGACCTGGGGATTTGACTCCCAGTTTCCAAGGACGTAACTTATTGCCATGTGTATGTGGGAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTCCCACATACTCTGATGATCCAGACGGTCGGGTCCATCTGAGACGGTCGGGTCCAGATATTCGTATCTGTCGAGTAGAGTGTGGGCTCAGATGTCGAGTAGAGTGTGGGCTGGATCATTCATGGCAAGCTTCCCCGCGAAAGCGGGGTTTTTTTTTTTAGCT |
| Олигонуклеотиды, 5'-3' | |
| Broc_F | GAGACGGTCGGGTCCAGATATTCGTATCTG |
| Broc_R | GAGCCCACACTCTACTCGACAGATACGAAT |
| 2xdBroc_F | GAGACGGTCGGGTCCATCTG |
| 2xdBroc_R | GAGCCCACACTCTACTCGACATC |
| Prom1338_F | CTAGAGTGACCGCGTCTGA |
| Term1338_R | AGCTAAAAAAAAAAACCCCGCTT |
| T7Pr1338 | GTTTTTTTTAATACGACTCACTATAGGACCGGGGAAACCCG |
| T7PrBroc | GTTTTTTTTAATACGACTCACTATAGGGAGACGGTCGGGTC |
| T7PrF30Broc | GTTTTTTTTAATACGACTCACTATAGGTTGCCATGTGTATG |
| pMV261_F | GATCTACGTGGCGAACTCCG |
| pMV261_R | ATGCCTGGCAGTCGATCGTA |
Для того, чтобы убедиться в возможности укладки модульной РНК в правильную вторичную структуру, при которой РНК может флуоресцировать при взаимодействии с DFHBI-1T, сконструированные модульные РНК синтезировали in vitro с использованием набора T7 RiboMAX™ Express Large Scale RNA Production System (Promega), разделяли в полиакриламидном геле, инкубировали в растворе флуорофора DFHBI-1Т и анализировали с помощью флуоресцентного визуализатора ZOE Fluorescent Cell Imager (Bio-Rad) с использованием зеленого канала прибора (рис. 1б). Максимальная флуоресценция наблюдалась для РНК MTS1338-F30-2xdBroccoli, поэтому дальнейшие эксперименты проводили с этим аптамером.
В составе шаттл-вектора pMV261, способного реплицироваться как в E. coli, так и в микобактериях [14], генетическая конструкция MTS1338-F30-2xdBroccoli была трансформирована в Myco-licibacterium smegmatis MC2 155. Эти непатогенные бактерии используются в качестве суррогатного организма при изучении M. tuberculosis, поскольку их геномы, а также метаболизм очень сходны, при этом M. smegmatis обладает значительно большей скоростью роста и гораздо легче подвергается генетической модификации [15]. M. smegmatis не содержит гена исследуемой малой РНК.
Для визуализации модульных РНК в бактериях внутри макрофагов проводили инфицирование клеточной культуры макрофагов мыши RAW 264.7 (ATCC® TIB-71™) штаммом M. smegmatis MTS1338-F30-2xdBroccoli. Макрофаги, выращенные на покровных стеклах в среде RPMI-1640, 10% FCS при 5% CO2, 37°C до конфлуэнтности 70%, инфицировали с MOI 10:1 (множественность инфекции – количество бактерий на один макрофаг). Инфицирование проводили в течение 1 ч. После инфекции среду удаляли, промывали макрофаги PBS и анализировали препараты на конфокальном микроскопе Eclipse TE2000 (Nikon, Япония) (рис. 2). На полученных изображениях наблюдалась колокализация зеленых (флуоресцирующие модульные РНК) и красных (бактерии) сигналов, что свидетельствует о том, что модульная РНК, содержащая MTS1338, синтезируется, складывается в корректную вторичную структуру и взаимодействует с флуорофором в микобактериях, поглощенных макрофагами. Также были отмечены зеленые сигналы в цитоплазме макрофагов, окружающие бактерии. Возможно, эти сигналы указывают на секрецию в цитоплазму макрофагов MTS1338 в составе модульной РНК, что будет проверено нами в дальнейшем.
Рис. 2.
Визуализация генетически кодируемой модульной РНК MTS1338-F30-2xdBroccoli внутри макрофагов RAW 264.7, инфицированных M. smegmatis. Схема эксперимента приведена слева; prom, term – промотор и терминатор соответственно. Конфокальная микроскопия (справа). Клеточные ядра окрашивали Hoechst 33258 (9 мкM) в течение 15 мин во время инфекции. Детекцию бактерий (красный) и бактериальных модульных РНК (зеленый) осуществляли путем добавления красного красителя мембран FM4-64 (8 мкM) в инкубационную среду бактерий за 30 мин до инфицирования макрофагов и флуорофора DFHBI-1T (40 мкM, согласно [12]) при визуализации.
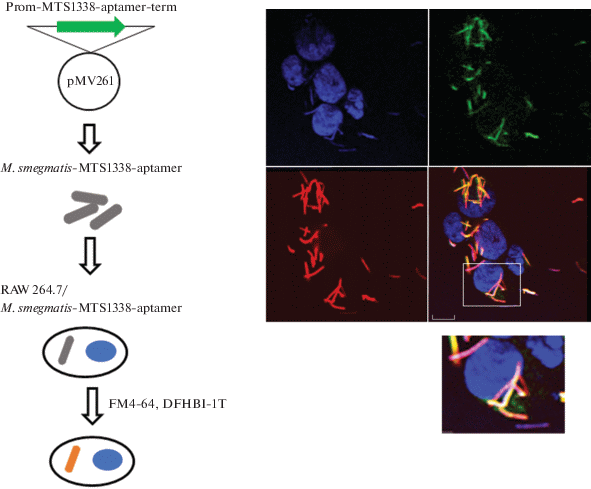
Таким образом, мы впервые продемонстрировали возможность детекции методом флуоресцентной микроскопии малых бактериальных РНК при персистировании бактерий внутри макрофагов. Данная система в дальнейшем будет использована в исследованиях по выявлению роли малых РНК M. tuberculosis в патогенезе туберкулеза, процессов секреции этих РНК в инфицированных макрофагах, поиска их эукариотических мишеней, а также молекулярных путей, на которые могут оказывать воздействие эти малые РНК.
Список литературы
Azhikina T.L., Ignatov D.V., Salina E.G. et al Role of Small Noncoding RNAs in Bacterial Metabolism // Biochemistry (Mosc). 2015. V. 80. № 13. P. 1633–1646.
Holmqvist E., Wagner E.G.H. Impact of bacterial sRNAs in stress responses // Biochem Soc Trans. 2017. V. 45. № 6. P. 1203–1212.
Ignatov D.V., Timoshina O., Logunova N.N. et al. Expression of Mycobacterium tuberculosis small RNAs in mice models of tuberculosis // Bioorg Khim. 2014. V. 40. № 2. P. 253–256.
Salina E.G., Grigorov A., Skvortsova Y. et al. MTS1338, A Small Mycobacterium tuberculosis RNA, Regulates Transcriptional Shifts Consistent With Bacterial Adaptation for Entering Into Dormancy and Survival Within Host Macrophages // Front Cell Infect Microbiol. 2019. V. 9. P. 405.
Gu H., Zhao C., Zhang T. et al. Salmonella produce microRNA-like RNA fragment Sal-1 in the infected cells to facilitate intracellular survival // Sci Rep. 2017. V. 7. № 1. P. 2392.
Pagliuso A., Tham T.N., Allemand E. et al. An RNA-Binding Protein Secreted by a Bacterial Pathogen Modulates RIG-I Signaling // Cell Host Microbe. 2019. V. 26. № 6. P. 823–835.
Tyagi S. Imaging intracellular RNA distribution and dynamics in living cells // Nat. Methods. 2009. V. 6. № 5. P. 331–338.
Bertrand E., Chartrand P., Schaefer M. et al. Localization of ASH1 mRNA particles in living yeast // Mol. Cell. 1998. V. 2. № 4. P. 437–445.
Babendure J.R., Adams S.R., Tsien R.Y. Aptamers switch on fluorescence of triphenylmethane dyes // J. Am. Chem. Soc. 2003. V. 125. № 48. P. 14716–14717.
Paige J.S., Wu K., Jaffrey S.R. RNA mimics of green fluorescent protein // Science. 2011. V. 333. № 6042. P. 642–646.
Filonov G.S., Moon J.D., Svensen N. et al. Broccoli: rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution // J. Am. Chem. Soc. 2014. V. 136. № 46. P. 16299–16308.
Filonov G.S., Jaffrey S.R. RNA Imaging with Dimeric Broccoli in Live Bacterial and Mammalian Cells // Curr. Protoc. Chem. Biol. 2016. V. 8. № 1. P. 1–28.
Zinskie J.A., Roig M., Janetopoulos C. et al. Live-cell imaging of small nucleolar RNA tagged with the broccoli aptamer in yeast // FEMS Yeast Res. 2018. V. 53. № 6. P. 333–337.
Stover C.K., de la Cruz V.F., Fuerst T.R. et al. New use of BCG for recombinant vaccines // Nature. 1991. V. 351. № 6326. P. 456–460.
Chaturvedi V., Dwivedi N., Tripathi R.P. et al. Evaluation of Mycobacterium smegmatis as a possible surrogate screen for selecting molecules active against multi-drug resistant Mycobacterium tuberculosis // J. Gen. Appl. Microbiol. 2007. V. 53. № 6. P. 333–337.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни


