Доклады Российской академии наук. Науки о жизни, 2022, T. 502, № 1, стр. 54-59
НАПРАВЛЕННАЯ ИНАКТИВАЦИЯ ГЕНОВ dhfr, glul, bak1, bax МЕТОДОМ МУЛЬТИПЛЕКСНОГО ГЕНОМНОГО РЕДАКТИРОВАНИЯ КЛЕТОК CHO
Н. А. Орлова 1, *, Л. К. Даянова 2, Е. А. Гаямова 1, М. В. Синегубова 1, С. В. Ковнир 1, И. И. Воробьев 1, 2
1 Институт биоинженерии им. академика
К.Г. Скрябина, ФИЦ “Фундаментальные основы биотехнологии” РАН
Москва, Россия
2 Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН
Москва, Россия
* E-mail: nobiol@gmail.com
Поступила в редакцию 01.11.2021
После доработки 05.11.2021
Принята к публикации 05.11.2021
- EDN: BAQHXY
- DOI: 10.31857/S2686738922010188
Аннотация
Линия клеток яичника китайского хомячка CHO широко используется в биофармацевтическом производстве. Редактирование генома позволяет улучшить ростовые свойства клеток, их ауксотрофность, работу систем индукции апоптоза и аутофагии. Одновременное редактирование многих генов позволяет получить линию с требуемым генотипом быстрее, чем при последовательных геномных нокаутах, но с меньшей вероятностью. При одновременном редактировании генов dhfr, glul, bak1, bax в геноме клеток CHO S получено 24 клона с признаками ауксотрофности по тимидину и глутамину. Среди них обнаружено пять dhfr+/- клонов, все они содержали нокаут одного или двух аллелей glul. В одном клоне было инактивировано сдвигом рамки 7 целевых аллелей из 8 и частично инактивирован второй аллель dhfr инсерцией триплета GAA, что снизило активность фермента в 2.5 раза. Вероятность одновременного нокаута обоих аллелей dhfr была увеличена до 50% при редактировании генома парой гидовых РНК, направленных к одному экзону гена dhfr.
Культивируемые клетки яичника китайского хомячка (СНО) широко применяются для получения терапевтических белков. Создание промышленных линий-продуцентов обычно предполагает использование метаболических селекционных маркеров – дигидрофолатредуктазы (DHFR) и/или глутаминсинтетазы (GS, ген Glul). Сублинии клеток CHO, с повреждениями генов соответствующих ферментов CHO, а именно клетки линий CHO DUX-B11 (dhfr+/–), CHO DG-44 (dhfr–/–), CHO K1SV (glul–/–), позволяют существенно увеличить эффективность отбора стабильно трансфицированных популяций или клонов клеток. Также для линий DUX-B11 и DG-44 становится возможной многостадийная геномная амплификация целевого гена под действием возрастающих концентраций ингибитора DHFR метотрексата (MTX). Линия DG-44, полученная при помощи двух последовательных раундов ненаправленного мутагенеза, несет множественные хромосомные аберрации (1, 2) и значительно отличается от интактных клеток CHO K1 по метаболическим свойствам. Ранее мы показали, что удельная продуктивность клеток CHO S, стабильно трансфицированных плазмидой, кодирующей цепи фолликулостимулирующего гормона человека (ФСГ), примерно вдвое выше, чем для DG-44, а копийность целевого гена в геноме CHO S вдвое меньше, чем у DG-44 (3). Интактные клетки CHO не обеспечивают эффективной амплификации трансгенов под действием MTX – 3 раунда амплификации позволили увеличить уровень секреции ФСГ в 2 раза для клеток CHO S, и в 4–8 раз для DG-44 и аналогичных белков. Сублинии CHO K1 или CHO S, содержащие нокауты всех аллелей генов dhfr и/или glul, но не содержащие значимого числа случайных мутаций и перестроек генома, представляют большой практический интерес. Такие сублинии ранее были получены как для dhfr (4), так и для glul (5) при помощи нуклеаз с доменом цинкового пальца (zinc finger nucleases, TALEN), но они не могут быть использованы в производстве из-за ограничений, налагаемых правообладателями. Сублинии клеток CHO с двойным нокаутом dhfr–/–glul–/– не описаны. Ранее мы показали, что котрансфекция клеток CHO плазмидами с маркерами устойчивости DHFR и GS позволяет существенно увеличить суммарную продуктивность клеток по сравнению с использованием одного селекционного маркера (6).
Помимо обеспечения возможности эффективной метаболической селекции, геномное редактирование клеток СНО может использоваться для изменения профиля пост-трансляционных модификаций секретируемых белков (например, для экспрессии афукозилилованных рекомбинантных антител (7)), метаболической инженерии и для улучшения ростовых характеристик культуры путем регуляции клеточного цикла, индукции апоптоза и аутофагии. В частности, оверэкспрессия пары белков Bcl-2 и Beclin-1 приводит к повышению клеточной плотности в долговременной культуре (8). При нокауте генов проапоптотических факторов BAX (BCL2 associated X), BAK1 (BCL2 antagonist/killer 1), при помощи эндонуклеаз с доменом цинкового пальца (9), наблюдалось увеличение титра IgG при препаративной транзиентной трансфекции в 3–4 раза.
Мы предположили, что геномное редактирование клеток CHO S ферментом spCas9 может проводиться в мультиплексном режиме, т.е. одновременно в отношении четырех генов – dhfr, glul, bax, bak1, используя по одной оптимальной гидовой РНК (гРНК) к каждому гену, что позволит сократить процедуру селекции отредактированных клонов (табл. 1). Для этого клетки трансфицировали плазмидой, кодирующей нуклеазу Cas9 и ген устойчивости к пуромицину, и смесью четырех плазмид, кодирующих гРНК, направленные к экзонам генов-мишеней. Гидовые РНК для нокаутов bax и bak1 описаны в (10), кандидатные гРНК для dhfr и glul рассчитывали алгоритмом CRISPY (11). Все гРНК собирали из пар олигонуклеотидов (Евроген, Россия) и клонировали по сайту BstV2I в плазмиду pKS-gRNA-BB (12), для экспрессии Cas9 использовали плазмиду pX459dU6, полученную путем удаления рестрикцией PciI-XbaI области U6 промотора из бифункциональной плазмиды pX459 (Ran, Hsu et al. 2013).
Таблица 1.
Названия и целевые последовательности исследуемых вариантов гРНК
| Целевой ген | Название гРНК | Последовательность специфической части гРНК и PAM-сайт * |
|---|---|---|
| BAK1 (BCL2 Antagonist/Killer 1) | BAK | GGAAGCCGGTCAAACCACGT TGG |
| BAX (BCL2 Associated X) | BAX | GCTGATGGCAACTTCAACTG GGG |
| DHFR (Dihydrofolate Reductase) | D1 | GGTGATCGCTGCTGCTGTCA TGG |
| D2 | GATGCCCATATTCTGGGACA CGG | |
| D3 | GCAAGAACGGAGACCTTCCC TGG | |
| D4 | GTCGCCGTGTCCCAGAATAT GGG | |
| GS (GLUL, Glutamate-Ammonia Ligase, Glutamine Synthetase) | G1 | GCCATGTATATCTGGGTTGAT TGG |
| G2 | GCGCTGCAAAACCCGCACCC TGG | |
| G3 | GCTTGATGTTTTTGTTCAAG TGG | |
| G6 | GGCCTCCTCGATGTGCCTGG TGG |
Клетки линии CHO S (Invitrogen, США) культивировали в среде ProCHO 5 (Lonza, Швейцария) c 8 мМ L-глутамина (Invitrogen), 2 мМ гипоксантина и 2 мМ тимидина (HT, Панэко, Россия). Нуклеофекцию проводили аппаратом Neon (Invitrogen). Клетки после трансфекции растили 48 ч при +32oС, затем на +37°C 3 дня с добавлением пуромицина (Gibco, США) от 8 до 16 мкг/мл, затем 48 ч без пуромицина. Поликлональные популяции анализировали или клонировали методом предельных разведений в среде SAFC EX-CELL CHO Cloning (Sigma, США). Клональные линии культивировали в среде ProCHO5 в 24-луночных планшетах, отобранные клоны масштабировали и культивировали в суспензионной культуре.
Тестовое редактирование генома проводили для четырех наборов плазмид, кодирующих гРНК – по одному варианту гРНК для генов DHFR и GS для каждой смеси (табл. 1). Выбор оптимальных гРНК проводили по деградации гетеродуплексов эндонуклеазой T7 (13) набором T7 Endonuclease I-based Mutation Detection with the EnGen® Mutation Detection Kit (New England Biolabs, США), а также по анализу гетерогенности данных секвенирования по Сэнгеру продуктов ПЦР целевых областей генов алгоритмами tIDE (Tracking of Indels by Decomposition) (14) и Synthego ICE (15).
Результаты теста на деградацию гетеродуплексов (рис. 1) ограниченно согласуются с результатами расчета долей измененных аллелей генов по данным секвенирования (табл. 2); для дальнейшего использования выбраны варианты гРНК с максимальной эффективностью редактирования по данным TIDE и Synthego ICE и, в случае DHFR, наличием видимых специфических продуктов распада гетеродуплекса.
Рис. 1.
Электрофореграммы продуктов деградации гетеродуплексов T7-эндонуклеазой. Разделение продуктов гидролиза в 1% агарозном геле, окраска бромистым этидием. Изображения инвертированы и контрастированы. а – ампликон целевой области гена dhfr, 488230-488774 в геномной сборке NW_003614442.1. б – ампликон первой целевой области гена glul, 1429912-1430484 в геномной сборке NW_003613921.1. в – ампликон второй целевой области гена glul, 1434716-1435536 в геномной сборке NW_003614442.1 . Обозначения: Cas9 – трансфекция клеток плазмидой pX459dU6, D1 – D4, G1 – G6 – трансфекция плазмидами, кодирующими соответствующие гРНК, CHO S – интактные клетки, +контроль – контрольные продукты ПЦР из состава набора.
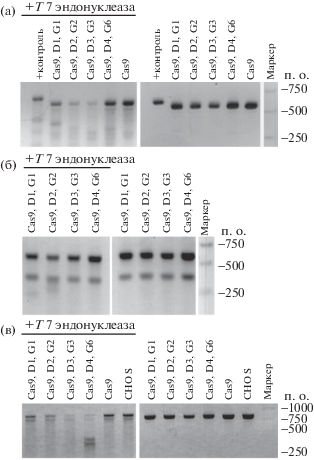
Таблица 2.
Эффективность вариантов гРНК, направленных к генам DHFR и GS
| Образец | Процент редактированных копий гена, % | Алгоритм | |||
|---|---|---|---|---|---|
| Ампликон | DHFR | GS1 | |||
| Праймер | F | R | F | R | |
| D1, G1 | 35 | 46 | T7d | ||
| 46 | 26 | T7p | |||
| 19.6 | 10.4 | 15 | 13.6 | TIDE | |
| 5 | 0 | 6 | 7 | SYN | |
| D2, G2 | 64 | 53 | T7d | ||
| 40 | 32 | T7p | |||
| 5.3 | 9.5 | 0 | 16.7 | TIDE | |
| 0 | н/о | 3 | 3 | SYN | |
| D3, G3 | 64 | 22 | T7d | ||
| 44 | 0 | T7p | |||
| 13.5 | 3.9 | 0.1 | 1.9 | TIDE | |
| 2 | 0 | 0 | 0 | SYN | |
| D4, G6 | 23 | 19* | T7d | ||
| 17 | 0* | T7p | |||
| 2.2 | н/о | 24.3 | 33.8 | TIDE | |
| 0 | н/о | 19 | 23 | SYN | |
* – гРНК G6 не направлена к области ампликона GS1, результат приведен как отрицательное контрольное значение. н/о – не определяли, F – секвенирование +цепи, R – секвенирование -цепи, T7d – степень распада ампликона под действием T7 эндонуклеазы, T7p – степень образования специфических продуктов распада гетеродуплекса, SYN – анализ по алгоритму Synthego ICE. Полужирным шрифтом отмечены максимальные значения показателей.
Мультиплексное редактирование генома клеток CHO S вели для гРНК D3, G6, BAK, BAX, селекцию вели в присутствии 8–16 мкг/мл пуромицина, для клонирования использовали популяции, полученные с 8 и 10 мкг/мл пуромицина. Первичный анализ клональных линий вели путем посева реплик колоний в лунки двух планшетов со средой, не содержащей глутамина, либо не содержащей HT. Из 80 активно делящихся клональных линий отобрано 24, потерявших способность к делению в отсутствие как глутамина, так и HT.
Из 24 линий только для 5 были обнаружены события продуктивного редактирования гена DHFR по данным секвенирования продуктов ПЦР, и только для данных 5 линий обнаружены события редактирования гена GS, все найденные события редактирования приведены в табл. 3. При мультиплексном редактировании генома клеток CHO с использованием четырех гРНК возможен нокаут 7 из 8 целевых аллелей. Эффективность метаболического отбора линий не превышает 20%; такая стадия отбора может быть пропущена без риска потери перспективных кандидатов.
Таблица 3.
Изменения целевых участков генома в клональных линиях, полученных при мультиплексном редактировании
| Код клональной линии | |||||
|---|---|---|---|---|---|
| Ген | 8/3* | 10/14* | 10/18 | 10/19* | 10/22* |
| DHFR | –16/0 | –10/0 | –4 | –3 | –5/+3 |
| GS | +1 | –27/ → 250** | –8 | +3/–3 | +184 |
| BAX | –15 | –15 | –15 | –15*** | +1 |
| BAK1 | –15/+3 | +1/0, +1/0 со сдвигом 8 | н/о | -13 | н/о |
Два числа, разделенных значком “/” – различные изменения в двух аллелях, одно число – одинаковое изменение в обоих аллелях, либо изменение одного аллеля и обширная делеция во втором аллеле; * – два независимых выделения геномной ДНК, результаты анализа полностью совпадают; ** – два продукта ПЦР различного размера, раздельный анализ секвенированием; *** – сдвиг начала делеции на +29 от PAM-сайта.
Поскольку однократное мультиплексное редактирование генома не позволило получить нокауты всех целевых аллелей, была исследована возможность повышения вероятности редактирования гена при помощи пары гРНК, направленных к одной области гена. Редактирование генома клеток CHO S повторно провели с гРНК D1 и D3, направленными к одному экзону dhfr и демонстрировавшими достаточную эффективность редактирования. Популяции клеток, полученные для концентраций пуромицина 8–14 мкг/мл, объединяли и клонировали, как указано выше.
Получено 66 активно делящихся клональных линий, для 19 линий с наибольшей скоростью деления проведено секвенирование целевой области гена dhfr (рис. 2). Эффективность получения генотипа dhfr–/– составила около 50%. При проверке ауксотрофности для данных 19 линий наибольшее ингибирование деления клеток в отсутствие HT обнаружено для клонов А2, А11, А15, А19; таким образом, метаболический тест не позволяет выявлять линии с генотипом dhfr–/– с приемлемой точностью.
Рис. 2.
Изменения в гене dhfr в клональных линиях, полученных при редактировании с парой гРНК. Обозначения: н/и – данные не интерпретируемы из-за множественных сдвигов рамки в аллелях; * – инсерция некодирующей ДНК E. coli; ** – посторонняя последовательность в одном из аллелей. Данные для каждого клона из групп с одинаковым генотипом приведены как “имя клона, изменения в PAM-сайте для гРНК D1, изменения в PAM-сайте гРНК D3”, n = 19. Остальные обозначения аналогично табл. 3.

Для линий 10/22 и А11 с генотипами dhfr+/– и dhfr–/– определили удельную ферментативную активность DHFR набором Dihydrofolate Reductase Assay Kit (Sigma) со специфическим ингибированием DHFR 1.5 мкМ MTX (Тэва, Израиль). Для линии 10/22 была обнаружена инсерция трех нуклеотидов, кодирующих Glu, в сохранившемся аллеле dhfr, что могло изменить удельную активность фермента. Активность DHFR для лизата интактных клеток CHO-S составила 1.99 ± 0.39 мЕд/мг; для линии 10/22 0.39 ± 0.16 мЕд/мг; для линии А11 –0.1 ± 0.21 мЕд/мг. Удельная активность DHFR для линии 10/22 составила 20% от активности в интактных клетках, таким образом, инсерция Glu уменьшила удельную активность DHFR в 2.5 раза, однако остаточная активность DHFR достоверно отличается от фонового значения. В случае линии A11 с генотипом dhfr–/– ферментативная активность DHFR не обнаруживается, что подтверждает корректность прямого анализа последовательностей редактированных областей генома.
В результате исследования способов проведения редактирования генома клеток CHO установлено, что мультиплексное редактирование позволяет получить до 7 событий нокаута для одной линии из 24 проанализированных, а редактирование одного гена при помощи пары близкорасположенных гРНК дает вероятность нокаута обоих аллелей около 50%. Проведение мультиплексного редактирования клеток CHO при помощи множественных пар гРНК будет предметом дальнейших исследований. Разработанный метод редактирования может быть применен для получения линий клеток CHO с нокаутами групп генов. Проведение мультиплексного редактирования генома клеток CHO с высокой вероятностью редактирования также может быть совмещено с оверэкспрессией одного или нескольких генов домашнего хозяйства, кодируемых плазмидами с различными селекционными маркерами.
Список литературы
Cao Y., Kimura S., Park J.Y., et al. Chromosome identification and its application in Chinese hamster ovary cells // BMC Proc. 2011. V. 5. Suppl 8:O8.
Derouazi M., Martinet D., Besuchet Schmutz N., et al. Genetic characterization of CHO production host DG44 and derivative recombinant cell lines // Biochem Biophys Res Commun. 2006. V. 340. № 4. P. 1069–1077.
Orlova N.A., Kovnir S.V., Khodak Y.A., et al. High-level expression of biologically active human follicle stimulating hormone in the Chinese hamster ovary cell line by a pair of tricistronic and monocistronic vectors // PloS one. 2019. V. 14. № 7. P. e0219434.
Santiago Y., Chan E., Liu P.Q., et al. Targeted gene knockout in mammalian cells by using engineered zinc-finger nucleases // Proceedings of the National Academy of Sciences of the United States of America. 2008. V. 105. № 15. P. 5809–5814.
Fan L., Kadura I., Krebs L.E., et al. Improving the efficiency of CHO cell line generation using glutamine synthetase gene knockout cells // Biotechnology and bioengineering. 2012. V. 109. № 4. P. 1007–1015.
Ковнир С.В., Орлова Н.А., Ходак Ю.А., и др. Управляемая коамплификация генов для получения рекомбинантных белков терапевтического назначения: исследование динамики инсерции и амплификации генетических кассет в геноме клеток яичника китайского хомячка при коэкспрессии пары совместимых плазмид // Бюлл. Эксп. Биол. Мед. 2017. Т. 163. № 2. С. 245–249.
Heffner K.M., Wang Q., Hizal D.B., et al. Glycoengineering of Mammalian Expression Systems on a Cellular Level // Adv Biochem Eng Biotechnol. 2021. V. 175. P. 37–69.
Lee J.S., Ha T.K., Park J.H., Lee G.M. Anti-cell death engineering of CHO cells: co-overexpression of Bcl-2 for apoptosis inhibition, Beclin-1 for autophagy induction // Biotechnology and bioengineering. 2013. V. 110. № 8. P. 2195–2207.
Macaraeg N.F., Reilly D.E., Wong A.W. Use of an anti-apoptotic CHO cell line for transient gene expression // Biotechnology progress. 2013. V. 29 № 4. P. 1050–1058.
Grav L.M., Lee J.S., Gerling S., et al. One-step generation of triple knockout CHO cell lines using CRISPR/Cas9 and fluorescent enrichment // Biotechnol. J. 2015. V. 10. № 9. P. 1446–1456.
Ronda C., Pedersen L.E., Hansen H.G., et al. Accelerating genome editing in CHO cells using CRISPR Cas9 and CRISPy, a web-based target finding tool // Biotechnology and bioengineering. 2014. V. 111. № 8. P. 1604–1616.
Tarasevich A., Filatov A., Pichugin A., Mazurov D. Monoclonal antibody profiling of cell surface proteins associated with the viral biofilms on HTLV-1 transformed cells // Acta Virol. 2015. V. 59. № 3. P. 247–256.
Sentmanat M.F., Peters S.T., Florian C.P., et al. A Survey of Validation Strategies for CRISPR-Cas9 Editing // Scientific reports. 2018. V. 8. № 1. P. 888.
Brinkman E.K., Chen T., Amendola M., van Steensel B. Easy quantitative assessment of genome editing by sequence trace decomposition // Nucleic acids research. 2014. V. 42. № 22. P. e168.
Hsiau T., Maures T., Waite K., et al. Inference of CRISPR Edits from Sanger Trace Data // bioRxiv. 2018. P. 251082.
Дополнительные материалы отсутствуют.
Инструменты
Доклады Российской академии наук. Науки о жизни


