Физиология растений, 2019, T. 66, № 5, стр. 339-353
Доставка CRISPR/Cas-компонентов в клетки высших растений для редактирования их геномов
Б. Р. Кулуев a, *, Г. Р. Гумерова a, Е. В. Михайлова a, Г. А. Геращенков a, Н. А. Рожнова a, З. Р. Вершинина a, А. В. Князев a, Р. Т. Матниязов a, Ан. Х. Баймиев a, Ал. Х. Баймиев a, А. В. Чемерис a
a Институт биохимии и генетики – обособленное структурное подразделение Федерального государственного бюджетного учреждения науки Уфимского федерального исследовательского центра Российской академии наук
Уфа, Россия
* E-mail: kuluev@bk.ru
Поступила в редакцию 05.08.2018
После доработки 10.02.2019
Принята к публикации 11.02.2019
Аннотация
Геномное редактирование на основе технологии CRISPR/Cas в биологии растений производится в трех основных вариантах: нокаутные мутации в виде инделов, вставка чужеродных фрагментов ДНК и однонуклеотидные замены путем дезаминирования азотистых оснований. Наиболее важными этапами CRISPR/Cas геномного редактирования являются выбор мишени, дизайн гидовых РНК, создание генно-инженерных конструкций и доставка CRISPR/Cas-компонентов в растительную клетку. Стремительное развитие событий в области редактирования растительных геномов с помощью CRISPR/Cas-систем требует более подробного рассмотрения последнего этапа, поэтому данный обзор посвящен описанию основных способов доставки CRISPR/Cas-компонентов в клетки высших растений. В первых работах по геномному редактированию разных видов растений доставка CRISPR/Cas-компонентов к месту редактирования производилась преимущественно с помощью Agrobacterium tumefaciens. Она предполагала встраивание Т-ДНК в геном и стабильную экспрессию CRISPR/Cas-компонентов или их транзиентную экспрессию в случае агроинфильтрации. Широкое распространение также получили способы доставки CRISPR/Cas-компонентов в клетки растений с использованием растительных вирусов, при котором последние в основном использовались для наработки в увеличенных количествах гидовой РНК, что заметно повышало эффективность редактирования. Для доставки CRISPR/Cas-компонентов в клетки растений предложено также использовать другую агробактерию – Agrobacterium rhizogenes, вызывающую образование волосовидных корней, что косвенно свидетельствует о произошедшем геномном редактировании и может помочь в отборе генетически измененных форм. Довольно распространенным способом получения растений с редактированным геномом являются биобаллистическая доставка в растительные экспланты генно-инженерных конструкций и различные технологии трансформации протопластов. В обзоре также рассмотрены некоторые вопросы принадлежности растений, полученных в ходе CRISPR/Cas-редактирования, к трансгенным растениям и ГМО. Отмечается ряд примеров, когда созданные с помощью CRISPR/Cas-технологии новые организмы, в которых не произошло внедрения чужеродной ДНК, за трансгенные не принимались, и вполне вероятно, что и в России такие растения не будут подпадать под запрет на возделывание ГМО.
ВВЕДЕНИЕ
Открытие у бактерий защитных систем на основе повторяющихся элементов CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) повлекло за собой разработку методов CRISPR/Cas геномного редактирования [1, 2], после чего в мире начался самый настоящий бум вокруг данной технологии. Причем первую из процитированных работ даже считают важнейшей публикацией в области молекулярной биологии в наступившем столетии [3] и это, наверное, справедливо, но останется ли она таковой надолго – уже другой вопрос.
CRISPR/Cas-технология геномного редактирования состоит из нескольких этапов, каждый из которых важен по-своему. Геномное редактирование начинается с выбора задач и мишеней, под которыми понимается, что предстоит сделать экспериментатору – нарушить ли работу какого-то конкретного гена либо изменить свойства кодируемого им белка, или внедрить в растение некий новый ген. Так, существует несколько вариантов геномного редактирования на основе системы CRISPR/Cas: 1) нокаутные мутации, при которых происходит нарушение какого-либо гена, в том числе негативно влияющего на важный для человека признак, сопровождаемое инсерциями или делециями ряда нуклеотидов в участке редактирования за счет негомологичного объединения репарируемых концов фрагментов ДНК в месте их двухцепочечного разрыва (нокаутные мутации обозначают как SDN-1, от Site-Directed Nucleas); 2) так называемый нокин (от knock-in) вариант геномного редактирования, когда за счет гомологичной рекомбинации в определенные участки генома по местам двухцепочных разрывов происходят встраивания иных нуклеотидных последовательностей в виде олигонуклеотидов или более протяженных фрагментов ДНК как нарушающих работу целевого гена, так и приводящих к появлению новых желательных признаков (нокин-варианты нынешнего CRISPR/Cas-редактирования фактически соответствуют SDN-2 и SDN-3 подходам); 3) однонуклеотидные замены, производимые путем дезаминирования азотистых оснований с помощью “сшитых” с каталитически неактивными нуклеазами соответствующих дезаминаз без образования двухцепочечных разрывов ДНК и без делеций или инсерций, обеспечивающие появление новых признаков либо изменение имеющихся, включая нокаутирование генов вследствие образования терминирующих кодонов. На следующем этапе под выбранные мишени и задачи осуществляется дизайн гидовых РНК (гидРНК) с помощью разнообразных компьютерных программ. Ранее нами был проведен достаточно подробный анализ таковых, в том числе ориентированных на редактирование геномов растений [4]. Задача данного этапа заключается в поиске наиболее оптимальных мест в выбранном гене или ином участке ДНК для проведения соответствующего варианта геномного редактирования с целью достижения наибольшей эффективности и точности этого процесса, сводя к минимуму редактирование нецелевых участков (off-target активность). Далее проводится создание генно-инженерных конструкций, несущих необходимые для редактирования гены подходящих Cas-нуклеаз и гидРНК, а также донорной ДНК, или образование из Cas-нуклеаз и гидРНК соответствующих рибонуклеопротеидных частиц, после чего становится необходимо доставить эти CRISPR/Cas-компоненты в растительную клетку. На заключительном этапе требуется проверка правильности редактирования генома с помощью тех или иных методов, включая полногеномное секвенирование.
В данной статье будет сделан акцент только на одном из этапов геномного редактирования, а именно на способах доставки CRISPR/Cas-компонентов к местам внесения изменений в геноме растений. В литературе имеются несколько обзорных статей, где определенное внимание уделено этим вопросам [5–9], однако стремительное развитие событий в области редактирования растительных геномов с помощью CRISPR/Cas-систем настоятельно требует рассмотреть данный этап геномного редактирования растений более подробно.
Собственно для геномного редактирования с помощью CRISPR/Cas-технологии требуется присутствие в участке редактирования комплекса из гидРНК и подходящей Cas-нуклеазы, однако их доставка в растительную клетку может производиться разными способами, которые можно подразделить на те, что ведут к образованию трансгенных растений, поскольку все или некоторые CRISPR/Cas-компоненты внедряются в геном редактируемого растения, и на те, в ходе которых возникают нужные мутации, а чужеродная ДНК в геноме не появляется. Последний вариант геномного редактирования можно назвать “бесследным”, так как производится оно без участия ДНК (в англоязычной литературе известным как DNA-free). В случае нокин варианта CRISPR/Cas-редактирования, приводящего к образованию по сути трансгенных растений (даже без встраивания участков ДНК, кодирующих Cas-нуклеазу и гидРНК), дополнительно требуется введение некой донорной ДНК, которая может быть представлена как в виде коротких олигонуклеотидов, так и довольно протяженных участков ДНК в двух- или одноцепочечном состоянии. В целом способы доставки CRISPR/Cas-компонентов для геномного редактирования можно разделить на две категории – прямые и непрямые [9]. В случае первых происходит непосредственно внедрение CRISPR/Cas-компонентов в растительную клетку за счет того или иного физического воздействия, а для вторых необходимо использование промежуточного “хозяина” в виде агробактерий и/или модифицированных растительных вирусов.
ДОСТАВКА CRISPR/Cas-КОМПОНЕНТОВ В РАСТИТЕЛЬНЫЕ КЛЕТКИ С ПОМОЩЬЮ Agrobacterium tumefaciens
Поскольку первые варианты растений с редактированными геномами мало отличались от получения привычных трансгенных растений, то неудивительно, что существующие способы создания последних были применены и для доставки CRISPR/Cas-компонентов в растительные клетки. Часто используемой системой для получения большинства трансгенных двудольных растений, а также некоторых видов однодольных, служили агробактерии Agrobacterium tumefaciens, несущие подходящие генно-инженерные конструкции на основе бинарных векторов. Этот же подход стал довольно широко применяться при проведении геномного редактирования. Так, уже в первых работах по геномному редактированию разных видов растений (Arabidopsis thaliana L., табака Nicotiana tabacum L., риса Oryza sativa L., сорго Sorghum bicolor (L.) Moench.), доставку CRISPR/Cas-компонентов в клетки производили с помощью A. tumefaciens, предполагая встраивание Т-ДНК и стабильную экспрессию трансгенов [10–14]. В отдельных работах тех лет осуществляли агроинфильтрацию [15], что приводило к транзиентной экспрессии Cas9/гидРНК, как, например, при редактировании генома табака Бентхама (Nicotiana benthamiana Domin) [16]. Некоторым преимуществом транзиентной экспрессии является теоретически меньшее число нецелевых сайтов, подвергнувшихся редактированию ввиду относительно кратковременной экспрессии трансгенов. Агроинфильтрация была применена для редактирования генома в условиях транзиентной экспрессии ряда видов цитрусовых, в результате чего было показано, что эффективность процесса повышалась за счет предварительно произведенной обработки листовой поверхности суспензией бактерий Xanthomonas citri subsp. citri [17].
Сообщалось о создании системы агробактериальных векторов, пригодных для эффективного мультиплексного редактирования как однодольных (O. sativa), так и двудольных (A. thaliana) растений с высокой эффективностью [18]. Успешное редактирование различных генных локусов показано для томата (Solanum lycopersicum L.), кукурузы (Zea mays L.), винограда (Vitis vinifera L.), маниока (Manihot esculenta Crantz) и хлопчатника (Gossypium hirsutum L.) после доставки с помощью A. tumefaciens соответствующих Т-ДНК в клетки этих видов растений [19–23]. Для трансгенного тополя Populus tremula × alba, полученного с помощью A. tumefaciens, несущей конструкции для геномного редактирования двух генов биосинтеза лигнина, была достигнута 100% эффективность создания намеченных мутаций [24]. Известно, что использование парных никаз Cas9 повышает точность редактирования, и чтобы убедиться в справедливости такого мнения, на растительных объектах была проведена трансформация растений риса (O. sativa) соответствующими конструкциями, находящимися в агробактерии A. tumefaciens, что полностью оправдало ожидания авторов [25].
Геномному нокаутному CRISPR/Cas-редактированию подвергли суспензионную культуру клеток BY2 табака N. tabacum, продуцирующую репортерный флуоресцентный белок, с таким расчетом, что три подобранных гидРНК должны были направить Cas-нуклеазу для разрушения кодирующего этот репортерный белок гена [26]. В 7 из 20 трансформантов, полученных с использованием A. tumefaciens, это удалось. Другими авторами также с помощью A. tumefaciens было произведено нокаутное редактирование сразу 14 локусов в генах, ответственных за синтез ксилоз и фукоз, что позволило получить трансгенную линию суспензионных клеток табака BY2, более пригодную для наработки в ней биофармацевтических продуктов в виде рекомбинантных (животных) белков [27].
Помимо наиболее часто используемой для геномного редактирования нуклеазы Cas9, существует немало и других аналогичных ферментов [28], среди которых нуклеаза Cas12, известная также как Cpf1. Она характеризуется тем, что для ее “нацеливания” на место редактирования достаточно значительно более короткой молекулы гидРНК. Сообщается также об использовании комплекса нуклеазы Cpf1 и гидРНК в виде рибозимного блока в составе бинарного вектора, доставляемого в редактируемые клетки риса (O. sativa) с помощью A. tumefaciens [29]. Ранее было показано успешное мультиплексное редактирование ряда генов у риса с использованием двух разных нуклеаз Cpf1 и соответствующих гидРНК, объединенных в разных бинарных векторах на основе pCambia, доставка которых также осуществлялась при помощи A. tumefaciens [30].
Отдельного внимания заслуживают работы, в которых проводилось не нокаутное редактирование, а однонуклеотидные замены при помощи каталитически неактивной нуклеазы Cas9, сшитой с соответствующими дезаминазами. Так, с помощью агробактериальной трансформации были получены трансгенные растения A. thaliana и рапса (Brassica napus L.) с точечной заменой A на G в том числе в гене фитоендесатуразы, что привело к формированию альбиносных фенотипов [31]. Сходное редактирование путем дезаминирования и превращения C в T в кодоне аминокислотного остатка Pro190 гена ацетолактатсинтазы арбуза (Citrullus lanatus (Thunb. Matsum. & Nakai)) привело к тому, что созданная форма приобрела устойчивость к гербициду трибенурону [32]. Причем доставка нужных CRISPR/Cas-компонентов производилась с помощью A. tumefaciens с образованием трансгенных арбузов, но в поколении T1 редактирующую конструкцию у некоторых растений удалось исключить путем отбора, что позволило авторам говорить о создании ими нетрансгенной формы арбуза, устойчивой к гербициду. Для первичного визуального отбора редактированных форм предлагается воспользоваться маркерным белком (например, в случае вектора pKAMA-ITACHI (pKIR) это красный флуоресцентный белок RFP), поскольку практически все (или подавляющее большинство) несущие данный маркерный признак растения будут характеризоваться намеченными мутациями [33].
ДОСТАВКА CRISPR/Cas-КОМПОНЕНТОВ В КЛЕТКИ РАСТЕНИЙ С ИСПОЛЬЗОВАНИЕМ РАСТИТЕЛЬНЫХ ВИРУСОВ
Использование растительных геминивирусов, подвергнутых генно-инженерным модификациям, позволяет достигнуть более высокой эффективности трансформации без стабильной встройки в геном (табл. 1) [34]. Геминивирусы, инфицирующие широкий круг как однодольных, так и двудольных растений, формируют большое семейство растительных вирусов Geminiviridae и представляют собой одноцепочечные ДНК-вирусы, которые после попадания в ядро растительной клетки становятся двухцепочечными и интенсивно реплицируются по механизму катящегося кольца с использованием ферментов хозяина. Однако ввиду малых размеров их геномов, колеблющихся в диапазоне от 2.5 до 3 тысяч пар нуклеотидов (т.п.н.), они даже после оставления в их геноме только участков, обеспечивающих репликацию, часто не могут нести в себе протяженные чужеродные фрагменты ДНК в виде, например, генов Cas-нуклеаз. Поэтому было предложено использовать геминивирусы прежде всего для наработки гидРНК в увеличенных количествах. Так, по аналогии с аббревиатурой VIGS (Virus Induces Gene Silencing) – давно применяющегося метода для намеренного замолкания генов [35, 36], один из вариантов геномного редактирования с использованием растительных вирусов было предложено называть VIGE – VIrus based gRNA delivery system for CRISPR/Cas9 mediated plant Genome Editing [37]. Его суть заключается в увеличении копийности гидРНК в ядре клетки путем доставки измененного геминивируса с помощью агроинфильтрации трансгенных растений, экспрессирующих ген нуклеазы Cas9. В этой работе использовался вирус курчавости листьев капусты CaLCuV (Cabbage Leaf Curl Virus), относящийся к роду Begomovirus, модифицированный геном которого содержал ген гидРНК и был встроен в Т-ДНК в составе бинарного агробактериального вектора, которым путем агроинфильтрации трансформировался табак N. benthamiana. Еще в ряде работ такой подход с использованием разных геминивирусов (BeYDV – вируса желтой карликовости фасоли, WDV – вируса карликовости пшеницы и др.) применялся для редактирования геномов различных растений – риса (O. sativa) [30], томата (S. lycopersicum) [38], картофеля (Solanum tuberosum L.) [39] и пшеницы (Triticum aestivum L.) [40].
Таблица 1.
Некоторые сведения об эффективности геномного редактирования при стабильной встройке и транзиентной экспрессии CRISPR/Cas-компонентов
| Вид растения | Способ доставки | Встраивание в геном | Эффективность редактирования |
|---|---|---|---|
| Sorghum bicolor | Agrobacterium tumefaciens | Стабильная встройка | 27.8% [10] |
| Arabidopsis thaliana | 70–71% [13, 14] | ||
| Oryza sativa | 83.3–91.6% [11] | ||
| Solanum lycopersicum | 83.6% [19] | ||
| Zea mays | 70% [20] | ||
| Vitis vinifera | 61.5% [21] | ||
| Manihot esculenta | 90–100% [22] | ||
| Populus tremula × alba | 100% [24] | ||
| Brassica napus | 52–66% [31] | ||
| Citrullus lanatus | 23% [32] | ||
| Solanum lycopersicum | 75% [38] | ||
| Citrus sp. | Транзиентная экспрессия | 20% [17] | |
| Nicotiana benthamiana | Вирусная транзиентная экспрессия | 85% [37] на 43–61% выше, чем в контроле [41] |
|
| Oryza sativa | 44–75% [23] | ||
| Arabidopsis thaliana | 57–63% [43] | ||
| Glycine max | Agrobacterium rhizogenes | Стабильная встройка | До 95% [48] 95% [49] 14.7–20.2% [51] |
| Medicago truncatula | Нет данных [50] | ||
| Lotus japonicus | 35% [53] | ||
| Salvia miltiorrhiza | 42.3% [54] 50% [55] |
||
| Taraxacum kok-saghyz | 88.9% [56] | ||
| Brassica carinata | Во всех трансгенных корнях [57] | ||
| Oryza sativa | Биобаллистика | Стабильная встройка | 14.5–20% [59] |
| Triticum aestivum | 23–38% [60] | ||
| Вирусная транзиентная экспрессия | В 12 раз выше, чем в безвирусном варианте [40] | ||
| Транзиентная экспрессия | 1–9.5% [63] | ||
| Биобаллистика РНП | 21.8–33.4% [66] | ||
| Zea mays | 47% [64] | ||
| Oriza sativa | Трансформация протопластов | Стабильная встройка | 3–8% [69] |
| Arabidopsis thaliana | Транзиентная экспрессия | 26% [12] | |
| Silene latifolia | 20% [74] | ||
| Oriza sativa | Трансформация протопластов РНП |
До 27.3% [70] | |
| Vitis vinifera | 0.1% [71] | ||
| Malus domestica | До 6.9% [71] | ||
| Petunia hybrida | 5.3–17.8% [72] | ||
| Solanum tuberosum | 25% [76] | ||
| Lactuca sativa | До 46% [77] | ||
| Glycine max | До 11.7% [78] |
Другой группой вирусов, использующихся для повышения эффективности редактирования геномов растений, служат одноцепочечные РНК-вирусы семейства Virgaviridae, инфицирующие около 400 видов растений из 50 семейств. Наиболее часто используемым является вирус погремковости табака TRV – Tobacco Rattle Virus из рода Tobravirus. Данный вирус имеет двухсегментный геном TRV1 и TRV2, причем субгеном TRV2 может быть модифицирован и способен нести чужеродные фрагменты, необходимые для редактирования геномов с помощью CRISPR/Cas-технологии. Хотя емкость TRV для чужеродной ДНК и выше чем у геминивирусов (около 2–3 т.п.н.), она все равно недостаточна для размещения в этом вирусе гена Cas-нуклеазы, и поэтому экспериментаторы вынуждены ограничиваться доставкой таким способом только гидРНК. Так, было сообщено об успешном редактировании гена фитоендесатуразы табака Бентхама путем совместной агроинфильтрации в это растение, уже экспрессирующее ген нуклеазы Cas9, двух штаммов агробактерий A. tumefaciens, содержащих соответственно нативный TRV1 субгеном и измененный субгеном TRV2, несущий участок, кодирующий гидРНК под сильным вирусным промотором PEBV (Pea Early-Browning Virus) [41, 42]. Упомянутый промотор вируса раннего побурения гороха PEBV вместе с вирусом TRV недавно был использован для аналогичной доставки гидРНК как в растения A. thaliana, так и того же вида табака N. benthamiana [43]. Векторы, созданные на основе геминивирусов, могут быть использованы не только для агробактериальной трансформации [34, 38], но и для биобаллистики. К примеру, с использованием вектора pWDV2 на основе вируса карликовости пшеницы, несущего как ген Cas9, так и гидРНК, была достигнута увеличенная в 12 раз эффективность геномного редактирования по сравнению с доставкой необходимых CRISPR/Cas-компонентов с помощью обычных векторов (табл. 1) [40]. Также следует отметить работу, в которой с помощью агробактерий, несущих вектор на основе геминивируса, содержащий одновременно последовательности гидРНК, донорной ДНК и нуклеазы Cas9, был осуществлен нокин целевого гена в растения томата (S. lycopersicum) [38], причем эффективность редактирования была на порядок выше, чем при использовании обычных бинарных векторов. Одни авторы отмечают, что с повышением эффективности геномного редактирования увеличения частоты off-target активности не происходит [38], тогда как другие не акцентируют на этом своего внимания [40].
ДОСТАВКА CRISPR/Cas-КОМПОНЕНТОВ В РАСТИТЕЛЬНЫЕ КЛЕТКИ С ПОМОЩЬЮ Agrobacterium rhizogenes
Для доставки CRISPR/Cas-компонентов в клетки растений также было предложено использовать другую агробактерию Agrobacterium rhizogenes [44], вызывающую, как известно, рост волосовидных корней (термин “волосовидные корни” для таких корней был предложен еще в начале прошлого века известным специалистом в области бактериального патогенеза растений И.Л. Сербиновым [45]). Было сообщено об успешном редактировании репортерного гена (которым служили разные варианты зеленого флуоресцентного белка) у растений томата (S. lycopersicum) с использованием бинарного вектора, несущего необходимые компоненты для CRISPR/Cas9-редактирования, причем первичный отбор форм для анализа производился на основе появляющихся волосовидных корней [44]. Справедливости ради следует отметить, что ранее этот вид агробактерий использовался для доставки компонентов для геномного редактирования с помощью нуклеазы “цинковых пальцев” [46]. Преимуществом использования A. rhizogenes по сравнению c A. tumefaciens служит как раз появление волосовидных корней, свидетельствующих о произошедшем редактировании генома опытного растения. Однако надо признать, что данный маркерный признак носит косвенный характер, поскольку внедрение rol-генов, вызывающих неопластический рост корней и расположенных в диких штаммах A. rhizogenes на отдельной Ri-плазмиде, и внедрение так называемой T-ДНК, несущей необходимые для редактирования ген Cas-нуклеазы и кассету для гидРНК, находящихся в специальном бинарном векторе, являются разными трансформационными событиями. Таким образом, появление волосовидных корней свидетельствует в первую очередь о том, что в растительную клетку, давшую начало их росту, попала чужеродная ДНК, что заметно сужает поиск редактированных форм среди массы потенциальных трансформантов. Позже на томате было также показано, что феномен появления волосовидных корней служит удобной системой для проверки функциональности сконструированных бинарных векторов для ведения геномного редактирования [47].
После сообщения об успешном использовании для доставки CRISPR/Cas-компонентов и предварительного анализа произведенного геномного редактирования с помощью агробактерии A. rhizogenes, довольно быстро стали появляться аналогичные публикации. В нескольких статьях сообщалось о применении этой агробактерии для доставки бинарных векторов с CRISPR/Cas-компонентами для нокаутного редактирования генома сои культурной (Glycine max (L.) Merr.) [48–51]. Так, было показано, что из 88 проанализированных образцов волосовидных корней в 95% обнаружились ожидаемые мутации, биаллельный характер которых выявлен для 8 из 9 используемых бинарных векторов [49]. Помимо сои в одной из этих работ доставка бинарных векторов, несущих компоненты для CRISPR/Cas-редактирования, осуществлялась также в другое бобовое растение – люцерну усеченную (Medicago truncatula Gaertn.) [50]. Произведенный авторами нокаут гена фитоендесатуразы привел к побелению листьев, что служило уже прямым подтверждением произошедшего изменения ДНК. Другими авторами было продемонстрировано довольно успешное редактирование генома сои с помощью как CRISPR/Cas-, так и TALEN-систем, доставляемых в клетки с использованием соответствующих бинарных векторов на основе pCambia 1301 с помощью A. rhizogenes, благодаря чему отбор трансформантов велся по фенотипу в виде волосовидных корней [52]. Еще одним растением семейства бобовых, геном которого был редактирован в нокаутном режиме путем доставки CRISPR/Cas-компонентов с помощью A. rhizogenes, явился лядвенец японский (Lotus japonicus L.) [53].
В двух статьях разных групп авторов сообщается о произведенном редактировании генома медицинского растения шалфея краснокорневищного (Salvia miltiorrhiza Bunge), для чего использовались разные штаммы A. rhizogenes с бинарными векторами, несущими CRISPR/Cas-компоненты [54, 55]. Причем в последней работе говорится, что 50% растений, регенерированных из трансгенных волосовидных корней, несли нужные нокаутные мутации. Ранее с успехом осуществлено редактирование генома каучуконосного вида одуванчика кок-сагыза (Taraxacum kok-saghyz Rodin) путем доставки с помощью A. rhizogenes CRISPR/Cas-компонентов в составе бинарного вектора для нокаутирования одного из генов биосинтеза инулина [56]. В данной работе было показано, что из 11 проанализированных волосовидных корней кок-сагыза, появившихся через три недели после трансформации, в 10 произошло намеченное редактирование и через 10 недель были получены полноценные растения кок-сагыза с нокаутированным геном 1-FFT.
Одной из целей редактирования генома эфиопской горчицы (Brassica carinata A. Braun) было нарушение гена, отвечающего за рост корневых волосков, что удалось осуществить с помощью бинарного вектора, несущего, помимо необходимых для этого CRISPR/Cas-компонентов, еще и ген репортерного зеленого флуоресцентного белка, что позволило дополнительно контролировать трансгенную природу образующихся под действием A. rhizogenes волосовидных корней [57]. Недавно сообщено о нокаутировании у картофеля клубненосного (S. tuberosum) гена, участвующего в биосинтезе стероидных гликоалкалоидов, придающих этому растению горечь и являющихся токсичными для многих организмов, что было осуществлено с помощью A. rhizogenes, несущей соответствующую бинарную конструкцию, а анализ произведенного редактирования проводился на появившихся волосовидных корнях [58].
Насколько нам известно, это практически все работы по редактированию геномов растений, в которых для доставки созданных генно-инженерных конструкций на основе различных бинарных векторов использовалась агробактерия A. rhizogenes. При этом происходило встраивание Т-ДНК из бинарных плазмид в геном редактируемых растений, и имела место стабильная экспрессия данных трансгенов, в результате чего в отдельных случаях происходили нецелевые мутации [51]. Несмотря на удобство первичного скрининга по появлению фенотипа волосовидных корней необходимо учитывать, что регенерация из них полноценных растений для разных видов может быть довольно проблематичной.
ДОСТАВКА CRISPR/Cas-КОМПОНЕНТОВ В РАСТИТЕЛЬНЫЕ КЛЕТКИ С ПОМОЩЬЮ БИОБАЛЛИСТИКИ
Довольно распространенным способом получения трансгенных растений, более часто используемым для трансформации представителей класса однодольных, является биобаллистическая доставка в растительные экспланты генно-инженерных конструкций, сорбированных на золотых частицах микронного и субмикронного размеров. Неудивительно, что биобаллистическая трансформация стала применяться и для редактирования геномов разных видов растений. Так, было сообщено о бомбардировке каллуса риса (O. sativa) плазмидами, содержащими ген нуклеазы Cas9 и гидРНК, нацеленной на редактирование гена фитоендесатуразы, о произошедшем нокауте которого можно было судить, в том числе по белесому фенотипу регенерированных растений риса [59]. Эта же группа авторов продолжила эксперименты по редактированию генома однодольных и в качестве объекта редактирования использовала гены, расположенные во всех трех субгеномах мягкой гексаплоидной пшеницы (T. aestivum), ответственные за аттрактацию этиологического агента, вызывающего мучнистую росу, и доставка CRISPR/Cas-компонентов в растительные клетки в их работе также осуществлялась с помощью биобаллистики [60, 61]. На примере риса показано, что с помощью биобаллистической бомбардировки каллусных тканей золотыми частицами, несущими соответствующие гены нуклеазы Cas9 и гидРНК, экспрессирующимися транзиентно, можно получить редактированные растения с мутациями в гене ацетолактатсинтазы, приводящими к появлению устойчивости к некоторым гербицидам [62]. В одной из работ способ редактирования растительных геномов с помощью транзиентной экспрессии CRISPR/Cas9-комплекса, доставляемого с помощью бомбардировки тканей золотыми частицами, получил название TECCDNA (Transiently Expressing CRISPR/Cas9 DNA) [63].
С целью создания растений с редактированным геномом, в который бы не происходило интеграции чужеродной ДНК, был разработан подход с использованием готового рибонуклеопротеидного (РНП) комплекса из соответствующей Cas-нуклеазы и гидРНК, сорбированных на золотых частицах, доставка которых осуществлялась с помощью биобаллистики. При этом необходимо отметить, что происхождение такого комплекса, формирующегося in vitro за короткое время при комнатной температуре, может быть различным, имея ввиду, что гидРНК может быть заранее наработана транскрипцией in vitro или синтезирована химическим путем в виде рибоолигонуклеотида, а соответствующая Cas-нуклеаза может быть наработана в системе in vivo и затем очищена должным образом, либо приобретена как биопрепарат у ряда фирм-производителей такой продукции.
Впервые биобаллистическая процедура доставки рибонуклеопротеидного комплекса (РНП) Cas9/гидРНК для растительных объектов была успешно применена при редактировании генома кукурузы (Z. mays) [64] в “протеолистической” (proteolistics) модификации данного метода [65]. Сообщалось об использовании для доставки такого редактирующего комплекса в растения пшеницы (в незрелые зародыши) бомбардировки тканей золотыми частицами с нанесенным на них рибонуклеопротеином из нуклеазы Cas9/гидРНК [66]. В своей следующей статье эти авторы привели подробный протокол действий по геномному редактированию мягкой пшеницы (T. aestivum) с помощью биобаллистической рибонуклеопротеидной трансформации [67]. Нокаутный и нокин варианты редактирования генома риса (O. sativa) были произведены с использованием Cpf1-нуклеазы, причем гены двух задействованных ферментов FnCpf1 и LbCpf1, кодирующие гидРНК участки, а также донорные фрагменты ДНК (для нокин экспериментов) находились в составе разных бинарных векторов [68]. Следует отметить, что использование для биобаллистической трансформации РНП существенно увеличивало эффективность геномного редактирования (табл. 1).
ДОСТАВКА CRISPR/Cas-КОМПОНЕНТОВ В РАСТИТЕЛЬНЫЕ КЛЕТКИ ТРАНСФОРМАЦИЕЙ ПРОТОПЛАСТОВ
Способ создания трансгенных растений через трансформацию протопластов с некоторыми вариациями был применен и для целей геномного редактирования различных растений. Так, в одной из ранних работ редактирование генома риса (O. sativa) проводилось путем полиэтиленгликольной трансформации протопластов этого вида растений специально созданными конструкциями, содержащими необходимые CRISPR/Cas-компоненты [69]. Наряду с использованием агробактериальной трансформации для редактирования геномов риса и A. thaliana через стабильно экспрессируемые нуклеазу Cas9 и гидРНК (о чем уже говорилось выше), описывалась и трансформация протопластов A. thaliana с целью транзиентной экспрессии аналогичных компонентов для редактирования генома этого растения [12]. В работе других авторов помимо доставки в растения риса CRISPR/Cas-компонентов с помощью A. tumefaciens также проводилась и трансформация протопластов, причем рибонуклеопротеидными частицами, приготовленными заранее in vitro [70]. Также с помощью рибонуклеопротеидных частиц, состоящих из гидРНК и Cas9-нуклеазы, были произведены направленные изменения геномов винограда (V. vinifera), яблони (Malus domestica Borkh.), петунии (Petunia Juss.), доставка редактирующих комплексов в клетки которых осуществлялась путем трансформации их протопластов [71, 72].
Проведено успешное CRISPR/Cas-редактирование двух генов пшеницы (T. aestivum) с помощью транзиентной экспрессии соответствующих конструкций, доставленных к месту редактирования путем трансформации протопластов [73]. Сообщается о транзиентной трансформации протопластов двудомного растения смолевки белой (Silene latifolia Poir.) генно-инженерными конструкциями, содержащими как CRISPR/Cas-систему редактирования, так и TALEN-систему [74].
Упомянутые выше работы ставили своей целью проведение по сути модельных экспериментов и ограничивались констатацией факта произошедшего редактирования. Поэтому нельзя не обратить отдельного внимания на исследования, в ходе которых созданы полноценные редактированные растения. Так, удалось провести мультиаллельное редактирование гена крахмалсинтазы путем транзиентной экспрессии CRISPR/Cas-компонентов, доставленных в протопласты картофеля (S. tuberosum), в виде последующей регенерации полноценных растений, несущих ожидаемые изменения генома [75]. В своей следующей работе эти авторы для трансформации протопластов картофеля с целью аналогичного CRISPR/Cas-редактирования использовали уже рибонуклеопротеидный комплекс [76]. Ранее подобные рибонуклеопротеидные комплексы с нуклеазой Cas9 доставлялись в протопласты A. thaliana, N. tabacum, салата (Lactuca sativa L.) и риса (O. sativa) путем полиэтиленгликольной трансформации также с последующей регенерацией растений [77]. В дальнейшем эти авторы для редактирования геномов G. max и N. tabacum использовали уже рибонуклеопротеидный комплекс, включающий нуклеазу Cpf1 [78].
Завершая рассмотрение доставки CRISPR/Cas-компонентов в протопласты растений, следует заметить, что помимо этапа получения протопластов под действием ряда ферментов, главным недостатком этого подхода служат трудности с регенерацией полноценных растений, которые для целого ряда видов до сих пор не преодолены. В то же время главным преимуществом данного направления исследований является возможность использования разрабатываемых технологий для получения полностью редактированных растений без “следов” чужеродной ДНК, так как предполагается получение регенерантов из единичных генетически измененных протопластов.
ДОСТАВКА CRISPR/Cas-КОМПОНЕНТОВ И ЭФФЕКТИВНОСТЬ ГЕНОМНОГО РЕДАКТИРОВАНИЯ
Эффективность геномного CRISPR/Cas-редактирования зависит от очень многих факторов, начиная с определения типа геномного редактирования (нокаутное, нокин, дезаминирование) и выбора мишени, дизайна гидРНК, доставки необходимых компонентов к месту редактирования (табл. 1). Наконец, она зависит от оказавшихся в клетке количеств этих компонентов, которые определяются как способом доставки, так и уровнем экспрессии доставленных генов, находящихся под контролем тех или иных промоторов, включая тканеспецифичные. Но поскольку темой статьи является доставка CRISPR/Cas-компонентов, то остальные моменты, среди которых создание разнообразных плазмидных конструкций (тем более что большое их число в готовом виде, включая варианты с оптимизированным кодонным составом, доступно через репозитарий Addgene) и анализ силы промоторов здесь будут оставлены без внимания.
Наиболее простым и проверенным способом доставки чужеродной ДНК внутрь растительных клеток является агробактериальная трансформация, осуществляемая как с помощью A. tumefaciens, так и A. rhizogenes. При этом может произойти встраивание чужеродной ДНК в ядерную ДНК растения-реципиента и в зависимости от используемых промоторов будет иметь место стабильная или индуцибельная экспрессия. Весьма часто в экспериментах по трансформации используются “сильные” промоторы, как, например, промотор 35S РНК вируса мозаики цветной капусты. Однако, если при проведении геномного редактирования созданное таким образом трансгенное растение будет конститутивно экспрессировать эти чужеродные для него элементы в виде Cas-нуклеазы и гидРНК в больших количествах, то это приведет, с одной стороны, к более полному редактированию целевых сайтов (табл. 1), достигая полной биаллельности мутаций, а с другой – повысится вероятность редактирования нецелевых сайтов. Так, на табаке N. tabacum была проведена специальная работа, в ходе которой производилась полиэтиленгликольная трансформация протопластов, а также создавались полноценные трансгенные растения [79]. Оказалось, что при редактировании двух генов табака эффективность возникновения мутаций составила от 16.2 до 20.3% при транзиентной экспрессии, тогда как в полученном трансгенном растении эффективность редакции одного гена составила 81.8%, а второго – 87.5%.
Учитывая нежелательность нецелевых мутаций, вполне логичным выглядит вариант со встраиванием в геном, например, только участков, кодирующих гидРНК, тогда как транскрипция гена Cas-нуклеазы происходит транзиентно с доставленной в клетку путем агроинфильтрации другой плазмиды, несущей данный ген. В этом случае требуется использование двух разных плазмидных конструкций, а для нокин вариантов – еще и донорной ДНК, которая может находиться на третьей плазмиде. Впрочем, при встраивании в геном любого из CRISPR/Cas-компонентов образуется трансгенное растение со всеми вытекающими последствиями. Хотя затем удаление временно требующихся для редактирования элементов из “отредактированного” растения может быть произведено, например, используя возвратные скрещивания. Но такие методы удаления трансгенов могут оказаться малоэффективными в случае двудомных и самофертильных видов растений или даже неприемлемыми для сельскохозяйственных культур, размножаемых вегетативно, поскольку размножение семенным путем приведет у них к потере ценных сортовых признаков. Также мало подходит такой способ для растений с продолжительным ювенильным периодом, однако некоторое ускорение процесса может обеспечить прививка редактированного растения на взрослый подвой. В генной инженерии растений для удаления маркерных генов применяют также специально разработанные методы. К примеру, для этого может быть использована рекомбинационная система Cre/loxP из бактериофага P1. При этом необходимый для последующего удаления маркерный ген фланкируется участками loxP, а Cre-рекомбиназа при транзиентной экспрессии в векторе, основанном, к примеру, на вирусе картофеля X, удаляет этот фрагмент ДНК [80].
Альтернативой создания трансгенных растений при проведении геномного редактирования является технология, в которой при нокаутировании или дезаминировании целевых мишеней не требуется использования молекул ДНК, а производится доставка непосредственно комплекса из нуклеазы и гидРНК (рис. 1). При этом их количества в месте редактирования могут быть довольно большими. Так, гидРНК может быть получена с помощью транскрипции в системе in vitro под контролем промотора Т7 РНК полимеразы с использованием специальной конструкции или синтезирована химическим путем. Нуклеазы Cas9 или Cas12 могут быть наработаны в E. coli с помощью экспрессионных векторов, несущих гены этих белков под контролем подходящего промотора, или приобретены из коммерческих источников, поскольку уже ряд фирм включили в свой ассортимент товаров данные позиции.
Рис. 1.
Упрощенная схема способов доставки CRISPR/Cas-компонентов для редактирования геномов растений с внедрением и без внедрения чужеродной ДНК. “Н” или “н” – различные Cas-нуклеазы; “Р” или “р” – гидовые РНК; “п” – РНП, представляющий собой рибонуклеопротеидный комплекс из молекул Cas-нуклеаз и гидовых РНК; “Д” – донорная ДНК. Заглавные буквы символизируют интеграцию соответствующих компонентов в геном редактируемого растения, тогда как их строчные варианты означают транзиентную экспрессию, не приводящую к встраиванию в геном чужеродной ДНК. Прямоугольники “Cas”, “РНК”, “РНП”, “ДНК” символизируют компоненты CRISPR/Cas-редактирования, под которыми понимаются соответствующие генно-инженерные конструкции (“Cas”, “РНК”); “РНП” – представляет собой рибонуклеопротеидный комплекс, формируемый in vitro; под “ДНК” понимается любая донорная ДНК как в виде олигонуклеотидов, так и протяженных молекул в одно- или двухцепочечных формах, в том числе в составе векторных конструкций, которые используются в нокин экспериментах. Сплошными линиями обозначены способы доставки CRISPR/Cas-компонентов, приводящие к встраиванию чужеродной ДНК, включая донорную ДНК, в геном редактируемого растения, а пунктирными линиями показаны способы, при которых имеет место транзиентная экспрессия отдельных CRISPR/Cas-компонентов или функционирует доставляемый РНП.
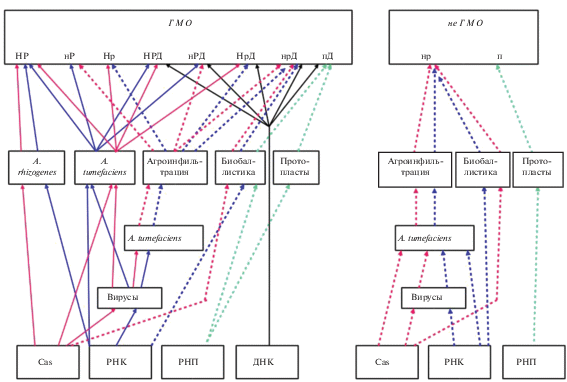
В целом, анализ литературы показывает, что стабильная встройка и экспрессия CRISPR/Cas-компонентов способствует высокому уровню эффективности редактирования в среднем от 50 до 100% (табл. 1). В то же время эффективность геномного редактирования при транзиентной экспрессии CRISPR/Cas-компонентов заметно ниже (от 1 до 47%). Эффективность редактирования существенно повышалась при использовании для транзиентной экспрессии генно-инженерных конструкций с вирусными регуляторами репликации. При биобаллистической трансформации эффективность редактирования была относительно низкой, особенно при транзиентной экспрессии, однако существенно повышалась при использовании для бомбардировки РНП комплексов (табл. 1).
Как уже говорилось выше, в настоящее время с использованием современных технологий трансформации растений при геномном CRISPR/Cas-редактировании в его разных вариантах могут получаться как нетрансгенные растения, обозначаемые на рисунке “нр” и “п”, так и трансгенные растения, обозначаемые как “НР”, “НРД”, “Нр”, “НрД”, “нР”, “нРД”, “нрД”, “пД”. Все эти варианты, безусловно, зависят от способа доставки CRISPR/Cas-компонентов, но это уже вторично, поскольку, например, нрД- и пД-растения (равно как и другие) могут быть созданы с помощью разных способов (рис. 1).
НОВЫЕ МЕТОДЫ СЕЛЕКЦИИ РАСТЕНИЙ И ПРОБЛЕМА ГМО
В последние годы в англоязычной литературе появились устойчивые словосочетания New Breeding Techniques (NBT) или New Plant Breeding Techniques (NPBT), под которыми подразумевается применение для современной селекции растений целой группы новых методов. Однако в мире до сих пор нет единой точки зрения – приводит ли использование этих новых методов селекции к созданию генетически-модифицированных растений в привычном понимании или таковыми их считать не следует. Так, в 2007 г. в Европе была создана специальная рабочая группа по новым технологиям “New Techniques Working Group”, в течение нескольких лет рассматривавшая восемь различных подходов к внесению изменений в генетический материал высших растений: Oligonucleotide-directed mutagenesis; Site-directed nucleases (SDN-1, SDN-2, SDN-3); Cisgenesis/intragenesis; Grafting on a GMO rootstok; Agro-infiltration; RNA-dependent DNA methylation; Reverse breeding; Synthetic genomics. В связи с темой данной статьи интерес представляет лишь использование нуклеаз, вызывающих двуцепочечные разрывы в редактируемой ДНК, причем, когда перед теми экспертами ставилась задача по анализу последствий создания растений с использованием новых методов селекции, то имелись ввиду мегануклеазы и нуклеазы “цинковых пальцев”, поскольку другие нуклеазные технологии для целей редактирования тогда еще не использовались. Позже в рассмотрение добавились TALE нуклеазы и Cas-нуклеазы. Причем варианты SDN-1, SDN-2 и SDN-3 распространились сейчас и на них, поскольку под ними имеются ввиду соответственно: 1) репарирование негомологичных концов без добавления репарирующей ДНК; 2) репарирование путем гомологичной рекомбинации с добавлением относительно короткой ДНК, несущей небольшие изменения по сравнению с заменяемой последовательностью; 3) репарирование путем гомологичной рекомбинации с внедрением довольно протяженного чужеродного участка ДНК [81]. По результатам работы упоминавшейся рабочей группы был подготовлен специальный доклад и статья, даже заголовок которой (“Transgenic or not? No simple answer!”) говорил о том, что вопрос, являются ли трансгенными растения, созданные с помощью новых селекционных технологий, остается открытым [82].
Существуют различные подходы к произведенной модификации. Либо рассматривается конечный продукт в виде измененного любым способом растения (Аргентина, Канада, США), либо способ, которым это изменение достигнуто (Европейский Союз, Российская Федерация). В этой связи надо заметить, что основные термины и определения, какие организмы считать генетически-модифицированными, предложены достаточно давно, еще до появления новых методов селекции и уже не в полной мере соответствуют реалиям сегодняшнего дня, тем более, если принимать во внимание бурно развивающуюся CRISPR/Cas-технологию редактирования геномов, в том числе в “бесследном” варианте. К тому же, помимо искусственно редактированных, существуют “природно-трансгенные растения” c чужеродными генами, попавшими в их геномы за счет естественных природных процессов [83, 84]. Поэтому остро необходим пересмотр требований к создаваемым с помощью генной инженерии, включающей геномное редактирование, растениям, где главенствующим должен быть не способ создания нового организма, а внесенные изменения, которые принципиально могут или не могут произойти в природе.
Уже есть ряд примеров, когда созданные с помощью CRISPR/Cas-технологии новые организмы, в которых не произошло внедрения чужеродной ДНК, за ГМО приняты не были. Так, в 2016 г. в отношении созданных с помощью CRISPR/Cas нокаутной технологии нового сорта шампиньона двуспорового (Agaricus bisporus (J.E.Lange) Imbach), не темнеющего на воздухе после сбора урожая благодаря тому, что в нем оказался нарушенным один из шести имеющихся генов полифенолоксидазы, а также сорта кукурузы (Z. mays) с нарушенным геном waxy, характеризующимся повышенным содержанием амилопектина, было решено, что они не являются ГМО и не требуют проведения соответствующих исследований на предмет их безопасности [85]. В 2017 г. в Канаде и США перечень таких редактированных растений, не подлежащих ГМО-контролю, пополнили рыжик (Camelina sativa (L.) Crantz), продуцирующий масло с увеличенным содержанием омега-3 компонента, соя (G. max), устойчивая к засухе и засолению, что достигнуто путем нарушения генов Drb2a и Drb2b, и однолетний злак щетинник зеленый (Setaria viridis (L.) P. Beauv), характеризующийся задержкой цветения, вызванной нарушением гомолога гена ID1 [86]. Такое же отношение к подобным растениям есть в некоторых других странах мира, включая Швецию, хотя в Европе в целом отношение к ГМО довольно суровое. Однако в январе 2018 г. после ходатайства, поданного Францией, генеральный адвокат Европейского суда M. Bobek определил, что организмы, полученные с помощью новых технологий редактирования генов, не должны попадать под правила Европейского Союза 2001 г. (Directive 2001/18/EC) по выращиванию и маркетингу генетически модифицированных продуктов питания. Тем не менее, 25 июля 2018 г. Европейский суд, заседавший в Люксембурге, постановил, что на все растения с редактированным геномом должны распространяться правила, действующие в отношении обычных трансгенных растений, чем вызвал серьезное разочарование научной общественности не только в Европе, но и во всем мире [87, 88]. При этом отмечается, что данное решение не имеет под собой никакой научной основы. Остается надеяться, что в России такие растения все же не будут подпадать под запрет на возделывание ГМО. При этом нельзя исключать, что со временем может появиться даже новая аббревиатура – “ГРО” (геном-редактированные организмы).
ЗАКЛЮЧЕНИЕ
Недавно в журнале Nature Reviews Molecular and Cell Biology была опубликована краткая заметка [89], в которой отмечается, что одним из “тонких мест” при редактировании геномов растений является доставка CRISPR/Cas-компонентов в растительные клетки и последующая регенерация целого растения. Причем это до некоторой степени взаимосвязанные вещи, учитывая, что далеко не все виды растений легко поддаются регенерации из культуры тканей. Именно поэтому в этой публикации подчеркивается необходимость разработки новых способов доставки CRISPR/Cas-компонентов в апикальную меристему или в пыльцевые зерна, чтобы для получения полноценных растений с редактированными геномами обходиться без этапа культивирования in vitro. В этой заметке также отмечается о том, что достигнутые в настоящее время результаты в области редактирования геномов растений – это лишь совсем малая их часть и следует ожидать более грандиозных успехов, включая, к примеру, придание небобовым растениям способности фиксировать атмосферный азот [89]. При этом справедливо замечается, что для широкомасштабного использования CRISPR/Cas-технологии в сельском хозяйстве предстоит преодоление не научных трудностей, а принятие общественностью этой технологии и правильное государственное регулирование. А способы доставки CRISPR/Cas-компонентов в клетки растений для редактирования их геномов также играют в этом немаловажную роль.
Работа выполнена в рамках государственных заданий № АААА-А16-116020350028-4, № AAAA-A19-119021190011-0 и при поддержке грантов Российского фонда фундаментальных исследований №№ 18-04-00118, 19-016-00117, 19-016-00139.
Авторы заявляют, что у них нет конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Список литературы
Jinek M., Chylinski K., Fonfara I., Hauer M., Doudna J.A., Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity // Science. 2012. V. 337. P. 816–821. https://doi.org/10.1126/science.1225829
Cong L., Ran F.A., Cox D., Lin S., Barretto R., Habib N., Hsu P.D., Wu X., Jiang W., Marraffini L.A., Zhang F. Multiplex genome engineering using CRISPR/Cas systems // Science. 2013. V. 339. P. 819–823. https://doi.org/10.1126/science.1231143
Puchta H. Applying CRISPR/Cas for genome engineering in plants: the best is yet to come // Curr. Opin. Plant Biol. 2017. V. 36. P. 1–8. https://doi.org/10.1016/j.pbi.2016.11.011
Чемерис Д.А., Кирьянова О.Ю., Геращенков Г.А., Кулуев Б.Р., Рожнова Н.А., Матниязов Р.Т., Баймиев Ан.Х., Баймиев Ал.Х., Губайдуллин И.М., Чемерис А.В. Биоинформатические ресурсы для CRISPR/Cas редактирования геномов // Биомика. 2017. Т. 9. С. 203–228.
Ma X., Zhu Q., Chen Y., Liu Y.G. CRISPR/Cas9 Platforms for genome editing in plants: developments and applications // Mol. Plant. 2016. V. 9. P. 961–974. https://doi.org/10.1016/j.molp.2016.04.009
Злобин Н.Е., Терновой В.В., Гребенкина Н.А., Таранов В.В. Сделать сложное проще: современный инструментарий для редактирования генома растений // Вавиловский журнал генетики и селекции. 2017. Т. 21. С. 104–111. https://doi.org/10.18699/VJ17.228
Кулуев Б.Р., Геращенков Г.А., Рожнова Н.А., Баймиев Ан.Х., Вершинина З.Р., Князев А.В., Матниязов Р.Т., Гумерова Г.Р., Михайлова Е.В., Никоноров Ю.М., Чемерис Д.А., Баймиев Ал.Х., Чемерис А.В. CRISPR/Cas редактирование геномов растений // Биомика. 2017. Т. 9. С. 155–182.
Liu X., Xie C., Si H., Yang J. CRISPR/Cas9-mediated genome editing in plants // Methods. 2017. V. 121-122. P. 94–102. https://doi.org/10.1016/j.ymeth
Ran Y., Liang Z., Gao C. Current and future editing reagent delivery systems for plant genome editing // Sci. China Life Sci. 2017. V. 60. P. 490–505. https://doi.org/10.1007/s11427-017-9022-1
Jiang W., Zhou H., Bi H., Fromm M., Yang B., Weeks D.P. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice // Nucleic Acids Res. 2013. V. 41: e188. https://doi.org/10.1093/nar/gkt780
Miao J., Guo D., Zhang J., Huang Q., Qin G., Zhang X., Wan J., Gu H., Qu L.J. Targeted mutagenesis in rice using CRISPR-Cas system // Cell Res. 2013. V. 23. P. 1233–1236. https://doi.org/10.1038/cr.2013.123
Feng Z., Zhang B., Ding W., Liu X., Yang D.L., Wei P., Cao F., Zhu S., Zhang F., Mao Y., Zhu J.K. Efficient genome editing in plants using a CRISPR/Cas system // Cell Res. 2013. V. 23. P. 1229–1232. https://doi.org/10.1038/cr.2013.114
Feng Z., Mao Y., Xu N., Zhang B., Wei P., Yang D.L., Wang Z., Zhang Z., Zheng R., Yang L., Zeng L., Liu X., Zhu J.K. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis // Proc. Natl. Acad. Sci. USA. 2014. V. 111. P. 4632–4637. https://doi.org/10.1073/pnas.1400822111
Fauser F., Schiml S., Puchta H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana // Plant J. 2014. V. 79. P. 348–359. https://doi.org/10.1111/tpj.12554
Schob H., Kunc C., Meins F. Silencing of transgenes introduced into leaves by agroinfiltration: a simple, rapid method for investigating sequence requirements for gene silencing // Mol. Gen. Genetics. 1997. V. 256. P. 581–585. https://doi.org/10.1007/s004380050604
Nekrasov V., Staskawicz B., Weigel D., Jones J.D., Kamoun S. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease // Nat. Biotechnol. 2013. V. 31. P. 691–693. https://doi.org/10.1038/nbt.2655
Jia H., Wang N. Xcc-facilitated agroinfiltration of citrus leaves: a tool for rapid functional analysis of transgenes in citrus leaves // Plant Cell Rep. 2014. V. 33. P. 1993–2001. https://doi.org/10.1007/s00299-014-1673-9
Ma X., Zhang Q., Zhu Q., Liu W., Chen Y., Qiu R., Wang B., Yang Z., Li H., Lin Y., Xie Y., Shen R., Chen S., Wang Z., Chen Y., et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants // Mol. Plant. 2015. V. 8. P. 1274–1284. https://doi.org/10.1016/j.molp.2015.04.007
Pan C., Ye L., Qin L., Liu X., He Y., Wang J., Chen L., Lu G. CRISPR/Cas9-mediated efficient and heritable targeted mutagenesis in tomato plants in the first and later generations // Sci. Rep. 2016. V. 6: e24765. https://doi.org/10.1038/srep24765
Char S.N., Neelakandan A.K., Nahampun H., Frame B., Main M., Spalding M.H., Becraft P.W., Meyers B.C., Walbot V., Wang K., Yang B. An Agrobacterium-delivered CRISPR/Cas9 system for high-frequency targeted mutagenesis in maize // Plant Biotechnol. J. 2017. V. 15. P. 257–268. https://doi.org/10.1111/pbi.12611
Nakajima I., Ban Y., Azuma A., Onoue N., Moriguchi T., Yamamoto T., Toki S., Endo M. CRISPR/Cas9-mediated targeted mutagenesis in grape // PLoS One. 2017. V. 12: e0177966. https://doi.org/10.1371/journal.pone.0177966
Odipio J., Alicai T., Ingelbrecht I., Nusinow D.A., Bart R., Taylor N.J. Efficient CRISPR/Cas9 genome editing of phytoene desaturase in Cassava // Front. Plant Sci. 2017. V. 8: e01780. https://doi.org/10.3389/fpls.2017.01780
Wang M., Mao Y., Lu Y., Tao X., Zhu J.K. Multiplex gene editing in rice using the CRISPR-Cpf1 system // Mol. Plant. 2017. V. 10. P. 1011–1013. https://doi.org/10.1016/j.molp.2017.03.001
Zhou X., Jacobs T.B., Xue L.J., Harding S.A., Tsai C.J. Exploiting SNPs for biallelic CRISPR mutations in the outcrossing woody perennial Populus reveals 4-coumarate:CoA ligase specificity and redundancy // New Phytol. 2015. V. 208. P. 298–301. https://doi.org/10.1111/nph.13470
Mikami M., Toki S., Endo M. Precision targeted mutagenesis via Cas9 paired nickases in rice // Plant Cell Physiol. 2016. V. 57. P. 1058–1068. https://doi.org/10.1093/pcp/pcw049
Mercx S., Tollet J., Magy B., Navarre C., Boutry M. Gene inactivation by CRISPR-Cas9 in Nicotiana tabacum BY-2 suspension cells // Front. Plant Sci. 2016. V. 7. https://doi.org/10.3389/fpls.2016.00040
Hanania U., Ariel T., Tekoah Y., Fux L., Sheva M., Gubbay Y., Weiss M., Oz D., Azulay Y., Turbovski A., Forster Y., Shaaltiel Y. Establishment of a tobacco BY2 cell line devoid of plant-specific xylose and fucose as a platform for the production of biotherapeutic proteins // Plant Biotechnol. J. 2017. V. 15. P. 1120–1129. https://doi.org/10.1111/pbi.12702
Murovec J., Pirc Ž., Yang B. New variants of CRISPR RNA-guided genome editing enzyme // Plant Biotechnol. J. 2017. V. 15. P. 917–926. https://doi.org/10.1111/pbi.12736
Li S., Zhang X., Wang W., Guo X., Wu Z., Du W., Zhao Y., Xia L. Expanding the scope of CRISPR/Cpf1-mediated genome editing in rice // Mol. Plant. 2018. V. 11. P. 995–998. https://doi.org/10.1016/j.molp.2018.03.009
Wang M., Lu Y., Botella J.R., Mao Y., Hua K., Zhu J.K. Gene targeting by homology-directed repair in rice using a geminivirus-based CRISPR/Cas9 system // Mol. Plant. 2017. V. 10. P. 1007–1010. https://doi.org/10.1016/j.molp.2017.03.002
Kang B.C., Yun J.Y., Kim S.T., Shin Y., Ryu J., Choi M., Woo J.W., Kim J.S. Precision genome engineering through adenine base editing in plants // Nat. Plants. 2018. V. 4. P. 427–431. https://doi.org/10.1038/s41477-018-0178-x
Tian S., Jiang L., Cui X., Zhang J., Guo S., Li M., Zhang H., Ren Y., Gong G., Zong M., Liu F., Chen Q., Xu Y. Engineering herbicide-resistant watermelon variety through CRISPR/Cas9-mediated base-editing // Plant Cell Rep. 2018. https://doi.org/10.1007/s00299-018-2299-0
Tsutsui H., Higashiyama T. pKAMA-ITACHI vectors for highly efficient CRISPR/Cas9-mediated gene knockout in Arabidopsis thaliana // Plant Cell Physiol. 2017. V. 58. P. 46–56. https://doi.org/10.1093/pcp/pcw191
Baltes N.J., Gil-Humanes J., Cermak T., Atkins P.A., Voytas D.F. DNA replicons for plant genome engineering // Plant Cell. 2014. V. 26. P. 151–163. https://doi.org/10.1105/tpc.113.119792
Kumagai M.H., Donson J., della-Cioppa G., Harvey D., Hanley K., Grill L.K. Cytoplasmic inhibition of carotenoid biosynthesis with virus-derived RNA // Proc. Natl. Acad. Sci. USA. 1995. V. 92. P. 1679–1683.
Ruiz M.T., Voinnet O., Baulcombe D.C. Initiation and maintenance of virus-induced gene silencing // Plant Cell. 1998. V. 10. P. 937–946. https://doi.org/10.1105/tpc.10.6.937
Yin K., Han T., Liu G., Chen T., Wang Y., Yu A.Y., Liu Y. A geminivirus-based guide RNA delivery system for CRISPR/Cas9 mediated plant genome editing // Sci. Rep. 2015. V. 5. https://doi.org/10.1038/srep14926
Čermák T., Baltes N.J., Čegan R., Zhang Y., Voytas D.F. High-frequency, precise modification of the tomato genome // Genome Biol. 2015. V. 6. https://doi.org/10.1186/s13059-015-0796-9
Butler N.M., Baltes N.J., Voytas D.F., Douches D.S. Geminivirus-mediated genome editing in potato (Solanum tuberosum L.) using sequence-specific nucleases // Front. Plant Sci. 2016. V. 7: e01045. https://doi.org/10.3389/fpls.2016.01045
Gil-Humanes J., Wang Y., Liang Z., Shan Q., Ozuna C.V., Sánchez-León S., Baltes N.J., Starker C., Barro F., Gao C., Voytas D.F. High-efficiency gene targeting in hexaploid wheat using DNA replicons and CRISPR/Cas9 // Plant J. 2017. V. 89. P. 1251–1262. https://doi.org/10.1111/tpj.13446
Ali Z., Abul-Faraj A., Piatek M., Mahfouz M.M. Activity and specificity of TRV-mediated gene editing in plants // Plant Signal. Behav. 2015. V. 10: e1044191. https://doi.org/10.1080/15592324.2015.1044191
Ali Z., Abul-faraj A., Li L., Ghosh N., Piatek M., Mahjoub A., Aouida M., Piatek A., Baltes N.J., Voytas D.F., Dinesh-Kumar S., Mahfouz M.M. Efficient virus-mediated genome editing in plants using the CRISPR/Cas9 system // Mol. Plant. 2015. V. 8. P. 1288–1291. https://doi.org/10.1016/j.molp.2015.02.011
Ali Z., Eid A., Ali S., Mahfouz M.M. Pea early-browning virus-mediated genome editing via the CRISPR/Cas9 system in Nicotiana benthamiana and Arabidopsis // V-irus Res. 2018. V. 244. P. 333–337. https://doi.org/10.1016/j.virusres.2017.10.009
Ron M., Kajala K., Pauluzzi G., Wang D., Reynoso M.A., Zumstein K., Garcha J., Winte S., Masson H., Inagaki S., Federici F., Sinha N., Deal R.B., Bailey-Serres J., Brady S.M. Hairy root transformation using Agrobacterium rhizogenes as a tool for exploring cell type-specific gene expression and function using tomato as a model // Plant Physiol. 2014. V. 166. P. 455–469. https://doi.org/10.1104/pp.114.239392
Сербинов И.Л. Бактериальный рак плодовых деревьев, ягодных кустарников и других садовых, а также сельскохозяйственных растений // Плодоводство. 1912. № 9. С. 787–795.
Curtin S.J., Zhang F., Sander J.D., Haun W.J., Starker C., Baltes N.J., Reyon D., Dahlborg E.J., Goodwin M.J., Coffman A.P., Dobbs D., Joung J.K., Voytas D.F., Stupar R.M. Targeted mutagenesis of duplicated genes in soybean with zinc-finger nucleases // Plant Physiol. 2011. V. 156. P. 466–473. https://doi.org/10.1104/pp.111.172981
Jacobs T.B., Martin G.B. High-throughput CRISPR vector construction and characterization of DNA modifications by generation of tomato hairy roots // J. Vis. Exp. 2016. V. 110. https://doi.org/10.3791/53843
Cai Y., Chen L., Liu X., Sun S., Wu C., Jiang B., Han T., Hou W. CRISPR/Cas9-mediated genome editing in soybean hairy roots // PLoS One. 2015. V. 10: e0136064. https://doi.org/10.1371/journal.pone.0136064
Jacobs T.B., LaFayette P.R., Schmitz R.J., Parrott W.A. Targeted genome modifications in soybean with CRISPR/Cas9 // BMC Biotechnol. 2015. V. 15: 16. https://doi.org/10.1186/s12896-015-0131-2
Michno J.M., Wang X., Liu J., Curtin S.J., Kono T.J., Stupar R.M. CRISPR/Cas mutagenesis of soybean and Medicago truncatula using a new web-tool and a modified Cas9 enzyme // GM Crops Food. 2015. V. 6. P. 243–252. https://doi.org/10.1080/21645698.2015.1106063
Sun X., Hu Z., Chen R., Jiang Q., Song G., Zhang H., Xi Y. Targeted mutagenesis in soybean using the CRISPR-Cas9 system // Sci. Rep. 2015. V. 5. https://doi.org/10.1038/srep10342
Du H., Zeng X., Zhao M., Cui X., Wang Q., Yang H., Cheng H., Yu D. Efficient targeted mutagenesis in soybean by TALENs and CRISPR/Cas9 // J. Biotechnol. 2016. V. 217. P. 90–97. https://doi.org/10.1016/j.jbiotec.2015.11.005
Wang L., Wang L., Tan Q., Fan Q., Zhu H., Hong Z., Zhang Z., Duanmu D. Efficient inactivation of symbiotic nitrogen fixation related genes in Lotus japonicus using CRISPR-Cas9 // Front. Plant Sci. 2016. V. 7: e01333. https://doi.org/10.3389/fpls.2016.01333
Li B., Cui G., Shen G., Zhan Z., Huang L., Chen J., Qi X. Targeted mutagenesis in the medicinal plant Salvia miltiorrhiza // Sci. Rep. 2017. V. 7. https://doi.org/10.1038/srep43320
Zhou Z., Tan H., Li Q., Chen J., Gao S., Wang Y., Chen W., Zhang L. CRISPR/Cas9-mediated efficient targeted mutagenesis of RAS in Salvia miltiorrhiza // Phytochemistry. 2018. V. 148. P. 63–70. https://doi.org/10.1016/j.phytochem.2018.01.015
Iaffalando B., Zhang Y., Cornish K. CRISPR/Cas9 genome editing of rubber producing dandelion Taraxacum kok-saghyz using Agrobacterium rhizogenes without selection // Ind. Crops Prod. 2016. V. 89. P. 356–362. https://doi.org/10.1016/j.indcrop.2016.05.029
Kirchner T.W., Niehaus M., Debener T., Schenk M.K., Herde M. Efficient generation of mutations mediated by CRISPR/Cas9 in the hairy root transformation system of Brassica carinata // PLoS One. 2017. V. 12: e0185429. https://doi.org/10.1371/journal.pone.0185429
Nakayasu M., Akiyama R., Lee H.J., Osakabe K., Osakabe Y., Watanabe B., Sugimoto Y., Umemoto N., Saito K., Muranaka T., Mizutani M. Generation of α-solanine-free hairy roots of potato by CRISPR/Cas9 mediated genome editing of the St16DOX gene // Plant Physiol. Biochem. 2018. V. 131. P. 70–77. https://doi.org/10.1016/j.plaphy.2018.04.026
Shan Q., Wang Y., Li J., Zhang Y., Chen K., Liang Z., Zhang K., Liu J., Xi J.J., Qiu J.L., Gao C. Targeted genome modification of crop plants using a CRISPR-Cas system // Nat. Biotechnol. 2013. V. 31. P. 686–688. https://doi.org/10.1038/nbt.2650
Wang Y., Cheng X., Shan Q., Zhang Y., Liu J., Gao C., Qiu J.L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew // Nat. Biotechnol. 2014. V. 32. P. 947–951. https://doi.org/10.1038/nbt.2969
Zhang Y., Bai Y., Wu G., Zou S., Chen Y., Gao C., Tang D. Simultaneous modification of three homoeologs of TaEDR1 by genome editing enhances powdery mildew resistance in wheat // Plant J. 2017. V. 91. P. 714–724. https://doi.org/10.1111/tpj.13599
Sun Y., Zhang X., Wu C., He Y., Ma Y., Hou H., Guo X., Du W., Zhao Y., Xia L. Engineering herbicide-resistant rice plants through CRISPR/Cas9-mediated homologous recombination of acetolactate synthase // Mol. Plant. 2016. V. 9. P. 628–631. https://doi.org/10.1016/j.molp.2016.01.001
Zhang Y., Liang Z., Zong Y., Wang Y., Liu J., Chen K., Qiu J.L., Gao C. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA // Nat. Commun. 2016. V. 7: e12617. https://doi.org/10.1038/ncomms12617
Svitashev S., Schwartz C., Lenderts B., Young J.K., Mark Cigan A. Genome editing in maize directed by CRISPR-Cas9 ribonucleoprotein complexes // Nat. Commun. 2016. V. 7: e13274. https://doi.org/10.1038/ncomms13274
Martin-Ortigosa S., Wang K. Proteolistics: a biolistic method for intracellular delivery of proteins // Transgenic Res. 2014. V. 23. P. 743–756. https://doi.org/10.1007/s11248-014-9807-y
Liang Z., Chen K., Li T., Zhang Y., Wang Y., Zhao Q., Liu J., Zhang H., Liu C., Ran Y., Gao C. Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexe // Nat. Commun. 2017. V. 8: e14261. https://doi.org/10.1038/ncomms14261
Liang Z., Chen K., Zhang Y., Liu J., Yin K., Qiu J.L., Gao C. Genome editing of bread wheat using biolistic delivery of CRISPR/Cas9 in vitro transcripts or ribonucleoproteins // Nat. Protoc. 2018. V. 13. P. 413–430. https://doi.org/10.1038/nprot.2017.145
Begemann M.B., Gray B.N., January E., Gordon G.C., He Y., Liu H., Wu X., Brutnell T.P., Mockler T.C., Oufattole M. Precise insertion and guided editing of higher plant genomes using Cpf1 CRISPR nucleases // Sci. Rep. 2017. V. 7. P. 11606. https://doi.org/10.1038/s41598-017-11760-6
Xie K., Yang Y. RNA-guided genome editing in plants using a CRISPR-Cas system // Mol. Plant. 2013. V. 6. P. 1975–1983. https://doi.org/10.1093/mp/sst119
Butt H., Eid A., Ali Z., Atia M.A.M., Mokhtar M.M., Hassan N., Lee C.M., Bao G., Mahfouz M.M. Efficient CRISPR/Cas9-mediated genome editing using a chimeric single-guide RNA molecule // Front. Plant Sci. 2017. V. 24: e01441. https://doi.org/10.3389/fpls.2017.01441
Malnoy M., Viola R., Jung M.H., Koo O.J., Kim S., Kim J.S., Velasco R., Nagamangala Kanchiswamy C. DNA-free genetically edited grapevine and apple protoplast using CRISPR/Cas9 ribonucleoproteins // Front. Plant Sci. 2016. V. 7: e01904. https://doi.org/10.3389/fpls.2016.01904
Subburaj S., Chung S.J., Lee C., Ryu S.M., Kim D.H., Kim J.S., Bae S., Lee G.J. Site-directed mutagenesis in Petunia × hybrida protoplast system using direct delivery of purified recombinant Cas9 ribonucleoproteins // Plant Cell Rep. 2016. V. 35. P. 1535–1544. https://doi.org/10.1007/s00299-016-1937-7
Kim D., Alptekin B., Budak H. CRISPR/Cas9 genome editing in wheat // Funct. Integr. Genomics. 2018. V. 18. P. 31–41. https://doi.org/10.1007/s10142-017-0572-x
Hudzieczek V., Cegan R., Cermak T., Bacovska N., Machalkova Z., Dolezal K., Plihalova L., Voytas D., Hobza R., Vyskot B. Agrobacterium rhizogenes-mediated transformation of a dioecious plant model Silene latifolia // New Biotechnol. 2018. V. 48. P. 20–28. https://doi.org/10.1016/j.nbt.2018.04.001
Andersson M., Turesson H., Nicolia A., Fält A.S., Samuelsson M., Hofvander P. Efficient targeted multiallelic mutagenesis in tetraploid potato (Solanum tuberosum) by transient CRISPR-Cas9 expression in protoplasts // Plant Cell Rep. 2017. V. 36. P. 117–128. https://doi.org/10.1007/s00299-016-2062-3
Andersson M., Turesson H., Olsson N., Fält A.S., Ohlsson P., Gonzalez M.N., Samuelsson M., Hofvander P. Genome editing in potato via CRISPR-Cas9 ribonucleoprotein delivery // Physiol. Plant. 2018. V. 164. P. 378–384. https://doi.org/10.1111/ppl.12731
Woo J.W., Kim J., Kwon S.I., Corvalán C., Cho S.W., Kim H., Kim S.G., Kim S.T., Choe S., Kim J.S. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins // Nat. Biotechnol. 2015. V. 33. P. 1162–1164. https://doi.org/10.1038/nbt.3389
Kim H., Kim S.T., Ryu J., Kang B.C., Kim J.S., Kim S.G. CRISPR/Cpf1-mediated DNA-free plant genome editing // Nat. Commun. 2017. V. 8. https://doi.org/10.1038/ncomms14406
Gao J., Wang G., Ma S., Xie X., Wu X., Zhang X., Wu Y., Zhao P., Xia Q. CRISPR/Cas9-mediated targeted mutagenesis in Nicotiana tabacum // Plant Mol. Biol. 2015. V. 87. P. 99–110. https://doi.org/10.1007/s11103-014-0263-0
Kopertekh L., Schiemann J. Marker removal in transgenic plants using Cre recombinase delivered with potato virus X // Methods Mol. Biol. 2017. V. 1642. P. 151–168. https://doi.org/10.1007/978-1-4939-7169-5_10
Sprink T., Eriksson D., Schiemann J., Hartung F. Regulatory hurdles for genome editing: process- vs. product-based approaches in different regulatory contexts // Plant Cell Rep. 2016. V. 35. P. 1493–1506. https://doi.org/10.1007/s00299-016-1990-2
Podevin N., Devos Y., Davies H.V., Nielsen K.M. Transgenic or not? No simple answer! New biotechnology-based plant breeding techniques and the regulatory landscape // EMBO Rep. 2012. V. 13. P. 1057–1061. https://doi.org/10.1038/embor.2012.168
Матвеева Т.В. Природно-трансгенные растения, как модель для изучения отсроченных экологических рисков возделывания ГМО // Экологическая генетика. 2015. Т. 13. № 2. С. 118–126. https://doi.org/10.17816/ecogen132118-126
Матвеева Т.В., Азарахш М. Генно-инженерно-модифицированные организмы, разрешенные к выращиванию и разведению в России // Экологическая генетика. 2016. Т. 14. № 4. С. 32–40. https://doi.org/10.17816/ecogen14432-40
Баймиев Ан.Х., Кулуев Б.Р., Вершинина З.Р. Князев А.В., Чемерис Д.А., Рожнова Н.А., Геращенков Г.А., Михайлова Е.В., Баймиев Ал.Х., Чемерис А.В. CRISPR/Cas редактирование геномов (растений) и общество // Биомика. 2017. Т. 9. С. 183–202.
Waltz E. With a free pass, CRISPR-edited plants reach market in record time // Nat. Biotechnol. 2018. V. 36. P. 6–7. https://doi.org/10.1038/nbt0118-6b
Callaway E. CRISPR plants now subject to tough GM laws in European Union // Nature. 2018. V. 560. P. 16. https://doi.org/10.1038/d41586-018-05814-6
Urnov F., Ronald P.C., Carroll D. A call for science-based review of the European court’s decision on gene-edited crops // Nature Biotechnology. 2018. V. 36. P. 800–802. https://doi.org/10.1038/nbt.4252
Gao C. The future of CRISPR technologies in agriculture // Nat. Rev. Mol. Cell Biol. 2018. V. 19. P. 275–276. https://doi.org/10.1038/nrm.2018.2
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


