Физиология растений, 2020, T. 67, № 3, стр. 227-245
Феномика растений: фундаментальные основы, программно-аппаратные платформы и методы машинного обучения
В. В. Демидчик a, b, *, А. Ю. Шашко a, В. Ю. Бондаренко a, Г. Н. Смоликова c, Д. А. Пржевальская a, М. А. Черныш a, Г. А. Пожванов c, А. В. Барковский d, И. И. Смолич a, А. И. Соколик a, М. Ю b, С. С. Медведев c
a Белорусский государственный университет
Минск, Беларусь
b Департамент сельского хозяйства Университета Фошаня
Фошань, Китай
c Санкт-Петербургский государственный университет
Санкт-Петербург, Россия
d Белорусский государственный университет информатики и радиоэлектроники
Минск, Беларусь
* E-mail: dzemidchyk@bsu.by
Поступила в редакцию 02.04.2019
После доработки 05.10.2019
Принята к публикации 07.10.2019
Аннотация
В последние годы сформировался новый раздел физиологии растений – феномика растений, который фокусируется на выявлении закономерностей организации и изменения растительных феномов, т.е. физических и биохимических характеристик, рассматриваемых как совокупность фенотипов растительного организма. Феномика – постгеномная дисциплина, активно использующая достижения геномной эры и биоинформатики. Она дополняет их стандартизированным и статистически значимым фактологическим материалом о фенотипах с высокой степенью детализации. Техника получения и анализа информации о фенотипах в феномике получила название фенотипирование. Широкое распространение получило высокопроизводительное фенотипирование, обеспечивающее цифровой автоматизированный анализ больших выборок данных. Наметившийся в последние годы прогресс в высокопроизводительном фенотипировании связан с развитием систем регистрации изображений в различных областях спектра, подходов культивирования растительных объектов в стандартизированных условиях, сенсорных технологий, робототехники, а также методов обработки и анализа данных, таких как компьютерное зрение и машинное обучение (искусственные нейронные сети). Феномные технологии имеют высокую информативность анализа, превосходя возможности человека, проводя измерения в гиперспектральном диапазоне, используя X-ray-томографию, сверхточные “термальные” имиджи, а также ряд других низкоинвазивных и прецизионных подходов. Получаемые с использованием феномных технологий массивы данных регистрируются и обрабатываются автоматически и лишены проблемы субъективной оценки и неадекватной статистической обработки. Предполагается, что фенотипирование может стать методологией, которая позволит создать цифровые модели процессов жизнедеятельности и “формирования” продуктивности растений на организменном уровне в связи с динамикой транскриптомов, протеомов и метаболомов. Феномика помогает исследователям трансформировать большое количество информации, получаемой от современных датчиков и сенсоров, в новые знания с помощью компьютерной обработки данных и моделирования, сокращая расстояние от фундаментальной науки до практического применения результатов в растениеводстве и селекции. Фенотипирование активно развивается как в лабораторных и в тепличных условиях, так и на открытых сельскохозяйственных площадках, лесных массивах и в реальных природных фитоценозах. В обзоре анализируется текущее состояние феномики растений с фокусом на технические аспекты, в частности, устройство аппаратно-программных комплексов фенотипирования – т.е. феномных платформ, а также на использование нейронных сетей в фенотипировании растительных организмов.
ВВЕДЕНИЕ
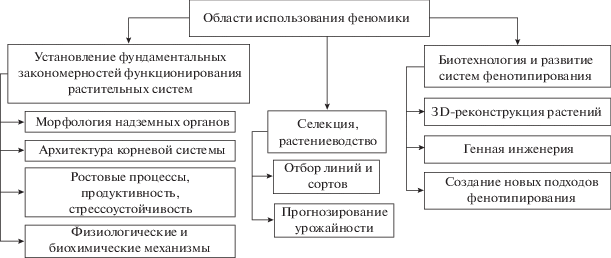
Фенотипирование, или высокопроизводительное фенотипирование (high-throughput phenotyping), в последние годы демонстрирует значительный прогресс и имеет большой потенциал для всех областей фундаментальной и прикладной биологии растений. Ее развитие привело к формированию “феномики растений” – фундаментального раздела физиологии растений, который концентрируется на выявлении закономерностей формирования, организации и изменения растительных феномов (совокупности фенотипов) во взаимосвязи с влиянием внешних факторов, характеристиками генотипа, паттернами экспрессии генов и функционального проявления белков (рис. 1). Феномика растений, ввиду ее стремительного развития, часто рассматривается как революционное направление не только фундаментальной, но и прикладной биологии, хотя в ее основе лежат принципы и подходы, заложенные несколькими столетиями исследований в области физиологии растений [1–3]. Использование феномных подходов позволяет выявить детерминанты продуктивности, идентифицировать механизмы устойчивости растений к патогенам и абиотическим стрессорам, произвести планирование урожая на сельскохозяйственных угодьях [4–7] (рис. 1). Применение феномных технологий в агро- и биоценозах позволяет более эффективно проводить экологический мониторинг и решать глобальные проблемы в области экологической безопасности.
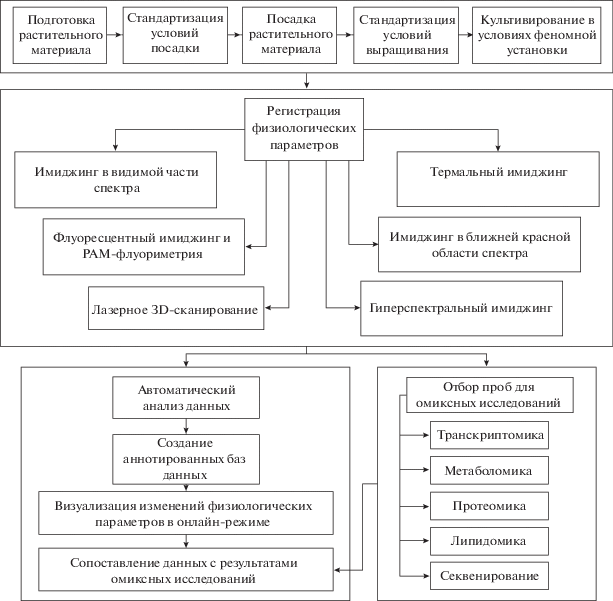
Бурное развитие технологий фенотипирования главным образом связано с прогрессом в регистрации цифровых изображений и методов оценки фенотипических характеристик с помощью систем компьютерного зрения и машинного обучения [8–10] (рис. 2). Большое внимание исследователей-физиологов направлено на совершенствование и дальнейшее развитие феномных платформ, сенсоров, робототехники, а также программного обеспечения на всех этапах фенотипирования.
Цель обзора – анализ информации о текущем состоянии феномики растений как уже неотъемлемого раздела фитофизиологии с фокусом на ключевые технические аспекты, такие как устройство и характеристики феномных платформ, а также использовании нейронных сетей для анализа данных, полученных при фенотипировании растений. Такой фокус обусловлен тем, что именно технический прогресс являлся “драйвером” развития современной феномики, в связи с чем понимание возможностей и будущего данного научного направления в значительной степени зависит от развития новых технологий фенотипирования, усовершенствования феномных платформ и прорывов в области искусственного интеллекта.
ФУНДАМЕНТАЛЬНЫЕ ПОНЯТИЯ И ОСОБЕННОСТИ РАЗВИТИЯ ФЕНОМИКИ
Феном – совокупность форм проявления организма. Он слагается из фенотипов – композиций количественных и качественных признаков живой системы на определенном этапе онтогенеза и в конкретных условиях обитания. Феномика изучает и сводит к закономерностям процессы формирования, изменения и регуляции фенотипических проявлений живых систем [11–13]. Она появилась на стыке биологии, физики, инженерии и компьютерных технологий и использует различные подходы пассивного и экспериментального наблюдения для описания фенотипов в виде массивов графических, физиологических и биохимических данных. Феномика получила большое развитие в связи с появлением доступной цифровой фото- и видеосъемки и необходимостью неинвазивного измерения большого количества характеристик растений в широкомасштабных скрининговых тестах [14–17]. Применение феномики в настоящее время все больше определяет успех молекулярно-генетических и селекционных работ, в особенности, на полевых культурах [6]. Некоторые исследователи полагают, что феномику растений можно считать физиологией растений, но в более современной “цифровой” и практически ориентированной форме [1].
Строение растительных организмов исключительно пластично и разнообразно в рамках проявления одного и того же генотипа. Оно в большей степени, чем у животных, зависит от условий внешней среды. Засуха, дефицит азота и низкая освещенность оказывают огромное влияние на количество и размер органов растения, описание разнообразия проявлений которых невозможно без современных цифровых методов. В этом отношении феномные подходы практически уникальны, так как впервые создают потенциальную возможность детально и несубъективно разобраться (до уровня выведения закономерностей) в широчайшем многообразии форм растительных фенотипов при воздействии на них сложных комбинаций динамично изменяющихся факторов среды [6, 18] (табл. 1).
Таблица 1.
Примеры феномного анализа в физиологии растений
| Объект | Параметры | Функции |
|---|---|---|
| Лист (побег) | Морфология, флуоресценция хлорофиллов | Установление механизмов ответов на засуху и засоление; визуализация внедрения патогена; тестирование взаимосвязи между увеличением площади листа и накоплением биомассы [38, 45, 92, 93] |
| Корень | Морфология | Тестирование изменений морфологии в ответ на фитогормоны; анализ развития корневой системы [47, 94, 95] |
| Семена | Морфология, всхожесть | Поиск корреляции между локусами количественных признаков и морфологическими особенностями семян и проростков; оценка качества семян [10, 49] |
| Цветки | Количество побегов, колосьев и зерен в колосе, количество цветков в соцветии | Поиск корреляции между параметрами соцветия и урожайностью; предсказание урожайности [96, 97] |
| Целое растение | Физиологическое состояние, урожайность | Модели оценки физиологического состояния и водообмена [98, 99] |
| Клетки водорослей | Скорость и частота биения жгутиков | Выявление закономерностей работы ресничек и жгутиков [100] |
Феномика растений использует достижения геномной эры и классической биоинформатики, дополняя их стандартизированным и статистически обработанным “количественным” фактологическим материалом с высокой степенью детализации. В качестве одной из перспективных ее задач рассматривается сведение цифровой информации о фенотипе к стандартам “гомологического” сравнения, концептуально схожим с рутинным сравнением нуклеотидных и белковых последовательностей [19, 20]. Получаемые с использованием феномных технологий массивы данных регистрируются и обрабатываются автоматически, поэтому, в отличие от классических методик, они не имеют проблемы субъективности и низкой статистической достоверности [21]. Феномные технологии высокоинформативны, превосходят возможности человека-оператора, в них вовлечены гиперспектральный анализ [22, 23], X-ray-томография [24], термальный имиджинг (thermal imaging) [25] и ряд других низкоинвазивных подходов (табл. 2). Высокоэффективное фенотипирование развивается не только в закрытых площадках, но также и на открытых сельскохозяйственных угодьях, лесных и других природных фитоценозах [4, 26]. Данный раздел так называемой полевой феномики (field phenomics) отличается внедрением программируемой автономной сельскохозяйственной техники, рельсовых систем, а также специализированных беспилотных летательных аппаратов – дронов, мультикоптеров, аэростатов и др. [3, 27, 28].
Таблица 2.
Методы регистрации изображений, используемые при фенотипировании растений
| Метод регистрации | Параметры | ||||
|---|---|---|---|---|---|
| датчик | результат, получаемый сенсором | первичные данные анализа информации с сенсора | измеряемые параметры фенотипа | условия проведения эксперимента | |
| Имиджинг в видимом диапазоне (RGB Imaging) | Камера, чувствительная к видимой области спектра | Изображение целого растения или его отдельных частей | RGB-изображения или изображения в оттенках серого | Площадь проекции, динамика роста, биомасса, показатели урожайности, архитектура (архитектоника) корня, скорость прорастания, время цветения и т.д. | Полевые, лабораторные (контролируемые) |
| Флуоресцентный имиджинг (Fluorescent Imaging) | Флуоресцентная камера, PAM-флуориметр | Изображение всего побега, его участка или тканей листа | Пиксельная карта излучаемой флуоресценции в красной и дальней красной областях, величина интенсивности светового потока в области флуоресценции хлорофилла | Фотосинтетический статус, квантовый выход, физиологическое состояние листа, архитектура побега | Полевые, лабораторные (контролируемые) |
| Термальный имиджинг (Thermal Imaging) | Камера, чувствительная к ближней инфракрасной области спектра (NIR-камера) | Пиксельная карта температуры поверхности в инфракрасном спектре | Изображения листьев или целых побегов, временные ряды | Температура поверхности листа, наличие и степень инвазии насекомых | Лабораторные (контролируемые) |
| Имиджинг в ближней инфракрасной области спектра (Near Infrared Imaging) | NIR-камера, мультиспектральный линейный сканер | Непрерывный или дискретный спектр в ближней инфракрасной области для каждого пикселя | Временные ряды или одномоментный анализ побегов и крон, точечная оценка семян | Параметры водного статуса, в т.ч. для семян | Лабораторные (контролируемые) |
| Гиперспектральный имиджинг (Hyperspectral Imaging) | NIR-камера, спектрометр, гиперспектральная камера | Непрерывный или дискретный спектр | Вегетационные циклы | Параметры водного статуса, физиологическое состояние, показатели роста | Лабораторные (контролируемые) |
| 3D-имиджинг (3D Imaging) | Система стереоскопических камер, камеры ‘Time-of-Flight’ |
Серия снимков всего побега при разных разрешениях | Карты глубины | Морфология семян, ориентация листьев, структура кроны, архитектура корня, линейные размеры растения | Полевые, лабораторные (контролируемые) |
| Лазерный имиджинг (Laser Imaging) | Лазерный сканер | Серия снимков всего побега при разных разрешениях | Карты глубины, 3D-облака точек |
Морфология и биомасса семян, ориентация листьев, структура кроны, архитектура корня, линейные размеры растения | Полевые, лабораторные (контролируемые) |
| Магнитно-резонансная томография (Magnetic Resonance Tomography) | Магнитно-резонансный томограф | Изображения с разрешением 200–500 нм |
Водные (1Н)-карты | Трехмерные морфометрические параметры, содержание воды | Лабораторные (контролируемые) |
| Позитронная эмиссионная томография (Positron Emission Tomography) | Позитронный эмиссионный детектор для изотопов с малым временем жизни (например, 11CO2) | Изображения с разрешением 1–2 нм | Карты движения радиодетектора, позитронные эмиссионные сигналы (совместная регистрация) | Разделение и скорость потоков жидкости | Лабораторные (контролируемые) |
| Компьютерная томография (Computer Tomography) | Рентгеновский компьютерный томограф, рентгеновский цифровой томограф | Изображения с разрешением около 100 нм | Вокселы и срезы тканей | Трехмерные морфометрические параметры, морфологические характеристики зерна | Лабораторные (контролируемые) |
Примечание. При создании таблицы были использованы данные, представленные в работе [37], с дополнениями и изменениями.
Возникновение и становление феномики растений напрямую связано с прогрессом в регистрации цифровых изображений и развитием компьютерной и системной биологии. Доступность получения, анализа, хранения и обработки цифровых RGB-изображений легла в основу создания первых феномных платформ – программно-аппаратных комплексов, адаптированных под конкретные экспериментальные нужды [29] (табл. 3). Выход за пределы RGB-диапазона, т.е. видимой части спектра электромагнитного излучения, в последние годы дополнительно расширил возможности фенотипирования [23, 30].
Таблица 3.
Характеристики основных программно-аппаратных платформ, применяемых для фенотипирования растений (феномных платформ)
| Платформа | Тип/Сенсоры | Объекты | Характеристика |
|---|---|---|---|
| LemnaTec (Аахен, Германия) | |||
| Lab Scanalyzer | Компактная модульная лабораторная система; МБС/FL, HS, NIR, TH, VIS | Небольшие растения, семена, плоды, водоросли | Три размерных конфигурации: 600 × 600× × 1128.5 мм, 600 × 600 × 1750 мм, 2059 × × 660 × 1290 мм. Ручная инсталляция растений; ПО для мониторинга скорости роста, биомассы, изменение архитектуры растения |
| Scanalyzer HTS | Модульная лабораторная система средних размеров; МБС/FL, HS, LS, NIR, TH, VIS | Лист растения, грибы, нематоды, личинки | Инсталляция широкого спектра культивационных сосудов. Возможность исследования светового цикла, движения объектов, и анализа живых клеток с использованием ПО LemnaLauncher |
| Phenocenter | Лабораторная конвейерная система с роботизированным манипулятором; ПР/FL, LS, NIR, VIS | Небольшие растения | Оборудуется боксами для фотосъемки, до 6 сенсоров в боксе. Основная задача – получение максимального полных и точных данных о феноме растения |
| Germination Scanalyzer | Модульная лабораторная система малых или средних размеров, МБС/LS, VIS | Семена растений | Оценивает размер, форму, окраску семян, длину корня и гипокотиля проростка, количество проросших семян, скорость прорастания, гомогенность, наличие болезней, посторонних объектов |
| Scanalyzer FIELD | Крупномасштабная полевая система, МБС/FL, HS, LS, NIR, TH, VIS | Злаковые и кормовые культуры, популяции растений | Рельсовая П-образная система шириной до 15 м либо стойка с мостом на монорельсовой платформе для автоматического сбора данных о росте, морфологии и физиологическом состоянии растений в полевых условиях. Сенсоры смонтированы на роботизированной руке-манипуляторе, удерживающей и подносящей к исследуемым растениям сенсорную “головку”. |
| Photon Systems Instruments (Драсов, Чехия) | |||
| PlantScreen™ Modular Systems | Лабораторная конвейерная система средних размеров; ПР/FL, HS, LS, NIR, TH, VIS | Травянистые растения | Флагман линейки платформ PSI. Производится контроль характеристик роста и развития растений, реакций на биотические и абиотические стрессоры. Имеет максимальное количество сенсоров среди всех систем PSI. В состав платформы также входят установки для взвешивания и полива. |
| PlantScreen™ Robotic XYZ Systems |
Лабораторная система средних размеров; МБС/FL, HS, TH, VLS | Небольшие растения | Содержит все типы доступных сенсоров и камер, способна к высокоточному перемещению в 3D-режиме в пределах объемной площадки размерами 4.5 × 4.5 × 4 м. Позволяет производить детальное изучение растений на площадке, находящихся в любых емкостях и имеющих любые геометрические параметры. |
| PlantScreen™ Compact Systems | Компактная лабораторная конвейерная система; ПР/FL, HS, LS, NIR, TH, VIS | Небольшие растения | Могут быть инсталлированы все виды лабораторных сенсоров. Растения культивируются в индивидуальных сосудах либо сгруппированы по лоткам в многоячеечных платах. Доступно управление условиями среды: интенсивностью и спектральным составом света, температурой, влажностью |
| PlantScreen™ Self Contained Systems | Компактная роботизированная система для теплиц; ПР/FL, HS, TH, VIS | Растения длиной до 50 см | Могут быть инсталлированы все виды лабораторных сенсоров. Автономный автоматизированный комплекс для изучения одиночных растений высотой до 50 см в почве или in vitro. Вегетационный сосуд вручную помещается в свето-изолированный бокс с полностью контролируемыми условиями, где проводится автоматический сбор и анализ данных через заданные интервалы времени |
| PlantScreen™ Field | Полевая система; МБС/FL, HS, TH, VIS | Злаковые и кормовые культуры | Могут быть инсталлированы все виды полевых сенсоров. Система схожа с LemnaTec Scanalyzer FIELD |
| Phenospex (Маастрихт, Нидерланды) | |||
| PlantEye F500 | Мультиспектральный 3D-сканер/VIS, NIR, LS | Небольшие растения | 3D-моделирование и получение спектральных изображений, автоматическая оценка морфологических параметров (высота растения, площадь листовой поверхности, биомасса, глубина проникновения света и др.), визуализация полученной информации в виде трехмерного облака точек |
| Phenospex FieldScan | Крупномасштабная полевая система; МБС/VIS, NIR, LS, FL, TH | Злаковые и кормовые культуры | Максимальный спектр полевых камер и сенсоров. Пропускная способность до 5000 растений в час. Ширина сканирования до 50 м, длина до 500 м, скорость движения до 15 м/мин, скорость сканирования 3 м/мин. Имеется возможность использования нескольких сканеров параллельно. Снабжена системой мониторинга и анализа метеоданных |
| CropDesign (Гент, Бельгия) | |||
| TraitMill | Смарт-теплица/VIS, NIR, LS | Злаковые культуры | Глубокое высокопроизводительное фенотипирование различных видов растений. Используются наиболее современные технологии автоматического сбора данных, статистической обработки, биоинформатики |
| Optimalog (Тур, Франция) | |||
| Phenopsis | Лабораторная система средних размеров/VIS, NIR | Небольшие растения, арабидопсис | Комплекс обеспечивает культивирование растений арабидопсиса в контролируемых условиях, автоматический полив и взвешивание до 500 опытных образцов в индивидуальных горшках, получение VIS-/NIR-изображений, их хранение и анализ; компоненты системы управляются пакетом Optima PLC через интернет |
| SMO и VIB (Экло, Гент, Бельгия) | |||
| WIWAM XY | Лабораторная система малых размеров/VIS, NIR, HS | Небольшие растения, арабидопсис | Модульная высокоточная система, устанавливаемая в ростовой камере. Точность полива достигает ±0.1 мл. Данные в системах WIWAM представляются в виде баз данных SQL, а все процессы управляются программным пакетом PIPPA |
| WIWAM Line | Лабораторная система средних размеров/VIS, NIR, HS | Небольшие растения | Позиционирование горшков, базовый робот-портал и станция для полива и взвешивания экспериментальных образцов. Изображения получают с расположенного в портале сбоку неинвазивного сенсора под различными углами |
| WIWAM Conveyor | Тепличная конвейерная система/VIS, NIR, HS | Крупные растения, злаковые культуры | В комплекс входят транспортная система, объединяющая растения с несколькими станциями взвешивания/полива, и феномные боксы, несущие ряд неинвазивных сенсоров |
| PHENOSCOPE (Хеерлен, Нидерланды) | |||
| PlantEye F500 | Мультиспектральный 3D-сканер/VIS, NIR, LS | Небольшие растения | Возможность получения мультиспектральных изображений, оценки площади листьев и угла их наклона, автоматического расчета вегетационных индексов |
| FieldScan | Мультиспектральный 3D-сканер; полевая система; МБС/VIS, NIR, LS | Крупные растения | Мультиспектральный сканер на подвижной П-образной раме позволяет фенотипировать десятки тысяч растений. ПО HortControl выполняет анализ и визуализацию измеряемых показателей, предоставляет доступ к данным по протоколам удаленного доступа |
| MobileDevice | Лабораторная система малого размера; МБС/VIS, NIR, LS | Небольшие растения | Одинарный или сдвоенный мультиспектральный 3D-сканер, передвигающийся вдоль балки в ростовой камере |
| DroughtSpotter | Гравиметрическая система | Небольшие растения | Оценка потери воды при транспирации; сочетание прецизионных весов и системы полива |
| FieldScale | Полевая гравиметрическая система | Небольшие растения | Система представляет собой полевую реализацию концепции DroughtSpotter |
Примечание. ПО – программное обеспечение; ПР – передвижение растений (растения двигаются к неподвижному сенсору/системе регистрации при помощи конвейерного или роботизированного манипулятора); МБС – мобильный блок сенсоров (растения неподвижны, передвигается блок с сенсорами/системами регистрации); FL (Fluorescent) – флуоресцентный датчик; HS (Hyperspectral) – датчик широкого диапазона электромагнитного излучения (~400–2700 нм) с количеством спектральных каналов ≥ 100 с разрешением 2–4 нм; LS (Laser scanner) – лазерный сканер для получения 3D-изображения; NIR (Near infra-red) – датчик света в ближней инфракрасной области спектра; TH (Thermal) – датчик излучения с длиной волны 9–14 нм; VIS (Visible light sensor) – датчик видимого света.
Сейчас прогресс в области фенотипирования связан с внедрением стационарных систем, когда растения и камеры обездвижены либо передвигается только регистрирующая аппаратура. Стационарные системы приходят на смену классическим конвейерным, в которых растения передвигаются по бегущей ленте или роликам. Стационарные системы позволяют минимизировать механическое воздействие сотрясения растений при перемещении, что критически важно для стандартизации измерений. Параллельно происходит усовершенствование программного обеспечения для управления феномными комплексами и анализа данных [4, 30, 31]. Активно идет технологическая революция, выражающаяся во внедрении методов машинного обучения и искусственных нейронных сетей, позволяющая перейти на качественно новый уровень анализа изображений. Внедрение искусственных нейронных сетей значительно расширяет биоинформационные возможности феномики как раздела фундаментальной биологии, поскольку обеспечивает пласт новых данных о механизмах функционирования организма (в особенности процессов формирования его органов), количественных и качественных показателей фенотипа в связи с набором биомассы и приобретением стрессоустойчивости [32, 33].
ОБЪЕКТЫ И ПРЕДМЕТЫ ИССЛЕДОВАНИЯ В ФЕНОМИКЕ РАСТЕНИЙ
Фенотипирование растений – это процедура оценки фенотипа растения по его размерам, форме, физиолого-биохимическим характеристикам в конкретных условиях внешней среды и активности генома [34, 35]. Фенотипирование возможно на всех уровнях организации организма: субклеточном, клеточном, тканевом, организменном и фито(агро)ценотическом. Современные методы фенотипирования обладают высокой производительностью, позволяют получать данные в режиме реального времени и анализировать информацию о целом спектре физиологических параметров (табл. 1). Эти методы дают возможность детально исследовать процессы роста, развития и размножения растений, фотосинтеза, дыхания, водообмена и минерального питания, механизмов регуляции продуктивности и стрессоустойчивости [1, 36, 37].
Феномика – это относительно молодая область знаний, поэтому круг используемых ею объектов и приложений пока ограничен, но быстро увеличивается. В настоящее время феномные исследования в большей степени сфокусированы на регистрации морфометрических и физиологических характеристик надземных частей растений, таких как листья, стебли, плоды, реже – цветки [38–40] (рис. 2). Слабее развиты платформы для анализа архитектоники корневой системы, хотя и в этом направлении в последние 8–10 лет очевиден огромный прогресс [41–43]. Основными объектами фенотипирования являются модельные и сельскохозяйственные виды высших растений, а также некоторые виды водорослей. Наибольшее количество платформ разработано для Arabidopsis thaliana L. Heynh (арабидопсиса) – важнейшего модельного объекта, своего рода “растительной дрозофилы”. Имеются платформы для более чем 20 видов злаковых, бобовых и овощных культур, а также для около 10 видов древесных растений. Среди последних фокус делается на тополь, яблоню и виноград. Небольшое количество платформ также развито для декоративных травянистых растений.
Фенотипирование побегов в основном направлено на получение информации о физических размерах, геометрии и движении листьев и других надземных органов растения, а также о таких функциях, как фотосинтез, дыхание и транспирация [1] (табл. 1). Полученные в последние годы с помощью феномного анализа имиджей флуоресценции хлорофилла, позволили построить фундаментальные модели “поведения” хлоропластов в связи с их фотохимической активностью [36, 44], выявить молекулярные механизмы воздействия на фотосинтетические процессы абиотических стрессоров и патогенов [45]. На основе фенотипирования количественных изменений площади листьев удалось создать модель формирования и развития розетки арабидопсиса, выявить центральные генетические механизмы, вовлеченные в этот процесс, установить взаимосвязь между увеличением площади листа, фотосинтетическими показателями и накоплением биомассы [38, 46].
Фенотипирование корневой системы позволяет производить мониторинг как макроскопических (размеры, архитектоника, ветвление), так и микроскопических характеристик (строение трихобластов, тканей, стели). Глобальные взаимосвязи между этими параметрами, раскрытые при помощи современного фенотипирования, оказались намного сложнее, чем ранее ожидалось [6]. 3D-моделирование архитектоники корня в последние годы успешно комбинируется (с целью обнаружения корреляций) с анализом генов, кодирующих рецепторные и другие системы, обеспечивающие быстрые ростовые реакции корня [47]. Также немаловажно отметить, что появились феномные комплексы для исследования роста и развития корневых систем в полевых условиях, например, платформа DIRT (от англ.: Digital Imaging of Root Traits), революционно расширившая в последние годы представления о развитии корневой системы в поле [48].
Разработан ряд методов анализа изображений семян для оценки их качества [49]. Системы фенотипирования позволяют автоматически оценивать форму, размеры и другие морфологические показатели различных типов семян, что выявило ряд совершенно новых закономерностей их формирования [50]. Среди феномных исследований семян также одним из активно развивающихся направлений является анализ генетических детерминант естественной изменчивости их всхожести и устойчивости к патогенам и к абиотическим стрессорам [49]. Для фенотипирования плодов развиты многочисленные методы автоматизированного анализа изображений, полученных с помощью таких сенсоров, как лидары (LIDAR, от англ.: Light Detection and Ranging). Например, феномная реконструкция виноградных гроздей на основе данных лидаров, позволила впервые создать 3D-модель гроздей с точностью до 0.025 мм, что имеет фундаментальное значение для исследований в области физиологии винограда [51].
ПЛАТФОРМЫ ФЕНОТИПИРОВАНИЯ РАСТЕНИЙ
Для автоматизации фенотипирования разработан широкий спектр автоматизированных “феномных платформ” – аппаратно-программных комплексов, обеспечивающих стандартизацию условий, инсталляцию растений, регистрацию изображений и информации от сенсоров, а также их обработку в режиме онлайн [9, 52–54]. Феномные платформы могут включать боксы для культивирования растений, измерительное оборудование, системы калибровки и перемещения растений, камер и сенсоров, системы автоматического взвешивания, полива, освещения, поддержания микроклимата, датчики контроля работоспособности и прохождения циклов, системы компьютерного управления процессами, хранения и обработки информации, а также серверы и программное обеспечение для более глубокого анализа изображений, базирующееся на системах машинного зрения и обучения. Количество регистрируемых и анализируемых параметров зависит от насыщенности феномных платформ регистрирующими устройствами – RGB-, гиперспектральными и тепловыми/термальными камерами, флуориметрами, 3D-сканерами, лидарами, X-RAY-, магнитно-резонансными, позитронно-эмиссионными томографами и др. [52] (табл. 3). Имеются как узкоспециализированные платформы, например, DIRT или SmartGrain, так и более широкопрофильные (линейки LemnaTec или PSI).
Основной принцип работы феномных платформ – сбор данных, характеризующих морфологические и физиологические параметры фенотипа, их обработка и визуализация. В большинстве случаев растения выращиваются с использованием элементов платформ: теплиц, стендов, конвейеров с вегетационными сосудами или автоматизированных стеллажей в полностью контролируемых условиях. С заданной периодичностью растения передвигаются в боксы с датчиками (конвейерный тип платформ) либо роботизированная система выносит датчики к остающимся неподвижными растениям (стационарный тип платформ).
Для платформ корневой феномики используются затемненные системы вертикальной гидропоники, плоские и цилиндрические сосуды с почвенными субстратами, гелевые вертикальные системы с различной степенью автоматизации и типами съемки [55]. Для построения структуры поверхности почвы с выращиваемыми растениями используется электротомография (ERT, от англ.: Electrical Resistance Tomography,) онлайн [52–54]; для неинвазивного картирования внутрипочвенной проводимости и построения картины распространения корней применяется техника электромагнитной индукции почвы (ESMI, EMI, от англ.: Electromagnetic Soil Inductance), с помощью почвенных радаров (GPR, от англ.: Ground Penetrating Radar) осуществляется картирование внутрипочвенных структур на основе рефракции и рассеивания импульсов высокочастотных радиоволн [55].
Наиболее популярными феномными платформами являются LemnaTec (Германия), Photon Systems Instruments (Чехия), Qubit Phenomics (Канада), Phenomix (Франция), Phenospex (Австралия), Delta-T Devices Ltd. (Великобритания), Heinz Walz (Германия), WPS (Голландия), CropDesign (BASF, отделение в Бельгии), WIWAM (Бельгия), Rothamsted Research (Великобритания) и VBCF (Австрия). На их долю приходится почти 100% феномного оборудования. Программное обеспечение для фенотипирования весьма разнообразно и постоянно адаптируется под новые задачи. Обзор программных продуктов и решений для феномики приводится порталом Plant Image Analysis (www.plant-image-analysis.org). Коммерческие феномные системы часто имеют закрытые коды, однако значительное количество программ уже являются бесплатными и имеют открытые исходные данные. Активно ведутся разработки систем машинного обучения (искусственных нейронных сетей) для приложений в области феномики, что кардинально повышает возможности феномных систем [9]. Прогнозируется, что искусственные нейронные сети вытеснят классические системы анализа изображений.
Ниже приводится информация о крупнейших феномных платформах. В значительной степени данные платформы, разрабатываемые крупными исследовательскими центрами и инновационными компаниями, инкорпорируются более мелкими производителями для создания спектра более узкоспециализированных систем. Детальные сведения о технических характеристиках платформ приводятся на сайтах компаний и исследовательских центров.
Платформы LemnaTec. Компания LemnaTec (г. Ахен, Германия) является старейшим и крупнейшим производителем и разработчиком инновационных феномных платформ для фенотипирования растений (www.lemnatec.com). Компания активно сотрудничает с университетами Германии и других стран, что позволяет поддерживать исключительно высокий уровень разрабатываемых и внедряемых технологий. LemnaTec имеет широкую гамму платформ для закрытых лабораторных помещений, теплиц, открытых опытных площадок и полевых условий, а также ведет разработку аэроплатформ. Детальная информация по платформам LemnaTec приведена в таблице 3. В последние годы, благодаря использованию феномных платформ LemnaTec, раскрыт ряд важнейших закономерностей функционирования растительных систем. Например, работы на базе платформы LemnaTec Lab Scanalyzer позволили выявить гены, ответственные за контроль толщины листа и приобретение листовой пластинкой различных морфологических форм у томатов [56]. С ее помощью также была разработана стратегия регистрации повреждения листьев сахарной свеклы и сои под действием гербицидов [57]. С использованием миниплатформы LemnaTec 3D-Scanalyzer установлена роль N-терминальной части белков DELLA в гиббереллин-зависимом поддержании покоя семян пшеницы [58]. С использованием платформы LemnaTec Phenocenter обнаружена уникальная система поддержания водного статуса у злаков [35] и создана технология оценки эффективности фотосинтеза для основных групп высших растений [59]. Системы на базе LemnaTec Scanalyzer FIELD, внедренные в полевую феномику, дали возможность впервые описать фенотипические проявления для большого числа генов и связать их активность с конкретными физиологическими процессами, такими как фотосинтез, дыхание, стрессоустойчивость и контроль архитектоники побегов [60].
Платформы Photon Systems Instruments (PSI). Данные платформы разрабатываются и производятся в г. Драсов (Чехия). Крупнейший Североамериканский разработчик и продавец феномных систем Qubit Phenomics (Канада) является отделом PSI. Отличительной чертой PSI является собственное производство камер и сенсоров. Камеры PSI FluorCam используют систему амплитудно-импульсной модуляции светового потока (PAM), совмещенную с высокочувствительными CCD-камерами и позволяющую измерять параметры флуоресценции хлорофилла (Fv/Fm, Y(II) и др.) в виде 2D-имиджа высокого разрешения. Оригинальные разработки PSI включают также флуориметры, RGB- и “флуоресцентные” камеры, LED-осветители, источники импульсного освещения, сенсоры и датчики физических параметров среды, конвейеры, рефрактометры, роботизированные манипуляторы, фотобиореакторы, инкубаторы для водорослей. На сегодняшний день PSI имеет широчайшую гамму феномных платформ различного уровня, созданных в основном на базе собственных разработок. PSI также представляет мобильные платформы в виде автономных тракторов и беспилотных аэрофеномных комплексов и уникальные контейнерные системы, располагающиеся на любой открытой площадке (в морских контейнерах). Платформы PSI формируются по модульному принципу для нужд конкретного исследования.
С использованием PSI-платформ и сенсоров уже в течение двух десятилетий проводятся исследования в различных направлениях физиологии, биотехнологии и агрохимии. Среди работ физиологической направленности можно отметить, выявление механизмов, лежащих в основе контроля структурной организации тилакоидной мембраны под действием света различной интенсивности, демонстрацию роли движения листьев в фотохимических реакциях на фоне различной обеспеченности водой, установление природы зависимых от температуры и от влажности изменений в фотохимических реакциях фотосинтеза у Nostoc commun [61]. Недавно Mishra с соавт. [62] с использованием оборудования PSI показали, что флуоресценция хлорофилла а может являться показателем дефицита воды в листьях высших растений. На базе платформ PSI Kamarainen с соавт. [63] продемонстрировали, что пиридин-нуклеотид-трансгидрогеназа PntAB необходима для оптимального роста и фотосинтеза при низкой освещенности у Synechocystis sp. Ferroni с соавт. [64] охарактеризовали структурно-функциональную организацию тилакоидной мембраны плауна Selaginella martensii, продемонстрировав, что при переходе от низкой степени освещенности к высокой архитектура тилакоидов изменяется от псевдоламинарной до преимущественно гранальной, при этом соотношения ФС II/ФС I изменяется в сторону ФС I. Важным примером работ с использованием платформы PSI PlantScreen™ Modular Systems также является раскрытие комплекса молекулярных механизмов, обеспечивающих устойчивость Pisum sativum L. к холоду [65].
Среди работ биотехнологической направленности можно отметить разработку методов получения и оптимизации роста фотосинтезирующей суспензионной культуры клеток Arabidopsis thaliana, установление закономерностей влияния плотности культуры Synechocystis sp. PCC 6803 на продукцию биомассы и эффективность использования света в фотобиореакторах [66], а также выявление механизмов акклиматизации Chromera velia к световом стрессу на уровне реорганизации мембран при культивировании в фотобиореакторах [67].
Phenospex – феномные платформы и одноименная компания из Нидерландов (с 2011 г.), производящая аппаратуру и программное обеспечение для регистрации и анализа изображений растений в автоматическом режиме (табл. 3). Особенностью Phenospex является активное использование 3D-сканеров и автоматизированных полевых платформ.
CropDesign – биотехнологическая компания, основанная в 1998 г. на базе Гентского университета и приобретенная концерном BASF в 2006 г. Производит платформы TraitMill, специализирующиеся на анализе трансгенных растений риса и отличающиеся широким спектром камер и сенсоров, передовыми технологиями обработки информации (табл. 3).
Phenopsis – феномная платформа компании Optimalog (Тур, Франция), разрабатываемая при содействии Национального института сельскохозяйственных исследований Франции. Комплекс включает оборудование для выращивания растений арабидопсиса в контролируемых условиях, автоматического полива и взвешивания до 500 растений, системы регистрации VIS/NIR-имиджей, программное обеспечение для хранения и анализа изображений (табл. 3). Компоненты системы управляются пакетом Optima PLC через интернет. Данные публикуются на сайте http://bioweb.supagro.inra.fr/phenopsis/.
WIWAM – феномные системы компании SMO (Экло, Бельгия). Системы обладают способностью поддержания физико-химических условий (температура, влажность, освещенность), полива и автоматического имиджинга. Данные представляются в виде баз данных SQL, а все процессы управляются программным пакетом PIPPA. WIWAM XY – небольшой модульный комплекс, состоящий из станции позиционирования, роботизированной руки и станции для взвешивания, полива и имиджинга (табл. 3). Недавно с помощью WIWAM XY Skiryczс с соавт. [68] показали, что снижение ростовой активности может обеспечить стратегию повышения производительности биомассы растений при стрессе, а Clauw с соавт. [69] выявили закономерности ответов шести экотипов арабидопсиса на т.н. “мягкую” засуху.
PHENOSCOPE – феномная система Национального института сельскохозяйственных исследований Франции, представляющая платформу для мониторинга сотен индивидуальных горшков с небольшими растениями [70]. Имеет автоматический полив и взвешивание в соответствии с пользовательским сценарием. Станции имиджинга работают в VIS- и IR-диапазонах (табл. 3).
СИСТЕМЫ КОМПЬЮТЕРНОГО ЗРЕНИЯ И МАШИННОГО ОБУЧЕНИЯ И ИХ ИСПОЛЬЗОВАНИЕ В ФЕНОМИКЕ РАСТЕНИЙ
Среди аналитических подходов в феномике особое место занимают методы компьютерного зрения и машинного обучения. Компьютерное зрение – технология, с помощью которой компьютеры/машины могут обнаруживать, отслеживать, классифицировать и автоматически идентифицировать объекты, извлекая данные из изображений и анализируя полученную информацию [10]. Цель технологий компьютерного зрения – принятие решений о реальных физических объектах и сценах, основываясь на воспринимаемых изображениях. Для этого разрабатываются и используются библиотеки паттернов и специальные алгоритмы распознавания конкретных объектов. Стандартная обработка при помощи подходов компьютерного зрения позволяет убрать фон, очертить границы объекта (листья, побеги, стебли и т.п.), устранить шумы и выполнить другие подготовительные процедуры, предшествующие глубокому анализу с использованием, например, технологии машинного обучения [71].
Современные методы анализа изображений направлены на то, чтобы не только получать напрямую из снимков полезную информацию, но и тренировать распознающие алгоритмы, делать анализ в процессе сбора данных и “фундаментально понимать” то, из чего состоит конкретное изображение [53]. В этом отношении прорывными характеристиками обладает так называемое машинное обучение. Машинное обучение – область информатики, которая фокусируется на создании и использовании алгоритмов, позволяющих обучаться на основе собственного “опыта”, а также на примерах и аналогиях. Машинное обучение является комплексным подходом к анализу данных и использует теорию вероятности, статистику, теории принятия решений, визуализации и оптимизации. Возможность обучения создает условия для автоматического повышения точности расчетов с течением времени на основе предыдущих результатов. Машинное обучение активно используется для решения таких задач, в которых структура данных и закономерности их взаимосвязей заранее не известны.
В машинном обучении применяются алгоритмы, которые способны дать информацию о зависимостях между получаемыми данными без написания специфического кода под каждую задачу. При обработке цифровой и графической информации методы машинного обучения обладают намного более высокой эффективностью и рядом других очевидных преимуществ по сравнению с традиционными подходами. Текущий прогресс в машинном обучении привел к созданию масштабируемых, надежных и гибких программных инструментов (R-пакетов, наборов инструментов Matlab и программных пакетов, таких как Theano, Caffe для архетипических алгоритмов) [72, 73]. Одним из методов машинного обучения являются нейронные сети и их более узкое направление – сверточные сети, используемые для высокоточного анализа изображений [74]. Методы машинного обучения применяются для решения проблем, которые ранее считались затруднительными для моделирования [75]. Можно отметить такие известные примеры, как создание систем распознавания лиц, обработки речи (Google, Apple Siri), распознавания языков (IBM Watson), потребительских прогнозов (Netflix). В биологии методы машинного обучения применяют для аннотации геномов [76], в разработке лекарственных средств, в приложениях клеточной биологии [77], в гистологии и медицине при идентификации тканей [78, 79].
Одним из основных преимуществ использования подходов машинного обучения для феномики растений является возможность одновременного изучения комбинации многочисленных параметров и/или факторов вместо того, чтобы анализировать каждый из них отдельно. Высокая размерность изображений отдельных растений в сочетании с их огромным количеством вызывают практически непреодолимые трудности для их анализа. Алгоритмы классической информатики не в состоянии анализировать гигантский объем графических данных, получаемых при фенотипировании с использованием современных сенсоров высокого разрешения (RGB, NIR, HySpec и др.), не рассматривая в принципе их комбинации. В этой связи в современной феномике происходит все более активное внедрение подходов машинного обучения для анализа изображений [8, 53, 80]. Пока они используются как модули или небольшие программные продукты при определении видов, сортов и физиологического состояния растений в ведущих феномных платформах [8, 81].
Наиболее эффективны при цифровой обработке имиджей растительных систем нейронные сети с архитектурой Mask R-CNN [82]. Они впервые позволили выделять на фотографиях контуры (“маски”) перекрывающихся и неполных растительных объектов. Современные версии нейронной сети Mask R-CNN способны дополнять границы изучаемого объекта при его фрагментарной видимости с высочайшей точностью. Значительные успехи достигнуты в использовании машинного обучения при анализе гиперспектральных изображений для определения степени зрелости и сортовых различий у томатов, перцев и яблок [83]. Сверточные нейронные сети успешно применены при выявлении механизмов контроля и детального описания стадий развития листьев [84], раскрытии взаимосвязей между генотипом и фенотипом у целого спектра видов [85], выявлении особенностей повреждения листьев кукурузы патогенами [86], установлении механизмов молекулярного контроля архитектоники соцветий [87], построении первой модели целого растения с использованием алгоритмов сегментации [88], прогнозировании динамики и интенсивности хлороза при дефиците железа [89]. Сверточные нейронные сети применимы и в полевых исследованиях, в частности, при оценке качества посевного материала в селекции широкого спектра сельскохозяйственных растений [10], выявлении количественных математических характеристик процесса цветения хлопка [90].
Большинство работ с использованием нейронных сетей базируется на анализе RGB-изображений, однако имеются исследования, анализирующие и более широкий диапазон электромагнитного спектра. Недавно Ludovisi с соавт. [28] на основе данных тепловизионных (термальных) изображений беспилотных летательных аппаратов выявили механизмы первичных реакций черного тополя (Populus nigra L.) на недостаток влаги. На базе сверточных сетей Grift с соавт. [91] впервые разработали технику измерения глубины распространения корней с использованием электротомографии почвенного субстрата.
В целом потенциал использования нейронных сетей в феномике можно оценить очень позитивно. Данный подход в значительной степени создает предпосылки для появления “цифровой” интегративной феномики и физиологии растений (plant integrative phenomics and physiology), что позволит связать клеточные физиологические механизмы с разнообразием фенотипов, а также конститутивными и индуцированными генетическими программами.
ЗАКЛЮЧЕНИЕ
Новая область физиологии растений – феномика растений – регистрирует, накапливает и математически анализирует массивы данных о фенотипе растительного организма. Это направление претерпевает стремительное развитие, благодаря чему открываются абсолютно новые возможности фундаментальных исследований взаимосвязи между генотипом и фенотипом, что критически важно для перехода к высокотехнологичному сельскому и лесному хозяйству. Создан и успешно развивается целый спектр феномных платформ, включающих в себя регистрирующие устройства, по своим характеристикам перекрывающие максимально возможный на сегодняшний день диапазон неинвазивно тестируемых характеристик фенотипа. Технологический прорыв в феномике растений привел к ряду важных открытий в области фундаментальной биологии и обозначил будущий прогресс в селекции и сельскохозяйственной биотехнологии. Следует отметить активное внедрение в феномику растений инновационных программных средств, инженерных решений, сенсорных и роботизированных систем. Несомненно, в ближайшее время еще большее применение в феномике растений найдет технология нейронных сетей (машинного обучения), что даст качественный скачок как в данной области, так и в биологии растений в целом. Также растет уверенность в том, что это приведет к переходу физиологии, селекции и биотехнологии растений на качественно новый уровень. Феномика растений вследствие внедрения цифровой обработки информации о разнообразии фенотипических проявлений растений потенциально может довести развитие физиологии растений до скоростей, с которыми в настоящее время развиваются геномика и молекулярная биология.
Работа выполнена при финансировании Государственного комитета по науке и технологиям Беларуси в рамках Государственной программы “Наукоемкие технологии и техника”, а также совместных проектов с Министерством науки и технологий Китая (CB02-07), Департамента науки и технологий провинции Гаундонг (2018YFD0201203) и Международной базы по науке и технологиям провинции Гуандонг (163-2018-XMZC-0001-05-0049).
Авторы заявляют об отсутствии конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Список литературы
Furbank R.T., Tester M. Phenomics – technologies to relieve the phenotyping bottleneck // Trends Plant Sci. 2011. V. 16. P. 635. https://doi.org/10.1016/j.tplants.2011.09.005
Bolger M., Schwacke R., Gundlach H., Schmutzer T., Chen J., Arend D., Oppermann M., Weise S., Lange M., Fiorani F., Spannagl M., Scholz U., Mayer K., Usadel B. From plant genomes to phenotypes // J. Biotechnol. 2017. V. 261. P. 46. https://doi.org/10.1016/j.jbiotec.2017.06.003
Lee S., Persson P. Financing from family and friends // Rev. Financ. Stud. 2016. V. 29. P. 2341. https://doi.org/10.1093/rfs/hhw031
White J.W., Andrade-Sanchez P., Gore M.A., Bronson K.F., Coffelt T.A., Conley M.M., Feldmann K.A., French A.N., Heun J.T., Hunsaker D.J., Jenks M.A., Kimball B.A., Roth R.L., Strand R.J., Thorp K.R. et al. Field-based phenomics for plant genetics research // F. Crop. Res. 2012. V. 133. P. 101. https://doi.org/10.1016/j.fcr.2012.04.003
Araus J.L., Cairns J.E. Field high-throughput phenotyping: the new crop breeding frontier // Trends Plant Sci. 2014. V. 19. P. 52. https://doi.org/10.1016/j.tplants.2013.09.008
Афонников Д.А., Генаев М.А., Дорошков А.В., Комышев Е.Г., Пшеничникова Т.А. Методы высокопроизводительного фенотипирования растений для массовых селекционно-генетических экспериментов // Генетика. 2016. V. 52. P. 788. https://doi.org/10.7868/S001667581607002X
Colaço A.F., Molin J.P., Rosell-Polo J.R., Escolà A. Application of light detection and ranging and ultrasonic sensors to high-throughput phenotyping and precision horticulture: current status and challenges // Hortic. Res. 2018. V. 5. P. 35. https://doi.org/10.1038/s41438-018-0043-0
König I.R., Auerbach J., Gola D., Held E., Holzinger E.R., Legault M.-A., Sun R., Tintle N., Yang H.-C. Machine learning and data mining in complex genomic data – a review on the lessons learned in Genetic Analysis Workshop 19 // BMC Genet. 2016. V. 17. P. S1. https://doi.org/10.1186/s12863-015-0315-8
Cheng L., Zhang B., He L., Ma F.F., Wang X.C., Li H.Y., Han Y.H. Constitutive down-regulation of SiSGR gene is related to green millet in Setaria italica // Russ. J. Plant Physiol. 2017. V. 64. P. 608. https://doi.org/10.1134/S1021443717040045
Uzal L.C., Grinblat G.L., Namías R., Larese M.G., Bianchi J.S., Morandi E.N., Granitto P.M. Seed-per-pod estimation for plant breeding using deep learning // Comput. Electron. Agric. 2018. V. 150. P. 196. https://doi.org/10.1016/j.compag.2018.04.024
Skelly D.A., Merrihew G.E., Riffle M., Connelly C.F., Kerr E.O., Johansson M., Jaschob D., Graczyk B., Shulman N.J., Wakefield J., Cooper S.J., Fields S., Noble W.S., Muller E.G.D., Davis T.N. et al. Integrative phenomics reveals insight into the structure of phenotypic diversity in budding yeast // Genome Res. 2013. V. 23. P. 1496. https://doi.org/10.1101/gr.155762.113
Orgogozo V., Morizot B., Martin A. The differential view of genotype-phenotype relationships // Front. Genet. 2015. V. 6. P. 179. https://doi.org/10.3389/fgene.2015.00179
Lobos G.A., Camargo A.V, del Pozo A., Araus J.L., Ortiz R., Doonan J.H. Editorial: Plant phenotyping and phenomics for plant breeding // Front. Plant Sci., 2017. V. 8. P. 2181. https://doi.org/10.3389/fpls.2017.02181
Gerlai R. Phenomics: fiction or the future? // Trends Neurosci. Elsevier, 2002. V. 25. P. 506. https://doi.org/10.1016/S0166-2236(02)02250-6
Deans A.R., Lewis S.E., Huala E, Anzaldo S.S., Ashburner M., Balhoff J.P., Blackburn D.C., Blake J.A., Burleigh J.G., Chanet B., Cooper L.D., Courtot M., Csösz S., Cui H., Dahdul W. et al. Finding our way through phenotypes // PLoS Biol. 2015. V. 13. P. e1002033. https://doi.org/10.1371/journal.pbio.1002033
Kochinke K., Zweier C., Nijhof B., Fenckova M., Cizek P., Honti F., Keerthikumar S., Oortveld M.A.W., Kleefstra T., Kramer J.M., Webber C., Huynen M.A., Schenck A. Systematic phenomics analysis deconvolutes genes mutated in intellectual disability into biologically coherent modules // Am. J. Hum. Genet. 2016. V. 98. P. 149. https://doi.org/10.1016/j.ajhg.2015.11.024
Awada L., Phillips P.W.B., Smyth S.J. The adoption of automated phenotyping by plant breeders // Euphytica. 2018 V. 214. P. 148. https://doi.org/10.1007/s10681-018-2226-z
Hay F.R., Timple S., Duijn B. Van. Can chlorophyll fluorescence be used to determine the optimal time to harvest rice seeds for long-term genebank storage? // Seed Sci. Res. 2015. V. 25. P. 321
Schnable J.C., Freeling M. Genes identified by visible mutant phenotypes show increased bias toward one of two subgenomes of maize // PLoS One. 2011. V. 6. P. e17855. https://doi.org/10.1371/journal.pone.0017855
Burleigh J.G., Alphonse K., Alverson A.J., Bik H.M., Blank C., Cirranello A.L., Cui Y., Daly M., Dietterich T.G., Gasparich G., Irvine J., Julius M., Kaufman S., Law E., Liu J. et al. Next-generation phenomics for the Tree of Life // PLoS Curr. 2013. https://doi.org/10.1371/currents.tol.085c713acafc8711b2ff7010a4b03733
Flood P.J., Kruijer W., Schnabel S.K., van der Schoor R., Jalink H., Snel J.F.H., Harbinson J., Aarts M.G.M. Phenomics for photosynthesis, growth and reflectance in Arabidopsis thaliana reveals circadian and long-term fluctuations in heritability // Plant Methods. 2016. V. 12. P. 14. https://doi.org/10.1186/s13007-016-0113-y
Henebry G.M. Making sense of remotely sensing vegetation // Bioscience. 2011. V. 61. P. 568. https://doi.org/10.1525/bio.2011.61.7.13
Thorp K.R., Gore M.A., Andrade-Sanchez P., Carmo-Silva A.E., Welch S.M., White J.W., French A.N. Proximal hyperspectral sensing and data analysis approaches for field-based plant phenomics // Comput. Electron. Agric. 2015. V. 118. P. 225. https://doi.org/10.1016/j.compag.2015.09.005
Hughes N., Askew K., Scotson C.P., Williams K., Sauze C., Corke F., Doonan J.H., Nibau C. Non-destructive, high-content analysis of wheat grain traits using X-ray micro computed tomography // Plant Methods. 2017. V. 13. P. 76. https://doi.org/10.1186/s13007-017-0229-8
Jones H.G., Serraj R., Loveys B.R., Xiong L., Wheaton A., Price A.H. Thermal infrared imaging of crop canopies for the remote diagnosis and quantification of plant responses to water stress in the field // Funct. Plant Biol. 2009. V. 36. P. 978. https://doi.org/10.1071/FP09123
Warner J.L., Denny J.C., Kreda D.A., Alterovitz G. Seeing the forest through the trees: uncovering phenomic complexity through interactive network visualization // J. Am. Med. Informatics Assoc. 2015. V. 22. P. 324. https://doi.org/10.1136/amiajnl-2014-002965
Gago J., Douthe C., Coopman R.E., Gallego P.P., Ribas-Carbo M., Flexas J., Escalona J., Medrano H. UAVs challenge to assess water stress for sustainable agriculture // Agric. Water Manag. 2015. V. 153. P. 9. https://doi.org/10.1016/j.agwat.2015.01.020
Ludovisi R., Tauro F., Salvati R., Khoury S., Mugnozza Scarascia G., Harfouche A. UAV-Based thermal imaging for high-throughput field phenotyping of black poplar response to drought // Front. Plant Sci., 2017. V. 8. P. 1681. https://doi.org/10.3389/fpls.2017.01681
Pereyra-Irujo G.A., Gasco E.D., Peirone L.S., Aguirrezábal L.A.N. GlyPh: a low-cost platform for phenotyping plant growth and water use // Funct. Plant Biol. 2012. V. 39. P. 905. https://doi.org/10.1071/FP12052
Yang W., Duan L., Chen G., Xiong L., Liu Q. Plant phenomics and high-throughput phenotyping: accelerating rice functional genomics using multidisciplinary technologies // Curr. Opin. Plant Biol. 2013. V. 16. P. 180. https://doi.org/10.1016/j.pbi.2013.03.005
Paproki A., Sirault X., Berry S., Furbank R., Fripp J. A novel mesh processing based technique for 3D plant analysis // BMC Plant Biol. 2012. V. 12. P. 63. https://doi.org/10.1186/1471-2229-12-63
Gandomi A., Haider M. Beyond the hype: Big data concepts, methods, and analytics // Int. J. Inf. Manage. 2015. V. 35. P. 137. https://doi.org/10.1016/j.ijinfomgt.2014.10.007
Chen M., Du X., Zhu Y., Wang Z., Hua S., Li Z., Guo W., Zhang G., Peng J., Jiang L. Seed fatty acid reducer acts downstream of gibberellin signalling pathway to lower seed fatty acid storage in Arabidopsis // Plant. Cell Environ. 2012. V. 35. P. 2155. https://doi.org/10.1111/j.1365-3040.2012.02546.x
Granier C., Vile D. Phenotyping and beyond: modelling the relationships between traits // Curr. Opin. Plant Biol. 2014. V. 18. P. 96. https://doi.org/10.1016/j.pbi.2014.02.009
Fahlgren N., Gehan M.A., Baxter I. Lights, camera, action: high-throughput plant phenotyping is ready for a close-up // Curr. Opin. Plant Biol. 2015. V. 24. P. 93. https://doi.org/10.1016/j.pbi.2015.02.006
Dutta S., Cruz J.A., Jiao Y., Chen J., Kramer D.M., Osteryoung K.W. Non-invasive, whole-plant imaging of chloroplast movement and chlorophyll fluorescence reveals photosynthetic phenotypes independent of chloroplast photorelocation defects in chloroplast division mutants // Plant J. 2015. V. 84. P. 428. https://doi.org/10.1111/tpj.13009
Li L., Zhang Q., Huang D. A review of imaging techniques for plant phenotyping // Sensors. 2014. V. 14. P. 20078. https://doi.org/10.3390/s141120078
Vanhaeren H., Gonzalez N., Inzé D. A journey through a leaf: phenomics analysis of leaf growth in Arabidopsis thaliana // Arab. B. 2015. V. 13. P. e0181. https://doi.org/10.1199/tab.0181
Gregory P.J., Bengough A.G., Grinev D., Schmidt S., Thomas W.B.T.B., Wojciechowski T., Young I.M. Root phenomics of crops: opportunities and challenges // Funct. Plant Biol. 2009. V. 36. P. 922. https://doi.org/10.1071/FP09150
Plazas M., AndÃojar I., Vilanova S., Gramazio P., Herraiz F.J., Prohens J. Conventional and phenomics characterization provides insight into the diversity and relationships of hypervariable scarlet (Solanum aethiopicum L.) and gboma (S. macrocarpon L.) eggplant complexes // Front. Plant Sci. 2014. V. 5. P. 318. https://doi.org/10.3389/fpls.2014.00318
Pound M.P., French A.P., Atkinson J.A., Wells D.M., Bennett M.J., Pridmore T. RootNav: navigating images of complex root architectures // Plant Physiol. 2013. V. 162. P. 1802. https://doi.org/10.1104/pp.113.221531
Galkovskyi T., Mileyko Y., Bucksch A., Moore B., Symonova O., Price C.A., Topp C.N., Iyer-Pascuzzi A.S., Zurek P.R., Fang S., Harer J., Benfey P.N., Weitz J.S. GiA Roots: software for the high throughput analysis of plant root system architecture // BMC Plant Biol. 2012. V. 12. P. 116. https://doi.org/10.1186/1471-2229-12-116
Guan Q., Wu J., Yue X., Zhang Y., Zhu J. A nuclear calcium-sensing pathway is critical for gene regulation and salt stress tolerance in Arabidopsis // PLoS Genet. 2013. V. 9. P. e1003755. https://doi.org/10.1371/journal.pgen.1003755
Smolikova G., Kreslavski V., Shiroglazova O., Bilova T., Sharova E., Frolov A., Medvedev S. Photochemical activity changes accompanying the embryogenesis of pea (Pisum sativum) with yellow and green cotyledons // Funct. Plant Biol. 2018. V. 45. P. 228. https://doi.org/10.1071/FP16379
Rolfe S.A., Scholes J.D. Chlorophyll fluorescence imaging of plant-pathogen interactions // Protoplasma. 2010. V. 247. P. 163. https://doi.org/10.1007/s00709-010-0203-z
Arvidsson S., Pérez-Rodríguez P., Mueller-Roeber B. A growth phenotyping pipeline for Arabidopsis thaliana integrating image analysis and rosette area modeling for robust quantification of genotype effects // New Phytol. 2011. V. 191. P. 895. https://doi.org/10.1111/j.1469-8137.2011.03756.x
Clark R.T., MacCurdy R.B., Jung J.K., Shaff J.E., McCouch S.R., Aneshansley D.J., Kochian L.V. Three-dimensional root phenotyping with a novel imaging and software platform // Plant Physiol. 2011. V. 156. P. 455. https://doi.org/10.1104/pp.110.169102
Das A., Schneider H., Burridge J., Ascanio A.K.M., Wojciechowski T., Topp C.N., Lynch J.P., Weitz J.S., Bucksch A. Digital imaging of root traits (DIRT): a high-throughput computing and collaboration platform for field-based root phenomics // Plant Methods. 2015. V. 11. P. 51. https://doi.org/10.1186/s13007-015-0093-3
Joosen R.V.L., Arends D., Willems L.A.J., Ligterink W., Jansen R.C., Hilhorst H.W.M. Visualizing the genetic landscape of Arabidopsis seed performance // Plant Physiol. 2012. V. 158. P. 570. https://doi.org/10.1104/pp.111.186676
Tanabata T., Shibaya T., Hori K., Ebana K., Yano M. SmartGrain: high-throughput phenotyping software for measuring seed shape through image analysis // Plant Physiol. 2012. V. 160. P. 1871. https://doi.org/10.1104/pp.112.205120
Schöler F., Steinhage V. Automated 3D reconstruction of grape cluster architecture from sensor data for efficient phenotyping // Comput. Electron. Agric. 2015. V. 114. P. 163. https://doi.org/10.1016/j.compag.2015.04.001
Perez-Sanz F., Navarro P.J., Egea-Cortines M. Plant phenomics: an overview of image acquisition technologies and image data analysis algorithms // Gigascience. 2017. V. 6. P. 1. https://doi.org/10.1093/gigascience/gix092
Zhang J., Naik H.S., Assefa T., Sarkar S., Reddy R.V.C., Singh A., Ganapathysubramanian B., Singh A.K. Computer vision and machine learning for robust phenotyping in genome-wide studies // Sci. Rep. 2017. V. 7. P. 44048. https://doi.org/10.1038/srep44048
Czedik-Eysenberg A., Seitner S., Güldener U., Koemeda S., Jez J., Colombini M., Djamei A. The ‘PhenoBox’, a flexible, automated, open-source plant phenotyping solution // New Phytol. 2018. V. 219. P. 808. https://doi.org/10.1111/nph.15129
Sandhu D., Coleman Z., Atkinson T., Rai K.M., Mendu V. Genetics and physiology of the nuclearly inherited yellow foliar mutants in soybean // Front. Plant Sci. 2018. V. 9. P. 471. https://doi.org/10.3389/fpls.2018.00471
Coneva V., Frank M., de Luis Balaguer M.A., Li M., Sozzani R., Chitwood D.H. Genetic architecture and molecular networks underlying leaf thickness in desert-adapted tomato // Plant Physiol. 2017 V.175. P. 376. https://doi.org/10.1104/pp.17.00790
Weber J.F., Kunz C., Peteinatos G.G., Santel H.-J., Gerhards R. Utilization of chlorophyll fluorescence imaging technology to detect plant injury by herbicides in sugar beet and soybean // Weed Technol. 2017. V. 31. P. 523. https://doi.org/10.1017/wet.2017.22
Van De Velde K., Chandler P.M., Van Der Straeten D., Rohde A. Differential coupling of gibberellin responses by Rht-B1c suppressor alleles and Rht-B1b in wheat highlights a unique role for the DELLA N-terminus in dormancy // J. Exp. Bot. 2017. V. 68. P.443. https://doi.org/10.1093/jxb/erw471
Cabrera-Bosquet L., Fournier C., Brichet N., Welcker C., Suard B., Tardieu F. High-throughput estimation of incident light, light interception and radiation-use efficiency of thousands of plants in a phenotyping platform // New Phytol. 2016. V. 212. P. 269. https://doi.org/10.1111/nph.14027
Cendrero-Mateo M.P., Muller O., Albrecht H., Burkart A., Gatzke S., Janssen B., Keller B., Körber N., Kraska T., Matsubara S., Li J., Müller-Linow M., Pieruschka R., Pinto F., Rischbeck P. et al. Field phenotyping: challenges and opportunities // Terrestrial Ecosystem Research Infrastructures / Ed. Chabbi A., Loescher H.W. Boca Raton, FL: CRC Press, 2017. P. 53
Puglielli G., Redondo-Gómez S., Gratani L., Mateos-Naranjo E. Highlighting the differential role of leaf paraheliotropism in two Mediterranean Cistus species under drought stress and well-watered conditions // J. Plant Physiol. 2017. V. 213. P. 199. https://doi.org/10.1016/j.jplph.2017.02.015
Mishra A., Heyer A.G., Mishra K.B. Chlorophyll fluorescence emission can screen cold tolerance of cold acclimated Arabidopsis thaliana accessions // Plant Methods. 2014. V. 10. P. 38. https://doi.org/10.1186/1746-4811-10-38
Kämäräinen J., Huokko T., Kreula S., Jones P.R., Aro E.-M., Kallio P. Pyridine nucleotide transhydrogenase PntAB is essential for optimal growth and photosynthetic integrity under low-light mixotrophic conditions in Synechocystis sp. PCC 6803 // New Phytol. 2017. V. 214. P. 194. https://doi.org/10.1111/nph.14353
Ferroni L., Suorsa M., Aro E., Baldisserotto C., Pancaldi S. Light acclimation in the lycophyte Selaginella martensii depends on changes in the amount of photosystems and on the flexibility of the light-harvesting complex II antenna association with both photosystems // New Phytol. 2016. V. 211. P. 554. https://doi.org/10.1111/nph.13939
Humplík J.F., Lazár D., Fürst T., Husičková A., Hýbl M., Spíchal L. Automated integrative high-throughput phenotyping of plant shoots: a case study of the cold-tolerance of pea (Pisum sativum L.) // Plant Methods. 2015. V. 11. P. 20. https://doi.org/10.1186/s13007-015-0063-9
Straka L., Rittmann B.E. Growth kinetics and mathematical modeling of Synechocystis sp. PCC 6803 under flashing light // Biotechnol. Bioeng. 2018. V. 116. P. 469. https://doi.org/10.1002/bit.26862
Belgio E., Trsková E., Kotabová E., Ewe D., Prášil O., Kaňa R. High light acclimation of Chromera velia points to photoprotective NPQ // Photosynth. Res. 2018. V. 135. P. 263. https://doi.org/10.1007/s11120-017-0385-8
Skirycz A., Vandenbroucke K., Clauw P., Maleux K., De Meyer B., Dhondt S., Pucci A., Gonzalez N., Hoeberichts F., Tognetti V.B., Galbiati M., Tonelli C., Van Breusegem F., Vuylsteke M., Inzé D. Survival and growth of Arabidopsis plants given limited water are not equal // Nat. Biotechnol. 2011. V. 29. P. 212. https://doi.org/10.1038/nbt.1800
Clauw P., Coppens F., De Beuf K., Dhondt S., Van Daele T., Maleux K., Storme V., Clement L., Gonzalez N., Inzé D. Leaf responses to mild drought stress in natural variants of Arabidopsis // Plant Physiol. 2015. V. 167. P. 800. https://doi.org/10.1104/pp.114.254284
Tisné S., Serrand Y., Bach L., Gilbault E., Ben Ameur R., Balasse H., Voisin R., Bouchez D., Durand-Tardif M., Guerche P., Chareyron G., Da Rugna J., Camilleri C., Loudet O. Phenoscope: an automated large-scale phenotyping platform offering high spatial homogeneity // Plant J. 2013. V. 74. P. 534. https://doi.org/10.1111/tpj.12131
Manay S., Cremers D., Byung-Woo Hong, Yezzi A.J., Soatto S. Integral invariants for shape matching // IEEE Trans. Pattern Anal. Mach. Intell. 2006. V. 28. P. 1602. https://doi.org/10.1109/TPAMI.2006.208
Bau J., Cardé R.T. Simulation modeling to interpret the captures of moths in pheromone-baited traps used for surveillance of invasive species: the gypsy moth as a model case // J. Chem. Ecol. 2016. V. 42. P. 877. https://doi.org/10.1007/s10886-016-0765-0
Chu K.-W., Lee W.-S., Cheng C.-Y., Huang C.-F., Zhao F., Lee L.-S., Chen Y.-S., Lee C.-Y., Tsai M.-J. Demonstration of Lateral IGBTs in 4H-SiC // IEEE Electron Device Lett. 2013. V. 34. P. 286. https://doi.org/10.1109/LED.2012.2230240
Mirowski P.W., LeCun Y., Madhavan D., Kuzniecky R. Comparing SVM and convolutional networks for epileptic seizure prediction from intracranial EEG // IEEE Workshop on Machine Learning for Signal Processing. 2008. P. 244.
Wiederschain G.Y. Data mining techniques for the life sciences // Biochem. 2011. V. 76. P. 494. https://doi.org/10.1134/S0006297911040158
Yip K.Y., Cheng C., Gerstein M. Machine learning and genome annotation: a match meant to be? // Genome Biol. 2013. V. 14. P. 205. https://doi.org/10.1186/gb-2013-14-5-205
Sommer C., Gerlich D.W. Machine learning in cell biology – teaching computers to recognize phenotypes // J. Cell Sci. 2013. V. 126. P. 5529. https://doi.org/10.1242/jcs.123604
Guyon I., Weston J., Barnhill S., Vapnik V. Gene selection for cancer classification using support vector machines // Mach. Learn. 2002. V. 46. P. 389. https://doi.org/10.1023/A:1012487302797
Zacharaki E.I., Wang S., Chawla S., Soo Yoo D., Wolf R., Melhem E.R., Davatzikos C. Classification of brain tumor type and grade using MRI texture and shape in a machine learning scheme // Magn. Reson. Med. 2009. V. 62. P. 1609. https://doi.org/10.1002/mrm.22147
Gehan M.A., Fahlgren N., Abbasi A., Berry J.C., Callen S.T., Chavez L., Doust A.N., Feldman M.J., Gilbert K.B., Hodge J.G., Hoyer J.S., Lin A., Liu S., Lizárraga C., Lorence A. et al. PlantCV v2: Image analysis software for high-throughput plant phenotyping // PeerJ. 2017. V. 5. P. e4088. https://doi.org/10.7717/peerj.4088
Gutiérrez S., Fernández-Novales J., Diago M.P., Tardaguila J. On-the-go hyperspectral imaging under field conditions and machine learning for the classification of grapevine varieties // Front. Plant Sci. 2018. V. 9. P.1102. https://doi.org/10.3389/fpls.2018.01102
Cai Z., Fan Q., Feris R.S., Vasconcelos N. A unified multi-scale deep convolutional neural network for fast object detection // Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 2016. P. 354.
El-Bendary N., El Hariri E., Hassanien A.E., Badr A. Using machine learning techniques for evaluating tomato ripeness // Expert Syst. Appl. 2015. V. 42. P. 1892. https://doi.org/10.1016/j.eswa.2014.09.057
Ubbens J., Cieslak M., Prusinkiewicz P., Stavness I. The use of plant models in deep learning: an application to leaf counting in rosette plants // Plant Methods. 2018. V. 14. P. 6. https://doi.org/10.1186/s13007-018-0273-z
Taghavi Namin S., Esmaeilzadeh M., Najafi M., Brown T.B., Borevitz J.O. Deep phenotyping: deep learning for temporal phenotype/genotype classification // Plant Methods. 2018. V. 14. P. 66. https://doi.org/10.1186/s13007-018-0333-4
DeChant C., Wiesner-Hanks T., Chen S., Stewart E.L., Yosinski J., Gore M.A., Nelson R.J., Lipson H. Automated identification of northern leaf blight-infected maize plants from field imagery using deep learning // Phytopathology. 2017. V. 107. P. 1426. https://doi.org/10.1094/PHYTO-11-16-0417-R
Xiong X., Duan L., Liu L., Tu H., Yang P., Wu D., Chen G., Xiong L., Yang W., Liu Q. Panicle-SEG: a robust image segmentation method for rice panicles in the field based on deep learning and superpixel optimization // Plant Methods. 2017. V. 13. P. 104. https://doi.org/10.1186/s13007-017-0254-7
Jin S., Su Y., Gao S., Wu F., Hu T., Liu J., Li W., Wang D., Chen S., Jiang Y., Pang S., Guo Q. Deep learning: Individual maize segmentation from terrestrial lidar data using faster r-cnn and regional growth algorithms // Front. Plant Sci. 2018. V. 9. P. 866. https://doi.org/10.3389/fpls.2018.00866
Bai G., Jenkins S., Yuan W., Graef G.L., Ge Y. Field-based scoring of soybean iron deficiency chlorosis using rgb imaging and statistical learning // Front. Plant Sci. 2018. V. 9. P. 1002. https://doi.org/10.3389/fpls.2018.01002
Xu R., Li C., Paterson A.H., Jiang Y., Sun S., Robertson J.S. Aerial images and convolutional neural network for cotton bloom detection // Front. Plant Sci. 2018. V. 8. P. 2235. https://doi.org/10.3389/fpls.2017.02235
Grift T.E., Novais J., Bohn M. High-throughput phenotyping technology for maize roots // Biosyst. Eng. 2011. V. 110. P. 40. https://doi.org/10.1016/j.biosystemseng.2011.06.004
Schnaubelt D., Schulz P., Hannah M.A., Yocgo R.E., Foyer C.H. A phenomics approach to the analysis of the influence of glutathione on leaf area and abiotic stress tolerance in Arabidopsis thaliana // Front. Plant Sci. 2013. V. 4. P. 416. https://doi.org/10.3389/fpls.2013.00416
Weraduwage S.M., Chen J., Anozie F.C., Morales A., Weise S.E., Sharkey T.D. The relationship between leaf area growth and biomass accumulation in Arabidopsis thaliana // Front. Plant Sci. 2015. V. 6. P. 167. https://doi.org/10.3389/fpls.2015.00167
Chopin J., Laga H., Huang C.Y., Heuer S., Miklavcic S.J. RootAnalyzer: A cross-section image analysis tool for automated characterization of root cells and tissues // PLoS One. 2015. V. 10. P. e0137655. https://doi.org/10.1371/journal.pone.0137655
Montenegro-Johnson T.D., Stamm P., Strauss S., Topham A.T., Tsagris M., Wood A.T.A., Smith R.S., Bassel G.W. Digital single-cell analysis of plant organ development using 3DCellAtlas // Plant Cell. 2015. V. 27. P. 1018. https://doi.org/10.1105/tpc.15.00175
Duan L., Yang W., Huang C., Liu Q. A novel machine-vision-based facility for the automatic evaluation of yield-related traits in rice // Plant Methods. 2011. V. 7. P. 44. https://doi.org/10.1186/1746-4811-7-44
Aquino A., Millan B., Gaston D., Diago M.-P., Tardaguila J. VitisFlower®: Development and testing of a novel android-smartphone application for assessing the number of grapevine flowers per inflorescence using artificial vision techniques Techniques // Sensors. 2015. V. 15. P. 21204. https://doi.org/10.3390/s150921204
Virlet N., Costes E., Martinez S., Kelner J.-J., Regnard J.-L. Multispectral airborne imagery in the field reveals genetic determinisms of morphological and transpiration traits of an apple tree hybrid population in response to water deficit // J. Exp. Bot. 2015. V. 66. P. 5453. https://doi.org/10.1093/jxb/erv355
Golzarian M.R., Frick R.A., Rajendran K., Berger B., Roy S., Tester M., Lun D.S. Accurate inference of shoot biomass from high-throughput images of cereal plants // Plant Methods. 2011. V. 7. P. 2. https://doi.org/10.1186/1746-4811-7-2
Fujita S., Matsuo T., Ishiura M., Kikkawa M. High-throughput phenotyping of chlamydomonas swimming mutants based on nanoscale video analysis // Biophys. J. 2014. V. 107. P. 336. https://doi.org/10.1016/j.bpj.2014.05.033
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений