Физиология растений, 2020, T. 67, № 6, стр. 578-598
Молекулярный диалог корня и побега с участием регуляторных пептидов и его роль в системном контроле развития растений
М. А. Лебедева a, *, Я. С. Яшенкова a, И. Е. Додуева a, Л. А. Лутова a
a Санкт-Петербургский государственный университет,
кафедра генетики и биотехнологии
Санкт-Петербург, Россия
* E-mail: m.a.lebedeva@spbu.ru
Поступила в редакцию 03.04.2020
После доработки 08.05.2020
Принята к публикации 08.05.2020
Аннотация
Процессы развития растений являются удивительно пластичными и в значительной степени находятся под контролем факторов окружающей среды. Это во многом обеспечивается существованием у растений регуляторных механизмов, контролирующих развитие на уровне целого организма, то есть системно. У растений выявлены регуляторные пептиды, синтезируемые в корнях под действием различных факторов, воздействующих на корневую систему, таких как изменение уровня макроэлементов (прежде всего, азота и фосфора) в почве, воздействие симбиотических микроорганизмов (почвенных бактерий ризобий, грибов арбускулярной микоризы), дефицит воды в почве. Среди системно действующих пептидов наиболее изученными являются пептиды CLE (CLAVATA3/EMBRYO SURROUNDING REGION-related) и CEP (C-TERMINALLY ENCODED PEPTIDES), которые способны перемещаться по ксилеме из корней в побег и запускать ответные реакции, связываясь со специфическими рецепторами, работающими во флоэме листа. Рассмотрению роли этих двух групп пептидов в молекулярном диалоге корня и побега и будет уделено основное внимание в обзоре.
ВВЕДЕНИЕ
Наземные растения состоят из двух основных частей – побеговой системы, участвующей в ассимиляции углерода из воздуха в ходе фотосинтеза, и корневой системы, осуществляющей поглощение воды и минеральных веществ из почвы. Для выживания в меняющихся условиях окружающей среды процессы роста и развития двух этих систем должны быть четко скоординированы на уровне целого организма, то есть системно. Такая координация подразумевает наличие у растений специализированных сигнальных молекул, способных к дальнему транспорту и опосредующих коммуникацию между различными органами растения. Как показали исследования последних лет, в роли таких сигнальных молекул могут выступать регуляторные пептиды, также известные как пептидные гормоны растений.
К настоящему времени выделено более 30 классов пептидных фитогормонов с разнообразными функциями [1, 2]. В нашем обзоре мы сосредоточим внимание на пептидах семейств CLE (CLAVATA3/EMBRYO SURROUNDING REGION-related) [3, 4] и CEP (C-TERMINALLY ENCODED PEPTIDES) [5], относящихся к группе посттрансляционно модифицируемых пептидов (ПТМП) и играющих зачастую антагонистические роли в развитии растений. Все ПТМП синтезируются в виде протяженного белка-предшественника, из которого в результате протеолиза и посттрансляционных модификаций образуется активный высоко мобильный пептид длиной около 12–15 аминокислот [1], который может действовать как местно, так и транспортироваться в другие органы растения, выступая в качестве сигнала дальнего действия в системном контроле развития. Дальний транспорт регуляторных пептидов осуществляется по проводящим тканям: ряд синтезируемых в корнях пептидов способен перемещаться по ксилеме из корней в побег, где они связываются со своими рецепторами, располагающимися во флоэме листьев. Ответный сигнал, от листьев к корням, передается по флоэме также с участием мобильных сигналов различной молекулярной природы. Существование такого “молекулярного диалога” корня и побега, было предсказано более 30 лет назад при изучении мутантов сои, формирующих избыточное число азотфиксирующих клубеньков (так называемых “суперклубенькообразующих” мутантов) при симбиозе с почвенными бактериями ризобиями [6]. Более того, у таких мутантов процесс клубенькообразования не подавляется при высоких концентрациях нитрата, в отличие от растений дикого типа, у которых образование клубеньков происходит только при отсутствии доступных источников азота в почве. Эксперименты с прививками показали, что суперклубенькообразующий фенотип (формирование избыточного числа клубеньков на корнях) в ряде случаев определяется генотипом побега: растения, побеговая часть которых была взята от суперклубенькообразующих мутантных растений и привита на корневую часть растений дикого типа, образовывали избыточное число клубеньков [6]. Этот результат позволил исследователям предположить, что ген, ответственный за контроль численности симбиотических клубеньков, работает в побеге, и его продукт дистанционно ограничивает количество образующихся клубеньков, то есть в основе такой регуляции лежит дальний транспорт сигнальных молекул, и такая регуляция осуществляется системно. Дальний транспорт регуляторных молекул у растений также можно обнаружить и при изучении локализации транскриптов генов и их белковых продуктов: в ряде случаев отсутствие транскриптов и наличие белковых продуктов генов в одном органе указывает на существование дальнего транспорта соответствующих белков. Наряду с этим, для визуализации дальнего транспорта ряда молекул по проводящим тканям используют флуоресцентные метки, а также введение в растения радиоактивно меченых синтетических аналогов регуляторных молекул. Кроме того, возможно выявление регуляторных пептидов непосредственно в ксилемном соке.
Существование системного контроля с участием мобильных пептидов было показано и для другого типа симбиотических взаимодействий – для развития арбускулярной микоризы, а также для процессов поглощения нитрата и ответа на засуху. Тогда как формирование азотфиксирующих клубеньков является своего рода адаптивной реакцией на недостаток азота в почве, поскольку процесс азотификсации с участием клубеньковых бактерий обогощает растение азот-содержащими соединениями, формирование симбиоза с грибами арбускулярной микоризы позволяет растениям получать из почвы большее количество минеральных веществ, в особенности фосфора. Таким образом, факторы, воздействующие на корневую систему и запускающие ответные системные реакции с участием дальнего транспорта регуляторных молекул, связаны с реализацией основных функций корня – минерального питания и поглощения воды, которые определяют физиологический статус растения как целостного организма, и в связи с этим регулируются на системном уровне. Конкретные механизмы, лежащие в основе “молекулярного диалога” корня и побега в регуляции симбиотических реакций, минерального питания и ответа на засуху, будут подробно рассмотрены в данном обзоре.
ПЕПТИДЫ CLE КАК СИСТЕМНЫЕ РЕГУЛЯТОРЫ РАЗВИТИЯ РАСТЕНИЙ
CLE – пептидные фитогормоны, которые получили известность главным образом как регуляторы активности меристем. Так, CLAVATA3 (CLV3), первый из охарактеризованных и наиболее изученный представитель этой группы, регулирует поддержание пула стволовых клеток в апикальной меристеме побега (ПАМ) [4], CLE40 выполняет аналогичные функции в апикальной меристеме корня (КАМ) [7], а CLE41/44 – в камбии [8]. Помимо регуляции стволовых клеток в меристемах, пептиды CLE участвуют также в контроле дифференциации определенных типов клеток и тканей [9, 10], в раннем эмбриогенезе [11], во взаимодействии с симбионтами [12, 13] и патогенами [14].
Гены CLE кодируют небольшие белки, состоящие из 100–150 аминокислотных остатков. Белки CLE включают N-концевой сигнальный домен, центральную вариабельную часть, а также консервативный С-концевой CLE-домен, при этом у некоторых белков может быть несколько CLE-доменов [15]. CLE-домен, состоящий из 12–13 аминокислотных остатков, соответствует зрелому функциональному пептиду CLE, который вырезается из белка-предшественника в результате протеолиза. Кроме того, для активности пептидов CLE необходимы определенные посттрансляционные модификации: так, остаток пролина в седьмом положении CLE-домена гидроксилируется с образованием гидроксипролина, к которому затем присоединяются три остатка арабинозы [16]. Было показано, что триарабинозилирование по гидрокиспролину усиливает сродство пептида CLV3 к рецептору CLV1 [17].
Пептиды CLE являются мобильными компонентами регуляторных систем WOX-CLAVATA, в состав которых также входят рецепторы пептидов CLE – серин-треониновые протеинкиназы с лейцин богатыми повторами в лиганд-связывающем домене (представители семейства LRR-RLK, Leucine-Rich Repeats containing Receptor-Like Kinases) [18], индуцируемый ими слабоизученный сигнальный путь [19] и мишень действия этого пути – гены семейства WUSСHEL-RELATED HOMEOBOX (WOX), кодирующие одно из важнейших семейств транскрипционных факторов (ТФ) растений [20, 21].
Пептиды CLE разделяют на несколько групп на основании последовательностей CLE-домена: так, Strabala с соавт. [22] разделили 32 пептида CLE Arabidopsis thaliana на группы A-F. Согласно экспериментальным данным, пептиды CLE в пределах одной группы выполняют близкие функции могут замещать друг друга у мутантов [22]. Более простой классификацией является деление пептидов CLE на группы A и B в зависимости от их эффекта на активность апикальных и латеральных меристем [8, 23]: пептиды обширной группы А ингибируют активность ПАМ и КАМ, не влияя на активность камбия, тогда как пептиды небольшой группы В (называемые также Tracheary elements Differentiation Inhibitory Factors, TDIF) стимулируют активность камбия, не влияя на активность ПАМ и КАМ.
Свободно диффундируя между клетками, пептиды CLE, тем не менее, действуют обычно локально: в апикальных меристемах и камбии они передвигаются в пределах соседних клеток (ближний транспорт). Так, в ПАМ ген CLV3 экспрессируется в двух верхних слоях клеток центральной зоны меристемы (L1–L2), тогда как пептид CLV3 перемещается в слой L3, где подавляет экспрессию гена WUSCHEL (WUS), ответственного за поддержания пула недифференцированных стволовых клеток [4]. Основным рецептором пептида CLV3 в ПАМ является рецепторная протеинкиназа CLAVATA1 (CLV1) [24], также CLV3 может связываться с действующими в ПАМ рецепторами RECEPTOR PROTEINKINASE 1 и 2 (RPK1 и RPK2) [25], а также с рецепторами, работающими в разных типах меристем и дифференцированных тканей – киназами BARELY ANY MERISTEM 1-3 (BAM1-3) [26] и рецепторным комплексом CLAVATA2/CORYNE (CLV2/CRN) [27]. Способность только к ближнему транспорту характерна также для центрального регулятора КАМ – пептида CLE40, который синтезируется в колумелле и связывается в КАМ с рецепторами CLV1 и ACR4 (ARABIDOPSIS CRINKLY 4), регулируя экспрессию гена WOX5 [7], и пептидов TDIF (CLE41/44 и CLE42), которые синтезируются во флоэме и рецептируются сенсорной киназой TDIF RECEPTOR/ PHLOEM INTERCALATED WITH XYLEM (TDR/PXY) в камбии; мишенью их действия является ген WOX4 [28].
В то же время, показано, что некоторые пептиды CLE также способны к дальнему транспорту из корней в листья по ксилеме, опосредуя ответ растений на взаимодействие с симбиотическими организмами (ризобиями, грибами арбускулярной микоризы), реакции на изменение содержания минеральных веществ (азота, фосфора) в почве, а также реакции, вызванные засухой.
1.1. ПЕПТИДЫ CLE В СИСТЕМНОМ КОНТРОЛЕ РАЗВИТИЯ СИМБИОТИЧЕСКИХ КЛУБЕНЬКОВ И МИКОРИЗАЦИИ
Пептиды CLE как компоненты системы авторегуляции клубенькообразования (AON)
Пептиды CLE вовлечены в системный контроль развития симбиотических клубеньков у бобовых растений [12, 13]. Формирование и функционирование азотфиксирующих клубеньков требует больших энергетических ресурсов растения, в связи с чем эти процессы находится под системным контролем со стороны растения. У бобовых растений известна система авторегуляции клубенькообразования (AON, autoregulation of nodulation), ограничивающая формирование избыточных симбиотических клубеньков. Мобильными компонентами AON являются определенные пептиды CLE. Синтезируемые в развивающихся на корнях клубеньках они транспортируются по ксилеме из корней в побег, где происходит их взаимодействие со специфическими рецепторами – CLAVATA1 (CLV1)-подобными киназами, работающими во флоэме листа: SUPER NUMERIC NODULES (MtSUNN) у люцерны M. truncatula, HYPER NODULATION ABERRANT ROOT FORMATION1 (LjHAR1) у лядвенца японского (Lotus japonicus), PsSYM29 у гороха посевного (Pisum sativum) и NODULE AUTOREGULATION RECEPTOR KINASE (GmNARK) у сои культурной Glycine max [29, 30]. Вследствие этого взаимодействия происходит выработка ответного сигнала, блокирующего формирование новых клубеньков на корнях по механизму отрицательной обратной связи. Мутанты по генам CLV1-подобных киназ формируют избыточное число клубеньков (суперклубенькообразование) [29, 31–33], причем, как показали эксперименты с прививками, этот признак определяется генотипом побега: если побеговую часть мутантного растения привить на корневую часть растения дикого типа, то после инокуляции ризобиями корни полученных растений будут демонстрировать суперклубенькообразование.
Пептиды CLE, являющиеся индукторами AON, были охарактеризованы у различных модельных бобовых растений: MtCLE13 и MtCLE12 у M. truncatula [13], LjCLE-RS1, LjCLE-RS2 и LjCLE-RS3 (от CLE-ROOT SIGNAL) у L. japonicus [12], GmRIC1 GmRIC2 (от RHIZOBIUM INDUCED CLE) у G. max [30, 34]. Активируемые при клубенькообразовании гены CLE также были выявлены и у гороха P. sativum [35], однако их роль в AON к настоящему времени еще не была изучена.
Как было показано у M. truncatula, экспрессия генов MtCLE13 и MtCLE12, индуцирующих AON, активируется довольно рано в ответ на ризобиальную инфекцию: через 6 и 48 часов после инокуляции, соответственно [36]. Согласно данным по анализу активности промотора, у люцерны ген pMtCLE13:GUS экспрессируется на ранних этапах развития примордия в пролиферирующих клетках коры и перицикла, а в зрелом клубеньке его экспрессия сосредоточена в апикальной части клубенька: в клетках меристемы, в зоне ранней инфекции, а также в окончаниях проводящих пучков. Похожий паттерн экспрессии наблюдался и для гена MtCLE12, однако согласно данным по активности pMtCLE12:GUS, его экспрессия запускается позже, в клетках уже сформированного примордия клубенька [13].
Для нормального функционирования пептидов CLE, вовлеченных в клубенькообразование, в частности, для их связывания с рецепторами, важны посттрансляционные модификации, гидроксилирование и триарабинозилирование консервативных остатков пролина в CLE-домене: у люцерны и гороха мутанты по генам, кодирующие гидроксипролин O-арабинозилтрансферазы (rdn1, root determined nodulation1 у M. truncatula и nod3, nodulation 3, у P. sativum), характеризуются суперклубенькообразующим фенотипом, который, как было показано с помощью прививок, определяется корневой частью растения [37, 38]. При этом у люцерны гидроксипролин-O-арабинозилтрансфераза RDN1, вероятно, необходима для модификации пептида MtCLE12, но не MtCLE13 [39].
Возможность дальнего транспорта пептидов CLE, вовлеченных в AON, была показана на основании влияния сверхэкспрессии генов CLE на мутантов по генам, кодирующих CLV1-подобную киназу у ряда бобовых растений [12, 13]. Так, у растений дикого типа, сверхэкспрессия симбиоз-специфичных генов CLE (в частности, генов MtCLE12 и MtCLE13 люцерны, генов LjCLE-RS1 и LjCLE-RS2 у лядвенца) в трансгенных корнях, полученных в результате трансформации с помощью Agrobacterium rhizogenes, ингибировала клубенькообразование не только на трансгенных корнях со сверхэкспрессией CLE, но и на нетрансгенных корнях. В отличие от этого, у мутантов Mtsunn люцерны и Ljhar1 лядвенца по генам CLV1-подобной киназы, которые, как было показано с помощью прививок, функционируют в побеге, сверхэкспрессия симбиоз-специфичных генов CLE (MtCLE12/MtCLE13 и LjCLE-RS1/LjCLE-RS2, соответственно) не подавляла образование клубеньков [12, 13]. Эти данные указывают на то, что побеговые CLV1-подобные киназы необходимы для рецепции пептидов CLE, поступающих из корней. Непосредственное перемещение по ксилеме и связывание с CLV1-подобной киназой было показано для пептида лядвенца LjCLE-RS2 [40]. Так, пептид LjCLE-RS2 был идентифицирован в ксилемном соке, собранном в побеге. Химически синтезированный пептид LjCLE-RS2 с триарабинозилированным гидроксипролином был способен подавлять клубенькообразование при его нанесении непосредственно на семядольные листья лядвенца. Более того, радиоактивно меченый синтетический LjCLE-RS2 специфично связывался с белком LjHAR1, наработанным в клетках табака [40]. Таким образом, пептид LjCLE-RS2, синтезируемый в корнях, поступает по ксилеме в побег, где связывается со своим рецептором – CLV1-подобной киназой LjHAR1. Предполагается, что у других бобовых растений пептиды CLE, вовлеченные в AON, действуют аналогичным образом, связываясь с CLV1-подобной рецепторной киназой в побеге, запускают каскад реакций, подавляющих формирование клубеньков.
Наряду с CLV1-подобной киназой, в состав рецепторного комплекса, связывающего поступающие из корней пептиды CLE, по-видимому, могут входить и другие белки. С помощью метода бимолекулярной флуоресцентной комплементации было показано, что белки MtCRN и MtCLV2 M. truncatula взаимодействуют с CLV1-подобной киназой MtSUNN [41], при этом мутация в гене MtCLV2 также обуславливает суперклубенькообразующий фенотип растений [33]. Однако с помощью аналогичного подхода для белков LjCLV2 и LjHAR1 у лядвенца не было выявлено прямого взаимодействия из-за их различной внутриклеточной локализации: белок LjHAR1 локализован на плазмалемме, а LjCLV2 – на мембране эндоплазматического ретикулума [33]. Для L. japonicus было показано, что с белком LjHAR1 взаимодействует рецептор-подобная киназа KLAVIER (KLV), при этом мутация в гене LjKLV также приводит к суперклубенькообразованию, обусловленному побеговой частью растения [42]. Эти данные в совокупности указывают на то, что киназы CRN, CLV2 и KLV могут участвовать в рецепции CLE, вовлеченных в AON.
Активация CLV1-подобной киназы при рецепции AON-индуцирующих пептидов CLE приводит к выработке ответного сигнала в побеге, дистанционно ингибирующего формирование клубеньков на корнях (англ. SDI, shoot-derived inhibitor) [30]. По-видимому, такой сигнал является комплексным, и его действие связано с изменением биосинтеза и транспорта гормонов из побега в корень. Так, показано, что активация CLV1-подобной киназы приводит к снижению транспорта ауксина из побега в корень [43]; наряду с этим известно, что жасмоновая кислота и абсцизовая кислота (АБК) также участвуют в подавлении клубенькообразования при активации AON [44, 45]. Кроме того, в побеге при индукции AON активируется экспрессия генов биосинтеза цитокинина, и транспортируемый из побега цитокинин подавляет формирование клубеньков на корнях [46].
Среди факторов, действующих после активации CLV1-подобной киназы – белок с неизвестной функцией TOO MUCH LOVE (TML), содержащий kelch-повторы (мотивы, ответственные за белок-белковые взаимодействия) и F-бокс (мотив, также ответственный за белок-белковые взаимодействия и встречающийся, в частности, у субъединиц убиквитин-лигазных комплексов, опосредующих убиквитин-зависимую деградацию белков-мишеней) [47]. Мутация в гене TML приводит к суперклубенькообразующему фенотипу, определяемому корневой частью растения, что указывает на то, что белок TML функционирует в корне [48]. Белок TML локализуется в ядре, и как предполагается, он может быть вовлечен в подавление клубенькообразования за счет убиквитин-зависимой деградации ТФ, регулирующих развитие клубеньков [48]. Как было показано для лядвенца, транскрипты гена LjTML являются прямыми мишенями микроРНК miR2111 in vivo [49]. Уровень miR2111 снижается в ответ на инфицирование корней ризобиями, причем как в корнях, так и в листьях инокулированных ризобиями растений [49]. Сверхэкспрессия гена MIR2111-3, предшественника miR2111, приводила к гиперинфекции и к увеличению количества клубеньков, большинство из которых характеризовались меньшими размерами и недоразвитием по сравнению с клубеньками дикого типа. Согласно данным по локализации активности промотора, ген MIR2111-3 экспрессируется преимущественно во флоэме листа, тогда как его прямые мишени in vivo, транскрипты TML, выявлены только в корнях, инокулированных ризобиями, но не в листьях. Ряд экспериментов с использованием механического отделения побеговой части растений от корневой, а также обнаружение зрелой miR2111 во флоэмном соке, позволили Tsikou с соавт. сделать вывод о системном действии miR2111 в регуляции клубенькообразования: транспортируемая из побега miR2111 вызывает деградацию транскриптов TML в отсутствии ризобий, тогда как при развитии ризобиальной инфекции становится возможным накопление транскриптов TML вследствие снижения уровня miR2111, что в итоге приводит к подавлению клубенькообразования [49]. Кроме того, у мутантов лядвенца по гену Ljhar1 при ризобиальной инфекции уровень miR2111 был повышен как в листьях, так и в корнях, по сравнению с растениями дикого типа. Эти данные указывают на то, что CLV1-подобная киназа, кодируемая геном LjHAR1, опосредует снижение уровня miR2111 при ризобиальной инфекции, что приводит к увеличению транскриптов TML и как следствие – к подавлению клубенькообразования [49]. Аналогичные результаты, свидетельствующие о AON-зависимом снижении уровня miR2111 при ризобиальной инфекции, были получены и для люцерны: у мутанта Mtsunn, несущего мутацию в гене, ортологичном LjHAR1, не наблюдалось снижения уровня miR2111 в побегах и корнях при ризобиальной инфекции, а ингибирование действия miR2111 с помощью искусственной антагонистической микроРНК было достаточным для комплементации суперклубенькообразующего фенотипа у мутанта Mtsunn (т.е. приводило к снижению количества клубеньков до уровня, характерного для растений дикого типа) [50].
Совсем недавно были получены данные о том, что у люцерны уровень miR2111 также регулируется с участием рецепторной киназы COMPACT ROOT ARCHITECTURE 2 (CRA2) – рецептора пептидов CEP в побеге [50]. Таким образом, микроРНК miR2111, по-видимому, является системным регулятором клубенькообразования, интегрирующим сигналы от рецепторов двух групп пептидных гормонов – CLE и СЕР (см. ниже), которые оказывают антагонистическое действие на развитие симбиотических клубеньков у бобовых растений.
Было показано, что за активацию экспрессии генов CLE при клубенькообразовании ответственен ТФ NODULE INCEPTION (NIN) – ключевой регулятор развития симбиотических клубеньков [51]. Как было показано для лядвенца с помощью метода иммунопреципитации хроматина (ChIP-seq), ТФ LjNIN напрямую связывается с промоторами генов LjCLE-RS1 и LjCLE-RS2, активируя их экспрессию [51]. При этом экспрессия как самого гена NIN, так и мишеней NIN – генов CLE, ингибирующих клубенькообразование, активируется при воздействии цитокинина [51, 52], на основании чего предполагают, что именно ТФ NIN опосредует активацию CLE в ответ на действие цитокинина [51]. У лядвенца сверэкспрессия LjNIN в трансгенных корнях, полученных в результате трансформации с помощью A. rhizogenes, приводила к системному подавлению клубенькообразования как на трансгенных корнях, сверхэкспрессирующих NIN, так и на нетрансгенных корнях, при этом такое подавление клубенькообразование сопровождалось увеличением уровня экспрессии генов LjCLE-RS1 и LjCLE-RS2. Системное ингибирование клубенькообразования при сверхэкспрессии NIN отсутствовало у мутантов Ljhar1 и Ljtml-1, что указывает на то, что системное подавление клубенькообразования с участием NIN опосредовано AON. Аналогично, сверхэкспрессия NIN системным образом подавляла экспрессию самого гена NIN, что было показано на основании активности промотора, слитого с репортерным геном GUS, при этом такое подавление отсутствовало у мутантов Ljhar1. Эти данные указывают на то, что AON негативно регулирует экспрессию NIN на транскрипционном уровне, что в свою очередь, приводит к подавлению экспрессии генов-мишеней ТФ NIN, ответственных за активацию кортикальных клеточных делений в ходе развития примордиев клубеньков, и к подавлению клубенькообразования [51].
Таким образом, ТФ NIN способствует запуску AON и в тоже время является мишенью системной регуляции клубенькообразования, пептиды CLE, синтезированные под влиянием ТФ NIN, связываясь с рецепторными CLV1-подобными киназами в побеге, способствуют выработке ответного сигнала, приводящего к подавлению формирования клубеньков, в том числе за счет подавления экспрессии NIN. В этом смысле регуляторный модуль NIN-CLE-СLV1-подобная киназа схож c системой WUS-CLV3-CLV1, работающей в апикальной меристеме побега, где ТФ WUS напрямую запускает экспрессию гена CLV3, продукт которого, в свою очередь, подавляет экспрессию WUS, действуя через рецепторные комплексы, включающие CLV1 [4].
Роль пептидов CLE в нитрат-опосредованном ингибировании клубенькообразования
Процесс клубенькообразования требует затрат энергетических ресурсов со стороны растения. В том случае, когда в почве содержатся доступные соединения азота, растение преимущественно будет использовать именно их для восполнения собственного минерального статуса. В связи с этим, в процессе эволюции у растений сформировался механизм, позволяющий осуществлять контроль развития клубеньков в зависимости от доступности азотсодержащих компонентов в почве, в особенности нитрата.
Ряд пептидов CLE, активируемых при симбиозе и запускающих AON, также индуцируется при обработке нитратом, и таким образом, опосредует нитрат-зависимое подавление развития симбиотических клубеньков [12, 53]. Так, у L. japonicus экспрессия двух из трех генов CLE, индуцируемых при симбиозе с ризобиями, LjCLE-RS2 и LjCLE-RS3, также индуцируется в ответ на обработку нитратом [12].
У сои Glycine max были выявлены 2 группы пептидов CLE, различающиеся по механизму активации экспрессии их генов. Наряду с индуцируемыми ризобиями пептидами группы RIC, GmRIC1 и GmRIC2, которые задействованы в системном контроле клубенькообразования с участием работающей в побеге CLV1-подобной киназы GmNARK, у сои также были выявлены нитрат-индуцируемые CLE, относящиеся к группе NIC (от англ. NITRATE INDUCED CLE) [54]. Экспрессия генов GmNIC1 и GmNIC2 активируется в ответ на присутствие нитрата в почве и приводит к ингибированию формирования клубеньков. В экспериментах с реципрокными прививками было показано, что у растений, корневая часть которых была получена от дикого типа, в значительной степени было снижено количество клубеньков при обработке нитратом, по сравнению с растениями, корневая часть которых имела генотип Gmnark [54]. Этот результат указывает на то, за нитратное ингибирование клубенькообразования ответственна рецепторная киназа GmNARK, функционирующая в корне. В соответствии с этим, в отличие от системно действующих пептидов RIC, пептиды GmNIC1 и GmNIC2 обеспечивают локальный ответ растения на обработку нитратом, поскольку у композитных растений, содержащих как трансгенные корни со сверхэкспрессией GmNIC1 и GmNIC2, полученные в результате трансформации с помощью A. rhizogenes, так и нетрансгенные корни, сверхэкспрессия генов GmNIC1 и GmNIC2 в трансгенных корнях не подавляла клубенькообразование на нетрансгенных корнях. Вместе с этим, сверхэкспрессия GmNIC1 и GmNIC2 подавляла клубенькообразование на корнях дикого типа, но не у мутантов по гену GmNARK [54, 55]. Эти результаты свидетельствуют о том, что пептиды GmNIC1 и GmNIC2 подавляют клубенькообразование локально через CLV1-подобную киназу GmNARK, функционирующую в корнях [54, 55]. Таким образом, в отличие от лядвенца, нитрат-зависимый механизм подавления развития симбиотических клубеньков у сои действует локально с участием корневой CLV1-подобной киназы. Какие конкретно механизмы лежат в основе работы корневой рецепции CLE с участием GmNARK и почему, в отличие от нитрат-активируемых пептидов GmNIC, активируемые ризобиями пептиды GmRIC действуют не через корневой рецептор, а системно, через работающий в побеге GmNARK, еще предстоит выяснить.
У лядвенца активация экспрессии нитрат-регулируемых CLE, в частности LjCLE-RS2, осуществляется с участием ТФ LjNRSYM1 (от англ. NITRATE UNRESPONSIVE SYMBIOSIS 1), относящегося к семейству NIN-подобных белков (англ. NLP от NIN-like proteins). ТФ NLP были описаны и у других растений, в частности у A. thaliana, как регуляторы ответа на нитрат [56], в их составе выделяют консервативный ДНК-связывающий домен RWP-RK, с помощью которого они взаимодействуют с нитрат-регулируемыми элементами NRE (nitrate response element) в регуляторных последовательностях нитрат-индуцируемых генов, а также домены PB1, обеспечивающие белок-белковые взаимодействия [57]. Мутант лядвенца Ljnrsym1 способен образовывать клубеньки при высоких концентрациях нитрата [58], что свидетельствует о ведущей роли гена LjNRSYM1 в нитрат-зависимом ингибировании клубенькообразования.
Однако, как ни странно, уровень экспрессии LjNRSYM1 не менялся при нитратной обработке и инокуляции ризобиями. Предполагается, что активность ТФ NRSYM1 регулируется за счет его посттрансляционных модификаций, индуцируемых обработкой растения нитратом [58]. Обнаружено, что к действию нитрата чувствителен N-концевой домен белков NLP: на примере белков NLP арабидопсиса показано, что обработка нитратом влияет на внутриклеточную локализацию белка NLP и стимулирует его накопление в ядре, определяемое фосфорилированием аминокислотного остатка в N-концевом домене [59] (см. раздел 1.2.). Для белка LjNRSYM1 также был описан схожий нитрат-опосредованный транспорт из цитоплазмы в ядро при воздействии нитрата [58]. Способность менять конформацию и перемещаться в ядро под действием нитрата отличает ТФ NLP от белков NIN, регулирующих развитие симбиотических клубеньков, которые в ходе эволюции утратили способность реагировать на нитрат [60]. Как было показано для белка LjNLP1 у лядвенца, он связывается NRE в промоторах генов-мишеней, структурно схожими с сайтами связывания белка LjNIN (NIN-binding sites, NBS) [60]. Белок LjNRSYM1 также напрямую связывается с NBS/NRE в промоторе гена LjCLE-RS2 [58].
Роль ТФ NLP в регуляции экспрессии нитрат-индуцируемых генов также была изучена у M. truncatula [61]. Показано, что белок MtNLP1, необходимый для активации нитрат-регулируемых генов, в частности гена нитрит-редуктазы NIR1 и гена NRT2.1, кодирующего нитратный транспортер, способен связываться с ТФ MtNIN. Авторы выдвигают модель, согласно которой ТФ NLP в неактивном состоянии преимущественно локализуется в цитоплазме и в ответ на сигнал о повышении концентрации нитрата в среде транспортируется в ядро, где, формируя гетеродимер с NIN, блокирует его действие, препятствуя активации экспрессии генов-мишеней и тем самым ингибируя формирование клубеньков [61].
Пептиды CLE как системные регуляторы формирования микоризы
Многие наземные растения способны образовывать симбиоз с грибами арбускулярной микоризы, и благодаря формированию микоризы растения способны получать большее количество минеральных веществ из почвы, в особенности фосфора. Совсем недавно у бобовых растений были выявлены пептиды CLE, активируемые при микоризации, а также при действии фосфата, которые системно подавляют дальнейшую микоризацию с участием тех же рецепторов, которые задействованы в системном контроле численности симбиотических клубеньков [62]. Так, у люцерны в результате анализа экспрессии генов, которые активируются при формировании симбиоза с микоризным грибом Rhizophagus irregularis, было выявлено повышение уровней экспрессии генов M-tCLE16, MtCLE45 и MtCLE53. Кроме того, было показано, что в условиях повышенного содержания фосфата в среде (2 мМ) активируется экспрессия генов MtCLE32 и MtCLE33. Сверхэкспрессия генов MtCLE33 и MtCLE53 приводит к подавлению микоризации в результате снижения уровня стриголактонов – активаторов развития арбускулярной микоризы, что указывает на то, что гены биосинтеза стриголактонов являются одной из мишеней пептидов CLE [62].
Интересно, что, в отличие от растений дикого типа, у мутантов по гену MtSUNN – ключевому компоненту AON, сверхэкспрессия генов MtCLE33 и MtCLE53 не приводила к снижению уровня микоризации и подавлению биосинтеза стриголактонов [62]. Таким образом, функции рецепторной киназы MtSUNN не ограничиваются только контролем бобово-ризобиального симбиоза, но также связаны с регуляцией другого типа симбиоза – арбускулярной микоризы. При этом в случае бобово-ризобиального симбиоза и арбускулярной микоризы в активации рецептора MtSUNN, задействованы разные пептиды CLE (MtCLE12/MtCLE13 и MtCLE33/MtCLE53, соответственно), и механизмы подавления симбиотических реакций, запускающиеся рецепторной киназой MtSUNN, также различаются при разных симбиотических взаимодействиях. Более того, показано, что мутация в гене MtSUNN также приводит к нарушениям процессов поглощения нитрата и его транспорта в побег, что было показано на основании транскриптомного анализа мутанта Mtsunn, а также на основании снижения содержания нитрата в побеге у мутанта Mtsunn, по сравнению с растениями дикого типа через 24 часа после добавления нитрата в среду [63]. Вопрос о том, какие именно факторы в каждом случае определяют специфичность реакций, активируемых центральным регулятором, киназой MtSUNN, остается пока неразрешенным.
1.2. НИТРАТ-ЗАВИСИМАЯ РЕГУЛЯЦИИ РАЗВИТИЯ БОКОВОГО КОРНЯ С УЧАСТИЕМ CLE И ДРУГИЕ НИТРАТ-РЕГУЛИРУЕМЫЕ ПРОЦЕССЫ РАЗВИТИЯ У РАСТЕНИЙ
Пептиды CLE опосредуют нитрат-зависимые реакции у растений не только при симбиозе, но также в других процессах развития. Известно, что нитрат оказывает влияние на формирование корневой системы [64, 65], регулирует активность меристемы побега и время цветения [66]. Так, у бобовых растений мутации в гене CLV1-подобной киназы, задействованной в AON, помимо суперклубенькообразования, также приводят к нарушению архитектуры корня: у мутантов по гену LjHar1 лядвенца наблюдается укорочение главного корня и увеличение количества боковых корней [67]. У арабидопсиса участие в ответе на нитрат предполагается для рецепторной киназы CLV1 – центрального регулятора развития ПАМ: у мутантов clv1, в отличие от растений дикого типа, при недостатке нитрата не наблюдается подавление развития боковых корней [64].
У арабидопсиса также были выявлены нитрат-регулируемые пептиды CLE, вовлеченные в контроль развития боковых корней [64]. Так, Araya с соавт. показали, что гены AtCLE1/3/4/7 у арабидопсиса активируются при недостатке нитрата и в основном экспрессируются в клетках перицикла; их сверхэкспрессия приводит к подавлению развития боковых корней. Сверхэкспрессия AtCLE3 приводила к подавлению роста боковых корней у растений дикого типа, однако у мутантов Atclv1 его ингибирующее действие на рост боковых корней не проявлялось [64]. На основании полученных результатов авторы предположили, что пептиды AtCLE1/3/4/7 через рецепторную киназу AtCLV1 подавляют рост боковых корней при недостатке азота. Поскольку белок AtCLV1 локализован во флоэме корня, по предположению авторов, регуляция роста боковых корней, опосредованная пептидами AtCLE, осуществляется локально с участием работающей в корнях рецепторной киназы AtCLV1, возможный дальний транспорт пептидов AtCLE в данной работе не изучался [64].
Вместе с этим, у арабидопсиса выявлены NIN-подобные ТФ NLP, AtNLP6 и AtNLP7, которые, как и их гомологи у бобовых растений, ответственны за активацию нитрат-регулируемых генов. Так, было показано, что AtNLP7 при наличии нитрата в среде транспортируется в ядро, где связывается c нитрат-регулируемыми элементами (NRE) в последовательностях генов-мишеней [57]. Механизм, обеспечивающий внутриядерное удержание NLP7 в этих условиях, связан с фосфорилированием AtNLP7 по остатку Ser205 in vivo за счет Ca2+-сенсорных протеинкиназ (CPKs) CPK10, CPK30 и CPK32 [68]. Нитрат-зависимая активация экспрессии c участием ТФ AtNLP6/7 описана для ряда генов, контролирующих разные аспекты развития растений: в частности, для гена циклина класса В CYCLINB1;1 [69] и ряда генов, ответственных за цветение [66].
Помимо пептидов CLE, роль молекул дальнего транспорта, синтезирующихся под влиянием нитрата, играют цитокинины. Известно, что в корне арабидопсиса уровень экспрессии генов IPT, контролирующих первую стадию биосинтеза цитокининов из аденозинфосфатов, находится в прямой зависимости от концентрации нитрата в среде [70]. Было показано, что дальний транспорт синтезированных в корне цитокининов в надземную часть растения определяет нитрат-зависимое расширение зоны экспрессии гена WUS, которое приводит к увеличению размера ПАМ [71].
1.3. ПЕПТИДЫ CLE КАК РЕГУЛЯТОРЫ ОТВЕТА НА ЗАСУХУ
При недостатке воды в почве растения испытывают стресс и реагируют на него закрытием устьиц, что снижает потерю влаги и помогает растениям переносить засушливый период. Эта реакция индуцируется АБК, синтезируемой в листьях при засухе. Как оказалось, биосинтез АБК в листьях регулируется с участием пептидов CLE, синтезируемых в корнях. Так, при оценке способности различных синтетических пептидов CLE арабидопсиса индуцировать биосинтез АБК в листьях было обнаружено, что обработка корней пептидом CLE25 стимулировала экспрессию гена AtNCED3 (NINE-CIS-EPOXYCAROTENOID DIOXYGENASE 3), кодирующего ключевой фермент биосинтеза АБК при засухе, а также как следствие – реакции закрытия устьиц в листьях. Способность пептида AtCLE25 транспортироваться из корня в побег была подтверждена с использованием радиоактивно меченого синтетического AtCLE25, накопление которого наблюдали в листьях после обработки корней. Активность промотора гена AtCLE25 наблюдалась в боковых корнях, в кончиках корней, в проводящих тканях листьев, а также в прокамбии корня. У мутантов по гену AtCLE25, полученных с помощью CRISPR-Cas9-опосредованного редактирования, а также у растений с РНК-интерференцией гена AtCLE25 при воздействии засухи не наблюдалась активация экспрессии гена AtNCED3, и как следствие, не происходило накопление АБК в листьях, характерное для растений дикого типа при дегидратации. Кроме того, мутанты по гену CLE25 демонстрировали большие потери воды и были более чувствительны к засухе, по сравнению с растениями дикого типа. Для выявления возможных рецепторов пептида AtCLE25 авторы оценили эффект его воздействия на серию мутантов с нарушениями в генах, кодирующих рецепторы пептидов CLE, и обнаружили, что у двойного мутанта bam1-5 bam3-3, также характеризующегося повышенной чувствительностью к засухе, не наблюдалась активация биосинтеза АБК в листьях при обработке пептидом AtCLE25. При этом, как было показано с помощью прививок, именно мутантный генотип побега определял чувствительность растения к воздействию AtCLE25. На основании этих данных авторы предположили, что рецепторами AtCLE25 в листьях могут быть рецепторные киназы BAM1 и BAM3 [72].
В недавнем исследовании также было показано, что наряду с его участием в реакциях в ответ на засуху, пептид AtCLE25 необходим для развития флоэмы: мутация в гене AtCLE25 приводила к задержке дифференцировки клеток флоэмы, при этом подавление функции AtCLE25 с помощью “дефектного” пептида, несущего замену консервативного глицина в 6 положении CLE-домена на треонин (G6T), также нарушало развитие флоэмы и обуславливало формирование укороченного корня [73]. С помощью анализа мутантов, не чувствительных к действию пептида AtCLE25, было показано, что вероятным рецептором AtCLE25 в корне является рецепторная киназа СLERK (CLE-RESISTANT RECEPTOR KINASE), формирующая комплекс с CLV2 [73]. Таким образом, наряду с участием в дальнем транспорте и системном контроле реакций на засуху через рецепторы BAM1/BAM3 в листе, пептид AtCLE25 также действует локально в корне через рецепторный комплекс СLERK/CLV2, регулируя формирование флоэмы. При этом, по-видимому, для дальнего транспорта AtCLE25 по ксилеме в побег в ходе ответа на засуху, описанного в работе Takahashi с соавт. [72], необходимо перемещение этого пептида из флоэмы в клетки ксилемы.
2. ПЕПТИДЫ CEP КАК СИСТЕМНЫЕ РЕГУЛЯТОРЫ РАЗВИТИЯ
У Arabidopsis пептиды CEP были идентифицированы in silico при скрининге белковых семейств, содержащих структуру, характерную для ПТМП – короткий консервативный домен вблизи С-конца. Созревание СЕР, как и пептидов CLE, связано с протеолизом и посттрансляционными модификациями в консервативном домене (гидроксилированием остатков пролина) [5]. Зрелые пептиды СЕР состоят из 15 аминокислотных остатков и содержат 2–4 остатка гидроксипролина. В отличие от CLE, которые обнаружены даже у зеленых водорослей [74], пептиды СЕР найдены только у семенных растений: так, у Arabidopsis выявлено 15 генов CEP [75].
Функции пептидов СЕР связаны, по-видимому, в основном с развитием корневой системы, в том числе – с контролем развития боковых корней и клубеньков и ответа на абиотический стресс, в особенности на дефицит азота, при этом они способны действовать системно на дальних расстояниях от места своего синтеза (см. ниже).
Регуляция экспрессии CEP с участием нитрата и других факторов
У арабидопсиса гены семейства CEP экспрессируются преимущественно в корне и стебле, активируясь при недостатке азота в среде и при действии других стрессовых факторов, таких, как повышенное содержание хлорида натрия в среде и избыток углекислого газа. Для гена AtCEP3 арабидопсиса была показана наиболее сильная активация в корнях при недостатке азота в среде. Более того, мутант по гену AtCEP3 характеризовался более развитой корневой системой при действии различных стрессовых факторов, в частности, при недостатке азота и при засолении, по сравнению с растениями дикого типа; тогда как при росте на стандартной среде его отличия от дикого типа не проявлялись [76]. Эти данные позволяют предположить роль пептидов СЕР, в особенности AtСЕР3, как негативных регуляторов развития корневой системы в стрессовых условиях.
2.1. РЕЦЕПТОРЫ CEP – СИСТЕМНЫЕ РЕГУЛЯТОРЫ ОТВЕТА НА НЕДОСТАТОК АЗОТА
В работе Tabata с соавт. были выявлены рецепторы пептидов CEP: радиоактивно меченый синтетический пептид AtCEP1 арабидопсиса специфично связывался in vitro с киназами семейства LRR-RLK AtCEPR1 и AtCEPR2 (CEP RECEPTOR), при этом мутанты по генам AtCEPR1/2 были нечувствительны к обработке пептидом CEP1 при оценке его влияния на рост корня [77]. Активность промоторов AtCEPR1 и AtCEPR2 наблюдалась в проводящей системе листьев, главного корня и боковых корней. Двойные мутанты cerp1 cerp2 характеризовались увеличением длины боковых корней, при этом схожий фенотип, но в меньшей степени проявления, наблюдался и у одиночного мутанта cerp1, но не у мутанта cerp2, на основании чего авторы предположили, что именно CEPR1 играет определяющую роль в рецепции AtCEP1 и запуске последующих реакций, приводящих к подавлению роста корня. Помимо этого, у мутантов cerp1 cerp2 также наблюдался ряд признаков, характерных для растений, испытывающих голодание по азоту: накопление антоциана, бледно-зеленый цвет листьев, укорочение побега, в связи с чем авторы предположили, что AtCEP1 и его рецепторы могут регулировать экспрессию генов, вовлеченных в поглощение и накопление азота в растениях. И действительно, у мутантов cerp1 cerp2 была значительно снижена экспрессия гена нитратного транспортера NRT2.1, и в меньшей степени генов NRT3.1 и NRT1.1. Обработка экзогенным AtCEP1 приводила к активации экспрессии гена NRT2.1 даже в условиях высокого содержания азота у растений дикого типа, но не у мутантов по гену AtCEPR1. Таким образом, пептид AtCEP1 опосредует ответ на недостаток азота в среде, действуя через рецептор AtCERP1 [77].
Также ранее с использованием системы “split root”, при которой корневую систему растения разделяют на две части и помещают в разные условия (например, одну часть корневой системы – в среду с высоким содержанием азота, другую – с низким содержанием азота), было показано, что в гетерогенной по содержанию азота среде, голодание по азоту в одной части корневой системы может приводить к стимуляции развития боковых корней и к усилению экспрессии генов нитратных транспортеров в той части корневой системы, которая находится в среде с высоким содержанием азота, для компенсации недостатка по азоту у растения в целом [78]. Как оказалось, такая системная реакция опосредована действием пептидов CEP и их рецепторов [77]. Так, в отличие от дикого типа, у мутантов cepr1 cepr2 не наблюдалось системной активации экспрессии генов нитратных транспортеров NRT2.1, NRT3.1 и NRT1.1 в корнях, находящихся в среде высоким содержанием азота (с добавлением 10 мМ ${\text{NO}}_{3}^{ - }$), при использовании системы “split root”. Более того, в среде c высоким содержанием азота при обработке одной части корневой системы синтетическим пептидом AtСEP1 наблюдалась активация экспрессии гена NRT2.1 как в обработанных, так и в не обработанных AtCEP1 корнях, что указывает на то, что AtCEP1 опосредует системную активацию экспрессии генов нитратных транспортеров.
Поскольку гены AtCEPR1 и AtCEPR2 экспрессируются как в корне, так и в надземной части растений, возник вопрос о том, действует ли пептид AtCEP1 на транспорт нитратов местно или же транспортируется в побег. Было показано, что системное действие AtCEP1 осуществляется через рецепторы, работающие в побеге, поскольку в эксперименте с прививками побеговой части от двойных мутантов cepr1-1 cepr2-1 на корневую часть растений дикого типа обработка части корней пептидом AtCEP1 в системе “split root” не вызывала активации экспрессии NRT2.1 ни в одной из частей корневой системы [77].
Какие же факторы опосредуют передачу сигнала от рецепторов CEP из побега в корень? В работе Ohkubo с соавт. было показано, что у арабидопсиса при обработке корней пептидом AtCEP1 в клетках флоэмы листа происходит зависимая от AtCEPR1 активация экспрессии генов CEP DOWNSTREAM 1 (CEPD1) и CEPD2, кодирующих полипептиды семейства 10 глутаредоксинов III класса [79]. Анализ сверхэкспрессии генов AtC-EPD1 и AtCEPD2, а также двойных мутантов cepd1 cepd2, показал, что AtCEPD1 и AtCEPD2 ответственны за системную активацию в корнях экспрессии гена нитратного транспортера NRT2.1, но не двух других генов, NRT3.1 и NRT1.1, экспрессия которых, как было показано ранее, активируется при обработке пептидом AtCEP1 [77, 79]. Активность промоторов AtCEPD1 и AtCEPD2 наблюдалась во флоэме листа, при этом в корнях транскриптов генов AtCEPD1 и AtCEPD2 не было выявлено даже после обработки пептидом CEP1. Вместе с этим, гибридный белок GFP-AtCEPD1 локализовался в проводящей системе корня, в клетках флоэмы, а также в клетках эндодермы, и его количество увеличивалось как при обработке пептидом AtCEP1, так и при недостатке азота в среде [77]. Эти данные свидетельствуют о том, что полипептиды AtCEPD выступают в роли мобильных сигнальных молекул, транспортирующихся по флоэме из побега в корень в ответ на активацию рецептора AtCEPR1 в побеге. Этот вывод был дополнительно подтвержден экспериментами с прививками, где гибридный белок GFP-AtCEPD1 выявляли в корневой части растения, несущей мутации в генах AtCEPD (cepd1-1 cepd2-1), в случае, когда в побеговой части растения экспрессировался гибридный ген GFP-AtCEPD1 [79].
Кроме того, совсем недавно Ota c соавт. обнаружили у арабидопсиса еще два гена, имеющих большой процент сходства с AtCEPD1, названных AtCEPD-like 1 (CEPDL1) и CEPDL2 [80]. Их сверхэкспрессия также приводила к активации экспрессии NRT2.1 в корнях, даже в среде с высоким содержанием азота (10 мМ NH4+, 10 мМ ${\text{NO}}_{3}^{ - }$), при этом анализ транскриптома растений со сверхэкспрессией AtCEPDL2 показал, что AtCEPDL2 стимулирует экспрессию генов, кодирующих так называемые нитратные транспортеры с высокой аффинностью (high-affinity nitrate uptake), такие как NRT2.1, NRT2.2, NRT2.4, NRT3.1, а также NRT1.5, осуществляющий загрузку нитрата в ксилему для его транспорта из корня в побег [80]. Как и его гомологи AtCEPD1 и AtCEPD2, белок AtCEPDL2 транспортируется из побега в корень по флоэме, что было выявлено на основании локализации активности промоторов и оценки флуоресценции гибридного белка GFP-AtCEPDL2. Однако в отличие от AtCEPD1 и AtCEPD2, экспрессия AtCEPDL2 в побеге активировалась при недостатке азота независимо от активности рецептора AtCEPR1 и даже выявлялась в побегах, отделенных от корней, при низких концентрациях азота, на основании чего авторы предположили, что экспрессия AtCEPDL2 регулируется содержанием азота в побеге [80].
Таким образом, у растений существует сложная регуляция системы, ответственной за поглощения нитрата в корне, индукция которой осуществляется параллельно двумя путями: при недостатке содержания азота в побеге – с участием AtCEPDL2, а при недостатке азота в почве – с участием синтезирующихся в корнях мобильных пептидов AtCEP и их рецепторов AtCEPR, работающих в побеге [80].
Пептиды CEP как локальные регуляторы развития боковых корней
Наряду с системными реакциями на недостаток азота, пептиды CEP, как предполагается, участвуют также в локальной регуляции развития боковых корней. Так, у арабидопсиса экспрессия гена AtCEP5 подавляется ауксином и ассоциирована с ранними этапами развития бокового корня: AtCEP5 экспрессируется в клетках перицикла рядом с флоэмным полюсом, т.е. рядом с сайтами инициации бокового корня, но не в них самих (у арабидопсиса инициация развития бокового корня происходит в клетках перицикла напротив ксилемного полюса). В этих же клетках, согласно данным Roberts с соавт. [81], экспрессируется ген AtCEPR1, который кодирует предполагаемый рецептор пептида AtCEP5, поскольку мутанты cepr1 были нечувствительны к экзогенной обработке синтетическим пептидом AtCEP5. На основании данных по сверхэкспрессии и РНК-интерференции гена AtCEP5, а также результатов экзогенной обработки растений пептидом AtCEP5, показано, что AtCEP5 подавляет развитие боковых корней). У мутанта по гену AtCEPR1 (также известному как XIP1, от англ. XYLEM INTERMIXED WITH PHLOEM 1) наблюдалось снижение числа боковых корней, на основании чего авторы предположили, что пептид AtCEP5 локально подавляет развитие боковых корней, негативно регулируя работу рецептора AtCEPR1/XIP1 – позитивного регулятора их развития [81]. Роль пептидов CEP в локальной регуляции развития боковых корней была также показана для люцерны: cверхэкспрессия гена MtCEP1 в культуре бородатых корней в отсутствии побега подавляла развитие боковых корней [82].
2.2. РОЛЬ CEP В КЛУБЕНЬКООБРАЗОВАНИИ
Азотфиксирующие клубеньки формируются на корнях бобовых растений при симбиозе с почвенными бактериями ризобиями при недостатке азота в почве, и в этом отношении их формирование также является реакцией на дефицит азота. У модельного бобового растения M. trunсatula обработка пептидом MtCEP1, а также сверхэкспрессия MtCEP1 стимулировала формирование клубеньков и при этом подавляла закладку боковых корней [83]. Более того, эффектом обработки экзогенным пептидом MtCEP1, а также сверхэкспрессии MtCEP1 является формирование так называемых сайтов ССР (circumferential cell proliferations) – периодически расположенных “вздутий” на корне, возникающих в результате пролиферации клеток перицикла, коры и эпидермиса. В части таких сайтов наблюдали блокированные в развитии боковые корни, а из части сайтов CCP при инокуляции ризобиями развивались функционирующие симбиотические клубеньки [83]. Клубеньки, формирующиеся при сверэкспрессии MtCEP1 и обработке MtCEP1, развивались быстрее, характеризовались большими размерами и более высокой нитрогеназной активностью, по сравнению с контролем, даже при высокой концентрации нитрата в среде (до 25 мМ) [83].
У люцерны был охарактеризован мутант cra2 (compact root system architecture 2), характеризующийся укорочением корня и увеличение числа боковых корней [84]. Кроме того, у мутанта cra2 было также снижено число образующихся симбиотических клубеньков, при этом эффект подавления клубенькообразование наблюдался уже на ранних этапах развития примордия, на 1–3 день после инокуляции, и проявлялся независимо от влияния cra2 на укорочение корня. Ген MtCRA2 экспрессируется в проводящей системе побега, корня, в примордиях боковых корней и клубеньков, а в зрелых клубеньках – в апикальной меристеме и в периферических проводящих пучках. Эксперименты с использованием прививок показали, что MtCRA2 локально (на уровне корневой части растения) регулирует развитие боковых корней и системно (с участием побеговой части растения) – развитие симбиотических клубеньков [84]. Ген MtCRA2 кодирует рецептор-подобную киназу с лейцин-богатыми повторами (LRR-RLK) и демонстрирует наибольшее сходство с геном арабидопсиса AtXIP1 [84], продуктом которого является рецептор пептидов CEP [77]. Мутант по гену MtCRA2 оказался нечувствительным к действию MtCEP1. В работе Mohd-Radzman с соавт. было показано, что эффект MtCEP1 на развитие симбиотических клубеньков и боковых корней у люцерны зависит от MtCRA2, что наряду со сходством с AtXIP1, указывает на возможную роль MtCRA2 в рецепции MtCEP [82].
Обработка пептидом MtCEP1 ряда мутантов с нарушением развития клубеньков, дефектных по компонентам сигнального каскада, активируемого ризобиями, не была способна индуцировать клубенькообразование, но при этом вызывала подавление развития боковых корней, что указывает на существование независимых путей регуляции развития клубеньков и боковых корней с участием MtCEP1. Так, у суперклубенькообразующего мутанта люцерны Mtsunn с нарушением AON, обработка MtCEP1 приводила к еще большему увеличению числа клубеньков и к уменьшению количества боковых корней по сравнению с контролем, при этом наблюдалось увеличение границ зоны корня, в пределах которой происходит закладка клубеньков [82].
В противоположность этому, у другого суперклубенькообразующего мутанта, Mtein2/sickle (skl), характеризующегося подавлением передачи сигнала этилена, увеличением числа сайтов инфицирования корня ризобиями (гиперинфекцией) и формированием избыточного числа незрелых клубеньков, часто “слитых” друг с другом, обработка пептидом MtCEP1 или сверхэкспрессия у такого мутанта гена MtCEP1 не вызывала увеличения числа клубеньков или расширения границ зоны клубенькообразования в пределах корня. Продукт гена MtEIN2 – ортолог белка EIN2 арабидопсиса, центрального регулятора клеточного ответа на этилен [85]; у люцерны он также является негативным регулятором клубенькообразования и опосредует негативное влияние этилена на закладку и развитие симбиотических клубеньков. Следует отметить, что обработка пептидом MtCEP1 растений дикого типа также приводила к гиперинфекции ризобиями и увеличению доли “слитых” клубеньков, причем закладка большего процента клубеньков наблюдалась напротив флоэмного полюса (в отличие от контроля, где закладка клубеньков происходит напротив ксилемного полюса). Все эти признаки также характерны для мутантов skl [86]. На основании этих данных и данных по совместной обработке пептидом MtCEP1 и предшественником этилена ACC, авторы предположили, что стимулирующий эффект MtCEP1 на клубенькообразование может быть связан с подавлением MtEIN2-опосредованного этиленового сигналинга [82]. В совокупности, данные полученные в работе Mohd-Radzman с соавт., указывают на то, что пептид MtCEP1 подавляет ингибирующий эффект этилена на закладку клубеньков, зависимым от MtCRA2 способом [82]. Таким образом, MtCEP1 и его вероятный рецептор MtCRA2 регулируют закладку боковых корней и клубеньков за счет активации двух различных регуляторных путей: первый путь связан с системной (через функционирующий в побеге MtCRA2) активацией клубенькообразования за счет подавления этилен-MtEIN2-опосредованного ингибирования клубенькообразования; второй путь связан с локальным (через функционирующий в корне MtCRA2) ингибированием закладки боковых корней, не зависящим от этиленового сигналинга.
Кроме того, совсем недавно был описан еще один механизм, за счет которого MtCEP1 и его предполагаемый рецептор MtCRA2 способны стимулировать клубенькообразование [50]. Gautrat с соавт. идентифицировали у люцерны M. truncatula гены MtCEPD1 и MtCEPD2, гомологи генов AtCEPDs, продуктами которых у арабидопсиса являются мобильные полипептиды, передающие сигнал от рецепторов CEP из побега в корень [50]. Гены MtCEPD1 и MtCEPD2 активировались при недостатке нитрата у дикого типа, но не у мутанта cra2 люцерны, но при этом в отличие от арабидопсиса, у которого транскрипты AtCEPD1 и AtCEPD2 обнаруживались только в побеге, транксрипты MtCEPD1 и MtCEPD2 у люцерны были выявлены как в побеге, так и в корне, что указывает на возможное участие MtCEPD1 и MtCEPD2 в локальной регуляции ответа на недостаток нитрата в корне [50]. В ответ на инокуляцию ризобиями, уровень экспрессии MtCEPD1 и MtCEPD2 в побеге не изменялся, а в корнях наблюдалось, в противоположности ожиданиям авторов, увеличение экспрессии MtCEPD1 и MtCEPD2, при этом такая активация не зависела от MtCRA2. Таким образом, выявленные авторами гены MtCEPD1 и MtCEPD2 не являются системными регуляторами клубенькообразования в пути, активируемом MtCEP1 и MtCRA2, а, вероятно, задействованы в локальной регуляции ответа на нитрат и инокуляцию ризобиями в пределах корня. В этой же работе авторы проверили возможное участие в MtCEP1-активируемом сигнальном пути другого ранее выявленного системного регулятора – микроРНК miR2111, которая у лядвенца в отсутствии ризобий транспортируется из побега в корень и опосредует деградацию транскриптов гена TML – ингибитора клубенькообразования, таким образом, делая корень “восприимчивым” к формированию симбиоза с ризобиями [49]. Экспрессия выявленных Gautrat с соавт. генов-предшественников miR2111 у люцерны подавлялась в ответ на инокуляцию ризобиями, и так же, как это ранее было описано для miR2111 лядвенца, снижала содержание транскриптов генов MtTML1/MtTML2, при этом такая регуляция зависела от активности рецептора SUNN – ключевого компонента AON у люцерны [50].
У мутанта cra2 была значительно снижена экспрессия генов-предшественников miR2111 даже до инокуляции ризобиями, что сопровождалось увеличением количества транскриптов одного из гомологов TML у люцерны, MtTML1 – предполагаемой мишени miR2111. Сверхэкспрессия miR2111 приводила к увеличению числа клубеньков у мутанта cra2 до уровня, сопоставимого с диким типом. Кроме того, уровень экспрессии генов-предшественников miR2111 и содержание зрелых miR2111 были повышены при недостатке азота, а также при сверхэкспрессии MtCEP1 [50]. На основании полученных результатов авторы сделали вывод о том, что пептид CEP1 и его вероятный рецептор CRA2 опосредуют активацию экспрессии miR2111 в отсутствии инокуляции ризобиями и при низком содержании азота в среде. Согласно данным, полученным с помощью прививок, для такой регуляции необходима активность MtCRA2 в побеге. Таким образом, уровень miR2111, мобильной микроРНК, транспортирующейся из побега в корень и стимулирующей клубенькообразование, контролируется с участием двух регуляторных путей: пути CEP-CRA2, позитивно регулирующего клубенькообразование, и пути CLE-SUNN, подавляющего развитие клубеньков, а также с учетом содержания азота в почве и присутствия азотфиксаторов – ризобий (рис. 1).
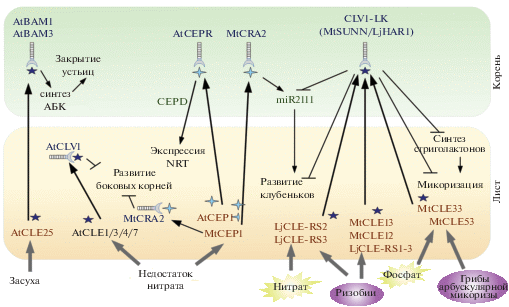
Рис. 1.
Участие пептидов CLE и CEP в системном контроле развития симбиотических клубеньков у люцерны. Ризобии, выделяя Nod-факторы, активируют сигнальный каскад, приводящий к формированию примордиев симбиотических клубеньков. ТФ NIN активирует экспрессию генов CLE. Пептиды CLE (MtCLE12, MtCLE13) транспортируются из корней по ксилеме в побег, где во флоэме листа они связываются с CLV1-подобными киназами (MtSUNN). Триарабинозилирование CLE осуществляется ферментами гидроксипролин-O-арабинозилтрансферазами, кодируемыми генами RDN. Активация рецепторов CLE в побеге запускает ответный сигнал, приводящий к подавлению клубенькообразования, в частности, за счет подавления экспрессии NIN. Транспортируемый из побега цитокинин системно подавляет образование клубеньков. MtSUNN подавляет экспрессию генов miR2111. miR2111 – мобильный фактор, подавляющий экспрессию гена TML – негативного регулятора клубенькообразования. Нитрат подавляет развитие клубеньков. Нитрат опосредует накопление в ядре ТФ NLP. NLP1 у люцерны подавляет работу NIN за счет прямого взаимодействия с ним, что, вероятно, блокирует активацию экспрессии генов-мишеней NIN, и как следствие – ведет к подавлению клубенькообразования. При недостатке в почве нитрата активируется экспрессия генов CEP (MtCE-P1). MtCEP1 транспортируется из корней в побег, где во флоэме листа узнается специфическим рецептором – MtCRA2. Активация MtCRA2 в побеге стимулирует клубенькообразование за счет подавления этиленового ответа в корне. Также MtCRA2 активирует накопление miR2111 в побеге, что способствует развитию симбиотичских клубеньков. Гены CEPD активируеются при недостатке нитрата как в листьях, так и в корнях люцерны. У люцерны CEPD, по-видимому, дейтствуют на уровне корня. Экспрессия CEPD также активируется в корнях при действии ризобий, однако их участие в клубенькообразовании остается не исследованным. Таким образом, MtCEP1 – стимулирует, а MtCLE-12/13 подавляют клубенькообразование, действуя через рецепторы, работающие в побеге (MtCRA2 и MtSUNN, соответственно).

3. ДРУГИЕ ПРИМЕРЫ СЕКРЕТИРУЕМЫХ ПЕПТИДОВ РАСТЕНИЙ, СПОСОБНЫХ К ДАЛЬНЕМУ ТРАНСПОРТУ ПО КСИЛЕМЕ
В геноме арабидопсиса идентифицировано около 1000 генов, предположительно кодирующих белки-предшественники пептидов [87]. Функции многих из этих пептидов еще не охарактеризованы, и возможно, что некоторые из них также способны транспортироваться по проводящей системе и опосредовать коммуникацию между разными органами растения. Протеомный анализ ксилемного сока является перспективным подходом для выявления потенциально мобильных пептидов, однако этот подход имеет ряд технических ограничений, связанных с необходимостью получения достаточного количества материала для исследований, и до недавнего времени был применим только к достаточно крупным модельным растениям, таким как соя, кукуруза, капуста [88, 89]. В работе Okamoto с соавт. был проведен пептидомный анализ ксилемного сока сои Glycine max для выявления потенциальных пептидов, участвующих в дальнем транспорте по ксилеме [90]. В результате было выявлено семь пептидов длиной от 11 до 19 аминокислот, обозначенных авторами как XAP 1-7 (xylem sap-associated peptides), молекулы которых имели одну или более посттрансляционную модификацию: гликозилирование, сульфатирование и/или гидроксилирование (табл. 1). Так, пептиды XAP1 и XAP3 были сульфатированы по остатку тирозина и были представлены в относительно большом количестве в ксилемном соке, а пептиды XAP4 и XAP6 оказались гомологами охарактеризованных ранее пептидов, для которых был показан транспорт по ксилеме: XAP4, несущий гидроксилированные остатки пролина и триарабинозилированный гидроксипролин в положении семь, оказался представителем семейства пептидов CLE, а пептид XAP6 c гидроксилированным пролином в положении одиннадцать – представителем пептидов CEP. Для этих четырех пептидов, XAP1, XAP3, XAP4 и XAP6, была подтверждена способность транспортироваться по ксилеме. Путем внесения специфических мутаций в последовательности, кодирующие пептиды XAP1, XAP3, XAP4 и XAP6, были получены мутантные формы пептидов, которые по масс-спектрометрическим показателям можно было отличить от нативных пептидов, присутствующих в растениях. Мутантные формы пептидов XAP1, XAP3, XAP4 и XAP6, синтезированных в трансгенных корнях сои, полученных в результате трансформации с помощью A. rhizogenes, были обнаружены в ксилемном соке побега, что доказывает способность этих пептидов перемещаться по ксилеме из корня в побег [90]. Кроме того, авторы изучили влияние различных факторов среды на экспрессию генов, кодирующих данные пептиды у сои, и показали, что экспрессия XAP1 активировалась при избыточном увлажнении, XAP4 активировался как при избытке влаги, так и при инокуляции ризобиями, экспрессия гена XAP6 подавлялась при избытке азота и при избыточном увлажнении, а экспрессия XAP3 значимо не менялась при воздействии внешних факторов среды, но в незначительной степени снижалась при избыточном увлажнении [90].
Таблица 1.
Пептиды, выявленные в кислемном соке сои и люцерны
| Семейство пептидов | Последовательность пептида | ID гена | Название гена M. truncatula, ближайшего гомолога гена G. max у M. truncatula по номенклатуре de Bang с соавт. [91] |
|---|---|---|---|
| CLE | RLSPGGPDAHHH | Glyma13g24026.1 (XAP4)* | CLE42 |
| HVVPSGPNPLHN | Medtr1g100733.1 (MtCLE5)** | CLE5 | |
| RKVPTGSNPLHN | Medtr5g085990.1 (MtCLE17) | CLE17 | |
| CEP | DFKPTDPGHSPGVG | Glyma01g39170.1 (XAP6A)* Glyma05g16420.2 (XAP6B)* |
CEP12 |
| AFQPTTPGNSPGVGH | Medtr5g030490.1** (MtCEP1-D1) |
CEP1 | |
| EFQKTNPGHSPGVGH | Medtr5g030490.1** (MtCEP1-D2) |
CEP1 | |
| AFRPTTPGHSPGIGH | AFK45165.1 (MtCEP2)** | CEP2 | |
| AFRPTTPGHSPGVGH | Medtr5g017710.1 (MtCEP5)** | CEP5 | |
| AFRPTTPGNSPGVGH | AC233112_1014.1 (MtCEP8)** | CEP8 | |
| PSY | DYEGTGANKDHDPKSPGGA | Glyma19g29590.1 (XAP1)* | PSY9 |
| DYEGTGANKDHDPKSPGGP | Glyma16g03960.1 (XAP2)* | PSY9 | |
| DYDEAGPNPRH | Glyma04g41240.2 (XAP5A)* Glyma06g13610.1 (XAP5B)* |
PSY4 | |
| DYSPSGPNGRHTP | Glyma05g00980.2 (XAP7A)* Glyma17g10910.1 (XAP7B)* |
PSY6 | |
| DYGGTGANTDHEPKPPR | Medtr7g086770.1 (MtXAP1a)** | PSY10 | |
| DYEGPGANKEHNPKSPGNG | Medtr6g027480.1 (MtXAP1b)** | PSY9 | |
| DYPGTGPNHHHDPKSPG | Medtr6g027470.1 (MtXAP1c)** | PSY3 | |
| DYDEAGPNPKHSKKPGKG | Medtr3g491850.1 (MtXAP5 )** | PSY4 | |
| DYAPSGANGRHTP | Medtr4g108700.1 (MtXAP7)** | PSY6 | |
| CIF | DYGRYDPSPSLSKPPFK | Glyma13g27110.1 (XAP3A)* Glyma12g36411.1 (XAP3B)* |
CIF1 |
| DYGRYDPSPTFSKPP | Medtr8g037800.1 (MtXAP3)** | CIF1 |
Позднее, de Bang с соавт. опубликовали работу по идентификации генов, кодирующих предполагаемые регуляторные пептиды, в геноме люцерны M. truncatula, в результате чего было охарактеризовано около двух тысяч генов, отнесенных к 46 различным семействам регуляторных пептидов [91]. Анализируя последовательности пептидов люцерны, полученных в данном исследовании, мы обнаружили близкие гомологи пептидов XAP1, XAP3, XAP4 и XAP 6 (табл. 1).
Пептид XAP1 по последовательности оказался близок пептиду люцерны из семейства PSY (PLANT PEPTIDE CONTAINING SULFATED TYROSINE). Известно, что представитель этой группы пептидов у арабидопсиса, пептид PSY1 (AtPSY1), стимулирует рост корня, в частности, за счет активации протонных помп и закисления апопласта [92]. Рецептором AtPSY1 является рецепторная киназа PSY1R (PSY1 RECEPTOR), работающая в виде гетеродимера с SERK1 (SOMATIC EMBRYOGENESIS RECEPTOR KINASE1), мишенью действия которой является мембранная H+-АТФаза AHA2 [93]. Интересно, что фитопатоген Xanthomonas oryzae также продуцирует сульфатированные пептиды, схожие по последовательности с PSY, способные имитировать рост-стимулирующее действие эндогенных пептидов PSY [94]. Ранее для этой группы пептидов не был описан дальний транспорт, однако, в связи с обнаружением транспорта PSY-подобного пептида по ксилеме у сои, представляет интерес более детальное изучение пептидов PSY в развитии растений и в дальнем транспорте, а также в ответе на различные внешние стимулы.
Пептид XAP3 по последовательности оказался близок пептиду люцерны семейства CIF (CASPARIAN STRIP INTEGRITY FACTOR). У арабидопсиса пептиды CIF1 и CIF2 регулируют развитие поясков Каспари клеток эндодермы [95]. Кроме того, экспрессия генов CIF у арабидопсиса меняется в зависимости от рН среды и концентрации солей железа в среде, при этом мутанты арабидопсиса cif1-1cif2-1 характеризуются пониженной устойчивостью к солям железа [95]. Обнаружение CIF-подобного пептида в ксилемном соке сои может указывать на его возможное участие в системном контроле развития растений в ответ на воздействие неблагоприятных факторов среды.
Интересно, что обнаруженный в ксилемном соке сои пептид XAP4 по последовательности оказался идентичным CLE-домену пептида люцерны MtCLE42. Ранее было показано, что экспрессия гена MtCLE42 активируется при развитии симбиотических клубеньков, но экзогенная обработка несущим посттрансляционные модификации пептидом MtCLE42 не подавляла клубенькообразование у люцерны, в отличие от его гомологов MtCLE12 и MtCLE13, на основании чего предполагается, что данный пептид не вовлечен в AON [36].
В другой работе с использованием культуры бородатых корней люцерны M. truncatula был также идентифицирован ряд секреторных пептидов, присутствующих в ксилемном соке [96]. Также как и при анализе ксилемного сока сои авторам удалось показать наличие в ксилемном соке секретируемых пептидов, которые можно отнести к семействам CLE, CEP, CIF и PSY (табл. 1). Кроме того, в данной работе была изучена биологическая активность идентифицированных пептидов. В частности, обработка экзогенным пептидом MtCLE17, который был обнаружен в ксилемном соке, а также пептидом MtCLE5, который по последовательности оказался очень близким CLE-домену TDIF, подавляла рост главного корня и стимулировал образование боковых корней [96]. Однако такой эффект ранее не был описан для предполагаемого гомолога MtCLE5 – пептида TDIF арабидопсиса, кодируемого генами AtCLE41/AtCLE44 [97]. Обработка пептидами из группы PSY, сульфатированными по тирозину пептидами, обозначенными авторами как MtXAP1 и MtXAP5, приводила к подавлению развития боковых корней [96].
Таким образом, в ходе пептидомных исследований ксилемного сока сои и люцерны были выявлены пептиды семейств CLE и CEP, для которых ранее была показана роль в системном контроле процессов развития, а также представители двух других семейств – пептиды PSY и пептиды CIP, для которых ранее не был показан дальний транспорт по ксилеме. Обнаружение пептидов PSY и CIP в ксилемном соке у двух проанализированных видов бобовых растений указывает на возможное участие этих малоизученных к настоящему времени пептидов в дальнем транспорте по ксилеме и в системном контроле процессов развития, и, несомненно, заслуживает дальнейшего детального изучения.
ЗАКЛЮЧЕНИЕ
Таким образом, представители двух семейств пептидных регуляторов, CLE и CEP, синтезируются в корнях при воздействии различных факторов, таких, как доступность воды и минеральных веществ, присутствие в почве микроорганизмов, а также способны транспортироваться из корней в наземную часть растения, где во флоэме листа их распознают специфические рецепторы (рис. 2).
Рис. 2.
Регуляторные пептиды и их рецепторы, опосредующие системные реакции у растений. Пептид AtCLE25 синтезируется в корнях при засухе, перемещается в побег, где связывается со своими предполагаемыми рецепторами AtBAM1/AtBAM3. В ответ активируется экспрессия гена NCED3, вовлеченного в биосинтез АБК – гормона, стимулирующего закрытие устьиц. При недостатке азота в почве у арабидопсиса активируется экспрессия AtCLE1/3,4,7, продукты которых связываются с рецептором AtCLV1, предположительно работающем в побеге, и подавляют развитие боковых корней. Недостаток азота также активирует экспрессию CEP. У арабидопсиса AtCEP1 транспортируется в побег, где связывается с рецептором – AtCEPR, в результате чего посредством мобильных белков CEPD в корень передается ответный сигнал, стимулирующий экспрессию генов нитратных транспортеров NRT. У люцерны при недостатке азота активируется экспрессия MtCEP1. MtCEP1 транспортируется в побег, где связывается с рецептором MtCRA2 и опосредует ответные реакции, активирующие клубенькообразование на корнях: подавление этиленового ответа и активацию экспрессии генов микроРНК miR2111. Кроме того, через работающую в корнях рецепторную киназу MtCRA2 пептид MtCEP1 подавляет развитие боковых корней. Высокое содержание нитрата активирует нитрат-регулируемые CLE у бобовых растений. Продукты активируемых нитратом генов LjCLE-RS2,3 действуя через CLV1-подобную киназу LjHAR1, работающую в побеге, подавляют клубенькообразование. Кроме того, экспрессия генов LjCLE-RS2,3, а также LjCLE-RS1, активируется при взаимодействии растения с ризобиями. Их продукты перемещаются по ксилеме в листья, где во флоэме листа взаимодействуют с LjHAR1 и запускают ответные реакции, подавляющие дальнейшее формирование симбиотических клубеньков. В результате взаимодействия с ризобиями у люцерны происходит активация генов MtCLE12,13, продукты которых перемещаются в побег и связываются c киназой MtSUNN. MtSUNN подавляет клубенькообразование опосредованно через гормоны, а также за счет уменьшения уровня miR2111, перемещающейся из побега в корень и подавляющей экспрессию TML – ингибитора клубенькообразования. У люцерны при микоризации и обработке фосфатом активируются гены MtCLE53 и MtCLE33, соответственно, продукты которых по механизму обратной связи подавляют микоризацию, действуя через рецепторную киназу MtSUNN в побеге. В ответ происходит подавление биосинтеза стриголактонов – гормонов, стимулирующих развитие микоризы. Таким образом, пептиды CLE (MtCLE33, MtCLE53) и их рецептор – MtSUNN являются компонентами системы авторегуляции микоризации.
Разнообразие генов, кодирующих пептиды CLE и CEP у растений, достаточно велико, и, по-видимому, их появление в ходе эволюции способствовало возникновению новых регуляторных путей, в том числе, системной регуляции, обеспечивающей лучшую приспособленность растения к меняющимся условиям среды. Интересно, что гены, кодирующие пептиды CEP и CLE, были обнаружены не только у растений, но также и у организмов, тесно с ними взаимодействующими. Так, у паразитических нематод, вызывающих разнообразные разрастания тканей растений, были обнаружены гены, кодирующие пептиды CLE и CEP, продукты которых биологически активны в тканях растений, и даже способны узнаваться рецепторами растения-хозяина, “мимикрируя” под эндогенные регуляторы растений [98, 99]. Кроме того, в геноме грибов арбускулярной микоризы (у четырех видов рода Rhizophagus и одного вида рода Gigaspora) также были обнаружены гены, кодирующие пептиды CLE и оказывающие влияние на развитие корневой системы растения-хозяина, также, вероятно, за счет взаимодействия с растительными рецепторами [100].
По-видимому, рецепторы пептидов CLE и CEP у растений обладают достаточно широкой специфичностью и способны связываться с целым рядом пептидов, различающихся по аминокислотной последовательности, в связи с чем пептиды, индуцируемые при воздействии различных факторов, могут восприниматься рецепторными комплексами, включающими одни и те же белки. Например, у люцерны CLV1-подобная киназа MtSUNN, как предполагается, ответственна за восприятие пептидов MtCLE12/MtCLE13, активируемых ризобиями, а также пептидов MtCLE33/MtCLE53, активируемых при развитии арбускулярной микоризы (рис. 2).
Корневая система растений выполняет “сенсорную” функцию, “улавливая” воздействие различных факторов (изменение уровня минеральных веществ в почве, ее увлажненности, воздействие микроорганизмов) и стимулирует выработку регуляторных пептидов, которые передают информацию о таких изменениях побеговой системе. Рецепторы регуляторных пептидов, поступающих по ксилеме из корней – в побег, локализованы во флоэме листа, и они способны интегрировать различные сигналы, поступающие от подземной части растения. Клетки флоэмы листа, таким образом, являются своего рода “центрами управления”, которые запускают ответные реакции и регулируют процессы развития растений и их минеральное питание на системном уровне. Поскольку в рецепции поступающих из корней пептидов задействованы рецепторные киназы, работающие во всей совокупности листьев растений, то, таким образом, сигнальные молекулы, поступающие из побега – в корень, также представляет собой некий “коллективный” ответ побега на сигналы, полученные от корневой системы. Такой обмен сигналами между побеговой и корневой системами за счет дальнего транспорта регуляторных молекул лежит в основе системной регуляции процессов развития и жизнедеятельности растения на организменном уровне.
Изучение молекулярных основ такой системной регуляции является важным как с фундаментальной точки зрения, так с практической, поскольку понимание механизмов регуляции минерального питания и влияния макроэлементов на рост и развитие растений позволит сформировать новые принципы в сельском хозяйстве, направленные на оптимизацию количества вносимых минеральных удобрений с целью повышения урожайности сельскохозяйственно значимых культур.
Работа выполнена при поддержке гранта Российского фонда фундаментальных исследований № 19-116-50095 (Экспансия).
Авторы заявляют, что у них нет конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Список литературы
Oh E., Seo P.J., Kim J. Signaling peptides and receptors coordinating plant root development // Trends Plant Sci. 2018. V. 23. P. 337. https://doi.org/10.1016/j.tplants.2017.12.007
Ганчева М.С., Маловичко Ю.В., Полюшкевич Л.О., Додуева И. Е., Лутова Л.А. Пептидные гормоны растений // Физиология растений. 2019. Т. 66. С. 83.
Opsahl-Ferstad H.G., Le Deunff E., Dumas C., Rogowsky P.M. ZmEsr, a novel endosperm-specific gene expressed in a restricted region around the maize embryo // The Plant Journal. 1997. V. 12. P. 235.
Fletcher J.C., Brand U., Running M.P., Simon R., Meyerowitz E.M. Signaling of cell fate decisions by CLAVATA3 in Arabidopsis shoot meristems // Science. 1999. V. 283. P. 1911.
Ohyama K., Ogawa M., Matsubayashi Y. Identification of a biologically active, small, secreted peptide in Arabidopsis by in silico gene screening, followed by LC-M-S-based structure analysis // Plant J. 2008. V. 55. P. 152. https://doi.org/10.1111/j.1365-313X.2008.03464.x
Delves A.C., Mathews A., Day D.A., Carter A.S., Carroll B.J., Gresshoff P.M. Regulation of the soybean-Rhizobium nodule symbiosis by shoot and root factors // Plant Physiol. 1986. V. 82. P. 588.
Stahl Y., Wink R.H., Ingram G.C., Simon R. A signaling module controlling the stem cell niche in Arabidopsis root meristems // Curr. Biol. 2009. V. 19. P. 909.
Ito Y., Nakanomyo I., Motose H., Iwamoto K., Sawa S., Dohmae N., Fukuda H. Dodeca-CLE peptides as suppressors of plant stem cell differentiation // Science. 2006. V. 313. I. 5788. P. 842.
Vatén A., Soyars C.L., Tarr P.T., Nimchuk Z.L., Bergmann D.C. Modulation of asymmetric division diversity through cytokinin and SPEECHLESS regulatory interactions in the arabidopsis stomatal lineage // Dev. Cell. 2018. V. 47: 53.e5.
Ren S.C., Song X.F., Chen W.Q., Lu R., Lucas W.J., Liu C.M. CLE25 peptide regulates phloem initiation in Arabidopsis through a CLERK-CLV2 receptor complex // J. Integr. Plant Biol. 2019. V. 61. P. 1043.
Fiume E., Fletcher J.C. Regulation of Arabidopsis embryo and endosperm development by the polypeptide signaling molecule CLE8 // Plant Cell. 2012. V. 24. P. 1000.
Okamoto S., Ohnishi E., Sato S., Takahashi H., Nakazono M., Tabata S., Kawaguchi M. Nod factor/nitrate-induced CLE genes that drive HAR1-mediated systemic regulation of nodulation // Plant Cell Physiol. 2009. V. 50. P. 67.
Mortier V., Den Herder G., Whitford R., Van de Velde W., Rombauts S., D’Haeseleer K., Holsters M., Goormachtig S. CLE peptides control Medicago truncatula nodulation locally and systemically // Plant Physiol. 2010. V. 153. P. 222.
Kiyohara S., Sawa S. CLE signaling systems during plant development and nematode infection // Plant Cell Physiol. 2012. V. 53. P. 1989.
Oelkers K., Goffard N., Weiller G.F., Gresshoff P.M., Mathesius U., Frickey T. Bioinformatic analysis of the CLE signaling peptide family // BMC Plant Biol. 2008. V. 8. P. 1. https://doi.org/10.1186/1471-2229-8-1
Matsubayashi Y. Posttranslationally modified small-peptide signals in plants // Annu. Rev. Plant Biol. 2014. V. 65. P. 385.
Ohyama K., Shinohara H., Ogawa-Ohnishi M., Matsubayashi Y. A glycopeptide regulating stem cell fate in Arabidopsis thaliana // Nat. Chem. Biol. 2009. V. 8. P. 578.
Hazak O., Hardtke C.S. CLAVATA 1-type receptors in plant development // J. Exp. Bot. 2016. V. 67. P. 4827.
Yamaguchi Y.L, Ishida T., Sawa S. CLE peptides and their signaling pathways in plant development // J. Exp. Bot. 2016. V. 67. P. 4813.
Dolzblasz A., Nardmann J., Clerici E., Causier B., van der Graaff E., Chen J., Davies B., Werr W., Laux T. Stem cell regulation by Arabidopsis WOX genes // Mol. Plant. 2016. V. 9. P. 1028.
Dodueva I.E., Tvorogova V.E., Azarakhsh M., Lebedeva M.A., Lutova L.A. Plant stem cells: Unity and diversity // Russ. J. Genet. Appl. Res. 2017. V. 7. P. 385. https://doi.org/10.1134/S2079059717040025
Strabala T.J., O’donnell P.J., Smit A.M., Ampomah-Dwamena C., Martin E.J., Netzler N. Nieuwenhuizen N.J., Quinn B.D., Foote H.C., Hudson K.R. Gain-of-function phenotypes of many CLAVATA3/ESR genes, including four new family members, correlate with tandem variations in the conserved CLAVATA3/ESR domain // Plant Physiol. 2006. V. 140. P. 1331.
Whitford R., Fernandez A., De Groodt R., Ortega E., Hilson P. Plant CLE peptides from two distinct functional classes synergistically induce division of vascular cells // Proc. Natl. Acad. Sci. U. S. A. 2008. V. 105. I. 47. P. 18625.
Clark S.E., Williams R.W., Meyerowitz E.M. The CLAVATA1 gene encodes a putative receptor kinase that controls shoot and floral meristem size in Arabidopsis // Cell. 1997. V. 89. P. 575.
Kinoshita A., Betsuyaku S., Osakabe Y., Mizuno S., Nagawa S., Stahl Y., Simon R., Yamaguchi-Shinozaki K., Fukuda H., Sawa S. RPK2 is an essential receptor-like kinase that transmits the CLV3 signal in Arabidopsis // Development. 2010. V. 137. I. 22. P. 3911.
DeYoung B.J., Bickle K.L., Schrage K.J., Muskett P., Patel K., Clark S.E. The CLAVATA1-related BAM1, BAM2 and BAM3 receptor kinase-like proteins are required for meristem function in Arabidopsis // Plant J. 2006. V. 45. P. 1.
Bleckmann A., Weidtkamp-Peters S., Seidel C., Simon R. Stem cell signaling in Arabidopsis requires CRN to localize CLV2 to the plasma membrane // Plant Physiol. 2010. V. 152. P. 166.
Qiang Y., Wu J., Han H., Wang G. CLE peptides in vascular development // J. Integr. Plant Biol. 2013. V. 55. P. 389.
Searle I.R., Men A.E., Laniya T.S., Buzas D.M., Iturbe-Ormaetxe I., Carroll B.J., Gresshoff P.M. Long-distance signaling in nodulation directed by a CLAVATA1-like receptor kinase // Science. 2003. V. 299. I. 5603. P.109.
Reid D.E., Ferguson B.J., Hayashi S., Lin Y.-H., Gresshoff P.M. Molecular mechanisms controlling legume autoregulation of nodulation // Ann. Bot. 2011. V. 789. https://doi.org/10.1093/aob/mcr205
Nishimura R., Hayashi M., Wu G.J., Kouchi H., Imaizumi-Anraku H., Murakami Y., Kawasaki S., Akao S., Ohmori M., Nagasawa M., Harada K., Kawaguchi M. HAR1 mediates systemic regulation of symbiotic organ development // Nature. 2002. V. 420. I. 6914. P. 426.
Schnabel E., Journet E.P., de Carvalho-Niebel F., Duc G., Frugoli J. The Medicago truncatula SUNN gene encodes a CLV1-like leucine-rich repeat receptor kinase that regulates nodule number and root length // Plant Mol Biol. 2005. V. 58. I. 6. P. 809.
Krusell L., Sato N., Fukuhara I., Koch B.E., Grossmann C., Okamoto S., Oka-Kira E., Otsubo Y., Aubert G., Nakagawa T., Sato S., Tabata S., Duc G., Parniske M., Wang T.L., Kawaguchi M., Stougaard J. The Clavata2 genes of pea and Lotus japonicus affect autoregulation of nodulation // Plant J. 2011. V. 65. I. 6. P. 861.
Lim C.W., Lee Y.W., Hwang C.H. Soybean nodule-enhanced CLE peptides in roots act as signals in GmNARK-mediated nodulation suppression // Plant Cell Physiol. 2011. V. 52. 1613. https://doi.org/10.1093/pcp/pcr091
Samorodova A.P., Tvorogova V.E., Tkachenko A.A., Potsenkovskaya E.A., Lebedeva M.A., Tikhonovich I.A., Lutova L.A. Agrobacterial tumors interfere with nodulation and demonstrate the expression of nodulation-induced CLE genes in pea // J. Plant Physiol. 2018 V. 221. P. 94.
Imin N., Patel N., Corcilius L., Payne R.J., Djordjevic M.A. CLE peptide tri-arabinosylation and peptide domain sequence composition are essential for SUNN-dependent autoregulation of nodulation in Medicago truncatula // New Phytol. 2018. V. 218. P. 73. https://doi.org/10.1111/nph.15019
Postma J.G., Jacobsen E., Feenstra W.J. Three pea mutants with an altered nodulation studied by genetic analysis and grafting // J. Plant Physiol. 1988. V. 132. P. 424. https://doi.org/10.1016/S0176-1617(88)80056-7
Schnabel E.L., Kassaw T.K., Smith L.S., Marsh J.F., Oldroyd G.E., Long S.R., Frugoli J.A. The ROOT DETERMINED NODULATION1 gene regulates nodule number in roots of Medicago truncatula and defines a highly conserved, uncharacterized plant gene family // Plant Physiol. 2011. V. 157. P. 328. https://doi.org/10.1104/pp.111.178756
Kassaw T., Nowak S., Schnabel E., Frugoli J. ROOT DETERMINED NODULATION1 is required for M. truncatula CLE12, but not CLE13, peptide signaling through the SUNN receptor kinase // Plant Physiol. 2017. V. 174. P. 2445. https://doi.org/10.1104/pp.17.00278
Okamoto S., Shinohara H., Mori T., Matsubayashi Y., Kawaguchi M. Root-derived CLE glycopeptides control nodulation by direct binding to HAR1 receptor kinase // Nat. Commun. 2013. V. 4. P. 2191.
Crook A.D., Schnabel E.L., Frugoli J.A. The systemic nodule number regulation kinase SUNN in Medicago truncatula interacts with MtCLV2 and MtCRN // Plant J. 2016. V. 88. I. 1. P. 108.
Miyazawa H., Oka-Kira E., Sato N., Takahashi H., Wu G.-J., Sato S., Hayashi M., Betsuyaku S., Nakazono M., Tabata S., Harada K., Sawa S., Fukuda H., Kawaguchi M. The receptor-like kinase KLAVIER mediates systemic regulation of nodulation and non-symbiotic shoot development in Lotus japonicus // Development. 2010. V. 137. P. 4317. https://doi.org/10.1242/dev.058891
van Noorden G.E., Ross J.J., Reid J.B., Rolfe B.G., Mathesius U. Defective long-distance auxin transport regulation in the Medicago truncatula super numeric nodules mutant // Plant Physiol. 2006. V. 140. P. 1494. https://doi.org/10.1104/pp.105.075879
Kinkema M., Gresshoff P.M. Investigation of downstream signals of the soybean autoregulation of nodulation receptor kinase GmNARK // Mol. Plant Microbe Interact. 2008 V. 21. I. 10. P. 1337.
Cheng C., Li C., Wang D., Zhai L., Cai Z. The soybean GmNARK affects ABA and salt responses in transgenic Arabidopsis thaliana // Front. Plant Sci. 2018. V. 9. P. 514.
Sasaki T., Suzaki T., Soyano T., Kojima M., Sakakibara H., Kawaguchi M. Shoot-derived cytokinins systemically regulate root nodulation // Nat. Commun. 2014. V. 5. P. 4983.
Andrade M.A., González-Guzmán M., Serrano R., Rodrí-guez P.L. A combination of the F-box motif and kelch repeats defines a large Arabidopsis family of F‑box proteins // Plant Mol Biol. 2001. V. 46. I. 5. P. 603.
Takahara M., Magori S., Soyano T., Okamoto S., Yoshida C., Yano K., Sato S., Tabata S., Yamaguchi K., Shigenobu S., Takeda N., Suzaki T., Kawaguchi M. TOO MUCH LOVE, a novel kelch repeat-containing F-box protein, functions in the long-distance regulation of the legume–rhizobium symbiosis // Plant Cell Physiol. 2013. V. 54. P. 433. https://doi.org/10.1093/pcp/pct022
Tsikou D., Yan Z., Holt D.B., Abel N.B., Reid D.E., Madsen L.H., Bhasin H., Sexauer M., Stougaard J., Markmann K. Systemic control of legume susceptibility to rhizobial infection by a mobile microRNA // Science. 2018. V. 362. I. 6411. P. 233.
Gautrat P., Laffont C., Frugier F. Compact root architecture 2 promotes root competence for nodulation through the miR2111 systemic effector // Curr. Biol. 2020. V. 30. I. 7. P. 1339. https://doi.org/10.1016/j.cub.2020.01.084
Soyano T., Hirakawa H., Sato S., Hayashi M., Kawaguchi M. NODULE INCEPTION creates a long-distance negative feedback loop involved in homeostatic regulation of nodule organ production // PNAS. 2014. V. 111. P. 14607. https://doi.org/10.1073/pnas.1412716111
Mortier V., De Wever E., Vuylsteke M., Holsters M., Goormachtig S. Nodule numbers are governed by interaction between CLE peptides and cytokinin signaling // Plant J. 2012. V. 70. P. 367. https://doi.org/10.1111/j.1365-313X.2011.04881.x2012
Nishida H., Suzaki T. Nitrate-mediated control of root nodule symbiosis // Curr. Opin. Plant Biol. 2018. V. 44. P. 129. https://doi.org/10.1016/j.pbi.2018.04.006
Reid D.E., Ferguson B.J., Gresshoff P.M. Inoculation- and nitrate-induced CLE peptides of soybean control NARK-dependent nodule formation // Mol. Plant Microbe Interact. 2011. V. 24. P. 606. https://doi.org/10.1094/MPMI-09-10-0207
Lim C.W., Lee Y.W., Lee S.C., Hwang C.H. Nitrate inhibits soybean nodulation by regulating expression of CLE genes // Plant Science. 2014. V. 229. P. 1. https://doi.org/10.1016/j.plantsci.2014.08.014
Schauser L., Wieloch W., Stougaard J. Evolution of NIN-like proteins in Arabidopsis, rice, and Lotus japonicus // J. Mol. Evol. 2005. V. 60. I. 2. P. 229.
Konishi M., Yanagisawa S. Identification of a nitrate-responsive cis-element in the Arabidopsis NIR1 promoter defines the presence of multiple cis-regulatory elements for nitrogen response // Plant J. 2010. V. 63. I. 2. P. 269.
Nishida H., Tanaka S., Handa Y., Ito M., Sakamoto Y., Matsunaga S., Betsuyaku S., Miura K., Soyano T., Kawaguchi M., Suzaki T. A NIN-LIKE PROTEIN mediates nitrate-induced control of root nodule symbiosis in Lotus japonicus // Nat. Commun. 2018. V. 499. https://doi.org/10.1038/s41467-018-02831-x
Marchive C., Roudier F., Castaings L., Bréhaut V., Blondet E., Colot V., Meyer C., Krapp A. Nuclear retention of the transcription factor NLP7 orchestrates the early response to nitrate in plants // Nat. Commun. 2013. V. 4. P. 1713.
Suzuki W., Konishi M., Yanagisawa S. The evolutionary events necessary for the emergence of symbiotic nitrogen fixation in legumes may involve a loss of nitrate responsiveness of the NIN transcription factor // Plant Signaling Behav. 2013. V. 8: e25975. https://doi.org/10.4161/psb.25975
Lin J.-S., Li X., Luo Z., Mysore K.S., Wen J., Xie F. NIN interacts with NLPs to mediate nitrate inhibition of nodulation in Medicago truncatula // Nat. Plants. 2018. V. 4. P. 942. https://doi.org/10.1038/s41477-018-0261-3
Müller L.M., Flokova K., Schnabel E., Sun X., Fei Z., Frugoli J., Bouwmeester H.J., Harrison M.J. A CLE–SUNN module regulates strigolactone content and fungal colonization in arbuscular mycorrhiza // Nat. Plants. 2019. V. 5. P. 933. https://doi.org/10.1038/s41477-019-0501-1
Lagunas B., Achom M., Bonyadi-Pour R., Pardal A.J., Richmond B.L., Sergaki C., Vázquez S., Schäfer P., Ott S., Hammond J., Gifford M.L. Regulation of resource partitioning coordinates nitrogen and Rhizobia responses and autoregulation of nodulation in Medicago truncatula // Mol. Plant. 2019. V. 12. P. 833. https://doi.org/10.1016/j.molp.2019.03.014
Araya T., Miyamoto M., Wibowo J., Suzuki A., Kojima S., Tsuchiya Y.N., Sawa S., Fukuda H., von Wirén N., Takahashi H. CLE-CLAVATA1 peptide-receptor signaling module regulates the expansion of plant root systems in a nitrogen-dependent manner // PNAS. 2014. V. 111. P. 2029. https://doi.org/10.1073/pnas.1319953111
Sun C.-H., Yu J.-Q., Hu D.-G. Nitrate: A crucial signal during lateral roots development // Front. Plant Sci. 2017. V. 8. https://doi.org/10.3389/fpls.2017.00485
Olas J.J., Dingenen J.V., Abel C., Działo M.A., Feil R., Krapp A., Schlereth A., Wahl V. Nitrate acts at the Arabidopsis thaliana shoot apical meristem to regulate flowering time // New Phytol. 2019. V. 223. P. 814. https://doi.org/10.1111/nph.15812
Wopereis J., Pajuelo E., Dazzo F.B., Jiang Q., Gresshoff P.M., De Bruijn F.J., Stougaard J., Szczyglowski K. Short root mutant of Lotus japonicus with a dramatically altered symbiotic phenotype // Plant J. 2000. V. 23. P. 97. https://doi.org/10.1046/j.1365-313x.2000.00799.x
Liu K.H., Niu Y., Konishi M., Wu Y., Du H., Sun Chung H., Li L., Boudsocq M., McCormack M., Maekawa S., Ishida T., Zhang C., Shokat K., Yanagisawa S., Sheen J. Discovery of nitrate-CPK-NLP signalling in central nutrient-growth networks // Nature. 2017. V. 545 P. 311.
Guan P., Ripoll J.-J., Wang R., Vuong L., Bailey-Steinitz L.J., Ye D., Crawford N.M. Interacting TCP and NLP transcription factors control plant responses to nitrate availability // Proc. Natl. Acad. Sci. U. S. A. 2017. V. 114. P. 2419. https://doi.org/10.1073/pnas.1615676114
Hirose N., Takei K., Kuroha T., Kamada-Nobusada T., Hayashi H., Sakakibara H. Regulation of cytokinin biosynthesis, compartmentalization and translocation // J. Exp. Bot. 2008. V. 59. P. 75.
Landrein B., Formosa-Jordan P., Malivert A., Schuster C., Melnyk C.W., Yang W., Turnbull C., Meyerowitz E.M., Locke J.C.W., Jönsson H. Nitrate modulates stem cell dynamics in Arabidopsis shoot meristems through cytokinins // PNAS. 2018. V. 115. P. 1382. https://doi.org/10.1073/pnas.1718670115
Takahashi F., Suzuki T., Osakabe Y., Betsuyaku S., Kondo Y., Dohmae N., Fukuda H., Yamaguchi-Shinozaki K., Shinozaki K. A small peptide modulates stomatal control via abscisic acid in long-distance signalling // Nature. 2018. V. 556. P. 235. https://doi.org/10.1038/s41586-018-0009-2
Ren S.-C., Song X.-F., Chen W.-Q., Lu R., Lucas W.J., Liu C.-M. CLE25 peptide regulates phloem initiation in Arabidopsis through a CLERK-CLV2 receptor complex // J. Integr. Plant Biol. 2019. V. 61. P. 1043. https://doi.org/10.1111/jipb.12846
Goad D.M., Zhu C., Kellogg E.A. Comprehensive identification and clustering of CLV3/ESR-related (CLE) genes in plants finds groups with potentially shared function // New Phytol. 2017. V. 2. P. 605.
Ogilvie H.A., Imin N., Djordjevic M.A. Diversification of the C-TERMINALLY ENCODED PEPTIDE (CEP) gene family in angiosperms, and evolution of plant-family specific CEP genes // BMC Genomics. 2014. V. 15. P. 870.
Delay C., Chapman K., Taleski M., Wang Y., Tyagi S., Xiong Y., Imin N., Djordjevic M.A. CEP3 levels affect starvation-related growth responses of the primary root // J. Exp. Bot. 2019. V. 70. I. 18. P. 4763-4774.
Tabata R., Sumida K., Yoshii T., Ohyama K., Shinohara H., Matsubayashi Y. Perception of root-derived peptides by shoot LRR-RKs mediates systemic N-demand signaling // Science. 2014. V. 346. I. 6207. P. 343.
Ruffel S., Krouk G., Ristova D., Shasha D., Birnbaum K.D., Coruzzi G.M. Nitrogen economics of root foraging: transitive closure of the nitrate-cytokinin relay and distinct systemic signaling for N supply vs. demand // Proc. Natl. Acad. Sci. U. S. A. 2011. V. 108. I. 45. P. 18524.
Ohkubo Y., Tanaka M., Tabata R., Ogawa-Ohnishi M., Matsubayashi Y. Shoot-to-root mobile polypeptides involved in systemic regulation of nitrogen acquisition // Nature Plants. 2017. V. 3. P. 1. https://doi.org/10.1038/nplants.2017.29
Ota R., Ohkubo Y., Yamashita Y., Ogawa-Ohnishi M., Matsubayashi Y. Shoot-to-root mobile CEPD-like 2 integrates shoot nitrogen status to systemically regulate nitrate uptake in Arabidopsis // Nat. Commun. 2020. V. 11. I. 1. P. 641.
Roberts I., Smith S., Stes E., De Rybel B., Staes A., van de Cotte B., Njo M.F., Dedeyne L., Demol H., Lavenus J., Audenaert D., Gevaert K., Beeckman T., De Smet I. CEP5 and XIP1/CEPR1 regulate lateral root initiation in Arabidopsis // J. Exp. Bot. 2016. V. 67. I. 16. P. 4889.
Mohd-Radzman N.A., Laffont C., Ivanovici A., Patel, N., Reid D., Stougaard J., Frugier F., Imin N., Djordjevic M.A. Different pathways act downstream of the CEP peptide receptor CRA2 to regulate lateral root and nodule development // Plant Physiol. 2016. V. 171. I. 2536. https://doi.org/10.1104/pp.16.00113
Imin N., Mohd-Radzman N.A., Ogilvie H.A., Djordjevic M.A. The peptide-encoding CEP1 gene modulates lateral root and nodule numbers in Medicago truncatula // J. Exp. Bot. 2013. V. 64. P. 5395. https://doi.org/10.1093/jxb/ert369
Huault E., Laffont C., Wen J., Mysore K.S., Ratet P., Duc G., Frugier F. Local and systemic regulation of plant root system architecture and symbiotic nodulation by a receptor-like kinase // PLoS Genet. 2014. V. 10(12): e1004891.
Ju C., Chang C. Mechanistic insights in ethylene perception and signal transduction // Plant Physiol. 2015. V. 169. I. 1. P. 85.
Penmetsa R.V., Uribe P., Anderson J., Lichtenzveig J., Gish J.C., Nam Y.W., Engstrom E., Xu K., Sckisel G., Pereira M., Baek J.M., Lopez-Meyer M., Long S.R., Harrison M.J., Singh K.B., Kiss G.B., Cook D.R. The Medicago truncatula ortholog of Arabidopsis EIN2, sickle, is a negative regulator of symbiotic and pathogenic microbial associations // Plant J. 2008. V. 55. I. 4. P. 580.
Lease K.A., Walker J.C. The Arabidopsis unannotated secreted peptide database, a resource for plant peptidomics // Plant Physiol. 2006. V. 142. I. 3. P. 831.
Kehr J., Buhtz A., Giavalisco P. Analysis of xylem sap proteins from Brassica napus // BMC Plant Biol. 2005 V. 5. P. 11.
Alvarez S., Marsh E.L., Schroeder S.G., Schachtman D.P. Metabolomic and proteomic changes in the xylem sap of maize under drought // Plant Cell Environ. 2008. V. 31. I. 3. P. 325.
Okamoto S., Suzuki T., Kawaguchi M., Higashiyama T., Matsubayashi Y. A comprehensive strategy for identifying long-distance mobile peptides in xylem sap // Plant J. 2015. V. 84. P. 611. https://doi.org/10.1111/tpj.13015
de Bang T.C., Lundquist P.K., Dai X., Boschiero C., Zhuang Z., Pant P., Torres-Jerez I., Roy S., Nogales J., Veerappan V., Dickstein R., Udvardi M.K., Zhao P.X., Scheible W.-R. Genome-wide identification of Medicago peptides involved in macronutrient responses and nodulation //Plant Physiol. 2017. V. 175. P. 1669.
Amano Y., Tsubouchi H., Shinohara H., Ogawa M., Matsubayashi Y. Tyrosine-sulfated glycopeptide involved in cellular proliferation and expansion in Arabidopsis // Proc. Natl. Acad. Sci. U. S. A. 2007. V. 104. I. 46. P. 18333.
Fuglsang A.T., Kristensen A., Cuin T.A., Schulze W.X., Persson J., Thuesen K.H., Ytting C.K., Oehlenschlæger C.B., Mahmood K., Sondergaard T.E., Shabala S., Palmgren M.G. Receptor kinase-mediated control of primary active proton pumping at the plasma membrane // Plant J. 2014. V. 80. P. 951.
Pruitt R.N., Joe A., Zhang W., Feng W., Stewart V., Schwessinger B., Dinneny J.R., Ronald P.C. A microbially derived tyrosine-sulfated peptide mimics a plant peptide hormone // New Phytol. 2017. V. 215. I. 2. P. 725.
Nakayama T., Shinohara H., Tanaka M., Baba K., Ogawa-Ohnishi M., Matsubayashi Y. A peptide hormone required for Casparian strip diffusion barrier formation in Arabidopsis roots // Science. 2017. V. 355. I. 6322. P. 284.
Patel N., Mohd-Radzman N.A., Corcilius L., Crossett B., Connolly A., Cordwell S.J., Ivanovici A., Taylor K., Williams J., Binos S., Mariani M., Payne R.J., Djordjevic M.A. Diverse peptide hormones affecting root growth identified in the Medicago truncatula secreted peptidome // Mol. Cell. Proteomics. 2018. V. 17. P. 160. https://doi.org/10.1074/mcp.RA117.000168
Hirakawa Y., Shinohara H., Welke K., Irle S., Matsubayashi Y., Torii K.U., Uchida N. Cryptic bioactivity capacitated by synthetic hybrid plant peptides // Nat. Commun. 2017. V. 8. P. 14318.
Wang X., Mitchum M.G., Gao B., Li C., Diab H., Baum T.J., Hussey R.S., Davis E.L. A parasitism gene from a plant-parasitic nematode with function similar to CLAVATA3/ESR (CLE) of Arabidopsis thaliana // Mol. Plant Pathol. 2005. V. 6. I. 2. P. 187.
Eves-Van Den Akker S., Lilley C.J., Yusup H.B., Jones J.T., Urwin P.E. Functional C-TERMINALLY ENCODED PEPTIDE (CEP) plant hormone domains evolved de novo in the plant parasite Rotylenchulus reniformis // Mol. Plant Pathol. 2016. V. 17. I. 8. P. 1265.
Le Marquer M., Bécard G., Frei Dit Frey N. Arbuscular mycorrhizal fungi possess a CLAVATA3/embryo surrounding region-related gene that positively regulates symbiosis // New Phytol. 2019. V. 222. I. 2. P. 1030.
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


