Физиология растений, 2021, T. 68, № 3, стр. 258-267
Выбор референсных генов для нормализации данных количественной ПЦР в реальном времени у двух форм березы повислой
Ю. Л. Мощенская a, Н. А. Галибина a, *, Т. В. Тарелкина a, К. М. Никерова a, О. В. Чирва a, Л. Л. Новицкая a
a Институт леса — обособленное подразделение Федерального исследовательского центра
“Карельский научный центр Российской академии наук”
Петрозаводск, Россия
* E-mail: galibina@krc.karelia.ru
Поступила в редакцию 28.07.2020
После доработки 06.08.2020
Принята к публикации 06.08.2020
Аннотация
Изучили пригодность пяти генов – GAPDH1, Actin1, Ef1a(1), Ef1a(2), 18SрРНК для нормирования данных экспрессии целевых генов методом обратной транскрипции с последующей ПЦР в реальном времени. С целью обнаружения стабильно экспрессирующихся референсных генов использовали листья и ткани ствола у B. pendula var. pendula и B. pendula var. carelica. Стабильность экспрессии потенциальных референсных генов оценивали с помощью двух пакетов программ NormFinder и BestKeeper. С использованием как стабильно, так и менее стабильно экспрессирующихся референсных генов сравнили экспрессию гена, кодирующего инвертазу клеточной стенки в тканях ствола деревьев Betula pendula Roth в период активного камбиального роста. Показано, что лучшие результаты получают при использовании комбинация двух генов GAPDH1 и Ef1a(1).
ВВЕДЕНИЕ
Береза повислая (Betula pendula Roth), как пионерная порода бореальных лесов Евразии, представляет собой важный источник древесного сырья в Северной Европе [1]. Ценность березы – в ее незаменимости для производства натуральных отделочных материалов (фанера), технологичности производства из нее целлюлозы и гораздо большей, чем у сосны и ели, скорости наращивания древесной массы. Помимо практической значимости, данный вид представляет собой уникальный объект для познания механизмов ксилогенеза и флоэмогенеза, поскольку включает формы, существенно отличающиеся по текстуре древесины. У обычной березы повислой (B. pendula var. pendula) образуется типичная для вида прямослойная древесина, у карельской березы (B. pendula var. carelica) в результате нарушения дифференцировки камбиальных инициалей локально формируется аномальная (узорчатая) древесина [2]. Публикация полного генома B. pendula [1] открыла широкие возможности для изучения профиля экспрессии генов в различных органах и тканях и установления их функции [3–7].
По сравнению с другими методами анализа экспрессии генов, метод обратной транскрипции с последующей ПЦР в реальном времени (qRT-PCR) является предпочтительным в связи с его высокой чувствительностью, специфичностью и меньшим временем, затрачиваемым на эксперимент [8]. Для объективной оценки уровней экспрессии целевых генов необходимо нормирование результатов qRT-PCR по отношению к референсным генам. Пригодный для нормализации референсный ген должен иметь стабильную экспрессию в различных тканях и в различных экспериментальных условиях [9–13]. В качестве референсных, как правило, выбирают гены, участвующие в поддержании клеток, регулирующие основные метаболические функции/пути, такие как синтез компонентов цитоскелета, гликолитический путь, синтез субъединиц рибосом, синтез факторов элонгации, свертывание белков и деградация белков [13]. Вариабельность экспрессии любых референсных генов не является постоянной и в различных экспериментальных условиях может сильно изменяться, поэтому выбор генов для нормализации данных является необходимым этапом эксперимента, и даже применение широко используемых в современных исследованиях референсных генов требует предварительной оценки их стабильности [8, 13–16]. Подбор подходящих референсных генов для изучения экспрессии генов-интереса на растениях разных форм березы повислой (B. pendula var. pendula и var. carelica) ранее не проводился.
С целью выявления референсных генов, пригодных для нормирования уровня транскриптов, мы проанализировали стабильность экспрессии генов ACT1, 18S рРНК, GAPDH1, Ef1a(1), Ef1a(2) в разных тканях (лист, флоэма, ксилема) у двух форм березы повислой, различающихся по структуре образующейся древесины.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал и отбор образцов. Объектами исследования были 13–14-летние деревья B. pendula var. pendula и B. pendula var. carelica. Деревья B. pendula var. pendula (“обычная” береза) имели типичную для вида прямослойную древесину со слабо выраженной текстурой. Поскольку при любом варианте скрещивания родительских форм в потомстве карельской березы появляются особи, как с узорчатой, так и безузорчатой текстурой древесины, среди деревьев карельской березы подбирали экземпляры (1) с высокой степенью узорчатости древесины (узорчатые растения) и (2) растения, у которых признаки аномального строения древесины отсутствовали (безузорчатые растения). Растения выращены из семян, полученных от контролируемого опыления плюсовых деревьев (Forelia OY, Финляндия). Число растений в каждой группе составило 3 штуки. Все растения произрастали на Агробиологической станции КарНЦ РАН в 2 км от Петрозаводска (61°45′ с.ш., 34°20′ в.д.).
Для анализа отбирали листья с удлиненных побегов, ткани ствола (флоэма и ксилема). Образцы флоэмы и ксилемы отбирали на высоте 1.3 м от поверхности земли. У карельской березы выбирали участки с наибольшей степенью проявления структурных аномалий проводящих тканей. Для отбора образцов на стволе вырезали окошки 2 × 6 см и отделяли кору от древесины. С обнаженной поверхности древесины бритвенным лезвием соскабливали тонкий слой, куда входили дифференцирующиеся клетки ксилемы и наружные слои прироста ксилемы текущего года. С внутренней поверхности коры препарировали комплекс тканей, включающий камбиальную зону, проводящую флоэму и самые внутренние слои непроводящей флоэмы. В дальнейшем эти ткани обозначены как “флоэма”. Весь растительный материал замораживали в жидком азоте и хранили в низкотемпературной морозильной камере при –80°C. Отбор образцов тканей ствола контролировали под световым микроскопом. Анализ срезов показал, что для всех исследуемых растений был характерен активный камбиальный рост.
Выделение и очистка суммарной РНК, синтез кДНК. Для выделения суммарной РНК навеску растительной ткани (50–100 мг) растирали в жидком азоте. Процедуру выделения проводили с использованием двух лизирующих буферов (на основе цетилтриметиламмонийбромида (ЦТАБ) и додецилсульфата натрия (ДДС)) и хлороформ-изоамиловой смеси (24 : 1) по методике, описанной М. Су с соавт. [18], дополнительно лизат ткани обрабатывали ингибитором РНКаз для предотвращения возможной деградации проб и ДНКазой (“Синтол”, Россия) для избавления от примеси геномной ДНК. Соотношение ткань/буфер составило 1 : 10. Состав экстракционного ЦТАБ буфера: 100 мМ Tris–HCl (pH 8.0); 25 мМ ЭДТА, 2 M NaCl, 2% CTAB, 2% PVP, 2% меркаптэтанол (добавляют перед использованием); экстракционного ДДС буфера: 10 мМ Tris–HCl, 1 мМ ЭДТА, 1 M NaCl, 0.5% ДДС.
Осаждение РНК проводили 100% изопропанолом. Осадок РНК растворяли в необходимом объеме свободной от РНКаз воды. Целостность полученной РНК анализировали при помощи метода электрофореза в 1% агарозном геле. Определение концентрации РНК проводили спектрофотометрически (планшетный спектрофотометр SPECTROstar NANO, “BMG Labtech”, Германия) по поглощению на длине волны 260 нм. Чистоту полученного препарата оценивали по соотношению оптических плотностей, измеренных при 230, 260 и 280 нм (А260/А280 и А260/А230). Отсутствие примесей геномной ДНК подтверждали с помощью постановки ПЦР с использованием в качестве матрицы полученного препарата РНК.
кДНК синтезировали согласно протоколу производителя, используя набор реактивов для обратной транскрипции MMLV RT kit (“Евроген”, Россия) в 20 мкл реакционной смеси, содержащей 6 мкл РНК-матрицы (1 мкг), 1 мкл Oligo(dT) праймера и 1 мкл случайного праймера (Randoom (dN)10-primer) (20 мкМ), 1 мкл обратной транскриптазы (MMLV ревертазы) (100 ед.), 4 мкл 5х буфера для синтеза первой цепи кДНК (280 мМ Трис-HCl, pH 8.7; 37 5 мМ КСl; 30 мМ MgCl2), 2 мкл смеси dNTP (10 мМ каждого), 2 мкл DDT (20 мМ), 3 мкл стерильной, свободной от РНКаз воды. Реакцию обратной транскрипции проводили на амплификаторе QuantStudio 5 (“Thermo scientific”, США).
Поиск генов в геноме березы повислой методами биоинформатики. В качестве референсных генов были выбраны гены рибосомной РНК 18S (18S рРНК), актина (ACT1), фактора элонгации 1ɑ (Ef1a(1), Ef1a(2)), глицеральдегид-3-фосфатдегидрогеназы (GAPDH1) – как наиболее часто используемые для этой цели [18–20 и др.].
Поиск генов проводили в геноме березы повислой (Betula pendula Roth), опубликованном на портале CoGe [1]. С этой целью были получены нуклеотидные последовательности CDS и соответствующие им белковые последовательности генов Arabidopsis thaliana и Populus trichocarpa из баз данных The Arabidopsis Information Resource (TAIR) (release 13, https://www.arabidopsis.org) и Phytozome (release 3.0, http://www.phytozome.net/ poplar). Указанные последовательности использовали в качестве поискового запроса BLAST по геному B. pendula (release 1.2, https://genomevolution.org/coge) для выявления гомологичных последовательностей.
Предсказание структуры белков березы повислой выполняли с использованием ресурса National Center for Biotechnology Information (NCBI) (http:// www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml). Филогенетический анализ проводили с помощью программы MEGA X [21]. Множественное выравнивание последовательностей осуществляли с помощью алгоритма ClustalW. Филогенетическое древо было построено с использованием метода присоединения ближайшего соседа (Neighbor-Joining method), оценку достоверности кластеризации проводили методом bootstrap с 1000 повторами [22, 23]. Определение процента идентичности/сходства последовательностей выполняли с помощью ресурса EMBOSS Needle (https:// www.ebi.ac.uk/Tools/psa/emboss_needle/).
Подбор праймеров для qRT-PCR и их проверка. Специфические праймеры (“Синтол”, Россия) для амплификации участков исследуемых референсных генов конструировали с помощью программного обеспечения Beacon Designer 8.21 (PREMIER Biosoft). С целью подтвердить, что получен единственный продукт реакции, проводили анализ кривой плавления, а также электрофорез продуктов количественной ПЦР в 8% акриламидном геле с окраской бромистым этидием.
Условия количественной ПЦР в режиме реального времени. Амплификацию образцов проводили на амплификаторе QuantStudio 5 (“Thermo scientific”, США), используя наборы для амплификации с интеркалирующим красителем SYBR Green – qPCRmix-HS SYBR (“Евроген”, Россия). ПЦР-РВ проводили в реакционной смеси объемом 25 мкл, содержащей 5 мкл qPCRmix-HS SYBR, по 1 мкл прямого и обратного праймера (0.4 мкМ) (“Синтол”, Россия), 2 мкл матрицы кДНК, 16 мкл деионизированной, свободной от нуклеаз воды. Конечное содержание кДНК в реакционной среде для всех образцов составляло 100 нг, согласно рекомендации протокола производителя. ПЦР проводили при следующих условиях: 95°C в течение 5 мин, затем 40 циклов по 95°C в течение 15 с для денатурации, 55°C в течение 30 с для отжига и 72°C в течение 30 с для элонгации. Для каждой пары праймеров применяли отрицательный контроль – постановка ПЦР в отсутствие матрицы кДНК.
Анализ стабильности экспрессии генов. Специфичность полученных продуктов ПЦР оценивали с помощью анализа кривых плавления. Для определения эффективности (Е) ПЦР проводили c каждой парой праймеров на серии 10-кратных разведений (10–1, 10–2, 10–3, 10–4 и 10–5) кДНК. Для определения значений Сt строили калибровочные кривые, используя программное обеспечение Excel, в соответствии с коэффициентами корреляции R и значениями наклона кривой (slope, к), которые можно рассчитать по калибровочным кривым значений Ct для каждого гена. Эффективность ПЦР-амплификации (Е) рассчитывали по формуле E = (10^(–1/к) – 1) × 100.
Поиск подходящего референсного гена проводили с помощью двух пакетов программ NormFinder и BestKeeper, анализируя стабильность экспрессии каждого претендента на эту роль. Исходные значения Ct переводили в формат, необходимый для анализа в NormFinder. Максимальный уровень экспрессии (наименьшее значение Ct) принимали равным 1. Относительные уровни экспрессии рассчитывали, используя формулу: 2–ΔgCt, в которой ΔgCt – это наименьшее значение Ct, вычтенное из соответствующего значения Ct для каждого гена и каждого образца. Отобранные NormFinder гены анализировали с помощью BestKeeper с учетом непреобразованных значений Сt и эффективности амплификации.
Проверка референсных генов. С использованием потенциальных референсных генов сравнили относительную экспрессию гена CWIN1, кодирующего изоформу апопластной инвертазы (АпИнв), в тканях ствола растений обычной березы и узорчатых растений карельской березы. Ранее было показано, что возрастание узорчатости древесины у карельской березы коррелирует с увеличением активности АпИнв [4]. Относительный уровень транскриптов генов CWIN1 (RQ) вычисляли по формуле:
Статистическая обработка данных. Статистическая обработка данных осуществлялась в среде Microsoft Excel и PAST. На диаграммах приведены средние значения с учетом трехкратной биологической и трехкратной аналитической повторностей и их стандартные отклонения. Выборки проверялись на нормальность с использованием критерия Шапиро-Уилка. Для оценки различий использовали критерий Манна-Уитни (обозначение на графиках латинскими буквами). Статистически значимыми считали различия при P < 0.05.
РЕЗУЛЬТАТЫ
Оценка качества РНК. Оценка качества полученной РНК при помощи электрофореза в 1% агарозном геле с визуализацией бромистым этидием позволила выявить отчетливое разделение полос 18S и 28S рРНК, что свидетельствует о хорошем качестве суммарной РНК. Отношение оптических плотностей, измеренные на длинах волн 260 и 230 нм (А260/А230), показывает уровень загрязнения препарата РНК полисахаридами и полифенолами, а их отношение при 260 и 280 нм (А260/А280) – загрязнение образца белками. Полученные нами для всех органов и тканей значения А260/А230 были выше 2, а значения А260/А280 составляли 1.97–2.0 (табл. 1), что говорит об отсутствии загрязнения препарата РНК полифенолами, полисахаридами и белками. Отсутствие примеси геномной ДНК в полученных препаратах подтверждали также постановкой ПЦР-РВ с использованием в качестве матрицы полученной суммарной РНК, при этом не было зафиксировано появление ПЦР продукта после 40 циклов реакции.
Таблица 1.
Спектрофотометрические показатели качества суммарной РНК, полученной из растений двух форм Betula pendula Roth.
| Объект | Ткань/орган | А260/А280 | А260/А230 |
|---|---|---|---|
| Betula pendula var. pendula | Лист | 1.980 ± 0.002 | 2.033 ± 0.020 |
| Флоэма | 1.968 ± 0.005 | 2.036 ± 0.011 | |
| Ксилема | 1.985 ± 0.006 | 2.022 ± 0.001 | |
| Betula pendula var. carelica | Лист | 1.986 ± 0.002 | 2.069 ± 0.020 |
| Флоэма | 1.972 ± 0.002 | 2.070 ± 0.021 | |
| Ксилема | 1.979 ± 0.003 | 2.057 ± 0.019 |
Поиск последовательностей потенциальных референсных генов и подбор праймеров для количественной ПЦР в режиме реального времени. В геноме березы повислой нам не удалось идентифицировать последовательности, обладающие достаточно высоким уровнем сходства с последовательностями 18S рРНК других видов. В базе данных NCBI была найдена частичная последовательность 18S рРНК березы повислой (номер доступа в базе данных NCBI GU476453.1) длиной 1493 п.н., которая была использована для конструирования праймеров для количественной ПЦР [24]. На основе последовательности GU476453.1 нами были сконструированы праймеры для ПЦР-РВ, используемые в данной работе (табл. 3).
Таблица 2.
Результаты попарного выравнивания аминокислотных последовательностей белков B. pendula и A. thaliana
| ID гена, кодирующего белок B. pendula | Ближайший гомологичный белок A. thaliana (номер локуса в TAIR) | Процент идентичности аминокислотных последовательностей белков B. pendula и A. thaliana |
|---|---|---|
| Bpev01.c0427.g0027.m0001 | Actin 1 (AT2G37620) | 93.8 |
| Bpev01.c1225.g0009.m0001 | Actin 7 (AT5G09810) | 98.7 |
| Bpev01.c0556.g0017.m0001 | Actin 7 (AT5G09810) | 97.6 |
| Bpev01.c0029.g0002.m0001 | Actin 11 (AT3G12110) | 97.1 |
| Bpev01.c0564.g0031.m0001 | Actin 11 (AT3G12110) | 96.8 |
| Bpev01.c0365.g0049.m0001 | Actin 12 (AT3G46520) | 97.1 |
| Bpev01.c0052.g0178.m0001 | Сytosolic GAPDH 1 (AT3G04120) | 89.1 |
| Bpev01.c1040.g0016.m0001 | Сytosolic GAPDH 1 (AT3G04120) | 89.4 |
| Bpev01.c1513.g0002.m0001 | Сytosolic GAPDH 1 (AT3G04120) | 87.9 |
| Bpev01.c0288.g0008.m0001 | EF1a (AT1G07940) | 95.3 |
| Bpev01.c0437.g0013.m0001 | EF1a (AT1G07940) | 94.2 |
| Bpev01.c0437.g0013.m0002 | EF1a (AT1G07940) | 94.0 |
| Bpev01.c0437.g0018.m0001 | EF1a (AT1G07940) | 94.2 |
В геноме березы повислой было идентифицировано по 6 генов, кодирующих белки, гомологичные актину и GAPDH, и 4 гена, кодирующих белки, гомологичные Ef1a A. thaliana и P. trichocarpa (рис. 1). Аминокислотные последовательности предсказанных белков B. pendula продемонстрировали высокий процент идентичности с последовательностями гомологичных белков A. thaliana (табл. 2). Для дальнейшего анализа были отобраны гены из каждого семейства и сконструированы специфичные пары праймеров (табл. 3). Размер продуктов количественной ПЦР для всех исследуемых генов находился в диапазоне 94–188 п.н., а температура отжига праймеров имела диапазон от 52 до 60°C.
Рис. 1.
Филогенетические деревья, построенные на основе сравнительного анализа аминокислотных последовательностей известных белков Arabidopsis thaliana (светлые точки) и Populus trichocarpa (серые точки) и потенциальных белков Betula pendula (черные точки). а – актин, б – GAPDH, в – Ef1a. Длина ветвей пропорциональна генетической дистанции (число замещений на сайт). Существенные значения bootstrap (процент деревьев, в которых ассоциированные таксоны кластеризуются вместе) для 1000 выборок показаны в основании ветви. Рядом с названиями белков A. thaliana и P. trichocarpa указаны коды доступа в базах данных TAIR и Phytozome.
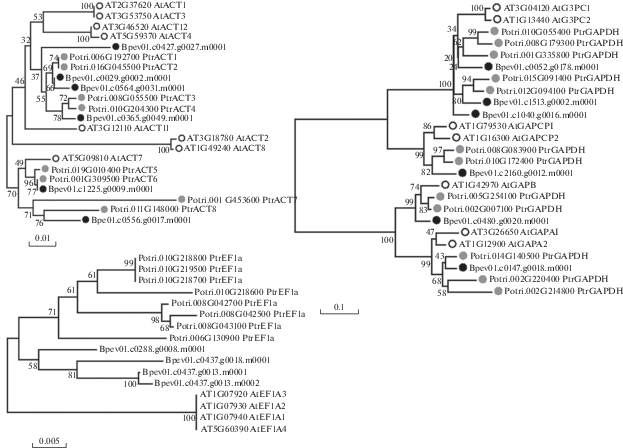
Таблица 3.
Описание референсных (18S рРНК, Actin1, Ef1а(1), Ef1a(2), GapDH1) и целевого (CWIN1) генов. Тm – температуры плавления; R2 – коэффициент корреляции порогового цикла и логарифма концентрации ДНК матрицы
| Ген | Локус/номер доступа (NCBI) | Праймер (5′ → 3′) (прямой / обратный) |
Длина продукта (п.н.) |
Tm, °C | Эффективность амплификации, % |
R2 |
|---|---|---|---|---|---|---|
| 18S рРНК | GU476453.1 | CTACTATGGTGGTGACGGGTGAC CGCCTGCTGCCTTCCTTGG |
94 | 60 | 98.8 | 0.993 |
| Actin1 | Bpev01.c0427.g0027 | GGTGGTGAATGAGTAGCC TTCTTTCCCTTTATGCC |
167 | 54 | 100.7 | 0.998 |
| Ef1а(1) | Bpev01.c0437.g0013.m0001 | TGTGCTGTTCTTATCATTGACTCC TTCCTCGTACCTTGCCTTGG |
145 | 54 | 98.7 | 0.989 |
| Ef1a(2) | Bpev01.c0437.g0018.m0001 | GCCACACCTGCCACATTGC AGCGAAAGTCTCCACCACCATAG |
188 | 52 | 98.5 | 0.999 |
| GapDH1 | Bpev01.c1040.g0016.m0001 | AGAATACAAGCCAGAACTCAAC CTCTACCACCTCTCCAATCC |
188 | 54 | 98.7 | 0.984 |
| CWIN1 | Bpev01.c0333.g0031.m0001 | AGTGCCCCGATTTCTTCCCTG GTCCACCTGCCCCTTGTCCG |
181 | 55 | 99.0 | 0.990 |
Определение эффективности и специфичности амплификации ПЦР. Прежде чем оценивать стабильность экспрессии изучаемых референсных генов, мы установили эффективность амплификации в ходе ПЦР. Полученные значения эффективности (E) находились в диапазоне от 98.5 до 100% для всех рассмотренных генов (табл. 3). В ходе оценки специфичности ПЦР при помощи гель-электрофореза в 8% акриламидном геле были обнаружены ампликоны ожидаемой длины и показано отсутствие димеров праймеров. При анализе кривых плавления продуктов ПЦР с каждой парой праймеров был выявлен один пик и не обнаружено неспецифических продуктов реакции (рис. 2).
Рис. 2.
Специфичность амплификации и кривые плавления продуктов ПЦР потенциальных референсных генов.

Анализ стабильности экспрессии потенциальных референсных генов. Значения пороговых циклов (Ct), полученные для различных образцов, позволили выявить существенные различия в уровне транскрипции исследуемых генов (рис. 3а). Полученные для 5 генов значения Ct находились в диапазоне от 8 до 35 циклов. Ген 18S рРНК имел самые низкие значения Ct (9.9), что говорит о наиболее высоком уровне транскрипции. Самый низкий уровень экспрессии был показан для гена GAPDH1 (27.7).
Рис. 3.
Значения Ct референсных генов (а) и стабильность их экспрессии, определенная с помощью программы NormFinder (б).
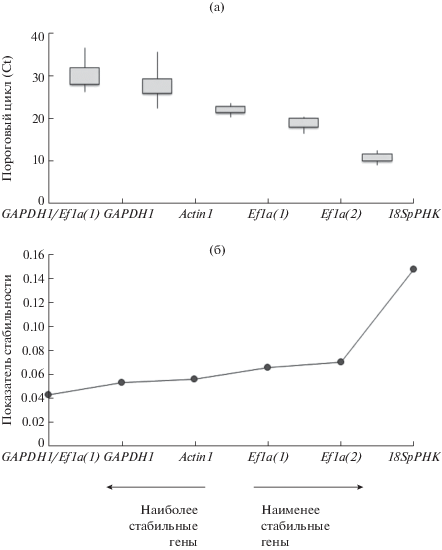
Анализ стабильности экспрессии потенциальных референсных генов при помощи программы NormFinder позволил выявить, что наилучшими референсными генами для проведения ПЦР на различных образцах B. pendula Roth являются GAPDH1 и Actin1, имеющие сходные значения показателя стабильности (0.054 и 0.056, соответственно), затем располагались Ef1a(1) и Ef1a(2) (0.067 и 0.071, соответственно) и 18SрРНК (0.148) (рис. 3б).
Используемая нами программа NormFinder оценивает стабильность экспрессии на основе математической модели [25]. Данная программа выстраивает исследуемые гены на основе их минимальной меж- и внутригрупповой вариации. Поскольку в данной работе по оценке пригодности референсных генов мы использовали образцы разных форм березы повислой, то выборка образцов (n = 30) была разделена на две группы (n = 15) – образцы B. pendula var. pendula и B. pendula var. carelica. В результате оценки стабильности экспрессии потенциальных референсных генов и их внутри- и межгрупповой вариации, референсные гены GAPDH1 и Ef1a(1) были предложены программой NormFinder как лучшая комбинация двух генов. Показатель стабильности данных генов составил при этом 0.044.
Оценка референсных генов при помощи программы BestKeeper основана на анализе пороговых значений Ct, по которым рассчитывается коэффициент вариации (CV), стандартное отклонение (SD) и коэффициент корреляции (r) для каждого гена в ряду данных [26]. Наименьшие значения SD и наиболее близкий к 1 показатель r имеют наиболее стабильно экспрессирующиеся гены. По результатам анализа NormFinder исключили наименее стабильно экспрессирующийся ген 18SрРНК, остальные гены ранжировали в соответствии со значениями CV ± SD в образцах. Анализ исследуемых генов при помощи программы BestKeeper показал, что наиболее стабильными генами с наименьшим показателем SD являются гены Act-in1, GAPDH1, Ef1a(1), наименее стабильным оказался ген Ef1a(2).
Таким образом, анализ стабильности экспрессии референсных генов при помощи двух пакетов программ показал сходные результаты.
Экспрессия гена, кодирующего апопластную инвертазу, в тканях ствола двух форм березы повислой относительно потенциальных референсных генов. В результате экспериментальной проверки потенциальных референсных генов было показано, что использование для расчета относительного уровня транскрипции референсных генов с разной стабильностью экспрессии может приводить к значительному искажению результатов. При использовании стабильных референсных генов (GAPDH1/Ef1a(1); GAPDH1 и Ef1a(1)) закономерности в распределении экспрессии CWIN1 у двух форм березы повислой были схожи. Так, у карельской березы, по сравнению с “обычной” березой, относительный уровень транскриптов CWIN1 был в 3.5–4 и 20–30 раз больше в тканях ксилемы и флоэмы, соответственно (рис. 4а, б, в), что согласуется с нашими предыдущими данными [4].
Рис. 4.
Относительные уровни экспрессии (отн. ед) целевого гена (CWIN1.1), рассчитанные посредством нормализации на различные референсные гены в ксилеме деревьев Betula pendula Roth. var pendula (светлые столбики) и var carelica (темные столбики). Бары – стандартные отклонения, рассчитанные на основании трех биологических и трех аналитических повторностей.

При использовании в качестве внутреннего контроля ACT1 и наименее стабильного гена Ef1a(2) достоверных отличий в экспресии CWIN1 в ксилеме между растениями карельской и “обычной” березой не наблюдали (рис. 4г, д)
Относительный уровень транскрипции гена CWIN1 при использовании в качестве референсного гена 18SрРНК был близок к нулю, что значительно усложняет оценку различий в экспрессии целевого гена. Кроме того, как и в случае с ACT1 и Ef1a(2), не наблюдали отличий между узорчатой древесиной B. pendula var. carelica и прямоствольной древесиной B. pendula var. pendula (рис. 4е).
ОБСУЖДЕНИЕ
Универсального референсного гена, имеющего стабильную экспрессию у разных видов растений, не существует. Ранее для растений B. pendula при нормализации данных количественной ПЦР-РВ использовали гены Actin [4, 6, 29], Tubulin [5, 28], GAPDH [27] и 18SрРНК [7, 30]. В проведенном исследовании у двух форм березы повислой была впервые оценена пригодность пяти генов – GAPDH1, Actin1, Ef1a(1), Ef1a(2), 18SрРНК для использования в качестве референсных при постановке qRT-PCR, основываясь на стабильности их экспрессии.
Результаты исследования уровня экспрессии потенциальных референсных генов были проанализированы при помощи программ NormFinder и BestKeeper. Данные программы используют различные алгоритмы оценки стабильности референсных генов, в связи с чем возможны некоторые различия в результатах. В нашем исследовании данные, полученные при помощи данных программ, различались незначительно. Было показано, что из числа изученных генов наиболее стабильно экспрессируемыми в образцах B. pendula являются Actin1, GAPDH1, Ef1a(1), при этом гены GAPDH1 и Ef1a(1) были предложены программой NormFinder как лучшая комбинация двух референсных генов и имели наиболее низкое значение показателя стабильности.
Согласно результатам нашего исследования, наименее стабильно экспрессирующимся оказался ген 18SрРНК. Несмотря на частое использование этого гена в качестве референсного, встречаются и противоположные данные, согласно которым 18SрРНК не может служить в качестве внутреннего контроля, поскольку он транскрибируется РНК-полимеразой I, тогда как мРНК синтезируются РНК-полимеразой II [8]. Согласно полученным данным, 18SрРНК имеет самые низкие значения Сt, что при расчетах приводит к очень низким значениям относительного уровня экспрессии целевого гена и искажению результатов. На основании вышеперечисленных данных ген 18SрРНК был исключен из числа возможных референсных генов для проведения ПЦР-РВ на образцах B. pendula.
Помимо проверки стабильности экспрессии референсных генов нами была проверена надежность и целесообразность использования описанных генов в качестве референсных. Показано, что использование различных генов в качестве референсных без предварительной оценки стабильности их экспрессии может приводить к значительному искажению данных об уровне транскрипции целевого гена.
Финансовое обеспечение исследований осуществлялось из средств федерального бюджета на выполнение государственного задания Карельского научного центра РАН (Институт леса КарНЦ РАН) и при частичной финансовой поддержке Российского фонда фундаментальных исследований (№ 19-04-00622_a).
Авторы заявляют об отсутствии конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Список литературы
Salojärvi J., Smolander O.-P., Nieminen K., Rajaraman S., Safronov O., Safdari P., Lamminmäki A., Immanen J., Lan T., Tanskanen J., Rastas P., Amiryousefi A., Jayaprakash B., Kammonen J.I., Hagqvist R. et al. Genome sequencing and population genomic analyses provide insights into the adaptive landscape of silver birch // Nat. Genet. 2017. V. 49. P. 904. https://doi.org/10.1038/ng.3862
Novitskaya L.L., Kushnir F.V. The role of sucrose in regulation of trunk tissue development in Betula pendula Roth // Plant Growth Regul. 2006. V. 25. P. 18. https://doi.org/10.1007/s00344-004-0419-2
Moshchenskaya Y.L., Galibina N.A., Topchieva L.V., Novitskaya L.L. Expression of genes encoding sucrose synthase isoforms during anomalous xylogenesis in Karelian birch // Russ. J. Plant Physiol. 2017. V. 64. P. 616. https://doi.org/10.1134/S1021443717030104
Galibina N.A., Novitskaya L.L., Nikerova K.M., Moshchenskaya Y.L., Borodina M.N., Sofronova I.N. Apoplastic invertase activity regulation in the cambial zone of Karelian birch // Russ. J. Dev. Biol. 2019. V. 50. P. 20. https://doi.org/10.1134/S1062360419010028
Alonso Serra J., Safronov O., Lim K.J., Fraser Miller S.J., Blokhina O.B., Campilho A., Chong S.L., Fagerstedt K., Haavikko R., Helariutta Y., Immanen J., Kangasjärvi J., Kauppila T.J., Lehtonen M., Ragni L. et al. Tissue specific study across the stem reveals the chemistry and transcriptome dynamics of birch bark // New Phytol. 2019. V. 222. P. 1816. https://doi.org/10.1111/nph.15725
Novitskaya L.L., Tarelkina T.V., Galibina N.A., Moshchenskaya Y.L., Nikolaeva N.N., Nikerova K.M., Podgornaya M.N., Sofronova I.N., Semenova L.I. The formation of structural abnormalities in Karelian birch wood is associated with auxin inactivation and disrupted basipetal auxin transport // Plant Growth Regul. 2020. V. 39. P. 378. https://doi.org/10.1007/s00344-019-09989-8
Qu C., Bian X., Han R., Jiang J., Yu Q., Liu G. Expression of BpPIN is associated with IAA levels and the formation of lobed leaves in Betula pendula ‘Dalecartica’ // J. For. Res. 2020. V. 31. P. 87. https://doi.org/10.1007/s11676-018-0865-5
Zhou L.N., Yao W.F., Liu J., Shang J., Shan M.Q., Zhang L., Ding A.W. Protective effect of different solvent extracts from platycladi cacumen carbonisatum on LPS-induced human umbilical vein endothelial cells damage // China journal of Chinese materia medica. 2013. V. 38. P. 3933.
Nicot N., Hausman J.F., Hoffmann L., Evers D. Housekeeping gene selection for real-time RT-PCR normalisation in potato during biotic and abiotic stress // J. Exp. Bot. 2005. V. 56. P. 2907. https://doi.org/10.1093/jxb/eri285
Exposito-Rodriguez M., Borges A.A., Perez A.B., Perez J.A. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process // BMC Plant Biol. 2008. V. 8: e131. https://doi.org/10.1186/1471-2229-8-131
Paolacci A.R., Tanzarella O.A., Porceddu E., Ciaffi M. Identification and validation of reference genes for quantitative RT-PCR normalization in wheat // BMC Molecular Biol. 2009. V. 10: e11. https://doi.org/10.1186/1471-2199-10-11
Li M.Y., Wang F., Jiang Q., Wang G.L., Tian C., Xiong A.-S. Validation and Comparison of Reference Genes for qPCR Normalization of Celery (Apium graveolens) at Different Development Stages // Front. Plant Sci. 2016. V. 7: e313. https://doi.org/10.3389/fpls.2016.00313
Jaiswal P.S., Kaur N., Randhawa G. Identification of reference genes for real-time PCR gene expression studies during seed development and under abiotic stresses in Cyamopsis tetragonoloba (L.) Taub. // bioRxiv. 2018: e313437. https://doi.org/10.1101/313437
Chandna R., Augustine R., Bisht N.C. Evaluation of candidate reference genes for gene expression normalization in Brassica juncea using real time quantitative RT-PC-R // PLoS ONE. 2012. V. 7: e36918. https://doi.org/10.1371/journal.pone.0036918
Manoli A., Sturaro A., Trevisan S., Quaggiotti S., Nonis A. Evaluation of candidate reference genes for qPCR in maize // J. Plant Physiol. 2012. V. 169. P. 807. https://doi.org/10.1016/j.jplph.2012.01.019
Reddy P.S., Rao T.S.R.B., Sharma K.K., Vadez V. Genomewide identification and characterization of the aquaporin gene family in Sorghum bicolor (L.) // Plant Gene. 2015. V. 1. P. 8. https://doi.org/10.1016/j.plgene.2014.12.002
Су М., Цзан В., Яо Н., Хуан М. Выделение высококачественной РНК из различных тканей Populus // Физиология растений. 2009. Т. 56. С. 791.
Han X., Lu M., Chen Y., Zhan Z., Cui Q., Wang Y. Selection of reliable reference genes for gene expression studies using real-time PCR in tung tree during seed development // PLoS ONE. 2012. V. 7: e43084. https://doi.org/10.1371/journal.pone.0043084
Yang M., Wu S., You W., Jaisi A., Xiao Y. Selection of Reference Genes for Expression Analysis in Chinese Medicinal Herb Huperzia serrata // Front. Pharmacol. 2019. V. 10. P. 1. https://doi.org/10.3389/fphar.2019.00044
Zhou J., Yuan J., Long H., Tan X. Expression profiles of a tung tree phosphate transporter cdna and structural characteristics of the encoded protein // Cytol. Genet. 2019. V. 53. P. 162. https://doi.org/10.3103/S0095452719020105
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms // Mol. Biol. Evol. 2018. V. 35. P. 1547. https://doi.org/10.1093/molbev/msy096
Felsenstein J. Confidence limits on phylogenies: an approach using the bootstrap // Evolution. 1985. V. 39. P. 783. https://doi.org/10.1111/j.1558-5646.1985.tb00420.x
Saitou N., Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees // Mol. Biol. Evol. 1987. V. 4 P. 406. https://doi.org/10.1093/oxfordjournals.molbev.a040454
Zhang W., Wei R., Chen S., Jiang J., Li H., Huang H., Yang G., Wang S., Wei H., Liu G. Functional characterization of CCR in birch (Betula platyphylla × Betula pendula) through overexpression and suppression analysis // Physiol. Plant. 2015. V. 154. P. 283. https://doi.org/10.1111/ppl.12306
Andersen C.L., Jensen J.L., Orntoft T.F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets // Cancer Res. 2004. V. 64. P. 5245. https://doi.org/10.1158/0008-5472.CAN-04-0496
Кулакова Н.В., Болотова Т.А., Ханаев И.В., Черногор Л.И., Беликов С.И. Выбор референсных генов для количественной ПЦР в режиме реального времени в губке Lubomirskia baicalensis // Internetion journal of applied and fundamental research. 2016. V. 1. P. 360.
Гродецкая Т.А., Евлаков П.М., Исаков И.Ю. Анализ экспрессии генов стрессоустойчивости в условиях воздействия засухи на растения березы в центрально-черноземном регионе // Лесотехнический журнал. 2020. № 2. С. 23. https://doi.org/10.34220/issn.2222-7962/2020.2/3
Keinanen S.I., Hassinen V.H., Karenlampi S.O., Tervahauta A.I. Isolation of genes up-regulated by copper in a copper-tolerant birch (Betula pendula) clone // Tree Physiol. 2007. V. 27. P. 1243. https://doi.org/10.1093/treephys/27.9.1243
Morales L.O., Tegelberg R., Brosche M., Keinanen M., Lindfors A., Aphalo P.J. Effects of solar UV-A and UV-B radiation on gene expression and phenolic accumulation in Betula pendula leaves // Tree Physiol. 2010. V. 30. P. 923. https://doi.org/10.1093/treephys/tpq051
Kosonen M., Lännenpää M., Ratilainen M., Kontunen-Soppela S., Julkunen-Tiitto R. Decreased anthocyanidin reductase expression strongly decreases silver birch (Betula pendula) growth and alters accumulation of phenolics // Physiol. Plantarum. 2015. V. 155. P. 384. https://doi.org/10.1111/ppl.12324
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


