Физиология растений, 2022, T. 69, № 3, стр. 261-271
Фотопериод-зависимые механизмы инициации цветения Arabidopsis thaliana L. и Zea mays L.
А. В. Щенникова *
Институт биоинженерии Федерального исследовательского центра
“Фундаментальные основы биотехнологии” Российской академии наук
Москва, Россия
* E-mail: shchennikova@yandex.ru
Поступила в редакцию 02.06.2021
После доработки 03.08.2021
Принята к публикации 04.08.2021
- EDN: TSPAQF
- DOI: 10.31857/S0015330322020166
Аннотация
Одомашнивание растений включает их адаптацию к различным климатическим условиям, в том числе к изменениям фотопериода и температуры. Многие генетические локусы одомашнивания ортологичны у отдаленных таксонов. Основные молекулярно-генетические механизмы цветения детально охарактеризованы у модельного растения Arabidopsis thaliana L., что составляет основу для изучения и построения моделей цветения других видов растений. Одомашнивание и селекция кукурузы Zea mays L. привели к тому, что сегодня у данного вида наблюдается большое разнообразие реакций на продолжительность светового дня при сохранении основного каркаса генетической сети, контролирующей чувствительность к фотопериоду. В обзоре рассмотрены особенности реакции на фотопериод растений кукурузы в сравнении с модельным видом Arabidopsis, включая фенологические гены для селекции кукурузы, являвшиеся мишенью отбора в процессе адаптации Zea mays к новым эколого-географическим условиям.
ВВЕДЕНИЕ
На сегодняшний день основными источниками питания человека являются зерновые, главные из которых – пшеница, рис, кукуруза и другие культуры. Согласно прогнозам, таковыми они останутся и в будущем в условиях непрерывного роста населения мира и меняющегося климата [1]. Критическими факторами при выращивании зерновых являются время перехода растения к репродуктивному развитию и длительность фазы налива зерна. Оба фактора чувствительны к циркадным ритмам, связанным с суточными и сезонными колебаниями таких параметров как фотопериод, температура и др. [2].
Чувствительные к фотопериоду растения делятся на виды длинного (LD) и короткого (SD) дня, цветение которых наступает при, соответственно, превышении и снижении пороговых значений продолжительности светового дня, определяемых, в первую очередь, регионом происхождения вида [3]. Одомашненные виды зерновых, давшие начало современным сельскохозяйственным культурам, проходили через дополнительную селекцию человеком с целью получения сортов, способных пройти полный цикл развития до созревания семян за пределами эколого-географической ниши, освоенной диким предком. Доместикация трав Poaceae произошла, предположительно, около 15 тыс. лет назад в Восточном Средиземноморье, и последующая селекция в различных эколого-географических зонах привела к диверсификации видов зерновых и возможности их выращивания в самых разных сезонно-климатических условиях [1, 4, 5].
При описании механизмов доместикации растений широко используется понятие генетических локусов одомашнивания, многие из которых ортологичны у отдаленных таксонов [6]. Это позволяет переносить генетические данные, полученные на одних видах растений, на другие виды (включая сельскохозяйственные культуры) в процессе их изучения.
Наиболее детально основные генетические и молекулярные механизмы фотопериод-зависимого цветения охарактеризованы у модельного растения Arabidopsis thaliana L., являющегося растением LD [7]. Полученные знания составили основу для изучения и построения моделей ответа на фотопериод других видов растений, чему значительно способствовало секвенирование и аннотация их геномов и транскриптомов. С помощью такого подхода были идентифицированы ортологи генов, ассоциированных с фотопериодом, у однодольных культур, как LD (пшеница Triticum aestivum L. и ячмень Hordeum vulgare L.), так и SD (кукуруза Zea mays L., 1753 и рис Oryza sativa L.). Характеристика данных генов позволила выделить функционально как консервативные, так и дивергентные гены времени цветения, а также предложить возможные регуляторные механизмы, которые в ответ на эндогенные и экзогенные сигналы определяют пластичность перехода зерновых культур к репродуктивному развитию.
Кукуруза (Zea mays L., 1753) имеет тропическое происхождение и, как следствие, теплолюбива и приспособлена к короткому световому дню [8]. Одомашнивание и селекция данной культуры привели к тому, что сегодня у данного вида наблюдается большое разнообразие реакций на продолжительность светового дня при сохранении основного каркаса генетической сети, контролирующей чувствительность к фотопериоду. При продвижении на север, в более высокие широты, сокращается период вегетации Z. mays за счет короткого лета и, как следствие, значительно снижается урожай зерна. Поэтому селекционный процесс все последнее столетие сводился к постепенной адаптации данной культуры к условиям северного климата методом инбридинга и отбора в традиционных популяциях без существенного изменения их генетического полиморфизма.
Настоящий обзор сфокусирован на анализе и обобщении существующей на сегодняшний день информации о зависимости генетической регуляции цветения Z. mays от длины дня с целью возможного практического использования этих данных в характеристике видового разнообразия Z. mays и селекции новых линий и сортов зерновой кукурузы для возделывания в умеренных и высоких широтах. В обзоре рассмотрены особенности реакции растений Z. mays на фотопериод в сравнении с модельным видом Arabidopsis, включая гены, предположительно являвшиеся мишенями при одомашнивании и селекции Z. mays, в том числе при адаптации вида к новым эколого-географическим условиям выращивания.
ИНИЦИАЦИЯ ЦВЕТЕНИЯ Arabidopsis thaliana: СИГНАЛЬНЫЙ ПУТЬ ФОТОПЕРИОДА
Известная концепция инициации цветения предполагает существование гормоноподобных стимулятора и ингибитора (так называемых флоригенов) цветения, лучшее снабжение апекса побега ассимилятами при индуктивных условиях, а также комплексное влияние условий окружающей среды [9]. Молекулярно-биологический контекст перехода растения к репродуктивному развитию означает наличие сигнальных путей, опосредованных множеством генов, стимулирующих или угнетающих цветение [7].
Стимуляция и угнетение цветения Arabidopsis происходит в условиях, соответственно, LD (14–16 часов) и SD (8–10 часов) [3, 10]. Центральным регулятором фотопериодического пути считается локализованный в листьях транскрипционный фактор (ТФ) CONSTANS (CO) – ядерный белок с цинковым пальцем B-бокса (BBX) с С-концевым ДНК-связывающим доменом CCT (CO, CO-like и TIMING OF CAB EXPRESSION 1) (рис. 1) [11].
Рис. 1.
Схема контроля времени цветения Arabidopsis. Сигнальный путь фотопериода в условиях длинного дня (LD). Стрелки указывают на активацию транскрипции/цветения; стрелки с плоским концом указывают на репрессию транскрипции/цветения.
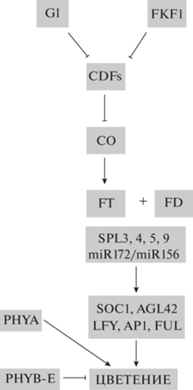
В конце световой фазы происходит инициация экспрессии генов GIGANTEA (GI) и FLAVIN BINDING KELCH REPEAT F-BOX PROTEIN 1 (FKF1) [12]. Белки FKF1 и GI образуют комплекс, активирующий опосредованную протеасомами деградацию группы белков CYCLING DOF FACTORs (CDFs), которые являются репрессорами транскрипции гена CO при SD [12, 13]. За счет этого происходит активация CO. Наивысший уровень экспрессии гена CO в световой фазе LD фотопериода и стабильность белка CO исключительно в световой период приводят к максимальному накоплению белка в вечернее время перед наступлением сумерек [14–16]. ТФ CO, совместно с другими партнерами, контролирует корректную транскрипцию флоригена FLOWERING LOCUS T (FT) [17–19]. Важное место среди партнеров занимают: ТФ NF-Y (состоит из трех субъединиц NF-YA, NF-YB и NF-YC; модуль CO–NF-Y активирует транскрипцию FT); белки B-Box19 (BBX19) и TOE1 (связываются с CO с образованием неактивного комплекса, что приводит к отсутствию индукции FT); несколько транскрипционных репрессоров семейства DELLA (связываясь с CO, блокируют его активность в отсутствие фитогормонов гиббереллинов; уровень экспрессии FT снижается); B-box микробелки (MicroProteins, MPs) miP1a и miP1b, которые опосредуют взаимодействие с белком-корепрессором TOPLESS (происходит не только инактивация CO, но и его превращение в репрессор FT) [19].
Накопление белка FT является сигналом к цветению Arabidopsis [17]. Считается, что ТФ FT перемещается из листьев через флоэму в апикальную меристему побега (АМП), где связывается с ТФ FLOWERING LOCUS D (FD) [17, 18, 20]. Исследования O. sativa показали, что для связывания с FD1 (гомолог FD) белок Hd3a (гомолог FT) формирует комплекс с белками 14-3-3 [18]. Тройной модуль FT-14-3-3-FD активирует транскрипцию OsMADS15, гомолога AP1 Arabidopsis, что приводит к цветению [18]. У Arabidopsis модуль FT-FD взаимодействует с белками SQUAMOSA PROMOTER-BINDING PROTEIN (SBP)-LIKE 3 (SPL3), SPL4, SPL5 и SPL9, индуцируя экспрессию генов идентичности цветковой меристемы LEAFY (LFY), APETALA1 (AP1) и FRUITFULL (FUL), а в случае SPL9 – также гены SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1 (SOC1) и AGAMOUS-LIKE 42 (AGL42) [21–27]. Белки SPL непосредственно повышают уровень экспрессии микроРНК miR172, которая участвует в подавлении транскрипции гена репрессора FT – APETALA2 (AP2), что приводит к цветению [21, 28–30]. В то же время другой тип микроРНК, miR156, подавляет транскрипцию генов SPL, кодирующие или 3'-нетранслируемые области которых содержат последовательности, комплементарные miR156 [31, 32]. Модуль miR172/miR156/SPL определяет зависящий от возраста регуляторный путь цветения и высоко консервативен у наземных растений [22, 30, 33–36].
Среди фоторецепторов Arabidopsis на инициацию цветения положительно влияют фитохром PHYA, фототропины PHOT1 и PHOT2, криптохромы CRY1 и CRY2, рецептор синего света FLAVIN-BINDING KELCH REPEAT F-BOX 1 (FKF1), а также рецептор ультрафиолета UVR8 [6, 12, 14, 37–39]. В подавлении зацветания участвуют фитохромы PHYB, PHYC, PHYD и PHYE, а также рецепторы синего света ZEITLUPE (ZTL) и LOV KELCH PROTEIN 2 (LKP2) [6, 14, 40–44]. FKF1, ZTL и LKP2 участвуют в регуляции стабильности белка-репрессора CO: сверхэкспрессия LKP2 или ZTL, так же как мутация fkf1, приводит к позднему цветению в условиях LD за счет подавления экспрессии CO и FT [45–47]. Интересно, что PHYB также может функционировать и как температурный рецептор [48], что подчеркивает наличие корреляции между воздействием фотопериода и температуры на инициацию цветения растения.
ИНИЦИАЦИЯ ЦВЕТЕНИЯ КУКУРУЗЫ Zea mays
Важнейшим фенологическим этапом развития зерновых является инициация и продолжительность цветения, также известные как дата и фаза колошения, которые напрямую влияют на урожайность зерна и должны укладываться в определенный сезон во избежание абиотических (холод, мороз, жара и засуха) и биотических (грибы, бактерии, вирусы, нематоды или насекомые) стрессов.
Получение и анализ последовательностей геномов зерновых – O. sativa [49], H. vulgare [50], T. aestivum [51] и Z. mays [52], позволили идентифицировать и охарактеризовать многие фенологические гены. Было показано, что сезонное цветение трав контролируется, за некоторым исключением, генами, ортологичными генам цветения Arabidopsis. При этом ключевые регуляторные гены фотопериодического контроля цветения консервативны у SD-растений O. sativa и LD-растений T. aestivum и H. vulgare, но регуляция этих генов часто противоположная [53]. Также выявлены многие функционально дивергентные (по отношению к Arabidopsis) гены, участвующие в цветении зерновых [54]. Кроме того, обнаружены значительные различия между злаками, выращиваемыми в умеренном и тропическом климате [54].
Кукуруза Z. mays является тропическим растением SD и, в отличие от T. aestivum и H. vulgare, не требует яровизации. Кукуруза (Z. mays ssp. mays) была одомашнена около 9 тыс. лет назад из дикой травы теосинте (Z. mays ssp. parviglumis), произрастающей в тропиках Центральной Америки (юго-западная часть Мексики) и являющейся растением SD [8]. В процессе одомашнивания теосинте была адаптирована к более прохладным умеренным регионам Северной Америки и Европы [55]. После доместикации селекция Z. mays была обусловлена, в частности, отбором по генам и локусам для адаптации времени цветения к новым условиям роста [56]. Сегодня, сорта кукурузы, чувствительные к фотопериоду, выращивают в тропических регионах, тогда как сорта умеренного климата нейтральны к продолжительности дня, и их цветение стимулируется почти исключительно за счет скоординированного действия генов автономных путей, регулируемых эндогенными сигналами [56]. В целом, современные сорта и линии Z. mays демонстрируют огромное естественное разнообразие по времени цветения, благодаря высокому уровню диверсификации фенологических генов [56, 57].
Текущие знания о генах цветения Z. mays основаны на результатах анализа локусов количественных признаков (QTL), с использованием различных популяций, мутагенеза и сравнительных исследований с Arabidopsis и O. sativa [55, 57, 58]. Основные фенологические гены перечислены в табл. 1.
Таблица 1.
Гены цветения кукурузы
| Ген кукурузы | Ортолог гена у Arabidopsis |
|---|---|
| ZmFKF1a (GRMZM2G107945) ZmFKF1b (GRMZM2G106363) |
FKF1 (AT1G68050) |
| ZmPRR37 (GRMZM2G033962, GRMZM2G005732) | PRR3 (AT5G60100) PRR7 (AT5G02810) |
| ZmGHD8 (GRMZM2G444073) | NF-YB2 (AT5G47640) NF-YB3 (AT4G14540) |
| ZmCCT (GRMZM5G868285) | Нет |
| CONZ1 (GRMZM2G405368) | CO (AT5G15840) |
| gigz1A/GI1 (GRMZM2G107101) gigz1B/GI2 (GRMZM5G844173) |
GI (AT1G22770) |
| ZmDLF1 (GRMZM2G067921) | FD (AT4G35900) |
| ZmMADS1 (GRMZM2G171365) ZmMADS69 (GRMZM2G171650) |
SOC1 (AT2G45660) |
| ZmPHYA1 (GRMZM2G157727) ZmPHYA2 (GRMZM2G181028) ZmPHYB1 (GRMZM2G124532) ZmPHYB2 (GRMZM2G092174) ZmPHYC1 (GRMZM2G057935) ZmPHYC2 (GRMZM2G129889; GRMZM2G129913) |
PHYA (AT1G09570) PHYB (AT2G18790) PHYC (AT5G35840) |
| ZCN8 (GRMZM2G179264) ZCN7 (GRMZM2G141756) ZCN14 (GRMZM2G373928) ZCN12 (GRMZM2G103666) |
FT (AT1G65480) |
| ZmZCN6 (GRMZM2G132880) | TFL1 (AT5G03840) |
| ZFL1 (GRMZM2G098813) ZFL2 (GRMZM2G180190) |
LFY (AT5G61850) |
| ID1 (GRMZM2G011357) | Нет |
| ZmSPL25 (GRMZM2G414805) | SPL3 (AT2G33810) SPL4 (AT1G53160) SPL5 (AT3G15270) SPL9 (AT2G42200) |
| ZMM4 (GRMZM2G032339) ZMM15 (GRMZM2G553379) ZMM24 (GRMZM2G087095) ZMM31 (GRMZM2G071620) ZAP1 (GRMZM2G148693) |
AP1 (AT1G69120) FUL (AT5G60910) |
Ключевым геном автономного пути цветения Z. mays (особенно важным в случае сортов умеренного климата) является INDETERMINATE 1 (ID1), специфичный для однодольных. Данный ген экспрессируется в листьях, кодирует ТФ с цинковым пальцем C2H2-типа и способен активировать переход растения к цветению независимо от фотопериода [59]. Уровни транскрипта и белка ID1 остаются стабильными в течение светового цикла, а мутации в ID1 приводят к увеличению продолжительности вегетативной фазы [60]. Показано, что ID1 контролирует модификации хроматина в локусах, кодирующих флоригены Z. mays, и может регулировать цветение посредством метилирования гистонов [56].
Комплекс FKF1/GI, который способствует активации транскрипции гена CO у Arabidopsis в зависимости от фотопериода [12, 13], по всей видимости, имеется и у кукурузы. В геноме Z. mays обнаружены гомологи генов Arabidopsis, как CO (CONZ1) и GI (GI1 и GI2), суточный профиль экспрессии которых зависит от фотопериода [61], так и FKF1 (ZmFKF1a и ZmFKF1b) [62]. В условиях LD ZmGI способствует подавлению цветения, так как мутация gi1 вызывает раннее цветение в условиях LD, и это противоположно функции GI у Arabidopsis [53, 63]. ZmFKF1a и ZmFKF1b выявлены у инбредных линий Z. mays, тогда как у образцов теосинте присутствует только ZmFKF1b [62]. При этом у инбредных линий обнаружены восемь сайтов положительной селекции на ZmFKF1b, соответствующих тропическим и умеренным группам кукурузы, что указывает на ключевую роль ZmFKF1b в адаптивной эволюции Z. mays [62].
Гомологи флоригена FT также присутствуют во множестве в геноме Z. mays. Анализ 15 FT-подобных генов ZEA CENTRORADIALIS (ZCN) определил флориген – ZCN8, играющий центральную роль в инициации цветения Z. mays [64]. Мутация zcn8 приводит к задержке цветения, а сверхэкспрессия ZCN8 восстанавливает мутантный фенотип ft Arabidopsis до дикого типа [65]. Регулирование гена ZCN8, а также второго предполагаемого флоригена ZCN7, находится под контролем белка ID1 [56].
Другой ТФ ZmNF-YA3 семейства Nuclear factor-Y (NF-Y), способствует раннему цветению, взаимодействуя с CO-подобным белком и FLOWERING PROMOTING FACTOR 1 (FPF1), а также с промотором FT-like 12 Z. mays [66].
В сложной генетической сети контроля инициации цветения Z. mays задействованы многочисленные QTL, в основном, с небольшим эффектом [57, 67]. Так, локус Vegetative to generative transition 1 (Vgt1) имеет в своей основе cis-регуляторный элемент Rap2.7, кодирующий АР2-доменный ТФ [68]. Ген ZCN8 составляет основу QTL qDTA8, определяющего время цветения. Данный локус постоянно обнаруживался во многих экспериментальных популяциях теосинте. Ген экспрессируется во флоэме зрелых листьев только у тропической кукурузы в условиях SD [64]. Еще один QTL qDTA3-2, включающий ген ZmMADS69, ассоциирован с различием во времени цветения между Z. mays и теосинте [69]. Через регуляторный модуль Rap2.7-ZCN8, ТФ ZmMADS69 действует как активатор цветения, подавляя экспрессию гена репрессора цветения Rap2.7 и тем самым ослабляя репрессию флоригена ZCN8 [69]. Сверхэкспрессия гена Zm‑MADS69 вызывает раннее цветение, в то время как его инактивация – обратный эффект. Показано, что по мере распространения Z. mays в широты умеренного климата селекцией была затронута 5'-регуляторная область ZmMADS69 [69]. И ZmMADS69, и Rap2.7 картируются в пределах хорошо охарактеризованных QTL времени цветения, предполагая, что аллельные варианты этих генов отвечают за часть существующего разнообразия признака времени цветения у Z. mays умеренного пояса [68, 70].
В контроле экспрессии флоригена ZCN8 участвуют также белки ID1 и DELAYED FLOWERING 1 (DLF1, bZIP-ТФ) [64, 71, 72]. ТФ ID1 активирует экспрессию гена DLF1 (гомолог FD Arabidopsis), и синтезируемый белок DLF1 взаимодействует в АМП с белком ZCN8, формируя модуль DLF1/ZCN8 (подобно FT/FD Arabidopsis, о котором писалось выше) [64, 71]. Эктопическая экспрессия DLF1 приводит к раннему цветению, тогда как выключение гена значительно его задерживает [71].
Постепенное распространение Z. mays в северном направлении сопровождалось появлением вариаций cis-регуляторных последовательностей промотора ZCN8, которые модифицировали профиль связывания различных ТФ [73, 74]. Анализ ассоциаций у широкой выборки инбредных линий кукурузы позволил идентифицировать в промоторе ZCN8 мононуклеотидный полиморфизм SNP-1245 (предположительно, происходящий от Zea mays ssp. mexicana), наиболее сильно коррелирующий с ранним цветением и связыванием с активатором цветения ТФ ZmMADS1 (гомолог SOC1 Arabidopsis) [74]. Предполагается, что SNP-1245 являлся мишенью отбора во время раннего одомашнивания Z. mays. Перед SNP-1245 находится независимый ассоциативный блок, не имеющий отношения к QTL qDTA8 [74]. Данный блок содержит Indel-2339 и считается сайтом внедрения аллеля раннего цветения SNP-1245 в раноцветущий гаплотип при адаптации к высоким широтам. На это указывает, в числе прочего, низкая частота (5%) аллеля Indel-2339Del в современных инбредных линиях тропической кукурузы и более высокая его частота (30%) у кукурузы умеренного пояса [74]. Таким образом, в процессе отбора в промоторной области ZCN8 произошли два тесно связанных события – появление SNP-1245 и Indel-2339 [74].
В условиях длинного дня репрессия ZCN8 опосредована CCT-доменным ТФ ZmCCT, который входит в один из основных QTL времени цветения (oft10) [64, 75]. ZmCCT связывается с промотором гена ZmEhd1, подавляя его транскрипцию, что снижает экспрессию ZCN8 [76]. Кроме того, ZmCCT подавляет транскрипцию флоригенов ZCN7, ZCN12 и ZCN14, генов BBX24 и ZmPRR37, и MADS-box гена ZAP1 (гомолог АР1), регулируя таким образом сложную сеть для инициации и поддержания перехода растения от вегетативной к репродуктивной фазе [76].
Более 85% генома Z. mays составляют различные транспозоны [52], которые играли и играют важную роль в доместикации и селекции данной культуры. К примеру, охарактеризованы аллели ZmCCT9 [77] и ZmCCT10 [70, 78, 79], независимые вставки транспозонов в регуляторные области которых снижают экспрессию ZmCCT9 и ZmCCT10 и, как следствие, чувствительность Z. mays к LD-фотопериоду [78, 79].
У различных аллелей ZmCCT10 были выявлены две транспозонные вставки (5.1 т.п.н. – в промоторной области, 4.2 т.п.н. – в интроне). Данные вставки приводят к противоположным эффектам. Аллель со вставкой 5.1 т.п.н. CACTA-подобного транспозона в промоторе ускоряет цветение в условиях умеренного климата (LD), тогда как в тропических регионах (SD) не оказывает никакого эффекта по сравнению с нулевым аллелем (без вставок) [76]. Напротив, аллель со вставкой 4.2 т.п.н. в интроне ускоряет время цветения в тропическом регионе, но не в регионе с умеренным климатом [76]. Предполагается, что существует неизвестный белок, который может индуцироваться изменением длины дня и связываться с промотором ZmCCT10, усиливая его экспрессию в умеренном климате. Транспозонная вставка 5.1 т.п.н. может препятствовать связыванию неизвестного белка в высокоширотных регионах, тогда как в низкоширотной области данный белок может либо не экспрессироваться, либо экспрессироваться на низком уровне [76]. В свою очередь, вставка 4.2 т.п.н. может индуцировать транскрипцию антисмысловых РНК, что будет вызывать репрессирование хроматина, включая метилирование ДНК, H3K9me2 и деацетилирование гистонов, а затем подавлять транскрипцию гена ZmCCT10 как в низко-, так и в высокоширотных регионах. В сравнении с аллелем со вставкой 5.1 т.п.н., ZmCCT10 со вставкой 4.2 т.п.н. показывает равную и более низкую экспрессию в высоко- и низкоширотных регионах, соответственно [76].
Еще один мобильный элемент, Harbinger-like, расположенный за 57 т.п.н. до гена ZmCCT9, связан с еще одним QTL времени цветения, qDTA9 [67, 77]. Так как ZmCCT9 подавляет экспрессию флоригена ZCN8, данный элемент выступает как репрессор ZmCCT9, способствуя цветению в условиях LD. Предполагается, что Harbinger-like элемент возник после появления вставки 5.1 т.п.н. в промоторе ZmCCT10 [70, 76].
В инициации цветения Z. mays важная роль принадлежит также фитохром-опосредованному сигнальному пути в совокупности с регуляторным модулем miR172/miR156/SPL, как это показано на примере Arabidopsis [21]. Геном Z. mays содержит по паре паралогичных генов, кодирующих три типа фитохромов, PHYA1, PHYA2, PHYB1, PHYB2, PHYC1 и PHYC2 [80], а также 30 генов ZmSPL [81]. Анализ сверхэкспрессии ZmPHYC1 и ZmPHYC2 и двойного нокаута zmphyC1 zmphyC2 выявил консервативную функцию генов ZmPHYC в негативной регуляции времени цветения в условиях LD [82]. Данные анализа генов ZmSPL подтверждают, что 18 из них могут являться прямыми мишенями для zma-miR156s [81, 83–85], а ZmSPL25 участвует в контроле времени цветения и региональной адаптации у сортов кукурузы умеренного климата [86]. У Z. mays найдено 12 членов семейства MIR156 и показано, что сверхэкспрессия двух тандемных генов MIR156 задерживает цветение [83].
После активации флоригенов и других факторов инициации цветения происходит активация генов идентичности меристем соцветия и цветка Z. mays, таких как ZEA FLORICAULA/LEAFY (ZFL1 и ZFL2) (гомологи LFY) и MADS-box гены ZMM4, ZMM15, ZMM24 и ZMM31 (AP1/FUL подсемейство) [87, 88].
На рис. 2 приведена возможная схема инициации цветения Z. mays под влиянием фотопериода короткого дня.
Рис. 2.
Схема контроля времени цветения Z. mays. Сигнальный путь фотопериода короткого дня (SD). Стрелки указывают на активацию транскрипции/цветения; стрелки с плоским концом указывают на репрессию транскрипции/цветения. Серые блоки соответствуют генам, ортологи которых имеются в геноме Arabidopsis. В рамке – дивергентные гены цветения, идентифицированные в геноме Z. mays и отсутствующие у Arabidopsis.

Продвижение культуры в высокие широты сопряжено с понижением диапазона температур и, следовательно, с чувствительностью кукурузы к холоду, в частности, способностью цвести при пониженных температурах. Хотя Z. mays, в отличие от T. aestivum, не требует яровизации [8], она исходно является тропическим растением и должна быть чувствительна к холоду. Поскольку основные площади возделывания кукурузы находятся в регионах с теплым климатом, в основном изучаются механизмы устойчивости данной культуры к повышенным (выше 35°С) температурам [6, 66, 89, 90]. О связи цветения Z. mays с пониженными температурами известно только, что флоригены ZCN12 и ZCN8 играют совместную центральную роль в адаптации кукурузы при продвижении с юга на север в связи со сменой не только продолжительности дня, но и температурного градиента [91].
Исследования видов, адаптированных к северным, околополярным зонам роста, показали, что устойчивость к холоду и заморозкам может быть ассоциирована с накоплением сахаров [92]. Связь с углеводным обменом наблюдается и в случае ответа растений на изменение длины дня. Так, при культивировании в условиях непрерывного освещения (имитация условий околополярных широт) агрокультуры демонстрируют снижение активности фотосинтеза из-за накопления высоких концентраций крахмала в листьях [92]. Все это может свидетельствовать о связи эффектов изменения фотопериода и понижения температуры при адаптационном продвижении Z. mays с юга на север, а также об участии в данном процессе эволюционных изменений генов углеводного метаболизма.
ЗАКЛЮЧЕНИЕ
В настоящем обзоре изложены сведения о регуляции инициации цветения у растений кукурузы Z. mays, включая воздействие фотопериода, и ее возможной связи с адаптацией данной культуры к условиям увеличенной продолжительности дня в процессе доместикации и селекции при продвижении из тропиков в высокие широты. Общим для различных растений в основной схеме молекулярно-генетического контроля времени перехода к репродуктивной фазе является требование к вышестоящим регуляторам определять экспрессию FT-подобных генов, но количество паралогов и относительный вклад таких регуляторов в значительной степени отличается у Z. mays в сравнении с модельным растением A. thaliana. Имеются свидетельства связи между изменением фотопериода и понижением температуры при адаптационном продвижении Z. mays с юга на север. Обобщенные в обзоре данные могут быть использованы для оценки современного разнообразия вида в рамках существующих коллекций образцов, инбредных линий и сортов Z. mays, а также в селекции раноцветущих линий и сортов зерновой кукурузы для возделывания в умеренных и высоких широтах.
Работа выполнена при поддержке Российского научного фонда (грант № 21-16-00008) и Министерства науки и образования Российской Федерации.
Авторы заявляют об отсутствии конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследования.
Список литературы
Hill C.B., Li C. Genetic Architecture of Flowering Phenology in Cereals and Opportunities for Crop Improvement // Front. Plant Sci. 2016. V. 7: 1906. https://doi.org/10.3389/fpls.2016.01906
Craufurd P.Q., Wheeler T.R. Climate change and the flowering time of annual crops // J. Exp. Bot. 2009. V. 60. P. 2529.
Greenup A., Peacock W.J., Dennis E.S., Trevaskis B. The molecular biology of seasonal flowering-responses in Arabidopsis and the cereals // Ann. Bot. 2009. V. 103. P. 1165.
von Zitzewitz J., Szucs P., Dubcovsky J., Yan L., Francia E., Pecchioni N., Casas A., Chen T.H., Hayes P.M., Skinner J.S. Molecular and structural characterization of barley vernalization genes // Plant Mol. Biol. 2005. V. 59. P. 449.
Gawroński P., Ariyadasa R., Himmelbach A., Poursarebani N., Kilian B., Stein N., Steuernagel B., Hensel G., Kumlehn J., Sehgal S.K., Gill B.S., Gould P., Hall A., Schnurbusch T. A distorted circadian clock causes early flowering and temperature-dependent variation in spike development in the Eps-3Am mutant of einkorn wheat // Genetics. 2014. V. 196. P. 1253.
Cao S., Luo X., Xu D., Tian X., Song J., Xia X., Chu C., He Z. Genetic architecture underlying light and temperature mediated flowering in Arabidopsis, rice, and temperate cereals // New Phytol. 2021. V. 230. P. 1731.
Song Y.H., Kubota A., Kwon M.S., Covington M.F., Lee N., Taagen E.R., Laboy Cintrón D., Hwang D.Y., Akiyama R., Hodge S.K., Huang H., Nguyen N.H., Nusinow D.A., Millar AJ, Shimizu K.K. et al. Molecular basis of flowering under natural long-day conditions in Arabidopsis // Nat. Plants. 2018. V. 4. P. 824.
Matsuoka Y., Vigouroux Y., Goodman M.M., Sanchez G.J., Buckler E., Doebley J. A single domestication for maize shown by multilocus microsatellite genotyping // Proc. Natl. Acad. Sci. USA. 2002. V. 99. P. 6080.
Bernier G., Havelange A., Houssa C., Petitjean A., Lejeune P. Physiological signals that induce flowering // Plant Cell. 1993. V. 5. P. 1147.
Pidkowich M.S., Klenz J.E., Haughn G.W. The making of a flower: control of floral meristem identity in Arabidopsis // Trends Plant Sci. 1999. V. 4. P. 64.
Putterill J., Robson F., Lee K., Simon R., Coupland G. The CONSTANS gene of Arabidopsis promotes flowering and encodes a protein showing similarities to zinc finger transcription factors // Cell. 1995. V. 80. P. 847.
Sawa M., Nusinow D.A., Kay S.A., Imaizumi T. FKF1 and GIGANTEA complex formation is required for day-length measurement in Arabidopsis // Science. 2007. V. 318. P. 261.
Fornara F., Panigrahi K.C., Gissot L., Sauerbrunn N., Rühl M., Jarillo J.A., Coupland G. Arabidopsis DOF transcription factors act redundantly to reduce CONSTANS expression and are essential for a photoperiodic flowering response // Dev. Cell. 2009. V. 17. P. 75.
Valverde F., Mouradov A., Soppe W., Ravenscroft D., Samach A., Coupland G. Photoreceptor regulation of CONSTANS protein in photoperiodic flowering // Science. 2004. V. 303. P. 1003.
Jang S., Marchal V., Panigrahi K.C., Wenkel S., Soppe W., Deng X.W., Valverde F., Coupland G. Arabidopsis COP1 shapes the temporal pattern of CO accumulation conferring a photoperiodic flowering response // EMBO J. 2008. V. 27. P. 1277.
Lazaro A., Mouriz A., Piñeiro M., Jarillo J.A. Red Light-Mediated Degradation of CONSTANS by the E3 Ubiquitin Ligase HOS1 Regulates Photoperiodic Flowering in Arabidopsis // Plant Cell. 2015. V. 27. P. 2437.
Corbesier L., Vincent C., Jang S., Fornara F., Fan Q., Searle I., Giakountis A., Farrona S., Gissot L., Turnbull C., Coupland G. FT protein movement contributes to long-distance signaling in floral induction of Arabidopsis // Science. 2007. V. 316. P. 1030.
Taoka K., Ohki I., Tsuji H., Furuita K., Hayashi K., Yanase T., Yamaguchi M., Nakashima C., Purwestri Y.A., Tamaki S., Ogaki Y., Shimada C., Nakagawa A., Kojima C., Shimamoto K. 14-3-3 proteins act as intracellular receptors for rice Hd3a florigen // Nature. 2011. V. 476. P. 332.
Brambilla V., Fornara F. Y flowering? Regulation and activity of CONSTANS and CCT-domain proteins in Arabidopsis and crop species // Biochim. Biophys. Acta Gene Regul. Mech. 2017. V. 1860. P. 655.
Jaeger K.E., Wigge P.A. FT protein acts as a long-range signal in Arabidopsis // Curr. Biol. 2007. V. 17. P. 1050.
Wang J.W., Czech B., Weigel D. miR156-regulated SPL transcription factors define an endogenous flowering pathway in Arabidopsis thaliana // Cell. 2009. V. 138. P. 738.
Wu G., Park M.Y., Conway S.R., Wang J.W., Weigel D., Poethig R.S. The sequential action of miR156 and miR172 regulates developmental timing in Arabidopsis // Cell. 2009. V. 138. P. 750.
Mathieu J., Yant L.J., Mürdter F., Küttner F., Schmid M. Repression of flowering by the miR172 target SMZ // PLoS Biol. 2009. V. 7: e1000148. https://doi.org/10.1371/journal.pbio.1000148
Yamaguchi A., Wu M.F., Yang L., Wu G., Poethig R.S., Wagner D. The microRNA-regulated SBP-Box transcription factor SPL3 is a direct upstream activator of LEAFY, FRUITFULL, and APETALA1 // Dev. Cell. 2009. V. 17. P. 268.
Yant L., Mathieu J., Dinh T.T., Ott F., Lanz C., Wollmann H., Chen X., Schmid M. Orchestration of the floral transition and floral development in Arabidopsis by the bifunctional transcription factor APETALA2 // Plant Cell. 2010. V. 22. P. 2156.
Jung J.H., Seo P.J., Kang S.K., Park C.M. miR172 signals are incorporated into the miR156 signaling pathway at the SPL3/4/5 genes in Arabidopsis developmental transitions // Plant Mol. Biol. 2011. V. 76. P. 35.
Jung J.H., Lee H.J., Ryu J.Y., Park C.M. SPL3/4/5 Integrate Developmental Aging and Photoperiodic Signals into the FT-FD Module in Arabidopsis Flowering // Mol. Plant. 2016. V. 9. P. 1647.
Jung J.H., Seo Y.H., Seo P.J., Reyes J.L., Yun J., Chua N.H., Park C.M. The GIGANTEA-regulated microRNA172 mediates photoperiodic flowering independent of CONSTANS in Arabidopsis // Plant Cell. 2007. V. 19. P. 2736.
Schwarz S., Grande A.V., Bujdoso N., Saedler H., Huijser P. The microRNA regulated SBP-box genes SPL9 and SPL15 control shoot maturation in Arabidopsis // Plant Mol. Biol. 2008. V. 67. P. 183.
Wang J.W. Regulation of flowering time by the miR156-mediated age pathway // J. Exp. Bot. 2014. V. 65. P. 4723.
Gandikota M., Birkenbihl R.P., Höhmann S., Cardon G.H., Saedler H., Huijser P. The miRNA156/157 recognition element in the 3' UTR of the Arabidopsis SBP box gene SPL3 prevents early flowering by translational inhibition in seedlings // Plant J. 2007. V. 49. P. 683.
Guo A.Y., Zhu Q.H., Gu X., Ge S., Yang J., Luo J. Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family // Gene. 2008. V. 418. P. 1.
Xie K., Wu C., Xiong L. Genomic organization, differential expression, and interaction of SQUAMOSA promoter-binding-like transcription factors and microRNA156 in rice // Plant Physiol. 2006. V. 142. P. 280.
Yang Z., Wang X., Gu S., Hu Z., Xu H., Xu C. Comparative study of SBP-box gene family in Arabidopsis and rice // Gene. 2008. V. 407. P. 1.
Yang L., Conway S.R., Poethig R.S. Vegetative phase change is mediated by a leaf-derived signal that represses the transcription of miR156 // Development. 2011. V. 138. P. 245.
Poethig R.S. Vegetative phase change and shoot maturation in plants // Curr. Top. Dev. Biol. 2013. V. 105. P. 125.
Kong Y., Zheng Y. Phototropin is partly involved in blue-light-mediated stem elongation, flower initiation, and leaf expansion: a comparison of phenotypic responses between wild Arabidopsis and its phototropin mutants // Environ. Exp. Bot. 2020. V. 171: e103967. https://doi.org/10.1016/j.envexpbot.2019.103967
Liu H., Liu B., Zhao C., Pepper M., Lin C. The action mechanisms of plant cryptochromes // Trends Plant Sci. 2011. V. 16. P. 684.
Arongaus A.B., Chen S., Pireyre M., Glockner N., Galvao V.C., Albert A., Winkler J.B., Fankhauser C., Harter K., Ulm R. Arabidopsis RUP2 represses UVR8-mediated flowering in noninductive photoperiods // Genes Dev. 2018. V. 32. P. 1332.
Monte E., Alonso J.M., Ecker J.R., Zhang Y., Li X., Young J., Austin-Phillips S., Quail P.H. Isolation and characterization of phyC mutants in Arabidopsis reveals complex crosstalk between phytochrome signaling pathways // Plant Cell. 2003. V. 15. P. 1962.
Devlin P.F., Robson P.R., Patel S.R., Goosey L., Sharrock R.A., Whitelam G.C. Phytochrome D acts in the shade-avoidance syndrome in Arabidopsis by controlling elongation growth and flowering time // Plant Physiol. 1999. V. 119. P. 909.
Devlin P.F., Patel S.R., Whitelam G.C. Phytochrome E influences internode elongation and flowering time in Arabidopsis // Plant Cell. 1998. V. 10. P. 1479.
Kim W.Y., Fujiwara S., Suh S.S., Kim J., Kim Y., Han L., David K., Putterill J., Nam H.G., Somers D.E. ZEITLUPE is a circadian photoreceptor stabilized by GIGANTEA in blue light // Nature. 2007. V. 449. P. 356.
Schultz T.F., Kiyosue T., Yanovsky M., Wada M., Kay S.A. A role for LKP2 in the circadian clock of Arabidopsis // Plant Cell. 2001. V. 13. P. 2659.
Nelson D.C., Lasswell J., Rogg L.E., Cohen M.A., Bartel B. FKF1, a clock-controlled gene that regulates the transition to flowering in Arabidopsis // Cell. 2000. V. 101. P. 331.
Somers D.E., Kim W.Y., Geng R. The F-box protein ZEITLUPE confers dosage-dependent control on the circadian clock, photomorphogenesis, and flowering time // Plant Cell. 2004. V. 16. P. 769.
Takase T., Nishiyama Y., Tanihigashi H., Ogura Y., Miyazaki Y., Yamada Y., Kiyosue T. LOV KELCH PROTEIN2 and ZEITLUPE repress Arabidopsis photoperiodic flowering under non-inductive conditions, dependent on FLAVIN-BINDING KELCH REPEAT F-BOX1 // Plant J. 2011. V. 67. P. 608.
Gil K.E., Park C.M. Thermal adaptation and plasticity of the plant circadian clock // New Phytol. 2019. V. 221. P. 1215.
Goff S.A. Collaboration on the rice genome // Science. 2002. V. 296: 45. https://doi.org/10.1126/science.296.5565.45c
International Barley Genome Sequencing Consortium, Mayer K.F., Waugh R., Brown J.W., Schulman A., Langridge P., Platzer M., Fincher G.B., Muehlbauer G.J., Sato K., Close T.J., Wise R.P., Stein N. A physical, genetic and functional sequence assembly of the barley genome // Nature. 2012. V. 91. P. 711.
International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome // Science. 2014. V. 345: 1251788. https://doi.org/10.1126/science.1251788
Schnable P.S., Ware D., Fulton R.S., Stein J.C., Wei F.S., Pasternak S., Liang C.Z., Zhang J.W., Fulton L., Graves T.A., Minx P., Reily A.D., Courtney L., Kruchowski S.S., Tomlinson C. et al. The B73 maize genome: complexity, diversity, and dynamics // Science. 2009. V. 326. P. 1112.
Hayama R., Yokoi S., Tamaki S., Yano M., Shimamoto K. Adaptation of photoperiodic control pathways produces short-day flowering in rice // Nature. 2003. V. 422. P. 719.
Calixto C.P., Waugh R., Brown J.W. Evolutionary relationships among barley and Arabidopsis core circadian clock and clock-associated genes // J. Mol. Evol. 2015. V. 80. P. 108.
Chardon F., Virlon B., Moreau L., Falque M., Joets J., Decousset L., Murigneux A., Charcosset A. Genetic architecture of flowering time in maize as inferred from quantitative trait loci meta-analysis and synteny conservation with the rice genome // Genetics. 2004. V. 168. P. 2169.
Mascheretti I., Turner K., Brivio R.S., Hand A., Colasanti J., Rossi V. Florigen-Encoding Genes of Day-Neutral and Photoperiod-Sensitive Maize Are Regulated by Different Chromatin Modifications at the Floral Transition // Plant Physiol. 2015. V. 168. P. 1351.
Buckler E.S., Holland J.B., Bradbury P.J., Acharya C.B., Brown P.J., Browne C., Ersoz E., Flint-Garcia S., Garcia A., Glaubitz J.C., Goodman M.M., Harjes C., Guill K., Kroon D.E., Larsson S. et al. The genetic architecture of maize flowering time // Science. 2009. V. 325. P. 714.
Steinhoff J., Liu W., Reif J.C., Della Porta G., Ranc N., Würschum T. Detection of QTL for flowering time in multiple families of elite maize // Theor. Appl. Genet. 2012. V. 125. P. 1539.
Colasanti J., Tremblay R., Wong A.Y., Coneva V., Kozaki A., Mable B.K. The maize INDETERMINATE1 flowering time regulator defines a highly conserved zinc finger protein family in higher plants // BMC Genomics. 2006. V. 7: 158. https://doi.org/10.1186/1471-2164-7-158
Wong A.Y., Colasanti J. Maize floral regulator protein INDETERMINATE1 is localized to developing leaves and is not altered by light or the sink/source transition // J. Exp. Bot. 2007. V. 58. P. 403.
Miller T.A., Muslin E.H., Dorweiler J.E. A maize CONSTANS-like gene, conz1, exhibits distinct diurnal expression patterns in varied photoperiods // Planta. 2008. V. 227. P. 1377.
Liu L., Wu Y., Liao Z., Xiong J., Wu F., Xu J., Lan H., Tang Q., Zhou S., Liu Y., Lu Y. Evolutionary conservation and functional divergence of the LFK gene family play important roles in the photoperiodic flowering pathway of land plants // Heredity (Edinb). 2018. V. 120. P. 310.
Bendix C., Mendoza J.M., Stanley D.N., Meeley R., Harmon F.G. The circadian clock-associated gene gigantea1 affects maize developmental transitions // Plant Cell Environ. 2013. V. 36. P. 1379.
Meng X., Muszynski M.G., Danilevskaya O.N. The FT-like ZCN8 Gene Functions as a Floral Activator and Is Involved in Photoperiod Sensitivity in Maize // Plant Cell. 2011. V. 23. P. 942.
Lazakis C.M., Coneva V., Colasanti J. ZCN8 encodes a potential orthologue of Arabidopsis FT florigen that integrates both endogenous and photoperiod flowering signals in maize // J. Exp. Bot. 2011. V. 62. P. 4833.
Su H., Cao Y., Ku L., Yao W., Cao Y., Ren Z., Dou D., Wang H., Ren Z., Liu H., Tian L., Zheng Y., Chen C., Chen Y. Dual functions of ZmNF-YA3 in photoperiod-dependent flowering and abiotic stress responses in maize // J. Exp. Bot. 2018. V. 69. P. 5177.
Li Y.X., Li C., Bradbury P.J., Liu X., Lu F., Romay C.M., Glaubitz J.C., Wu X., Peng B., Shi Y., Song Y., Zhang D., Buckler E.S., Zhang Z., Li Y., Wang T. Identification of genetic variants associated with maize flowering time using an extremely large multi-genetic background population // Plant J. 2016. V. 86. P. 391.
Salvi S., Corneti S., Bellotti M., Carraro N., Sanguineti M.C., Castelletti S., Tuberosa R. Genetic dissection of maize phenology using an intraspecific introgression library // BMC Plant Biol. 2011. V. 11: 4. https://doi.org/10.1186/1471-2229-11-4
Liang Y., Liu Q., Wang X., Huang C., Xu G., Hey S., Lin H.Y., Li C., Xu D., Wu L., Wang C., Wu W., Xia J., Han X., Lu S. et al. ZmMADS69 functions as a flowering activator through the ZmRap2.7-ZCN8 regulatory module and contributes to maize flowering time adaptation // New Phytol. 2019. V. 221. P. 2335.
Ducrocq S., Giauffret C., Madur D., Combes V., Dumas F., Jouanne S., Coubriche D., Jamin P., Moreau L., Charcosset A. Fine mapping and haplotype structure analysis of a major flowering time quantitative trait locus on maize chromosome 10 // Genetics. 2009. V. 183. P. 1555.
Muszynski M.G., Dam T., Li B., Shirbroun D.M., Hou Z., Bruggemann E., Archibald R., Ananiev E.V., Danilevskaya O.N. delayed flowering1 Encodes a basic leucine zipper protein that mediates floral inductive signals at the shoot apex in maize // Plant Physiol. 2006. V. 142. P. 1523.
Minow M.A.A., Ávila L.M., Turner K., Ponzoni E., Mascheretti I., Dussault F.M., Lukens L., Rossi V., Colasanti J. Distinct gene networks modulate floral induction of autonomous maize and photoperiod-dependent teosinte // J. Exp. Bot. 2018. V. 69. P. 2937.
Alter P., Bircheneder S., Zhou L.Z., Schlüter U., Gahrtz M., Sonnewald U., Dresselhaus T. Flowering Time-Regulated Genes in Maize Include the Transcription Factor ZmMADS1 // Plant Physiol. 2016. V. 172. P. 389.
Guo L., Wang X., Zhao M., Huang C., Li C., Li D., Yang C.J., York A.M., Xue W., Xu G., Liang Y., Chen Q., Doebley J.F., Tian F. Stepwise cis-Regulatory Changes in ZCN8 Contribute to Maize Flowering-Time Adaptation // Curr. Biol. 2018. V. 28. P. 3005.
Stephenson E., Estrada S., Meng X., Ourada J., Muszynski M.G., Habben J.E., Danilevskaya O.N. Over-expression of the photoperiod response regulator ZmCCT10 modifies plant architecture, flowering time and inflorescence morphology in maize // PLoS One. 2019. V. 14: e0203728. https://doi.org/10.1371/journal.pone.0203728
Zhong S., Liu H., Li Y., Lin Z. Opposite response of maize ZmCCT to photoperiod due to transposon jumping // Theor. Appl. Genet. 2021. https://doi.org/10.1007/s00122-021-03862-7
Huang C., Sun H., Xu D., Chen Q., Liang Y., Wang X., Xu G., Tian J., Wang C., Li D., Wu L., Yang X., Jin W., Doebley J.F., Tian F. ZmCCT9 enhances maize adaptation to higher latitudes // Proc. Natl. Acad. Sci. USA. 2018. V. 115. P. E334.
Hung H.Y., Shannon L.M., Tian F., Bradbury P.J., Chen C., Flint-Garcia S.A., McMullen M.D., Ware D., Buckler E.S., Doebley J.F., Holland J.B. ZmCCT and the genetic basis of day-length adaptation underlying the postdomestication spread of maize // Proc. Natl. Acad. Sci. USA. 2012. V. 109. P. E1913.
Yang Q., Li Z., Li W., Ku L., Wang C., Ye J., Li K., Yang N., Li Y., Zhong T., Li J., Chen Y., Yan J., Yang X., Xu M. CACTA-like transposable element in ZmCCT attenuated photoperiod sensitivity and accelerated the postdomestication spread of maize. // Proc. Natl. Acad. Sci. USA. 2013. V. 110. P. 16969.
Sheehan M.J., Farmer P.R., Brutnell T.P. Structure and expression of maize phytochrome family homeologs // Genetics. 2004. V. 167. P. 1395.
Wei H., Zhao Y., Xie Y., Wang H. Exploiting SPL genes to improve maize plant architecture tailored for high-density planting // J. Exp. Bot. 2018. V. 69. P. 4675.
Li Q., Wu G., Zhao Y., Wang B., Zhao B., Kong D., Wei H., Chen C., Wang H. CRISPR/Cas9-mediated knockout and overexpression studies reveal a role of maize phytochrome C in regulating flowering time and plant height // Plant Biotechnol. J. 2020. V. 18. P. 2520.
Chuck G., Cigan A.M., Saeteurn K., Hake S. The heterochronic maize mutant Corngrass1 results from overexpression of a tandem microRNA // Nat. Genet. 2007. V. 39. P. 544.
Chuck G., Whipple C., Jackson D., Hake S. The maize SBP-box transcription factor encoded by tasselsheath4 regulates bract development and the establishment of meristem boundaries // Development. 2010. V. 137. P. 1243.
Chuck G.S., Brown P.J., Meeley R., Hake S. Maize SBP-box transcription factors unbranched2 and unbranched3 affect yield traits by regulating the rate of lateral primordia initiation // Proc. Natl. Acad. Sci. USA. 2014. V. 111. P. 18775.
Unterseer S., Pophaly S.D., Peis R., Westermeier P., Mayer M., Seidel M.A., Haberer G., Mayer K.F., Ordas B., Pausch H., Tellier A., Bauer E., Schön C.C. A comprehensive study of the genomic differentiation between temperate Dent and Flint maize // Genome Biol. 2016. V. 17: 137. https://doi.org/10.1186/s13059-016-1009-x
Bomblies K., Doebley J.F. Pleiotropic effects of the duplicate maize FLORICAULA/LEAFY genes zfl1 and zfl2 on traits under selection during maize domestication // Genetics. 2006. V. 172 P. 519.
Danilevskaya O.N., Meng X., Selinger D.A., Deschamps S., Hermon P., Vansant G., Gupta R., Ananiev E.V., Muszynski M.G. Involvement of the MADS-box gene ZMM4 in floral induction and inflorescence development in maize // Plant Physiol. 2008. V. 147. P. 2054.
Zeng W., Shi J., Qiu C., Wang Y., Rehman S., Yu S., Huang S., He C., Wang W., Chen H., Chen C., Wang C., Tao Z., Li P. Identification of a genomic region controlling thermotolerance at flowering in maize using a combination of whole genomic re-sequencing and bulked segregant analysis // Theor. Appl. Genet. 2020. V. 133. P. 2797.
Galic V., Franic M., Jambrovic A., Ledencan T., Brkic A., Zdunic Z., Simic D. Genetic Correlations Between Photosynthetic and Yield Performance in Maize Are Different Under Two Heat Scenarios During Flowering // Front. Plant Sci. 2019. V. 10: 566. https://doi.org/10.3389/fpls.2019.00566
Castelletti S., Coupel-Ledru A., Granato I., Palaffre C., Cabrera-Bosquet L., Tonelli C., Nicolas S.D., Tardieu F., Welcker C., Conti L. Maize adaptation across temperate climates was obtained via expression of two florigen genes // PLoS Genet. 2020. V. 16: 1008882. https://doi.org/10.1371/journal.pgen.1008882
Lütz C. Cell physiology of plants growing in cold environments // Protoplasma. 2010. V. 244. P. 53.
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


