Физиология растений, 2023, T. 70, № 6, стр. 601-611
Влияние мозаичного нокаутирования гена фитоиндесатуразы NtPDS на биосинтез каротиноидов у Nicotiana tabacum L.
А. В. Нежданова a, *, М. А. Слугина a, А. В. Кулакова a, А. М. Камионская a, Е. З. Кочиева a, А. В. Щенникова a
a Институт биоинженерии Федерального исследовательского центра
Фундаментальные основы биотехнологии Российской академии наук
Москва, Россия
* E-mail: anna-negdanova@mail.ru
Поступила в редакцию 17.04.2023
После доработки 27.04.2023
Принята к публикации 28.04.2023
- EDN: CXCKHG
- DOI: 10.31857/S001533032360033X
Аннотация
Каротиноиды крайне важны как незаменимые участники фотосинтеза и фотозащиты, а также роста, развития и стрессовой реакции растений. Фермент фитоиндесатураза (PDS; EC 1.3.5.5) катализирует первую фазу десатурации 15-цис-фитоина (предшественника всех каротиноидов). В работе впервые исследовано влияние нокаута PDS в геноме Nicotiana tabacum L. с использованием системы CRISPR-Cas9 на активность нижестоящих генов пути биосинтеза каротиноидов. Получены девять трансгенных линий табака с мозаичным редактированием гена NtPDS – три варианта инделей (350^351→ins^g#, 350^351→ins^t# и t351→del(1nt)#) в области экзона II, приводящих к синтезу укороченной, нефункциональной версии белка. Линии характеризовались пятнистой, зелено-белой пигментацией листьев, изменениями времени инициации цветения и морфологических характеристик, а также снижением содержания каротиноидов и хлорофиллов в ткани листа. Продемонстрировано увеличение уровня транскриптов гена фитоинсинтазы NtPSY2 у позднецветущих линий в сравнении с контролем. Редактированная линия L29 с самым поздним цветением характеризовалась существенным подъемом уровня транскриптов нижестоящих структурных генов каротиногенеза. Снижение содержания каротиноидов в листьях мозаично редактированных растений сопровождалось падением уровня экспрессии MADS-box гена NtSEP1, продукт которого предположительно участвует в регуляции транскрипции генов биосинтеза каротиноидов. Полученные данные могут быть использованы для дальнейших исследований регуляции биосинтеза каротиноидов и апокаротиноидов у Пасленовых культур.
ВВЕДЕНИЕ
Каротиноиды (пигменты различных оттенков желтого, оранжевого и красного) образуются de novo у фото- и хемосинтезирующих организмов: у некоторых видов бактерий, архей и грибов, а также у водорослей и растений; остальные организмы (включая животных и человека) каротиноиды не синтезируют, но потребляют с пищей и метаболизируют [1]. Для фотосинтезирующих организмов данные пигменты крайне важны как незаменимые участники фотосинтеза и фотозащиты. Каротиноиды собирают энергию света и осуществляют синглет-синглетную передачу энергии электронного возбуждения хлорофиллам. Одновременно они поглощают избыточную энергию хлорофиллов, защищая организм от неблагоприятного воздействия света высокой интенсивности. Нефотохимическое тушение происходит при ферментативном преобразовании производных α- и β-каротинов, ксантофиллов (виолаксантин, антераксантин и эпоксид лютеина), в деэпоксидные ксантофиллы (зеаксантин и лютеин). Кроме того, образующиеся в процессе фотосинтеза активные формы кислорода нейтрализуются каротиноидами как антиоксидантами за счет физических и/или химических реакций [1, 2].
У растений блокировка синтеза каротиноидов приводит к нарушениям в работе фотосинтетического аппарата и защите от фотоингибирующего и оксидативного стрессов [3]. Более того, это отменяет образование α- и β-каротинов – основных источников фитогормонов стриголактонов и абсцизовой кислоты (АБК), необходимых для регуляции различных аспектов роста, развития и стрессовой реакции организма [4, 5], равно как и витамина А в рационе человека и животных [6].
С учетом вышесказанного, нарушение синтеза каротиноидов может иметь критические последствия вплоть до гибели растения. К примеру, на этом основано действие хлорсодержащих отбеливающих гербицидов (таких как норфлуразон), которые приводят растение к гибели, ингибируя биосинтез каротиноидов [2, 7]. Гербицид действует на стадии первой десатурации, когда начинается преобразование предшественника каротиноидов (15-цис-фитоин) в окрашенный хромофор 9,15,9'-три-цис-ζ-каротина под действием фитоиндесатуразы (PDS; EC 1.3.5.5). Фермент катализирует симметричное введение первых двух из четырех двойных связей, необходимых для образования полностью транс-ликопина [2, 7]. Молекулы PDS собираются в каталитически активные гомотетрамеры, образующие гидрофобный туннель, который связывает 15-цис-фитоин (высоко гидрофобный субстрат) и пластохинон (косубстрат). Механизм действия гербицида связан с эффективностью его конкуренции с пластохиноном за сайт связывания в туннеле PDS-тетрамера [2]. Исследование PDS выявило несколько сайтов, мутации в которых препятствуют взаимодействию с карбонилом норфлуразона; введение таких мутаций обеспечивает устойчивость растения к хлорсодержащим гербицидам с сохранением биосинтеза каротиноидов [2].
Критичность корректной работы PDS продемонстрирована и для запасающих органов растения. Так, снижение уровня транскриптов PDS в плодах томата не только сокращает количество каротиноидов, но также приводит к падению экспрессии ключевого для созревания MADS-box гена RIPENING INHIBITOR (RIN) и генов, являющихся мишенями фактора транскрипции (ТФ) RIN [8]. Это может быть следствием прямого взаимодействия белков PDS и RIN, либо, под влиянием дефицита каротиноидов, роста синтеза других метаболитов (например, АБК) и белков, которые подавляют транскрипцию RIN [8]. В свою очередь, сверхэкспрессия RIN в растениях томата с мутацией rin приводит к повышению экспрессии PDS и накоплению каротиноидов [9]. Отметим, что среди подтвержденных мишеней ТФ RIN находятся многие гены каротиногенеза (Phytoene synthase PSY1, 15-cis-zeta-carotene-isomerase Z-ISO, Lycopene beta-cyclase LCY-b, Zeta-carotene desaturase ZDS, Carotenoid isomerase CrtISO) [10, 11].
То, что ингибирование активности PDS приводит к “отбеливанию” в норме зеленой фотосинтезирующей ткани, позволило исследователям использовать PDS как репортерный ген для оценки эффективности CRISPR-Cas9-редактирования генома растений [12, 13], а также для тестирования системы вирус-индуцированного подавления генов (VIGS) [14]. Транзиентное VIGS-замалчивание гена PDS с целью обесцвечивания хлорофилл-содержащих тканей существенно облегчает визуализацию флуоресценции слитых с GFP белков в листьях растений и представляет собой альтернативу конфокальной лазерной сканирующей микроскопии [15]. PDS-эффект используется не только для отработки различных методов, но и в целях исследования различных аспектов развития и адаптации растения. Так, показано, что нарушение функции хлоропластов за счет подавления фитоиндесатуразы усиливает системное накопление переносимого тлей вируса в листьях Nicotiana benthamiana Domin [16].
В данном исследовании мы использовали систему CRISPR-Cas9 для выключения гена PDS в геноме Nicotiana tabacum L. с целью изучения защитного/адаптационного ответа растения путем изменения активности других генов пути каротиногенеза, а также MADS-box генов, кодирующих гомологи ТФ RIN. Нами были получены независимые трансгенные линии, химерные по нокауту PDS за счет однобуквенных вставок/делеций (со сбивкой рамки считывания) в экзоне II гена. Выключение гена приводило к нарушениям фенофаз и других морфологических характеристик растения, а также вызывало альбиносность листовой ткани, снижение содержания каротиноидов и хлорофиллов. У редактированных линий наблюдалось падение уровня экспрессии гена PDS и рост экспрессии других генов каротиногенеза (PSY2, ZISO, CRTISO, CrtB1, LCYE, NSY и VDE). Значительные изменения наблюдались в уровне экспрессии стресс-чувствительных генов, кодирующих MADS-доменные факторы транскрипции (N. tabacum AGL8, MADS23, SEP1, SEP2 и SEP3). Полученные результаты подчеркивают важную роль фермента PDS в метаболизме каротиноидов, фотосинтезе и фотозащите у табака, а также предполагают возможное опосредованное участие PDS в регуляции стресс-чувствительных MADS-box генов.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал. Объектом исследования стал табак N. tabacum L. сорт Samsun. Для получения стерильной культуры in vitro семена тепличных растений стерилизовали в парах хлора. Открытые микропробирки с семенами (~10–20 мг) помещали в стеклянный эксикатор над емкостью с 50–100 мл хлорсодержащего моющего средства (типа “Доместос”) с добавлением 2–3 мл концентрированной HCl. Эксикатор плотно закрывали крышкой и оставляли на 2 ч, после чего семена помещали на чашки Петри с агаризованной (0.7%) средой MS (Murashige and Skoog basal nutrient medium including vitamins; M5519, Sigma-Aldrich, США) с добавлением 10 г/л сахарозы (S9378; Sigma-Aldrich). Проростки переносили в 0.5 л пластиковые контейнеры с той же средой и выращивали in vitro в климатической камере в условиях длинного фотопериода (16 ч день/8 ч ночь, 21°C) в течение 3–4 недель до фазы 5–7 листа.
Конструкция для CRISPR-Cas9 редактирования. С целью выявления наиболее консервативных участков гена PDS (N. tabacum NCBI Gene ID LOC107816873) кДНК гена сравнивали с гомологичными последовательностями видов табака (N. attenuata LOC109223965; N. tomentosiformis LOC104107035; N. sylvestris LOC104248773; N. benthamiana LC543533.1) и Cyanobacteria bacterium J055 (RFHR01000124.1). Область 5'-конца гена, включавшую два первых экзона NtPDS (360 нуклеотидов от ATG-кодона), использовали для подбора направляющей (guid; g) РНК с помощью web-программ CRISPRdirect (http://crispr.dbcls.jp) и CRISPR MultiTargeter (http://www.multicrispr.net/basic_input.html).
Конструкцию (для CRISPR/Cas9-введения случайных мутаций в кодирующую последовательность NtPDS, соответствующую выбранной gРНК) собирали на основе бинарного вектора p201N_Cas9 (AddGene, Watertown, MA, США; кат. № 59175; https://www.addgene.org/) в соответствии с протоколом (https://media.addgene.org/data/plasmids/47/ 47024/47024-attachment_rNUYUYROwjgZ.pdf) [17]. Т-область вектора включала ген Cas9 (под двойным промотором CaMV35S), ген NPTII (устойчивость к канамицину; для селекции трансгенных растений) и сайт I-PpoI для клонирования кассеты экспрессии gРНК (управляемой промотором U6.6 Medicago truncatula), предварительно собранной в челночном векторе pUC_gРНК (AddGene Кат. № 47024) [17]. Корректность сборки плазмиды p201N-NtPDS-gRNA проверяли методами ПЦР и рестрикции согласно [17] и использовали для трансформации штамма Agrobacterium tumefaciens LBA4404. Агробактерию, содержащую пустой вектор p201N, применяли для трансформации растений в качестве контроля.
Трансформация растений табака. Для трансформации использовали экспланты культуры in vitro табака (нарезанные секторами (~0.5 × 0.5 см) пластины верхних листьев с предварительно удаленными жилками).
Суспензию агробактерии для инфицирования эксплантов получали из свежей культуры агробактерии, выращенной в течение ночи (+28°С, 150 rpm) в 5 мл жидкой среды LB, содержащей рифампицин (Rif, 12.5–25.0 мг/л) и канамицин (Km, 100 мг/л). Культуру центрифугировали (1500 g, 10 мин), осадок суспендировали в 20 мл жидкой среды А2 (MS, 3% сахароза; pH 5.5) в стерильной чашке Петри, где замачивали экспланты (40 мин в темноте; верхней поверхностью листа вверх) в суспензии агробактерии (p201N – контроль; содержащие плазмиду p201N-NtPDS-gRNA – опыт). Инфицированные экспланты просушивали с помощью фильтровальной бумаги, выкладывали верхней стороной листа вниз на чашки Петри с агаризованной (0.7%) средой А2 и выдерживали в темноте при комнатной температуре в течение 2 сут. Далее экспланты отмывали от агробактерии (15 мин в жидкой А2 среде, содержащей карбенициллин (Cb) 500 мг/л), просушивали на фильтровальной бумаге и помещали на чашки с агаризованной (0.7%) средой А3 (MS, 2% глюкоза, 0.1 мг/л NAA (1-Naphthaleneacetic acid), 1 мг/л BAP (6-Benzylaminopurine), Km 100 мг/л и Cb 500 мг/л). Раз в две недели экспланты перекладывали на свежую среду А3. Образующиеся побеги срезали с каллуса, укореняли на среде А3 без BAP, адаптировали к грунту и тепличным условиям.
Анализ целевого участка гена NtPDS. Геномную ДНК экстрагировали из целого листа каждой трансгенной линии Т0 с использованием набора ZR-96 Plant/Seed DNA Kit (Zymo Research, США). Область 178 п. н. гена NtPDS, содержащую целевой сайт gРНК, амплифицировали методом ПЦР с использованием праймеров PDS2exF и PDS3exR (табл. 1) при следующих условиях: начальная денатурация 5 мин при 95°С, 35 циклов (30 с при 95°С, 30 с при 59°С, 1 мин при 72°С), 10 мин при 72°С. Продукты ПЦР ожидаемого размера выделяли из агарозного геля с помощью набора QIA-EX® II Gel Extraction Kit (QIAGEN, Германия), клонировали в вектор pGEM®-T Easy (pGEM®-T Easy Vector System I, Promega, США) и секвенировали на секвенаторе ДНК ABI Prism 377 (Applied Biosystems, США). Полученные последовательности ДНК анализировали на возможные полиморфизмы с помощью программы MEGA 7.0 (https://www.megasoftware.net/).
Таблица 1.
Последовательности праймеров
| Ген N. tabacum (NCBI ID) | Последовательность (5' → 3') |
|---|---|
| Амплификация редактируемой области гена NtPDS | |
| NtPDS (LOC107816873) | TGCATTGATTATCCAAGACCAG GTTTGTGACCAGCATCTGC |
| РВ-ПЦР | |
| NtPDS (LOC107816873) | GCTCTTCCTGCGCCATTAAATG CTTGCTTTCTCATCCAGTCCTT |
| NtPSY2 (LOC107772713; LOC107761716) | AACTGGACTCTGCTAGTAGATG GCACAAGAGATTTTGCATAAGCA |
| NtZDS (LOC107772271) | CAGCAGATACGTGATTTGGAT ACAGTCAATGAATCCAAGAGCA |
| NtCRTISO (LOC107832559; LOC107791326) | ATGAAGCAAAGAAAGAGCTTGT GCAAGGTATCGTCTATGTGTCT |
| NtCrtRB1 (LOC107809222; LOC107822278) | ACTATGGTTTCTTCCATAAAGGCCT GCGGCCACCTTCCTTAAATAAG |
| NtVDE (LOC107780507; LOC107763628) | CCTGACGAAACGGAATGTCAG AGTCACCAACATCAGATTTACGA |
| NtWUS (NtWUSCHEL-like [LOC107796712; LOC107812471; MG843891.1; JQ686923.1]) | CTTCTCATGGTGTACTGGCC CAGTTCCTCATAATCGTCTACTAG |
| NtSEP1 (SEPALLATA 1-like [LOC107776641; LOC107789627]; гомолог гена Arabidopsis SEPALLATA 1) | GCAACATGCTCAATCTCAGG TTGGGCATTTGTTACTGCTG |
| NtSEP2 (NtMADS6-like [LOC107821033; LOC107818774]; гомолог генов Arabidopsis AGL6 и SEPALLATA 2) | AATAATGGCGGAACAGATGG TGGATCAGGTTCACATTCCA |
| NtSEP3 (LOC107773468; LOC107764357; JQ686934.1; гомолог гена Arabidopsis AGL9) | TCACTTGAGAGGCAGCTTGA CATCGCCCTGAGTTTGAGTT |
| NtAGL8 (LOC107777773; гомолог гена Arabidopsis AGL8 [836212]) | ATCAGTGGGATCAGCAGAACCA ACCTCCTTCCACTTCTCCATTATC |
| NtMADS23-like (LOC107825287; гомолог генов S. tuberosum MADS23-like [LOC102593715] и Arabidopsis ANR1 [LOC9321977]) | CAATTGGCAACTCCAACTTCTGAG CGTCCTTTCTTACACGAACACCA |
| actin-7 (LOC107831145) | ACAGTGTCTGGATTGGAGGATC GAAGCACTTCCTGTGGACAATG |
Анализ экспрессии генов. Для анализа использовали ткань верхушечных листьев побега, достигшего фазы развития 8–10 листьев. Из ткани листа (~0.2–0.5 г), предварительно измельченной растиранием в ступке в жидком азоте, выделяли суммарную РНК с помощью набора RNeasy Plant Mini Kit (QIAGEN, Германия). Удалив возможные примеси геномной ДНК (RNase-free DNase set, QIAGEN, Германия), препарат анализировали на качество (электрофорез в 1.5% агарозном геле) и использовали для синтеза (с праймером oligo-dT) первой цепи кДНК (GoScript Reverse Transcription System, Promega, США). Концентрации РНК и кДНК измеряли флуориметрически (Qubit® Fluorometer, Thermo Fisher Scientific).
Количественную ПЦР в реальном времени (РВ-ПЦР) проводили в двух биологических и трех технических повторах на приборе CFX96 Real-Time PCR Detection System (Bio-Rad Laboratories, США) с использованием 3.0 нг кДНК, ген-специфических праймеров (табл. 1) и смеси SYBR Green RT-PCR (Синтол, Россия) при следующих условиях: исходная денатурация 5 мин при 95°С, далее 40 циклов (15 с при 95°С; 40 с при 60°С). Полученные данные нормализовали по экспрессии референсного гена Nicotiana tabacum Actin-7 (LOC107831145) [18].
Анализ содержания каротиноидов, хлорофиллов и антоцианов. В ткани листа трансгенных/редактированных и контрольных растений измеряли количество (мг/г сырого веса) хлорофиллов (a и b), каротиноидов (сумма) и антоцианов (сумма) согласно [19]. Спектры поглощения регистрировали на спектрофотометре Eppendorf BioSpectrometer® basic (Eppendorf, Германия).
Структурный и статистический анализ. Для сопутствующего работе структурного анализа последовательностей ДНК и белка использовали программы NCBI-BLAST и NCBI-CDD (https:// blast.ncbi.nlm.nih.gov/) и MEGA 7.0 (https://www.megasoftware.net/). Статистическую обработку экспрессионных и биохимических данных проводили с помощью программы Graph Pad Prism v. 8 (https://www.graphpad.com, США). Результаты выражали как среднее значение ± стандартное отклонение (SD) на основе двух биологических и трех технических повторов. Для оценки различий применяли критерий Стьюдента; P < 0.01 считали показателем статистической значимости.
РЕЗУЛЬТАТЫ
Трансгенные линии табака N. tabacum с редактированным геном NtPDS. Для редактирования гена NtPDS в качестве gРНК была выбрана последовательность 5'-GAGATTGTTATTGCTGGTGCAGG-3' (в положении 334–356 п. н. от ATG-кодона, кДНК, конец экзона II). Была собрана плазмидная конструкция p201N-NtPDS-gRNA и введена в штамм A. tumefaciens LBA4404, который был использован для трансформации листовых эксплантов табака сорта Samsun.
В качестве контроля использовали нетрансгенные растения (WT) и растения, трансформированные пустым вектором (p201N). Каких-либо морфологических отличий от WT-контроля растения p201N не имели. В сравнении с WT, укоренившиеся на канамицине регенеранты Т0NtPDS-gRNA имели измененную в разной степени окраску листа – от полностью белой (такие регенеранты гибли после образования корней и первых истинных листьев) до зеленой. Из полученных 27 жизнеспособных трансгенных (укоренившихся, канамицин-резистентных) линий 18 формировали зеленые листья, и девять – мозаично альбиносные (зеленые с белыми пятнами) (рис. 1). Цветки всех трансгенных линий не имели каких-либо заметных фенотипических отклонений.
Рис. 1.
Трансгенные растения табака N. tabacum с мозаично нокаутированным геном фитоиндесатуразы NtPDS (б, в, д) в сравнении с нетрансгенным контролем (а, г).

Полученные трансгенные растения NtPDS-gRNA формировали две группы, характеризующиеся заметной мозаичной альбиносностью листьев (I) и ее отсутствием (II). В свою очередь, каждая группа делилась на три подгруппы: рано цветущие (А) (2 и 4 линии, соответственно), поздно цветущие (Б) (4 и 1) и цветущие, как контроль (В) (3 и 10). Кроме того, трансгенные линии (I) и (II) отличались от нетрансгенного контроля нарушением ряда других морфологических параметров (табл. 2).
Таблица 2.
Характеристики трансгенных растений табака в сравнении с нетрансгенным контролем.
| Образец | Варианты аллеля NtPDS | Дни до цветения | Высота, см | Листья, шт. | Цветки, шт. | Коробочки, шт. | Вес семян, г | |
|---|---|---|---|---|---|---|---|---|
| WT | Сроки цветения (vs. WT) | WT | 122.50 ± 17.80 | 133.80 ± 15.20 | 40.80 ± 7.80 | 13.70 ± 5.60 | 11.10 ± 5.12 | 0.11 ± 0.05 |
| Группа (I) | (А) Норма: L2, L16, L24 | 350^351→ins^g# 350^351→ins^t# t351→del(1nt)# 341t→c 351t→c |
121.00 ± 15.00 | 83.33 ± 13.02 | 32.33 ± 2.33 | 5.00 ± 3.00 | 3.67 ± 2.12 | 0.11 ± 0.03 |
| (Б) Ранние: L3, L7 | 61.50 ± 3.30 | 65.5 ± 4.50 | 36.5 ± 3.50 | 9.00 ± 3.00 | 6.50 ± 1.50 | 0.07 ± 0.01 | ||
| (В) Поздние: L13, L28–30 | 304.25 ± 94.56 | 86.00 ± 17.05 | 44.50 ± 11.50 | 6.00 ± 3.00 | 4.50 ± 2.50 | 0.08 ± 0.02 | ||
| Группа (II) | (А) Норма: L1, L11, L14, L15, L17, L19, L20, L22, L26, L27 | 341t→c 351t→c Нет SNPs или инделей |
117.75 ± 15.75 | 73.25 ± 13.75 | 28.25 ± 2.25 | 4.75 ± 3.75 | 4.00 ± 3.00 | 0.11 ± 0.04 |
| (Б) Ранние: L5, L18, L21, L23 | 65.30 ± 12.50 | 72.70 ± 9.50 | 26.70 ± 3.57 | 9.90 ± 3.50 | 7.30 ± 3.00 | 0.11 ± 0.03 | ||
| (В) Поздние: L12 | 297.00 ± 0.00 | 121.00 ± 0.00 | 42.00 ± 0.00 | 8.00 ± 0.00 | 6.00 ± 0.00 | 0.12 ± 0.00 | ||
Так, в сравнении с WT, линии группы (I) формировали укороченный за счет сокращения междоузлий стебель и меньше цветков/коробочек (все три подгруппы А–В), а также меньше листьев (А и Б) и семян (Б и В). Подгруппа Б цвела раньше (в ~2 раза) и подгруппа В позже (в ~2.5 раза), чем WT (табл. 2).
Линии группы (II), несмотря на отсутствие визуально регистрируемой альбиносности листьев, характеризовались сходными с (I) изменениями в сравнении с WT, за исключением количества семян (вес семян на одну коробочку не отличался от контроля) (табл. 2).
Анализ целевого сайта NtPDS в геноме трансгенных линий (клонирование и секвенирование) обнаружил различные варианты мутаций (однонуклеотидные индели со сдвигом рамки считывания и замены (SNP)) в редактируемом участке гена. Было показано, что в группе (I) из 10 клонов каждой линии 10–20% содержат пять вариантов измененного аллеля NtPDS: SNPs 341t→c и 351t→c; индели t351→del(1nt)#, 350^351→ins^g# или 350^351→ins^t#. Анализ клонов, полученных для одного нежизнеспособного растения с почти полностью белыми листьями, показал вставку 350^351→ins^g# в 9 случаях из 10 секвенированных. В случае растений группы (II) среди 10 клонов каждой линии были обнаружены только варианты с SNPs (рис. 2, табл. 2).
Рис. 2.
Результаты секвенирования (сверху) и выравнивания (снизу) целевого сайта гена фитоиндесатуразы NtPDS с указанием обнаруженных полиморфизмов (замены и индели даны в соответствии с табл. 2).
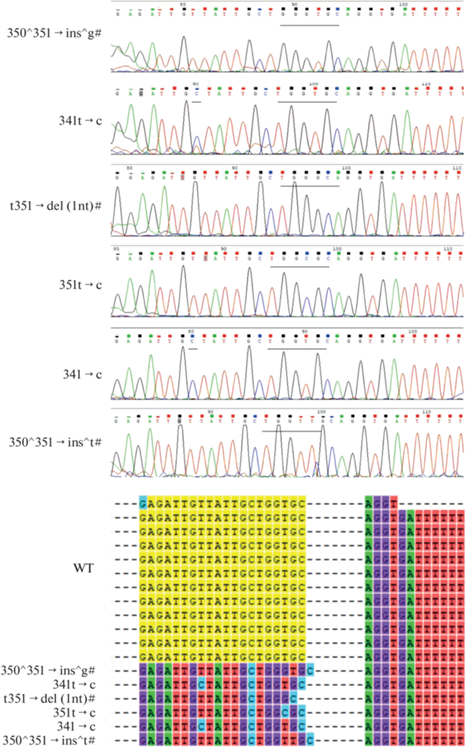
Таким образом, однонуклеотидные индели со сдвигом рамки считывания легко детектировались у линий табака группы (I), которые формировали мозаично обесцвеченные листья. У растений группы (II) индели не обнаружены. Учитывая аналогичные группе (I) отличия растений группы (II) от контроля, данный факт может быть связан с существенно меньшим количеством редактированных клеток и секвенированием числа клонов, недостаточного для обнаружения мутаций.
Структурный анализ редактированной последовательности NtPDS показал, что все три инделя приводят к сбою рамки считывания. Образуются преждевременные стоп-кодоны и, как следствие, значительно укороченные и измененные на С-конце варианты белка: 154 (в случае 350^351→ins^g# и 350^351→ins^t#) и 144 (t351→del(1nt)#) аминокислотных (а. о.) остатка при корректном размере NtPDS 582 а. о. В случае инсерций первый преждевременный стоп-кодон образуется через 111 нуклеотидов после сбивки рамки считывания, и С-конец мутантного белка состоит из 37 некорректных а. о. остатков [CRSGWFVYSKISGRCWSQTAIAGGKRCPRWKGSCMER]. При делеции неправильный С-конец формируется из 27 а. о. [QVWVVCLQQNIWQMLVTNRYCWRQEMS].
Среди обнаруженных SNPs, один (t351→c) является синонимичным, тогда как второй (t341→c) приводит к нейтральному замещению V114→A в N-концевой области функционального домена PLN02612 (расположен на участке 21–578 а. о. белка NtPDS).
Содержание пигментов в линиях табака дикого типа и трансгенных/редактированных линиях. Воздействие инделей в последовательности гена фитоинсинтазы NtPDS на морфологию растений, включая пигментацию фотосинтезирующей ткани, предполагает нарушение биосинтеза каротиноидов. Для оценки такой возможности был проведен сравнительный анализ содержания каротиноидов (сумма), хлорофиллов (a и b) и антоцианов (сумма) в листьях табака дикого типа (WT) и двух трансгенных поздних линий L28 и L29 из группы (I). Кроме фенотипического проявления нокаута NtPDS, выбор линий основывался на значительном их отличии от контроля по времени инициации цветения: 346 (L28) и 461 (L29) день против 122 дней в случае WT.
В результате было показано, что в сравнении с контролем количество как каротиноидов, так и хлорофиллов в редактированных линиях уменьшилось в среднем в ~1.5 раза, тогда как содержание антоцианов не изменилось (табл. 3).
Таблица 3.
Содержание пигментов в ткани листа контрольных и опытных (позднецветущих) растений
| Растение* | Содержание пигментов (мг/г сырого веса) | |||
|---|---|---|---|---|
| сумма каротиноидов | хлорофилл a | хлорофилл b | сумма антоцианов | |
| WT | 0.142 ± 0.012 | 0.394 ± 0.024 | 0.169 ± 0.012 | 0.030 ± 0.004 |
| L28 | 0.094 ± 0.021 | 0.263 ± 0.031 | 0.117 ± 0.017 | 0.024 ± 0.002 |
| L29 | 0.095 ± 0.027 | 0.263 ± 0.035 | 0.119 ± 0.021 | 0.028 ± 0.002 |
Примечание: * содержание каротиноидов и хлорофиллов в альбиносной ткани листа регенеранта (рис. 1в) носит следовый характер.
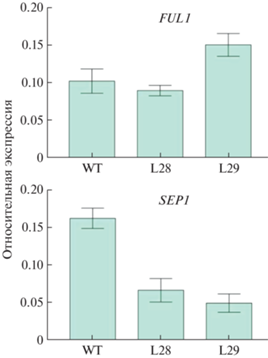
Экспрессия генов, связанных с биосинтезом каротиноидов, в листьях табака дикого типа и трансгенных/редактированных линий. Известно, что количество субстрата и/или продукта метаболических путей влияет на экспрессию соответствующих генов [8, 20]. С учетом химерности редактирования полученных линий L28 и L29 мы проверили, во-первых, уровень транскриптов полноразмерного гена NtPDS, и, во-вторых, как влияет изменение его экспрессии, а также содержания каротиноидов, на экспрессию генов NtPSY2, NtCrtISO, NtZDS, NtLCYB, NtCrtRB1 и NtVDE в листьях WT, L28 и L29.
Результаты показали, что в сравнении с WT уровень транскриптов NtPDS снизился в ~2.0 раза у линии L28 и поднялся в ~2.2 раза у L29, тогда как экспрессия NtCrtRB1 у обеих линий L28 и L29 не изменилась. Уровень транскриптов остальных анализируемых генов существенно вырос у линии L29: в ~1.7 (NtLCYB), ~4.0 (NtCrtISO), ~2.9 (NtPSY2), ~1.9 (NtZDS) и ~2.6 (NtVDE) раз. В то же время в листьях L28 он остался неизменным, за исключением NtPSY2, экспрессия которого увеличилась в ~1.7 раза (рис. 3).
Рис. 3.
Экспрессия структурных генов пути биосинтеза каротиноидов NtPDS, NtPSY2, NtCrtISO, NtZDS, NtLCYB, NtCrtR-B1 и NtVDE в листьях контрольного нетрансгенного растения (WT) и позднецветущих мозаично редактированных линий L28 и L29. * Р < 0.01 – статистически значимое различие от значений WT-контроля.
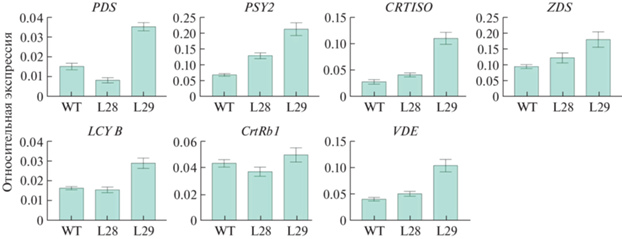
Таким образом, взаимосвязи между уровнями транскриптов NtPDS (суммарно нередактированного и редактированных), экспрессией нижестоящих генов каротиногенеза и содержанием каротиноидов в листе не наблюдалось.
Экспрессия регуляторных MADS-box генов в листьях табака дикого типа и трансгенных/редактированных линий. Из исследований томата Solanum lycopersicum L. известно, что промотор многих структурных генов каротиногенеза содержит сайт связывания с MADS-доменными белками. В тканях плода томата транскрипция этих генов находится под контролем ТФ RIN (подсемейство SEPALLATA (SEP) MADS-доменных белков), и уровень экспрессии гена RIN взаимосвязан с содержанием каротиноидов [8, 21]. В вегетативной ткани, кроме SEP, в ответ на стресс активируются гены и других MADS-доменных ТФ (к примеру, гомологи Arabidopsis thaliana AGAMOUS-LIKE 8 (AGL8) и ARABIDOPSIS NITRATE REGULATED 1 (ANR1)) [22, 23].
С учетом этих данных, мы провели оценку влияния изменений в каротиногенезе редактированных растений на экспрессию MADS-box генов NtSEP1, NtSEP2, NtSEP3 (представлены 3 клады подсемейства SEP), NtAGL8 и NtMADS23 (гомолог ANR1). В результате были обнаружены транскрипты NtSEP1 и NtAGL8, тогда как остальные анализируемые гены не экспрессировались в листе растений WT, L28 или L29. При этом уровень транскриптов NtSEP1 у L28 и L29 был более чем в 2 раза ниже, чем в контроле; NtAGL8 экспрессировался у WT, L28 и L29 приблизительно на одном уровне (рис. 4).
ОБСУЖДЕНИЕ
Метаболизм каротиноидов регулируется в растении с помощью известного и хорошо охарактеризованного ферментативного механизма, в котором фитоиндесатураза PDS катализирует первую стадию модификации 15-цис-фитоина – предшественника всех каротиноидов [1, 24, 25]. Различные исследования показывают, что как подавление каталитической активности PDS, так и нокаут/замалчивание кодирующего его гена, критично для растения, поскольку приводит к нарушениям фотосинтеза, фотозащиты и гормонального баланса [2, 12–14]. Уровень экспрессии гена PDS напрямую связан с эффективностью каротиногенеза во всех тканях растения и количеством накапливаемых каротиноидов в запасающих органах [26].
В данном исследовании ген PDS был нокаутирован в геноме табака N. tabacum (сорт Samsun) с помощью CRISPR/Cas9-системы. Девять независимых трансгенных линий формировали листья со слабо пигментированными участками, мозаично локализованными на зеленом фоне (рис. 1б, д). Регенеранты с почти полным обесцвечиванием листьев (рис. 1в) были нежизнеспособными. Подобные изменения соответствовали ранее показанному фенотипу растений с отсутствием транскрипции гена PDS [14] или экспрессией нефункционального варианта белка PDS [12, 13].
Наблюдаемый паттерн пигментации наглядно свидетельствовал о мозаичности факта нокаутирования гена, что считается обычным при CRISPR/Cas9-редактировании с использованием агробактериальной трансформации растения [27]. Мозаичность способствовала выживанию регенерантов, в отличие от побегов с почти полностью белой тканью листа (рис. 1в), что еще раз подчеркивает критичность роли PDS для поддержания жизненно важных процессов в растении.
Предположительно лежащее в основе полученного фенотипа нарушение биосинтеза каротиноидов было подтверждено результатами биохимического и генетического анализа. Существенное сокращение количества каротиноидов в листовой ткани (табл. 3) согласовалось с присутствием однонуклеотидных инделей в экзоне II гена PDS (табл. 2, рис. 2), приводящих к синтезу коротких вариантов белка. По меньшей мере, их размер (144 или 154 а. о.) исключал формирование функционального гомотетрамера PDS. Так, на примере риса Oryza sativa показано, что PDS-гомодимеры в тетрамерном комплексе стабилизируются четырьмя водородными связями между Thr207, Trp216 и Arg220 одного мономера и карбонильными атомами кислорода Pro239, Leu235 и Gln263 соседнего мономера (то есть между а. о., расположенными после 200 а. о.) [2].
Таким образом, нефункциональные, нокаутированные версии PDS табака прекращали синтез каротиноидов в самом начале пути, в результате чего в хлоропластах редактированных клеток отсутствовали ксантофиллы (непосредственные участники работы фотосинтетического аппарата и фотозащиты растения [1]), что, вероятно, нарушало функцию хлоропластов [15]. Более того, там отсутствовали побочные продукты каротиногенеза – фитогормоны АБК и стриголактоны, необходимые для регуляции различных аспектов развития [4, 5], что может объяснить другие (помимо мозаичной пигментации листа) отличия редактированных линий от контроля (табл. 2).
Экспрессионный анализ линий L28 и L29, отличающихся от WT-контроля и друг от друга серьезным сдвигом по времени инициации цветения (L28/L29/WT – 346/461/122 дней), обнаружил существенные изменения уровня транскриптов NtP-DS и NtPSY2 у обеих линий, и всех генов, кроме NtCrtRB1, у линии L29 (наиболее поздней) (рис. 3). Серьезное отставание по цветению линий L28 и L29 от контроля могло быть отражением нарушений как фотосинтеза/фотозащиты (следствие дефицита ксантофиллов), так и физиологических процессов (следствие дефицита фитогормонов). К примеру, для апокаротиноида АБК показано как положительное, так и отрицательное воздействие на время цветения растения [28]. Рост уровня транскриптов почти всех генов каротиногенеза (рис. 3) сопровождается пониженным (в сравнении с WT) содержанием каротиноидов (табл. 3) в листьях линии L29. Это допускает влияние количества субстрата и/или продукта каротиногенеза на генную экспрессию, как это предполагается для различных метаболических путей [8, 20]. Повышенной экспрессии NtPSY2 в нередактированных клетках линии L28 оказалось, вероятно, достаточно для обеспечения пороговых (для цветения) значений продуктов каротиноидного пути. Существенное отставание линии L29 не только от контроля, но и от линии L28, возможно потребовало активации не только гена фитоинсинтазы NtPSY2, но и других нижестоящих генов пути (рис. 3), в особенности NtLCYB и NtVDE, играющих критичные роли в биосинтезе ксантофиллов и, следовательно, фотосинтезе и фотозащите [1].
Поскольку время цветения (как и другие аспекты развития) связано с фотопериодом и фотосинтезом [29, 30], существенное изменение содержания каротиноидов является для растения стрессом. Таким образом, дифференциальная экспрессия генов каротиногенеза в ответ на мозаичный нокаут NtPDS может свидетельствовать о вынужденной адаптации растения к изменению содержания продуктов каротиноидного пути посредством компенсаторного увеличения их синтеза в нередактированных клетках.
Повышение уровня транскриптов генов каротиногенеза может происходить за счет активации их регуляторов, какими, к примеру, могут быть MADS-доменные ТФ [10, 11]. Так, в плодах томата транскрипция гена PDS находится под контролем MADS-доменного ТФ RIN; экспрессия гена RIN прямо зависит либо от уровня транскриптов PDS, либо от содержания каротиноидов [8]. Ген RIN входит в подсемейство SEP, отдельные члены которого (SEP1 и SEP4) транскрибируются не только в плодах, но и в листьях [31]. В подтверждение предполагаемых корреляций с содержанием каротиноидов [8], листья обеих проанализированных линий L28 и L29 отличались от WT существенным снижением уровня транскриптов NtSEP1 (рис. 4). С учетом вышесказанного, а также роли MADS-доменных ТФ в АБК-опосредованном ответе растений на абиотические стрессы [22, 32, 33], дифференциальная экспрессия гена NtSEP1 (рис. 4) предполагает его участие в регуляции биосинтеза каротиноидов в листьях табака (в том числе, в ответ на стрессы).
Таким образом, в данном исследовании мы нокаутировали ген фитоинсинтазы NtPDS в геноме табака N. tabacum с использованием системы CRISPR-Cas9 и проанализировали полученные трансгенные линии на содержание каротиноидов в совокупности с экспрессией структурных генов пути синтеза каротиноидов и, возможно, участвующих в их регуляции MADS-box генов. Полученные результаты мозаичного редактирования гена свидетельствуют о том, что индели (350^351→ins^g#, 350^351→ins^t# и t351→del(1nt)#) в экзоне II гена NtPDS приводят к образованию нефункционального белка NtPDS, что изменяет метаболизм каротиноидов, время инициации цветения и морфологические характеристики растений табака. Дифференциальная экспрессия NtSEP1 в контрольных нетрансгенных растениях и позднецветущих редактированных линиях L28 и L29 предполагает участие ТФ NtSEP1 в регуляции биосинтеза каротиноидов и побочных продуктов пути, в том числе в ответ на стресс.
Исследование выполнено при финансовой поддержке Российского научного фонда (грант № 19-16-00016) и Министерства науки и высшего образования РФ. В работе использована экспериментальная установка искусственного климата (ЭУИК, Федерального исследовательского центра Биотехнологии Российской академии наук).
Авторы заявляют об отсутствии конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследования.
Список литературы
Maoka T. Carotenoids as natural functional pigments // J. Nat. Med. 2020. V. 74. P. 1. https://doi.org/10.1007/s11418-019-01364-x
Brausemann A., Gemmecker S., Koschmieder J., Ghisla S., Beyer P., Einsle O. Structure of phytoene desaturase provides insights into herbicide binding and reaction mechanisms involved in carotene desaturation. Structure // 2017. V. 25. P. 1222. https://doi.org/10.1016/j.str.2017.06.002
Demmig-Adams B., Adams W.W. Antioxidants in photosynthesis and human nutrition // Science. 2002. V. 298. P. 2149. https://doi.org/10.1126/science.1078002
Chen K., Li G.J., Bressan R.A., Song C.P., Zhu J.K., Zhao Y. Abscisic acid dynamics, signaling, and functions in plants // J. Integr. Plant Biol. 2020. V. 62. P. 25. https://doi.org/10.1111/jipb.12899
Mashiguchi K., Seto Y., Yamaguchi S. Strigolactone biosynthesis, transport and perception // Plant J. 2021. V. 105. P. 335. https://doi.org/10.1111/tpj.15059
Babu R., Rojas N.P., Gao S., Yan J., Pixley K. Validation of the effects of molecular marker polymorphisms in LcyE and CrtRB1 on provitamin A concentrations for 26 tropical maize populations // Theor. Appl. Genet. 2013. V. 126. P. 389. https://doi.org/10.1007/s00122-012-1987-3
Wagner T., Windhövel U., Römer S. Bansformation of tobacco with a mutated cyanobacterial phytoene desaturase gene confers resistance to bleaching herbicides // Z. Naturforsch. C. J. Biosci. 2002. V. 57. P. 671. https://doi.org/10.1515/znc-2002-7-821
Naing A.H., Kyu S.Y., Pe P.P.W., Park K.I., Lee J.M., Lim K.B., Kim C.K. Silencing of the phytoene desaturase (PDS) gene affects the expression of fruit-ripening genes in tomatoes // Plant Methods. 2019. V. 15: 110. https://doi.org/10.1186/s13007-019-0491-z
Dong T., Chen G., Tian S., Xie Q., Yin W., Zhang Y., Hu Z. A non-climacteric fruit gene CaMADS-RIN regulates fruit ripening and ethylene biosynthesis in climacteric fruit // PLoS One. 2014. V. 9: e95559. https://doi.org/10.1371/journal.pone.0095559
Fujisawa M., Nakano T., Ito Y. Identification of potential target genes for the tomato fruit-ripening regulator RIN by chromatin immunoprecipitation // BMC Plant Biol. 2011. V. 11: 26. https://doi.org/10.1186/1471-2229-11-26
Li S., Xu H., Ju Z., Cao D., Zhu H., Fu D., Grierson D., Qin G., Luo Y., Zhu B. The RIN-MC fusion of MADS-box transcription factors has transcriptional activity and modulates expression of many ripening genes // Plant Physiol. 2018. V. 176. P. 891. https://doi.org/10.1104/pp.17.01449
Chen L., Li W., Katin-Grazzini L., Ding J., Gu X., Li Y., Gu T., Wang R., Lin X., Deng Z., McAvoy R.J., Gmitter F.G., Deng Z., Zhao Y., Li Y. A method for the production and expedient screening of CRISPR/Cas9-mediated non-transgenic mutant plants // Hortic. Res. 2018. V. 5: 13. https://doi.org/10.1038/s41438-018-0023-4
Bánfalvi Z., Csákvári E., Villányi V., Kondrák M. Generation of transgene-free PDS mutants in potato by Agrobacterium-mediated transformation // BMC Biotechnol. 2020. V. 20: 25. https://doi.org/10.1186/s12896-020-00621-2
Xu H., Xu L., Yang P., Cao Y., Tang Y., He G., Yuan S., Ming J. Tobacco rattle virus-induced PHYTOENE DESATURASE (PDS) and Mg-chelatase H subunit (ChlH) gene silencing in Solanum pseudocapsicum L. // PeerJ. 2018. V. 6: e4424. https://doi.org/10.7717/peerj.4424
Zhang L., Gase K., Baldwin I., Gális I. Enhanced fluorescence imaging in chlorophyll-suppressed tobacco tissues using virus-induced gene silencing of the phytoene desaturase gene // Biotechniques. 2010. V. 48. P. 125. https://doi.org/10.2144/000113345
DeBlasio S.L., Rebelo A.R., Parks K., Gray S.M., Heck M.C. Disruption of chloroplast function through downregulation of phytoene desaturase enhances the systemic accumulation of an aphid-borne, phloem-restricted virus // Mol. Plant Microbe Interact. 2018. V. 31. P. 1095. https://doi.org/10.1094/MPMI-03-18-0057-R
Jacobs T.B., LaFayette P.R., Schmitz R.J., Parrott W.A. Targeted genome modifications in soybean with CRISPR/Cas9 // BMC Biotechnology. 2015. V. 15: 16. https://doi.org/10.1186/s12896-015-0131-2
Nezhdanova A.V., Slugina M.A., Dyachenko E.A., Kamionskaya A.M., Kochieva E.Z., Shchennikova A.V. Analysis of the structure and function of the tomato Solanum lycopersicum L. MADS-box gene SlMADS5 // Vavilovskii Zhurnal Genet Selektsii. 2021. V. 25. P. 492. https://doi.org/10.18699/VJ21.056
Filyushin M.A., Dzhos E.A., Shchennikova A.V., Kochieva E.Z. Dependence of pepper fruit colour on basic pigments ratio and expression pattern of carotenoid and anthocyanin biosynthesis genes // Russian Journal of Plant Physiology. 2020. V. 67. P. 1054. https://doi.org/10.1134/S1021443720050040
Meugnier E., Rome S., Vidal H. Regulation of gene expression by glucose // Curr. Opin. Clin. Nutr. Metab. Care. 2007. V. 10. P. 518. https://doi.org/10.1097/MCO.0b013e3281298fef
Slugina M.A. Transcription factor RIPENING INHIBITOR and its homologs in regulation of fleshy fruit ripening of various plant species // Russian Journal of Plant Physiology. 2021. V. 68. P. 783. https://doi.org/10.1134/S1021443721050186
Castelán-Muñoz N., Herrera J., Cajero-Sánchez W., Arrizubieta M., Trejo C., García-Ponce B., Sánchez M.P., Álvarez-Buylla E.R., Garay-Arroyo A. MADS-box genes are key components of genetic regulatory networks involved in abiotic stress and plastic developmental responses in plants // Front. Plant Sci. 2019. V. 10: 853. https://doi.org/10.3389/fpls.2019.00853
Zhang Y., Cai W., Wang A., Huang X., Zheng X., Liu Q., Cheng X., Wan M., Lv J., Guan D., Yang S., He S. MADS-box protein AGL8 interacts with chromatin-remodeling component SWC4 to activate thermotolerance and environment-dependent immunity in pepper // J. Exp. Bot. 2023. erad092. https://doi.org/10.1093/jxb/erad092
Cazzonelli C.I., Pogson B.J. Source to sink: regulation of carotenoid biosynthesis in plants // Trends Plant Sci. 2010. V. 15. P. 266. https://doi.org/10.1016/j.tplants.2010.02.003
Quian-Ulloa R., Stange C. Carotenoid Biosynthesis and Plastid Development in Plants: The Role of Light // Int. J. Mol. Sci. 2021. V. 22: 1184. https://doi.org/10.3390/ijms22031184
Yan P., Gao X.Z., Shen W.T., Zhou P. Cloning and expression analysis of phytoene desaturase and ζ-carotene desaturase genes in Carica papaya // Mol. Biol. Rep. 2011. V. 38. P. 785. https://doi.org/10.1007/s11033-010-0167-2
Cui Y., Zhao J., Gao Y., Zhao R., Zhang J., Kong L. Efficient multi-sites genome editing and plant regeneration via somatic embryogenesis in Picea glauca // Front. Plant Sci. 2021. V. 12: 751891. https://doi.org/10.3389/fpls.2021.751891
Shu K., Luo X., Meng Y., Yang W. Toward a molecular understanding of abscisic acid actions in floral transition // Plant Cell Physiol. 2018. V. 59. P. 215. https://doi.org/10.1093/pcp/pcy007
Song Y.H., Ito S., Imaizumi T. Flowering time regulation: photoperiod- and temperature-sensing in leaves // Trends Plant Sci. 2013. V. 18. P. 575. https://doi.org/10.1016/j.tplants.2013.05.003
Yang G., Li W., Fan C., Liu M., Liu J., Liang W., Wang L., Di S., Fang C., Li H., Ding G., Bi Y., Lai Y. Genome-wide association study uncovers major genetic loci associated with flowering time in response to active accumulated temperature in wild soybean population // BMC Genomics. 2022. V. 23: 749. https://doi.org/10.1186/s12864-022-08970-2
Parenicová L., de Folter S., Kieffer M., Horner D.S., Favalli C., Busscher J., Cook H.E., Ingram R.M., Kater M.M., Davies B., Angenent G.C., Colombo L. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis: new openings to the MADS world // Plant Cell. 2003. V. 15. P. 1538. https://doi.org/10.1105/tpc.011544
Li X., Yu B., Wu Q., Min Q., Zeng R., Xie Z., Huang J. OsMADS23 phosphorylated by SAPK9 confers drought and salt tolerance by regulating ABA biosynthesis in rice // PLoS Genet. 2021. V. 17: e1009699. https://doi.org/10.1371/journal.pgen.1009699
Zhao P.X., Zhang J., Chen S.Y., Wu J., Xia J.Q., Sun L.Q., Ma S.S., Xiang C.B. Arabidopsis MADS-box factor AGL16 is a negative regulator of plant response to salt stress by downregulating salt-responsive genes // New Phytol. 2021. V. 232. P. 2418. https://doi.org/10.1111/nph.17760
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений