Генетика, 2019, T. 55, № 12, стр. 1466-1470
Скорость эволюции митохондриального гена цитохрома b согласно анализa недавней (около 12 000 лет) изоляции гольцов Salvelinus озера Кроноцкое
С. В. Шедько *
Федеральный научный центр Биоразнообразия наземной биоты Восточной Азии
Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
* E-mail: shedko@biosoil.ru
Поступила в редакцию 06.12.2018
После доработки 01.02.2019
Принята к публикации 11.02.2019
Аннотация
На основе анализа генетических отличий популяций гольцов Salvelinus с известным временем изоляции (граница верхнего плейстоцена–голоцена) определена скорость нуклеотидных замещений для митохондриального гена цитохрома b – 3.11 × 10–8 замен на позицию/год (95%-ный доверительный интервал: 2.59 × 10–8–3.64 × 10–8). Эта оценка примерно в 5 раз выше той, что получена для данного гена у лососевых рыб при рассмотрении длительных интервалов времени (филогенетической скорости) – 0.66 × 10–8 (0.56 × 10–8–0.77 × 10–8). Таким образом показано, что правило падения оценок скорости нуклеотидных замещений в мтДНК по мере продвижения от недавних отметок времени к древним, отнесенным на миллионы лет в прошлое, распространяется также и на линию лососевых рыб.
Скорость нуклеотидных замещений в последовательности ДНК (μ) – одна из важнейших статистик в молекулярной филогенетике и популяционной генетике, которая позволяет датировать и исследовать процесс эволюции на видовом или популяционном уровне, а также приходить к абсолютным оценкам важных популяционных показателей. Сравнительно недавно было установлено [1], что для одной и той же группы организмов скорость нуклеотидных замещений, рассчитанная для длительных (млн или десятки млн лет) интервалов времени, не совпадает с оценками μ, полученными при анализе коротких (десятки–сотни тысяч лет) периодов дивергенции. Оценки скорости мутирования, полученные при рассмотрении родословных внутри популяции, оказываются выше, чем скорость нуклеотидных замещений, оцененная при расхождении популяций одного вида, а последняя, в свою очередь, выше, чем скорость, рассчитанная для давно разделившихся видов (филогенетическая скорость). Поэтому используемая калибровка скорости эволюции ДНК должна, по возможности, находиться в соответствии с временным масштабом решаемых задач. Для популярного при изучении различных аспектов биологии и эволюции лососевых рыб (Salmonidae) генетического маркера – митохондриальной ДНК (мтДНК) – оценки филогенетической скорости были получены во многих работах, начиная с 90-х годов прошлого века [2, 3]. Однако для интервалов времени порядка 104 лет, важных для популяционных исследований лососевых, таких оценок до сих пор получено не было. Проведенный в настоящей работе анализ имеющихся данных по изменчивости мтДНК гольцов Salvelinus из озера Кроноцкое [4, 5] позволил этот пробел отчасти закрыть.
Кроноцкое озеро – крупнейшее пресноводное озеро на п-ове Камчатка с площадью водного зеркала порядка 240 км2 и максимальной глубиной около 150 м. Озеро находится на высоте 373 м над уровнем моря, с которым соединяется через вытекающую из него р. Кроноцкая, длиной около 40 км. Неподалеку от истока реки, примерно на траверзе воображаемой линии, соединяющей вершины двух находящихся в 8–10 км крупных вулканов – Кроноцкой сопки (высота – 3.5 км, диаметр основания – 16 км) и сопки Крашенинникова (высота – около 1.9 км), имеется порожистый участок, непроходимый для мигрирующих вверх по течению рыб. Известно, что озеро появилось в результате вулканической деятельности. В самом конце верхнего плейстоцена–голоцене (около 12 000 лет назад) произошла серия извержений Кроноцкой сопки и лавовые потоки суммарной мощностью более 200 м полностью погребли речную долину р. Пра-Лиственничной. Возникшая лавовая плотина привела к образованию Кроноцкого озера [6, 7]. Именно тогда скорее всего и возникла серия водопадов и порогов, сделавших невозможным миграцию рыб вверх по течению р. Кроноцкая. Озеро Кроноцкое, как крупный изолированный водоем с наличием разнообразных ниш, послужил ареной для бурного формообразования. В настоящее время из рыб его населяют две формы жилой нерки-кокани, производные от проходной нерки, а также целый букет форм гольцов, производных от проходной мальмы Salvelinus malma – белый, носатый, длинноголовый и др. [8, 9]. Недавние исследования дифференциации кроноцких гольцов по последовательностям двух участков мтДНК (D-петли и Cytb) показали малые генетические расстояния между формами гольцов (0.0034 замещений/позицию и менее), но, одновременно, существенные различия между ними в составе и частоте гаплотипов мтДНК [4, 5]. Установлено, что уникальные для озерных гольцов гаплотипы отличаются одной-двумя заменами от массовых гаплотипов, встречающихся также у проходной мальмы из р. Кроноцкая. Сделан вывод, что из двух ранее предложенных сценариев – серии последовательных вселений в озеро проходной мальмы и ее дифференциации [8] и симпатрического формообразования [10] – наиболее вероятен второй [4, 5].
На наш взгляд, эти изолированные популяции кроноцких гольцов представляют собой удобный случай для расчета скорости эволюции мтДНК гольцов на коротком временном отрезке. Она была оценена двумя различными способами.
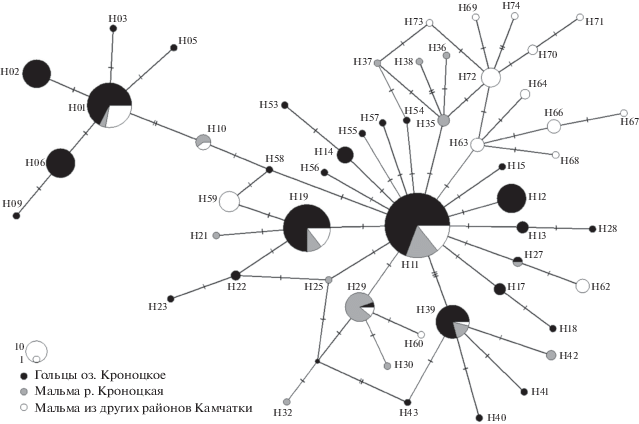
Первый способ [11] основывался на подсчете накопленных нуклеотидных замен, которые появились в мтДНК особей-основателей популяции уже после их изоляции. Понятно, что мутации могли возникать как у изолированных гольцов из озера, так и у мальмы из р. Кроноцкая. Так как последняя могла обмениваться генами с мальмой из других рек п-ова Камчатки, то у мальмы из р. Кроноцкая отделить автохтонно возникшие новые гаплотипы от гаплотипов, полученных в результате притока генов из соседних популяций, с уверенностью невозможно. Поэтому объектом анализа выступили только озерные гольцы. Из двух исследованных у кроноцких гольцов участков мтДНК только фрагмент гена Cytb обладал необходимым уровнем изменчивости. Выявленные у гольцов из оз. Кроноцкое и мальмы из р. Кроноцкая варианты нуклеотидных последовательностей фрагмента гена Cytb (1015 пн) получены из Gen Bank (JN868278–868489). Гаплотипы фрагмента гена Cytb выделены с помощью пакета Fabox [12] и приведены в соответствие с оригинальными гаплотипами из работы [4]. К ним были добавлены новые гаплотипы, обнаруженные в дополнительных выборках кроноцких гольцов, а также у мальмы из различных районов Камчатки [5]. Из той же работы были взяты и выборочные частоты гаплотипов гена Cytb. Далее методом TCS [13], реализованным в программе PopART [14], была построена филогенетическая сеть (рис. 1). На ее основе были установлены предковые для озерных гольцов гаплотипы. Предковым гаплотипом считался тот, который являлся общим для гольцов из озера, с одной стороны, и р. Кроноцкая и/или остальной Камчатки, с другой. Затем были подсчитаны отличия производных гаплотипов с учетом частоты их встречаемости и получена оценка среднего числа накопленных нуклеотидных различий – ρ. Доверительный интервал показателя ρ был установлен в 1000 циклах бутстрепа, произведенных с помощью пакета boot [15] в среде программирования R. Деление значения ρ на длину фрагмента Cytb и время изоляции (12 000 лет) давало искомую оценку скорости нуклеотидных замещений (μ), выраженную в числе различий на позицию/год/линию.
Расчеты, проведенные по первому способу, не принимали во внимание то, что изоляция речных гольцов от озерных может быть неполной [16]. Проникновение в популяцию мальмы из р. Кроноцкая генов от озерных гольцов могло, в принципе, сказаться на результатах расчетов (повлиять на интерпретацию предковых и производных гаплотипов и соответственно на оценку μ). Поэтому в дополнение к первому мы применили иной подход, в котором расчет скорости нуклеотидных замещений производился при использовании модели изоляции с миграцией (IM-модель), предназначенной для коалесцентного анализа дивергенции популяций одного вида или недавно разделившихся видов, обменивающихся генами [17]. В этой модели в момент времени t предковая популяция разделяется на две дочерних, после чего они обмениваются генами со скоростью m. Модель изначально была разработана для анализа данных по одному нерекомбинирующему локусу (мтДНК, к примеру) в рамках правдоподобного и байесовского подходов, когда расчет всех ее параметров производится совместно при использовании метода Монте-Карло по схеме марковских цепей (Markov Chain Monte Carlo, MCMC). В настоящей работе была задействована одна из последних реализаций IM-модели – компьютерная программа IMa2p, позволяющая проводить вычисления в параллельном режиме [18]. Эта модель была применена для анализа дивергенции в двух парах популяций, по которым имелись показательные выборки: носатый голец (n = 80) – мальма р. Кроноцкая (n = 57) и белый голец (n = 119) – мальма р. Кроноцкая. MCMC-симуляции генных генеалогий производились на основе эволюционной модели HKY со стартовым равномерным распределением вероятностей (uniform prior: –m 40 –q 40 –t 40) путем параллельного пуска 20 цепей Маркова с протяженностью в 2 000 000 шагов, с отбором каждого сотого из них и отжигом начального (не достигшего конвергенции) фрагмента каждой цепи (burn-in) длиной в 200 000 шагов. В результате для искомых параметров эффективный размер выборок (Effective Sample Size, ESS) составлял 1000 и более. Собственно, сам расчет скорости нуклеотидных замещений проводился следующим образом. На выходе программы IMa2p бралась оценка параметра времени дивергенции t0, представляющего собой произведение t × μ (t – время, прошедшее с момента разделения популяций, выраженное в числе поколений, μ – нейтральная скорость мутирования, выраженная в числе замен на локус на поколение). Отсюда оценку μ, выраженную в числе замен в год на позицию, получали путем деления оценки t0 на время дивергенции (12000 лет) и затем на длину (1015 пн) фрагмента ДНК.
Филогенетическая сеть (TCS-нетворк) для 53 гаплотипов гена Cytb, найденых у 232 кроноцких озерных гольцов, 57 мальм из р. Кроноцкая и 77 мальм из других районов Камчатки, приведена на рис. 1. Из 29 гаплотипов, выявленных у четырех форм озерных гольцов, к предковым было отнесено шесть: H01, H11, H19, H27, H29 и H39. Производные гаплотипы идентифицированы у 83 особей. Семьдесят восемь из них отличались от ближайшего к ним предкового гаплотипа по одной нуклеотидной позиции и пять – по двум. Среднее отличие от предкового гаплотипа отсюда составило 0.379 замен на ген (95%-ный доверительный интервал, рассчитанный методом бутстрепа – 0.315–0.444). Таким образом, скорость нуклеотидных замещений оказалась равной 3.11 × 10–8 (=0.379/1015/12 000) замен на позицию/год (95%-ный доверительный интервал: 2.59 × 10–8–3.64 × 10–8).
Результаты IM-анализа дифференциации популяций озерных гольцов и мальмы из р. Кроноцкая показали, что скорость двусторонней миграции генов (в мутационных единицах на поколение на генную копию) в паре сравнения носатый голец– мальма р. Кроноцкая существенно не отличается от нуля. В паре белый голец–мальма она, согласно тесту отношения оценок правдоподобия, достоверно выше нуля – Λ = 5.32 (P < 0.05). При этом миграция была асимметрична: скорость притока генов в популяцию белого гольца из популяции мальмы р. Кроноцкая составляла незначительную, близкую к нулю, величину – 0.02, в то время как в обратном направлении она была на два порядка выше – 1.70 (95%-ный интервал наивысшей плотности апостериорного распределения, HPD: 0.14–4.70).
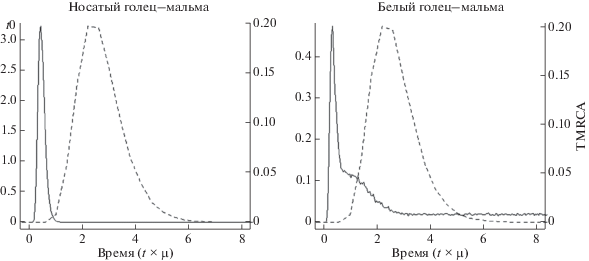
Время, прошедшее с момента разделения популяций озерных и речных гольцов (t0), согласно полученным оценкам, невелико и составляет примерно 15% от времени, прошедшего с момента существования последнего общего предка (TMRCA) гаплотипов Cytb в объединенной выборке этих гольцов – 0.42 (95%-ный интервал HPD: 0.18–0.90) для пары мальмы с носатым гольцом и 0.34 для пары с белым гольцом (рис. 2). Отсюда скорость нуклеотидных замещений оказывается равной 3.45 × 10–8 замен на позицию/год (0.42/12000/1015) для пары носатый голец–мальма (95%-ный интервал HPD: 1.48 × 10–8–7.39 × 10–8) и 2.79 × 10–8 (0.34/12 000/1015) для пары белый голец–мальма (нижняя граница 95%-ного интервала HPD – 1.81 × 10–8). Эти оценки, как можно видеть, оказались весьма близки к оценке μ, полученной первым способом.
Рис. 2.
Апостериорная кривая плотности распределения двух параметров – времени, прошедшего с момента разделения популяций (t0, сплошная линия), и времени, прошедшего с момента существования наиболее близкого общего предка гаплотипов гена Cytb (TMRCA, пунктир), рассчитанных для двух пар сравнения кроноцких озерных гольцов и мальмы из р. Кроноцкая.
С другой стороны, филогенетическая скорость эволюции гена Cytb, рассчитанная с помощью программы BEAST v. 1.8.4 [19] при тех же условиях, что и ранее [20], для филогенетического ствола видов Salvelinus (интервал времени – 7–19 млн лет назад, от момента его обособления и до базальной дихотомии линий гольцов) составила 0.63 × 10–8 замен/позицию/год на линию (95%-ный интервал HPD: 0.41 × 10–8–0.85 × 10–8). Такая же скорость получена для филогенетического дерева Salmonidae в целом (включая все три его основные линии – лососей, сигов и хариусов, разделившиеся порядка 42 млн лет назад [20]) – 0.66 × 10–8 (95%-ный интервал HPD: 0.56 × 10–8–0.77 × 10–8). Обе эти оценки примерно в 5 раз ниже оценок скорости эволюции гена Cytb, полученных для масштаба времени изоляции кроноцких гольцов. Таким образом, результаты настоящей работы показывают, что правило падения оценок скорости нуклеотидных замещений в мтДНК по мере продвижения от недавних отметок времени к древним, удаленным на миллионы лет в прошлое [1], распространяется и на линию лососевых рыб.
Проведенная в настоящей работе настройка “молекулярных часов” для популярного генетического маркера позволит по-другому взглянуть на плейстоцен-голоценовую историю расселения, дивергенции и динамику численности популяций лососевых рыб.
Исследование проводилось в рамках темы НИР № АААА-А17-117062710083-0. Результаты получены с использованием оборудования ЦКП “Дальневосточный вычислительный ресурс” ИАПУ ДВО РАН (https://www.cc.dvo.ru).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Список литературы
Ho S.Y.W., Phillips M.J., Cooper A., Drummond A.J. Time dependency of molecular rate estimates and systematic overestimation of recent divergence times // Mol. Biol. Evol. 2005. V. 22. № 7. P. 1561–1568. https://doi.org/10.1093/molbev/msi145
Шедько С.В. О скорости эволюции митохондриальной ДНК лососевых рыб // Журн. эволюцион. биохимии и физиологии. 1991. Т. 27. № 2. С. 249–254.
Smith G.R. Introgression in fishes: significance for paleontology, cladistics, and evolutionary rates // Syst. Biol. 1992. V. 41. № 1. P. 41–57. https://doi.org/10.1093/sysbio/41.1.41
Сенчукова А.Л., Павлов С.Д., Мельникова М.Н., Мюге Н.С. Генетическая дифференциация гольцов (род Salvelinus) из озера Кроноцкое на основе анализа митохондриальной ДНК // Вопр. ихтиол. 2012. Т. 52. № 4. P. 489–499.
Senchukova A.L., Mugue N.S., Pavlov S.D., Mel’nikova M.N. On the origin of charrs of the genus Salvelinus of the Kronotskoe Lake and their relationships with other charr populations of the Kamchatka peninsula // J. Ichthyol. 2013. V. 53. № 10. P. 840–848. https://doi.org/10.1134/S0032945213100093
Мелекесцев И.В., Брайцева О.А., Эрлих Э.Н., Кожемяка Н.Н. Вулканические горы и равнины // История развития рельефа Сибири и Дальнего Востока. Камчатка, Курильские и Командорские острова. М.: Наука, 1974. С. 162–233.
Гущенко И.И. Вулкан Кроноцкий // Действующие вулканы Камчатки: в 2-х т. Т. 2. М.: Наука, 1991. С. 52–61.
Викторовский Р.М. Механизмы видообразования у гольцов Кроноцкого озера. М.: Наука, 1978. 112 с.
Есин Е.В., Маркевич Г.Н. Гольцы рода Salvelinus азиатской части Северной Пацифики: происхождение, эволюция и современное разнообразие. Петропавловск-Камчатский: Камчатпресс, 2017. 188 с.
Савваитова К.А. Арктические гольцы. М.: Агропромиздат, 1989. 224 с.
Forster P., Harding R., Torroni A., Bandelt H.-J. Origin and evolution of Native American mtDNA variation: a reappraisal // Am. J. Hum. Genet. 1996. V. 59. № 4. P. 935–945.
Villesen P. FaBox: an online toolbox for fasta sequences // Mol. Ecol. Notes. 2007. V. 7. № 6. P. 965–968. https://doi.org/10.1111/j.1471-8286.2007.01821.x
Clement M., Posada D., Crandall K.A. TCS: A computer program to estimate gene genealogies // Mol. Ecol. 2000. V. 9. № 10. P. 1657–1659. https://doi.org/10.1046/j.1365-294x.2000.01020.x
Leigh J.W., Bryant D. PopART: Full-feature software for haplotype network construction // Methods Ecol. Evol. 2015. V. 6. № 9. P. 1110–1116. https://doi.org/10.1111/2041-210X.12410
Canty A., Ripley B. boot: Bootstrap R (S-Plus) Functions. R package version 1.3-20. 2017. 117 p.
Павлов С.Д., Кузищин К.В., Груздева М.А. и др. Фенетическое разнообразие и пространственная структура гольцов (Salvelinus) озерно-речной системы Кроноцкая (восточная Камчатка) // Вопр. ихтиол. 2013. Т. 53. № 6. С. 645–670. https://doi.org/10.7868/S004287521306009X
Nielsen R., Wakeley J. Distinguishing migration from isolation: a Markov chain Monte Carlo approach // Genetics. 2001. V. 158. № 2. P. 885–896.
Sethuraman A., Hey J. IMa2p – parallel MCMC and inference of ancient demography under the isolation with migration (IM) model // Mol. Ecol. Resours. 2016. V. 16. № 1. P. 206–215. https://doi.org/10.1111/1755-0998.12437
Drummond A.J., Suchard M.A., Xie D., Rambaut A. Bayesian phylogenetics with BEAUti and the BEAST 1.7 // Mol. Biol. Evol. 2012. V. 29. № 8. P. 1969–1973. https://doi.org/10.1093/molbev/mss075
Шедько С.В., Мирошниченко И.Л., Немкова Г.А. Филогения лососевых рыб (Salmoniformes: Salmonidae) и ее молекулярная датировка: анализ мтДНК-данных // Генетика. 2013. Т. 49. № 6. С. 718–734. https://doi.org/10.7868/S0016675813060118
Дополнительные материалы отсутствуют.