Генетика, 2019, T. 55, № 2, стр. 136-145
Сравнительный геномный анализ плазмиды вирулентности Salmonella enterica подвид enterica серотип Enteritidis
А. В. Раков 1, *, Ф. Н. Шубин 1
1 Научно-исследовательский институт эпидемиологии и микробиологии
им. Г.П. Сомова
690087 Владивосток, Россия
* E-mail: rakovalexey@gmail.com
Поступила в редакцию 16.03.2018
После доработки 15.05.2018
Принята к публикации 17.03.2018
Аннотация
Некоторые серотипы Salmonella enterica подвид enterica содержат серотипоспецифические плазмиды вирулентности. Доступность полных последовательностей плазмиды вирулентности S. Enteritidis (pSEV) позволила проследить ее эволюционные изменения. Нами были изучены плазмиды вирулентности S. Enteritidis из различных штаммов для выявления эволюции pSEV и определения предковой плазмиды и ее точного размера. Сравнение всех доступных последовательностей плазмид вирулентности S. Enteritidis показало, что они консервативны и ограничены в размерах. Эти размеры колебались от 59 336 до 59 374 пн, а более половины плазмид имели размер, равный 59 372 пн. Генный состав плазмиды является консервативным и состоит из 81 открытой рамки считывания с небольшой псевдогенизацией плазмид, размер которой отличался от 59 372 пн. Предположено, что прототипом плазмиды вирулентности S. Enteritidis pSEV является плазмида из SEJ-подобного предкового штамма с размером 56 372 пн и таким же нуклеотидным и генным составом, как S. Enteritidis штамм SEJ. Последовательность данной плазмиды может быть использована в качестве референсной для всех будущих работ по плазмиде вирулентности S. Enteritidis.
Salmonella enterica подвид enterica серотип Enteritidis (S. Enteritidis) остается первым по значимости серотипом в этиологии сальмонеллезной инфекции у человека во многих странах мира [1] и в том числе России. Широко известно, что некоторые серотипы Salmonella enterica подвид enterica содержат серотипоспецифические плазмиды вирулентности [2]. Общим для всех известных плазмид вирулентности S. enterica подвид enterica является оперон spvRABCD размером 7.8 тпн, необходимый для развития системной инфекции на животных моделях [3]. S. Enteritidis содержит плазмиду вирулентности размером 59 тпн, обозначенную pSEV (plasmid Salmonella Enteritidis Virulence) [4]. По данным 25-летнего мониторинга за S. Enteritidis в РФ, около 94% клинических штаммов и 89% штаммов из окружающей среды содержат плазмиду вирулентности (наши неопубликованные данные).
Поскольку плазмиды вирулентности сальмонелл имеют значительный размер, их копийность низка (2–3 копии на клетку) и поэтому они более стабильны по сравнению с плазмидами меньшего размера [5]. Напротив, некоторые авторы полагают, что из-за малого числа копий плазмиды вирулентности их случайное распределение в дочерние клетки приводило бы к очень частой утрате плазмиды [6], однако этого не происходит. Гены на плазмиде вирулентности в основном хорошо охарактеризованы, но значимость некоторых из них до сих пор остается неясной.
У некоторых серотипов сальмонелл плазмиды вирулентности различны по размеру (например, S. Choleraesuis и S. Dublin) [4, 7]. Хорошо известно, что плазмиды вирулентности сальмонелл содержат специфичные репликоны FIB(S) и FII(S), которые представляют собой подгруппу плазмид IncF, без каких-либо генов антибиотикорезистентности [8]. В последние годы плазмиды, сочетающие в себе часть плазмиды вирулентности вместе с генами антибиотикорезистентности, обнаружены у некоторых серотипов сальмонелл (S. Typhimurium, S. Enteritidis, S. Dublin, S. Choleraesuis). Конъюгативные плазмиды, несущие кассеты вирулентности и антибиотикорезистентности, переносимые интегроном, были выделены в клинических изолятах S. Typhimurium [9]. Гибридная плазмида вирулентности–антибиотикорезистентности S. Enteritidis pUO-SeVR1 была обнаружена в клинических изолятах из Испании в 2003 г. [10]. Показано, что в эволюции таких гибридных плазмид S. Enteritidis основную роль играет инсерционная последовательность IS26 [11]. Слияние R‑плазмид с плазмидами вирулентности имеет большое значение для эволюции высоковирулентных и антибиотикорезистентных клонов S. Enterica [12].
Технологии секвенирования ДНК значительно продвинулись за последние десять лет, а экспоненциальный рост баз данных секвенированных геномов обеспечил возможность для сравнительного анализа геномных последовательностей. Тем не менее все еще существует проблема правильной аннотации геномов [13]. Большинство плазмидных и хромосомных последовательностей, доступных в GenBank, аннотировано автоматически без какой-либо последующей проверки. Этим продиктована необходимость в применении новых усовершенствованных методов для реаннотации с последующей проверкой перед выполнением любого сравнительного анализа геномов.
Предполагается, что S. Enteritidis приобрела плазмиду вирулентности путем горизонтального переноса от эволюционно далеких серотипов [14], но изучение эволюции плазмиды pSEV в S. Enteritidis до настоящего времени не было проведено. За последние несколько лет секвенированы плазмиды вирулентности из различных изолятов S. Enteritidis [15–17].
Цель данной работы – сравнительное изучение последовательностей плазмид вирулентности различных штаммов S. Enteritidis для оценки вариабельности размеров и состава плазмиды вирулентности, нахождения возможного предка pSEV и определение ее точного размера.
Бактериальные штаммы и плазмиды. Полные последовательности плазмид вирулентности 13 штаммов S. Enteritidis были отобраны из базы нуклеотидов NCBI (http://www.ncbi.nlm.nih.gov/ nuccore/) (табл. 1). Для целей сравнения в анализ были включены последовательности двух предположительно гибридных плазмид вирулентности–антибиотикорезистентности: pFORC7 (Acc. No. CP009767) и pSEN-BT (Acc. No. LN879484), из штаммов S. Enteritidis FORC_007 и D7795 соответственно [18], поскольку они имели в своем составе spv-оперон. Для подтверждения принадлежности используемых штаммов к серотипу S. Enteritidis использован сайт SeqSero для проверки последовательности хромосомы, где это было возможно [19].
Таблица 1.
Штаммы и плазмиды S. Enteritidis, использованные в работе
| Обозначение плазмид | Обозначение штамма | Размер, пн | Число генов NCBI (CDS) |
Число генов
(реаннотация: GeneMark/ AMIGene/ Prodigal/RAST) |
Число генов (majority vote)/ Псевдогенов | Номер в GenBank | Источник получения | Страна | Год | Литературный источник | Группа несовместимости |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Нет | SEJ | 59 372 | 75/77 | 89/74/78/108 | 81/0 | CP008927 | н.д. | н.д. | н.д. | [15] | IncFII(S)/ IncFIB(S) |
| Нет | Durban | 59 372 | 75 | 89/74/78/108 | 81/0 | CP007508 | Больной | Южная Африка | 2013 | [32] | IncFII(S)/ IncFIB(S) |
| pSEN | P125109 | 59 372 | 70 | 90/74/79/109 | 81/0 | HG970000 | н.д. | н.д. | н.д. | [33] | IncFII(S)/ IncFIB(S) |
| н.д. | OLF-SE2-98984-6 | 59 372 | N/A | 88/74/78/108 | 81/0 | CP011840 | Окр. среда | Канада | 2000 | [16] | IncFII(S)/ IncFIB(S) |
| н.д. | OLF-SE3-98983-4 | 59 372 | N/A | 90/74/78/108 | 81/0 | CP011843 | Окр. среда | Канада | 2000 | [16] | IncFII(S)/ IncFIB(S) |
| pSE9-641 | EC20090641 | 59 372 | 71 | 89/74/78/108 | 81/0 | KT317611 | Куры | Канада | 2008 | [17] | IncFII(S)/ IncFIB(S) |
| pSE12-5 | EC20120005 | 59 372 | 71 | 88/74/78/107 | 81/0 | KT317613 | Куры | Канада | 2011 | [17] | IncFII(S)/ IncFIB(S) |
| pCMCC 50041 | CMCC50041 | 59 373 | 75/71 | 90/75/79/109 | 80/1 | CP013098 | н.д. | Дания | н.д. | [34] | IncFII(S)/ IncFIB(S) |
| pSLA5 | LA5 | 59 374 | 103 | 92/76/81/109 | 77/4 | HE663166 | Куры | н.д. | н.д. | [35] | IncFII(S)/ IncFIB(S) |
| plasmid00 | CDC_2010K_0968 | 59 369 | 75/71 | 89/74/76/107 | 79/2 | CP007529 | н.д. | США | 2010 | [36] | IncFII(S)/ IncFIB(S) |
| pSENV | LK5 | 59 368 | 84 | 91/74/77/105 | 80/1 | JN885080 | н.д. | н.д. | н.д. | [14] | IncFII(S)/ IncFIB(S) |
| pSE12-2 | EC20120002 | 59 363 | 73 | 88/75/83/106 | 80/1 | KT317612 | Куры | Канада | 2012 | [17] | IncFII(S)/ IncFIB(S) |
| pCFSAN 000006 | 18569 | 59 336 | 62/75 | 95/77/87/113 | 73/8 | CP011395 | Куры | Мексика | н.д. | [36] | IncFII(S)/ IncFIB(S) |
| pSEN-BT | D7795 | 85 310 | 108 | 116/110/112/145 | н.д. | LN879484 | Больной | Малави | н.д. | [18] | IncFII(S)/ IncFIB(S)/ IncQ1 |
| pFORC7 | FORC_007 | 101 428 | 131 | 131/125/124/148 | н.д. | CP009767 | Морепродукт | Южная Корея | 2014 | Lee*, 2015 | IncFIB(S)/ IncX1 |
Реаннотация. Автоматическая реаннотация была выполнена с помощью программ GeneMarkS [20], Prodigal [21], AMIGene [22] и сервера RAST [23]. Комбинация нескольких программ для автоматической аннотации с применением мажоритарной системы с проверкой вручную позволила повысить точность предсказаний [24, 25]. Инсерционные последовательности (IS-элементы) и гены антибиотикорезистентности определяли с помощью программ IS Finder [26] и ResFinder [27] соответственно. Определение группы несовместимости проводили на сервере PlasmidFinder [8].
Сравнительная геномика. Выравнивание последовательностей полных плазмид выполняли в программах Mauve [28] и Kalign2 [29]. Транслированные кодирующие последовательности сравнивали с последовательностями известных белков с использованием BLASTP [30]. Ортологи определяли как транслированные кодирующие последовательности с гомологией >90% с известными последовательностями белков в базе данных NCBI. Филогенетическое родство плазмид выявляли с помощью программы MEGA6 [31].
Секвенирование ДНК показало, что точный размер плазмид вирулентности S. Enteritidis варьирует от 59 336 до 59 374 пн. Более половины плазмид (7 из 13) имели размер 59 372 пн. Эти данные соответствуют описанным ранее, в которых было установлено, что размер pSEV составляет 59 372 пн [12]. Нами было показано, что размер может изменяться в определенных пределах, в зависимости от наличия инделов. Все исследованные плазмиды имели два специфичных для плазмид вирулентности сальмонелл репликона – FIB(S) и FII(S) [8].
После множественного выравнивания с помощью программ Mauve и Kalign2 было установлено, что плазмида вирулентности из генома S. Enteritidis SEJ идентична консенсусной последовательности и использовалась в последующем в качестве референсной, что положительным образом повлияло на качество результатов реаннотации. Для автоматической реаннотации использованы четыре программы: GeneMarkS, Prodigal, AMIGene и RAST. Число выявленных открытых рамок считывания (ORF) приведено в табл. 1. Наименьшее число ORF было получено при использовании AMIGene, а наибольшее – при использовании RAST. Мы сравнили результаты оригинальной аннотации с полученной нами реаннотацией с применением мажоритарного подхода для более корректного определения позиции старт-кодонов выявленных ORF. Число ORF для плазмид вирулентности размером 59 372 пн определено нами как 81, в то время как в оригинальных аннотациях оно значительно колебалось – от 62 (для плазмиды штамма pCFSAN000006) до 103 (для плазмиды штамма pSLA5). Два генома плазмиды вирулентности (штаммы OLF-SE2-98984-6 и OLF-SE3-98983-4) были изначально депонированы неаннотированными, а также не имели названия для плазмид вирулентности. Кроме того, не было обнаружено никаких различий по нуклеотидному составу среди обеих плазмид. Список 81 ORF (предполагаемых кодирующих последовательностей) представлен в табл. 2. Из них белки с известной функцией составили 54 (66.7%), предполагаемые и гипотетические белки – 19 (23.4%) и мобильные элементы – 8 (9.9%). Поиск гомологов подтвердил наличие следующих кластеров (оперонов) генов: pef-оперон (плазмид-кодируемые фимбрии), неполный tra-оперон [5], spv-оперон, а также гены, кодирующие белки деления плазмиды (parA/parB), белки, участвующие в SOS-репарации (psiA/psiB, umuC/umuD), и белки системы токсин–антитоксин II типа (ccdA/ccdB).
Таблица 2.
Кодируемые последовательности (открытые рамки считывания) плазмиды вирулентности штамма S. Enteritidis SEJ
| Номер п.п. | Расположение кодирующих последовательностей (в пн) от точки начала репликации |
Ген (гомолог или синоним) | Кодируемый продукт |
|---|---|---|---|
| 1 | 463...624 | Резольваза | |
| 2 | 712...966 | ygiW | Предполагаемый белок внутренней мембраны |
| 3 | 1770...2072 | pefB(papB) | Регуляторный белок основной субъеденицы фимбрий |
| 4 | 2347...2871 | pefA | Основная субъединица фимбрий |
| 5 | 3098...5506 | pefC(fimD) | Ашер-белок фимбрий |
| 6 | 5499...6191 | pefD(faeE) | Шаперон фимбрий |
| 7 | 6209...6766 | Предполагаемый белок внутренней мембраны | |
| 8 | 6856...7803 | Гипотетический белок | |
| 9 | 8063...8275 | pefI | Регулятор транскрипции FaeA-подобного семейства |
| 10 | 8260...8610 | rcsB | Регулятор транскрипции GerE-подобного семейства |
| 11 | 8589...8732 | Гипотетический белок | |
| 12 | 8854...9510 | srgA | Белок бисульфидного обмена DsbA |
| 13 | 9697...10542 | yaeT | Белок наружной мембраны YaeT |
| 14 | 10632...11189 | rck | Белок наружной мембраны Rck |
| 15 | 11455...12201 | envY(gadX) | Терморегуляторный порин EnvY |
| 16 | 12298...12519 | Белок наружной мембраны | |
| 17 | 13375...14244 | repA | Инициатор репликации плазмиды incFII семейства RepA |
| 18 | 14564...14809 | Репликативный белок | |
| 19 | 15009...15188 | Фосфолипаза D | |
| 20 | 15205...15489 | nuc | Эндонуклеаза |
| 21 | 15611...16063 | dsbA | Оксидоредуктаза DsbA |
| 22 | 16180...16602 | finO | Белок ингибирования конъюгации IncF плазмиды FinO |
| 23 | 16864...17196 | traV | Белок системы конъюгации IncF плазмиды TraV |
| 24 | 17193...17408 | trbD | Белок конъюгационного переноса TrbD |
| 25 | 17395...17991 | traP | Белок системы конъюгации IncF плазмиды TraP |
| 26 | 17981...19402 | traB | Белок системы конъюгации IncF плазмиды TraB |
| 27 | 19402...20142 | traK | Белок системы конъюгации IncF плазмиды TraK (секретин) |
| 28 | 20129...20695 | traE | Белок системы конъюгации IncF плазмиды TraE |
| 29 | 20717...21028 | traL | Белок системы конъюгации IncF плазмиды TraL |
| 30 | 21043...21405 | traA | Белок системы конъюгации IncF плазмиды TraA |
| 31 | 21447...21629 | traY | Регулятор системы конъюгации IncF плазмиды TraY |
| 32 | 21768...22139 | traJ | Регулятор системы конъюгации IncF плазмиды TraJ |
| 33 | 22647...23027 | traM | Белок передачи сигнала скрещивания для конъюгации IncF плазмиды TraM |
| 34 | 23020...23226 | Гипотетический белок | |
| 35 | 23443...23913 | Литическая транс-гликозилаза | |
| 36 | 24436...24726 | yubP | Гипотетический белок |
| 37 | 24773...25042 | Гипотетический белок | |
| 38 | 25552...26151 | psiA | Белок ингибирования SOS-репарации |
| 39 | 26145...26309 | psiB | Белок ингибирования SOS-репарации |
| 40 | 26352...28049 | parB | Белок деления плазмиды ParB |
| 41 | 28374...28796 | umuD(lexA) | Репаративный белок UmuD |
| 42 | 28796...30070 | umuC(dinB1) | Репаративный белок UmuC |
| 43 | 30152...31126 | parB(virB) | Белок деления плазмиды ParB |
| 44 | 31126...32331 | parA | Белок деления плазмиды ParA |
| 45 | 32746...33687 | Транспозаза | |
| 46 | 33684...34289 | Гипотетический белок | |
| 47 | 34346...34747 | Гипотетический белок | |
| 48 | 34865...35374 | yacC | Экзонуклеаза |
| 49 | 35367...36482 | tlpA(smc) | Суперспиральный белок TlpA |
| 50 | 36740...37228 | Предполагаемый цитоплазматический белок | |
| 51 | 37883...38623 | cynT | Углеродная ангидраза |
| 52 | 38830...39390 | rlgA(hin) | Резольваза RlgA |
| 53 | 39374...41038 | Транспозаза IS481 | |
| 54 | 41028...41996 | exeA | АТФаза (cистемы секреции II типа) |
| 55 | 42185...43222 | Транспозаза IS630 | |
| 56 | 43430...43555 | Гипотетический белок | |
| 57 | 43830...44723 | spvR(mkaC) | Активатор транскрипции генов вирулентности spv |
| 58 | 44897...45061 | rrrD | Лизоцим профага Gifsy-2 RrrD |
| 59 | 45235...46002 | spvA(mkaB) | Вирулентный белок SpvA |
| 60 | 46184...47959 | spvB | Актин-АДФ-рибозилтрансфераза SpvB |
| 61 | 48240...48965 | spvC(ospF) | Фосфотрининлиаза SpvC |
| 62 | 49227...49877 | spvD | Вирулентный белок SpvD |
| 63 | 50004...50429 | Транспозаза IS200 | |
| 64 | 50486...50836 | Транспозаза IS605 | |
| 65 | 50903...51319 | Инвазин/интимин | |
| 66 | 51343...51888 | lysM | Пептидогликан-связывающий белок LysM |
| 67 | 51619...51906 | Гипотетический белок | |
| 68 | 51986...52156 | Гипотетический белок | |
| 69 | 52183...52668 | Белок внутренней мембраны | |
| 70 | 52662...53150 | Регулятор транскрипции семейства CaiF/GrlA | |
| 71 | 53175...53888 | yahA | Дигуанилатциклаза/фосфодиэстераза |
| 72 | 53897...54679 | xerD | Тирозин-рекомбиназа |
| 73 | 54714...55235 | Предполагаемый цитоплазматический белок | |
| 74 | 55232...55522 | Цитоплазматический белок | |
| 75 | 55524...55829 | ccdB | Токсин СcdB |
| 76 | 55831...56049 | ccdA | Антитоксин CcdA |
| 77 | 56376...56504 | Гипотетический белок | |
| 78 | 56758...57246 | Гипотетический белок | |
| 79 | 57380...57604 | Гипотетический белок | |
| 80 | 57730...57918 | Предполагаемый цитоплазматический белок | |
| 81 | 59148...59267 | Гипотетический белок |
Сравнение геномов показало, что плазмиды вирулентности из нескольких штаммов S. Enteritidis имели мутации (сдвиг рамки считывания), которые могут приводить к возможному образованию псевдогенов. В табл. 3 приведен список замен аминокислот и их возможный эффект на трансляцию белка. Плазмиды S. Enteritidis штаммов Durban, OLF-SE2-98984-6, OLF-SE3-98983-4 имели только мутации в межгенных регионах и не имели мутаций, которые могли бы повлиять на состав белков. Поэтому функционально они являются такими же, как и S. Enteritidis штамм SEJ, и имеют такой же размер.
Таблица 3.
Список мутаций в генах исследованных плазмид вирулентности S. Enteritidis
| Обозначение штамма | Обозначение белка (в SEJ) | Замена аминокислоты (позиция в пн)/индел (позиция в пн) | Возможный эффект |
|---|---|---|---|
| Durban | – | – | – |
| OLF-SE2-98984-6 | – | – | – |
| OLF-SE3-98983-4 | – | – | – |
| EC20090641 | Белок системы конъюгации IncF плазмиды TraB | V18568I | Функциональный белок |
| EC20120002 | Гипотетический белок | 15980delGATTTCCA Сдвиг рамки |
Нефункциональный белок |
| CDC_2010K_0968 | Регулятор транскрипции FaeA-подобного семейства | 8128delCG Сдвиг рамки |
Нефункциональный белок |
| CDC_2010K_0968 | Гипотетический белок | 9540delC Сдвиг рамки |
Нефункциональный белок |
| CMCC50041 | Интеграза | E42,707R Q42,747S |
Возможно функциональный белок |
| CMCC50041 | Активатор транскрипции генов вирулентности spv | 44537insG Сдвиг рамки |
Нефункциональный белок |
| EC20120005 | Белок бисульфидного обмена DsbA | T9,206M G9,212D R9,251H A9,287G S9,427G |
Возможно функциональный белок |
| EC20120005, LA5, P125109, 18569 | Гипотетический белок | L9,543P A9,580T |
Возможно функциональный белок |
| LA5, P125109, 18569 | Гипотетический белок | G6,952S | Функциональный белок |
| LA5 | Гипотетический белок | 7349insA Сдвиг рамки |
Нефункциональный белок |
| LA5 | Белок бисульфидного обмена DsbA | 9253insCCCG Сдвиг рамки |
Нефункциональный белок |
| LA5, P125109, 18569 | Белок наружной мембраны YaeT | D10,125N R10,234Q A10,366D D10,384G |
Возможно функциональный белок |
| LA5, P125109, 18569 | Белок системы конъюгации IncF плазмиды TraE | I20,165N | Возможно функциональный белок |
| LA5 | Гипотетический белок | 23052insN Сдвиг рамки |
Нефункциональный белок |
| LA5, P125109, 18569 | Гипотетический белок | E40,984K | Функциональный белок |
| LA5 | Актин-АДФ-рибозилтрансфераза SpvB | 46386delC Сдвиг рамки |
Нефункциональный белок |
| 18569 | Белок системы конъюгации IncF плазмиды TraB | 18781insG Сдвиг рамки |
Нефункциональный белок |
| 18569 | Белок передачи сигнала скрещивания для конъюгации IncF плазмиды TraM | 22874delC Сдвиг рамки |
Нефункциональный белок |
| 18569 | Белок ингибирования SOS-репарации psiA | 25688insG 25923insT 25966delC 26006delGGGCA 26022delG 26037delGC 26088delAGGC 26114delTTAA Сдвиг рамки |
Нефункциональный белок |
| 18569 | Белок ингибирования SOS-репарации psiB | 26247delTGGGG 26260delA 26270delG Сдвиг рамки |
Нефункциональный белок |
| 18569 | Белок деления плазмиды ParB | 26721insTTT 26939delGC 27165delC GT27,408delC 27779delCAG Сдвиг рамки |
Нефункциональный белок |
| 18569 | Репаративный белок UmuD | 28422delG 28512delG 28630delC 28699delC 28720delG Сдвиг рамки |
Нефункциональный белок |
| 18569 | Репаративный белок UmuC | 29358delG 29492delC 29704delC 29940delG 30069delA Сдвиг рамки |
Нефункциональный белок |
| 18569 | Белок деления плазмиды ParA | 31286insG 31348insT Сдвиг рамки |
Нефункциональный белок |
| LK5 | Гипотетический белок | A4,124V | Функциональный белок |
| LK5 | Регулятор транскрипции FaeA-подобного семейства | G8,123D 8125delGCG(H) H8,199Q |
Нефункциональный белок |
| LK5 | АТФаза (cистемы секреции II типа) | V41,981L | Функциональный белок |
Гибридная плазмида pSEN-BT из штамма S. Enteritidis D7795 содержит репликоны FIB(S) и FII(S), а также репликон IncQ1. Она имеет часть генов плазмиды вирулентности вместе с детерминантами антибиотикорезистентности для аминогликозидов (strAB), бета-лактамов (blaTEM-1B), сульфаниламидов (sul1 и sul2), тетрациклина (tetA) и триметоприма (dfrA7). Гибридная плазмида pFORC7 из штамма S. Enteritidis FORC_007 содержит только один репликон FIB(S) от плазмиды вирулентности и репликон IncX1. Однако нам не удалось обнаружить какие-либо детерминанты антибиотикорезистентности в плазмиде pFORC7 (табл. 1).
Полные последовательности 13 плазмид pSEV использовали для построения дерева по алгоритму максимального правдоподобия (ММП) в программе MEGA6 [31]. Неукорененное ММП-дерево показало, что предком плазмиды вирулентности в центре дерева является последовательность, идентичная последовательности S. Enteritidis штамма SEJ (рис. 1). Это подтверждает данные о том, что данная плазмида идентична консенсусной при множественном выравнивании. Кроме того, она не имеет мутаций и псевдогенов при сравнении с остальными 12 последовательностями плазмид. Из дерева видно, что две наиболее генетически отдаленные плазмиды это плазмиды вирулентности штаммов S. Enteritidis 18569 и LK5.
Рис. 1.
Неукорененное ММП-дерево, показывающее родство исследованных плазмид вирулентности штаммов S. Enteritidis.
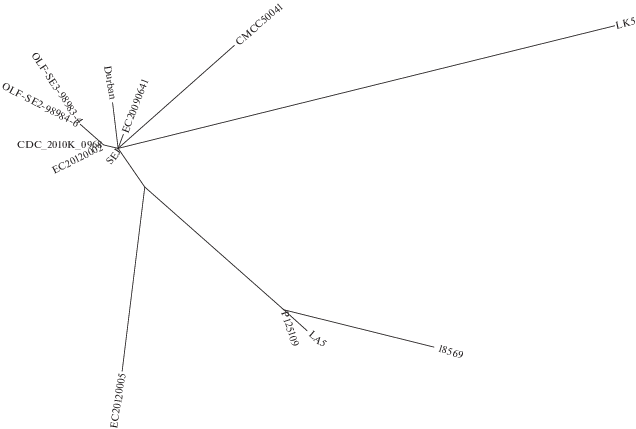
Сравнение всех доступных к настоящему времени последовательностей плазмиды вирулентности S. Enteritidis показало, что они ограничены в размерах. Эти размеры варьируют от 59 336 до 59 374 пн, при этом более половины плазмид имеют размер, равный 59 372 пн. Состав генов является довольно консервативным и состоит из 81 открытой рамки считывания с признаками псевдогенизации некоторых генов у плазмид, отличающихся по размеру от 59 72 пн.
Таким образом, можно утверждать, что предком плазмиды вирулентности S. Enteritidis (pSEV) является плазмида из SEJ-подобного штамма [15] размером 56 372 пн с таким же нуклеотидным и генным составом, как и S. Enteritidis штамм SEJ. Последовательность этой плазмиды может быть использована как референсная плазмида вирулентности pSEV для всех последующих работ с использованием плазмиды вирулентности S. Enteritidis.
Таким образом, установлено, что плазмида вирулентности S. Enteritidis (pSEV) является консервативной, что затрудняет использование ее в качестве молекулярно-генетического маркера в методах, основанных на ПЦР и секвенировании.
Список литературы
Hendriksen R.S., Vierira A.R., Karlsmose S. et al. Global monitoring of Salmonella serovar distribution from the World Health Organization Global Foodborne Infections Network Country Data Bank: results of quality assured laboratories from 2001 to 2007 // Foodborne Pathog. Dis. 2011. V. 8. № 8. P. 887–900. doi 10.1089/ fpd.2010.0787
Popoff M.Y., Miras I., Coynault C. et al. Molecular relationships between virulence plasmids of Salmonella serotypes typhimurium and dublin and large plasmids of other Salmonella serotypes // Ann. Microbiol. (Paris). 1984. V. 135A. № 3. P. 389–398.
Gulig P.A., Danbara H., Guiney D.G. et al. Molecular analysis of spv virulence genes of the Salmonella virulence plasmids // Mol. Microbiol. 1993. V. 7. № 6. P. 825–830.
Chu C., Hong S.F., Tsai C. et al. Comparative physical and genetic maps of the virulence plasmids of Salmonella enterica serovars typhimurium, enteritidis, choleraesuis, and dublin // Infect. Immun. 1999. V. 67. № 5. P. 2611–2614.
Rotger R., Casadesús J. The virulence plasmids of Salmonella // Int. Microbiol. 1999. V. 2. № 3. P. 177–184.
Sengupta M., Austin S. Prevalence and significance of plasmid maintenance functions in the virulence plasmids of pathogenic bacteria // Infect. Immunol. 2011. V. 79. № 7. P. 2502–2509. doi 10.1128/IAI.00127-11
Chu C., Feng Y., Chien A.C. et al. Evolution of genes on the Salmonella Virulence plasmid phylogeny revealed from sequencing of the virulence plasmids of S. enterica serotype Dublin and comparative analysis // Genomics. 2008. V. 92. № 5. P. 339–343. doi 10.1016/ j.ygeno.2008.07.010
Carattoli A., Zankari E., García-Fernández A. et al. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing // Antimicrob. Agents Chemother. 2014. V. 58. № 7. P. 3895–3903. doi 10.1128/AAC.02412-14
Guerra B., Soto S., Helmuth R. et al. Characterization of a self-transferable plasmid from Salmonella enterica serotype typhimurium clinical isolates carrying two integron-borne gene cassettes together with virulence and drug resistance genes // Antimicrob. Agents Chemother. 2002. V. 46. № 9. P. 2977–2981.
Rodríguez I., Guerra B., Mendoza M.C. et al. pUO-SeVR1 is an emergent virulence-resistance complex plasmid of Salmonella enterica serovar Enteritidis // J. Antimicrob. Chemother. 2011. V. 66. № 1. P. 218–220. doi 10.1093/jac/dkq386
García V., García P., Rodríguez I. et al. The role of IS26 in evolution of a derivative of the virulence plasmid of Salmonella enterica serovar Enteritidis which confers multiple drug resistance // Infect. Genet. Evol. 2016. V. 45. P. 246–249. doi 10.1016/j.meegid.2016.09.008
Rychlik I., Gregorova D., Hradecka H. Distribution and function of plasmids in Salmonella enterica // Vet. Microbiol. 2006. V. 112. № 1. P. 1–10.
Schnoes A.M., Brown S.D., Dodevski I. et al. Annotation Error in Public Databases: Misannotation of Molecular Function in Enzyme Superfamilies // PLoS Comput. Biol. 2009. V. 5. № 12. P. e1000605. doi 10.1371/journal.pcbi.1000605
Feng Y., Liu J., Li Y.G. et al. Inheritance of the Salmonella virulence plasmids: mostly vertical and rarely horizontal // Infect. Genet. Evol. 2012. V. 12. № 5. P. 1058–1063. doi 10.1016/j.meegid.2012.03.004
Bishop-Lilly K.A., Frey K.G., Daligault H.E. et al. Complete genome sequence of Salmonella enterica subsp. enterica serovar Enteritidis Strain SEJ // Genome Announc. 2014. V. 2. № 5. P. e01084–14. doi 10.1128/genomeA.01084-14
Ogunremi D., Devenish J., Amoako K. et al. High resolution assembly and characterization of genomes of Canadian isolates of Salmonella Enteritidis // BMC Genomics. 2014. V. 15. P. 713. doi 10.1186/1471-2164-15-713
Rehman M.A., Ziebell K., Nash J.H. et al. Complete genome sequences of 16 Canadian strains of Salmonella enterica subsp. enterica serovar Enteritidis // Genome Announc. 2014. V. 2. № 2. P. e00330–14. doi 10.1128/ genomeA.00330-14
Feasey N.A., Hadfield J., Keddy K.H. et al. Distinct Salmonella Enteritidis lineages associated with enterocolitis in high-income settings and invasive disease in low-income settings // Nat. Genet. 2016. V. 48. № 10. P. 1211–1217. doi 10.1038/ng.3644
Zhang S., Yin Y., Jones M.B. et al. Salmonella serotype determination utilizing high-throughput genome sequencing data // J. Clin. Microbiol. 2015. V. 53. № 5. P. 1685–1692. doi 10.1128/JCM.00323-15
Besemer J., Lomsadze A., Borodovsky M. GeneMarkS: a self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions // Nucl. Acids Res. 2001. V. 29(12). P. 2607–2618.
Hyatt D., Chen G.L., Locascio P.F. et al. Prodigal: prokaryotic gene recognition and translation initiation site identification // BMC Bioinformat. 2010. V. 11. P. 119. doi 10.1186/1471-2105-11-119
Bocs S., Cruveiller S., Vallenet D. et al. AMIGene: Annotation of MIcrobial Genes // Nucl. Acids Res. 2003. V. 31. № 13. P. 3723–3726.
Aziz R.K., Bartels D., Best A.A. et al. The RAST Server: rapid annotations using subsystems technology // BMC Genomics. 2008. V. 9. P. 75. doi 10.1186/1471-2164-9-75
Ederveen T.H., Overmars L., van Hijum S.A. Reduce manual curation by combining gene predictions from multiple annotation engines, a case study of start codon prediction // PLoS One. 2013. V. 8. № 5. P. e63523. doi 10.1371/journal.pone.0063523
Wall M.E., Raghavan S., Cohn J.D. et al. Genome majority vote improves gene predictions // PLoS Comput. Biol. 2011. V. 7. № 11. P. e1002284. doi 10.1371/journal.pcbi.1002284
Siguier P., Perochon J., Lestrade L. et al. ISfinder: the reference centre for bacterial insertion sequences // Nucl. Acids Res. 2006. V. 34. P. D32–36. doi 10.1093/ nar/gkj014
Zankari E., Hasman H., Cosentino S. et al. Identification of acquired antimicrobial resistance genes // J. Antimicrob. Chemother. 2012. V. 67. № 11. P. 2640–2644. doi 10.1093/jac/dks261
Darling A.C., Mau B., Blattner F.R. et al. Mauve: multiple alignment of conserved genomic sequence with rearrangements // Genome Res. 2004. V. 14. № 7. P. 1394–1403. doi 10.1101/gr.2289704
Lassmann T., Frings O., Sonnhammer E.L. Kalign2: high-performance multiple alignment of protein and nucleotide sequences allowing external features // Nucl. Acids Res. 2009. V. 37. № 3. P. 858–865. doi 10.1093/nar/gkn1006
Altschul S.F., Gish W., Miller W. et al. Basic local alignment search tool // J. Mol. Biol. 1990. V. 215. № 3. P. 403–410. doi 10.1016/S0022-2836(05)80360-2
Tamura K., Stecher G., Peterson D. et al. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0 // Mol. Biol. Evol. 2013. V. 30. № 12. P. 2725–2729. doi 10.1093/molbev/mst197
Russell D.A., Bowman C.A., Hatfull G.F. Genome sequence of Salmonella enterica subsp. enterica Strain Durban // Genome Announc. 2014. V. 2. № 3. P. e00399–14. doi 10.1128/genomeA.00399-14
Langridge G.C., Fookes M., Connor T.R. et al. Patterns of genome evolution that have accompanied host adaptation in Salmonella // Proc. Natl Acad. Sci. USA. 2015. V. 112. № 3. P. 863–868. doi 10.1073/ pnas.1416707112
Li Q., Wang X., Yin K. et al. Genetic analysis and CRISPR typing of Salmonella enterica serovar Enteritidis from different sources revealed potential transmission from poultry and pig to human // Int. J. Food Microbiol. 2018. V. 266. P. 119–125. doi 10.1016/j.ijfoodmicro.2017.11.025
Grépinet O., Rossignol A., Loux V. et al. Genome sequence of the invasive Salmonella enterica subsp. enterica serotype enteritidis strain LA5 // J. Bacteriol. 2012. V. 194. № 9. P. 2387–2388. doi 10.1128/JB.00256-12
Allard M.W., Luo Y., Strain E. et al. On the evolutionary history, population genetics and diversity among isolates of Salmonella Enteritidis PFGE pattern JEGX01.0004 // PLoS One. 2013. V. 8. № 1. P. e55254. doi 10.1371/journal.pone.0055254
Дополнительные материалы отсутствуют.


