Генетика, 2019, T. 55, № 6, стр. 655-664
Профиль экспрессии генов унипортеров семейства SWEET в процессе развития ловчих аппаратов плотоядного растения Nepenthes sp.
М. А. Филюшин 1, *, Е. З. Кочиева 1, 2, А. В. Щенникова 1, А. В. Белецкий 1, А. В. Марданов 1, Н. В. Равин 1, 2, К. Г. Скрябин 1, 2
1 Федеральный исследовательский центр “Фундаментальные основы биотехнологии”
Российской академии наук
119071 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова,
кафедра биотехнологии
119991 Москва, Россия
* E-mail: michel7753@mail.ru
Поступила в редакцию 26.06.2018
После доработки 23.07.2018
Принята к публикации 04.09.2018
Аннотация
В результате анализа транскриптомов ловчего аппарата (на трех стадиях развития) и листа плотоядного растения Nepenthes sp. были идентифицированы 20 кДНК генов семейства SWEET, кодирующих унипортеры сахаров классов I–IV. Структура белков NSWEET в целом соответствовала схеме 3-1-3, характерной для SWEET-белков эукариот. Вариабельность экспрессии генов NSWEET в зрелом листе и на трех стадиях развития ловчего аппарата указывала на возможное разнообразие функций данных генов. Было высказано предположение об участии транспортеров класса I (NSWEET2d, 2f и 2h) в экспорте сахаров из листа в точку закладки “ловчей” меристемы, а белков NSWEET1, 2a (класс I), 4a, 4b, 4d (II) и 12c (III) – в доставке сахаров для начального развития ловушки. На последующих стадиях их могут сменять NSWEET2b, 2c, 2e, 2i, 2j (класс I), 4c (II) и 12b (III), доставляя гексозы и сахарозу в растущий кувшинчик. В сформированном ловчем аппарате белки NSWEET12a (III), 2g (I) и 16 (IV) могут экспортировать сахара из пищеварительной жидкости в лист.
Растворимые сахара, образующиеся у растений в процессе фотосинтеза, являются основным источником энергии и материалом для биосинтеза многих клеточных соединений, участвуют в поддержании гомеостаза и в формировании ответной реакции на биотические и абиотические стрессы [1–3]. Доставка сахаров из фотосинтезирующих клеток в гетеротрофные ткани и органы, не способные самостоятельно их синтезировать (например, ткани цветка, корни, плоды, семена), осуществляется специфичными транспортными белками семейств MST (Monosaccharide Transporter), SUT (Sucrose Transporter) и SWEET (Sugars Will Eventually be Exported Transporters) [4]. MST-белки осуществляют транспорт моносахаридов, SUT-белки транспортируют молекулы сахарозы, а белки недавно идентифицированного семейства SWEET осуществляют транспорт как моно-, так и дисахаридов [4, 5].
Белки SWEET являются трансмембранными и функционируют как унипортеры, осуществляя транспорт сахаров через клеточную мембрану и опосредуя отток сахарозы из клеток паренхимы флоэмы в апопласт [3, 6]. К настоящему времени данные белки и кодирующие их гены идентифицированы и описаны как у прокариот, так и у эукариот, включая виды животных и растений [3, 5, 7–9].
У прокариот белки SWEET состоят из трех α‑спиральных трансмембранных мотивов (ТМ), образующих домен MtN3 [5]. Предполагается, что в ходе эволюции генная дупликация привела к появлению эукариотических белков SWEET, включающих два домена MtN3, которые соединены седьмым трансмембранным мотивом (такая схема названа 3-1-3) [5, 10]. У ряда растений выявлены белки SWEET с числом трансмембранных мотивов от 3 до 14 [11–13]. При этом организация белков имеет в основе схему 3-1 -3, но в зависимости от количества ТМ – разное число MtN3-доменов.
В зависимости от транспортируемых сахаров белки SWEET растений делят на четыре группы [14]. Транспортеры групп I и II переносят гексозы, белки группы III являются эффективными транспортерами сахарозы, а белки группы IV расположены в вакуолярной мембране и транспортируют фруктозу [8, 11, 15]. Считается, что различная локализация и углеводная специфичность этих белков обусловливают многообразие их физиологических функций: участие в развитии пыльцы, синтез нектара, формирование семян и обеспечение устойчивости к абиотическим и биотическим стрессам [3, 11].
Наиболее подробно функции белков SWEET изучены у Arabidopsis thaliana. Показано, что некоторые члены данного семейства белков могут быть активны только в вакуолярной или только в клеточной мембране, а некоторые присутствуют в обеих мембранах. Например, белки AtSWEET11 и AtSWEET12 обеспечивают отток сахарозы из паренхимальных клеток флоэмы в апопласт [8], а AtSWEET16 и AtSWEET17 участвуют в экспорте фруктозы из вакуоли и играют ключевую роль в стимуляции двунаправленного транспорта фруктозы в тонопласте корней для поддержания гомеостаза фруктозы в цитозоле [15–17]. Еще более специфичны транспортеры AtSWEET5, AtSWEET8 и AtSWEET13, транскрипты генов которых обнаружены только в пыльце [18]. Белок AtSWEET9 и его гомологи у видов двудольных являются нектар-специфичными транспортерами, осуществляющими отток сахаров в процессе продукции нектара [19].
Если у большинства растений первичным источником углеводов является фотосинтез, то у плотоядных растений наряду с фотосинтезом сахара образуются в процессе переваривания жертвы в ловчих аппаратах [20]. Плотоядность развилась независимо в нескольких линиях высших растений, у которых в процессе эволюции ловчие аппараты возникли как результат преобразования листа [21].
Для исследования механизмов хищничества у растений уже более 100 лет в качестве модели используют виды Nepenthes [22]. В первую очередь изучают ферментный состав пищеварительной жидкости ловчих аппаратов (кувшинчиков), в частности хитиназы, расщепляющие хитиновые оболочки насекомых до простых углеводов и азотистых соединений [23, 24]. Транспортные белки, включая семейство SWEET, у представителей рода Nepenthes ранее не исследовались. Только у одного вида плотоядных растений рода Cephalotus были идентифицированы гены SWEET [20]. Поэтому целью данной работы стала идентификация и характеристика генов семейства SWEET у плотоядного растения Nepenthes sp. на основе транскриптомных данных.
МАТЕРИАЛЫ И МЕТОДЫ
Поиск последовательностей, кодирующих белки семейства SWEET, осуществляли в транскриптомах ловчего аппарата Nepenthes sp. (на трех стадиях развития – зачаток кувшинки (P1; длина около 1 см), молодая неоткрытая кувшинка (P2; 3–4 см; промежуточная стадия развития), открытая кувшинка (P3; 7–11 см; конечная стадия)) и взрослого листа, в трех биологических повторностях. Лист был взят в связи с тем, что ловчий аппарат является эволюционной модификацией данного органа. С учетом наличия развитого воскового слоя листьев и кувшинчиков, затрудняющего экстракцию нуклеиновых кислот, для выделения РНК из тканей Nepenthes была разработана методика с использованием СТАВ-буфера (наши неопубликованные данные). Препараты РНК очищали от возможных примесей ДНК с помощью набора RNase-freeDNaseSet (Qiagen, США). Концентрацию РНК измеряли с помощью флуориметра Qubit 4, качество оценивали по результатам электрофореза в агарозном геле. Препараты РНК после пробоподготовки были секвенированы с помощью Illumina MiSeq (ФИЦ Биотехнологии РАН). Сборку и картирование чтений, а также идентификацию белок-кодирующих генов в контигах осуществляли аналогично [25]. Уровень экспрессии генов рассчитывали как число картированных на их последовательности чтений транскриптома на миллион чтений, деленное на длину гена. Тепловую карту экспрессии строили с помощью онлайн-программы Heatmapper (http:// www2.heatmapper.ca/).
Для идентификации последовательностей транспортеров SWEET, определения их доменной структуры и поиска гомологов использовали базу данных NCBI (http://blast.ncbi.nlm.nih.gov/). Консервативные мотивы определяли с помощью программы МЕМЕ 4.12.0 (Multiple Expectation Maximization for Motif Elicitation) (http://meme-suite.org/tools/meme) [26].
Сравнительный анализ аминокислотных последовательностей и построение дендрограммы осуществляли методом максимального правдоподобия (Maximum likelihood) в программе MEGA7.0 [27]. Для кластерного анализа из базы NCBI были взяты последовательности мРНК 17 известных генов SWEET модельного вида A. thaliana.
РЕЗУЛЬТАТЫ
Были получены 12 транскриптомов Nepenthes sp. – ловчего аппарата (кувшинчика) на трех стадиях развития (P1, P2 и P3) и листа, в трех биологических повторах каждый объект. Всего по результатам аннотации анализируемых транскриптомов было идентифицировано 20 последовательностей, кодирующих белки SWEET (табл. 1). Последовательности были депонированы в NCBI (коды доступа MH544206–MH544225). Сходство с известными последовательностями растительных белков SWEET из базы NCBI составило 57–80%.
Таблица 1.
Идентифицированные транскрипты генов семейства SWEET в анализируемых транскриптомах Nepenthes sp. и их гомологи в базе данных NCBI
| № п/п | Транскрипт | Белок, а.о. | Гомолог в NCBI | Идентичность, % | Трансмембранные мотивы |
|---|---|---|---|---|---|
| 1 | NSWEET1 | 327 | XP_021760946 bidirectional sugar transporter SWEET1-like [Chenopodium quinoa] | 68 | 1–7 |
| 2 | NSWEET2a* | 237 | XP_017234423 bidirectional sugar transporter SWEET2a-like [Daucuscarota subsp. sativus] | 73 | 1–7 |
| 3 | NSWEET2b* | 112 | XP_018830079 bidirectional sugar transporter SWEET2a-like [Juglans regia] | 77 | 1–3 |
| 4 | NSWEET2c | 172 | XP_002269484 bidirectional sugar transporter SWEET2a [Vitis vinifera] | 74 | 3–7 |
| 5 | NSWEET2d* | 205 | XP_002269484 bidirectional sugar transporter SWEET2a [Vitis vinifera] | 70 | 2–7 |
| 6 | NSWEET2e* | 172 | XP_002269484 bidirectional sugar transporter SWEET2a [Vitis vinifera] | 69 | 1–5 |
| 7 | NSWEET2f* | 196 | XP_009376722 bidirectional sugar transporter SWEET2-like [Pyrus × bretschneideri] | 63 | 2–7 |
| 8 | NSWEET2g* | 236 | XP_009376722 bidirectional sugar transporter SWEET2-like [Pyrus × bretschneideri] | 71 | 1–7 |
| 9 | NSWEET2h | 133 | XP_021842582 bidirectional sugar transporter SWEET2 [Spinacia oleracea] | 69 | 1–3 |
| 10 | NSWEET2i | 131 | XP_021773915 bidirectional sugar transporter SWEET2-like [Chenopodium quinoa] | 74 | 2–5 |
| 11 | NSWEET2j | 240 | XP_008363645 bidirectional sugar transporter SWEET2-like [Malus domestica] | 68 | 1–7 |
| 12 | NSWEET2k | 223 | XP_008363645 bidirectional sugar transporter SWEET2-like [Malus domestica] | 66 | 1–6 |
| 13 | NSWEET4a* | 258 | XP_010695602 bidirectional sugar transporter SWEET4-like [Beta vulgaris subsp. vulgaris] | 66 | 1–7 |
| 14 | NSWEET4b | 157 | XP_021757527 bidirectional sugar transporter SWEET4-like [Chenopodium quinoa] | 73 | 1–5 |
| 15 | NSWEET4c | 113 | XP_010695602 bidirectional sugar transporter SWEET4-like [Beta vulgaris subsp. vulgaris] | 57 | 5–7 |
| 16 | NSWEET4d | 100 | XP_010695602 bidirectional sugar transporter SWEET4-like [Beta vulgaris subsp. vulgaris] | 73 | 6–7 |
| 17 | NSWEET12a* | 263 | XP_021775815 bidirectional sugar transporter SWEET12-like [Chenopodium quinoa] | 64 | 1–7 |
| 18 | NSWEET12b | 126 | XP_021775815 bidirectional sugar transporter SWEET12-like [Chenopodium quinoa] | 58 | 6–7 |
| 19 | NSWEET12c | 108 | XP_021775815 bidirectional sugar transporter SWEET12-like [Chenopodium quinoa] | 73 | 1–3 |
| 20 | NSWEET16* | 299 | XP_012088980 bidirectional sugar transporter SWEET16 [Jatropha curcas] | 80 | 1–7 |
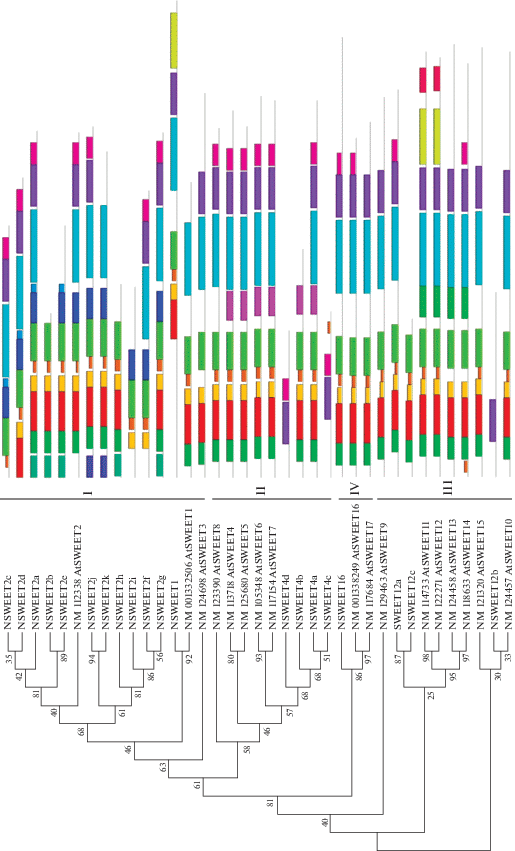
Филогенетический анализ последовательностей кодируемых белков классифицировал транскрипты и определил, что идентифицированные последовательности гомологичны белкам A. thaliana SWEET1, SWEET2, SWEET4, SWEET12 и SWEET16 и представляют все четыре известных класса белков SWEET (рис. 1).
Рис. 1.
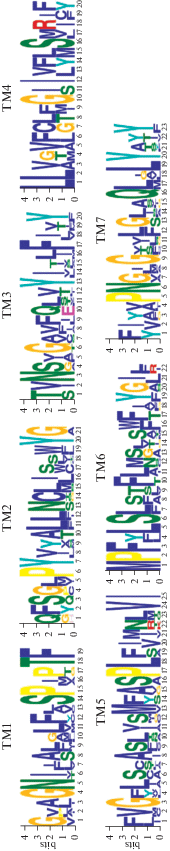
Филогенетическое дерево, построенное на основе эволюционного анализа 37 аминокислотных последовательностей белков семейства SWEET. Длина ветвей оценивается в генетической дистанции (число замещений на сайт). Существенные значения bootstrap (процент деревьев, в которых ассоциированные таксоны кластеризуются вместе) для 1000 выборок показаны в основании ветви. Напротив названий белков, взятых в анализ из базы данных NCBI, указан код доступа в NCBI. Справа от дендрограммы дана схема консервативных мотивов, определенных с помощью программы MEME 4.12.0. Линии соответствуют вариабельным участкам, окрашенные боксы – консервативным мотивам. См. текст.
Структурный анализ кДНК NSWEET и кодируемых ими белков показал, что девять транскриптов являются полноразмерными (кодируемые ими белки имеют старт- и стоп-кодоны), а остальные 11 – неполными (табл. 1). Выравнивание аминокислотных последовательностей, кодируемых полноразмерными кДНК, выявило наличие изоформ, различающихся числом трансмембранных мотивов (табл. 1). Поиск консервативных последовательностей обнаружил участки, гомологичные известным трансмембранным мотивам белков SWEET, определенным ранее у других видов растений [5, 12, 28]. На рис. 2 показаны консенсусные последовательности мотивов 1–7 в белках NSWEET.
Биоинформационный анализ позволил определить возможные профили экспрессии генов NSWEET в ловчем аппарате на трех стадиях развития и в листе (рис. 3, 4). Было обнаружено, что гены класса I (NSWEET2a, NSWEET2c, NSWEET2d, NSWEET2e, NSWEET2g и NSWEET2j) и класса IV (NSWEET16) транскрибируются как в листе, так и в ловчих аппаратах на всех трех стадиях развития. В листе самый высокий уровень транскрипции детектирован для NSWEET2с (класс I), в зачатке кувшинчика и неоткрытом кувшинчике – для NSWEET4a (класс II), а в полностью сформированном, открытом кувшинчике – для NSWEET12a (класс III). Транскрипты двух генов класса I (NSWEET2b и NSWEET2i), а также всех выявленных генов классов II (NSWEET4a, NSWEET4b, NSWEET4c и NSWEET4d) и III (NSWEET12a, NSWEET12b и NSWEET12c) в листе полностью отсутствуют. При этом все идентифицированные гены экспрессируются в кувшинчиках на одной, двух или всех трех стадиях развития. В открытом кувшинчике (P3) не обнаружено транскриптов NSWEET1 (I), NSWEET2h (I), NSWEET2i (I), NSWEET2k (I), NSWEET4b (II), NSWEET4d (II), NSWEET12b (III) и NSWEET12c (III). В зачатке кувшинчика (P1) не выявлено транскриптов NSWEET2i (I), экспрессия которого в целом низка и ограничена кувшинчиком второй стадии развития (P2). Транскрипты NSWEET4b (II) присутствуют только на начальной стадии развития кувшинчика (P1).
Рис. 3.
Профили экспрессии генов SWEET Nepenthes sp. на основе нормализованных транскриптомных данных. По оси абсцисс указаны идентифицированные гены SWEET; по оси ординат отложено нормализованное число чтений в транскриптомах.
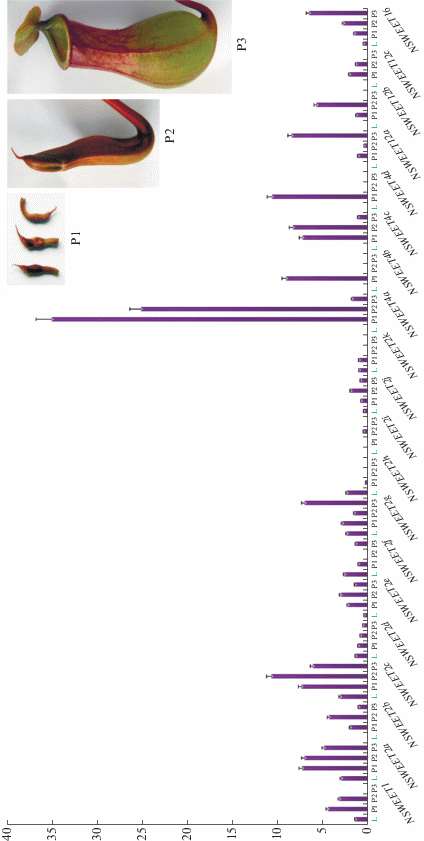
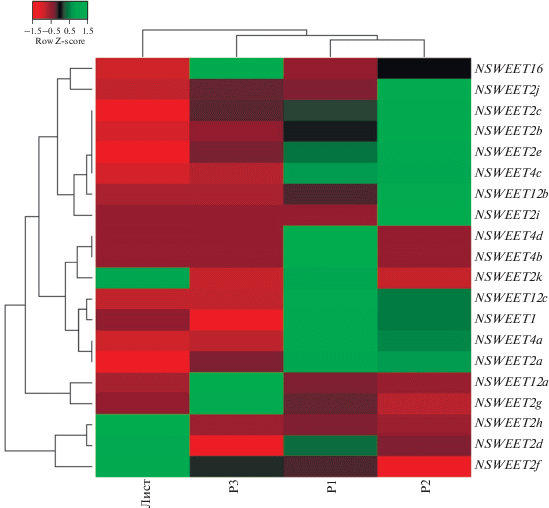
Рис. 4.
Тепловая карта экспрессии генов SWEET Nepenthes sp., построенная с использованием программы Heatmapper (http://www2.heatmapper.ca/). Различия в уровнях экспрессии отражены при помощи цвета. Слева дана дендрограмма, показывающая степень сходства профилей экспрессии генов SWEET Nepenthes. Дендрограмма сверху отражает уровень сходства транскрипционных профилей генов SWEET в листе и ловчем аппарате на трех стадиях развития (P1, P2, P3).
По динамике экспрессии идентифицированные гены NSWEET можно поделить на две группы: у первой – уровень экспрессии падает по мере развития кувшинчика, у второй – растет (рис. 3). В процессе формирования на кончике листа ловчего аппарата наиболее резкое снижение уровня транскрипции наблюдалось у генов первой группы для NSWEET1 (I), NSWEET4b (II), NSWEET4a (II), NSWEET4d (II), NSWEET4c (II), NSWEET12b (III) и NSWEET12c (III). В то время как у генов второй группы NSWEET2g (I), NSWEET12a (III) и NSWEET16 (IV) был выявлен резкий рост экспрессии.
Анализ тепловой карты (рис. 4) выявил наибольшее сходство профилей экспрессии генов SWEET в зачатке (P1) и неоткрытом кувшинчике (P2). Для каждого из идентифицированных генов, кроме NSWEET2k, максимальный уровень экспрессии выявлен только на одной из стадий развития ловчего аппарата (P1 – гены NSWEET1, 2a (I), 4a, 4b, 4d (II), 12c (III); P2 – NSWEET2b, 2c, 2e, 2i, 2j (I), 4c (II), 12b (III); P3 – NSWEET16 (IV), 12a (III), 2g (I)) либо в листе (NSWEET2d, 2f и 2h (I)). Уровень экспрессии NSWEET2k был одинаков в листе и зачатке кувшинчика (рис. 3 и 4).
ОБСУЖДЕНИЕ
Возникновение хищничества у растений является результатом их эволюционной адаптации к окружающей среде, обедненной питательными веществами. Плотоядные растения рода Nepenthes представлены почти 120 видами. На кончике листа они формируют усик с пассивной ловушкой в форме кувшинчика для ловли и переваривания насекомых и мелких позвоночных. Данный уникальный ловчий аппарат имеет секреторные железки, которые продуцируют белки для пищеварительной жидкости [29].
Исследования плотоядных растений сфокусированы в основном на анализе белкового состава пищеварительной жидкости, накапливающейся в нижней трети кувшинчика и в которой содержатся ферменты для переваривания (хитиназы, пероксидазы, непентезины, непрозины и другие ферменты метаболизма жиров, нуклеиновых кислот, полисахаридов и белков) насекомых [20, 29]. Кувшинчик состоит из трех функционально различных частей: нижней (glandular zone) с секретирующими железками, верхней, скользкой (slippery zone) за счет покрытия воском, и крышечки (lid), закрывающей кувшинчик после попадания жертвы внутрь [29].
Интересно, что адаптация растений к условиям обитания, обедненным питательными веществами или освещением, может приводить к де-эволюции (микогетеротрофы), когда цветковое растение за ненужностью перестает формировать вегетативные надземные органы [25, 30–32]. В случае плотоядных растений эволюция пошла дальше, в результате чего путем модификации листа сформировался ловчий аппарат (как ранее возникли из листа цветковые органы), снабженный, как и цветок, железками, отличающимися от нектарников составом секрета. Помимо этого, железки бифункциональны – они не только продуцируют пищеварительную жидкость, но и экспортируют оттуда полученные после переваривания питательные вещества, в том числе углеводы [29].
Транспорт сахаров осуществляется, как было сказано выше, в том числе двунаправленными транспортерами семейства SWEET [6]. Изучение генов SWEET у Camellia sinensis обнаружило, что наиболее активно они экспрессируются в цветках и семенах и, таким образом, тесно связаны с экспортом сахаров из фотосинтезирующих тканей к репродуктивным органам [28]. Многие из генов вовлечены в ответ растения на абиотический стресс [28]; кроме того, их экспрессию могут индуцировать симбиотические бактерии и грибы для усиления собственного питания [7].
При закладке и последующем формировании нового органа растения резко активируется экспорт сахаров в точку роста. Зрелый лист является фотосинтезирующим органом и, следовательно, экспортером быстрых углеводов. Можно предположить, что при инициации “ловчей” меристемы на кончике листа и последующем развитии кувшинчика происходит отток сахаров из листа в растущий на его кончике ловчий аппарат. При этом сформированная ловушка с функционирующими железками по мере переваривания жертвы становится экспортером углеводов, как и лист.
В настоящей работе в транскриптомах плотоядного растения Nepenthes sp. впервые были идентифицированы 20 кДНК генов семейства SWEET, кодирующих трансмембранные транспортеры сахаров. Структура кодируемых белков в целом соответствовала ранее описанной схеме 3-1 -3, характерной для SWEET-белков эукариот (табл. 1) [7]. Было показано, что экспрессия идентифицированных генов в зрелом листе и на трех стадиях развития кувшинчика вариабельна, что свидетельствует о разнообразии функциональной роли данных генов (рис. 3, 4). Полученные экспрессионные данные позволяют предположить, что белки класса I, NSWEET2d, 2f и 2h, принимают участие в экспорте гексоз из листа, в том числе в точку возможной закладки “ловчей” апикальной меристемы.
Максимальная экспрессия в зачатке кувшинчика генов NSWEET1, 2a (класс I), 4a, 4b, 4d (II) и 12c (III) предполагает участие кодируемых ими транспортеров в доставке гексоз и сахарозы для роста ловушки. На последующих стадиях их может сменять другая группа генов, NSWEET2b, 2c, 2e, 2i, 2j (класс I), 4c (II) и 12b (III), продукты которых доставляют гексозы и сахарозу в растущий кувшинчик. Высокий уровень экспрессии генов NSWEET12a (III), 2g (I) и 16 (IV) только в полностью сформированном ловчем аппарате может указывать на участие кодируемых ими транспортеров в экспорте сахаров из пищеварительной жидкости функционирующего кувшинчика обратно в лист.
Работа выполнена при финансовой поддержке гранта Российского научного фонда № 14-24-00175 и с использованием экспериментальной установки искусственного климата ЭУИК.
Список литературы
Lemoine R., La Camera S., Atanassova R. et al. Source-to-sink transport of sugar and regulation by environmental factors // Front Plant Sci. 2013. V. 4. Article 272. https://doi.org/10.3389/fpls.2013.00272
Chen L.-Q., Cheung L.S., Feng L. et al. Transport of sugars // Annu. Rev. Biochem. 2015. V. 84. P. 865–894. https://doi.org/10.1146/annurev-biochem-060614-033904
Feng C.Y., Han J.X., Han X.X., Jiang J. Genome-wide identification, phylogeny, and expression analysis of the SWEET gene family in tomato // Gene. 2015. V. 573. № 2. P. 261–272. https://doi.org/10.1016/j.gene.2015.07.055
Eom J.S., Chen L.Q., Sosso D. et al. SWEETs, transporters for intracellular and intercellular sugar translocation // Curr. Opin. Plant Biol. 2015. V. 25. P. 53–62. https://doi.org/10.1016/j.pbi.2015.04.005
Hu L.-P., Zhang F., Song S.-H. et al. Genome-wide identification, characterization, and expression analysis of the SWEET gene family in cucumber // J. Integrative Agriculture. 2017. V. 16. № 7. P. 1486–1501. https://doi.org/10.1016/S2095-3119(16)61501-0
Chen L.-Q. SWEET sugar transporters for phloem transport and pathogen nutrition // New Phytol. 2014. V. 201. № 4. P. 1150–1155. https://doi.org/10.1111/nph.12445
Chen L.-Q., Hou B.-H., Lalonde S. et al. Sugar transporters for intercellular exchange and nutrition of pathogens // Nature. 2010. № 468. P. 527–532.
Chen L.-Q., Qu X.-Q., Hou B.-H. et al. Sucrose efflux mediated by SWEET proteins as a key step for phloem transport // Science. 2012. V. 335. P. 207–211. https://doi.org/10.1126/science.1213351
Yuan M., Wang S. Rice MtN3/saliva/SWEET family genes and their homologs in cellular organisms // Mol. Plant. 2013. V. 6. P. 665–674. https://doi.org/10.1093/mp/sst035
Xuan Y.H., Hu Y.B., Chen L.Q. et al. Functional role of oligomerization for bacterial and plant SWEET sugar transporter family // Proc. Natl Acad. Sci. USA. 2013. V. 110. P. E3685–E3694. https://doi.org/10.1073/pnas.1311244110
Patil G., Valliyodan B., Deshmukh R. et al. Soybean (Glycine max) SWEET gene family: insights through comparative genomics, transcriptome profiling and whole genome re-sequence analysis // BMC Genomics. 2015. V. 16. Article 520. https://doi.org/10.1186/s12864-015-1730-y
Gao Y., Wang Z.Y., Kumar V. et al. Genome-wide identification of the SWEET gene family in wheat // Gene. 2018. V. 642. P. 284–292. https://doi.org/10.1016/j.gene.2017.11.044
Guo C., Li H., Xia X. et al. Functional and evolution characterization of SWEET sugar transporters in Ananas comosus // Biochem. Biophys. Res. Commun. 2018. V. 496. № 2. P. 407–414. https://doi.org/10.1016/j.bbrc.2018.01.024
Chandran D. Co-option of developmentally regulated plant SWEET transporters for pathogen nutrition and abiotic stress tolerance // IUBMB Life. 2015. V. 67. № 7. P. 461–471. https://doi.org/10.1002/iub.1394
Klemens P.A., Patzke K., Deitmer J. et al. Over expression of the vacuolar sugar carrier AtSWEET16 modifies germination, growth, and stress tolerance in Arabidopsis // Plant Physiol. 2013. V. 163. P. 1338–1352. https://doi.org/10.1104/ pp.113.224972
Chardon F., Bedu M., Calenge F. et al. Leaf fructose content is controlled by the vacuolar transporter SWEET17 in Arabidopsis // Curr. Biol. 2013. V. 23. P. 697–702. https://doi.org/10.1016/j.cub.2013.03.021
Guo W.J., Nagy R., Chen H.Y. et al. SWEET17, a facilitative transporter, mediates fructose transport across the tonoplast of Arabidopsis roots and leaves // Plant Physiol. 2014. V. 164. P. 777–789. https://doi.org/10.1104/ pp.113.232751
Sun M.X., Huang X.Y., Yang J. et al. Arabidopsis RPG1 is important for primexine deposition and functions redundantly with RPG2 for plant fertility at the late reproductive stage // Plant Reproduction. 2013. V. 26. P. 83–91. https://doi.org/10.1007/s00497-012-0208-1
Lin I.W., Sosso D., Chen L.-Q. et al. Nectar secretion requires sucrose phosphate synthases and the sugar transporter SWEET9 // Nature. 2014. V. 508. P. 546–549.
Fukushima K., Fang X., Alvarez-Ponce D. et al. Genome of the pitcher plant Cephalotus reveals genetic changes associated with carnivory // Nat. Ecol. Evol. 2017. V. 1. № 3. Article 59. https://doi.org/10.1038/s41559-016-0059
Fukushima K., Fujita H., Yamaguchi T. et al. Oriented cell division shapes carnivorous pitcher leaves of Sarracenia purpurea // Nat. Communicat. 2015. № 6. Article 6450. https://doi.org/10.1038/ncomms7450
Frazier C.K. The enduring controversies concerning the process of protein digestion in Nepenthes (Nepenthaceae) // Carnivorous Plant Newsletter. 2000. V. 29. P. 56–61.
Rottloff S., Miguel S., Biteau F. et al. Proteome analysis of digestive fluids in Nepenthes pitchers // Ann. Bot. 2016. V. 117. P. 479–495. https://doi.org/10.1093/aob/mcw001
Saganová M., Bokor B., Stolárik T., Pavlovič A. Regulation of enzyme activities in carnivorous pitcher plants of the genus Nepenthes // Planta. 2018. V. 248(2). P. 451–464. https://doi.org/10.1007/s00425-018-2917-7
Ravin N.V., Gruzdev E.V., Beletsky A.V. et al. The loss of photosynthetic pathways in the plastid and nuclear genomes of the non-photosynthetic mycoheterotrophic eudicot Monotropa hypopitys // BMC Plant Biology. 2016. V. 16. Suppl. 3. P. 153–161. https://doi.org/10.1186/s12870-016-0929-7
Bailey T.L., Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers // Proc. Sec. Intern. Conf. Intelligent Systems Mol. Biology. 1994. P. 28–36.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Wang L., Yao L., Hao X. et al. Tea plant SWEET transporters: expression profiling, sugar transport, and the involvement of CsSWEET16 in modifying cold tolerance in Arabidopsis // Plant Mol. Biol. 2018. V. 96. № 6. P. 577–592. https://doi.org/10.1007/s11103-018-0716-y
Miguel S., Hehn A., Bourgaud F. Nepenthes: State of the art of an inspiring plant for biotechnologists // J. Biotechnol. 2018. V. 265. P. 109–115. https://doi.org/10.1016/j.jbiotec.2017.11.014
Филюшин М.А., Решетникова Н.М., Кочиева Е.З., Скрябин К.Г. Внутривидовая вариабельность ITS-последовательностей у паразитического растения Monotropa hypopitys L. из популяций европейской части России // Генетика. 2015. Т. 51. № 11. С. 1330–1334.
Shulga O.A., Shchennikova A.V., Beletsky A.V. et al. Transcriptome-wide characterization of the MADS-box family in pinesap Monotropa hypopitys reveals flowering conservation in non-photosynthetic myco-heterotrophs // J. Plant Growth Regulat. 2017. https://doi.org/10.1007/s00344-017-9772-9
Shchennikova A.V., Slugina M.A., Beletsky A.V. et al. The YABBY genes of leaf and leaf-like organ polarity in leafless plant Monotropa hypopitys // Intern. J. Genomics. 2018. V. 15. Article ID 7203469. https://doi.org/10.1155/ 2018/7203469
Дополнительные материалы отсутствуют.