Генетика, 2019, T. 55, № 6, стр. 665-674
Дивергенция видов Oxytropis секции Verticillares (Fabaceae) степной флоры Байкальской Сибири на основе анализа хлоропластной ДНК
А. Б. Холина 1, *, М. М. Козыренко 1, Е. В. Артюкова 1, Д. В. Санданов 2
1 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
2 Институт общей и экспериментальной биологии Сибирского отделения
Российской академии наук
670047 Улан-Удэ, Россия
* E-mail: kholina@biosoil.ru
Поступила в редакцию 31.07.2018
После доработки 18.10.2018
Принята к публикации 19.11.2018
Аннотация
Анализ полиморфизма нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хлоропластной ДНК у видов Oxytropis lanata, O. myriophylla, O. oxyphylla, O. selengensis, O. stukovii секции Verticillares степной флоры Байкальской Сибири показал, что популяции характеризуются высоким гаплотипическим (0.634–1.000) и относительно низким (0.0003–0.0045) нуклеотидным разнообразием. Статистически значимые значения генетических дистанций между популяциями одного вида изменяются от 0.19382 до 0.43449. Выявлены маркерные нуклеотидные замены и индели у видов O. lanata, O. gracillima, O. interposita, O. pumila и O. mongolica, которые указывают на значительную дивергенцию их хлоропластных геномов. Слабая дифференциация (ΦST = 0.137, P < 0.0001) между видами O. oxyphylla, O. selengensis, O. stukovii и O. bargusinensis, а также низкие значения генетических дистанций (0.08939–0.32339), соответствующие межпопуляционным, отсутствие дифференцирующих молекулярных маркеров, формирование единой гаплогруппы в медианной сети и наличие общих гаплотипов свидетельствуют о том, что эти виды представляют собой генетически однородную группу, образовавшуюся, вероятно, в результате относительно недавней и быстрой дивергенции от общего предка, а также о высокой степени межвидовой гибридизации.
В растительности Байкальской Сибири флора степей занимает особое положение [1–4]. Степи составляют только 7–9% площади региона [4], при этом степной комплекс является наиболее флористически богатым – включает 666 видов и подвидов сосудистых растений, в то время как горный и лесной комплексы – 550 и 638 соответственно [3]. Степные участки встречаются в основном в лесостепной полосе и лишь на юге Байкальской Сибири становятся зональными [1]. Экосистемы степей являются центром новейшего эндемизма и реликтовости Внутренней Азии [4, 5]. В составе степной флоры Байкальской Сибири наибольшее количество таксонов отмечено для рода Oxytropis DC. (30 видов и один подвид), из них девять видов: O. bargusinensis Peschk., O. lanata (Pall.) DC., O. lasiopoda Bunge, O. myriophylla (Pall.) DC., O. oxyphylla (Pall.) DC., O. prostrata (Pall.) DC., O. selengensis Bunge, O. stukovii Palib., O. turczaninovii Jurtzev – относятся к cекции Verticillares DC. (=Baicalia Bunge) подрода Oxytropis рода Oxytropis DC. [3]. Виды cекции Verticillares (типовой вид О. oxyphylla) характеризуются мутовчатым расположением листочков и представляют собой довольно резко обособленную группу, образованную, по мнению Положий [6], на основе перспективного макромутанта. Принято считать, что секция Verticillares является дериватной от других видов Oxytropis [7, 8], в частности, представляет одну из ветвей в эволюции секции Orobia Bunge [7]. Горно-степные виды секции, встречающиеся большей частью в Забайкалье, характеризуются рядом признаков (например, одногнездные бобы и значительная экологическая амплитуда), указывающих на отсутствие высокой специализации [9]. На основе этого автор предположил, что степи Забайкалья и примыкающих районов Северо-Западной Монголии являются центром происхождения видов секции Verticillares, обособление которой произошло в начале плейстоцена в более южных районах, а в последующие криоксеротические эпохи благоприятные условия способствовали дальнейшему развитию видов этой секции и продвижению их на север.
Некоторые виды секции Verticillares близки к представителям секции Orobia, реже к видам секций Хеrobia Bunge и Polyadena Bunge. Так, по мнению Г.А. Пешковой [10], О. bargusinensis возникла от смешения О. turczaninovii (секция Verticillares) и О. sylvatica (Pall.) DC. (секция Orobia), к последнему виду также близка О. tompudae М. Pop. (секция Verticillares), а О. oxyphylla гибридизирует с О. leptophylla (Pall.) DC. из секции Xerobia и с О. ircutensis М. Pop. (=О. baicalia (Pall.) Pers.) из секции Orobia. В.Н. Сипливинский [11] считал, что вид О. interposita Sipl. является промежуточным между секциями Verticillares и Polyadena. Л.И. Малышев [12] отмечал, что еще чаще наблюдается скрещивание видов, относящихся к одной секции, в результате чего могли возникнуть расы (подвиды), разновидности и экологические формы. По мнению авторов, изучающих флору Сибири [10, 13, 14], внутри секции Verticillares О. oxyphylla легко гибридизирует с О. lanata; О. prostrata – вероятно, гибрид между ними, потерявший опушение на бобах; О. stukovii является промежуточным видом между О. prostrata и О. mongolica Kom.; О. selengensis может быть мелкоцветковой формой О. oxyphylla.
Для изучения генетической изменчивости, популяционной структуры, видовой идентификации, филогенетических связей и дивергенции видов Oxytropis и других представителей сем. Fabaceae широко используются молекулярные маркеры ядерного и хлоропластного геномов [15–26]. Так, на основе анализа межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хлоропластной ДНК (хпДНК) нами было исследовано генетическое разнообразие и дивергенция популяций эндемичных видов O. chankaensis Jurtz. [15], O. glandulosa Turcz. [20] и O. bargusinensis, O. interposita и O. triphylla (Pall.) Pers. [21]. Подобные исследования для видов O. lanata, O. myriophylla, O. oxyphylla, O. selengensis, O. stukovii, O. pumila Fisch. ex DC. и O. gracillima Bunge секции Verticillares ранее не проводились.
Цель настоящей работы – изучение генетического разнообразия природных популяций и степени дивергенции хлоропластного генома у видов Oxytropis секции Verticillares степной флоры Байкальской Сибири по данным полиморфизма межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG (хпДНК).
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследования служили 135 растений из природных популяций O. lanata, O. myriophylla, O. oxyphylla, O. selengensis, O. stukovii, O. pumila и O. gracillima cекции Verticillares подрода Oxytropis степной флоры Байкальской Сибири, Монголии и Алтая (табл. 1). Для оценки дивергенции видов этой секции в филогенетические анализы были также включены полученные нами ранее нуклеотидные последовательности представителей O. mongolica [19], O. bargusinensis, O. interposita и O. tompudae, а также O. glabra (Lam.) DC. секции Mesogaea Bunge подрода Phacoxytropis Bunge в качестве внешней группы [21] (табл. 1). Названия видов, секций и подродов приняты по Л.И. Малышеву [27].
Таблица 1.
Список видов Oxytropis, коды популяций, информация о местонахождении, гаплотипы и номера доступа в GenBank нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК
| Вид | Код популяции |
Происхождение образца (число образцов) |
Гаплотип | Номер доступа в GenBank | ||
|---|---|---|---|---|---|---|
| psbA–trnH | trnL–trnF | trnS–trnG | ||||
| O. lanata (Pall.) DC. | LAN1 | Республика Бурятия, окр. с. Унэгэтэй (20) | V1–V18 | LT994841–LT994858 | LT994895–LT994912 | LT994949–LT994966 |
| LAN2 | Республика Бурятия, Баргузинский р-н, окр. с. Уржил (15) | |||||
| LAN3 | Монголия, Центральный аймак, окр. сомона Аргалант (5) | |||||
| O. myriophylla (Pall.) DC. | MYR1 | Забайкальский край, Могойтуйский р-н, окр. с. Цугол (5) | V19–V26 | LT994859–LT994866 | LT994913–LT994920 | LT996002–LT996009 |
| MYR2 | Республика Бурятия, Хоринский р-н, окр. с. Удинск (21) | |||||
| MYR3 | Республика Бурятия, Еравнинский р-н, окр. с. Комсомольское (8) | |||||
| O. oxyphylla (Pall.) DC. | OXY1 | Забайкальский край, Агинский р-н, окр. оз. Ножий (5) | V27–V51 | LT994867–LT994891 | LT994921–LT994945 | LT996010–LT996034 |
| OXY2 | Республика Бурятия, Селенгинский р-н, окр. с. Новоселенгинск (16) | |||||
| OXY3 | Республика Бурятия, Еравнинский р-н, окр. с. Комсомольское (8) | |||||
| OXY4 | Монголия, Булганский аймак, окр. сомона Рашаант (4) | |||||
| O. selengensis Bunge | SEL | Республика Бурятия, Заиграевский р-н, окр. с. Унэгэтэй (18) | V27, V38, V52–V56 | MH174937, LT996052–LT996057 | LT996045–LT996051 | LT996038–LT996044 |
| O. stukovii Palib. | STUK | Забайкальский край, Агинский р-н, окр. оз. Ножий (4) | V27, V31, V57 | LT994892–LT994894 | LT994946–LT994948 | LT996035–LT996037 |
| O. gracillima Bunge | GRAC | Монголия, Центральный аймак, окр. сомона Аргалант (5) | V58–V62 | MH174938, LT996058–LT996061 | LT996062–LT996066 | LT996067–LT996071 |
| O. pumila Fisch. ex DC. | PUM | Республика Алтай, Кош-Агачский р-н, Чуйская степь (1) | V63 | MH174939 | LT996251 | LT996252 |
| O. mongolica Kom. | MONG | Республика Тува, Тандинский р-н, западный берег оз. Хадан (1) | H63* | LN898501 | LN898613 | LN898625 |
| O. bargusinensis Peschk. | BARG | Республика Бурятия, Курумканский р-н, долина р. Алла (15) и Баргузинский р-н, окр. с. Уржил (15) | H11–H22** | LT856473–LT856478; LT856483–LT856488 |
LT856506–LT856511; LT856516–LT856521 |
LT856539–LT856544; LT856549–LT856554 |
| O. interposita Sipl. | INTER | Республика Бурятия, Курумканский р-н, долина р. Алла (14) | H23–H27** | LT856489–LT856493 | LT856522–LT856526 | LT856555–LT856559 |
| O. tompudae M. Pop. | TOMP | Республика Бурятия, Курумканский р-н, окр. с. Майск (8) | T1–T6** | LT856566–LT856571 | LT856579–LT856584 | LT856592–LT856597 |
| O. glabra (Lam.) DC. | GLAB | Республика Бурятия, Иволгинский р-н, окр. с. Оронгой (1) | G1** | LT856572 | LT856585 | LT856598 |
Методы выделения ДНК, амплификации и прямого секвенирования межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG приведены в наших предыдущих работах [19, 21, 28]. Нуклеотидные последовательности прямых и обратных цепей определяли на генетическом анализаторе ABI 3130 (Applied Biosystems, США) и собирали с помощью пакета программ Staden Package [29]. Для каждого образца последовательности трех регионов выравнивали вручную с использованием программы SeaView v. 4 [30]. Гаплотипическое (h) и нуклеотидное (π) разнообразие, парные генетические дистанции (FST), уровень дифференциации и распределение генетической изменчивости внутри и между популяциями (индекс фиксации ΦST, анализ молекулярной дисперсии, AMOVA) и тесты на популяционную стабильность (Tajima’s D и Fu’s Fs) рассчитывали с помощью программы Arlequin v. 3.5 [31]. Анализ распределения парных нуклеотидных различий (mismatch distribution) проводили в программе DnaSP v. 5.0 [32]. Генеалогические связи гаплотипов анализировали методом медианного связывания (Median Joining, MJ) в программе NetWork v. 5.0 [33], кодируя каждую делецию или вставку, независимо от их размера, как единичное мутационное событие.
Работа проводилась с использованием оборудования ЦКП “Биотехнология и генетическая инженерия” ФНЦ Биоразнообразия ДВО РАН.
РЕЗУЛЬТАТЫ
Анализ полиморфизма нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК показал, что длина каждого из регионов у исследуемых образцов разная вследствие присутствия инсерций/делеций (инделей), моно- и динуклеотидных повторов. Длина объединенных последовательностей трех регионов 135 представителей видов O. lanata, O. myriophylla, O. oxyphylla, O. selengensis, O. stukovii, O. pumila и O. gracillima после выравнивания составила 2475 сайта, из них 2349 мономорфных и 22 вариабельных. Десять нуклеотидных замен были информативны согласно методу максимальной экономии. Анализ полученной матрицы выявил 63 гаплотипа (V1–V63), из них три гаплотипа были общими: V27 у видов O. oxyphylla, O. selengensis и O. stukovii; V31 у O. oxyphylla и O. stukovii; V38 у O. oxyphylla и O. selengensis (табл. 1).
Все популяции характеризовались относительно низким нуклеотидным и высоким гаплотипическим разнообразием, кроме популяции SEL O. selengensis (табл. 2). Согласно результатам AMOVA, более 75% всей генетической изменчивости приходится на внутрипопуляционную составляющую у O. lanata (ΦST = 0.245, P < 0.0001) и O. oxyphylla (ΦST = 0.229, P < 0.003). Крайне низкое и статистически незначимое значение индекса фиксации (ΦST = 0.008, P > 0.37) у O. myriophylla указывает на отсутствие дифференциации между популяциями. Анализ генетических дистанций (FST) между популяциями одного вида показал (табл. 3), что величины FST между всеми популяциями O. myriophylla и между парами популяций OXY1–OXY2 и OXY3–OXY4 O. oxyphylla очень малы и статистически незначимы, а наименьшее (0.19382) и наибольшее (0.43449) статистически значимые (P < 0.0001) значения FST определены между популяциями O. lanata.
Таблица 2.
Генетическое разнообразие и популяционная стабильность у видов Oxytropis по данным хпДНК
| Вид и код популяции | Разнообразие (SD) | Тест на популяционную стабильность | ||
|---|---|---|---|---|
| гаплотипическое | нуклеотидное | Tajima’s D | Fu’s Fs | |
| O. lanata | ||||
| LAN1 | 0.895 (0.051) | 0.0010 (0.0006) | –0.84303 ns | –4.90331* |
| LAN2 | 0.895 (0.052) | 0.0010 (0.0006) | –1.00161 ns | –3.44483* |
| LAN3 | 0.900 (0.161) | 0.0017 (0.0012) | –0.81650 ns | –1.55426 ns |
| В целом | 0.923 (0.023) | 0.0012 (0.0007) | –1.16189 ns | –12.82981* |
| O. myriophylla | ||||
| MYR1 | 0.700 (0.218) | 0.0003 (0.0003) | 0.00000 | –0.82920 ns |
| MYR2 | 0.767 (0.057) | 0.0007 (0.0005) | 0.00000 | –0.52438 ns |
| MYR3 | 0.821 (0.101) | 0.0008 (0.0005) | 0.00000 | –0.03355 ns |
| В целом | 0.786 (0.042) | 0.0007 (0.0005) | 0.00000 | –1.6968 ns |
| O. oxyphylla | ||||
| OXY1 | 0.900 (0.161) | 0.0007 (0.0005) | –0.97256 ns | –1.40478 ns |
| OXY2 | 0.958 (0.036) | 0.0023 (0.0013) | –0.46739 ns | –3.75465* |
| OXY3 | 0.964 (0.077) | 0.0045 (0.0026) | 0.50437 ns | 0.11267 ns |
| OXY4 | 1.000 (0.177) | 0.0042 (0.0029) | 1.45884 ns | –0.18978 ns |
| В целом | 0.981 (0.012) | 0.0034 (0.0018) | –1.20403 ns | –11.32100** |
| O. selengensis, SEL | 0.634 (0.127) | 0.0009 (0.0006) | –1.51077* | –1.09311 ns |
| O. stukovii, STUK | 0.833 (0.222) | 0.0005 (0.0005) | 0.00000 | –0.65789 ns |
| O. gracillima, GRAC | 1.000 (0.126) | 0.0042 (0.0027) | –0.81650 ns | –0.31199 ns |
Примечание. SD – стандартное отклонение; * P < 0.05; ** P < 0.001; ns – незначимое. Код популяции см. табл. 1.
Таблица 3.
Генетические дистанции (FST) между популяциями видов Oxytropis lanata, O. myriophylla и O. oxyphylla по данным хпДНК
| Код популяции |
LAN1 | LAN2 | LAN3 | MYR1 | MYR2 | MYR3 | OXY1 | OXY2 | OXY3 |
|---|---|---|---|---|---|---|---|---|---|
| LAN2 | 0.19382* | – | |||||||
| LAN3 | 0.19868** | 0.43449* | – | ||||||
| MYR1 | 0.72032* | 0.70231* | 0.73799* | – | |||||
| MYR2 | 0.72416* | 0.69208* | 0.76576* | –0.01255 ns | – | ||||
| MYR3 | 0.70647* | 0.67393* | 0.72246* | 0.04762 ns | 0.01166 ns | – | |||
| OXY1 | 0.91232* | 0.91079* | 0.89928* | 0.94643* | 0.92185* | 0.92278* | – | ||
| OXY2 | 0.85033* | 0.83463* | 0.81535* | 0.81025* | 0.84937* | 0.81492* | 0.07287 ns | – | |
| OXY3 | 0.79317* | 0.76259* | 0.69164* | 0.67702* | 0.78910* | 0.70269* | 0.30425** | 0.28229* | – |
| OXY4 | 0.84281* | 0.82252* | 0.74437* | 0.73904* | 0.82958* | 0.75674* | 0.32269** | 0.28509** | –0.01926 ns |
Примечание. * P < 0.0001; ** 0.009 < P < 0.05; ns – незначимое. Уровень значимости определен на основе 1023 пермутаций. Код популяции см. табл. 1.
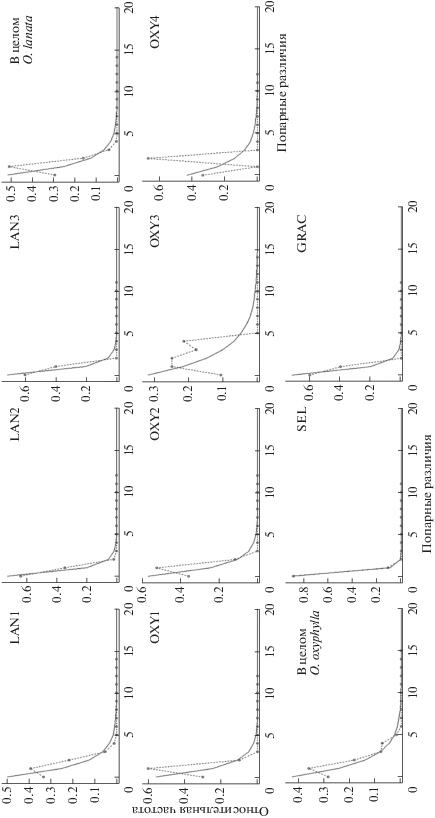
Для реконструкции демографических историй популяций проведен анализ распределения частот парных нуклеотидных различий между гаплотипами для всех популяций, кроме популяций O. myriophylla и O. stukovii, так как в их последовательностях нуклеотидных замен нет. Гистограммы показывают (рис. 1), что только в популяциях OXY3 и OXY4 выявлены значительные различия между ожидаемым и наблюдаемым распределением парных нуклеотидных различий и бимодальный характер наблюдаемого распределения. Во всех остальных популяциях, а также в объединенных выборках каждого из видов O. lanata и O. oxyphylla, две кривые совпадают или показывают относительно хорошее совпадение и имеют унимодальный характер распределения (рис. 1). Значения Tajima’s D и Fu’s Fs (табл. 2) в большинстве популяций, за исключением OXY3, отрицательные, но статистически значимые значения Tajima’s D отмечены только для популяции SEL O. selengensis, а Fu’s Fs – для популяций LAN1 и LAN2 O. lanata, OXY2 O. oxyphylla и для объединенных выборок каждого из видов.
Рис. 1.
Распределение частот величин попарных различий между нуклеотидными последовательностями межгенных спейсеров хпДНК в популяциях видов Oxytropis. Пунктирная линия показывает наблюдаемое распределение, сплошная линия – ожидаемое распределение при модели постоянного популяционного размера. Код популяции см. табл. 1.
Для оценки дивергенции видов Oxytropis секции Verticillares в анализ были включены полученные нами ранее последовательности образцов O. mongolica [19], O. bargusinensis, O. tompudae и O. interposita [21] (табл. 1). Длина объединенных последовательностей 188 образцов 11 видов составила 2484 сайта, из них 2336 мономорфных и 33 вариабельных. Пятнадцать нуклеотидных замен были информативны согласно методу максимальной экономии. Межвидовые генетические дистанции (FST) приведены в табл. 4. Высокие и статистически значимые значения FST определены между парами видов O. lanata, O. myriophylla, O. oxyphylla, O. gracillima, O. interposita и O. tompudae; высокие и незначимые FST между всеми парами, образованными каждым из видов O. pumila и O. mongolica со всеми другими (табл. 4). Генетические дистанции между парами O. oxyphylla–O. stukovii, O. selengensis–O. stukovii и O. oxyphylla–O. bargusinensis очень малы и статистически незначимы, за исключением последней, а между O. oxyphylla–O. selengensis, O. selengensis–O. bargusinensis и O. stukovii–O. bargusinensis величины FST находятся на уровне межпопуляционных значений (табл. 3, 4). Анализ молекулярной дисперсии выявил слабую дифференциацию между видами O. oxyphylla, O. selengensis, O. stukovii и O. bargusinensis (ΦST = 0.137, P < 0.0001), основная доля всей генетической изменчивости (более 86%) приходится на внутривидовую изменчивость и только около 13% – на межвидовую (ΦST = 0.137, P < 0.0001).
Таблица 4.
Генетические дистанции (FST) между видами Oxytropis lanata (1), O. myriophylla (2), O. oxyphylla (3), O. selengensis (4), O. stukovii (5), O. gracillima (6), O. bargusinensis (7), O. interposita (8), O. tompudae (9), O. pumila (10), O. mongolica (11) по данным хпДНК
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | |
|---|---|---|---|---|---|---|---|---|---|---|
| 2 | 0.67563* | – | ||||||||
| 3 | 0.78156* | 0.77768* | – | |||||||
| 4 | 0.88894* | 0.91770* | 0.10515* | – | ||||||
| 5 | 0.88734* | 0.92734* | 0.00520 ns | –0.01037 ns | – | |||||
| 6 | 0.81469* | 0.84348* | 0.76361* | 0.89158* | 0.82679* | – | ||||
| 7 | 0.86259* | 0.88186* | 0.08939** | 0.32339* | 0.31773** | 0.87579* | – | |||
| 8 | 0.74145* | 0.72343* | 0.74864* | 0.89490* | 0.88309* | 0.71406* | 0.86487* | – | ||
| 9 | 0.70156* | 0.72838* | 0.70864* | 0.87429* | 0.83348* | 0.67759* | 0.84445* | 0.67524* | – | |
| 10 | 0.91186 ns | 0.94797 ns | 0.59389 ns | 0.88512 ns | 0.93778 ns | 0.78205 ns | 0.79854 ns | 0.90017 ns | 0.83033 ns | – |
| 11 | 0.87122 ns | 0.90659 ns | 0.78899 ns | 0.94483 ns | 0.97028 ns | 0.68323 ns | 0.91268 ns | 0.85661 ns | 0.76984 ns | 1.00000 ns |
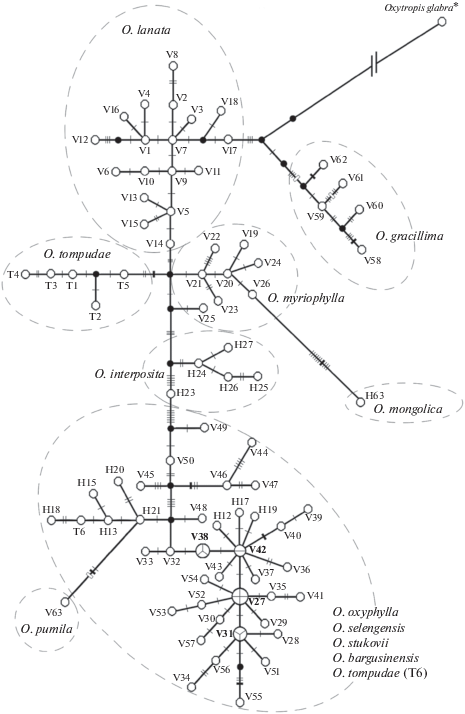
Для определения генеалогических связей между гаплотипами в матрицу выявленных гаплотипов семи исследуемых видов Oxytropis были добавлены последовательности гаплотипов O. mongolica (H63), O. bargusinensis (H11–H22), O. interposita (H23–H27), O. tompudae (T1–T6) и O. glabra (G1) в качестве внешней группы (табл. 1). Общая длина выровненных последовательностей 92 гаплотипов трех регионов хпДНК составила 2580 сайтов, из них 2156 мономорфных и 44 вариабельных. Десять нуклеотидных замен были информативны согласно методу максимальной экономии. Выявлено четыре общих гаплотипа: гаплотип V27, который присутствовал у O. oxyphylla, O. selengensis и O. stukovii, оказался идентичен гаплотипу H16 O. bargusinensis; гаплотип V31, выявленный у O. oxyphylla и O. stukovii, идентичен гаплотипу H22 O. bargusinensis; гаплотип V38, общий у O. oxyphylla и O. selengensis, идентичен гаплотипу H14 O. bargusinensis; гаплотип V42 O. oxyphylla идентичен гаплотипу H11 O. bargusinensis (табл. 1). На рис. 2 представлена медианная сеть, в которой все гаплотипы O. lanata, O. myriophylla, O. gracillima, O. interposita и пять из шести гаплотипов O. tompudae образуют гаплогруппы согласно видовой принадлежности. Все гаплотипы O. lanata имеют две маркерные нуклеотидные замены (А в позиции 1196 и С в позиции 2291), гаплотипы O. interposita – три (Т в позициях 1287 и 1723, G в позиции 1513), а гаплотипы O. gracillima – одну (Т в позиции 403) и вставку пяти нуклеотидов (AGTAA в позиции 1759), отсутствующую у всех других. Гаплотип V63 O. pumila связан с гаплотипом H21 O. bargusinensis через три мутационных шага, два из которых: вставка семи нуклеотидов (GAATTAT, позиции 1435–1441) и делеция пяти нуклеотидов (ACCTT, позиции 1796–1800), отличают O. pumila от всех остальных. Гаплотип H63 O. mongolica связан с гаплотипом V26 O. myriophylla через десять мутационных шагов, из них шесть нуклеотидных замен (T в позициях 158 и 1382, A в позициях 1457 и 1765, G в позиции 1704 и C в позиции 1715), вставка шести нуклеотидов (TATATC, позиции 1383–1388) и делеция двух нуклеотидов (AT, позиции 1146–1147) отличают O. mongolica от всех остальных. Одну большую гаплогруппу формируют все гаплотипы видов O. oxyphylla, O. selengensis, O. stukovii, O. bargusinensis и гаплотип T6 O. tompudae. В ней присутствуют хорошо выраженные “звездчатые” структуры с одним из общих гаплотипов V27, V31, V38 и V42 в центре, которые связаны друг с другом через один мутационный шаг (рис. 2), что свидетельствует о недавнем происхождении популяций на исследованной части ареала.
Рис. 2.
Генеалогическая сеть гаплотипов видов Oxytropis, построенная с помощью MJ-метода. Размер окружностей гаплотипов отражает частоту их встречаемости, маленькие черные кружки – медианные векторы, поперечные тонкие штрихи на ветвях – число мутационных событий, черные толстые штрихи – вставка нуклеотидов; белые толстые штрихи – делеция нуклеотидов. Пунктирной линией объединены гаплотипы одного вида или группы видов. * Мутации для O. glabra, используемой в качестве внешней группы, не рассматриваются и не указаны.
ОБСУЖДЕНИЕ
Проведенное нами изучение популяций O. lanata, O. myriophylla, O. oxyphylla, O. selengensis, O. stukovii и O. gracillima секции Verticillares по данным полиморфизма межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК выявило высокое гаплотипическое (h изменялось от 0.634 до 1.000) и относительно низкое нуклеотидное (π от 0.0003 до 0.0045) разнообразие (табл. 2). Уровень генетического разнообразия по данным изменчивости этих же регионов сравним с таковым в популяциях O. glandulosa, расположенных в Баргузинском (h = 0.703, π = 0.0036) и Курумканском районах Бурятии (h = 0.911, π = 0.0059) [20], у эндемичных видов O. bargusinensis (h = 0.844, π = 0.0013), O. interposita (h = 0.769, π = 0.0015) и O. triphylla (h = 0.915, π = 0.0020) [21], а также у O. chankaensis (h = 0.718, π = 0.0005) по данным изменчивости psbA–trnH, trnL–trnF, trnS–trnG и petG–trnP хпДНК [15]. Сочетание высоких значений гаплотипического разнообразия при низких значениях нуклеотидного разнообразия предполагает быстрый рост популяции из древней популяции с низким эффективным размером [34], при этом временной интервал должен быть достаточным для восстановления гаплотипического разнообразия посредством мутаций, но недостаточным для накопления значительных различий в последовательностях.
Отсутствие популяционной дифференциации у O. myriophylla (ΦST = 0.008, P > 0.37), как и у O. bargusinensis (ΦST = 0.027, P > 0.20) [21], обусловлено, вероятно, не прекращающимся обменом генами через цепь промежуточных локальных местообитаний. Унимодальное распределение парных нуклеотидных различий в большинстве исследуемых популяций видов O. lanata, O. oxyphylla, O. selengensis и O. gracillima (рис. 1) свидетельствует о возможной недавней демографической экспансии [35, 36] или о пространственном расширении с высоким уровнем генного потока между соседними популяциями [37]. Демографические сценарии для объединенных выборок каждого из видов O. lanata и O. oxyphylla подтверждаются также высокими и статистически значимыми (P < 0.05) отрицательными значениями Fu’s Fs (–12.82981 и –11.32100 соответственно). Бимодальный характер наблюдаемого распределения в популяциях OXY3 и OXY4 O. oxyphylla указывает на то, что они, вероятно, находятся в демографическом равновесии.
Высокие генетические дистанции между видами O. lanata, O. myriophylla, O. oxyphylla, O. gracillima, O. interposita и O. tompudae (табл. 4), а также выявленные маркерные нуклеотидные замены и индели у O. lanata, O. gracillima, O. interposita, O. pumila и O. mongolica указывают на существенную дифференциацию их хлоропластных геномов. Объединение всех гаплотипов видов O. oxyphylla, O. selengensis, O. stukovii, O. bargusinensis и гаплотипа T6 O. tompudae в одну гаплогруппу (рис. 2) не случайно. Проведенное нами ранее [21] изучение филогенетических связей O. triphylla, O. bargusinensis, O. interposita с близкородственными видами подтвердило сформированное на основе морфологических признаков представление о происхождении O. bargusinensis и O. tompudae в результате межсекционной гибридизации и показало, что для гаплотипа T6 O. tompudae родитель по материнской линии был близок к O. bargusinensis, и это могли быть O. oxyphylla или O. turczaninovii. Обнаруженные общие гаплотипы у видов O. oxyphylla, O. selengensis, O. stukovii и O. bargusinensis (табл. 1, см. “Результаты”), присутствие в медианной сети “звездчатых” структур с одним из общих гаплотипов в центре (рис. 2), слабая дифференциация между этими видами (ΦST = 0.137), которая оказалась ниже, чем между популяциями у видов O. lanata (ΦST = 0.245) и O. oxyphylla (ΦST = 0.229), низкие значения генетических дистанций (табл. 3, 4), соответствующие межпопуляционным, а также отсутствие дифференцирующих молекулярных маркеров – все в целом указывает на общность происхождения этих четырех видов, их относительно недавнюю дивергенцию и на высокую степень межвидовой гибридизации.
Полученные результаты, с одной стороны, подтверждают мнения авторов, основанные на морфологических признаках, что О. selengensis может быть мелкоцветковой формой O. oxyphylla с низкими простертыми цветоносами [13] или эколого-географической расой O. oxyphylla [27], что некоторые экземпляры О. selengensis, возможно, гибридного происхождения, так как совмещают признаки O. oxyphylla [14], и, с другой стороны, ставят под сомнение предположение о том, что O. stukovii – это промежуточная раса между O. prostrata и О. mongolica [13]. Для решения вопроса о таксономическом статусе O. selengensis и O. stukovii необходимо исследование расширенной выборки растений вышеперечисленных видов.
Исследование выполнено при финансовой поддержке гранта РФФИ (проект № 16-04-01399) и программы Президиума РАН “Дальний Восток” (проект № 18-4-011).
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Пешкова Г.А. Степная флора Байкальской Сибири. М.: Наука, 1972. 207 с.
Пешкова Г.А. Флорогенетический анализ степной флоры гор Южной Сибири. Новосибирск: Наука, 2001. 192 с.
Малышев Л.И., Пешкова Г.А. Особенности и генезис флоры Сибири (Предбайкалье и Забайкалье). Новосибирск: Наука, 1984. 364 с.
Намзалов Б.Б. К вопросу о реликтах во флоре и растительности степных экосистем Байкальской Сибири // Растительный мир Азиатской России. 2012. № 2. С. 94–100.
Намзалов Б.Б. Байкальский фитогеографический узел как новейший центр эндемизма Внутренней Азии // Сиб. экол. журн. 2009. Т. 16. № 4. С. 563–571.
Положий А.В. К вопросу о происхождении и эволюции рода Oxytropis (Fabaceae) // Бот. журн. 2003. Т. 88. № 10. С. 55–59.
Юрцев Б.А. Основные пути эволюции остролодочников секции Baicalia Bge. // Бот. журн. 1964. Т. 49. № 5. С. 634–648.
Васильченко И.Т. К вопросу о генезисе рода остролодочник Oxytropis DC. // Бот. журн. 1965. Т. 50. № 3. С. 313–323.
Положий А.В. Флорогенетический анализ остролодочников Средней Сибири // Уч. зап. Томск. гос. ун-та. Биология и почвоведение. 1965. № 51. С. 18–38.
Пешкова Г.А. Флора Центральной Сибири. Т. 2. Новосибирск: Наука, 1979. С. 623–624.
Сипливинский В.Н. Флористические находки в северо-восточном Прибайкалье // Новости систематики высших растений. М.; Л.: Наука, 1966. С. 272–291.
Малышев Л.И. Фенетика в секции Verticillares рода Oxytropis (Fabaceae) // Бот. журн. 2007. Т. 92. № 6. С. 793–807.
Попов М.Г. Флора Средней Сибири. Т. 1. М.; Л.: Изд-во. АН СССР, 1957. С. 347.
Положий А.В. Oxytropis DC. // Флора Сибири. Новосибирск: Наука, 1994. Т. 9. С. 74–151.
Artyukova E.V., Kozyrenko M.M., Kholina A.B., Zhuravlev Yu.N. High chloroplast haplotype diversity in the endemic legume Oxytropis chankaensis may result from independent polyploidization events // Genetica. 2011. V. 139. № 2. P. 221–232. https://doi.org/10.1007/s10709-010-9539-8
Archambault A., Stromvik M.V. Evolutionary relationships in Oxytropis species, as estimated from the nuclear ribosomal internal transcribed spacer (ITS) sequences point to multiple expansions into the Arctic // Botany. 2012. V. 90. № 8. P. 770–779. https://doi.org/10.1139/B2012-023
Gao L., Lu P., Jin F. et al. TrnL-F sequences analysis and molecular phylogeny of 10 species of Oxytropis // Acta Bot. Boreali-Occident. Sin. 2013. № 2. P. 266–271.
Lu P., Gao L., Jin F., En He-Ba-Ya-Er. Molecular phylogeny of 10 species of Oxytropis based on psbA–trnH sequences // Acta Bot. Yunnanica. 2014. № 3. P. 279–284.
Холина А.Б., Козыренко М.М., Артюкова Е.В. и др. Филогенетические взаимоотношения видов Oxytropis DC. subg. Oxytropis и Phacoxytropis (Fabaceae) Азиатской России на основе анализа нуклеотидных последовательностей межгенных спейсеров хлоропластного генома // Генетика. 2016. Т. 52. № 8. С. 895–909.
Kholina A., Kozyrenko M., Artyukova E. et al. Plastid DNA variation of the endemic species Oxytropis glandulosa Turcz. (Fabaceae) // Turk. J. Bot. 2018. V. 42. P. 38–50. https://doi.org/10.3906/bot-1706-11
Холина А.Б., Козыренко М.М., Артюкова Е.В., Санданов Д.В. Современное состояние популяций эндемичных видов Oxytropis Байкальской Сибири и их филогенетические связи по данным секвенирования маркеров хлоропластной ДНК // Генетика. 2018. Т. 54. № 7. С. 795–806.
Tekpinar A., Karaman Erkul S., Aytac Z., Kaya Z. Phylogenetic relationships among native Oxytropis species in Turkey using trnL intron, trnL-F IGS, and trnV intron cpDNA regions // Turk. J. Bot. 2016. V. 40. P. 472–479. https://doi.org/10.3906/bot-1506-45
Shavvon R.S., Kazempour-Osaloo S., Maassoumi A.A. et al. Increasing phylogenetic support for explosively radiating taxa: The promise of high-throughput sequencing for Oxytropis (Fabaceae) // J. Syst. Evol. 2017. V. 55. № 4. P. 385–404. https://doi.org/10.1111/jse.12269
Zhang M.-L., Wen Z.-B., Hao X.-L. et al. Taxonomy, phylogenetics and biogeography of Chesneya (Fabaceae), evidenced from data of three sequences, ITS, trnS-trnG, and rbcL // Biochem. Syst. Ecol. 2015. V. 63. P. 80–89. http://dx.doi.org/https://doi.org/10.1016/j.bse.2015.09.017.
Xie Y.-P., Meng Y., Sun H., Nie Z.-L. Molecular phylogeny of Gueldenstaedtia and Tibetia (Fabaceae) and their biogeographic differentiation within Eastern Asia // PLoS One. 2016. V. 11. № 9. e0162982. https://doi.org/10.1371/ journal.pone.0162982
Артюкова Е.В., Козыренко М.М. Филогенетические отношения Oxytropis chankaensis Jurtz. и Oxytropis oxyphylla (Pall.) DC. (Fabaceae) по данным секвенирования ITS рибосомного оперона ядерной ДНК и межгенных спейсеров хлоропластного генома // Генетика. 2012. Т. 48. № 2. С. 186–193.
Малышев Л.И. Разнообразие рода Остролодка (Oxytropis) в Азиатской России // Turczaninowia. 2008. Т. 11. № 4. С. 5–141.
Артюкова Е.В., Холина А.Б., Козыренко М.М., Журавлев Ю.Н. Анализ генетической изменчивости редкого эндемичного вида Oxytropis chankaensis Jurtz. (Fabaceae) на основе RAPD маркеров // Генетика. 2004. Т. 40. № 7. С. 877–884.
Bonfeld J.K., Smith K.F., Staden R. A new DNA sequence assembly program // Nucl. Acids Res. 1995. V. 23. P. 4992–4999. https://doi.org/10.1093/nar/23.24.4992
Gouy M., Guindon S., Gascuel O. SeaView version 4: A multiplatform graphical user interface for sequence alignment and phylogenetic tree building // Mol. Biol. Evol. 2010. V. 27. P. 221–224. https://doi.org/10.1093/molbev/msp259
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btp187
Bandelt H.-J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48.
Avise J.C. Phylogeography: The History and Formation of Species. Cambridge, MA: Harvard Univ. Press, 2000. 441 p.
Slatkin M., Hudson R.R. Pairwise comparisons of mitochondrial DNA sequences in stable and exponentially growing populations // Genetics. 1991. V. 129. P. 555–562.
Rogers A.R., Harpending H. Population growth makes waves in the distribution of pairwise genetic differences // Mol. Biol. Evol. 1992. V. 9. № 3. P. 552–569.
Excoffier L. Patterns of DNA sequence diversity and genetic structure after a range expansion: lessons from the infinite-island model // Mol. Ecol. 2004. V. 13. P. 853–864.
Дополнительные материалы отсутствуют.


