Генетика, 2020, T. 56, № 11, стр. 1304-1319
Синтез флавоноидных пигментов в зерновке у представителей Poaceae: общие закономерности и исключения в гомологических рядах Н.И. Вавилова
К. В. Стрыгина *
Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
* E-mail: k.strygina@vir.nw.ru
Поступила в редакцию 03.05.2020
После доработки 05.06.2020
Принята к публикации 17.06.2020
Аннотация
Флавоноидные пигменты представляют группу вторичных метаболитов растений. Интерес к данным соединениям обусловливается широким спектром их биологических свойств, поэтому насыщение флавоноидами съедобных частей растений, в частности зерновок злаков, сегодня является актуальной задачей. Синтез флавоноидных пигментов можно наблюдать в семенной кожуре, перикарпе, алейроновом слое и эндосперме зерна у представителей большинства злаковых культур (кукуруза Zea mays L., рис Oryza sativa L., пшеница мягкая Triticum aestivum L., ячмень Hordeum vulgare L., рожь Secale cereale L., сорго Sorghum bicolor (L.) Moench. и др.), что согласуется с Законом гомологических рядов в наследственной изменчивости Н.И. Вавилова. Однако по некоторым признакам у отдельных видов изменчивости не наблюдается: например, у некоторых представителей трибы Triticeae не найдены формы, синтезирующие антоцианы в алейроновом слое. Гомологические ряды не выявлены и для некоторых уникальных признаков. Например, соединения, относящиеся к группе 3-дезоксиантоцианидинов, имеющие нехарактерный для злаков желто-оранжевый цвет, синтезируются в перикарпе только у сорго. В данном обзоре представлена информация о генетическом разнообразии и особенностях биосинтеза флавоноидных пигментов в зерновке у злаковых культур, характеризующих природу наблюдаемых закономерностей и исключений из Закона Н.И. Вавилова.
Семейство Poaceae Barnhart (Мятликовые; альтернативное название Злаки или Злаковые, Gramineae Juss.) представляет крупную группу однодольных растений: согласно последним данным, оно насчитывает около 12 000 видов, которые подразделяются на 2 клады, 12 подсемейств, 7 супертриб, 52 трибы, 90 подтриб и 768 родов [1–5]. По своей численности Poaceae является пятым семейством покрытосеменных растений после Астровых (Asteraceae Bercht. & J. Presl, альтернативное название Compositae Giseke), Орхидных (Orchidaceae Juss.), Бобовых (Fabaceae Lindl., альтернативное название Leguminosae Juss., nom. cons.) и Мареновых (Rubiaceae Juss.) [6].
Фенотипически варьирующие от трав до гигантских бамбуков злаки заселяют все основные массивы суши. Ареалы преобладания злаков занимают до 40% поверхности Земли и включают умеренные пастбища, тропические саванны и пахотные земли [7].
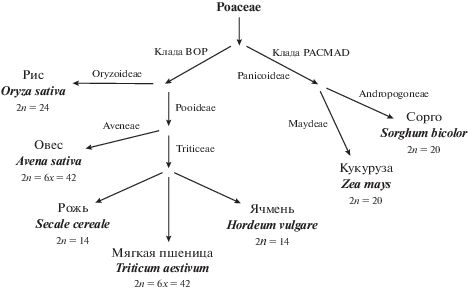
Злаковые представляют особый интерес для хозяйственной деятельности человека. Их экономическая значимость связана с несколькими областями, включая производство продуктов питания, кормопроизводство, перерабатывающую промышленность, производство источников энергии, а также озеленение [7]. Для большей части населения планеты злаки представляют существенную долю рациона питания. Наиболее важными продовольственными культурами для человека являются рис (Oryza L.), кукуруза (Zea L.), пшеница (Triticum L.), ячмень (Hordeum L.), рожь (Secale L.), овес (Avena L.) и сорго (Sorghum Moench) (рис. 1). Такие культуры, как просо (Panicum L.), мятлик (Poa L.), полевичка (Eragrostis Wolf), тимофеевка (Phleum L.), плевел (Lolium L.) и овсяница (Festuca L.), являются важной частью городского и пригородного ландшафта и составной частью рациона домашних животных [8]. Кроме того, представители Poaceae служат в качестве строительного материала (бамбук Bambusa Schreb. и Phyllostachys Siebold & Zucc.) [9]. Также в последнее время в качестве источника сырья для биомассы и биотоплива стали использоваться мискантус (Miscanthus Anderss.), арундо (Arundo L.) и сахарный тростник (Saccharum L.) [8].
Важными особенностями эволюции геномов представителей семейства Poaceae являются частые случаи межвидовой гибридизации, высокая частота полиплоидии, широкий диапазон изменения размера генома (содержание ДНК 2C варьирует от 0.7 в Chloris gayana Kunth до 27.6 в Lygeum spartum Loefl. ex L.) и изменения в базовом числе хромосом (х = 2–14 и 18), что демонстрируется на примере хозяйственно ценных культур на рис. 1 [4, 10–14]. Интересно, что при этом виды семейства Poaceae в пределах рода имеют, как правило, идентичное значение базового числа хромосом: в большинстве родов х = 10 (как у кукурузы Z. mays L. или сорго S. bicolor (L.) Moench) или 7 (как у мягкой пшеницы T. aestivum L. или овса A. sativa L.) (рис. 1).
Но несмотря на различия, многие морфологические признаки у злаков, включая наличие окраски различных частей растения, соответствуют Закону гомологических рядов в наследственной изменчивости, который был сформулирован Н.И. Вавиловым, согласно которому генетически тесно связанные виды и роды обычно характеризуются сходными рядами генетической изменчивости [15]. Так, сходные признаки окраски, связанной в том числе с пигментами флавоноидами, встречаются у всех важных для человека продовольственных культур [16].
Пигменты флавоноиды представляют группу вторичных растительных метаболитов фенольной природы. Данные соединения ответственны за окраску вегетативных и генеративных органов многих видов высших растений, а также играют роль в метаболизме как регуляторы роста и защиты от факторов абиотического и биотического стресса [17–21].
Обнаруженная в первой половине XX в. биоактивность флавоноидов вызвала большой интерес к этим соединениям. Растительная пища, богатая определенными флавоноидами, служит источником полезных для здоровья человека и домашних животных соединений [22–24], поэтому пигментированные за счет накопления флавоноидов зерновки злаков, которые отличаются длительностью хранения, доступностью и частым употреблением, перспективны в качестве функциональных продуктов и пищевых красителей [16, 25–29], а насыщение зерновок злаков флавоноидами и выведение новых сортов и линий методами традиционной и ускоренной маркер-ориентированной селекции, основанной на анализе генов биосинтеза данных соединений, сегодня является актуальной задачей [30–39].
Биосинтез флавоноидов представляет собой один из самых полно охарактеризованных метаболических путей у растений [40]. Благодаря работе более 20 структурных генов, кодирующих ферменты пути биосинтеза флавоноидов, известно огромное множество различных соединений этого класса [17]. Среди основных групп флавоноидов за красную, красно-коричневую, красно-фиолетовую, голубую и розовую окраску различных органов и тканей представителей семейства Poaceae в основном отвечают флобафены, проантоцианидины (конденсированные танины) и антоцианы (рис. 2). Данные соединения могут синтезироваться и накапливаться в вегетативных органах, а также в семенной кожуре, перикарпе, алейроновом слое и эндосперме зерна злаков [41–43]. Наличие такой фенотипической повторяемости, согласно Закону гомологических рядов, часто является следствием гомологии в последовательностях ДНК, кодирующих регуляторные и структурные гены биосинтеза флавоноидных пигментов, что предоставляет возможность с помощью методов генетики и биоинформатики обнаруживать важные с точки зрения селекции гены.
Рис. 2.
Путь биосинтеза флавоноидных пигментов зерновок злаковых растений. Прямоугольником выделены классы соединений, возле стрелок – основные ферменты биосинтеза. Базовая структура флавоноидов показана на примере флаванонов.
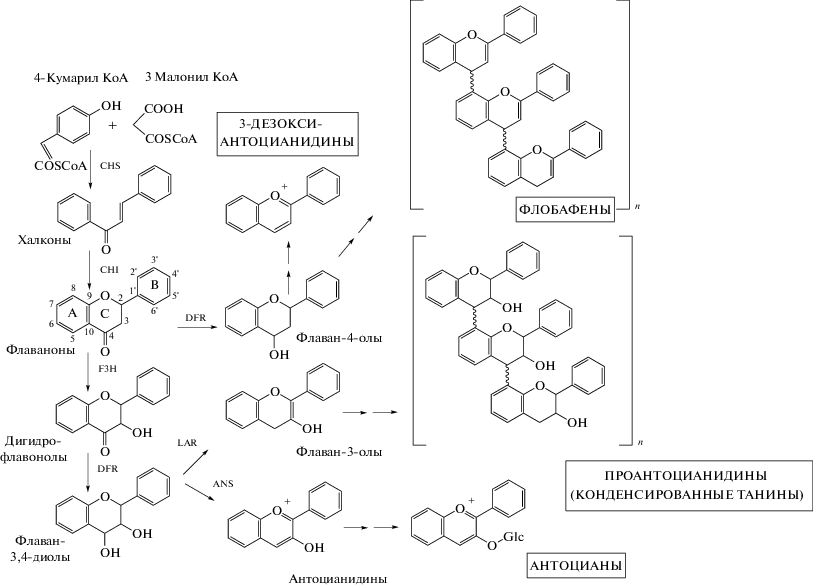
Однако наряду с общими закономерностями в гомологических рядах Н.И. Вавилова для ряда важных зерновых культур, таких как пшеница мягкая T. aestivum, ячмень H. vulgare L., рожь S. cereale L., рис O. sativa L., кукуруза Z. mays и сорго S. bicolor, наблюдаются исключения по признаку пигментация зерновки, проявляющиеся как закрепившиеся мутации в ключевых генах биосинтеза флавоноидов или как появление в геноме их копий. Например, отсутствие антоцианов в алейроновом слое у некоторых представителей трибы Triticeae Dumort. (Пшеницевые) или синтез желто-оранжевых 3-дезоксиантоцианидинов в перикарпе у сорго является уникальным для семейства Poaceae. В данном обзоре собрана информация, касающаяся особенностей биосинтеза флавоноидов в зерне у разных представителей Poaceae.
АНТОЦИАНЫ
Среди злаков распространены фенотипы с красно-фиолетовой (черной), голубой (серой, зеленой) и розовой пигментацией различных слоев зерновки [44]. Данная окраска обусловливается накоплением флавоноидных пигментов антоцианов (рис. 2).
Антоцианы представляют значительный интерес благодаря своей антиоксидантной активности. Известно, что антоцианы обладают противовоспалительными и антиканцерогенными свойствами, а также ослабляют диабет, ожирение, гипергликемию, гипертонию и сердечно-сосудистые заболевания [31, 45–47].
В зернах злаков антоцианы накапливаются в основном в наружных слоях зерна, придавая зерновке красно-фиолетовый цвет [48–58]. Например, наибольшая концентрация антоциановых пигментов в кукурузе Z. mays обнаруживается в перикарпе, тогда как в алейроновом слое содержится небольшая фракция антоцианов [59]. При этом накапливать голубые антоцианы в алейроновом слое зерна, характерные для ячменя и ржи, многие виды злаков не способны [60].
Среди нескольких сотен различных антоцианов в продуктах питания обычно содержатся агликоны шести антоцианидинов: цианидины, дельфинидины, петунидины, пеонидины, пеларгонидины и мальвидины [17, 61, 62]. Наиболее распространенными антоцианами злаков являются цианидин-3-глюкозид, цианидин-3-рутинозид (красно-фиолетовая окраска), пеларгонидин-3-глюкозид (розовая окраска), дельфинидин-3-рутинозид и дельфинидин 3-глюкозид (голубая окраса) [39, 58, 59, 63, 64].
Синтез антоцианов и их тканеспецифическое накопление контролируются работой регуляторных генов, кодирующих факторы транскрипции bHLH, Myb и WD40 [31]. При этом более важными с позиции разработки маркеров для селекции являются гены Myb и bHLH; мутации в структурных генах, а также в гене WD40 ввиду их консервативности выявляются редко [65].
Гены биосинтеза антоцианов в перикарпе
Гены биосинтеза красно-фиолетовых антоцианов (производных цианидина) в перикарпе хорошо изучены для многих злаков [66]. Лучше всего регуляция данного пути биосинтеза исследована на примере кукурузы Z. mays [52, 67, 68]. Помимо структурных генов, в геноме кукурузы выявлены ключевые гены синтеза антоцианов в перикарпе – ген B1 (хромосома 2) семейства bHLH, а также ген Pl1 (хромосома 6), который принадлежит к семейству генов Myb [69–72]. Также у кукурузы расшифрована последовательность гена Pac1, кодирующего белок WD40, необходимого для активации генов биосинтеза антоцианов в разных слоях зерновки [73, 74].
У риса O. sativa локусы на хромосомах 1, 3 и 4 (гены Kala1, Kala3 и Kala4, соответственно) обеспечивают антоциановую пигментацию перикарпа [75]. Kala1 кодирует фермент DFR (дигидрофлаванол-4-редуктаза), участвующий в пути синтеза как антоцианов, так и проантоцианинов (см. ниже) [76]. Ген Kala4 кодирует белок bHLH; Kala3, вероятно, является фактором транскрипции семейства Myb [77–79].
Установлено, что ключевые гены синтеза антоцианов в перикарпе ячменя H. vulgare Ant2 и Ant1 – это гены bHLH и Myb, которые располагаются в хромосомах 2H (ген Myc1) и 7H (ген Mpc1-H1) в коллинеарных локусах хромосом риса O. sativa [60, 80–82]. У пшеницы T. aestivum ортологичные гены типа bHLH (Myc-A1) и Myb (Mpc1-A1, Mpc1-B1 и Mpc1-D1) локализованы в хромосомах 2A и 7A, 7B и 7D и соответствуют ключевым локусам Pp3 и Pp-A1, Pp-B1 и Pp-D1 [43, 83, 84]. Кроме того, для данных культур известны WD40-кодирующие гены, транскрипционно активные как в перикарпе, так и в алейроновом слое и локализованные в шестой гомеологической группе хромосом, однако аллельный полиморфизм этих генов выявлен не был [85, 86].
Известно, что у ржи S. cereale гены Vi1–Vi6 необходимы для синтеза флавоноидных пигментов во всем растении, в том числе в перикарпе; локализация известна только для гена Vi1 – хромосома 7R [87, 88]. Также у ржи известен локус, вовлеченный в синтез пигментов перикарпа – ген Vs, картированный в хромосоме 2R в коллинеарном регионе хромосом 2H ячменя и 2A пшеницы и, вероятно, являющийся геном bHLH [43, 48].
Гены биосинтеза антоцианов в алейроновом слое
Появление голубой (зеленой) окраски алейронового слоя у ржи S. cereale связывают с доминантными локусами A, R и B (позже стал называться C) [48]. При этом ген A, необходимый для окраски всего растения, вероятно, является структурным геном, а B и R – регуляторными, необходимыми для появления окраски только в алейроне [88–91]. Кроме того, гены A и B являются тесно сцепленными, а гены A и R – слабо сцепленными и расположенными в порядке B–A–R, однако их хромосомная локализация неясна [91]. Оригинальные формы ржи с идентифицированными генами сохраняются только в Петергофской генетической коллекции, основанной В.С. Федоровым [92].
Окраску алейронового слоя ячменя H. vulgare связывают с наличием пяти функциональных генов Blx. Три гена (Blx1, Blx3, Blx4) тесно сцеплены и расположены на хромосоме 4HL; Blx2 расположен на хромосоме 7HL [93]. Цвет алейронового слоя может быть изменен с голубого до розового тогда, когда комплементарные доминантные аллели присутствуют в локусах Blx1, Blx2, Blx3 и Blx5, а в локусе Blx4 – рецессивный аллель. К настоящему моменту стало известно, что гены Blx1 и Blx3 кодируют факторы транскрипции Myb (ген Mpc1-H3) и bHLH (ген Myc2), а Blx4 – это структурный ген F3'5'H-1, кодирующий фермент флавоноид-3',5'-гидроксилазу (F3'5'H), мутации в котором блокируют синтез дельфинидина, благодаря чему в алейроне могут накапливаться только розовые производные пеларгонидина [85, 94, 95]. Таким образом, у ячменя выявлены все гены-кандидаты хромосомы 4HL для признака “голубая окраска зерна” [65].
Биосинтез антоцианов в алейроновом слое мягкой пшеницы находится под контролем генов Ba [96]. Однако в отличие от ячменя мягкая пшеница может иметь голубую окраску алейронового слоя только за счет замены хромосомы четвертой гомеологической группы или ее части на хромосомы родственных видов, способных продуцировать голубой пигмент в зерновке: диплоидной пшеницы T. monococcum subsp. boeoticum Boiss (дикая форма) или T. monococcum subsp. monococcum L. (доместицированная форма) [96, 97], тетраплоидной пшеницы T. turgidum (L.) subsp. turgidum (L.) convar. durum (Desf.) [98] или диплоидного пырея Thinopyrum intermedium (Host) Barkworth & D.R. Dewey или Th. ponticum (Podp.) Z.-W. Liu & R.-C. Wang [37, 96, 99–103]. Ген Ba1 хромосомы 4B мягкой пшеницы, перенесенный в геном мягкой пшеницы из Th. ponticum (обозначен ThMYC4E), оказался гомологичным генам четвертой гомеологической группы хромосом T. aestivum и кодирующим фактор транскрипции bHLH-типа [99–101, 103].
Анализ филогенетических взаимоотношений и синтении показал, что локус, содержащий кластер генов Myb-bHLH-F3'5'H в четвертой гомеологической группе хромосом, возникший в результате дупликаций генов, закрепился в ходе естественного и искусственного отбора только у отдельных представителей трибы Triticeae [94, 95, 104–106].
Исключением служат антоцианы, синтезируемые в алейроновом слое кукурузы Z. mays, поскольку филогенетически кукуруза далека от трибы Triticeae (рис. 1). Однако гены кукурузы, контролирующие синтез пигментов в данной ткани, как и у представителей трибы Triticeae, являются паралогичными копиями регуляторных генов, активирующих перикарп-специфический синтез антоцианов. У кукурузы известны два локуса R1 (хромосома 10) и C1 (хромосома 9), которые необходимы для пигментации алейронового слоя. В дополнение к данным регуляторам известен локус Pr1 (хромосома 5), благодаря которому под контролем генов C1 и R1 вместо фиолетовых цианидиновых пигментов происходит накопление розовых (красных) производных пеларгонидина в алейроновом слое [69–71]. Оказалось, что ген R1 – bHLH-кодирующий, а C1 – Myb-кодирующий, являющиеся результатом независимой дупликации генов B1 и Pl1 (см. выше) [70, 71, 107]. Ген Pr1 оказался структурным геном F3'H1, кодирующим фермент флавоноид-3'-гидроксилазу (F3'H), рецессивное состояние которого приводит к появлению фенотипа с розовым зерном [108].
Таким образом, путь биосинтеза антоцианов наиболее хорошо изучен и полно охарактеризован среди путей биосинтеза флавоноидов злаков. В перспективе на основании имеющихся данных, а также благодаря появляющимся данным, касающимся полногеномного секвенирования представителей Poaceae, по гомологии возможно выявить полный набор генов, регулирующих появление антоцианов в зерне у тех хозяйственно ценных злаков, для которых эти гены еще неизвестны.
ПРОАНТОЦИАНИДИНЫ
Проантоцианидины представляют собой метаболиты, синтезируемые растениями в виде олигомеров или полимеров флаван-3-олов, которые состоят в основном из катехина, эпикатехина, галлокатехина и эпигаллокатехина (рис. 2) [109, 110]. Благодаря своим антиоксидантным, противоопухолевым и иммуностимулирующим свойствам проантоцианидины являются полезными для здоровья человека соединениями [111, 112].
Путь биосинтеза проантоцианидинов, подобно другим путям вторичного метаболизма у растений, находится под сложным контролем множества регуляторных генов на уровне транскрипции [110]. Эти регуляторные гены подразделяются на несколько различных семейств, наиболее значимые из которых транскрипционные факторы bHLH, Myb и WD40, активирующие работу структурных генов биосинтеза проантоцианидинов, а также других флавоноидов, таких как антоцианы (см. выше) [113, 114].
К настоящему моменту известно, что только некоторые генотипы риса O. sativa, пшениц, ячменя H. vulgare, сорго S. bicolor, отдельных видов проса и кормовых трав (дагусса Eleusine coracana (L.) Gaertn., плевел L. perenne L. и овсяница F. arundinacea Schreb.) способны накапливать проантоцианидины в перикарпе и семенной кожуре, придавая зерновке красно-коричневую окраску [43, 115–122].
Гены биосинтеза проантоцианидинов в перикарпе и семенной кожуре
У риса O. sativa проантоцианидины накапливаются в перикарпе [122]. При этом в красном рисе их концентрация намного выше, чем в красно-фиолетовом (черном) и коричневом [123, 124]. Биосинтез проантоцианидинов в перикарпе риса контролируется генами Rc и Rd, расположенными на хромосомах 7 и 1 соответственно [125–127]. При этом было известно, что доминантные варианты Rc и Rd придают зерну красную окрасу, при сочетании доминантного Rc и рецессивного rd пигментация зерна становится коричневой, а аллели rc и rd или rc и Rd дают обычные зерна белого цвета. В 2006 г. благодаря двум независимым работам стало известно, что белок Rc – это фактор транскрипции типа bHLH, а Rd – это фермент DFR [125, 126].
У пшеницы T. aestivum и ячменя H. vulgare проантоцианидины накапливаются в семенной кожуре [60, 116, 128]. Проантоцианидиновый цвет зерновки пшеницы контролируется доминантными аллелями генов R-1, расположенных на длинных плечах трех гомеологичных хромосом 3A, 3B и 3D [43]. Кроме того, красно-коричневый цвет зерна – это аддитивный признак, и его интенсивность зависит от количества доминантных аллелей генов R-1 [102]. В 2011 г. было установлено, что R-1 кодируют регуляторные факторы Myb, регулирующие экспрессию структурных генов биосинтеза флавоноидов [132].
Для ячменя известно 11 независимых локусов, ассоциированных с фенотипом зерна, не содержащим проантоцианидины. Шесть из них – Ant13, 17, 18, 21, 22 и 25 – общие в пути биосинтеза антоцианов и проантоцианидинов, мутации в них приводят к отсутствию данных пигментов [130]. Мутации в пяти других (Ant19, 26, 27, 28 и 29) приводят к прекращению синтеза проантоцианидинов в зерне [116, 131]. Из них ген Ant28 локализован в дистальном районе хромосомы 3H в коллинеарной области хромосом 3H пшеницы. Оказалось, что он, как и R-1, кодирует фактор транскрипции Myb [129, 132]. Ортологичные гены Ant28 и R-1 были переименованы как Hvmyb10 и Tamyb10 соответственно [60, 132].
Для сорго S. bicolor было известно несколько локусов, связанных с накоплением проантоцианидинов в разных слоях зерновки. У генотипов с доминантными аллелями генов в локусах B1 и B2 проантоцианидины синтезируются в семенной кожуре [133, 134]. Доминантные аллели B1, B2 и S обусловливают накопление проантоцианидинов в перикарпе и в семенной кожуре [135]. У сорго цвет перикарпа – красный, желтый или белый – определяется локусами R и Y [135]. Доминантный ген Y придает желтый цвет перикарпу; при потере его функции зерно становится белым. Доминантный ген R отвечает за красную окраску перикарпа, но только в присутствии доминантного Y [136].
Позже у сорго были выявлены гены Y1 и Tan1, расположенные на хромосомах 1 и 4 соответственно. Y1 кодирует фактор транскрипции Myb и контролирует ранние гены в пути синтеза флавоноидов; потеря функции Y1 приводит к появлению белой окраски [137, 138]. Также известно, что генотипы сорго с функциональным аллелем гена Y1 синтезируют красные пигменты флаван-4-олы и флобафены (см. ниже) [138, 139]. Tan1 кодирует белок WD40 и может регулировать ранние и поздние гены синтеза антоцианов и проантоцианидинов [140, 141]. Дополнительно были выявлены кандидаты других генов синтеза флавоноидов в зерне, но их функции нужно подтвердить [142]. Таким образом, для сорго путь биосинтеза проантоцианидинов к настоящему моменту является наиболее хорошо изученным.
3-ДЕЗОКСИАНТОЦИАНИДИНЫ
С молекулярной точки зрения 3-дезоксиантоцианидины – это антоцианидины без гидроксильной группы в положении 3 С-кольца (рис. 2). 3-Дезоксиантоцианидины являются редким типом антоцианидинов, которые синтезируются в мхах, папоротниках и некоторых цветковых растениях и придают растениям оранжево-красный и сине-фиолетовый цвет. Подобно антоцианидинам, 3-дезоксиантоцианидины встречаются в виде агликонов, гликозидов и гидроксилированных производных [17, 143]. Однако в основном 3-дезоксиантоцианидины обнаруживаются в растительных тканях в форме агликона, тогда как антоцианидины в основном существуют в форме гликозида (антоцианы) и конъюгированы с другими молекулами [144].
Отсутствие гидроксильной группы в положении С-3 придает 3-дезоксиантоцианидинам уникальные свойства. Они являются мощными природными антиоксидантами с целым рядом потенциальных преимуществ для здоровья, включая профилактику рака [144].
3-Дезоксиантоцианидины, синтезируемые сорго S. bicolor – желтый апигенинидин и оранжевый лютеолинидин, служащие в роли фитоалексинов, – являются уникальными молекулами для злаков [62, 145–147]. Так, зерно сорго является единственным известным источником 3-дезоксиантоцианидинов в семействе Poaceae, за исключением вегетативных тканей кукурузы (Z. mays) и сахарного тростника (Saccharum sp.) [148, 149].
Гены биосинтеза 3-дезоксиантоцианидинов в перикарпе
С молекулярной точки зрения в сорго биосинтез 3-дезоксиантоцианидинов регулируется геном Y1, кодирующим белок типа Myb (см. выше). Ген Y1 высокогомологичен Myb-гену кукурузы P1, контролирующему синтез 3-дезоксиантоцианинов и флобафенов в растении (см. ниже) [150]. Генотипы сорго с функциональным аллелем гена Y1 способны к биосинтезу желтых 3-дезоксиантоцианидинов и красных флобафенов (см. ниже) [139, 151]. Кроме того, в геноме сорго были выявлены ключевые структурные гены, необходимые для синтеза 3-дезоксиантоцианидинов в вегетативных органах: гены DFR3 и FNR (флаванон-4-редуктаза) контролируют появление пигментации листьев сорго при патогенной инфекции [152, 153], ген F3'Н2 вовлечен в патоген-специфический синтез 3-дезоксиантоцианидинов в мезокотиле и листьях [154, 155]. Участие этих генов в синтезе 3-дезоксиантоцианидинов в зерне пока не подтвердилось.
ФЛОБАФЕНЫ
Флобафены (полимеры бесцветных флаван-4-олов или 3-деоксифлавоноидов) являются нерастворимыми в воде пигментами, отвечающими за красный цвет перикарпа у некоторых однодольных растений (рис. 2) [40]. Флобафены играют важную роль в устойчивости растений к различным патогенным микроорганизмам [156, 157]. Однако в отличие от 3-дезоксиантоцианидинов в настоящее время нет данных о биологической активности флобафенов с точки зрения пользы для здоровья человека.
Благодаря своему структурному сходству с 3‑дезоксиантоцианидинами регуляция биосинтеза флобафенов в растениях опосредуется схожим набором генов [151]. Однако модельное растение Arabidopsis thaliana (L.) Heynh. не способно к синтезу флобафенов, поэтому изучение пути биосинтеза данных пигментов, в отличие от многих других классов флавоноидов, происходит на таких растениях, как кукуруза и сорго.
Гены биосинтеза флобафенов в перикарпе и семенной оболочке
У кукурузы Z. mays синтез флобафенов в перикарпе зерна осуществляется благодаря экспрессии Myb-кодирущего гена P1 (хромосома 1), который контролирует работу структурных генов CHS (халконсинтаза), CHI (халконфлаванонизомераза), F3'H, DFR и FLS (флавонолсинтаза) в ограниченном наборе тканей [150, 158–162]. Помимо этого, в геноме кукурузы существует транс-модификатор Ufo1 (хромосома 10), изменяющий экспрессию гена P1 так, что синтез флобафенов запускается и в вегетативных тканях, которые обычно не накапливают значительных количеств пигмента, в результате чего появляются растения с оранжевой или красной окраской листьев и стеблей [163, 164].
В сорго S. bicolor соответствующий Myb-ген Y1, вовлеченный в синтез проантоцианидинов, 3‑дезоксиантоцианидинов (см. выше) и флобафенов, в перикарпе регулирует экспрессию структурных генов пути биосинтеза данных пигментов: CHS, CHI и DFR [138, 139, 151, 165].
Таким образом, к настоящему моменту мы обладаем недостаточным количеством данных, касающихся биосинтеза ряда флавоноидов в зерновках злаковых растений. Наиболее хорошо изученным и полно охарактеризованным среди флавоноидов является путь синтеза антоцианов. Наименее исследованными в этом плане остаются пути биосинтеза уникальных флобафенов и 3-дезоксиантоцианидинов. Также до сих пор неясно, вовлечены ли факторы транскрипции bHLH и WD40 в регуляцию синтеза пигментов флобафенов и 3‑дезоксиантоцианидинов наравне с факторами Myb. Однако благодаря развитию методов генетики и биоинформатики в скором времени удастся существенно продвинуться в изучении путей биосинтеза данных пигментов в семействе Poaceae.
ЗАКЛЮЧЕНИЕ
Синтез флавоноидных пигментов у представителей большинства культур семейства Poaceae можно наблюдать в различных слоях зерна. Однако некоторые признаки являются уникальными, такие как синтез 3-дезоксиантоцианидинов в перикарпе у сорго или отсутствие антоцианов в алейроновом слое у некоторых представителей трибы Triticeae (например, у мягкой пшеницы T. aestivum). Четко прослеживается, что в основе фенотипической изменчивости большинства видов семейства Poaceae лежат мутации как в регуляторных, так и в структурных генах биосинтеза флавоноидов, тогда как изменчивость по признакам окраски у пшеницы T. aestivum базируется на аллельном разнообразии регуляторных генов Myb и bHLH. Вероятно, дуплицированные копии структурных генов, возникшие в геноме пшеницы вследствие аллополиплоидизации, не успели претерпеть существенных изменений (неофункциолизацию или псевдогенизацию, что имеет место у палеополиплоидов, например кукурузы Z. mays), тогда как регуляторные гены успели дивергировать у диплоидных доноров пшеницы до аллополиплоидизации [166]. Сам факт участия структурных генов биосинтеза флавоноидных пигментов в изменчивости диплоидных злаков на уровне фенотипа, несмотря на большую степень консервативности структурных генов по сравнению с регуляторными, объясняется, вероятно, меньшим давлением отбора на эти гены в сравнении с видами растений, привлекающих насекомых для перекрестного опыления [167].
Статья подготовлена в рамках государственного задания ВИР согласно тематическому плану НИР по теме № 0481-2019-0001 “Геномные и постгеномные технологии для выявления новых генетических маркеров селекционно значимых свойств и новых аллельных вариантов хозяйственно ценных генов в генофонде культурных растений и их диких родичей”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Список литературы
Kellogg E.A. Flowering plants. Monocots. Poaceae // The Families and Genera of Vascular Plants / Ed. Kubitski K. Cham: Springer Intern., 2015. V. 13. 416 p. https://doi.org/10.1007/978-3-319-15332-2
Soreng R.J., Peterson P.M., Romaschenko K. et al. A worldwide phylogenetic classification of the Poaceae (Gramineae) // J. Syst. Evol. 2015. V. 53. № 2. P. 117–137. https://doi.org/10.1111/jse.12150
Soreng R.J., Peterson P.M., Romaschenko K. et al. A worldwide phylogenetic classification of the Poaceae (Gramineae) II: An update and a comparison of two 2015 classifications // J. Syst. Evol. 2017. V. 55. № 4. P. 259–290. https://doi.org/10.1111/jse.12262
Strömberg C.A.E. Evolution of grasses and grassland ecosystems // Annu. Rev. Earth Planet. Sci. 2011. V. 39. P. 517–544. https://doi.org/10.1146/annurev-earth-040809-152402
Tzvelev N.N. The system of grasses (Poaceae) and their evolution // Bot. Rev. 1989. V. 55. № 3. P. 141–203. https://doi.org/10.1007/BF02858328
The Plant List (2013). Version 1.1. Publ. Internet (accessed 1st January) [Electronic resource]. URL: http://www.theplantlist.org/ (дата обращения: 01.03.2020).
Gibson D.J. Grasses and Grassland Ecology. Oxford, UK: Oxford Univ. Press, 2009. 315 p. https://doi.org/10.1093/aob/mcp219
Hodkinson T.R. Evolution and taxonomy of the grasses (Poaceae): A model family for the study of species-rich groups // Annu. Plant Rev. 2018. V. 1. № 1. https://doi.org/10.1002/9781119312994.apr0622
Kellogg E.A. Evolutionary history of the grasses // Plant Physiol. 2001. V. 125. № 3. P. 1198–1205. https://doi.org/10.1104/pp.125.3.1198
Hilu K.W. Phylogenetics and chromosomal evolution in the Poaceae (grasses) // Aust. J. Bot. 2004. V. 52. № 1. P. 13–22. https://doi.org/10.1071/BT03103
de Wet J.M.J. Chromosome numbers of a few South African grasses // Cytologia (Tokyo). 1954. V. 19. № 2–3. P. 97–103. https://doi.org/10.1508/cytologia.19.97
Plant DNA C-values Database. Release 7.1, April 2019 [Electronic resource]. URL: https://cvalues.science.kew.org/ (дата обращения: 01.03.2020).
Murat F., Xu J.H., Tannier E. et al. Ancestral grass karyotype reconstruction unravels new mechanisms of genome shuffling as a source of plant evolution // Genome Res. 2010. V. 20. P. 1545–1557. https://doi.org/10.1101/gr.109744.110
Wang Z., Wang J., Pan Y. et al. Reconstruction of evolutionary trajectories of chromosomes unraveled independent genomic repatterning between Triticeae and Brachypodium // BMC Genomics. 2019. V. 20. P. 180. https://doi.org/10.1186/s12864-019-5566-8
Вавилов Н.И. Закон гомологических рядов в наследственной изменчивости. Сельхозгиз, 1935. 56 c.
Abdel-Aal E.S.M., Young J.C., Rabalski I. Anthocyanin composition in black, blue, pink, purple, and red cereal grains // J. Agric. Food Chem. 2006. V. 54. № 13. P. 4696–4704. https://doi.org/10.1021/jf0606609
Grotewold E. The Science of Flavonoids. N.Y.: Springer, 2006. 273 p. https://doi.org/10.1007/978-0-387-28822-2
Lepiniec L., Debeaujon I., Routaboul J.M. et al. Genetics and biochemistry of seed flavonoids // Annu. Rev. Plant Biol. 2006. V. 57. P. 405–430. https://doi.org/10.1146/annurev.arplant.57.032905.105252
Landi M., Tattini M., Gould K.S. Multiple functional roles of anthocyanins in plant-environment interactions // Environ. Exp. Bot. 2015. V. 119. P. 4–17. https://doi.org/10.1016/j.envexpbot.2015.05.012
Gould K.S. Nature’s Swiss army knife: The diverse protective roles of anthocyanins in leaves // J. Biomed. Biotechnol. 2004. V. 2004. № 5. P. 314–320. https://doi.org/10.1155/S1110724304406147
Lee D.W., Gould K.S. Why leaves turn red: pigments called anthocyanins probably protect leaves from light damage by direct shielding and by scavenging free radicals // Am. Sci. 2002. V. 90. № 6. P. 524–531.
Polonskiy V., Loskutov I., Sumina A. Biological role and health benefits of antioxidant compounds in cereals // Biol. Commun. 2020. V. 65. № 1. P. 53–67. https://doi.org/10.21638/spbu03.2020.105
Sánchez-Moreiras A.M., Weiss O., Roger M.R. Allelopathic evidence in the Poaceae // Bot. Rev. 2004. V. 69. № 3. P. 300–319. https://doi.org/10.1663/0006-8101(2003)069[0300:AE ITP]2.0.CO;2
Nemzer B., Lin Y., Huang D. Antioxidants in sprouts of grains // Sprouted Grains. Elsevier Inc. 2019. P. 55–68. https://doi.org/10.1016/B978-0-12-811525-1.00003-8
Khoo H.E., Azlan A., Tang S.T. et al. Anthocyanidins and anthocyanins: colored pigments as food, pharmaceutical ingredients, and the potential health benefits // Food Nutr. Res. 2017. V. 61. № 1. P. 1361779. https://doi.org/10.1080/16546628.2017.1361779
Abdel-Aal E.S.M., Hucl P., Rabalski I. Compositional and antioxidant properties of anthocyanin-rich products prepared from purple wheat // Food Chem. 2018. V. 254. P. 13–19. https://doi.org/10.1016/j.foodchem.2018.01.170
Shipp J., Abdel-Aal E. Food applications and physiological effects of anthocyanins as functional food ingredients // Open Food Sci. J. 2010. V. 4. P. 7–22. https://doi.org/10.2174/1874256401004010007
Yoshinaga K., Yakahashi K.Y.K. Liquor with pigments of red rice // J. Brew. Soc. Japan. 1986. V. 81. P. 337–342.
Yang Z., Zhai W. Identification and antioxidant activity of anthocyanins extracted from the seed and cob of purple corn (Zea mays L.) // Innov. Food Sci. Emerg. Technol. 2010. V. 11. № 1. P. 169–176. https://doi.org/10.1016/j.ifset.2009.08.012
Garg M., Chawla M., Chunduri V. et al. Transfer of grain colors to elite wheat cultivars and their characterization // J. Cereal Sci. 2016. V. 71. P. 138–144. https://doi.org/10.1016/j.jcs.2016.08.004
Khlestkina E.K., Shoeva O.Y., Gordeeva E.I. et al. Anthocyanins in wheat grain: genetic control, health benefit and bread-making quality // Current Challenges in Plant Genetics, Genomics, Bioinformatics, and Biotechnology. 2019. V. 24. P. 15–18. https://doi.org/10.18699/ICG-PlantGen2019-02
Usenko N.I., Khlestkina E.K., Asavasanti S. et al. Possibilities of enriching food products with anthocyanins by using new forms of cereals // Foods Raw Mater. 2018. V. 6. № 1. P. 128–135. https://doi.org/10.21603/2308-4057-2018-1-128-135
Zong Y., Xi X., Li S. et al. Allelic variation and transcriptional isoforms of wheat TaMYC1 gene regulating anthocyanin synthesis in pericarp // Front. Plant Sci. 2017. V. 8. P. 1–12. https://doi.org/10.3389/fpls.2017.01645
Gordeeva E.I., Shoeva O.Y., Khlestkina E.K. Marker-assisted development of bread wheat near-isogenic lines carrying various combinations of purple pericarp (Pp) alleles // Euphytica. 2014. № 203. P. 469–476. https://doi.org/10.1007/s10681-014-1317-8
Khlestkina E.K., Usenko N.I., Gordeeva E.I. et al. Evaluation of wheat products with high flavonoid content: justification of importance of marker-assisted development and production of flavonoid-rich wheat cultivars // Vavilovskii Zhurnal Genetiki i Selektsii. 2017. V. 21. № 5. P. 545–553. https://doi.org/10.18699/VJ17.25-o
Gordeeva E.I., Glagoleva A.Y., Kukoeva T.V. et al. Purple-grained barley (Hordeum vulgare L.): Marker-assisted development of NILs for investigating peculiarities of the anthocyanin biosynthesis regulatory network // BMC Plant Biol. 2019. V. 19. № 52. https://doi.org/10.1186/s12870-019-1638-9
Gordeeva E., Badaeva E., Yudina R. et al. Marker-assisted development of a blue-grained substitution line carrying the Thinopyrum ponticum chromosome 4Th(4D) in the spring bread wheat Saratovskaya 29 background // Agronomy. 2019. V. 9. № 11. https://doi.org/10.3390/agronomy9110723
Syed Jaafar S.N., Baron J., Siebenhandl-Ehn S. et al. Increased anthocyanin content in purple pericarp × × blue aleurone wheat crosses // Plant Breed. 2013. V. 132. № 6. P. 546–552. https://doi.org/10.1111/pbr.12090
Knievel D.C., Abdel-Aal E.S.M., Rabalski I. et al. Grain color development and the inheritance of high anthocyanin blue aleurone and purple pericarp in spring wheat (Triticum aestivum L.) // J. Cereal Sci. 2009. V. 50. № 1. P. 113–120. https://doi.org/10.1016/j.jcs.2009.03.007
Winkel-Shirley B. Flavonoid biosynthesis. A colorful model for genetics, biochemistry, cell biology, and biotechnology // Plant Physiol. 2001. V. 126. № 2. P. 485–493. https://doi.org/10.1104/pp.126.2.485
Adzhieva V.F., Babak O.G., Shoeva O.Y. et al. Molecular genetic mechanisms of the development of fruit and seed coloration in plants // Russ. J. Genet. Appl. Res. 2016. V. 6. № 5. P. 537–552. https://doi.org/10.1134/S2079059716050026
Kushnak G.D. Utilizing linkages of genetic male sterile and aleurone color genes in hybrid barley (Hordeum vulgare L.) systems. Montana State Univ., 1974.
Khlestkina E.K. Genes determining the coloration of different organs in wheat // Russ. J. Genet. Appl. Res. 2013. V. 3. № 1. P. 54–65. https://doi.org/10.1134/S2079059713010085
Ficco D.B.M., De Simone V., Colecchia S.A. et al. Genetic variability in anthocyanin composition and nutritional properties of blue, purple, and red bread (Triticum aestivum L.) and durum (Triticum turgidum L. ssp. turgidum convar. durum) Wheats // J. Agr. Food Chem. 2014. V. 62. № 34. P. 8686–8695. https://doi.org/10.1021/jf5003683
Sytar O., Bośko P., Živčák M. et al. Bioactive phytochemicals and antioxidant properties of the grains and sprouts of colored wheat genotypes // Molecules. 2018. V. 23. № 9. P. 2282. https://doi.org/10.3390/molecules23092282
Dangles O., Fenger J.A. The chemical reactivity of anthocyanins and its consequences in food science and nutrition // Molecules. 2018. V. 23. № 8. P. 1970. https://doi.org/10.3390/molecules23081970
Goufo P., Trindade H. Rice antioxidants: phenolic acids, flavonoids, anthocyanins, proanthocyanidins, tocopherols, tocotrienols, γ-oryzanol, and phytic acid // Food Sci. Nutr. 2014. V. 2. № 2. P. 75–104. https://doi.org/10.1002/fsn3.86
Voylokov A.V., Lykholay A.N., Smirnov V.G. Genetic control of anthocyanin coloration in rye // Russ. J. Genet. Appl. Res. 2015. V. 5. № 3. P. 262–267. https://doi.org/10.1134/S207905971503020X
Pihlava J.M., Hellström J., Kurtelius T., Mattila P. Flavonoids, anthocyanins, phenolamides, benzoxazinoids, lignans and alkylresorcinols in rye (Secale cereale) and some rye products // J. Cereal Sci. 2018. V. 79. P. 183–192. https://doi.org/10.1016/j.jcs.2017.09.009
Dedio W., Hill R.D., Evans L.E. Anthocyanins in the pericarp and coleoptiles of purple wheat // Can. J. Plant Sci. 1972. V. 52. № 6. P. 981–983.
Zykin P.A., Andreeva E.A., Lykholay A.N. et al. Anthocyanin composition and content in rye plants with different grain color // Molecules. 2018. V. 23. № 4. P. 948. https://doi.org/10.3390/molecules23040948
Petroni K., Pilu R., Tonelli C. Anthocyanins in corn: a wealth of genes for human health // Planta. 2014. V. 240. № 5. P. 901–911. https://doi.org/10.1007/s00425-014-2131-1
Awika J.M., Rooney L.W., Waniska R.D. Anthocyanins from black sorghum and their antioxidant properties // Food Chem. 2005. V. 90. № 1–2. P. 293–301. https://doi.org/10.1016/j.foodchem.2004.03.058
Su X., Rhodes D.H., Xu J. et al. Phenotypic diversity of anthocyanins in sorghum accessions with various pericarp pigments // J. Nutr. Food Sci. 2017. V. 7. № 4. https://doi.org/10.4172/2155-9600.1000610
Kim M.-K., Kim H., Koh K. et al. Identification and quantification of anthocyanin pigments in colored rice // Nutri. Res. and Pract. 2008. V. 2. № 1. P. 46–49. https://doi.org/10.4162/nrp.2008.2.1.46
Dedio W., Hill R.D., Evans L.E. Anthocyanins in the pericarp and coleoptiles of purple-seeded rye // Can. J. Plant Sci. 1972. V. 52. № 6. P. 981–983.
Mullick D.B., Faris D.G., Brink V.C., Acheson R.M. Anthocyanins and anthocyanidins of the barley pericarp and aleurone tissues // Can. J. Plant Sci. 1958. V. 38. № 4. P. 445–456. https://doi.org/10.4141/cjps58-071
Zhu F. Anthocyanins in cereals: Composition and health effects // Food Res. Int. 2018. V. 109. P. 232–249. https://doi.org/10.1016/j.foodres.2018.04.015
Salinas Moreno Y., Sánchez G.S., Hernández D.R., Lobato N.R. Characterization of anthocyanin extracts from maize kernels // J. Chromatogr. Sci. 2005. V. 43. № 9. P. 483–487. https://doi.org/10.1093/chromsci/43.9.483
Шоева О.Ю., Стрыгина К.В., Хлесткина Е.К. Гены, контролирующие синтез флавоноидных и меланиновых пигментов ячменя // Вавиловский журн. генетики и селекции. 2018. Т. 22. № 3. С. 333–342. https://doi.org/10.18699/VJ18.369
Jackman R.L., Smith J.L. Anthocyanins and betalains // Natural Food Colorants / Eds Hendry G.A.F., Houghton J.D. Springer, Boston, MA, 1996. P. 244–309. https://doi.org/10.1007/978-1-4615-2155-6_8
Wu X., Prior R.L. Identification and characterization of anthocyanins by high-performance liquid chromatography-electrospray ionization-tandem mass spectrometry in common foods in the United States: Vegetables, nuts, and grains // J. Agric. Food Chem. 2005. V. 53. № 8. P. 3101–3113. https://doi.org/10.1021/jf0478861
Fossen T., Slimestad R., Andersen O.M. Anthocyanins from maize (Zea mays) and reed canarygrass (Phalaris arundinacea) // J. Agric. Food Chem. 2001. V. 49. № 5. P. 2318–2321. https://doi.org/10.1021/jf001399d
Abdel-Aal E.S.M., Hucl P. Composition and stability of anthocyanins in blue-grained wheat // J. Agric. Food Chem. 2003. V. 51. № 8. P. 2174–2180. https://doi.org/10.1021/jf021043x
Стрыгина К.В. Регуляция тканеспецифической экспрессии генов биосинтеза флавоноидов у видов трибы Triticeae. Новосибирск: ИЦиГ СО РАН, 2018. 157 с.
Dwivedi S.L., Upadhyaya H.D., Chung I.M. et al. Exploiting phenylpropanoid derivatives to enhance the nutraceutical values of cereals and legumes // Front. Plant Sci. 2016. V. 7. P. 763. https://doi.org/10.3389/fpls.2016.00763
Ford R.H. Inheritance of kernel color in corn: Explanations & investigations // Am. Biol. Teach. 2000. V. 62. № 3. P. 181–188. https://doi.org/10.2307/4450870
Rhoades M.M. The effect of varying gene dosage on aleurone colour in maize // J. Genet. 1936. V. 33. № 3. P. 347–354. https://doi.org/10.1007/BF02982890
Li T., Zhang W., Yang H. et al. Comparative transcriptome analysis reveals differentially expressed genes related to the tissue-specific accumulation of anthocyanins in pericarp and aleurone layer for maize // Sci. Rep. 2019. V. 9. № 1. P. 2485. https://doi.org/10.1038/s41598-018-37697-y
Chandler V.L., Radicella J.P., Robbins T.P. et al. Two regulatory genes of the maize anthocyanin pathway are homologous: isolation of B utilizing R genomic sequences. // Plant Cell. 1989. V. 1. № 12. P. 1175–1183. https://doi.org/10.1105/tpc.1.12.1175
Dooner H.K., Robbins T.P., Jorgensen R.A. Genetic and developmental control of anthocyanin biosynthesis // Annu. Rev. Genet. 1991. V. 25. P. 173–199.
Pilu R., Piazza P., Petroni K. et al. Pl-Bol3, a complex allele of the anthocyanin regulatory Pl1 locus that arose in a naturally occurring maize population // Plant J. 2003. V. 36. № 4. P. 510–521. https://doi.org/10.1046/j.1365-313X.2003.01898.x
Carey C.C. Mutations in the pale aleurone color1 regulatory gene of the Zea mays anthocyanin pathway have distinct phenotypes relative to the functionally similar TRANSPARENT TESTA GLABRA1 gene in Arabidopsis thaliana // Plant Cell. 2004. V. 16. № 2. P. 450–464. https://doi.org/10.1105/tpc.018796
Selinger D.A., Chandler V.L. A mutation in the pale aleurone color1 gene identifies a novel regulator of the maize anthocyanin pathway // Plant Cell. 1999. V. 11. № 1. P. 5–14. https://doi.org/10.1105/tpc.11.1.5
Maeda H., Yamaguchi T., Omoteno M. et al. Genetic dissection of black grain rice by the development of a near isogenic line // Breed. Sci. 2014. V. 64. № 2. P. 134–141. https://doi.org/10.1270/jsbbs.64.134
Shih C.H., Chu H., Tang L.K. et al. Functional characterization of key structural genes in rice flavonoid biosynthesis // Planta. 2008. V. 228. P. 1043–1054. https://doi.org/10.1007/s00425-008-0806-1
Oikawa T., Maeda H., Oguchi T. et al. The birth of a black rice gene and its local spread by introgression // Plant Cell. 2015. V. 27. № 9. P. 2401–2414. https://doi.org/10.1105/tpc.15.00310
Rahman M.M., Lee K.E., Kang S.G. Allelic gene interaction and anthocyanin biosynthesis of purple pericarp trait for yield improvement in black rice // J. Life Sci. 2016. V. 26. № 6. P. 727–736. https://doi.org/10.5352/JLS.2016.26.6.727
Sun X., Zhang Z., Chen C. et al. The C-S-A gene system regulates hull pigmentation and reveals evolution of anthocyanin biosynthesis pathway in rice // J. Exp. Bot. 2018. V. 69. № 7. P. 1485–1498. https://doi.org/10.1093/jxb/ery001
Cockram J., White J., Zuluaga D.L. et al. Genome-wide association mapping to candidate polymorphism resolution in the unsequenced barley genome // PNAS. 2010. V. 107. № 50. P. 21611–21616. https://doi.org/10.1073/pnas.1010179107
Shoeva O.Y., Mock H.P., Kukoeva T.V. et al. Regulation of the flavonoid biosynthesis pathway genes in purple and black grains of Hordeum vulgare // PLoS One. 2016. V. 11. № 10. P. e0163782. https://doi.org/10.1371/journal.pone.0163782
Himi E., Taketa S. Isolation of candidate genes for the barley Ant1 and wheat Rc genes controlling anthocyanin pigmentation in different vegetative tissues // Mol. Genet. Genomics. 2015. V. 290. P. 1287–1298. https://doi.org/10.1007/s00438-015-0991-0
Shoeva O., Gordeeva E., Khlestkina E. The regulation of anthocyanin synthesis in the wheat pericarp // Molecules. 2014. V. 19. № 12. P. 20266–20279. https://doi.org/10.3390/molecules191220266
Jiang W., Liu T., Nan W. et al. Two transcription factors TaPpm1 and TaPpb1 co-regulate anthocyanin biosynthesis in purple pericarps of wheat // J. Exp. Bot. 2018. V. 69. № 10. P. 2555–2567.https://doi.org/10.1093/jxb/ery101
Strygina K.V., Börner A., Khlestkina E.K. Identification and characterization of regulatory network components for anthocyanin synthesis in barley aleurone // BMC Plant Biol. 2017. V. 17. P. 184. https://doi.org/10.1186/s12870-017-1122-3
Strygina K.V., Khlestkina E.K. Structural and functional organization and evolution of the WD40 genes involved in the regulation of flavonoid biosynthesis in the Triticeae tribe // Russian J. Genet. 2019. V. 55. № 11. P. 1398–1405. https://doi.org/10.1134/S1022795419110152
Lykholay A.N., Vladimirov I.A., Andreeva E.A. et al. Genetics of anthocyaninless rye // Rus. J. Genet. 2014. V. 50. P. 1102–1106. https://doi.org/10.1134/S1022795414100081
Федоров В.С. Генетика ржи (Secale cereale L.). Наследование антоциановой окраски, воскового налета и ветвистоколосости у ржи // Исследования по генетике. 1964. Т. 2. С. 100–110.
Dumon A.G. Contribution à la génétique et à lamélioration du seigle (Secale cereale L.) // Agricultura (Louvain). 1947. V. 45. P. 213–223.
Федоров В.С. Генетика ржи (Secale cereale L.). Ксении // Исследования по генетике. 1961. Т. 1. С. 116–121.
Watkins R., White W.J. The inheritance of anthocyanins in rye (Secale cereale L.) // Can. J. Genet. Cytol. 1964. V. 6. № 4. P. 403–410. https://doi.org/10.1139/g64-051
Войлоков А.В., Соснихина С.П., Тихенко Н.Д. и др. Петергофская коллекция ржи и ее использование в генетических исследованиях // Экол. генетика. 2018. Т. 16. № 2. С. 40–49. https://doi.org/10.17816/ecogen16240-49
Finch R.A., Simpson E. New colours and complementary colour genes in barley // Z. Pflanzenzücht. 1978. Bd 81. № 1. S. 40–53.
Strygina K.V., Khlestkina E.K. Structural and functional divergence of the Mpc1 genes in wheat and barley // BMC Evol. Biol. 2019. V. 19. P. 45. https://doi.org/10.1186/s12862-019-1378-3
Vikhorev A.V., Strygina K.V., Khlestkina E.K. Duplicated flavonoid 3'-hydroxylase and flavonoid 3',5'-hydroxylase genes in barley genome // PeerJ. 2019. V. 7. P. e6266. https://doi.org/10.7717/peerj.6266
Zeven A.C. Wheats with purple and blue grains: a review // Euphytica. 1991. V. 56. P. 243–258. https://doi.org/10.1007/BF00042371
Liu X., Feng Z., Liang D. et al. Development, identification, and characterization of blue-grained wheat-Triticum boeoticum substitution lines // J. Appl. Genet. 2020. V. 61. P. 169–177. https://doi.org/10.1007/s13353-020-00553-9
Ficco D.B.M., Mastrangelo A.M., Trono D. et al. The colours of durum wheat: a review // Crop Pasture Sci. 2014. V. 65. P. 1–15. https://doi.org/10.1071/CP13293
Zhao S., Xi X., Zong Y. et al. Overexpression of THMYC4E enhances anthocyanin biosynthesis in common wheat // Int. J. Mol. Sci. 2020. V. 21. № 1. P. 137. https://doi.org/10.3390/ijms21010137
Li N., Li S., Zhang K. et al. ThMYC4E, candidate blue aleurone 1 gene controlling the associated trait in Triticum aestivum // PLoS One. 2017. V. 12. № 7. P. e0181116. https://doi.org/10.1371/journal.pone.0181116
Liu L., Luo Q., Li H. et al. Physical mapping of the blue-grained gene from Thinopyrum ponticum chromosome 4Ag and development of blue-grain-related molecular markers and a FISH probe based on SLAF-seq technology // Theor. Appl. Genet. 2018. V. 131. № 11. P. 2359–2370. https://doi.org/10.1007/s00122-018-3158-7
Lachman J., Martinek P., Kotíková Z. et al. Genetics and chemistry of pigments in wheat grain – A review // J. Cereal Sci. 2017. V. 74. P. 145–154. https://doi.org/10.1016/j.jcs.2017.02.007
Li J., Lang T., Li B. et al. Introduction of Thinopyrum intermedium ssp. trichophorum chromosomes to wheat by trigeneric hybridization involving Triticum, Secale and Thinopyrum genera // Planta. 2017. V. 245. № 6. P. 1121–1135. https://doi.org/10.1007/s00425-017-2669-9
Strygina K.V., Khlestkina E.K. MYC gene family in cereals: Transformations during evolution of hexaploid bread wheat and its relatives // Mol. Biol. 2017. V. 51. P. 674–680. https://doi.org/10.1134/S0026893317050181
Strygina K.V., Khlestkina E.K. Myc-like transcriptional factors in wheat: Structural and functional organization of the subfamily I members // BMC Plant Biol. 2019. V. 19. P. 50. https://doi.org/10.1186/s12870-019-1639-8
Jia Y., Selva C., Zhang Y. et al. Uncovering the evolutionary origin of blue anthocyanins in cereal grains // Plant J. 2019. V. 101. № 5. P. 1057–1074. https://doi.org/10.1111/tpj.14557
Cone K.C., Cocciolone S.M., Burr F.A. et al. Maize anthocyanin regulatory gene pl is a duplicate of c1 that functions in the plant // Plant Cell. 1993. V. 5. № 12. P. 1795–1805. https://doi.org/10.1105/tpc.5.12.1795
Sharma M., Cortes-Cruz M., Ahern K.R. et al. Identification of the Pr1 gene product completes the anthocyanin biosynthesis pathway of maize // Genetics. 2011. V. 188. № 1. P. 69–79. https://doi.org/10.1534/genetics.110.126136
De Bruyne T., Pieters L., Deelstra H. et al. Condensed vegetable tannins: Biodiversity in structure and biological activities // Biochem. Syst. Ecol. 1999. V. 27. № 4. P. 445–459. https://doi.org/10.1016/S0305-1978(98)00101-X
He F., Pan Q.H., Shi Y. et al. Biosynthesis and genetic regulation of proanthocyanidins in plants // Molecules. 2008. V. 13. № 10. P. 2674–2703. https://doi.org/10.3390/molecules13102674
Rauf A., Imran M., Abu-Izneid T. et al. Proanthocyanidins: A comprehensive review // Biomed. Pharmacother. 2019. V. 116. P. 108999. https://doi.org/10.1016/j.biopha.2019.108999
Gunaratne A., Wu K., Li D. et al. Antioxidant activity and nutritional quality of traditional red-grained rice varieties containing proanthocyanidins // Food Chem. 2013. V. 138. № 2–3. P. 1153–1161. https://doi.org/10.1016/j.foodchem.2012.11.129
Marles M.A.S., Ray H., Gruber M.Y. New perspectives on proanthocyanidin biochemistry and molecular regulation // Phytochemistry. 2003. V. 64. № 2. P. 367–383. https://doi.org/10.1016/S0031-9422(03)00377-7
Gonzalez A., Zhao M., Leavitt J.M. et al. Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seedlings // Plant J. 2008. V. 53. № 5. P. 814–827. https://doi.org/10.1111/j.1365-313X.2007.03373.x
Zhu F. Proanthocyanidins in cereals and pseudocereals // Crit. Rev. Food Sci. Nutr. 2019. V. 59. № 10. P. 1521–1533. https://doi.org/10.1080/10408398.2017.1418284
Aastrup S., Outtrup H., Erdal K. Location of the proanthocyanidins in the barley grain // Carlsberg Res. Commun. 1984. V. 49. P. 105–109. https://doi.org/10.1007/BF02913969
Fraser K., Collette V., Hancock K.R. Characterization of proanthocyanidins from Seeds of perennial ryegrass (Lolium perenne L.) and tall fescue (Festuca arundinacea) by liquid chromatography-mass spectrometry // J. Agric. Food Chem. 2016. V. 64. № 35. P. 6676–6684. https://doi.org/10.1021/acs.jafc.6b02563
Mccallum J.A., Walker J.R.L. Proanthocyanidins in wheat bran // Cereal Chem. 1990. V. 67. № 3. P. 282–285.
Dykes L., Rooney L.W. Phenolic compounds in cereal grains and their health benefits // Cereal Food World. 2007. V. 52. № 3. P. 105–111. https://doi.org/10.1094/CFW-52-3-0105
Gu L., Kelm M.A., Hammerstone J.F. et al. Screening of foods containing proanthocyanidins and their structural characterization using LC-MS/MS and thiolytic degradation // J. Agric. Food Chem. 2003. V. 51. № 25. P. 7513–7521. https://doi.org/10.1021/jf034815d
Kristiansen K.N. Biosynthesis of proanthocyanidins in barley: genetic control of the conversion of dihydroquercetin to catechin and procyanidins // Carlsberg Res. Commun. 1984. V. 49. № 5. P. 503. https://doi.org/10.1007/BF02907552
Reddy V.S., Dash S., Reddy A.R. Anthocyanin pathway in rice (Oryza sativa L.): identification of a mutant showing dominant inhibition of anthocyanins in leaf and accumulation of proanthocyanidins in pericarp // Theor. Appl. Genet. 1995. V. 91. P. 301–312. https://doi.org/10.1007/BF00220892
Limtrakul (Dejkriengkraikul) P., Semmarath W., Mapoung S. Anthocyanins and proanthocyanidins in natural pigmented rice and their bioactivities // Phytochemicals in Human Health. 2019. https://doi.org/10.5772/intechopen.86962
Hosoda K., Sasahara H., Matsushita K. et al. Anthocyanin and proanthocyanidin contents, antioxidant activity, and in situ degradability of black and red rice grains // Asian-Australas. J. Anim. Sci. 2018. V. 31. № 8. P. 1213–1220. https://doi.org/10.5713/ajas.17.0655
Furukawa T., Maekawa M., Oki T. et al. The Rc and Rd genes are involved in proanthocyanidin synthesis in rice pericarp // Plant J. 2007. V. 49. № 1. P. 91–102. https://doi.org/10.1111/j.1365-313X.2006.02958.x
Sweeney M.T., Thomson M.J., Pfeil B.E. et al. Caught red-handed: Rc encodes a basic helix-loop-helix protein conditioning red pericarp in rice // Plant Cell. 2006. V. 18. P. 283–294. https://doi.org/10.1105/tpc.105.038430
Rahman M.M., Naher M.S., Sikdar M.S.I. et al. Genetic characterization of red pericarp trait in rice // JST. 2015. V. 13. P. 118–121.
Kohyama N., Chono M., Nakagawa H. et al. Flavonoid compounds related to seed coat color of wheat // Biosci. Biotech. Biochem. 2017. V. 81. № 11. P. 2112–2118. https://doi.org/10.1080/09168451.2017.1373589
Himi E., Noda K. Red grain colour gene (R) of wheat is a Myb-type transcription factor // Euphytica. 2005. V. 143. P. 239–242. https://doi.org/10.1007/s10681-005-7854-4
Himi E., Taketa S. Promising target locus for attaining anthocyanin/proanthocyanidin-free plants without pleiotropic reduction of grain dormancy // Genome. 2015. V. 58. P. 43–53. https://doi.org/10.1139/gen-2014-0189
Jende-Strid B., Lundqvist U. Diallelic tests of anthocyanin-deficient mutants // Barley Genet. Newsl. 1978. № 8. P. 57–59.
Himi E., Maekawa M., Miura H., Noda K. Development of PCR markers for Tamyb10 related to R-1, red grain color gene in wheat // Theor. Appl. Genet. 2011. V. 122. № 8. P. 1561–1576. https://doi.org/10.1007/s00122-011-1555-2
Dykes L., Rooney L.W. Sorghum and millet phenols and antioxidants // J. Cereal Sci. 2006. V. 44. № 3. P. 236–251. https://doi.org/10.1016/j.jcs.2006.06.007
Hahn D.H., Rooney L.W., Earp C.F. Tannins and phenols of sorghum // Cereal Food World. 1984. V. 29. № 12. P. 776–779.
Rhodes D.H., Hoffmann L., Jr., Rooney W.L. et al. Genome-wide association study of grain polyphenol concentrations in global sorghum [Sorghum bicolor (L.) Moench] germplasm // J. Agric. Food Chem. 2014. V. 62. № 45. P. 10916–10927. https://doi.org/10.1021/jf503651t
Doggett H. Sorghum (2nd ed.), tropical agricultural series // Longmans. 1988. P. 512. https://doi.org/10.1017/S002185960008477X
Morris G.P., Rhodes D.H., Brenton Z. et al. Dissecting genome-wide association signals for loss-of-function phenotypes in sorghum flavonoid pigmentation traits // G3. 2013. V. 3. № 11. P. 2085–2094. https://doi.org/10.1534/g3.113.008417
Boddu J., Svabek C., Ibraheem F. et al. Characterization of a deletion allele of a sorghum Myb gene yellow seed1 showing loss of 3-deoxyflavonoids // Plant Sci. 2005. V. 169. № 3. P. 542–552. https://doi.org/10.1016/j.plantsci.2005.05.007
Chopra S., Gevens A., Svabek C. et al. Excision of the Candystripe1 transposon from a hyper-mutable Y1-cs allele shows that the sorghumY1 gene controls the biosynthesis of both 3-deoxyanthocyanidin phytoalexins and phlobaphene pigments // Physiol. Mol. Plant Pathol. 2002. V. 60. № 6. P. 321–330. https://doi.org/10.1006/pmpp.2002.0411
Wu Y., Li X., Xiang W. et al. Presence of tannins in sorghum grains is conditioned by different natural alleles of Tannin1 // PNAS. 2012. V. 109. № 26. P. 10281–10286. https://doi.org/10.1073/pnas.1201700109
Kawahigashi H., Nonaka E., Mizuno H. et al. Classification of genotypes of leaf phenotype (P/tan) and seed phenotype (Y1 and Tan1) in tan sorghum (Sorghum bicolor) // Plant Breed. 2016. V. 135. № 6. P. 683–690. https://doi.org/10.1111/pbr.12426
Habyarimana E., Dall’Agata M., De Franceschi P. et al. Genome-wide association mapping of total antioxidant capacity, phenols, tannins, and flavonoids in a panel of Sorghum bicolor and S. bicolor × S. halepense populations using multi-locus models // PLoS One. 2019. V. 14. № 12. P. e0225979. https://doi.org/10.1371/journal.pone.0225979
Fossen T., Slimestad R., Øvstedal D.O. et al. Anthocyanins of grasses // Biochem. Syst. Ecol. 2002. V. 30. № 9. P. 855–864. https://doi.org/10.1016/S0305-1978(02)00028-5
Xiong Y., Zhang P., Warner R.D. et al. 3-Deoxyanthocyanidin colorant: Nature, health, synthesis, and food applications // Compr. Rev. Food Sci. F. 2019. V. 18. № 5. P. 1533–1549. https://doi.org/10.1111/1541-4337.12476
Pale E., Kouda-Bonafos M., Nacro M. et al. 7-O-methylapigeninidin, an anthocyanidin from Sorghum caudatum // Phytochemistry. 1997. V. 45. № 5. P. 1091–1092. https://doi.org/10.1016/S0031-9422(97)00099-X
Awika J.M., Rooney L.W., Waniska R.D. Properties of 3-deoxyanthocyanins from sorghum // J. Agric. Food Chem. 2004. V. 52. № 14. P. 4388–4394. https://doi.org/10.1021/jf049653f
Dykes L., Seitz L.M., Rooney W.L., Rooney L.W. Flavonoid composition of red sorghum genotypes // Food Chem. 2009. V. 116. № 1. P. 313–317. https://doi.org/10.1016/j.foodchem.2009.02.052
Winefield C.S., Lewis D.H., Swinny E.E. et al. Investigation of the biosynthesis of 3-deoxyanthocyanins in Sinningia cardinalis // Physiol. Plantarum. 2005. V. 124. № 4. P. 419–430. https://doi.org/10.1111/j.1399-3054.2005.00531.x
McMullen M.D., Snook M., Lee E.A. et al. The biological basis of epistasis between quantitative trait loci for flavone and 3-deoxyanthocyanin synthesis in maize (Zea mays L.) // Genome. 2001. V. 44. № 4. P. 667–676. https://doi.org/10.1139/g01-061
Zhang P., Chopra S., Peterson T. A segmental gene duplication generated differentially expressed myb-homologous genes in maize // Plant Cell. 2000. V. 12. № 12. P. 2311–2322. https://doi.org/10.1105/tpc.12.12.2311
Ibraheem F., Gaffoor I., Chopra S. Flavonoid phytoalexin-dependent resistance to anthracnose leaf blight requires a functional yellow seed1 in Sorghum bicolor // Genetics. 2010. V. 184. № 4. P. 915–926. https://doi.org/10.1534/genetics.109.111831
Kawahigashi H., Kasuga S., Sawada Y. et al. The sorghum gene for leaf color changes upon wounding (P) encodes a flavanone 4-reductase in the 3-deoxyanthocyanidin biosynthesis pathway // G3. 2016. V. 6. № 5. P. 1439–1447. https://doi.org/10.1534/g3.115.026104
Liu H., Du Y., Chu H. et al. Molecular dissection of the pathogen-inducible 3-deoxyanthocyanidin biosynthesis pathway in sorghum // Plant Cell Physiol. 2010. V. 51. № 7. P. 1173–1185. https://doi.org/10.1093/pcp/pcq080
Shih C.H., Chu I.K., Yip W.K. et al. Differential expression of two flavonoid 3'-hydroxylase cDNAs involved in biosynthesis of anthocyanin pigments and 3-deoxyanthocyanidin phytoalexins in sorghum // Plant Cell Physiol. 2006. V. 47. № 10. P. 1412–1419. https://doi.org/10.1093/pcp/pcl003
Mizuno H., Yazawa T., Kasuga S. et al. Expression level of a flavonoid 3′-hydroxylase gene determines pathogen-induced color variation in sorghum // BMC Res. Notes. 2014. V. 7. P. 761. https://doi.org/10.1186/1756-0500-7-761
Pilu R., Cassani E., Sirizzotti A. et al. Effect of flavonoid pigments on the accumulation of fumonisin B1 in the maize kernel // J. Appl. Genet. 2011. V. 52. P. 145–152. https://doi.org/10.1007/s13353-010-0014-0
Landoni M., Puglisi D., Cassani E. et al. Phlobaphenes modify pericarp thickness in maize and accumulation of the fumonisin mycotoxins // Sci. Rep. 2020. V. 10. P. 1417. https://doi.org/10.1038/s41598-020-58341-8
Sharma M., Chai C., Morohashi K. et al. Expression of flavonoid 3’-hydroxylase is controlled by P1, the regulator of 3-deoxyflavonoid biosynthesis in maize // BMC Plant Biol. 2012. V. 12. P. 196. https://doi.org/10.1186/1471-2229-12-196
Dong X., Braun E.L., Grotewold E. Functional conservation of plant secondary metabolic enzymes revealed by complementation of Arabidopsis flavonoid mutants with maize genes // Plant Physiol. 2001. V. 127. № 1. P. 46–57. https://doi.org/10.1104/pp.127.1.46
Grotewold E., Drummond B.J., Bowen B., Peterson T. The myb-homologous P gene controls phlobaphene pigmentation in maize floral organs by directly activating a flavonoid biosynthetic gene subset // Cell. 1994. V. 76. № 3. P. 543–553. https://doi.org/10.1016/0092-8674(94)90117-1
Ferreyra M.L.F., Rius S., Emiliani J. et al. Cloning and characterization of a UV-B-inducible maize flavonol synthase // Plant J. 2010. V. 62. № 1. P. 77–91. https://doi.org/10.1111/j.1365-313X.2010.04133.x
Grotewold E., Athma P., Peterson T. Alternatively spliced products of the maize P gene encode proteins with homology to the DNA-binding domain of myb-like transcription factors // PNAS. 1991. V. 88. № 11. P. 4587–4591. https://doi.org/10.1073/pnas.88.11.4587
Chopra S., Cocciolone S.M., Bushman S. et al. The maize unstable factor for orange1 is a dominant epigenetic modifier of a tissue specifically silent allele of pericarp color1 // Genetics. 2003. V. 163. № 3. P. 1135–1146.
Wittmeyer K., Cui J., Chatterjee D. et al. The dominant and poorly penetrant phenotypes of maize Unstable factor for orange1 are caused by DNA methylation changes at a linked transposon // Plant Cell. 2018. V. 30. № 12. P. 3006–3023. https://doi.org/10.1105/tpc.18.00546
Boddu J., Jiang C., Sangar V. et al. Comparative structural and functional characterization of sorghum and maize duplications containing orthologous Myb transcription regulators of 3-deoxyflavonoid biosynthesis // Plant Mol. Biol. 2006. V. 60. P. 185–199. https://doi.org/10.1007/s11103-005-3568-1
Glagoleva A.Y., Shoeva O.Y., Khlestkina E.K. Structural and functional divergence of homoeologous genes in allopolyploid plant genomes // Vavilovskii Zhurnal Genetiki i Selektsii = Vavilov Journal Genet. and Breeding. 2016. V. 20. № 6. P. 823–831. https://doi.org/10.18699/VJ16.204
Shoeva O.Y., Glagoleva A.Y., Khlestkina E.K. The factors affecting the evolution of the anthocyanin biosynthesis pathway genes in monocot and dicot plant species // BMC Plant Biol. 2017. V. 17. Suppl 2. P. 256. https://doi.org/10.1186/s12870-017-1190-4
Ji Q., Xu X., Wang K. Genetic transformation of major cereal crops // Int. J. Dev. Biol. 2013. V. 57. P. 495–508. https://doi.org/10.1387/ijdb.130244kw
Bennett M.D., Bhandol P., Leitch I.J. Nuclear DNA amounts in angiosperms and their modern uses – 807 new estimates // Ann. Bot. 2000. V. 86. № 4. P. 859–909. https://doi.org/10.1006/anbo.2000.1253
Arumuganathan K., Earle E.D. Nuclear DNA content of some important plant species // Plant Mol. Biol. Rep. 1991. V. 9. P. 208–218. https://doi.org/10.1007/BF02672069
Дополнительные материалы отсутствуют.