Генетика, 2020, T. 56, № 11, стр. 1320-1332
Трихомы у высших растений: гомологические ряды в наследственной изменчивости и молекулярно-генетические механизмы
Н. А. Швачко 1, *, Т. В. Семилет 1, Н. Г. Тихонова 1
1 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
* E-mail: n.shvachko@vir.nw.ru
Поступила в редакцию 08.05.2020
После доработки 08.06.2020
Принята к публикации 17.06.2020
Аннотация
Разнообразие трихом в растительном мире чрезвычайно велико: это опушение листовых пластин и колосковых чешуй у злаков, плодов и черешков у плодовых, трихомы в виде колючек у розы и огурца, в виде ворсинок у росянки или волокон у хлопчатника. Трихомы различаются по форме, размеру, структуре, местоположению, способности секретировать и т.д. Разнообразные трихомы объединяет общая основная функция – защита растений от воздействия различных биотических и абиотических факторов окружающей среды. Искусственный отбор иногда работает против развития трихом. Например, в селекции плодовых отдается предпочтение голоплодным формам. Среди дикорастущих видов также встречаются формы, контрастные по наличию/отсутствию трихом. В настоящем обзоре проведено сравнение механизмов образования трихом у разных видов, чтобы понять, насколько широко в пределах таксонов разного уровня простираются гомологические ряды в наследственной изменчивости. Обобщены данные по морфологии трихом, проведено сравнение данных о генах, определяющих изменчивость, выявлены ряды генов-ортологов. Сопоставление с данными о генных сетях, вовлеченных в развитие трихом, указываeт на то, что, несмотря на общность молекулярных механизмов развития трихом у всех высших растений, роль в изменчивости, наблюдаемой у разных таксонов, играют мутации разных компонент этой генной сети.
В процессе эволюции в связи с динамическими изменениями условий окружающей среды различные виды растений приобретают изменчивость по физиологическим и морфологическим признакам, что способствует большей пластичности видов, позволяющей им приспосабливаться в широком диапазоне изменений окружающей среды. К одному из важных морфологических признаков, изменчивость которого широко представлена в разных таксонах высших растений, относится наличие или отсутствие трихом, их строение и выполняемые функции. Формирование трихом является сложным процессом, происходящим в эпидермисе вегетативных и генеративных органов, который подразделяется на несколько фаз: фаза инициации, фаза дифференциации и развития трихом. Признаки, связанные с наличием или отсутствием опушения той или иной части растения, нередко используются для определения таксономической принадлежности и для описания сортов при их официальной регистрации (https:// www.upov.int/test_guidelines/en/list.jsp). Характер наследования опушения хорошо изучен у хозяйственно значимых видов растений еще в начале ХХ в. [1–5], показано сцепление с другими морфологическими признаками [6, 7]. Век спустя локусы, контролирующие наличие/отсутствие трихом, были картированы при помощи молекулярных маркеров [8–11]. К настоящему времени на молекулярном уровне хорошо изучены генные сети, регулирующие развитие трихом у модельных объектов однодольных (Oriza sativa L.) и двудольных (Arabidopsis thaliana (L.) Heynh.) растений [12]. С помощью in silico анализа генных сетей, участвующих в развитии трихом более 30 видов высших растений, установлено, что число генов в ортологических группах у высших растений, варьирует от 79 у Lotus japonicus (Regel) K. Larsen до 303 у Malus domestica Borkh. [12]. Однако взаимосвязь этих идентифицированных по гомологии нуклеотидных последовательностей у разных видов пока не сопоставлена с рядами фенотипической изменчивости, что является критичным для определения селекционно значимых генов. Сопоставление рядов гомологической изменчивости на уровне фено- и генотипа, понимание, насколько широко в пределах таксонов разного уровня простираются эти гомологические ряды – фундаментальная основа для ускоренного создания селекционных форм, адаптированных к неблагоприятным условиям окружающей среды и/или имеющих требуемый товарный вид. В настоящем обзоре рассматривается происхождение трихом и приводится сравнение генетических механизмов разнообразного проявления данного фенотипического признака у филогенетически удаленных таксонов.
МОРФОЛОГИЯ И ФУНКЦИИ ТРИХОМ
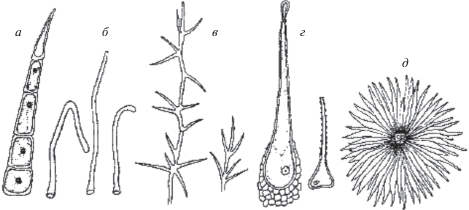
Трихомы (волоски) – специализированные выросты клеток поверхностного слоя эпидермиса, расположенные в наземных и подземных частях растений [13, 14]. Трихомы чрезвычайно разнообразны по своей форме, расположению на растении, выполняемым функциям, размерам и структуре (рис. 1). Каждая из этих особенностей трихомы является важным таксономическим признаком и для некоторых культур обусловливает селекционную значимость. Известны два основных типа трихом: нежелезистые (простые) и железистые [15, 16]. Основная функция нежелезистых трихом – защита растения от воздействия различных биотических и абиотических факторов окружающей среды. Трихомы защищают растения от насекомых и вредителей [17, 18], избыточного ультрафиолетового излучения [19], формируют микроклимат и защищают растение от перепадов температуры окружающей среды [20, 21]. Трихомы являются местами детоксикации [22, 23] и используются растением для выведения избытков тяжелых металлов [24]. Нежелезистые трихомы являются несекретирующими и могут быть как одноклеточными, так и многоклеточными. Например, плоды киви (Actinidia deliciosa (A. Chev.) C.F. Liang & A.R. Ferguson) покрыты двумя типами трихом: крупными многоярусными волосками и маленькими одноклеточными волосками. Крупные трихомы различаются у разных видов киви по расположению клеток [25]. Для культурных сортов характерно различие трихом по размеру и количеству слоев клеток. Кроме того, фенотип может меняться в процессе онтогенеза. Так, у одного из представителей рода Actinidia – A. аrguta (Siebold & Zucc.) Planch. ex Miq. плоды имеют трихомы только в зародышевом состоянии, по мере созревания плода трихомы исчезают. Трихомы зрелого плода A. deliciosa одревесневают [25]. Одноклеточные трихомы встречаются на плодах хлопчатника и персика, листьях арабидопсиса (A. thaliana), у представителей родов Ribes L. (смородины), Fragaria L. (земляники), а многоклеточные – у плодов киви. У некоторых видов растений, таких как смородина, киви и другие плодово-ягодные культуры, могут встречаться различные типы трихом на одном органе: кроющие, железистые, простые (игольчатые и жгутикообразные) и многоклеточные (пельтатные) [18]. У растений рода Ribes простые трихомы располагаются только на абаксиальной поверхности вдоль жилок [26, 27]. У представителей рода Rosaceae Juss. (яблоня, мушмула, груша и т.д.) трихомы могут располагаться на листовой пластине, вдоль жилок, на черешках, цветоножках, на плодах [28, 29]. Трихомы плодовых растений могут количественно видоизменяться в процессе развития растения. Например, на ранней стадии развития околоплодника малины простые трихомы мясистые и принадлежат к игольчатому типу, тогда как на средней стадии развития околоплодника малины трихомы характеризуются удлинением клеток, тонкой вершиной и тонким основанием. Это категория простых нежелезистых трихом, тип – жгутикообразный [30]. У однодольных растений по большей части определяются нежелезистые трихомы, которые располагаются на вегетативных (стебель, листья) и генеративных (колосковые чешуи) частях растений [31–36].
Рис. 1.
Типы трихом: а, в – простые многоклеточные; б – простые одноклеточные; г – железистые; д – простые звездчатые (цит. по [14]).
В отличие от простых волосков железистые трихомы являются только многоклеточными и могут значительно различаться по своему строению или по секреторным веществам. Железистые трихомы имеют различные варианты базовой структуры, которые включают в себя следующие компоненты: терминальная секреторная головка с одной или несколькими секреторными клетками; стебелек из одной или нескольких клеток; основание от одной до нескольких клеток; а иногда и шейные клетки между секреторной головкой и стебельком [15, 16]. Железистые трихомы преимущественно выполняют выделительную и защитную функции благодаря одной или нескольким секреторным клеткам, расположенным на конце одно- или многоклеточного стебля [13]. Они накапливают большое количество вторичных метаболитов, участвующих в защите растения от патогенных микроорганизмов [37, 38], а также привлекают насекомых-опылителей [16, 39, 40]. Железистые трихомы являются источником промышленно важных ингредиентов – ароматизаторов и фармацевтических препаратов [40]. Железистые трихомы более характерны для двудольных растений, но встречаются и у однодольных, например у сорта риса Tn1 были описаны мелкие железистые трихомы, встречающиеся вокруг устьиц [41]. Некоторые двудольные растения наряду с железистыми трихомами имеют и простые трихомы. На листовых пластинках томата отмечается наличие железистых и нежелезистых трихом. У томата описано восемь видов трихом, среди которых четыре железистых: тип I, тип IV, тип VI и тип VII. Железистые трихомы типа VI являются наиболее распространенными и секретируют в основном терпеноиды, тогда как тип I и тип IV участвуют в биосинтезе ацилового сахара [42]. Трихомы типа VII имеют одноклеточный стебель и многоклеточную ягодообразную головку и, по-видимому, не участвуют в биосинтезе вторичных метаболитов. Тип VI встречается почти у всех видов Solanaceae [43]. Одним из видоизменений трихом являются, например, колючки у малины и розы (Rubus, Rosa), которые представляют собой модифицированные железистые трихомы, продолжившие свой рост и в конечном итоге изменившиеся до их окончательного морфологического вида – колючек, выростов эпидермальной ткани [44, 45].
Таким образом, как у однодольных, так и у двудольных растений были обнаружены морфологически сходные типы трихом: простые и железистые, покрывающие вегетативные и/или генеративные органы растений и выполняющие сходную защитную функцию, повышающую адаптивный потенциал растений. Исследования, проводимые на морфологическом уровне, позволяют прослеживать гомологические ряды в наследственной изменчивости по признакам, связанным с развитием трихом, в соответствии с законом, сформулированным Николаем Ивановичем Вавиловым еще в 1920 г.
НАСЛЕДОВАНИЕ ГЕНОВ, ОПРЕДЕЛЯЮЩИХ ИЗМЕНЧИВОСТЬ НА УРОВНЕ ФЕНОТИПА
Однодольные
К.А. Фляксбергер, рассматривая закон гомологических рядов в наследственной изменчивости Н.И. Вавилова в отношении пшениц, подчеркивал, что гомологические ряды – не построение системы пшениц, а “интегрирование явлений полиморфизма”. В монографии “Пшеницы” им было выявлено, что опушенные листья встречаются среди всех видов пшениц, а признак наличие/отсутствие опушенности колоса встречается у всех видов пшениц, кроме Triticum timopheevi (Zhuk.) Zhuk., у которого не были обнаружены неопушенные формы [46]. Сегодня, с учетом развития исследований морфологических признаков на молекулярном уровне, можно найти объяснения тому, что наряду с параллелизмом в наследовании признаков, связанных с опушением, имеются и некоторые различия между разными таксонами по типу и количественным характеристикам трихом. Такие различия между представителями семейства злаков в свое время выявил Н.И. Вавилов в 1923 г. [7]. В своей классификации типов опушения Н.И. Вавилов выделял три группы злаковых растений в зависимости от количества и типа простых трихом: мохнатое, бархатистое и реснитчатое. Разработанные компьютерные методы высокопроизводительного фенотипирования на основе автоматического анализа изображений сгиба листа [47, 48] позволили провести сравнение широкого спектра видов родов Triticum и Aegilops разного уровня плоидности [36]. Среди пшениц разного уровня плоидности образцы с неопушенными листьями отсутствуют среди диплоидных форм пшеницы (2n = 14; геном АА), но встречаются намного чаще среди тетраплоидов (2n = 28; геномы ААВВ, AAGG), чем у гексаплоидов (2n = 42; геном ААВВDD); реснитчатый тип опушения характерен для диплоидов и практически не встречается у гексаплоидов, у последних чаще всего встречается бархатистый тип; установлено, что на плотность опушения влияют преимущественно геномы A и B, тогда как D-геном влияет на длину и плотность трихом, а также индекс опушения [36]. Диплоидные носители генома AA характеризуются большим разнообразием плотности опушения и имеют преимущественно короткие трихомы. Например, виды T. boeoticum Boiss и T. urartu Thumanjan ex Gandilyan имеют плотное опушение, в то время как T. monococcum L. отличается низкой плотностью опушения. Представители рода Aegilops L., носители геномов SS и DD характеризуются длинным, но редким опушением, аллотетраплоидные виды имеют различный тип опушения. У гексаплоидных видов с геномом AABBDD наблюдается умеренная длина трихом, однако сильно варьирует плотность опушения [36].
К пониманию молекулярно-генетических механизмов наследования разных типов опушения у пшеницы исследователи шли путем классического генетического анализа [1–3, 6, 7]; сведения о хромосомной локализации локусов, контролирующих развитие трихом, появились вследствие анализа нуллитетрасомных линий [49], моносомного анализа [31] и изучения дителосомных линий [32, 34]. С использованием молекулярно-генетического картирования появились сведения о точной внутрихромосомной локализации [33–35, 50]. В итоге было установлено расположение генов, контролирующих опушение листовой пластинки пшеницы, в длинном плече хромосомы 4B (Hl1) и коротком плече хромосомы 7В (Hl2). Doroshkov et al. исследовали фенотипическое проявление генов Hl1 и Hl2 и было впервые показано, что эти гены по-разному влияют на длину трихом и плотность опушения у пшеницы (Hl1 в большей степени связан с инициацией трихом и их ростом; Hl2 в большей степени влияет на длину трихом) [51]. У культурного ячменя доминантные аллели, ответственные за наличие трихом листовой пластинки, выявлены в длинном плече хромосомы 3H, а в длинном плече хромосомы 4H был обнаружен и картирован ген (Hsh), отвечающий за образование трихом листового влагалища [33, 52, 53]. У ржи ген Hp1 (Hairy peduncle 1) картирован на длинном плече хромосомы 5R [50]. Следует отметить, что участки хромосом 4BL пшеницы, 4HL ячменя и 5RL ржи коллинеарны [54].
У риса ген gl1 (glabrous leaf blade), контролирующий развитие трихом на листовой пластинке и цветковой чешуе, был картирован в коротком плече хромосомы 5 [55–57]. Это хромосомное плечо соответствует короткому плечу хромосомы 1Н ячменя [58], а, следовательно, ввиду синтении геномов ячменя и пшеницы и короткому плечу хромосомы 1A пшеницы, на котором был картирован ген опушения колосковой чешуи Hg [8]. Позже было установлено, что развитие трихом на листовой пластинке и цветковой чешуе регулируется фактором WOX, который кодируется геном GLR1 (Glabrous Rice 1), аллельным гену gl1 [41, 59].
Также в хромосоме 6 риса картирован еще один ген (GL6), контролирующий опушение листовой пластинки [60], для которого определены пять генов-кандидатов [61]. Хромосома 6 риса коллинеарна короткому плечу хромосомы 7Н ячменя [58], а, следовательно, ввиду синтении геномов ячменя и пшеницы и короткому плечу хромосомы 7В пшеницы, на котором был картирован ген опушения листовой пластинки Hl2 [34].
У кукурузы ген Mhl1 (Macrohairless 1), контролирующий развитие трихом на листовой пластинке, локализован в длинном плече хромосомы 9 вблизи маркеров umc1120 и umc95 [62]. Ген-кандидат для локуса Mhl1 пока не выделен, однако стоит отметить, что длинное плечо хромосомы 9 кукурузы соответствует хромосоме 6 риса; кроме того, маркер umc1120 тесно сцеплен с локусом sus1, район локализации которого коллинеарен району короткого плеча хромосомы 7 пшеницы [63].
У кукурузы был выделен ген OCL4, кодирующий транскрипционный фактор из семейства HD-ZIP IV, который влияет на развитие трихом [64]. Этот ген не соответствует ни одному из указанных гомологичных рядов (табл. 1). Данный ген располагается в хромосоме 1 кукурузы, которая имеет участки, коллинеарные с хромосомами риса 3, 8 и 10 [65], что не совпадает с хромосомной локализацией GLR1 или GL6 риса.
Таблица 1.
Гомологичные серии генов, определяющих изменчивость, связанную с наличием/отсутствием трихом у представителей сем. Poaceae
| Гомологичный ряд (кодируемый фактор) | Пшеница | Ячмень | Рожь | Рис | Кукуруза |
|---|---|---|---|---|---|
| Ряд 1 (не изв.) | Hl1 | Hsh | Hp1 | – | – |
| Ряд 2 (не изв.) | Hl2 | – | – | GL6 | Mhl1 |
| Ряд 3 (WOX) | Hg | – | – | GLR1 | – |
Таким образом, среди злаковых растений выделяются три предполагаемых гомологичных ряда генов, определяющих изменчивость, выявляемую на морфологическом уровне (табл. 1); большую часть этих генов еще предстоит расшифровать, чтобы доказать общее происхождение генов в пределах каждой серии.
Двудольные
Модельным объектом двудольных растений является арабидопсис (A. thaliana), для которого идентифицировано множество генов, контролирующих развитие трихом [66, 67]. Так, мутация в гене GL3 приводит к меньшему количеству трихом и уменьшению ветвления. Ген EGL3 оказывает умеренное влияние на количество трихом. Однако двойные мутанты gl3 egl3 обладают “голым” фенотипом. ТТ8, контролирующий биосинтез антоцианов и проантоцианидинов в вегетативных тканях и оболочке семян [68–70], также контролирует развитие трихом на краях листовых пластинок у A. thaliana [70]. Белок WD, кодируемый геном TTG1 A. thaliana, рассматривается как важнейший регулятор образования трихом на листьях, стеблях и корнях [71]. Растения арабидопсиса, мутантные по гену ttg1, безантоциановые и не имеют трихом [71, 72].
Работы на модельном объекте способствовали пониманию генетических механизмов контроля фенотипической изменчивости, связанной с развитием трихом у хозяйственно ценных видов. Так, гомологи гена TTG1 A. thaliana были выявлены у других двудольных и однодольных видов растений. Гомолог TTG1, выделенный из M. domestica, демонстрировал 80.2% идентичности A. thaliana TTG1 по нуклеотидам и 90.7% схожести по аминокислотному составу [73]. При трансформации в мутанты ttg1 A. thaliana гомологи яблони смогли восстановить рост трихом, выработку антоцианов в молодых проростках и проантоцианидинов в семенах. Гомологи гена TTG1 A. thaliana [71] были выявлены у Perilla frutescens (L.) Britton (PFWD) [74], Gossypium hirsutum L. (TTG1) [75], Matthiola incana (L.) W.T. Aiton (TTG1) [76], а также (PAC1) у Zea mays L. и др. [77]. Предположительно ген MdTTG1 принадлежит к семейству генов WD40. Аминокислотная последовательность MdTTG1 показывает высокую степень идентичности с другими белками двудольных растений, такими как PhAN11 [78], GhTTG1, AtTTG1 и PFWD. У растений чая (Camellia sinensis (L.) O. Kuntze) идентифицирован ген CsTTG1, который кодирует белок WDR и гомологичен AtTTG1 [79]. Установлена его роль не только в формировании трихом растений чая, но и в изменении плотности трихом листьев растений чая при различных уровнях экспрессии CsTTG1. Дальнейшие исследования показали, что CsTTG1 имеет идентичность 91.23% к AcTTG1, но функция AcTTG1 в Actinidia chinensis Planch. пока не известна. Вместе с тем CsTTG1 высоко гомологичен гену VcTTG1, который участвует в синтезе антоцианов Viccinium corymbosun L. [79]. Для гена RrGL1, выявленного у плодов розы, установлена гомология с GL1 A. thaliana [80] (табл. 2). Факторы транскрипции GLABROUS1 (GL1), TTG1 и ENHANCER OF GLABRA3 (GL3) были выявлены у сладкой полыни (Artemisia annua L.) как способствующие инициации железистых трихом [81]. Интересно, что образование волокон хлопчатника (G. hirsutum), напоминающих трихомы A. thaliana, контролируется GaMYB2, который демонстрирует высокую гомологию последовательности с GL1 и может восстанавливать образование трихом у мутанта арабидопсиса gl1 [82]. Кроме того, Wang et al. [83] выявили, что в инициации трихом (волокон хлопчатника) участвуют гены GhHOX1, GhMYC1 (гомолог GL3 арабидопсиса) [83, 84] и гомологи гена GL2 в арабидопсисе GbML1, GhHD1, GhHOX [83, 85] (табл. 2). Данные исследования показывают сходные пути регуляции инициации трихом арабидопсиса и хлопчатника.
Таблица 2.
Гомологичные серии генов, определяющих изменчивость, связанную с наличием/отсутствием трихом у представителей класса двудольных растений
| Гомологичный ряд (кодируемый фактор) | Арабидопсис | Огурец | Хлопчатник | Роза | Яблоня | Полынь |
|---|---|---|---|---|---|---|
| Ряд 1 (WD40) | TTG1 | – | GhTTG1 | RrTTG1 | MdTTG1 | TTG1 |
| Ряд 2 (R2R3-MYB) | GL1 | CsGL1 | GaMYB2 | RrGL1 | – | GL1 |
| Ряд 3 (GLABROUS2) | GL2 | CsGL2 | GbML1 | – | – | – |
| Ряд 4 (bHLH) | GL3/EGL3 | TRIL/CsGL3 | GhMYC1/GhHOX1 | – | – | GL3 |
Изучение простых трихом плодов персика показало моногенный характер наследования. Локус, контролирующий данный признак, был картирован в дистальной части хромосомы 5 [25]. Ген-кандидат, PpeMYB25 (ppa023143m), для этого локуса у растений с отсутствующим опушением плода имеет протяженную инсерцию в экзоне 3. Это уникальное мутационное событие привело к формированию рецессивного признака нектарина (отсутствие опушения плода), которое было закреплено в процессе искусственного отбора. Разработанный на основе этого гена функциональный маркер (indelG) позволяет на ранней стадии отобрать растения по признаку персик/нектарин [25].
Огурец (Cucumis sativus L.) характеризуется наличием трихом двух типов: железистых и простых. Классический генетический анализ и последующие молекулярные исследования показали, что за дифференцировку клеток эпидермиса и развитие простых трихом огурца ответственны следующие гены: CsGL3, TRIL, MICT, TBH и CsGL1 [86, 87]. Выявленные гены огурца были картированы на хромосоме 6. Ген Csa6M514870 кодирует фактор транскрипции, принадлежащей к семейству HD-ZIP [88]. В огурце гены TRIL/CsGL3 являются положительными регуляторами развития трихом, мутантные растения tril/csgl3 имеют фенотип без трихом [89].
У представителя семейства Rosaceae – Rosa rugose идентифицирован ген RrTTG1, связанный с развитием трихом; мутации в данном гене могут использоваться в селекции розоцветных для получения растений с отсутствием шипов или их небольшим количеством. Китайские исследователи [30] на основе морфологического сходства жгутикообразных трихом околоплодника розы (R. roxburghii Tratt) с многоклеточными трихомами плодов огурца дикого типа [87] выдвинули гипотезу, что инициация и развитие трихом как у розы, так и у огурца, возможно, контролируются двумя генами, причем доминантное состояние обоих генов контролирует наличие жестких колючек, тогда как предполагаемые носители данных генов в рецессивном состоянии имеют мягкие, практически незаметные колючки. Данная гипотеза подтвердилась для розы, но не для огурца [89].
Таким образом, на основании литературных данных можно говорить о наличии как минимум четырех гомологичных рядов генов двудольных растений, определяющих изменчивость по наличию/отсутствию трихом (табл. 2), что свидетельствует о сходных путях регуляции инициации трихом данных двудольных растений. Однако предстоят еще дополнительные исследования для выявления и дальнейшей расшифровки генов этих гомологичных рядов у других видов двудольных растений.
ГЕННЫЕ СЕТИ, ВОВЛЕЧЕННЫЕ В ФОРМИРОВАНИЕ И РАЗВИТИЕ ТРИХОМ
Простые трихомы
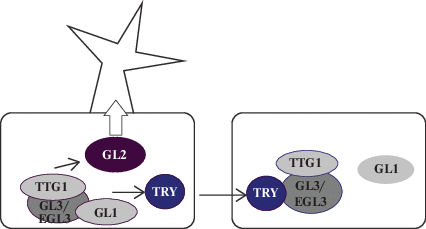
Развитие простых трихом у арабидопсиса регулируется обширной транскрипционной сетью, включающей несколько групп транскрипционных факторов (ТФ), основными из которых являются R2R3-MYB, helix-loop-helix (bHLH) и белок WD40-повтор (WDR). Эти три группы ТФ образуют тримерный MBW-комплекс: R2R3-MYB (GL1, WER, MYB82, MYB5 или MYB23), bHLH (TT8, EGL3, GL3 или MYC1), WD40 (TTG1), активирующий экспрессию гомеодоменового белка GL2 (GLABROUS2), который в свою очередь индуцирует образование трихом [12, 66, 72, 73, 90, 91] (рис. 2). Анализ генома арабидопсиса показывает, что семейство R2R3-MYB состоит из 126 генов, которые подразделяются на 25 подгрупп [92]. Семейство bHLH является одним из самых больших и известных ТФ и состоит из 160 генов, подразделяющихся в арабидопсисе на 12 подгрупп [93]. Факторы bHLH действуют как места стыковки для ряда регуляторных белков в комплексе MBW. За связывание с bHLH наряду с R2R3-MYB конкурируют R3-MYB, образуя, таким образом, репрессорный комплекс и действуя как негативные регуляторы развития трихом [92, 94]. К таким R3-MYB-репрессорам относятся: TRY (TRIPTYCHON) [95]; CPC (CAPRICE) [96]; ETC1, ETC2 и ETC3 (ENHANCER OF TRY 1–3) [97], TCL1 и TCL2 (TRICHOMELESS 1 и 2) [72, 98, 99].
Рис. 2.
Регуляторная модель инициации трихом на листьях эпидермиса Arabidopsis. Комплекс GLABRA1 (GL1)–GLABRA3 (GL3)/ENHANCER GLABRA3 (EGL3)–TRANS-PARENT TESTA GLABRA1 (TTG1) активирует экспрессию GLABRA2 (GL2) и TRIP-TYCHON (TRY). GL2 индуцирует дифференцировку трихом. Белок TRY перемещается в соседние клетки и конкурирует с GL1 за сайт связывания комплекса. Комплекс TRY-GL3/EGL3-TTG1 не способствует ни экспрессии GL2, ни инициации трихом (цит. по [113]).
Белки WD40 содержат высококонсервативные аминокислотные тандемные повторы (число повторов 40–43), обычно имеющие на конце Trp-Asp (WD). Они участвуют в регуляции ряда процессов, включая клеточный цикл, дифференцировку клеток и передачу сигналов клетки [100]. У арабидопсиса ген TTG1 (TRANSPARENT TESTA GLABRA1) является единственной копией семейства WD40 (WDR) [71, 72].
Еще одной группой ТФ, участвующей в формировании трихом на стеблях и цветках соцветия арабидопсиса, являются C2H2 (белки цинковые пальцы) [101, 102]. C2H2 представляют собой крупное семейство, состоящее из 176 регуляторных белков, участвующих в многочисленных физиологических процессах растений [103].
Кроме основных групп ТФ важную роль в развитии трихом у арабидопсиса играют фитогормоны, включая гиббереллины (GA), цитокинины (CK) и жасмоновые кислоты (JA) [72, 101, 104, 105]. Таким образом, в многочисленных работах по изучению инициации развития простых трихом арабидопсиса выявлена сложная регуляторная сеть, контролирующая этот процесс.
Молекулярно-генетические механизмы, лежащие в основе образования трихом однодольных, остаются в значительной степени невыясненными. Лучше других эти механизмы изучены на модельном для однодольных растений объекте – рисе. Образование трихом у риса контролируется несколькими факторами транскрипции: AP2/ERF, HL6 (Hairy Leaf 6). Основным регулятором инициации трихом риса является гомеодоменовый белок OsWOX3B, образующий комплекс с HL6. Активирующий комплекс OsWOX3B–HL6 регулирует как инициацию, так и удлинение трихом [106, 107].
Железистые трихомы
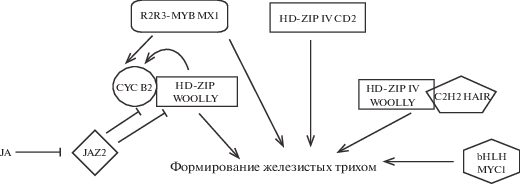
Механизмы инициации образования железистых трихом остаются менее изученными [72]. По литературным источникам в качестве одного из основных модельных объектов для изучения генной регуляции инициации и развития железистых трихом используется томат (Solanum lycopersicum L., сем. Solanaceae).
Образование железистых трихом у томата регулируется группой транскрипционных факторов, основные из которых относятся к семействам R2R3-MYB, HD-ZIP IV, bHLH и C2H2 [42, 108]. Транскрипционные факторы SlMX1 (MIXTA – семейство R2R3-MYB) [109], SlMYC1 (MYELOCYTOMATOSIS-RELATED1 – семейство bHLH) [110], SlH (HAIR – семейство C2H2) и SlCD2 (CUTIN DEFICIENT2 – семейство HD-ZIP IV) [108] были охарактеризованы как положительные регуляторы инициации железистых трихом томата. Важным регулятором этого процесса также является транскрипционный фактор HD-ZIP IV WOOLLY (Wo) [108]. Доминирующие точечные мутации в С-концевой части Wo отвечают за фенотип мутантов с увеличенной плотностью трихом [111]. Wo усиливает инициацию железистых трихом I типа [111]. Заметим, что фактор HD-ZIP уже упоминался в связи с развитием трихом (простых) у кукурузы и огурца [64].
Прямое взаимодействие между белками C2H2 SlH и транскрипционным фактором HD-ZIP IV Wo образует комплекс, инициирующий развитие железистых трихом томата [101, 112, 113]. Этот процесс регулируется и растительными гормонами, такими как жасмонаты (JA). У томатов избыточная экспрессия сигнального репрессора JA SlJAZ2 снижает плотность железистых трихом, указывая на то, что SlJAZ2 является негативным регулятором инициации этого типа трихом (рис. 3) [108].
ЗАКЛЮЧЕНИЕ
Несмотря на все разнообразие трихом в растительном мире, они выполняют сходную функцию и способствуют адаптационной способности растений к изменяющимся условиям окружающей среды. У однодольных и двудольных растений выявлено большое количество генов, участвующих в образовании трихом. При этом количество генов, определяющих изменчивость по признакам, связанным с наличием/отсутствием трихом, ограничено, и они относятся к нескольким семействам транскрипционных факторов, инициирующих развитие трихом растений. Механизмы образования простых и железистых трихом являются отчасти сходными. Прослеживаются гомологические ряды изменчивости. Однако не для всех видов растений выявлены гены-кандидаты, разные аллельные состояния которых определяют наличие или отсутствие трихом на той или иной части растения. Результаты сравнительного анализа механизмов образования трихом у разных видов растений способствуют заполнению “пробелов” в гомологических рядах изменчивости, что является основой для целенаправленного улучшения адаптации сортов культурных растений к неблагоприятным факторам окружающей среды (с наличием трихом, обеспечивающих в ряде случаев устойчивость к био- и абиотическим стрессорам) или получения сортов с надлежащим товарным качеством (голоплодные, без колючек и т.д.).
Статья подготовлена в рамках государственного задания ВИР согласно тематическому плану НИР по теме № 0481-2019-0001 “Геномные и постгеномные технологии для выявления новых генетических маркеров селекционно значимых свойств и новых аллельных вариантов хозяйственно ценных генов в генофонде культурных растений и их диких родичей”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Список литературы
Tschermak E. Über Züchtung neuer Getreiderassen mittelst künstlicher Kreuzung // Zeitschr. Landw. Versuch. Oesterreich. 1901. Bd 4. S. 1029–1060.
Howard A., Howard G. On the inheritance of some characters in wheat. India: Dept. Agr. Mem., 1912. Bot. Ser. 5. P. 1–47.
Howard A., Howard G. On the inheritance of some characters in wheat. India: Dept. Agr. Mem. 1915. Bot. Ser. 7. P. 273–285.
Wellington R. Mendelian inheritance of epidermal characters in the fruit of Cucumis sativus // Science. 1913. V. 38. Is. 967. P. 61. https://doi.org/10.1126/science.38.967.61
Stewart R.T., Wentz J.B. Recessive glabrous character in soybeans // Agronomy J. 1926. V. 18. P. 997–1009.
Ваценко А.А. Наследование опушения чешуй и черной окраски колоса у твердых пшениц Triticum durum Desf. // ДАН СССР. 1934. Т. 4(5–6). С. 338–342.
Вавилов Н.И. Научные основы селекции пшеницы. М.; Л.: Сельхозгиз, 1935. С. 70–87.
Khlestkina E.K., Pshenichnikova T.A., Röder M.S. et al. Comparative mapping of genes for glume colouration and pubescence in hexaploid wheat (Triticum aestivum L.) // Theor. Appl. Genet. 2006. V. 113. P. 801–807. https://doi.org/10.1007/s00122-006-0331-1
Dobrovolskaya O., Pschenichnikova T.A., Arbuzova V.S. et al. Molecular mapping of genes determining hairy leaf character common wheat with respect to other species of the Triticeae // Euphytica. 2007. V. 155. P. 285–293. https://doi.org/10.1007/s10681-006-9329-7
Du W., Yu D., Fu S. Analysis of QTLs for the trichome density on the upper and downer surface of leaf blade in soybean (Glycine max (L.) Merr.) // Agricultural Sci. in China. 2009. V. 8. P. 529–537. https://doi.org/10.1016/S1671-2927(08)60243-6
Pan Y., Bo K., Cheng Z., Weng Y. The loss-of-function GLABROUS 3 mutation in cucumber is due to LTR-retrotransposon insertion in a class IV HD-ZIP transcription factor gene CsGL3 that is epistatic over CsGL1 // BMC Plant Biology. 2015. V. 15. P. 1–15.https://doi.org/10.1186/s12870-015-0693-0
Doroshkov A.V., Konstantinov D.K., Afonnikov D.A., Gunbin K.V. The evolution of gene regulatory networks controlling Arabidopsis thaliana L. trichome development // BMC Plant Biology. 2019. V. 19. P. 71–85.https://doi.org/10.1186/s12870-019-1640-2
Сауткина Т.А., Поликсенова В.Д. Морфология растений // Курс лекций: В 2 ч. Ч. 1. Минск: БГУ, 2004. 115 с.
Коровин О.А. Анатомия и морфология высших растений. Словарь терминов. М.: Дрофа, 2007. 286 с.
Werker E. Trichome diversity and development // Adv. Bot. Res. 2000. V. 31. P. 1–35. https://doi.org/10.1016/S0065-2296(00)31005-9
Wagner G.J., Wang E., Shepherd R.W. New approaches for studying and exploiting an old protuberance, the plant trichome // Annals Bot. 2004. V. 93. Is. 1. P. 3–11. https://doi.org/10.1093/aob/mch011
Levin D.A. The role of trichomes in plant defense // The Quarterly Rev. Biol. 1973. V. 48. Is. 1. Part 1. P. 3–15. https://doi.org/10.1086/407484
Huttunen P., Kärkkäinen K., Løe G. et al. Leaf trichome production and responses to defoliation and drought in Arabidopsis lyrata (Brassicaceae) // Annales Bot. Fennici. 2010. V. 47. Is. 3. P. 199–207.https://doi.org/10.5735/085.047.0304
Karabourniotis G., Papadopoulos K., Papamarkou M., Manetas Y. Ultraviolet-B radiation absorbing capacity of leaf hairs // Physiol. Plantarum. 1992. V. 86. Is. 3. P. 414–418. https://doi.org/10.1111/j.1399-3054.1992.tb01337.x
Johnson H.B. Plant pubescence: An ecological perspective // The Bot. Rev. 1975. V. 41. Is. 3. P. 233–258.https://doi.org/10.1007/BF02860838
Karabourniotis G., Liakopoulos G., Nikolopoulos D., Bresta P. Protective and defensive roles of non-glandular trichomes against multiple stresses: structure–function coordination // J. Forestry Res. 2020. V. 31. Is. 1. P. 1–12. https://doi.org/10.1007/s11676-019-01034-4
Gutierrez-Alcala G., Gotor C., Meyer A.J. et al. Glutathione biosynthesis in Arabidopsis trichome cells // Proc. Nat. Acad. Sci. USA. 2020. V. 97. Is. 20. P. 11108–11113. https://doi.org/10.1073/pnas.190334497
Salt D.E., Prince R.C., Pickering I.J. et al. Mechanisms of cadmium mobility and accumulation in Indian mustard // Plant Physiol. 1995. V. 109. P. 1427–1433.https://doi.org/10.1104/pp.109.4.1427
Domínguez-Solís J.R., López-Martín M.C., Ager F.J. et al. Increased cysteine availability is essential for cadmium tolerance and accumulation in Arabidopsis thaliana // Plant Biotechnol. J. 2004. V. 2. Is. 6. P. 469–476. https://doi.org/10.1111/j.1467-7652.2004.00092.x
Vendramin E., Pea G., Dondini L. et al. A unique mutation in a MYB gene cosegregates with the nectarine phenotype in peach // PLoS One. 2014. V. 9. Is. 3. P. 1–13. https://doi.org/10.1371/journal.pone.0090574
Рeзанова Т.А., Сорокопудов В.Н., Колесников Д.А. Морфологическая классификация трихом Ribes americanum Mill. (Grossulariaceae) // Науч. ведомости. 2010. Вып. 11. № 9(80). С. 5–10.
Кузнецова Т.А. Адаптационные изменения листовой поверхности Ribes nigrum L. в зависимости от различных условий водообеспеченности // Науч. ведомости. 2015. Вып. 31. № 9(206). С. 29–36.
Кумахова Т.Х., Воронков А.С., Башабова А.В., Рябченко А.С. Морфофункциональная характеристика листьев и плодов Maloideae (Rosaceae). Микроструктура поверхностных тканей // Тр. по прикладной ботанике, генетике и селекции. 2019. Т. 180. № 1. С. 105–112. https://doi.org/10.30901/2227-8834-2019-1-105-112
Николаенко В.В. Анатомо-морфологические особенности развития листового аппарата декоративной земляники под влиянием окружающей среды // Уч. зап. Крымского федерального ун-та им. В.И. Вернадского. 2010. Т. 23(62). № 2. С. 131–141.
Wang Y., Zhao M.Y., Xu Z.D. et al. Cloning and expression analysis of TTG1 gene related to Rosa rugosa trichomes formation // Am. J. Plant Sci. 2019. V. 10. P. 265–275. https://doi.org/10.4236/ajps.2019.1020
Майстренко О.И. Идентификация и локализация генов, контролирующих опушение листа молодых растений мягкой пшеницы // Генетика. 1976. Т. 12. № 5. С. 5–15.
Arbuzova V.S., Efremova T.T., Laikova L.I. et al. The development of precise genetic stocks in two wheat cultivars and their use in genetic analysis // Euphytica. 1996. V. 89. P. 11–15.
Korzun V., Malyshev S., Pickering R.A., Börner A. RFLP mapping of a gene for hairy leaf sheath using a recombinant line from Hordeum vulgare L. × Hordeum bulbosum L. cross // Genome. 1999. V. 42. Is. 5. P. 960–963.https://doi.org/10.1139/g99-021
Taketa S., Chang C.L., Ishii M., Takeda K. Chromosome arm location of the gene controlling leaf pubescence of a Chinese local wheat cultivar “Hong-mang-mai” // Euphytica. 2002. V. 125. P. 141–147. https://doi.org/10.1023/A:1015812907111
Dobrovolskaya O., Pschenichnikova T.A., Arbuzova V.S. et al. Molecular mapping of genes determining hairy leaf character common wheat with respect to other species of the Triticeae // Euphytica. 2007. V. 155. P. 285–293. https://doi.org/10.1007/s10681-006-9329-7
Pshenichnikova T.A., Doroshkov A.V., Simonov A.V. et al. Diversity of leaf pubescence in bread wheat and relative species // Genet. Resour. Crop. Evol. 2017. V. 64. P. 1761–1773. https://doi.org/10.1007/s10722-016-0471-3
Glas J.J., Schimmel B.C.J., Alba J.M. et al. Plant glandular trichomes as targets for breeding or engineering of resistance to herbivores // Int. J. Mol. Sci. 2012. V. 13. P. 17077–17103. https://doi.org/10.3390/ijms131217077
Tissier A. Glandular trichomes: what comes after expressed sequence tags // The Plant J. 2012. V. 70. P. 51–68. https://doi.org/10.1111/j.1365-313X.2012.04913.x
Wagner G.J. Secreting glandular trichomes: more than just hairs // Plant Physiol. 1991. V. 96. P. 675–679. doi 0032-0889/91/96/0675/05/$01.00/0
Schilmiller A.L., Last R.L., Pichersky E. Harnessing plant trichome biochemistry for the production of useful compounds // The Plant J. 2008. V. 54. P. 702–711. https://doi.org/10.1111/j.1365-313X.2008.03432.x
Zhang H., Wu K., Wang Y. et al. A WUSCHEL-like homeobox gene, OsWOX3B responses to NUDA/GL-1 locus in rice // Rice. 2012. V. 5(30). P. 1–10. https://doi.org/10.1016/j.ab.2012.03.030
Bergau N., Bennewitz S., Syrowatka F. et al. The development of type VI glandular trichomes in the cultivated tomato Solanum lycopersicum and a related wild species S. habrochaites // BMC Plant Biology. 2015. V. 15. P. 1–15. https://doi.org/10.1186/s12870-015-0678-z
Bennewitz S., Bergau N., Tissier A. QTL mapping of the shape of type VI glandular trichomes in tomato // Frontiers Plant Sci. 2018. P. 1–12. https://doi.org/10.3389/fpls.2018.01421
Kellogg A.A., Branaman T.J., Jones N.M. et al. Morphological studies of developing Rubus prickles suggest that they are modified glandular trichomes // Botany. 2011. V. 89. № 4. P. 217–226. https://doi.org/10.1139/b11-008
Wang D.-J., Zeng J.-W., Ma W.-T. Morphological and structural characters of trichomes on various organs of Rosa roxburghii // HortScience. 2019. V. 54. Is. 1. P. 45–51. https://doi.org/10.21273/HORTSCI13485-18
Фляксбергер К.А. Пшеницы. М.; Л.: Огиз, 1935. С. 202–203.
Дорошков А.В., Пшеничникова Т.А., Афонников Д.А. Анализ особенностей морфологии и наследования опушения листа пшеницы Triticum aestivum L. с помощью компьютерных методов фенотипирования // Генетика. 2011. Т. 47. № 6. С. 836–841.
Genaev M.A., Doroshkov A.V., Pshenichnikova T.A. et al. Extraction of quantitative characteristics describing wheat leaf pubescence with a novel image-processing technique // Planta. 2012. V. 236. P. 1943–1954. https://doi.org/10.1007/s00425-012-1751-6
Sears E.R. Nullisomic analysis in common wheat // Am. Nat. 1953. V. 87. P. 245–252.
Korzun V., Malyshev S., Kartel N. et al. A genetic linkage map of rye (Secale cereale L.) // Theor. Appl. Genet. 1998. V. 96. P. 203–208.
Doroshkov A.V., Afonnikov D.A., Dobrovolskaya O.B., Pshenichnikova T.A. Interactions between leaf pubescence genes in bread wheat as assessed by high throughput phenotyping // Euphytica. 2016. V. 207. Is. 3. P. 491–500. https://doi.org/10.1007/s10681-015-1520-2
Franckowiak J.D. Revised linkage maps for morphological markers in barley, Hordeum vulgare // Barley Genet. Newsl. 1997. V. 26. P. 9–21.
Lundqvist U.J., Franckowiak D., Konishi T. New and revised descriptions of barley genes // Barley Genet. Newsl. 1997. V. 26. P. 22–516.
Devos K.M., Atkinson M.D., Chinoy C.N. et al. Chromosomal rearrangements in the rye genome relative to that of wheat // Theor. Appl. Genet. 1993. V. 85. P. 673–680. https://doi.org/10.1007/BF00225004
Yu Z.H., McCouch S.R., Tanksley S.D. et al. Association of morphological and RFLP markers in rice (Oryza sativa L.) // Genome. 1995. V. 38. P. 566–574. https://doi.org/10.1139/g95-073
Wang D., Sun S.-X., Gao F.-Y. et al. Mapping a rice glabrous gene using simple sequence repeat markers // Rice Sci. 2009. V.16. P. 93–98. https://doi.org/10.1016/S1672-6308(08)60063-3
Li W., Wu J., Weng S. et al. Characterization and fine mapping of the glabrous leaf and hull mutants (gl1) in rice (Oryza sativa L.) // Plant Cell Rep. 2010. V. 29. P. 617–627. https://doi.org/10.1007/s00299-010-0848-2
Stein N., Prasad M., Scholz U. et al. A 1000-loci transcript map of the barley genome: new anchoring points for integrative grass genomics // Theor. Appl. Genet. 2007. V. 114. P. 823–839. https://doi.org/10.1007/s00122-006-0480-2
Li J., Yuan Y., Lu E., Yang L. Glabrous Rice 1, encoding a homeodomain protein, regulates trichome development in rice // Rice. 2012. V. 5. Is. 32. P. 1–10. https://doi.org/10.1186/1939-8433-5-32
Zeng Y.H., Zhu Y.S., Lian L. et al. Genetic analysis and fine mapping of the pubescence gene GL6 in rice (Oryza sativa L.) // Chin. Sci. Bull. 2013. V. 58. P. 2992–2999. https://doi.org/10.1007/s11434-013-5737-y
Hamaoka N., Yasui H., Yamagata Y. et al. A hairy-leaf gene, BLANKET LEAF, of wild Oryza nivara increases photosynthetic water use efficiency in rice // Rice. 2017. V. 10. P. 1–11. https://doi.org/10.1186/s12284-017-0158-1
Moose S.P., Lauter N., Carlson S.R. The maize macrohairless1 locus specifically promotes leaf blade macrohair initiation and responds to factors regulating leaf identity // Genetics. 2004. V. 166. Is. 3. P. 1451–1461. https://doi.org/10.1534/genetics.166.3.1451
Devos K.M., Chao S., Li Q.Y. et al. Relationship between chromosome 9 of maize and wheat homeologous group 7 chromosomes // Genetics. 1994. V. 138. P. 1287–1292.
Vernoud V., Laigle G., Rozier F. et al. The HD-ZIP IV transcription factor OCL4 is necessary for trichome patterning and anther development in maize // The Plant J. 2009. V. 59. P. 883–894.
Ahn S., Tanksley S.D. Comparative linkage maps of the rice and maize genomes // Proc. Natl Acad. Sci. USA. 1993. V. 90. P. 7980–7984.
Payne C.T., Zhang F., Lloyd A.M. GL3 encodes a bHLH protein that regulates trichome development in arabidopsis through interaction with GL1 and TTG1 // Genetics. 2000. V. 156. Is. 3. P. 1349–1362.
Zhang F. A network of redundant bHLH proteins functions in all TTG1-dependent pathways of Arabidopsis // Development. 2003. V. 130. Is. 20. P. 4859–4869. https://doi.org/10.1242/dev.00681
Nesi N., Debeaujon I., Jond C. et al. The TT8 gene encodes a basic helix-loop-helix domain protein required for expression of DFR and BAN genes in Arabidopsis siliques // The Plant Cell. 2000. V. 12. Is. 10. P. 1863–1878. https://doi.org/10.1105/tpc.12.10.1863
Baudry A., Heim M.A., Dubreucq B. et al. TT2, TT8, and TTG1 synergistically specify the expression of BANYULS and proanthocyanidin biosynthesis in Arabidopsis thaliana // The Plant J. 2004. V. 39. Is. 3. P. 366–380. https://doi.org/10.1111/j.1365-313X.2004.02138.x
Maes L., Inze D., Goossens A. Functional specialization of the TRANSPARENT TESTA GLABRA1 network allows differential hormonal control of laminal and marginal trichome initiation in Arabidopsis rosette leaves // Plant Physiology. 2008. V. 148. Is. 3. P. 1453–1464. https://doi.org/10.1104/pp.108.125385
Walker A.R., Davison P.A., Bolognesi-Winfield A.C. et al. The TRANSPARENT TESTA GLABRA1 locus, which regulates trichome differentiation and anthocyanin biosynthesis in Arabidopsis, encodes a WD40 repeat protein // The Plant Cell. 1999. V. 11. Is. 7. P. 1337–1349. https://doi.org/10.1105/tpc.11.7.1337
Pattanaik S., Patra B., Singh S.K., Yuan L. An overview of the gene regulatory network controlling trichome development in the model plant, Arabidopsis // Frontiers Plant Sci. 2014. P. 1–8. https://doi.org/10.3389/fpls.2014.00259
Brueggemann J., Weisshaar B., Sagasser M. A WD40-repeat gene from Malus × domestica is a functional homologue of Arabidopsis thaliana TRANSPARENT TESTA GLABRA1 // Plant Cell Rep. 2010. V. 29. Is. 3. P. 285–294. https://doi.org/10.1007/s00299-010-0821-0
Sompornpailin K., Makita Y., Yamazaki M. et al. A WD-repeat-containing putative regulatory protein in anthocyanin biosynthesis in Perilla frutescens // Plant Mol. Biol. 2002. V. 50. P. 485–495. https://doi.org/10.1023/A:1019850921627
Humphries J.A., Walker A.R., Timmis J.N., Orford S.J. Two WD-repeat genes from cotton are functional homologues of the Arabidopsis thaliana TRANSPARENT TESTA GLABRA1 (TTG1) gene // Plant Mol. Biol. 2005. V. 57. P. 67–81. https://doi.org/10.1007/s11103-004-6768-1
Dressel A., Hemleben V. Transparent testa glabra 1 (TTG1) and TTG1-like genes in Matthiola incana R. Br. and related Brassicaceae and mutation in the WD-40 motif // Plant Biol. 2009. V. 11. P. 204–212. https://doi.org/10.1111/j.1438-8677.2008.00099.x
Carey C.C., Strahle J.T., Selinger D.A., Chandler V.L. Mutations in the pale aleurone color1 regulatory gene of the Zea mays anthocyanin pathway have distinct phenotypes relative to the functionally similar TRANSPARENT TESTA GLABRA1 gene in Arabidopsis thaliana // Plant Cell. 2004. V. 16. P. 450–464. https://doi.org/10.1105/tpc.018796
Vetten de F.N., Quattrocchio J., Mol R., Koes R. The an11 locus controlling flower pigmentation in petunia encodes a novel WD-repeat protein conserved in yeast, plants, and animals // Genes Dev. 1997. V. 11. P. 1422–1434.
Sun B., Zhu Z., Liu R. et al. TRANSPARENT TESTA GLABRA1 (TTG1) regulates leaf trichome density in tea Camellia sinensis // Nordic J. Botany. 2020. V. 38. Is. 1. P. 1–10. https://doi.org/10.1111/njb.02592
Huang X., Yan H., Zhai L., Yi Y. GLABROUS1 from Rosa roxburghii Tratt regulates trichome formation by interacting with the GL3/EGL3 protein // Gene. 2019. V. 692. P. 60–67. https://doi.org/10.1016/j.gene.2018.12.071
Liu S., Tian N., Li J. et al. Isolation and identification of novel genes involved in artemisinin production from flowers of Artemisia annua using suppression subtractive hybridization and metabolite analysis // Planta Medica. 2009. V. 75. Is. 14. P. 1542–1547. https://doi.org/10.1055/s-0029-1185809
Wang S., Wang J.-W., Yu N. et al. Control of plant trichome development by a cotton fiber MYB gene // The Plant Cell. 2004. V. 16. P. 2323–2334. https://doi.org/10.1105/tpc.104.024844.1
Wang Z., Yang Z., Li F. Updates on molecular mechanisms in the development of branched trichome in Arabidopsis and non-branched in cotton // Plant Biotechnol. J. 2019. V. 17. P. 1706–1722. https://doi.org/10.1111/pbi.13167
Wang M., Wang P., Tu L. et al. Multi-omics maps of cotton fibre reveal epigenetic basis for staged single-cell differentiation // Nucl. Acids Res. 2016. V. 44. P. 4067–4079.
Zhang F., Zuo K., Zhang J. et al. An L1 box binding protein, GbML1, interacts with GbMYB25 to control cotton fibre development // J. Experim. Bot. 2010. V. 61. P. 3599–3613.
Li Q., Cao C., Zhang C. et al. The identification of Cucumis sativus Glabrous 1 (CsGL1) required for the formation of trichomes uncovers a novel function for the homeodomain-leucine zipper I gene // J. Experim. Bot. 2015. V. 66. Is. 9. P. 2515–2526. https://doi.org/10.1093/jxb/erv046
Liu X., Ezra B., Cai Y., Ren H. Trichome-related mutants provide a new perspective on multicellular trichome initiation and development in cucumber (Cucumis sativus L.) // Front. Plant Sci. 2016. V. 7. P. 1–9. https://doi.org/10.3389/fpls.2016.01187
Cui J.-Y., Miao H., Ding L.-H. et al. A new glabrous gene (csgl3) identifiedin trichome development in Cucumber (Cucumis sativus L.) // PLoS One. 2016. V. 11. Is. 2. P. 1–13. https://doi.org/10.1371/journal.pone.0148422
Yan X., Zhang X., Lu M. et al. De novo sequencing analysis of the Rosa roxburghii fruit transcriptome reveals putative ascorbate biosynthetic genes and EST-SSR markers // Gene. 2015. V. 561. P. 54–62. https://doi.org/10.1016/j.gene.2015.02.054
Marks M.D., Feldmann K.A. Trichome development in Arabidopsis thaliana. I. T-DNA tagging of the GLABROUS1 gene // The Plant Cell. 1989. V. 1. Is. 11. P. 1043–1050. https://doi.org/10.2307/3869021
Zhao H., Wang X., Zhu D. et al. A single amino acid substitution in IIIf subfamily of basic helix-loop-helix transcription factor AtMYC1 leads to trichome and root hair patterning defects by abolishing its interaction with partner proteins in Arabidopsis // J. Biol. Chemistry. 2012. V. 287. Is. 17. P. 14109–14121. https://doi.org/10.1074/jbc.M111.280735
Dubos C., Stracke R., Grotewold E. et al. MYB transcription factors in Arabidopsis // Trends in Plant Sci. 2010. V. 15. Is. 10. P. 573–581. https://doi.org/10.1016/j.tplants.2010.06.005
Heim M.A. The basic helix-loop-helix transcription factor family in plants: A genome-wide study of protein structure and functional diversity // Mol. Biol. Evol. 2003. V. 20. Is. 5. P. 735–747. https://doi.org/10.1093/molbev/msg088
Yang D.-L., Yao J., Mei C.-S. et al. Plant hormone jasmonate prioritizes defense over growth by interfering with gibberellin signaling cascade // Proc. Natl Acad. Sci. USA. 2012. V. 109. Is. 19. P. 1192–1200. https://doi.org/10.1073/pnas.1201616109
Schellmann S., Schnittger A., Kirik V. et al. TRIPTYCHON and CAPRICE mediate lateral inhibition during trichome and root hair patterning in Arabidopsis // The EMBO J. 2002. V. 21. Is. 19. P. 5036–5046.https://doi.org/10.1093/emboj/cdf524
Wada T., Tachibana T., Shimura Y., Okada K. Epidermal cell differentiation in Arabidopsis determined by a Myb homolog // CPC. Science. 1997. V. 277. Is. 5329. P. 1113–1116. https://doi.org/10.1126/science.277.5329.1113
Wester K., Digiuni S., Geier F. et al. Functional diversity of R3 single-repeat genes in trichome development // Development. 2009. V. 136. Is. 9. P. 1487–1496. https://doi.org/10.1242/dev.021733
Morohashi K., Zhao M., Yang M. et al. Participation of the Arabidopsis bHLH factor GL3 in trichome initiation regulatory events // Plant Physiology. 2007. V. 145. Is. 3. P. 736–746. https://doi.org/10.1104/pp.107.104521
Wang S., Chen J.-G. Regulation of cell fate determination by single-repeat R3 MYB transcription factors in Arabidopsis // Frontiers Plant Sci. 2014. V. 5. P. 1–12. https://doi.org/10.3389/fpls.2014.00133
Neer E.J., Schmidt C.J., Nambudripad R., Smith T.F. Erratum: The ancient regulatory-protein family of WD-repeat proteins // Nature. 1994. V. 371. Is. 6495. P. 297–300. https://doi.org/10.1038/371297a
Gan Y., Liu C., Yu H., Broun P. Integration of cytokinin and gibberellin signalling by Arabidopsis transcription factors GIS, ZFP8 and GIS2 in the regulation of epidermal cell fate // Development. 2007. V. 134. Is. 11. P. 2073–2081. https://doi.org/10.1242/dev.005017
Zhou Z., Sun L., Zhao Y. et al. Zinc Finger Protein 6 (ZFP6) regulates trichome initiation by integrating gibberellin and cytokinin signaling in Arabidopsis thaliana // New Phytologist. 2013. V. 198. Is. 3. P. 699–708. https://doi.org/10.1111/nph.12211
Englbrecht C.C., Schoof H., Böhm S. Conservation, diversification and expansion of C2H2 zinc finger proteins in the Arabidopsis thaliana genome // BMC Genomics. 2004. V. 5. Is. 1. P. 1–17. https://doi.org/10.1186/1471-2164-5-39
Maes L., Nieuwerburgh F.C.W.V., Zhang Y. et al. Dissection of the phytohormonal regulation of trichome formation and biosynthesis of the antimalarial compound artemisinin in Artemisia annua plants // New Phytologist. 2011. V. 189. Is. 1. P. 176–189. https://doi.org/10.1111/j.1469-8137.2010.03466.x
Yoshida Y., Sano R., Wada T. et al. Jasmonic acid control of GLABRA3 links inducible defense and trichome patterning in Arabidopsis // Development. 2009. V. 136 Is. 6. P. 1039–1048. https://doi.org/10.1242/dev.030585
Sun W., Gao D., Xiong Y. et al. An AP2/ERF transcription factor, interacts with OsWOX3B and regulates trichome formation in rice // Mol. Plant. 2017. V. 10. P. 1417–1433. https://doi.org/10.1016/j.molp.2017.09.0153
Xie Y., Yu X., Jiang S. et al. OsGL6, a conserved AP2 domain protein, promotes leaf trichome initiation in rice // Biochem. Biophys. Res. Communications. 2020. V. 522. Is. 2. P. 1–8. https://doi.org/10.1016/j.bbrc.2019.11.125
Chalvin C., Drevensek S., Dron M. et al. Genetic control of glandular trichome development // Trends in Plant Sci. 2020. V. 25. P. 477–487. https://doi.org/10.1016/j.tplants.2019.12.025
Ewas M., Gao Y., Wang S. et al. Manipulation of SlMXl for enhanced carotenoids accumulation and drought resistance in tomato // Sci. Bull. 2016. V. 61. Is. 18. P. 1413–1418. https://doi.org/10.1007/s11434-016-1108-9
Xu J., Herwijnen Z.O.V, Drдger D.B. et al. SlMYC1 regulates type VI glandular trichome formation and terpene biosynthesis in tomato glandular cells // The Plant Cell. 2018. V. 30. Is. 12. P. 2988–3005. https://doi.org/10.1105/tpc.18.00571
Yang C., Li H., Zhang J. et al. A regulatory gene induces trichome formation and embryo lethality in tomato // Proc. Natl Acad. Sci. USA. 2011. V. 108. Is. 29. P. 11836–11841. https://doi.org/10.1073/pnas.1100532108
Chang J., Yu T., Yang Q. et al. Hair, encoding a single C2H2 zinc-finger protein, regulates multicellular trichome formation in tomato // Plant J. 2018. V. 96. Is. 1. P. 90–102. https://doi.org/10.1111/tpj.14018
Tominaga-Wada R., Ishida T., Wada T. New insights into the mechanism of development of Arabidopsis root hairs and trichomes // Intern. Rev. Cell and Mol. Biol. 2011. V. 286. P. 67–106. https://doi.org/10.1016/B978-0-12-385859-7.00002-1
Дополнительные материалы отсутствуют.