Генетика, 2020, T. 56, № 12, стр. 1387-1398
Филогенетические отношения видов Oxytropis секции Arctobia Северо-Востока Азии по данным секвенирования межгенных спейсеров хлоропластного и ITS ядерного геномов
А. Б. Холина 1, *, М. М. Козыренко 1, Е. В. Артюкова 1, В. В. Якубов 1, М. Г. Хорева 2, Е. А. Андриянова 2, О. А. Мочалова 2
1 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
2 Институт биологических проблем Севера Дальневосточного отделения
Российской академии наук
685000 Магадан, Россия
* E-mail: kholina@biosoil.ru
Поступила в редакцию 21.01.2020
После доработки 15.04.2020
Принята к публикации 09.06.2020
Аннотация
Три межгенных спейсера (psbA–trnH, trnL–trnF, trnS–trnG) хлоропластной ДНК (хпДНК) и внутренний транскрибируемый спейсер ядерной рибосомальной ДНК (ITS рДНК) использованы для изучения генетического разнообразия и филогенетических отношений видов Oxytropis czukotica, O. exserta, O. gorodkovii, O. kamtschatica, O. mertensiana, O. nigrescens, O. pumilio, O. revoluta и O. susumanica секции Arctobia рода Oxytropis. По данным хпДНК большинство популяций характеризуются низким и средним гаплотипическим (h изменяется от 0.154 до 0.583) и низким нуклеотидным (π изменяется от 0.0002 до 0.0050) разнообразием. Анализ генеалогических связей хлоротипов показал четкое разделение исследуемых таксонов и генетическую близость O. nigrescens с O. susumanica и O. kamtschatica с O. exserta, последние два наиболее дивергированы от всех других. У O. czukotica выявлено три риботипа ITS рДНК, у O. nigrescens и O. susumanica – один общий риботип, у всех остальных – по одному индивидуальному риботипу. По данным нуклеотидного полиморфизма маркеров двух геномов подтвержден видовой статус таксонов O. czukotica, O. gorodkovii и O. pumilio. Мы предполагаем, что O. susumanica является внутривидовым таксоном O. nigrescens. Выявленные генетическое сходство и различия O. revoluta, O. exserta, O. kamtschatica и их филогенетические связи не соответствуют делению секции Arctobia на подсекции, поэтому необходимы дополнительные комплексные исследования.
Метаарктическая амфиберингийская секция Arctobia Bunge подрода Oxytropis рода Oxytropis DC. включает 16 видов и подвидов на севере Азии и в Северной Америке [1–7]. Впервые описание секции, включающей виды O. nigrescens (Pall.) Fisch., O. pumilio (Pall.) Ledeb. и O. arctobia Bunge, представил Бунге [8]. Позднее Юрцев [2] провел ревизию секции и разделил ее на пять подсекций – Arctobia, Kamtschaticae, Revolutae, Podocarpae и Mertensiana [2, 3, 5]. Наиболее сложной с таксономической точки зрения среди них является самая крупная подсекция Arctobia с центральным видом O. nigrescens, который вместе с группой близких к нему видов O. czukotica Jurtz., O. gorodkovii Jurtz., O. pumilio и O. bryophila (Greene) Jurtz. составляет комплекс O. nigrescens s. l. [3, 5, 7]. Высокая фенотипическая пластичность видов комплекса приводит к тому, что границы между ними не всегда четко определены, поэтому у исследователей существуют противоречия в оценке ранга этих таксонов. Так, большинство американских исследователей от Барнеби [1] до Уэлша [4] относят все североамериканские растения комплекса O. nigrescens s. l. к O. nigrescens; шведский ботаник Хультен [9] – к O. nigrescens с тремя подвидами (subsp. bryophila, pygmaea, arctobia) и при этом отмечает, что их статус требует уточнения. Юрцев [3, 5], авторы проекта “Панарктическая флора” [7] и другие ботаники [6, 10] принимают все таксоны комплекса в ранге близких видов, так как они имеют ряд различий в морфологии и плоидности. Кроме того, Юрцев отмечает [3, 5], что большинство видов имеют парапатрические ареалы: O. nigrescens s. str. – от Енисея до Колымы, O. czukotica – от Колымы до Берингова пролива, O. gorodkovii – в районе Берингова пролива и далее на восток, O. pumilio – на п-ове Камчатка и северных и средних Курилах, O. bryophila – в основном на территории Юкона и Аляске. Малышев в более поздней работе [11] приводит, следуя Хультену [9], O. czukotica как синоним O. pumilio. Проведенный комплексный анализ морфологических признаков, анатомии семян и полиморфизма нуклеотидных последовательностей маркеров ядерного (CNGC5, LE и TRPT) и хлоропластного (matK) геномов видов секции Arctobia Северной Америки [12] показал наличие двух видов из комплекса O. nigrescens s. l. – O. gorodkovii и O. bryophila. Автор приводит O. czukotica как синоним O. bryophila. Однако разрешающая способность использованных в этой работе молекулярных маркеров была слабой.
Для реконструкции филогенетических отношений видов Oxytropis были также использованы внутренний транскрибируемый спейсер ядерной рибосомальной ДНК (ITS рДНК) [13], ITS рДНК + + matK хлоропластной ДНК (хпДНК) [14] и ITS рДНК + trnL–trnF хпДНК [15], однако это не привело к разрешению взаимоотношений видов даже на уровне секций. Нами проведен ряд исследований, основанных на сравнительном анализе нуклеотидного полиморфизма межгенных спейсеров psbA–trnH + trnL–trnF + trnS–trnG хпДНК, по результатам которых подтверждена видовая самостоятельность O. czukotica [16], изучены генетическое разнообразие и популяционная структура редких и эндемичных видов рода Oxytropis – O. chankaensis Jurtz. [17], O. triphylla (Pall.) Pers., O. bargusinensis Peschkova и O. interposita Sipl. [18], O. glandulosa Turcz. [19], пяти видов секции Orobia Bunge [20], реконструированы филогенетические связи внутри секций Xerobia Bunge [21] и Verticillares DC. [22]. Выявлены значительные различия в хлоропластном и ядерном (ITS рДНК) геномах O. ruthenica Vass. и O. kunashiriensis Kitam. секции Orobia, которые подтверждают самостоятельность этих видов, и установлено, что O. erecta Kom. и O. litoralis Kom. этой же секции представляют собой локальные фенотипы широко распространенного полиплоидного вида O. ochotensis Bunge [20]. Анализ генеалогических связей гаплотипов выявил четко обособленные эволюционные линии у O. glandulosa [19] и у O. ruthenica [20], что может указывать на активно идущие микроэволюционные процессы и/или наличие криптических видов.
Для Азиатской России Малышев [6] приводит девять видов секции Arctobia – O. bryophila, O. czukotica, O. exserta Jurtz., O. gorodkovii, O. kamtschatica Hult., O. mertensiana Turcz., O. nigrescens, O. pumilio, O. revoluta Ledeb. Кроме них он упоминает еще три таксона – O. siegizmundii N.S. Pavlova, O. susumanica Jurtz. и O. czerskii Jurtz., первый не имеет широкого ареала (только в Корякии, верховье р. Айнын на севере Пенжинской губы), второй и третий встречены лишь однажды (Сусуманский район Магаданской обл. и хребет Черского в верховье р. Индигирка соответственно), поэтому эти таксоны нуждаются в дополнительном изучении для признания самостоятельными видами. Большинство видов секции диплоиды: у O. exserta, O. mertensiana, O. nigrescens, O. pumilio и O. revoluta 2n = 16; у O. czukotica и O. gorodkovii 2n = 16 и 2n = 32, у O. kamtschatica 2n = 16 и 2n = 96 [6]; для O. bryophila, O. siegizmundii, O. susumanica и O. czerskii – данных нет.
Целью настоящей работы является изучение генетического разнообразия, популяционной структуры и реконструкция филогенетических отношений видов Oxytropis секции Arctobia северо-востока Азии по данным полиморфизма нуклеотидных последовательностей межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG хпДНК и ITS рДНК.
МАТЕРИАЛЫ И МЕТОДЫ
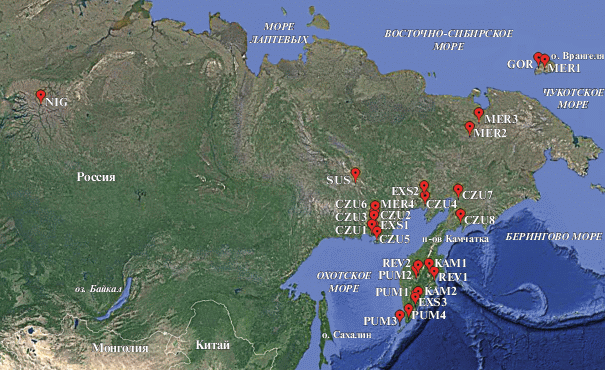
Материалом служили 170 растений видов O. czukotica (86 образцов), O. exserta (13), O. gorodkovii (3), O. kamtschatica (9), O. mertensiana (22), O. nigrescens (3), O. pumilio (17), O. revoluta (11) и O. susumanica (6) секции Arctobia подрода Oxytropis из 26 природных местонахождений (рис. 1). В данной работе нами использована таксономическая классификация секции Arctobia по Малышеву [6]. Размер выборки, код популяции и географические координаты даны в табл. 1.
Таблица 1.
Исследуемые популяции видов Oxytropis секции Arctobia и параметры генетического разнообразия по данным хпДНК
| Вид, местонахождение (число образцов) |
Координаты с.ш., в.д. |
Код | Гаплотип (число образцов) |
Разнообразие (стандартное отклонение) |
|
|---|---|---|---|---|---|
| гаплотипическое | нуклеотидное | ||||
| O. czukotica | |||||
| Магаданская обл., окр. г. Магадана, р. Медвежка (13) | 59.58°, 150.86° | CZU1 | A1 (12), A2 (1) | 0.154 (0.126) | 0.0004 (0.0003) |
| Магаданская обл., Хасынский р-н, гора Нух (15) | 60.36°, 151.32° | CZU2 | A3 (11), A4 (1), A5 (2), A6 (1) | 0.467 (0.148) | 0.0023 (0.0013) |
| Магаданская обл., Хасынский р-н, Сопка 1200 (7) | 60.32°, 151.25° | CZU3 | A7 (5), A8 (2) | 0.476 (0.171) | 0.0012 (0.0008) |
| Магаданская обл., Северо-Эвенский р-н, восточнее р. М. Вархалам (7) | 61.95°, 159.96° | CZU4 | A9 (3), A10 (4) | 0.571 (0.119) | 0.0002 (0.0002) |
| Магаданская обл., Ольский р-н, п-ов Кони, мыс Алевина (17) | 58.97°, 151.81° | CZU5 | A11 (2), A12 (1), A13 (4), A14 (8), A15 (1), A16 (1) | 0.743 (0.089) | 0.0011 (0.0007) |
| Магаданская обл., Хасынский р-н, гора Медвежья (5) | 61.13°, 151.47° | CZU6 | A6 (1), A7 (3), A17 (1) | 0.700 (0.218) | 0.0050 (0.0032) |
| Камчатский край, Пенжинский р-н, нижнее течение р. Пенжины (14) | 62.44°, 165.63° | CZU7 | A7 (9), A18 (3), A19 (2) | 0.560 (0.124) | 0.0003 (0.0002) |
| Камчатский край, Олюторский р-н, окр. пос. Тиличики (8) | 60.44°, 166.03° | CZU8 | A19 (7), A20 (1) | 0.250 (0.180) | 0.0012 (0.0008) |
| O. exserta | |||||
| Магаданская обл., Ольский р-н, окр. пос. Нюкля (7) | 59.57°, 151.23° | EXS1 | A21 (5), A22 (1), A23 (1) | 0.524 (0.209) | 0.0020 (0.0013) |
| Магаданская обл., Северо-Эвенский р-н, верхнее течение р. Гижига (2) | 62.72°, 159.83° | EXS2 | A21 (1), A24 (1) | – | – |
| Камчатка, Елизовский р-н, влк. Вилючинская сопка (4) | 52.71°, 158.28° | EXS3 | A25 (1), A26 (3) | – | – |
| O. gorodkovii | |||||
| Чукотка, о. Врангеля, бух. Сомнительная (3) | 71.15°, 179.29° | GOR | A27 (1), A28 (1), A29 (1) | – | – |
| O. kamtschatica | |||||
| Камчатка, влк. Ключевская сопка (4) | 56.06°, 160.62° | KAM1 | A30 (1), A31 (3) | – | – |
| Камчатка, влк. Авачинская сопка (5) | 53.26°, 158.84° | KAM2 | A32 (5) | 0.000 (0.000) | 0.0000 (0.0000) |
| O. mertensiana | |||||
| Чукотка, о. Врангеля, бух. Сомнительная (2) | 71.06°, 179.63° | MER1 | A33 (1), A34 (1) | – | – |
| Чукотка, Билибинский р-н, окр. оз. Липчиквыгытын (9) | 67.00°, 167.58° | MER2 | A34 (7), A35 (2), | 0.389 (0.164) | 0.0003 (0.0003) |
| Чукотка, Билибинский р-н, р. Малый Анюй (10) | 67.90°, 169.14° | MER3 | A35 (10) | 0.000 (0.000) | 0.0000 (0.0000) |
| Магаданская обл., Хасынский р-н, Ольское плато, верховье р. Ола (1) | 60.63°, 151.37° | MER4 | A33 (1) | – | – |
| O. nigrescens | |||||
| Красноярский край, плато Путорана, окр. оз. Аян (3) | 69.01°, 94.25° | NIG | A36 (1), A37 (1), A38 (1) | ||
| O. pumilio | |||||
| Камчатка, влк. Авачинская сопка (9) | 53.12°, 158.47° | PUM1 | A39 (6), A40 (1), A41 (1), A42 (1) | 0.583 (0.183) | 0.0005 (0.0004) |
| Камчатка, окр. пос. Эссо, Козыревский хребет (4) | 55.53°, 158.47° | PUM2 | A43 (1), A44 (1), A45 (1), A46 (1) | – | – |
| Курильский архипелаг, о-в Атласова, влк. Алаид (3) | 50.86°, 155.56° | PUM3 | A47 (3) | – | – |
| Камчатка, влк. Ильинский (1) | 51.50°, 157.20° | PUM4 | A48 (1) | – | – |
| O. revoluta | |||||
| Камчатка, Толбачинский дол, р-н горы Высокой (3) | 55.30°, 161.51° | REV1 | A49 (3) | – | – |
| Камчатка, окр. пос. Эссо, Козыревский хребет (8) | 55.88°, 158.80° | REV2 | A50 (7), A51 (1) | 0.250 (0.180) | 0.0002 (0.0002) |
| O. susumanica | |||||
| Магаданская обл., Сусуманский р-н, оз. Момонтай (6) | 63.72°, 148.12° | SUS | A52 (3), A53 (1), A54 (1), A55(1) | 0.800 (0.172) | 0.0006 (0.0005) |
Методы выделения ДНК, амплификации и секвенирования межгенных спейсеров psbA–trnH, trnL–trnF и trnS–trnG приведены в наших предыдущих работах [16, 18, 19]. Нуклеотидные последовательности прямых и обратных цепей определяли на генетическом анализаторе ABI 3500 (Applied Biosystems, США), затем редактировали и собирали с помощью пакета программ Staden Package 1.5 [23]. Для каждого образца последовательности регионов выравнивали в программе SeaView 4.7 [24]. Матрицу объединенных последовательностей использовали для расчета числа гаплотипов, гаплотипического (h) и нуклеотидного (π) разнообразия (для популяций с числом образцов пять и более), и анализа молекулярной дисперсии (AMOVA) в пакете программ Arlequin 3.5 [25]. Статистическую значимость (P) оценивали на основе 1023 пермутаций. Степень дивергенции на основе нуклеотидных замен (Dxy) определяли в программе DnaSP 5.0 [26]. Генеалогические связи гаплотипов хпДНК анализировали методом медианного связывания (Median-Joining, MJ) в программе Network 5.0 [27], кодируя каждую делецию или вставку, независимо от их размера, как единичное мутационное событие. В качестве внешней группы использовали полученные нами ранее [18] нуклеотидные последовательности межгенных спейсеров psbA–trnH, trnL–trnF, trnS–trnG хпДНК (номера в GenBank LT856572, LT856585, LT856598 соответственно) O. glabra секции Mesogaea Bunge подрода Phacoxytropis Bunge.
Регион ITS рДНК амплифицирован у 87 образцов: O. czukotica (33 образца), O. exserta (10), O. gorodkovii (3), O. kamtschatica (6), O. mertensiana (10), O. nigrescens (2), O. pumilio (13), O. revoluta (6) и O. susumanica (4), представляющих все выявленные в данной работе гаплотипы хпДНК, с праймерами ITS1 и ITS4, в реакционных условиях и температурном режиме, приведенных в работе [28]. Редактирование, сборку, выравнивание и анализ последовательностей ITS проводили с помощью программ, приведенных выше. Филогенетический анализ последовательностей проводили методом максимальной экономии (Maximum Parsimony, MP), используя эвристический поиск оптимальной топологии, в пакете программ PAUP 4.0b10 [29]. Достоверность порядка ветвления оценивали с помощью бутстреп-анализа 1000 альтернативных деревьев (Bootstrap Percentage, BP, %). В качестве внешней группы использовали последовательность O. glabra из GenBank (номер LC213354).
Работа проводилась с использованием оборудования ЦКП “Биотехнология и генетическая инженерия” ФНЦ Биоразнообразия ДВО РАН.
РЕЗУЛЬТАТЫ
Межгенные спейсеры psbA–trnH, trnL–trnF и trnS–trnG хпДНК были успешно амплифицированы и секвенированы для всех 170 образцов девяти таксонов. Нуклеотидные последовательности характеризуются относительно низкой нуклеотидной изменчивостью и разной длиной вследствие присутствия моно- и динуклеотидных повторов, коротких (от 4 до 7 нуклеотидов) и довольно протяженных (до 188 нуклеотидов) инделей. Общая длина объединенных последовательностей трех регионов составила 2579 сайтов (1–472, 473–1259 и 1260–2579 соответственно), из них 2192 мономорфных, 367 сайтов приходятся на индели и 20 вариабельных. 18 нуклеотидных замен (позиции 214, 389, 509, 702, 1160, 1314, 1437, 1534, 1680, 1947, 2008, 2012, 2024, 2025, 2027, 2215, 2403 и 2411) информативны согласно методу максимальной экономии, две замены (позиции 1919 и 2176) единичные.
Большинство популяций характеризуются низким и средним гаплотипическим разнообразием (h изменяется от 0.154 до 0.583), кроме CZU5 (h = 0.743) и CZU6 (h = 0.700) O. czukotica, SUS (h = 0.800) O. susumanica, и низким нуклеотидным (π изменяется от 0.0002 до 0.0050) разнообразием (табл. 1). Популяции KAM2 O. kamtschatica и MER3 O. mertensiana оказались мономорфными. Дивергенция нуклеотидных последовательностей (Dxy) между популяциями каждого из видов O. kamtschatica, O. mertensiana и O. revoluta отсутствует. У O. czukotica наибольшие значения Dxy (среднее число нуклеотидных различий (число фиксированных различий) и среднее число нуклеотидных замен на один сайт) определены между парами популяций CZU1–CZU6 и CZU5–CZU6 (1.600 (1) и 0.00067 соответственно), у O. exserta – 3.286 (3) и 0.00132 между EXS1–EXS3, у O. pumilio – 1.250 (1) и 0.00056 между PUM2 и каждой из популяций PUM1, PUM3 и PUM4. Согласно результатам AMOVA (табл. 2), у каждого из видов O. czukotica, O. exserta и O. kamtschatica основная доля всей генетической изменчивости (70–85%) приходится на межпопуляционные различия, которые достигают 94% у O. revoluta и более 97% у O. pumilio. Иерархический AMOVA девяти исследуемых видов показал высокую дифференциацию между видами – более 80% изменчивости объясняется межвидовыми различиями (табл. 2). В табл. 3 приведены значения дивергенции нуклеотидных последовательностей между видами. Максимальная дивергенция определена между парами, образованными каждым из видов O. kamtschatica и O. exserta со всеми другими, минимальная – между O. nigrescens и O. susumanica.
Таблица 2.
Распределение генетической изменчивости между группами Oxytropis секции Arctobia по данным хпДНК
| Группировка | Источник изменчивости | D.f. | Изменчивость, % | Индексы фиксации |
|---|---|---|---|---|
| O. czukotica | Между популяциями | 7 | 75.50 | FST = 0.75503* |
| Внутри популяций | 78 | 24.50 | ||
| O. exserta | Между популяциями | 2 | 70.31 | FST = 0.70314** |
| Внутри популяций | 10 | 29.69 | ||
| O. kamtschatica | Между популяциями | 1 | 84.64 | FST = 0.84642*** |
| Внутри популяций | 7 | 15.36 | ||
| O. mertensiana | Между популяциями | 3 | 69.97 | FST = 0.69973** |
| Внутри популяций | 18 | 30.03 | ||
| O. pumilio | Между популяциями | 3 | 97.62 | FST = 0.97624* |
| Внутри популяций | 13 | 2.38 | ||
| O. revoluta | Между популяциями | 1 | 93.86 | FST = 0.93857*** |
| Внутри популяций | 9 | 6.14 | ||
| O. nigrescens и O. susumanica | Между популяциями | 1 | 91.41 | FST = 0.91413*** |
| Внутри популяций | 7 | 8.59 | ||
| O. exserta и O. kamtschatica | Между группами | 1 | 54.03 | FCT = 0.54029 ns |
| Между популяциями внутри групп | 3 | 34.06 | FSC = 0.74085* | |
| Внутри популяций | 17 | 11.91 | FST = 0.88087* | |
| Девять исследуемых видов | Между группами | 8 | 80.73 | FCT = 0.80735* |
| Между популяциями внутри групп | 17 | 17.00 | FSC = 0.88218* | |
| Внутри популяций | 144 | 2.27 | FST = 0.97730* |
Таблица 3.
Нуклеотидная дивергенция между видами Oxytropis секции Arctobia по данным хпДНК
| Вид | O. czukotica | O. exserta | O. gorodkovii | O. kamtschatica | O. mertensiana | O. nigrescens | O. pumilio | O. revoluta | O. susumanica |
|---|---|---|---|---|---|---|---|---|---|
| O. czukotica | – | 9.549 (8) | 3.395 (2) | 11.395 (10) | 2.395 (1) | 2.729 (1) | 2.506 (0) | 2.395 (1) | 3.372 (1) |
| O. exserta | 0.00402 | – | 8.154 (8) | 6.462 (5) | 9.154 (9) | 9.487 (9) | 9.330 (8) | 9.154 (9) | 10.154 (10) |
| O. gorodkovii | 0.00142 | 0.00342 | – | 10.000 (10) | 3.000 (3) | 3.333 (3) | 2.176 (1) | 3.000 (3) | 4.000 (4) |
| O. kamtschatica | 0.00480 | 0.00260 | 0.00420 | – | 11.000 (11) | 11.333 (11) | 11.176 (10) | 11.000 (11) | 12.000 (12) |
| O. mertensiana | 0.00100 | 0.00386 | 0.00126 | 0.00463 | – | 2.333 (2) | 3.176 (2) | 2.000 (2) | 3.000 (3) |
| O. nigrescens | 0.00114 | 0.00399 | 0.00139 | 0.00476 | 0.00098 | – | 3.510 (2) | 2.333 (2) | 1.333 (1) |
| O. pumilio | 0.00112 | 0.00421 | 0.00098 | 0.00504 | 0.00143 | 0.00158 | – | 3.176 (2) | 4.176 (3) |
| O. revoluta | 0.00100 | 0.00384 | 0.00125 | 0.00461 | 0.00084 | 0.00097 | 0.00143 | – | 3.000 (3) |
| O. susumanica | 0.00141 | 0.00426 | 0.00167 | 0.00504 | 0.00126 | 0.00056 | 0.00187 | 0.00125 | – |
Анализ 170 последовательностей объединенной матрицы выявил 55 гаплотипов (хлоротипов), из них 30 (54.5%) уникальные (табл. 1). Последовательности трех регионов хпДНК каждого хлоротипа депонированы в GenBank (LR742876–LR743040). O. czukotica принадлежит 20 хлоротипов (A1–A20), O. exserta – 6 (A21–A26), O. gorodkovii – 3 (A27–A29), O. kamtschatica – 3 (A30–A32), O. mertensiana – 3 (A33–A35), O. nigrescens – 3 (A36–A38), O. pumilio – 10 (A39–A48), O. revoluta – 3 (A49–A51), O. susumanica – 4 (A52–A55). Общих хлоротипов у таксонов не обнаружено (табл. 1). В медианной сети генеалогических связей (рис. 2,а) хлоротипы формируют семь гаплогрупп (I–VII). Гаплогруппы I, II, V, VI, VII образованы хлоротипами согласно видовой принадлежности, гаплогруппа III объединяет хлоротипы O. nigrescens и O. susumanica, гаплогруппа IV – O. kamtschatica и O. exserta. Каждая из двух последних гаплогрупп имеет маркерные нуклеотидные замены и индели: III – С в позиции 1160 объединенной матрицы; IV – 6 нуклеотидных замен (G в позициях 509 и 2024, T в позициях 1437 и 1680, A в позиции 2012 и C в позиции 2025) и пять инделей, из них четыре коротких (от 1 до 7 нуклеотидов) и один длинный (до 100 нуклеотидов). В этих гаплогруппах хлоротипы формируют группы согласно таксономической принадлежности. Так, группы хлоротипов O. nigrescens и O. susumanica разделены восемью мутационными шагами, а O. kamtschatica и O. exserta – семью мутационными шагами. По результатам AMOVA (табл. 2), основная доля генетической изменчивости приходится на изменчивость между O. nigrescens и O. susumanica (более 91%). Иерархический AMOVA групп гаплотипов O. kamtschatica и O. exserta показал, что около 34% приходится на межпопуляционную компоненту внутри вида и около 12% – на внутрипопуляционную, а 54% изменчивости обусловлены различиями между видами, но эти различия статистически незначимые (табл. 2). Гаплогруппа IV наиболее генетически удалена от всех других гаплогрупп, что обусловлено высокой нуклеотидной дивергенцией O. exserta и O. kamtschatica от всех других видов (табл. 3). Например, от самого близкого хлоротипа А6 O. czukotica ее отделяют 20 мутационных шагов и пять инделей (рис. 2,а).
Рис. 2.
Филогенетические связи видов Oxytropis секции Arctobia. а – генеалогическая сеть гаплотипов хпДНК (А1–А55), построенная методом медианного связывания. Размер окружностей отражает частоту встречаемости гаплотипов, маленькие черные кружки – медианные векторы, поперечные тонкие линии на ветвях – мутационные события, черные толстые линии – индели. Цифры около тонких и толстых линий – число полиморфных сайтов и инделей. Пунктирной линией обозначены гаплогруппы I–VII и группы гаплотипов видов, образующих одну гаплогруппу. * Мутации для O. glabra, используемой в качестве внешней группы, не указаны и не рассматриваются; б – филогенетическое дерево риботипов ITS рДНК (R1–R10), построенное методом максимальной экономии. Числами в узлах ветвления обозначены значения индекса бутстрепа (выше 50%). Код популяции см. табл. 1. В качестве внешней группы использована ITS последовательность O. glabra (номер в GenBank LC213354).
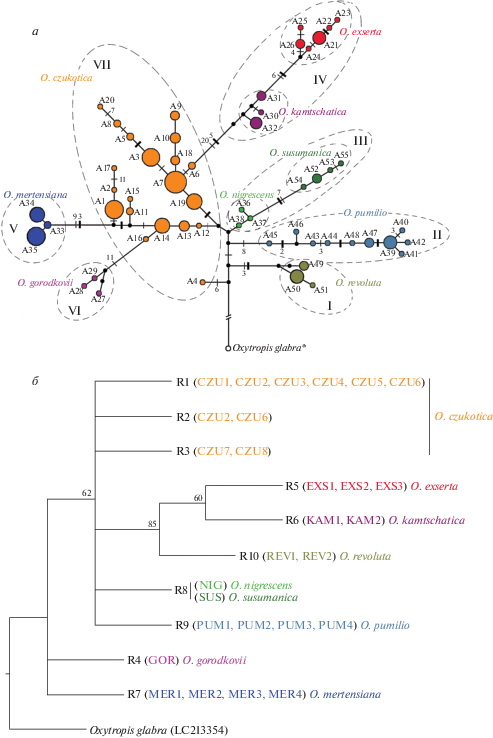
Выявлены видоспецифичные молекулярные маркеры у O. revoluta (A в позиции 214), O. gorodkovii (T в позиции 721), O. mertensiana (A в позиции 2411 и три коротких (4–6 нуклеотидов) инделя), O. kamtschatica (G в позициях 1314 и 2048, A в позициях 2073 и 2075, C в позиции 1534 и делеция трех нуклеотидов), O. exserta (T в позициях 2048, 2073, 2075 и вставка трех нуклеотидов). Таксоны O. nigrescens и O. susumanica имеют, как мы отмечали выше, одну общую замену. У O. czukotica и O. pumilio видоспецифичных маркеров нет, но необходимо отметить, что у последнего обнаружены специфичные для отдельных популяций маркеры. Так, в PUM1 обнаружена вставка семи нуклеотидов (AAGTATT, позиции 400–406), в PUM2 – нуклеотидная замена (С в позиции 828, у всех других А), в PUM1, PUM3 и PUM4 – делеция 188 нуклеотидов (позиции 707–894). В медианной сети распределение хлоротипов у O. pumilio соответствует популяционной принадлежности, а у O. czukotica – нет (рис. 2,а). Таким образом, анализ генеалогических связей хлоротипов выявил четкое разделение девяти видов Oxytropis секции Arctobia и показал генетическую близость O. nigrescens с O. susumanica и O. kamtschatica с O. exserta.
Нуклеотидные последовательности ITS рДНК получены у 87 образцов, представляющих все выявленные в данном исследовании гаплотипы хпДНК. Длина ITS составила 603 пн, кроме представителей видов O. exserta, O. kamtschatica и O. revoluta (602 пн), в последовательностях которых выявлена делеция одного нуклеотида (G в позиции 173). Из 603 сайтов 11 были вариабельными и информативными согласно методу максимальной экономии: шесть замен (позиции 76, 103, 116, 119, 160, 200) в ITS1 и пять (позиции 418, 427, 458, 484, 555) в ITS2. Выявлено 10 гаплотипов (риботипов), последовательности которых депонированы в GenBank (MN784407–MN784416). O. czukotica принадлежит три риботипа (R1–R3), O. nigrescens и O. susumanica имеют один общий риботип (R8), у всех остальных видов по одному индивидуальному риботипу: O. gorodkovii – R4, O. exserta – R5, O. kamtschatica – R6, O. mertensiana – R7, O. pumilio – R9, O. revoluta – R10. В результате филогенетического анализа риботипов МР-методом получено пять одинаково экономных деревьев, строгий консенсус которых представлен на рис. 2,б (длина – 20 шагов, индекс соответствия CI = 0.8500; индекс гомоплазии HI = 0.1500; индекс удерживания RI = 0.7000). Риботипы видов секции Arctobia отличаются четырьмя маркерными заменами от риботипа O. glabra секции Mesogaea, однако монофилетичность секции Arctobia не имеет статистически значимой поддержки. Обособленное положение занимают риботипы R4 O. gorodkovii и R7 O. mertensiana. Риботипы остальных видов формируют слабо поддержанную кладу (BP 62%), в которой только риботипы R10 O. revoluta, R5 O. exserta и R6 O. kamtschatica образуют филогруппу с высокой поддержкой (BP 87%). Риботип O. revoluta является сестринским к слабо поддержанной группе (BP 60%) риботипов O. exserta и O. kamtschatica. Филогенетические связи риботипов R1–R3 O. czukotica и R9 O. pumilio между собой и всеми другими риботипами в этой кладе остались неразрешенными (рис. 2,б).
ОБСУЖДЕНИЕ
Проведенный нами анализ полиморфизма нуклеотидных последовательностей межгенных спейсеров psbA–trnH + trnL–trnF + trnS–trnG хпДНК выявил, что большинство популяций исследуемых видов секции Arctobia характеризуются низким и средним гаплотипическим и низким нуклеотидным разнообразием, две популяции были мономорфными (табл. 1).
Невысокий уровень генетического разнообразия может быть обусловлен взаимным влиянием ряда факторов, таких как экологическая приуроченность, колебания численности и связанный с этим дрейф генов, эволюционная история видов. Например, растения O. czukotica – ацидофилы, обитают на кислых горных породах, растения O. gorodkovii – кальцефилы, характерны для карбонатных пород [2, 3], вследствие чего возникает фрагментированность популяции. Основная часть видов секции Arctobia обитает на территории, испытавшей интенсивное оледенение в плейстоцене [3], поэтому они перенесли резкое сокращение и последующее восстановление популяций из малого числа основателей. Определенный нами высокий (от 70 до 98%) уровень межпопуляционной изменчивости у O. czukotica, O. exserta, O. kamtschatica, O. revoluta и O. pumilio как раз и объясняется изоляцией популяций в совокупности с дрейфом генов.
В популяциях O. czukotica уровень гаплотипического разнообразия изменяется от низкого до высокого (табл. 1). У этого преимущественно тетраплоидного вида (в 28 местообитаниях на Чукотке, о-ве Врангеля и Колымском нагорье 2n = 32 [30] и только на горе Продолговатая и вулкане Ключевская сопка Камчатского края 2n = 16 [31]) отмечен высокий уровень морфологической изменчивости [3]. Большинство полиплоидных видов характеризуются более высоким уровнем генетического полиморфизма и, как следствие, большей устойчивостью к суровым условиям существования и повышенной способностью к адаптациям [32]. Вероятно, в результате полиплоидизации O. czukotica оказался достаточно устойчивым к климатическим флуктуациям плейстоцена [33], распространился на огромной территории и сохранил уровень полиморфизма предкового вида. Распределение хлоротипов у O. czukotica, не соответствующее популяционной принадлежности (рис. 2,а), обусловлено, скорее всего, непрерывным потоком генов между популяциями через цепь промежуточных локальных местообитаний. Филогенетические взаимоотношения риботипов ITS рДНК O. czukotica как внутривидовые, так и с другими видами секции остались неразрешенными (рис. 2,б). Это может быть связано с тем, что обычно процесс расхождения линий ядерной ДНК протекает дольше из-за ее бипарентального наследования и большей эффективной численности популяции [34].
Таксоны O. nigrescens и O. susumanica имеют разные хлоротипы (А36–А38 и А52–А55 соответственно) и общий риботип R8 (табл. 1, рис. 2) и являются потомками одной анцестральной формы. Учитывая минимальную нуклеотидную дивергенцию (табл. 3) и присутствие одной и той же маркерной нуклеотидной замены (С в позиции 1160), можно предположить, что узколокальный эндемик O. susumanica является внутривидовым таксоном широкоареального O. nigrescens. На основе морфологических признаков Малышев [6] считал O. susumanica аберрантом O. nigrescens. Для уточнения таксономического статуса O. susumanica необходимы дополнительные морфологические и генетические исследования расширенной выборки.
Четкое расхождение хлоротипов O. nigrescens, O. czukotica, O. gorodkovii и O. pumilio комплекса O. nigrescens s. l. северо-востока Азии (рис. 2,а), отсутствие общих хлоротипов, выявленные специфичные для отдельных популяций маркеры у O. pumilio и видоспецифичные у O. nigrescens и O. gorodkovii, наличие у всех индивидуальных риботипов ITS рДНК – все в целом согласуется с мнением ряда ботаников [3, 5–7, 10] о видовой самостоятельности таксонов O. gorodkovii, O. pumilio и O. czukotica и с результатами наших предыдущих молекулярно-генетических исследований [16].
Выявленное распределение в медианной сети хлоротипов O. revoluta, O. exserta и O. kamtschatica (рис. 2,а) не соответствует делению секции Arctobia на подсекции: первые два относятся к подсекции Revolutae, последний – к подсекции Kamtschaticae [2, 3]. Однако и ранее отмечалось, что по степени обособленности O. revoluta и O. exserta могли бы трактоваться как два монотипных ряда [2], что виды O. exserta и O. kamtschatica морфологически очень близки и часто неотличимы в фазе цветения [3]. Проведенное нами сравнение морфологических признаков, приведенных Павловой [10], показало, что по многим из них O. exserta более близка к O. kamtschatica, чем к O. revoluta. Так, растения O. exserta и O. kamtschatica – высотой до 15–20 см, с характерным обильным опушением, многоцветковыми соцветиями (до 5–6 цветков) и опушенными белыми волосками бобами с широкой брюшной перегородкой. Растения O. revoluta – высотой до 10 см, слабоопушенные или почти голые, с малоцветковыми соцветиями (2–3 цветка) и опушенными черными или рыжеватыми волосками бобами с узкой брюшной перегородкой. Выявленная высокая нуклеотидная дивергенция хпДНК O. exserta и O. kamtschatica от всех других видов (табл. 3), а также общие маркерные нуклеотидные замены и индели, объединение в одну гаплогруппу при сохранении видовой самостоятельности (рис. 2,а) и близость риботипов ITS рДНК (рис. 2,б) – все в совокупности указывает на необходимость пересмотра деления секции Arctobia на подсекции. Кроме того, риботип O. revoluta образует одну статистически высокоподдержанную филогруппу с риботипами O. exserta и O. kamtschatica, но обособляется раньше (рис. 2,б). Это противоречит представлениям Юрцева [2, 5], который полагал, что O. kamtschatica является наиболее близким видом к предковому типу секции Arctobia и O. revoluta произошла от O. exserta. Полученные нами данные свидетельствуют, что O. kamtschatica и O. exserta являются более молодыми видами, а O. czukotica, O. pumilio и O. revoluta – более древними.
Таким образом, данные, полученные на основе полиморфизма маркеров хлоропластного и ядерного геномов, согласуются с существующим мнением о видовом статусе таксонов O. czukotica, O. gorodkovii и O. pumilio. Очевидна необходимость пересмотра деления секции Arctobia на подсекции, а также проведения дополнительных исследований O. susumanica на основе комплексного анализа морфологических и генетических данных расширенной выборки растений для точного установления статуса этого таксона.
Авторы выражают благодарность И.Н. Поспелову и Е.Б. Поспеловой за предоставление образцов O. nigrescens с плато Путорана.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Barneby R.C. A revision of the North American species of Oxytropis DC. // Proc. Calif. Acad. Sci. 1952. V. 27. № 4. P. 177–312.
Юрцев Б.А. Конспект системы и новые таксоны секции Arctobia рода Oxytropis (Fabaceae) // Бот. журн. 1985. Т. 70. № 3. С. 394–397.
Юрцев Б.А. Oxytropis DC. // Арктическая флора СССР. Л.: Наука, 1986. Вып. 9. Ч. 2. С. 61–146.
Welsh S.L. Oxytropis DC. Names, basionyms, types, and synonyms. Flora North America project // Great Basin Nat. 1991. V. 51. № 4. P. 377–396. https://doi.org/10.2307/41712682
Yurtsev B.A. Survey of arctic legumes with emphasis on the species concept in Oxytropis // Norwegian Academy of Science and Letters. 1999. V. 38. P. 295–318.
Малышев Л.И. Разнообразие рода Остролодка (Oxytropis) в Азиатской России // Turczaninowia. 2008. Т. 11. № 4. С. 5–141.
Elven R., Murray D.M. Annotated checklist of the Panarctic Flora (PAF) vascular plants / Ed. Elven. R., 2011. Available from http://www.nhm.uio.no/english/research/infrastructure/paf.
Bunge Al. Species generis Oxytropis DC. // Mem. Acad. Sci. Petersb. (Sci. Phys. Math.). Ser. 7. 1874. V. 22. № 1. 166 p.
Hulten E. Flora of Alaska and Neighboring Territories. Stanford: Stanford Univ. Press, 1968. 1032 p.
Павлова Н.С. Остролодочник – Oxytropis DC. // Сосудистые растения советского Дальнего Востока. Л.: Наука, 1989. Т. 4. С. 236–280.
Малышев Л.И. Род Oxytropis DC. // Конспект флоры Азиатской России: Сосудистые растения. Новосибирск: Изд-во СО РАН, 2012. С. 237–248.
Meyers Z.J. A contribution to the taxonomy and phylogeny of Oxytropis section Arctobia (Fabaceae) in North America: Master Dissertation. Fairbanks, Alaska: Univ. Alaska Fairbanks, 2012. 155 p.
Archambault A., Strömvik M.V. Evolutionary relationships in Oxytropis species, as estimated from the nuclear ribosomal internal transcribed spacer (ITS) sequences point to multiple expansions into the Arctic // Botany. 2012. V. 90. № 8. P. 770–779. https://doi.org/10.1139/B2012-023
Tekpinar A., Karaman Erkul S., Aytac Z., Kaya Z. Phylogenetic relationships between Oxytropis DC. and Astragalus L. species native to an Old World diversity center inferred from nuclear ribosomal ITS and plastid matK gene sequences // Turk. J. Biol. 2016. V. 40. P. 250–263. https://doi.org/10.3906/biy-1502-5
Shavvon R.S., Kazempour-Osaloo S., Maassoumi A.A. et al. Increasing phylogenetic support for explosively radiating taxa: The promise of high-throughput sequencing for Oxytropis (Fabaceae) // J. Syst. Evol. 2017. V. 55. № 4. P. 385–404. https://doi.org/10.1111/jse.12269
Холина А.Б., Козыренко М.М., Артюкова Е.В. и др. Филогенетические взаимоотношения видов Oxytropis DC. subg. Oxytropis и Phacoxytropis (Fabaceae) Азиатской России на основе анализа нуклеотидных последовательностей межгенных спейсеров хлоропластного генома // Генетика. 2016. Т. 52. № 8. С. 895–909.
Artyukova E.V., Kozyrenko M.M., Kholina A.B., Zhuravlev Yu.N. High chloroplast haplotype diversity in the endemic legume Oxytropis chankaensis may result from independent polyploidization events // Genetica. 2011. V. 139. № 2. P. 221–232.
Холина А.Б., Козыренко М.М., Артюкова Е.В., Санданов Д.В. Современное состояние популяций эндемичных видов Oxytropis Байкальской Сибири и их филогенетические связи по данным секвенирования маркеров хлоропластной ДНК // Генетика. 2018. Т. 54. № 7. С. 795–806.
Kholina A., Kozyrenko M., Artyukova E. et al. Plastid DNA variation of the endemic species Oxytropis glandulosa Turcz. (Fabaceae) // Turk. J. Bot. 2018. V. 42. P. 38–50. https://doi.org/10.3906/bot-1706-11
Козыренко М.М., Холина А.Б., Артюкова Е.В. и др. Молекулярно-филогенетическая характеристика эндемичных дальневосточных близкородственных видов секции Orobia рода Oxytropis (Fabaceae) // Генетика. 2020. Т. 56. № 4. С. 421–432.
Холина А.Б., Козыренко М.М., Позднякова Т.Э. Генетическая изменчивость и филогенетические связи видов Oxytropis секции Xerobia (Fabaceae) степной флоры Прибайкалья // Изв. Санкт-Петербургского гос. аграрного ун-та. 2018. № 4(53). С. 38–45.
Холина А.Б., Козыренко М.М., Артюкова Е.В., Санданов Д.В. Дивергенция видов Oxytropis секции Verticillares (Fabaceae) степной флоры Байкальской Сибири на основе анализа хлоропластной ДНК // Генетика. 2019. Т. 55. № 6. С. 665–674.
Bonfeld J.K., Smith K.F., Staden R. A new DNA sequence assembly program // Nucl. Acids Res. 1995. V. 23. P. 4992–4999.
Gouy M., Guindon S., Gascuel O. SeaView version 4: A multiplatform graphical user interface for sequence alignment and phylogenetic tree building // Mol. Biol. Evol. 2010. V. 27. P. 221–224. https://doi.org/10.1093/molbev/msp259
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567.
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452.
Bandelt H.-J., Forster P., Röhl A. Median-Joining Networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48.
Mir B.A., Koul S., Kumar A. et al. Intraspecific variation in the internal transcribed spacer (ITS) regions of rDNA in Withania somnifera (Linn.) Dunal // Indian J. Biotechnol. 2010. V. 9. P. 325–328.
Swofford D.L. PAUP*: Phylogenetic analysis using parsimony (*and other methods): version 4.04. Sunderland: Sinauer Associate, 2003.
Жукова П.Г. Числа хромосом у некоторых видов семейства Fabaceae с северо-востока Азии // Бот. журн. 1983. Т. 68. № 7. С. 925–932.
Павлова Н.С., Пробатова Н.С., Соколовская А.П. Таксономический обзор семейства Fabaceae, числа хромосом и распространение на советском Дальнем Востоке // Комаровские чтения. 1989. Вып. XXXVI. С. 20–47.
Weiss-Schneeweiss H., Emadzade K., Jang T.-S., Schneeweiss G.M. Evolutionary consequences, constraints and potential of polyploidy in plants // Cytogenet Genome Res. 2013. V. 140. P. 137–150. https://doi.org/10.1159/000351727
Юрцев Б.А., Жукова П.Г. Полиплоидные ряды и таксономия (на материале анализа некоторых групп арктических бобовых) // Бот. журн. 1968. Т. 53. № 11. С. 1531–1542.
Jin D.-P., Lee J.-H., Xu B., Choi B.-H. Phylogeography of East Asian Lespedeza buergeri (Fabaceae) based on chloroplast and nuclear ribosomal DNA sequence variations // J. Plant Res. 2016. V. 129. P. 793–805. https://doi.org/10.1007/s10265-016-0831-2
Дополнительные материалы отсутствуют.