Генетика, 2020, T. 56, № 3, стр. 282-291
Селекция устойчивости к даптомицину метициллин-резистентного Staphylococcus aureus: роль гомо- и гетеро-мутаций
В. В. Гостев 1, 2, *, Ю. В. Сопова 3, 4, О. С. Калиногорская 1, И. А. Цветкова 1, С. В. Сидоренко 1, 2
1 Детский научно-клинический центр инфекционных болезней Федерального медико-биологического агентства
197022 Санкт-Петербург, Россия
2 Северо-Западный государственный медицинский университет им. И.И. Мечникова
191015 Санкт-Петербург, Россия
3 Санкт-Петербургский филиал Института общей генетики им. Н.И. Вавилова
Российской академии наук
198504 Санкт-Петербург, Россия
4 Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: guestvv11@gmail.com
Поступила в редакцию 27.03.2019
После доработки 18.06.2019
Принята к публикации 25.06.2019
Аннотация
Даптомицин – антибиотик для лечения инфекций, вызываемых мультирезистентными грамположительными патогенами. По мере роста устойчивости к другим антибиотикам следует ожидать увеличение объема потребления даптомицина, что создает потенциальную угрозу формирования устойчивости. Указанные факты обосновывают необходимость изучения возможных механизмов формирования резистентности. В работе проведена in vitro селекция устойчивости к даптомицину метициллин-резистентного штамма Staphylococcus aureus SA0420, принадлежащего к генетической линии ST239. После 40 пассажей мутантный штамм характеризовался увеличением МПК даптомицина с 1 до 64 мкг/мл, снижением времени удвоения клеток и скорости роста, индуцированная аутолитическая активность не изменялась. В ходе селекции при оценке популяционного разнообразия с использованием полногеномного секвенирования были выявлены как гомо-мутации (нуклеотидные замены в 96–100% ридов при выравнивании на контрольный геном в конкретной позиции), так и гетеро-мутации (альтернативные прочтения в 5–95% ридов). После пятого пересева была обнаружена мутация в кодоне S295L гена mprF, к 40-му пересеву дополнительно была обнаружена ранее не описываемая мутация в кодоне R50C. Новый вариант мутации A214T был идентифицирован в белке Cls2. На пятом пересеве в регуляторном гене клеточной стенки walK идентифицирована гетеро-мутация в кодоне Q493K, которая не детектировалась при последующих пассажах. Гетеро-мутации обнаруживались в генах ktrA и ntpA, кодирующих соответственно белок-переносчик ионов калия и транспортный белок для фосфатов. К 40-му пассажу селекции выявлены стоп-кодоны в гипотетическом гене yhfP (SACOL1927). Таким образом, формирование устойчивости к даптомицину происходит через формирование гетеро-популяций. После 5-, 20- и 40-го пассажей были обнаружены гомо- и гетеро-мутации в генах как с доказанной ролью в формировании устойчивости к даптомицину (mprF, cls2, walK), так и в потенциальных кандидатах (ktrA, ntpA и yhfP).
Метициллин-резистентные золотистые стафилококки (Methicillin-resistant Staphylococcus aureus, MRSA) являются важнейшими патогенами человека, характеризуются множественной устойчивостью к антибиотикам различных групп и рассматриваются как одна из угроз мировому здравоохранению [1]. Основой лечения инфекций, вызванных MRSA, являются антибиотики из классов гликопептидов (ванкомицин), липопептидов (даптомицин), оксазолидинонов (линезолид). Даптомицин – препарат, разрешенный для клинического использования в 2003 г., однако уже через год в США были описаны первые устойчивые изоляты стафилококков и энтерококков. Мишень действия этого антибиотика – цитоплазматическая мембрана грамположительных бактерий, а механизм действия аналогичен действию катионных противомикробных пептидов млекопитающих (Cationic Antimicrobial Peptides, CAMPs). Даптомицин способствует образованию кальций-зависимых каналов в мембране [2].
По результатам различных международных многоцентровых исследований, устойчивость к даптомицину регистрируется достаточно редко, и все описанные случаи в основном ассоциированы с MRSA [3, 4]. Среди коагулазоотрицательных стафилококков устойчивость к даптомицину практически не встречается и за последние несколько лет были опубликованы только единичные случаи резистентности [5, 6]. Самый распространенный механизм устойчивости к даптомицину связан с мутациями в гене mprF, который кодирует фосфатидилглицерол-лизилтрансферазу, участвующую в формировании положительно заряженного лизил-фосфатидилглицерола, входящего в состав мембран. Данный механизм описан как у S. aureus, так и среди коагулазоотрицательных стафилококков.
Гиперэкспрессия dlt-оперона, участвующего в формировании тейхоевых кислот, содержащих большое количество положительно заряженных остатков аланина, также способствует устойчивости [7, 8]. Мутации в фосфотидиотрансферазе (pgsA) и кардиолипинсинтазе (cls2) способствуют снижению количества фосфатидилглицерола, которое приводит к изменению физико-химических свойств (текучести) мембраны, что в значительной степени снижает ее взаимодействие с даптомицином [9, 10].
Наряду с этим у устойчивых к даптомицину стафилококков описаны мутации в регуляторном гене walK (yycG), участвующем в регуляции биосинтеза клеточной стенки, а также в генах, кодирующих субъединицы РНК-полимеразы rpoB и rpoC. В недавнем исследовании в ходе селекции in vitro были получены даптомицин-устойчивые мутанты S. epidermidis и S. capitis, основной механизм устойчивости авторы связывают с формированием мутаций также в генах walKR – регуляторах клеточной стенки [11].
Мутации в генах mprF, cls2, walKR обнаруживаются и у метициллин-чувствительных штаммов S. aureus, проявляющих устойчивость к даптомицину [12].
Мутации в регуляторных генах walKR и rpoB могут приводить к появлению устойчивости к другому антибиотику – ванкомицину [10, 13]. Связь между системами биосинтеза фосфолипидов и биосинтеза компонентов клеточной стенки, а также роль мутаций, приводящих к устойчивости, до конца остаются неизученными. В настоящей работе в процессе селекции устойчивости in vitro был получен даптомицин-резистентный мутант MRSA с высоким уровнем устойчивости. Проведена оценка фенотипических и генетических изменений, появившихся после селекции и приобретения устойчивости к даптомицину.
МАТЕРИАЛЫ И МЕТОДЫ
Бактериальный изолят
В работу был включен клинический изолят MRSA SA0420, чувствительный к даптомицину, из музея культур ФГБУ ДНКЦИБ ФМБА России, выделенный в 2012 г. и принадлежащий к генетической линии ST239.
Селекция устойчивости
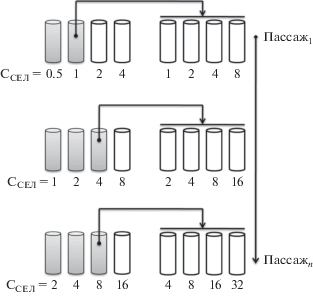
Селекцию проводили в 2 мл сердечно-мозгового бульона (bioMerieux, Франция) с добавлением кальция в конечной концентрации 50 мг/л и содержащего даптомицин (Molekula, Англия), со стартовым клеточным инокулюмом 109 КОЕ/мл. На первом этапе делали ряд разведений антибиотика с концентрацией от 1 до 8 мкг/мл и добавляли приготовленный инокулюм, культивировали в термостате при 37°С до появления роста (в среднем 48 ч) хотя бы в одной из концентраций. Полученную суспензию использовали для пересева на среды с увеличенной концентрацией даптомицина. Всего осуществлено 40 пассажей со ступенчатым увеличением концентраций антибиотика от 1 до 45 мкг/мл. Схема селекции представлена на рис. 1.
Оценка чувствительности к антибиотикам
Минимальные подавляющие концентрации (МПК) антибиотиков определяли методом серийных разведений в бульоне Мюллера-Хинтона (Bio-Rad, Франция).
Оценка скорости роста и индуцированной аутолитической активности
Скорость роста, ЛАГ-фазу оценивали путем измерения оптической плотности (OD600), используя планшетный ридер-шейкер инкубатор Infinite 200 Pro (Tecan, Австрия). Считывание OD600 проводили каждые 15 мин. ЛАГ-фазу оценивали как временной интервал от момента начала инкубации до момента 15%-ного увеличения OD600. Скорость роста оценивали по методике, приведенной в статье Hall et al. [14]. Определение времени удвоения клеток проводили в жидкой питательной среде в период экспоненциального роста культуры в двух точках через четыре часа путем высева на плотную питательную среду и подсчета КОЕ. Время удвоения клеток рассчитывали по формуле:
где Dt – время удвоения (мин); t – время между точками T1 и Т2; T1 и Т2 – соответственно количество КОЕ в начальной и конечной временных точках.
Индуцированная аутолитическая активность была проведена по методу Gustafson et al. [15] в опыте при добавлении 0.05% Тритона Х-100 и измерении OD600 в 96-луночном планшете в течение 5 ч, с интервалом считывания OD600 – 10 мин (инкубация при 30°С и орбитальном шейкировании – 142 rpm). Использовали восемь лунок на измерение. Все измерения были сделаны в трех повторностях.
Геномное секвенирование
Секвенирование штамма SA0420 было проведено до селекции (контрольный штамм) и в процессе селекции: после 5-, 20- и 40-го пассажей. Для получения лизатов ночной инокулюм осаждали центрифугированием, клеточный осадок лизировали в присутствии 5 мг/мл лизостафина (Sigma Aldrich, США) и 100 мг/мл лизоцима (Amresco, США) в трис-ЭДТА буфере в течение 60 мин при 37°С. Для выделения геномной ДНК из лизатов использовали набор GeneJET Genomic DNA Purification Kit (Thermo Fisher Scientific, Литва) по протоколу производителя. Полногеномное секвенирование проводили на платформе Miseq (Illumina, США). Использовали пул библиотек ДНК, приготовленных с помощью набора Nextera XT (Illumina kit, США). Все этапы секвенирования проводили в соответствии с протоколами производителя. Среднее покрытие геномов составило ×196.2. Полученные риды были депонированы в архив ридов SRA NCBI GenBank под следующими номерами: BioProject PRJNA325350, контрольный штамм (SRX1837665), мутант после пятого пересева (SRX5423494), мутант после 20-го пересева (SRX5423493), мутант после 40-го пересева (SRX2417051).
Биоинформатический анализ
Предварительно для всех полученных ридов были проведены удаление адаптерных последовательностей и фильтрация низкокачественных прочтений с использованием программ FastQC [16], trimmomatic [17]. Геном контрольного штамма был собран de novo, с использованием SPAdes [18]. На полученную сборку выравнивали риды этого же контрольного штамма, при этом все выявленные мутации и нуклеотидные гетеро-позиции отмечались как “ложные” и исключались из дальнейшего анализа при сравнении с мутантами для предотвращения интерпретации ложноположительных мутаций. Помимо этого, из анализа были исключены мобильные генетические элементы, профаговые области, адгезины, поверхностные белки вирулентности и протяженные нуклеотидные гомополимерные области, вследствие высокой скорости накопления неспецифических мутаций в этих локусах. Для выявления генетических изменений риды штаммов после 5-, 20- и 40-го пассажей выравнивали на контиги контрольного генома с использованием программ Bowtie2 [19], SAMtools [20]. Выявление гетеро-мутаций, анализ аллельной глубины, а также аннотацию генетических изменений осуществляли с помощью breseq [21]. За гетеро-мутации принимали долю альтернативных прочтений в конкретной позиции с частотой 5–95%. За гомо-мутации принимали 96–100%-ную частоту нуклеотидных замен в конкретной позиции при выравнивании ридов штаммов после 5-, 20- и 40-го пересевов на контиги контрольного штамма. Коррекцию аннотаций, а также оценку гипотетических белков проводили с помощью NCBI protein BLAST и UniProt [22], при этом использовали референс-геном S. aureus COL (GenBank: CP000046.1).
РЕЗУЛЬТАТЫ
Селекция устойчивости и фенотипические изменения
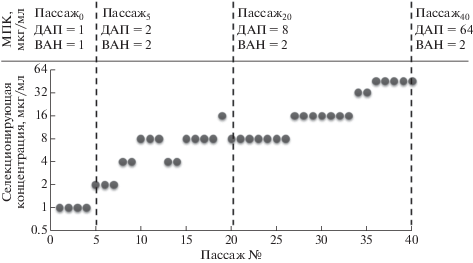
В процессе селекции штамма SA0420, ступенчато увеличивая концентрацию антибиотика в среде (с 1 до 45 мкг/мл), нам удалось получить резистентный фенотип и увеличение МПК к даптомицину с 1 до 64 мкг/мл (рис. 2).
Фенотипические характеристики контрольного штамма и устойчивого мутанта представлены в табл. 1. При оценке стабильности приобретенной устойчивости штамм дополнительно пассировали 10 раз на среде без антибиотика, при этом уровень МПК снижался до 16 мкг/мл. Параллельно с этим была проведена оценка чувствительности к 18 антибиотикам для оценки возможного влияния селекции устойчивости к даптомицину на чувствительность к другим препаратам (“seesaw”-эффекта). Так, было отмечено увеличение МПК с 1 до 2 мкг/мл только к одному антибиотику – ванкомицину, однако после десяти пассажей на среде без каких-либо антибиотиков МПК снизилась до исходного значения.
Таблица 1.
Изменение фенотипических свойств
| Характеристика | Контроль | Штамм после 40-го пересева |
|---|---|---|
| МПК ДАП, мкг/мл | 1 | 64 |
| МПК ВАН, мкг/мл | 1 | 2 |
| Индуцированная аутолитическая активность, % клеточного лизиса | 54 ± 13 | 67.6 ± 4 |
| Время удвоения клеток, мин | 26 ± 2 | 40 ± 5* |
| Скорость роста в экспоненциальной фазе μ, час–1 | 0.091 | 0.075* |
| ЛАГ-фаза, час | 2.5 | 2.4 |
| Пигментация колоний, цвет | Белый | Интенсивный желтый |
Индуцированная аутолитическая активность у мутантного штамма не изменилась. При оценке ростовых характеристик наблюдались увеличение времени удвоения клеток и снижение скорости роста, при этом ЛАГ-фаза не изменялась по сравнению с контролем. Колонии мутантного штамма после селекции характеризовались выраженным желтым цветом, что может быть связано с гиперпродукцией стафилоксантина, однако инструментальных измерений проведено не было.
Генотипические изменения
В процессе селекции были выявлены различные генетические события, при этом количество гетеро-мутаций было выше чем гомо-мутаций (табл. 2). Не было выявлено зависимости между глубиной гетеро-мутаций и покрытием генома, нуклеотидные гетеро-замены встречались с одинаковой частотой в областях как с высоким уровнем покрытия, так и с низким. Обнаруженные гетеро-мутации имели различную аллельную глубину от 5 до 90%, и в подавляющем большинстве случаев были уникальными для своего пересева. Мутации со 96–100%-ной глубиной не исчезали и сохранялись на протяжении всей селекции. Делеций и геномных перестроек обнаружено не было. Также не было обнаружено перехода гетеро-мутаций в гомо-мутации. Подавляющее большинство замен относилось к миссенс- и нонсенс-мутациям (табл. 3), также значительная часть мутаций была детектирована в промоторных областях. В частности, на пятом пересеве обнаружена нуклеотидная замена (C → A) в позиции –203 гипотетического гена (SACOL1752), вероятный белок этого гена имеет домены фосфодиэстеразы, а также DHHA1-домен, характеризующийся гидролазной активностью в отношении циклических сигнальных молекул c-di-AMP и c-di-GMP. Данная мутация сохранялась на протяжении всей селекции со 100%-ной аллельной глубиной. Значительное количество гетеро-мутаций в позициях –391 (G → A), –393 (A → T), –395 (G → A), –450 (A → T), –599 (C → T) обнаруживалось в ходе всей селекции в upstream области гена UgpQ (SACOL0031), предположительно участвующего в липидном обмене. Другие гетеро-мутации, обнаруженные в upstream областях, охватывали гены, кодирующие следующие белки: гипотетическая карбоксиэстераза PnbA (5-й пассаж, глубина 38%); альдегиддегидрогеназа SACOL2114 (20-й пассаж, глубина 8–11%); гипотетический переносчик остатков сахаров UhpT, SACOL0200 (20-й пересев, глубина 13.2%); и TarF (SACOL0239), участвующий в биосинтезе тейхоевых кислот (20-й пересев, глубина 7%).
Таблица 2.
Общее число генетических изменений у штамма MRSA SA0420 в ходе селекции устойчивости к даптомицину
| Мутации | Пересевы | ||
|---|---|---|---|
| 5 | 20 | 40 | |
| Миссенс | 1 | 1 | 3 |
| Гетеро-миссенс | 4 | 4 | 4 |
| Синонимичные (+гетеро-мутации) | 0 | 0 | 3 |
| Нонсенс | 0 | 0 | 1 |
| Гетеро-нонсенс | 0 | 1 | 1 |
| В upstream-областях | 1 | 1 | 1 |
| Гетеро-мутации в upstream-областях | 12 | 14 | 5 |
| Межгенные (+гетеро-мутации) | 11 | 9 | 15 |
Таблица 3.
Значимые мутации и гетеро-мутации, обнаруженные в различные этапы селекции штамма SA0420
| Биологическая функция | Ген | ЛокусA | DAP – RB | Мутации | Пассаж, % ридов с нуклеотидными заменами | |||
|---|---|---|---|---|---|---|---|---|
| 5 | 20 | 40 | ||||||
| Предполагаемая глутаматсинтаза | gltB2 | SACOL2469 | – | D154E | GAT → GAA | 15.9 | 11.2 | 0 |
| I153V | ATT → GTT | 0 | 15.8 | 0 | ||||
| Кардиолипинсинтаза | cls2 | SACOL2079 | + | A214T | GCT → ACT | 0 | 0 | 100 |
| Гипотетический протеин | – | SACOL1788 | – | S144T | TCA → ACA | 6.9 | 0 | 8.6 |
| Транспорт неорганических фосфатов | nptA | SACOL0088 | – | A32S | GCA → TCA | 12.1 | 0 | 0 |
| S33* | TCA → TAA | 0 | 12.4 | 0 | ||||
| Регулон клеточной стенки, регулятор | walK | yycG | + | Q493K | CAA → AAA | 6.2 | 0 | 0 |
| Биосинтез аминокислот | ilvC | ilvC | – | D258A | GAC → GCC | 0 | 5.9 | 0 |
| Транспорт ионов калия | ktrA | SACOL1096 | + | R220G | AGA → GGA | 0 | 7 | 0 |
| Мальтоза-связывающий транспортный белок | malE | SACOL0193 | – | D182Y | GAT → TAT | 0 | 0 | 5.3 |
| Предполагаемая ацил-CoA-ацетилтрансфераза | fadA | SACOL0211 | – | N135K | AAT → AAA | 0 | 0 | 5.2 |
| Гипотетический протеин | yfhP | SACOL1927 | – | E206* | GAA → TAA | 0 | 0 | 100 |
| Q182* | CAA → TAA | 0 | 0 | 6.7 | ||||
| Рибосомальный белок S11 (30S) | rpsK | rpsK | – | G90V | GGT → GTT | 0 | 0 | 38.4 |
| Биосинтез компонентов мембран | mprF | fmtC | + | S295L | TCA → TTA | 100 | 100 | 100 |
| R50C | CGT → TGT | 0 | 0 | 100 | ||||
После пятого пересева у тестируемого штамма была обнаружена аминокислотная замена S295L в белке MprF, которая сохранялась на протяжении всего периода селекции. К концу селекции в этом же белке дополнительно была обнаружена мутация, приводящая к замене в кодоне R50C. После 40-го пассажа в гене cls2 обнаруживалась мутация, обусловливающая аминокислотную замену в кодоне A214T. На пятом пересеве идентифицирована гетеро-мутация в гене walK, обусловливающая аминокислотную замену в кодоне Q493K.
Стоп-кодоны были обнаружены в гене, кодирующем гипотетический протеин YfhP. Значимые гетеро-мутации были обнаружены в генах ntpA и ktrA, кодирующих белки, вовлеченные в транспорт ионов. Помимо этого, различные гомо-мутации и гетеро-мутации были идентифицированы в рибосомальном белке (RpsK) и других белках метаболизма клетки (GltB2, IlvC, MalE, FadA), связь которых с устойчивостью к антибиотикам не установлена.
ОБСУЖДЕНИЕ
В настоящее время даптомицин используется при лечении инфекций, вызванных патогенами, устойчивыми к гликопептидам и оксазолидинонам. При этом по мере роста устойчивости к перечисленным антибиотикам следует ожидать увеличение объема потребления даптомицина, что создает потенциальную угрозу формирования устойчивости и к этому антибиотику. Также стоит отметить, что увеличение случаев снижения чувствительности к ванкомицину среди коагулазоотрицательных стафилококков может способствовать росту устойчивости к даптомицину и среди этой группы стафилококков. Указанные факты обосновывают необходимость изучения возможных путей и механизмов формирования резистентности к даптомицину.
В работу был включен штамм SA0420, относящийся к одной из доминирующих генетических линий – ST239, распространенной в России и мире. Полученные результаты могут быть использованы для предсказания формирования устойчивости к даптомицину у MRSA, и описанные мутации могут быть использованы в биоинформатических алгоритмах и базах данных о детерминантах устойчивости для предсказания устойчивости.
В ходе селекции удалось получить высокий уровень устойчивости к даптомицину с МПК = = 64 мкг/мл, при этом пограничным значением чувствительности является МПК = 1 мкг/мл. В исследовании были выявлены как хорошо охарактеризованные мутации, так и новые, ранее не описываемые, потенциально ассоциированные с устойчивостью к даптомицину. Полученный мутант характеризовался снижением скорости роста и нестабильным фенотипом. Данное явление может отражаться в выраженной биологической цене сопротивления (“fitness cost”), что согласуется с результатами других исследований [23, 24] при анализе фенотипических свойств даптомицин-устойчивых S. aureus. Чувствительность к другим антибиотикам у мутантного штамма не изменялась, за исключением ванкомицина, что еще раз подтверждает возможность формирования ассоциированной устойчивости к ванкомицину.
В настоящем исследовании мы использовали понятия “гомо-мутации” и “гетеро-мутации”, применяя их к клеточной популяции бактерий. Прокариоты представляют собой клеточную популяцию даже на уровне колонии, которая может быть в большей или меньшей степени гетерогенной. Геномное секвенирование с достаточным покрытием позволяет анализировать отдельные “прочтения” геномов, и, если в клеточной колонии формируются генетически разнородные клетки, глубокое покрытие таких прочтений позволяет проводить оценку данного феномена.
В ходе селекции были обнаружены мутации в генах с доказанной ролью устойчивости к даптомицину. В белке фосфатидилглицерол-лизилтрансфераза (MprF) уже после пятого пересева выявлена мутация, приводящая к аминокислотной замене S295L. Трансмембранный белок MprF состоит из нескольких доменов и обладает двойной активностью. Это транслоказная активность (N-терминальный конец, 1–586 а. к.) и синтазная активность (С-терминальный конец, 511–840 а. к.), выделяют также и бифункциональный домен: 276–357 а. к. [25]. Мутации в бифункциональном домене, включая аминокислотную позицию 295, достаточно хорошо изучены и ассоциированы с увеличением МПК к даптомицину до 1–4 мкг/мл [26, 27]. В настоящем исследовании появление этой мутации также сопровождалось незначительным увеличением МПК до 2 мкг/мл к пятому пересеву. К концу селекции в N-терминальном участке белка MprF, который выполняет функцию транслокации остатков лизил-фосфатидилглицерола, дополнительно была обнаружена ранее не описываемая аминокислотная замена в кодоне R50C. Ближайшей описанной мутацией в этом локусе является замена G61V, которая также была получена в работе при селекции на даптомицине, однако данная мутация также ассоциирована с незначительным увеличением МПК до 2 мкг/мл [26]. Возможно, комбинация аминокислотных замен S295L + R50C может способствовать усилению транслокации остатков лизил-фосфатидилглицерола, способствуя таким образом формированию значительного положительного заряда. Другая детерминанта, ассоциированная с устойчивостью, – ген cls2, кодирующий белок кардиолипинсинтазу. Отрицательно заряженный фосфолипид кардиолипин – важнейший компонент бактериальной мембраны, который участвует в связывании молекулы даптомицина и его олигомеризации [9]. Ранее было показано, что высокие концентрации этого компонента в мембране предотвращают формирование каналов под воздействием антибиотика [28]. К 40-му пассажу у мутанта SA0420 появилась ранее не описанная мутация A214T в белке Cls2, по-видимому играющая роль в значительном повышении МПК.
Белок WalK – важнейший регулятор метаболизма клеточной стенки, мутации в котором ассоциированы со снижением чувствительности как к даптомицину, так и ванкомицину [29]. Минорная гетеро-мутация (6% ридов) в WalK была детектирована в ранее не описанной позиции Q493K только на пятом пассаже, и в последующих пересевах эта замена не обнаруживалась.
Стоп-кодоны были обнаружены в гипотетическом протеине YfhP (SACOL1927). Роль этого белка в метаболизме прокариотической клетки, а также роль в формировании устойчивости к антибиотикам не изучена. Продукт этого гена содержит аминокислотный домен суперсемейства YdjM, белки этого суперсемейства участвуют в SOS-ответе, их активность регулируется белком LexA. Роль этой детерминанты в формировании устойчивости к даптомицину должна быть оценена в опытах по сайт-направленному мутагенезу.
В настоящем исследовании к 20-му пересеву была детектирована гетеро-мутация в гене ktrA. Белок KtrA является компонентом транспортной системы, участвующей в переносе ионов калия, принимает также участие в поляризации клеточной мембраны. В исследовании Gries et al. [30] было показано, что при делетировании этого гена у S. aureus наблюдалось увеличение чувствительности к агентам, действующим на цитоплазматическую мембрану (CAMPs, грамицидину и полимиксину). Помимо этого, белок KtrA является мишенью действия внутриклеточных сигнальных мессенджеров c-di-AMP. Эти сигнальные молекулы обеспечивают гомеостаз клеточной стенки и при повышении внутриклеточной концентрации способствуют устойчивости к препаратам, также действующим на клеточную стенку [31]. В этой связи KtrA может рассматриваться как потенциальная детерминанта, ассоциированная с устойчивостью к даптомицину. Транспортный белок NtpA входит в комплекс транспортной системы переноса фосфатов и опосредует развитие инфекционного процесса. Так, в исследовании Kelliher et al. [32] было показано, что при делеции гена ntpA стафилококки в значительной степени снижали способность к инфицированию у экспериментальных животных. Роль транспортера ntpA в формировании устойчивости к даптомицину не изучена, однако учитывая, что в этом гене в настоящем исследовании выявлен стоп-кодон, это может отражаться на снижении способности вызывать инфекционный процесс. В целом ряде работ было показано, что устойчивость к даптомицину коррелирует со снижением вирулентности. Так, в исследовании Cameron et al. [33] в опытах in vivo было продемонстрировано, что даптомицин-устойчивые штаммы проявляли меньший вирулентный потенциал по сравнению с чувствительными штаммами, но отличались свойством персистенции, т.е. длительным выделением патогена из очага без выраженных проявлений инфекционного процесса.
Ряд гетеро-мутаций были обнаружены в регуляторных upstream-областях, что может приводить к изменению транскриптома при формировании устойчивости. Потенциальными маркерами стали гены, участвующие в липидном обмене (UgpQ) и биосинтезе тейхоевых кислот (tarF). Ранее в аналогичном эксперименте по селекции устойчивости к даптомицину было показано, что происходит изменение транскрипционных профилей: увеличение биосинтеза мембранных фосфолипидов, жирных кислот и D-аланизация тейхоевых кислот [34]. Белок гипотетического гена SACOL1752, содержащий домен DHHA1, который, возможно, принимает участие в инактивации сигнальных молекул c-di-AMP, также может рассматриваться как потенциальный маркер, ассоциированный с устойчивостью к даптомицину.
Роль других мутаций, обнаруженных в генах метаболизма (gltB2, ilvC, malE, fadA), сложно оценить, не было описано связи этих генов с устойчивостью к антибиотикам. Скорее всего, изменения этих генов носят компенсаторный характер.
Настоящее исследование имеет несколько ограничений. Селекция была проведена только с представителем одной генетической линии – ST239, в этой связи сложно экстраполировать результаты в отношении других глобально распространенных генетических линий S. aureus. Однако на сегодняшний день нет экспериментальных данных о связи разных генетических линий с формированием какого-либо конкретного пути устойчивости к даптомицину. Это может являться предметом дальнейшего изучения проблемы резистентности к даптомицину. В работе не проведена оценка изменения транскриптома, что затрудняет интерпретацию данных о мутациях, обнаруженных в потенциальных промоторах. Изменение работы различных регуляторных сетей также может способствовать формированию устойчивости.
Формирование устойчивости через гетерорезистентность, скорее всего, носит универсальный характер, а изменчивость прокариот происходит через формирование изначально множества путей (мутаций), которые будут способствовать выживанию. Однако вопрос, по какому пути в дальнейшем пойдет эволюция того или иного механизма устойчивости, остается открытым. В ходе клеточной селекции в популяции останутся только те генотипы, которые будут максимально адаптированы к новым условиям. Это отражается в элиминации гетеро-мутаций или переходе их в постоянные гомо-мутации, т.е. закреплении в клеточной популяции, что было продемонстрировано в длительном эволюционном эксперименте на примере Escherichia coli [35].
Работа была выполнена в рамках гранта Российского научного фонда, проект № 18-75-10114 “Новые механизмы устойчивости Staphylococcus aureus к бета-лактамным и гликопептидным антибиотикам, связанные с внутриклеточными мессенджерами c-di-AMP и гетерорезистентностью”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Turner N.A., Sharma-Kuinkel B.K., Maskarinec S.A. et al. Methicillin-resistant Staphylococcus aureus: An overview of basic and clinical research // Nat. Rev. Microbiol. 2019. V. 17. № 4. P. 203–218. https://doi.org/10.1038/s41579-018-0147-4
Steenbergen J.N., Alder J., Thorne G.M., Tally F.P. Daptomycin: A lipopeptide antibiotic for the treatment of serious gram-positive infections // J. Antimicrob. Chemother. 2005. V. 55. № 3. P. 283–291. https://doi.org/10.1093/jac/dkh546
Stefani S., Campanile F., Santagati M. et al. Insights and clinical perspectives of daptomycin resistance in Staphylococcus aureus: A review of the available evidence // Int. J. Antimicrob. Agents. 2015. V. 46. № 3. P. 278–289. https://doi.org/10.1016/j.ijantimicag.2015.05.008
Streit J.M., Jones R.N., Sader H.S. Daptomycin activity and spectrum: a worldwide sample of 6737 clinical Gram-positive organisms // J. Antimicrob. Chemother. 2004. V. 53. № 4. P. 669–674. https://doi.org/10.1093/jac/dkh143
Duah M. Daptomycin for methicillin-resistant Staphylococcus epidermidis native-valve endocarditis: a case report // Ann. Clin. Microbiol. Antimicrob. 2010. V. 9. https://doi.org/10.1186/1476-0711-9-9
Hagiya H., Sugawara Y., Kimura K. et al. Emergence of daptomycin non-susceptible coagulase-negative Staphylococci in patients with cardiovascular device infections: Two cases report investigated by whole genome analysis // Medicine (Baltimore). 2018. V. 97. № 49. https://doi.org/10.1097/MD.0000000000013487
Bertsche U., Weidenmaier C., Kuehner D. et al. Correlation of daptomycin resistance in a clinical Staphylococcus aureus strain with increased cell wall teichoic acid production and D-alanylation // Antimicrob. Agents Chemother. 2011. V. 55. № 8. P. 3922–3930. https://doi.org/10.1128/AAC.01226-10
Cafiso V., Bertuccio T., Purrello S. et al. dltA overexpression: A strain-independent keystone of daptomycin resistance in methicillin-resistant Staphylococcus aureus // Int. J. Antimicrob. Agents. 2014. V. 43. № 1. P. 26–31. https://doi.org/10.1016/j.ijantimicag.2013.10.001
Tran T.T., Munita J.M., Arias C.A. Mechanisms of drug resistance: daptomycin resistance // Ann. N.Y. Acad. Sci. 2015. V. 1354. P. 32–53. https://doi.org/10.1111/nyas.12948
Bayer A.S., Schneider T., Sahl H.G. Mechanisms of daptomycin resistance in Staphylococcus aureus: role of the cell membrane and cell wall // Ann. N.Y. Acad. Sci. 2013. V. 1277. P. 139–158. https://doi.org/10.1111/j.1749-6632.2012.06819.x
Jiang J.H., Dexter C., Cameron D.R. et al. Evolution of daptomycin resistance in coagulase-negative Staphylococci involves mutations of the essential two-component regulator WalKR // Antimicrob. Agents Chemother. 2019. V. 63. № 3. https://doi.org/10.1128/AAC.01926-18
Kang K.M., Mishra N.N., Park K.T. et al. Phenotypic and genotypic correlates of daptomycin-resistant methicillin-susceptible Staphylococcus aureus clinical isolates // J. Microbiol. 2017. V. 55. № 2. P. 153–159. https://doi.org/10.1007/s12275-017-6509-1
Cui L., Isii T., Fukuda M. et al. An RpoB mutation confers dual heteroresistance to daptomycin and vancomycin in Staphylococcus aureus // Antimicrob. Agents Chemother. 2010. V. 54. № 12. P. 5222–5233. https://doi.org/10.1128/AAC.00437-10
Hall B.G., Acar H., Nandipati A., Barlow M. Growth rates made easy // Mol. Biol. Evol. 2014. V. 31. № 1. P. 232–238. https://doi.org/10.1093/molbev/mst187
Gustafson J.E., Berger-Bachi B., Strassle A., Wilkinson B.J. Autolysis of methicillin-resistant and -susceptible Staphylococcus aureus // Antimicrob. Agents Chemother. 1992. V. 36. № 3. P. 566–572. https://doi.org/10.1128/aac.36.3.566
Brown J., Pirrung M., McCue L.A. FQC Dashboard: integrates FastQC results into a web-based, interactive, and extensible FASTQ quality control tool // Bioinformatics. 2017. V. 33. № 19. P. 3137–3139 https://doi.org/10.1093/bioinformatics/btx373
Bolger A.M., Lohse M., Usadel B. Trimmomatic: A flexible trimmer for illumina sequence data // Bioinformatics. 2014. V. 30. № 15. P. 2114–2134. https://doi.org/10.1093/bioinformatics/btu170
Bankevich A., Nurk S., Antipov D. et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing // J. Comput. Biol. 2012. V. 19. № 5. P. 455–477. https://doi.org/10.1089/cmb.2012.0021
Langdon W. B. Performance of genetic programming optimised Bowtie2 on genome comparison and analytic testing (GCAT) benchmarks // BioData Min. 2015. V. 8. № 1. [pii]https://doi.org/10.1186/s13040-014-0034-034
Li H., Handsaker B., Wysoker A. et al. The sequence alignment/map format and SAMtools // Bioinformatics. 2009. V. 25. № 16. P. 2078–2087. https://doi.org/10.1093/bioinformatics/btp352
Barrick J.E., Colburn G., Deatherage D.E. et al. Identifying structural variation in haploid microbial genomes from short-read resequencing data using breseq // BMC Genomics. 2014. V. 15. https://doi.org/10.1186/1471-2164-15-1039
UniProt: a worldwide hub of protein knowledge // Nucl. Acids. Res. 2019. V. 47. № D1. P. D506–D515. https://doi.org/10.1093/nar/gky1049
Roch M., Gagetti P., Davis J. et al. Daptomycin resistance in clinical MRSA strains is associated with a high biological fitness cost // Front. Microbiol. 2017. V. 8. https://doi.org/10.3389/fmicb.2017.02303
Li S., Yin Y., Chen H. et al. Fitness cost of daptomycin-resistant Staphylococcus aureus obtained from in vitro daptomycin selection pressure // Front. Microbiol. 2017. V. 8. https://doi.org/10.3389/fmicb.2017.02199
Ernst C.M., Slavetinsky C.J., Kuhn S. et al. Gain-of-function mutations in the phospholipid flippase MprF confer specific daptomycin resistance // MBio. 2018. V. 9. № 6. https://doi.org/10.1128/mBio.01659-18
Peleg A.Y., Miyakis S., Ward D.V. et al. Whole genome characterization of the mechanisms of daptomycin resistance in clinical and laboratory derived isolates of Staphylococcus aureus // PLoS One. 2012. V. 7. № 1. https://doi.org/10.1371/journal.pone.0028316
Bayer A.S., Mishra N.N., Chen L. et al. Frequency and distribution of single-nucleotide polymorphisms within mprF in methicillin-resistant Staphylococcus aureus clinical isolates and their role in cross-resistance to daptomycin and host defense antimicrobial peptides // Antimicrob. Agents Chemother. 2015. V. 59. № 8. P. 4930–4937. https://doi.org/10.1128/AAC.00970-15
Zhang T., Muraih J.K., Tishbi N. et al. Cardiolipin prevents membrane translocation and permeabilization by daptomycin // J. Biol. Chemistry. 2014. V. 289. № 17. P. 11584–11591. https://doi.org/10.1074/jbc.M114.554444
Howden B.P., McEvoy C.R., Allen D.L. et al. Evolution of multidrug resistance during Staphylococcus aureus infection involves mutation of the essential two component regulator WalKR // PLoS Pathog. 2011. V. 7. № 11. https://doi.org/10.1371/journal.ppat.1002359
Gries C.M., Bose J.L., Nuxoll A.S. et al. The Ktr potassium transport system in Staphylococcus aureus and its role in cell physiology, antimicrobial resistance and pathogenesis // Mol. Microbiol. 2013. V. 89. № 4. P. 760–773. https://doi.org/10.1111/mmi.12312
Fahmi T., Port G.C., Cho K.H. c-di-AMP: An essential molecule in the signaling pathways that regulate the viability and virulence of gram-positive bacteria // Genes (Basel). 2017. V. 8. № 8. https://doi.org/10.3390/genes8080197
Kelliher J.L., Radin J.N., Grim K.P. et al. Acquisition of the phosphate transporter NptA enhances Staphylococcus aureus pathogenesis by improving phosphate uptake in divergent environments // Infect. Immun. 2018. V. 86. № 1. https://doi.org/10.1128/IAI.00631-17
Cameron D.R., Mortin L.I., Rubio A. et al. Impact of daptomycin resistance on Staphylococcus aureus virulence // Virulence. 2015. V. 6. № 2. P. 127–131. https://doi.org/10.1080/21505594.2015.1011532
Muller A., Grein F., Otto A. et al. Differential daptomycin resistance development in Staphylococcus aureus strains with active and mutated gra regulatory systems // Int. J. Med. Microbiol. 2018. V. 308. № 3. P. 335–348. https://doi.org/10.1016/j.ijmm.2017.12.002
Good B.H., McDonald M.J., Barrick J.E. et al. The dynamics of molecular evolution over 60,000 generations // Nature. 2017. V. 551. № 7678. P. 45–50. https://doi.org/10.1038/nature24287
Дополнительные материалы отсутствуют.