Генетика, 2020, T. 56, № 3, стр. 272-281
Мобилом клинических карбапенем-устойчивых изолятов Klebsiella pneumoniae
А. Е. Алексеева 1, *, Н. Ф. Бруснигина 1, Н. А. Гординская 2
1 Нижегородский научно-исследовательский институт эпидемиологии и микробиологии
им. академика И.Н. Блохиной
603950 Нижний Новгород, Россия
2 Приволжский исследовательский медицинский университет
603005 Нижний Новгород, Россия
* E-mail: a.e.alexeeva79@mail.ru
Поступила в редакцию 07.03.2019
После доработки 25.04.2019
Принята к публикации 05.06.2019
Аннотация
Дана характеристика структуры мобилома клинических изолятов Klebsiella pneumoniae, принадлежащих к эпидемически распространенному сиквенс-типу 395 и продуцирующих бета-лактамазы расширенного спектра действия CTX-M-15 и OXA-48 карбапенемазы. По данным анализа in silico в мобиломе всех штаммов установлено наличие Col-подобной плазмиды и репликонов групп несовместимости R и L/M. Проведен анализ генетического окружения генов blaOXA-48 и blaСТХ-М-15 и описаны варианты мобильных структур, участвующих в распространении этих генов. В структуре мобилома штамма K. pneumoniae KP1083 обнаружены дополнительная плазмида HI1B/FIB, нуклеотидная последовательность которой имеет филогенетическое родство с плазмидой pNDM-MAR, а также широкий спектр мобильных структур, сцепленных с детерминантами антибиотикорезистентности, в частности инсерционные структуры ISCR1 и две интегразы, относящиеся к интегронам класса 1.
В Российской Федерации, как и во всем мире, бактерии вида K. pneumoniae являются актуальными нозокомиальными патогенами, вызывающими инфекционные заболевания респираторного, желудочно-кишечного трактов, глаз, кожи, инфекции мочевыводящих путей, центральной нервной системы, а также первичные абсцессы печени [1–3]. Повышенное внимание исследователей к изучению клинических изолятов клебсиелл связано, в первую очередь, с биологическими свойствами данных микроорганизмов, в частности с их вирулентностью и высокой устойчивостью к широкому спектру антимикробных препаратов [1, 2, 4]. Антибиотикорезистентность клебсиелл, как правило, связана с наличием генетических детерминант, расположенных на мобильных элементах (плазмидах, транспозонах, интегронах, IS-элементах и т.д.) [3–7].
Исследователями разных стран мира показано присутствие в геноме клинических штаммов клебсиелл нескольких плазмид, часто комбинированных и принадлежащих одновременно к нескольким группам несовместимости [3, 5]. Однако хотелось бы отметить, что в Российской Федерации имеется существенный недостаток сведений, касающихся детальной характеристики мобилома нозокомиальных штаммов K. pneumoniae.
МАТЕРИАЛЫ И МЕТОДЫ
Объектом исследования были три карбапенем-резистентных клинических изолята K. pneumoniae KP59, KP314, KP1083, выделенных из раневой поверхности пациентов ожогового отделения клиники ФГБОУ “Приволжский исследовательский медицинский университет”. ДНК чистых культур K. pneumoniae выделяли с помощью коммерческого набора “АмплиПрайм ДНК-сорб-В” (ЦНИИЭ, Россия). Полногеномное секвенирование штаммов K. pneumoniae проводили на секвенаторе MiSeq (Illumina, США). При подготовке библиотеки ДНК для секвенирования использовали набор Nextera XT Sample Preparation kit (Illumina, США), секвенирование проводили с использованием набора MiSeq reagent kit v2 (Illumina, США) на 500 циклов. Сборку чтений de novo, полученных в результате секвенирования, осуществляли c помощью программного обеспечения SPAdes версия 3.9.1 [8]. Аннотирование проводили с использованием NCBI Prokaryotic Genome Annotation Pipeline (PGAP) (https://www.ncbi.nlm.nih.gov/ genome/annotation_prok/). С помощью сервиса BLASTN [9] осуществляли поиск гомологичных последовательностей, принадлежащих плазмидам, и подбор наиболее близких референс-последовательностей целых плазмид, депонированных в базе данных GenBank. Типирование по группам несовместимости плазмид in silico проводили с использованием web-сервиса PlasmidFinder [10]. Поиск детерминант антибиотикорезистентности и патогенности осуществляли с помощью базы данных Klebsiella Sequence Typing (http:// bigsdb.pasteur.fr/klebsiella/klebsiella.html) и сервера Center for Genomic Epidemiology (https:// cge.cbs.dtu.dk/services/). Поиск и характеристику мобильных элементов, связанных с детерминантами антибиотикорезистентности, проводили с помощью web-сервисов IS-finder [11] и INTEGRALL [12]. Для выявления гомологичных последовательностей мобильных структур относительно базы данных RefSeq (NCBI) [13] использовали сервис BLASTN.
Филогенетический анализ проводили с помощью web-сервиса REALPHY [14], построение дендрограмм осуществляли в программе MEGA версия 7.0.21 [15], алгоритм ближайшего соседа (Neighbor-Joining) [16] с бутстреп-поддержкой (1000 повторов).
РЕЗУЛЬТАТЫ
На основании полученных результатов сборки коротких чтений проведено типирование исследуемых штаммов K. pneumoniae KP59, KP314, KP1083, установлена принадлежность их сиквенс-типу 395 (ST395) и капсульному типу K2. Более подробная характеристика этих штаммов клебсиелл дана в ранее представленной публикации [17].
При анализе нуклеотидных последовательностей генома штаммов K. pneumoniae KP59, KP314, KP1083, проведенном с помощью web-сервиса PlasmidFinder, найдены репликоны групп несовместимости L/M и R. Кроме того, выявлен Col440I/440II-подобный репликон с уровнем гомологии около 90% относительно нуклеотидных последовательностей плазмид K. pneumoniae FDAARGOS_440 plasmid unnamed (номер GenBank CP023920.1 и CP023921.1). У штамма K. pneumoniae KP1083 дополнительно обнаружены репликоны IncHI1B и IncFIB, имеющие уровень идентичности 99.82 и 99.54% соответственно с репликонами плазмиды pNDM-MAR (номер GenBank JN420336.1), выявленной у штамма K. pneumoniae TCKpnC (Марокко, 2011) [18].
Принадлежность контигов конъюгативной плазмиде IncL/M определяли относительно референс-последовательности плазмиды pOXA-48 штамма K. pneumoniae Kp11978 (номер GenBank JN626286.1). В результате аннотирования установлено, что у всех трех штаммов данная плазмида несет только детерминанту OXA-48 карбапенемазы. У штамма K. pneumoniae KP1083 детерминанта OXA-48 входит в состав широко распространенного транспозона Tn1999 [19]. При анализе генетического окружения детерминанты blaOXA-48 штаммов клебсиелл KP59 и KP314 с помощью сервиса BLASTN установлено, что наиболее гомологичной является структура транспозона Tn1999.2, несущего ген OXA-48 карбапенемазы и выявленного впервые в плазмиде p11099 штамма E. cloacae 11099 (номер GenBank JN714122.1) [20]. На рис. 1 представлена дендрограмма нуклеотидных последовательностей плазмид IncL/M исследуемых штаммов и полногеномных последовательностей плазмид K. pneumoniae, депонированных в базу данных GenBank.
Рис. 1.
Дендрограмма, построенная на основании сравнения нуклеотидных последовательностей плазмид IncL/M штаммов K. pneumoniae.
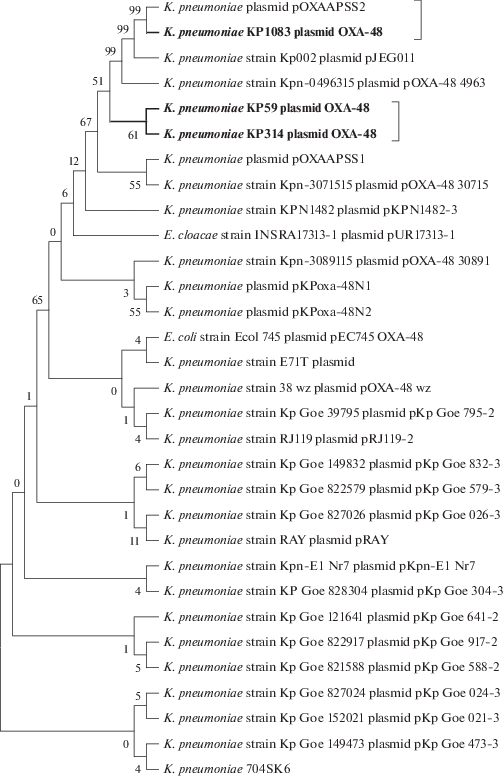
Результаты филогенетического анализа показали, что нуклеотидные последовательности плазмид IncL/M штаммов K. pneumoniae KP59, KP314 находятся на одной ветке, а штамма K. pneumoniae KP1083 группируются с плазмидной ДНК pOXAAPSS2 (номер GenBank KU159086.1), выделенной у штамма K. pneumoniae OXAAPSS2 (Санкт-Петербург, 2012 г.).
Референс-последовательностью для определения контигов, принадлежащих плазмиде IncR, являлась нуклеотидная последовательность плазмиды pKp_Goe_629-1 (номер GenBank CP018365.1). По данным аннотирования в структуре плазмиды IncR исследуемых штаммов K. pneumoniae обнаружено значительное количество детерминант антибиотикорезистентности, включая ген эпидемически значимой СТХ-М-15 цефалоспориназы. При анализе генетического окружения гена blaСТХ-М-15 с помощью web-сервиса IS-finder установлено, что у всех трех исследуемых штаммов в генетическую структуру, несущую детерминанту СТХ-М-15, входит делетированная последовательность мобильного элемента ISEcp1 длиной в 192 нуклеотида, через 48 нуклеотидов расположен ген blaСТХ-М-15, далее находится ген, предположительно кодирующий металлопротеин семейства WbuC, затем последовательность транспозона ∆Tn3 (контиги MRVH02000054.1, MRYN02000056.1, MRYM02000096.1 + + MRYM02000092.1). Вся структура фланкирована инвертированными инсерционными элементами длиной 188 пн, представляющими собой последовательности ∆IS26 транспозаз. Следует отметить, что высокогомологичная структура обнаружена только в референсной нуклеотидной последовательности плазмиды Kp_Goe_629-1 штамма K. pneumoniae Kp_Goe_62629 (Германия, 2015 г.) Менее гомологичные последовательности обнаружены в плазмидах других групп несовместимости, выделенных у различных штаммов бактерий семейства Enterobacteriaceae.
Сцеплены с мобильными элементами и другие детерминанты антибиотикорезистентности, локализованные группами на разных контигах, которые входят в структуру плазмиды IncR. В частности, на одном контиге расположены детерминанты пентапептидного белка QnrS1, определяющего устойчивость к хинолонам, и бета-лактамазы TEM-1. Ген qnrS1 фланкирован, c одной стороны, инсерционным элементом ISKpn19, а с другой – фрагментом последовательности транспозазы, предположительно относящейся к семейству IS3. Ген blaTEM-1 находится в составе ∆Tn2 транспозона длиной 2125 пн [21].
Гены, определяющие устойчивость к триметоприму (dfrA1), тетрациклину (tetA), сульфаниламидам (sul1), связаны с мобильным элементом IS6100.
С транспозазой семейства IS6 сцеплены гены аминогликозидацетилтрансферазы (aac(6')-Ib-cr), оксациллиназы OXA-1 (blaOXA-1), хлорамфениколацетилтрансферазы (catB3).
Дендрограмма нуклеотидных последовательностей плазмид IncR исследуемых штаммов K. pneumoniae относительно полногеномных последовательностей плазмид IncR K. pneumoniae, депонированных в базу данных GenBank, представлена на рис. 2.
Рис. 2.
Дендрограмма, построенная на основании сравнения нуклеотидных последовательностей плазмид IncR штаммов K. pneumoniae.

Результаты филогенетического анализа показали, что последовательности всех трех штаммов группируются в единый кластер только с плазмидой Kp_Goe_629-1 штамма K. pneumoniae Kp_Goe_62629.
Обнаруженная в структуре мобилома всех трех штаммов клебсиелл Col-подобная плазмида длиной 3.5–3.6 тыс. пн является высокогомологичной (99%) с нуклеотидной последовательностью короткой плазмиды pB1023 (номер GenBank JQ319770.1) штамма E. cloacae BB1092 и плазмиды plasmid4 (номер GenBank CP034043.1) штамма K. pneumoniae CRK0298. В структуре плазмиды выявлены только гены, ответственные за мобилизацию и репликацию. Относительно базы данных RefSeq NCBI (для таксона K. pneumoniae) высокогомологичные последовательности длиной от 3 тыс. до 7.3 тыс. пн обнаружены у более 30 штаммов клебсиелл, изолированных в различных странах мира, включая Российскую Федерацию.
Как уже было сказано выше, штамм K. pneumoniae KP1083 характеризуется наличием дополнительно двух репликонов HI1B и FIB, имеющих высокий уровень идентичности с соответствующими последовательностями плазмиды pNDM-MAR. Таким образом, мы предположили, что репликон IncFIB также входит в структуру плазмиды IncHI1B штамма K. pneumoniae KP1083. Подбор контигов, относящихся к плазмиде IncHI1B/FIB исследуемого штамма, проводился относительно последовательности pNDM-MAR. При анализе нуклеотидной последовательности плазмиды IncHI1B/FIB обнаружены гены аэробактина (iucABCDiutA), а также гены, определяющие устойчивость к ионам теллура (terEX) (табл. 1).
Таблица 1.
Общая характеристика мобилома штаммов Klebsiella pneumoniae
| Штамм | Группы несовместимости плазмид | Референс-геномы плазмид | Номера контигов в базе данных GenBank | Детерминанты антибиотикорезистентности | Детерминанты патогенности |
|---|---|---|---|---|---|
| KP59 | IncL/M | JN626286.1 | MRVH02000033.1, MRVH02000046.1, MRVH02000060.1, MRVH02000070.1, MRVH02000072.1, MRVH02000080.1, MRVH02000100.1 | blaOXA-48 | |
| IncR | CP018365.1 | MRVH02000044.1, MRVH02000048.1, MRVH02000049.1, MRVH02000053.1, MRVH02000054.1, MRVH02000056.1, MRVH02000058.1, MRVH02000061.1, MRVH02000062.1, MRVH02000063.1, MRVH02000064.1, MRVH02000065.1, MRVH02000066.1, MRVH02000067.1, MRVH02000070.1, MRVH02000072.1, MRVH02000080.1, MRVH02000081.1, MRVH02000083.1, MRVH02000092.1 | blaOXA-1, blaТЕМ-1, blaСТХ-М-15, qnrS1, tet(A), sul1, dfrA1, aac(6')-Ib-cr, catB3, catA1 | ||
| Col440I/440II | JQ319770.1 | MRVH02000055.1 | |||
| KP314 | IncL/M | CP018461.1 | MRYN02000034.1, MRYN02000046.1, MRYN02000061.1, MRYN02000070.1, MRYN02000072.1, MRYN02000083.1, MRYN02000103.1 | blaOXA-48 | |
| IncR | CP018365.1 | MRYN02000044.1, MRYN02000050.1, MRYN02000051.1, MRYN02000055.1, MRYN02000056.1, MRYN02000058.1, MRYN02000062.1, MRYN02000063.1, MRYN02000064.1, MRYN02000066.1, MRYN02000067.1, MRYN02000071.1, MRYN02000073.1, MRYN02000083.1, MRYN02000084.1, MRYN02000092.1, MRYN02000111.1, MRYN02000093.1, MRYN02000099.1 | blaOXA-1, blaТЕМ-1, blaСТХ-М-15, qnrS1, tet(A), sul1, dfrA1, aac(6')-Ib-cr, catB3, catA1 | ||
| Col440I/440II | JQ319770.1 | MRYN02000057.1 | |||
| KP1083 | IncL/M | CP018461.1 | MRYM02000034.1, MRYM02000064.1 | blaOXA-48 | |
| IncR | CP018365.1 | MRYM02000059.1, MRYM02000066.1, MRYM02000075.1, MRYM02000076.1, MRYM02000080.1, MRYM02000089.1, MRYM02000090.1, MRYM02000091.1, MRYM02000092.1, MRYM02000094.1, MRYM02000096.1, MRYM02000099.1, MRYM02000104.1, MRYM02000107.1 | blaOXA-1, blaТЕМ-1, blaСТХ-М-15, qnrS1, tet(A), sul1, dfrA1, aac(6')-Ib-cr, catA1, catB3 | ||
| IncHI1B/FIB | JN420336.1 | MRYM02000026.1, MRYM02000033.1, MRYM02000039.1, MRYM02000046.1, MRYM02000049.1, MRYM02000054.1, MRYM02000072.1, MRYM02000081.1, MRYM02000097.1, MRYM02000123.1 | iucABCD, iutA, terEX | ||
| Col440I/440II | JQ319770.1 | MRYM02000083.1 | |||
| MRYM02000063.1 | ISCR1, armA, msrE, mphE | ||||
| MRYM02000086.1 | Int1, dfrA17, aadA5 | ||||
| MRYM02000093.1 | ∆Int1, aadB, aadA1 |
На рис. 3 представлены результаты филогенетического анализа относительно полногеномных последовательностей плазмид IncHI1B/FIB.
Рис. 3.
Дендрограмма, построенная на основании сравнения нуклеотидных последовательностей плазмид IncHI1В/FIB штаммов K. pneumoniae.
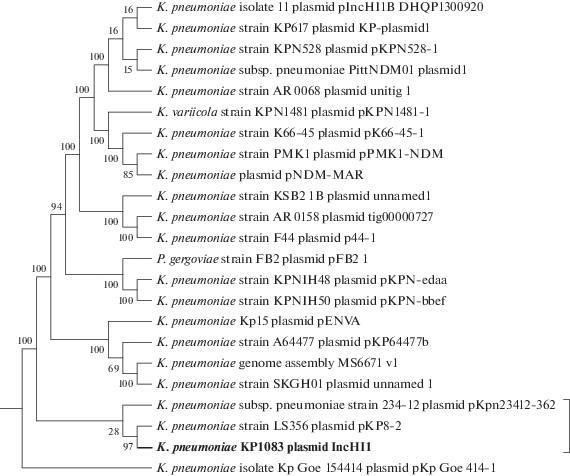
Полученные данные свидетельствуют, что последовательности плазмиды IncHI1B штамма K. pneumoniae KP1083 имеют высокое филогенетическое родство с нуклеотидной последовательностью плазмиды pKP8-2 (номер GenBank CP025638.1) штамма K. pneumoniae LS356 (Китай, 2011 г.).
Часть контигов, входящих в структуру мобилома штамма K. pneumoniae KP1083, не удалось отнести к какой-либо плазмиде с использованием сервиса BLASTN. Некоторые из них содержат мобильные элементы, сцепленные с детерминантами антибиотикорезистентности. Так, в контиге MRYM02000086.1 выявлен неполный интегрон первого класса, представленный только 5'CS-участком, включающим интегразу и гены устойчивости к триметоприму (dfrA17) и аминогликозидам (aadA5). В контиге MRYM02000075.1 обнаружен Tn3-подобный элемент, несущий транспозазу семейства TnAs3 и детерминанту устойчивости к фениколам (catA1). В структуре контига MRYM02000063.1 определены инсерционный элемент ISCR1 (insertion sequence common region) [22], ген метилазы 16S рРНК (armA), обеспечивающей устойчивость к аминогликозидам, IS4-подобный элемент (ISEc29 семейства транспозаз) с генами белка защиты мишени Msr(E) и макролид 2'-фосфотрансферазы (mphE), вся структура фланкирована инвертированными остатками IS6 транспозаз. В контиге MRYM02000093.1 обнаружена укороченная (127 пн) последовательность интегразы интегрона класса 1, с которой сцеплены гены аденилилтрансфераз: ant(2'')-Ia (aadB) и ant(3")-Ia (aadA1).
ОБСУЖДЕНИЕ
Характерной особенностью энтеробактерий, в том числе K. pneumoniae, является одновременное присутствие в структуре генома нескольких плазмид, принадлежащих к разным группам несовместимости и несущих детерминанты множественной лекарственной устойчивости. Известно, что многокопийные плазмиды могут иметь малый размер (1.3–3.75 тыс. пн), а низкокопийные – большой (105 тыс. пн и выше) [3, 5].
У всех трех исследуемых штаммов K. pneumoniae обнаружена Col-подобная плазмида, встречающаяся не только у представителей K. pneumoniae, но и у E. cloacae. Данная Col-подобная плазмида не несет в своей структуре детерминант патогенности и антибиотикорезистентности, однако по данным литературы некоторые группы Col-плазмид, в частности представители ColE1, характеризуются наличием таких детерминант за счет приобретения Tn3-подобных транспозонов [3].
Нуклеотидная последовательность плазмиды резистентности IncR, выявленная у всех исследуемых штаммов клебсиелл, содержит детерминанту blaСТХ-М-15. Обнаружение гена CTX-M-15 цефалоспориназы в структуре плазмиды, принадлежащей группе несовместимости R, является нехарактерным, поскольку согласно представленным в литературе сведениям данная детерминанта наиболее часто детектировалась в составе плазмид IncF (FIA, FIB, FII), IncI, IncN, IncHI2, IncL/M и IncK [5, 23]. Описанию генетического окружения гена CTX-M-15 цефалоспориназы посвящено значительное число работ, в которых показано наличие большого генетического разнообразия в участках ДНК бактерий семейства Enterobacteriaceae, прилегающих к генам blaCTX-M. Известно, что первичная мобилизация гена blaCTX-M-15 произошла в результате встраивания последовательности ISEcp1, в дальнейшем в структуре ISEcp1 отмечается появление делеции и инсерции последовательностей IS26, IS1, IS10, resTn2, resTn3 в разных ориентациях [5, 6, 23, 24]. В нашем исследовании выявлена конструкция, в которой последовательность ISEcp1 разрушена инсерционным элементом ∆IS26. При этом расстояние между ISEcp1 и геном blaCTX-M-15 в 48 пн сохранилось, что свидетельствует о продолжении процесса рекомбинационных перестроек, а не является новым случаем мобилизации. Необходимо отметить, что в структуре высокогомологичной последовательности плазмиды Kp_Goe_629-1 участок ∆ISEcp1-blaCTX-M-15-wbuC-∆Tn3 фланкирован целыми последовательностями IS26. Примеры разрушения мобильного элемента ISEcp1 инсерционной структурой IS26 описывались ранее в штаммах E. coli и K. pneumoniae, выделенных в Индии в 2003–2005 гг. [24]. Отметим, что высокогомологичные последовательности обнаружены в плазмидах штаммов клебсиелл других групп несовместимости, депонированных в базу данных GenBank.
Все три исследуемых нами штамма клебсиелл содержат детерминанту OXA-48 карбапенемазы, находящуюся в структуре плазмиды IncL/M, однако детальное изучение генетического окружения гена blaOXA-48 выявило отличия между штаммами, что подтверждается результатами филогенетического анализа и свидетельствует о разных источниках приобретения данной плазмиды.
Мобилом штамма K. pneumoniae KP1083 характеризуется более широким набором как плазмид, так и детерминант резистентности и патогенности. Согласно результатам филогенетического анализа нуклеотидная последовательность плазмиды IncHI1B/FIB данного штамма – наиболее близкородственная последовательность плазмиды вирулентности pKP8-2. Сходством является отсутствие детерминант антибиотикорезистентности в структурах обеих плазмид, однако есть и существенные отличия, например у плазмиды pKP8-2 отсутствует репликон FIB, а в структуре плазмиды IncHI1B/FIB исследуемого штамма – кластер генов катехол-содержащего сидерофора (iroBCDN) и регулятора мукоидного фенотипа rmpA. Отметим, что в единый кластер с плазмидами IncHI1B/FIB штамма K. pneumoniae KP1083 и pKP8-2 входит последовательность плазмиды pKpn23412_362 (номер GenBank CP011314.1) штамма K. pneumoniae 234-12 (Германия, 2011 г.), содержащая репликоны IncHI1B/FIB/Q1 [25] и несущая детерминанты множественной лекарственной устойчивости.
В мобиломе штамма K. pneumoniae KP1083 присутствуют контиги неустановленной локализации (плазмидной или хромосомной), нуклеотидная последовательность которых содержит детерминанты антибиотикорезистентности, сцепленные с мобильными элементами. Это связано с широким распространением данных мобильных структур, которые могут встречаться как в хромосоме, так и плазмидах бактерий – представителей семейства Enterobacteriaceae. Так, среди исследуемых штаммов клебсиелл лишь штамм K. pneumoniae KP1083 имеет структуру, содержащую интегразу, встречающуюся у интегронов первого класса. По данным литературы интегроны первого класса наиболее часто детектируются у штаммов K. pneumoniae, продуцирующих бета-лактамазы расширенного спектра [1, 2, 4, 6, 7]. Проведенное исследование in silico не позволило определить целую структуру интегрона, поскольку консервативными участками являются последовательноcти 5'CS (контиг MRYM02000086.1) и 3'CS (контиг MRYM02000104.1) концов, в то время как средняя часть вариабельна и может содержать любой набор кассетных генов [7, 26]. В результате поиска гомологичных последовательностей с помощью сервиса BLASTN относительно базы данных GenBank установлено, что интегроны класса 1, содержащие только кассетные гены dfrA17 и aadA5, имеют широкое распространение среди представителей семейства Enterobacteriaceae, в том числе бактерий родов Salmonella и Shigella, что подтверждается данными литературных источников [7, 22, 27].
Нуклеотидная последовательность еще одного контига штамма K. pneumoniae KP1083 по результатам аннотирования содержит укороченную последовательность гена интергазы интегрона класса 1, сцепленного с детерминантами aadB и aadA1. Описание подобной структуры интегрона первого класса для штаммов K. pneumoniae в литературе отсутствует. Однако анализ последовательности данного контига с помощью сервиса BLASTN относительно полных геномов, представленных в базе данных GenBank, позволил определить высокогомологичные последовательности (более 99%) в составе интегронов класса 1, выявленных у некоторых представителей разных видов бактерий. Например: в структуре плазмиды unitig_1 (номер GenBank CP021733.1) штамма E. coli AR_0114, который содержит кассетные гены aadB, aadA1, clmA6; в транспозоне Tn1412 P. aeruginosa 2293 (L36547.1); в хромосоме Trueperella pyogenes TP1 (номер GenBank CP033902.1); в транспозоне Tn2426 Shigella sonnei (номер GenBank M86913.1); в интегроне In547 P. aeruginosa Pae 2002 (номер GenBank KC879156.1) [28] и др.
Интересным фактом является присутствие нуклеотидной последовательности, содержащей инсерционную структуру ISCR1, с которой сцеплены гены трех детерминант антибиотикоустойчивости. Во многих работах показано, что ISCR может находиться в структуре интегрона, обеспечивая, таким образом, его мобилизацию [7, 22, 27, 28]. В частности, в результате поиска относительно базы данных RefSeq выявлен штамм K. pneumoniae kpneu013, у которого в последовательности одного контига (UWVW01000002.1) участок, содержащий ISCR1 и имеющий уровень идентичности 99% с последовательностью контига MRYM02000063.1, как раз находится в составе интегрона класса 1. Такая же последовательность выявлена и в составе интегрона класса 1 в плазмиде pPMK1-NDM штамма K. pneumoniae PMK1 (номер GenBank CP008933.1). Однако в базе данных описаны штаммы K. pneumoniae, у которых инсерционный элемент не является частью интегрона. В целом необходимо отметить, что высокогомологичная инсерционная структура ISCR1, сцепленная с детерминантами armA, msrE и mphE, имеет широкое распространение среди штаммов K. pneumoniae, выделенных в таких странах, как Непал, Турция, Китай, ЮАР, Филиппины и Пакистан.
Таким образом, результаты проведенного нами исследования выявили неоднородность структуры мобилома штаммов K. pneumoniae, выделенных у пациентов одного травматологического отделения медицинского учреждения и принадлежащих к одному сиквенс-типу. Мобилом одного штамма имеет более сложную структуру, в которой выявлено наличие инсерционной структуры ISCR1, и предположительно двух интегронов класса 1, несущих детерминанты антибиотикорезистентности. Один интегрон с кассетными генами dfrA17 и aadA5 имеет широкое распространение среди представителей разных видов бактерий и является, по-видимому, одним из наиболее “успешных” в распространении детерминант антибиотикорезистентности. Другой интегрон, содержащий гены aadB и aadA1, которые ранее обнаруживались в составе транспозонов, впервые выявлен в структуре мобилома K. pneumoniae. В связи с этим вероятно, что штамм K. pneumoniae KP1083 – источник распространения дополнительных детерминант антибиотикорезистентности среди штаммов не только K. pneumoniae, но и представителей других видов, относящихся к возбудителям нозокомиальных инфекций.
Исследование не имело спонсорской поддержки.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Paczosa M.K., Mecsas J. Klebsiella pneumoniae: going on the offense with a strong defense // Microbiol. Mol. Biol. Rev. 2016. V. 80. № 3. P. 629–661. https://doi.org/10.1128/MMBR.00078-15
Hou X.-H., Song X.-Y., Ma X.-B. et al. Molecular characterization of multidrug-resistant Klebsiella pneumoniae isolates // Braz. J. Microbiol. 2015. V. 46. № 3. P. 759–768. https://doi.org/10.1590/S1517-838246320140138
Ramirez M.S., Traglia G.M., Lin D.L. et al. Plasmid-mediated antibiotic resistance and virulence in gram-negatives: the Klebsiella pneumoniae paradigm // Microbiol. Spectr. 2014. V. 2. № 5. P. 1–15. https://doi.org/10.1128/microbiolspec.PLAS-0016-2013
Navon-Venezia S., Kondratyeva K., Carattoli A. Klebsiella pneumoniae: a major worldwide source and shuttle for antibiotic resistance // FEMS Microbiol. Rev. 2017. V. 41. № 3. P. 252–275. https://doi.org/10.1093/femsre/fux013
Фурсова Н.К., Прямчук С.Д., Абаев И.В. и др. Генетическое окружение генов blaCTX-M, локализованных на конъюгативных плазмидах нозокомиальных изолятов Enterobacteriaceae, выделенных в России в 2003–2007 гг. // Антибиотики и химиотерапия. 2010. Т. 55. № 11–12. С. 3–10.
Partridge S.R. Analysis of antibiotic resistance regions in gram-negative bacteria // FEMS Microbiol. Rev. 2011. V. 35. № 5. P. 820–855. https://doi.org/10.1111/j.1574-6976.2011.00277
Kaushik M., Kumar S., Kapoor R.K. et al. Integrons in Enterobacteriaceae: diversity, distribution and epidemiology // Int. J. Antimicrob. Agents. 2018. V. 51. № 2. P. 167–176. https://doi.org/10.1016/j.ijantimicag.2017.10.004
Bankevich A., Nurk S., Antipov D. et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing // J. Comput Biol. 2012. V. 19. № 5. P. 455–477. https://doi.org/10.1089/cmb.2012.0021
Altschup S.F., Gish W., Miller W. et al. Basic local alignment search tool // J. Mol. Biol. 1990. V. 215. № 3. P. 403–410. https://doi.org/10.1016/S0022-2836(05)80360-2
Carattoli A., Zankari E., Garcia-Fernandez A. et al. PlasmidFinder and pMLST: in silico detection and typing of plasmids // Antimicrob. Agents Chemother. 2014. V. 58. № 7. P. 3895–3903. https://doi.org/10.1128/AAC.02412-14
Siguier P., Perochon J., Lestrade L. et al. ISfinder: the reference centre for bacterial insertion sequences // Nucl. Acids Res. 2005. V. 34. P. 32–36. https://doi.org/10.1093/nar/gkj014
Moura A., Soares M., Pereira C. et al. INTEGRALL: a database and search engine for integrons, integrases and gene cassettes // Bioinformatics. 2009. V. 25. № 8. P. 1096–1098. https://doi.org/10.1093/bioinformatics/btp105
Tatusova T., Ciufo S., Fedorov B. et al. RefSeq microbial genomes database: new representation and annotation strategy // Nucl. Acids Res. 2014. V. 42. P. 553–559. https://doi.org/10.1093/nar/gkt1274
Bertels F., Silander O.K., Pachkov M.l. et al. Automated reconstruction of whole-genome phylogenies from short-sequence reads // Mol. Biol. Evol. 2014. V. 31. № 5. P. 1077–1088. https://doi.org/10.1093/molbev/msu088
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Saitou N., Nei M. The neighbor-joining method: A new method for reconstructing phylogenetic trees // Mol. Biol. Evol. 1987. № 4. P. 406–425. https://doi.org/10.1093/oxfordjournals.molbev.a040454
Алексеева А.Е., Бруснигина Н.Ф., Солнцев Л.А. и др. Молекулярное типирование клинических изолятов Klebsiella pneumoniae, продуцирующих бета-лактамазы расширенного спектра действия // Клин. лаб. диагн. 2017. Т. 62. № 11. С. 699–704. https://doi.org/10.18821/0869-2084-2017-62-11-699-704
Villa L., Poirel L., Nordmann P. et al. Complete sequencing of an IncH plasmid carrying the blaNDM-1, blaCTX-M-15 and qnrB1 genes // J. Antimicrob. Chemother. 2012. V. 67. № 7. P. 1645–1650. https://doi.org/10.1093/jac/dks114
Giani T., Conte V., Di Pilato V. et al. Escherichia coli from Italy producing OXA-48 carbapenemase encoded by a novel Tn1999 transposon derivative // Antimicrob. Agents Chemother. 2012. V. 56. № 4. P. 2211–2213. https://doi.org/10.1128/AAC.00035-12
Poirel L., Bonnin R. A., Nordmann P. Genetic features of the widespread plasmid coding for the carbapenemase OXA-48 // Antimicrob. Agents Chemother. 2012. V. 56. № 1. P. 559–562. https://doi.org/10.1128/AAC.05289-11
Bailey J.K., Pinyon J.L., Anantham S. et al. Distribution of the blaTEM gene and blaTEM-containing transposons in commensal Escherichia coli // J. Antimicrob. Chemother. 2011. V. 66. № 4. P. 745–751. https://doi.org/10.1093/jac/dkq529
Rui Y., Lu W., Li S. et al. Integrons and insertion sequence common region 1 (ISCR1) of carbapenem-non-susceptible Gram-negative bacilli in fecal specimens from 5000 patients in southern China // Int. J. Antimicrob. Agents. 2018. V. 52. № 5. P. 571–576. https://doi.org/10.1016/j.ijantimicag.2018.06.015
Прямчук С.Д. Идентификация специфичных маркеров для характеристики множественно-устойчивых госпитальных штаммов Enterobacteriaceae: Дис. … канд. биол. наук. Оболенск: Гос. науч. центр прикл. микробиологии и биотехнологии, 2011. 191 с.
Ensor V.M., Shahid M., Evans J.T. et al. Occurrence, prevalence and genetic environment of CTX-M beta-lactamases in Enterobacteriaceae from Indian hospitals // J. Antimicrob. Chemother. 2006. V. 58. № 6. P. 1260–1263. https://doi.org/10.1093/jac/dkl422
Becker L., Bunk B., Eller C. et al. Complete genome sequence of a CTX-M-15-producing Klebsiella pneumoniae outbreak strain from multilocus sequence type 514 // Genome Announc. 2015. V. 3. № 4. 2 p. https://doi.org/10.1128/genomeA.00742-15
Ильина Т.С. Мобильные ISCR-элементы: структура, функции и роль в создании, наращивании и распространении блоков бактериальных генов множественной резистентности к антибиотикам // Мол. ген. микробиол. вирусол. 2012. № 4. С. 3–13.
Tomova A., Ivanova L., Buschmann A.H. Plasmid-mediated quinolone resistance (PMQR) genes and class 1 integrons in quinolone-resistant marine bacteria and clinical isolates of Escherichia coli from an aquacultural area // Microb. Ecol. 2018. V. 75. № 1. P. 104–112. https://doi.org/10.1007/s00248-017-1016-9
Quiroga M.P., Arduino S.M., Merkier A.K. et al. Distribution and functional identification of complex class 1 integrons // Infect. Genet. Evol. 2013. V. 19. P. 88–96. https://doi.org/10.1016/j.meegid.2013.06.029
Дополнительные материалы отсутствуют.


