Генетика, 2020, T. 56, № 5, стр. 609-614
Эволюция гена Btr1-А у диплоидных видов пшениц рода Triticum L.
В. Ю. Вавилова 1, *, И. Д. Конопацкая 1, А. Г. Блинов 1, Н. П. Гончаров 1
1 Федеральный исследовательский центр Институт цитологии и генетики
Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
* E-mail: valeriya-vavilova@bionet.nsc.ru
Поступила в редакцию 05.08.2019
После доработки 02.09.2019
Принята к публикации 17.10.2019
Аннотация
При доместикации злаков важнейшими признаками были легкость обмолота (пленчатость–голозерность) и ломкость/неломкость колоса, и человек вел отбор на голозерность и неломкость колоса. У пшеницы выявлены три системы генов, контролирующих признак “ломкость колоса” и/или его выраженность. Одна из них обусловлена генами Btr1 (Non-brittle rachis), локализованными в хромосомах 3-й гомеологической группы. В данной работе были изучены последовательности 5'-нетранслируемого, кодирующего и 3'-нетранслируемого районов гена Btr1-A, расположенного в 3А хромосоме, у 30 образцов четырех диплоидных диких и возделываемых видов пшениц Triticum boeoticum, T. monococcum, T. sinskajae и T. urartu из Южной Европы, Закавказья и Малой Азии. Последовательность гена Btr1-A вида T. sinskajae была изучена впервые. Всего у изученных образцов диплоидных пшениц было установлено 11 гаплотипов этого гена, причем пять из них, а именно Нар12–Нар16, описаны впервые. Вариабельность ранее не описанных гаплотипов у T. boeoticum и T. urartu не затрагивает критическую замену в позиции 355 гена Btr1-A (G на А), характерную для ломкоколосых диплоидных пшениц. Последовательности этого гена у T. sinskajae и всех образцов T. monococcum были идентичны гаплотипу Нар9, выявленному у всех ранее изученных образцов T. monococcum. В позиции 355 гена Btr1-A у этого гаплотипа произошла замена G на А, что приводит к возникновению признака “неломкоколосость”. Обсуждается включение в голозерные полиплоидные виды пшениц вариантов гаплотипов диплоидных видов, обусловливающих у них признак “отсутствие ломкости колоса”.
Ломкость колоса (ломкоколосость) — ключевой хозяйственно важный признак пшениц, тесно связанный с процессом их доместикации и последующей селекции [1]. У диких видов пшениц созревший колос распадается на колоски, что обеспечивает эффективное распространение их семян [1–3]. Среди возделываемых пшениц диплоидный вид Triticum monococcum L. и тетраплоидные T. dicoccum (Schrank) Schuebl. и T. timopheevii (Zhuk.) Zhuk. характеризуются ломкоколосостью — после созревания колос у них ломается от механического воздействия [4]. У ди-, тетра- и гексаплоидных видов пшениц выраженность признака “ломкость колоса” имеет различную степень проявления [4]. В то же время созревший колос всех широко культивируемых в настоящее время полиплоидных видов пшениц не ломается, что облегчает сбор урожая и, как следствие, увеличивает количество собранного зерна. Три независимые системы генов контролируют признак “ломкость колоса” и его выраженность: Q [5–7], Tg [8], Btr [9, 10], причем две первые плейотропно контролируют еще и пленчатость–голозерность [5, 8, 11]. При этом ген Q контролирует признаки, по которым шла доместикация [5–7], а ген Tg встречается только у искусственно созданных амфиплоидов [8, 12]. Следует отметить, что у пшениц система генов Btr1 (Non-brittle rachis 1) наименее изучена.
Пукарендиш и соавт. [9] изучили гены Btr1-А и Btr2-А у диплоидных пшениц-однозернянок, культивируемой T. monococcum и дикой T. boeoticum Boiss. и выявили единичную нуклеотидную замену в гене Btr1-А (аллель btr1-Am с заменой G на А в позиции 355), приводящую к повышению прочности оси сложного колоса у культивируемых сортов. Позднее Чжао и соавт. проанализировали ген Btr1-А у ди- и полиплоидных видов пшениц и показали, что в отличие от дикого тетраплоидного вида T. dicoccoides (Körn. ex Aschers. et Graebn.) Schweinf. ген Btr1-А культивируемых полиплоидных пшениц содержит делецию из двух нуклеотидов, которая приводит к сдвигу рамки считывания и формированию укороченного нефункционального белка. Исследования на трансгенных линиях показали, что Btr1-А, вероятно, также оказывает влияние на морфологию колоса, размер и массу зерновок пшеницы [10].
В настоящем исследовании было изучено разнообразие гена Btr1-А у диплоидных видов пшениц T. boeoticum, T. monococcum, T. sinskajae A. Filat. et Kurk. и T. urartu Thum. ex Gandil., в том числе у образцов из ранее неизученных регионов (табл. 1). Для анализа было выбрано 30 образцов, для каждого из которых в условиях тепличного комплекса при стандартных условиях было выращено по 10 растений. Растения выращивались до полного созревания, затем для них был определен признак “ломкость колоса”. Колосья всех проанализированных образцов T. monococcum характеризуются пониженной ломкостью, а T. sinskajae – неломкостью колоса, в то время как все исследованные образцы T. urartu и T. boeoticum – ломким колосом. Кроме того, T. sinskajae – это единственный голозерный вид диплоидных пшениц [13].
Таблица 1.
Список проанализированных образцов диплоидных пшениц и выявленные гаплотипы гена Btr1-А
| № | Вид | Номер в генетическом банке | Место сбора | Гаплотип Btr1-А |
|---|---|---|---|---|
| 1 | T. boeoticum | AUS 19372, Australian Grain Genebank | Иран | Hap3 |
| 2 | T. boeoticum | к-14384, ВИР | Турция | Hap12 |
| 3 | T. boeoticum | IG 116198, ICARDA | Турция | Hap7 |
| 4 | T. boeoticum | AUS 19375, Australian Grain Genebank | Иран | Hap3 |
| 5 | T. boeoticum | к-61600, ВИР | Греция | Hap2 |
| 6 | T. boeoticum | G2511, University of California | Иран | Hap3 |
| 7 | T. boeoticum | G2523, University of California | Иран | Hap13 |
| 8 | T. boeoticum | к-33869, ВИР | Армения | Hap12 |
| 9 | T. boeoticum | к-18399*, ВИР | Турция | Hap12 |
| 10 | T. boeoticum | к-28132, ВИР | Армения | Hap7 |
| 11 | T. boeoticum | к-28280, ВИР | Азербайджан, Нахичевань | Hap12 |
| 12 | T. boeoticum | FN353, ICG | Не известно | Hap7 |
| 13 | T. boeoticum | FN346, ICG | Не известно | Hap8 |
| 14 | T. boeoticum | FN340, ICG | Не известно | Hap7 |
| 15 | T. boeoticum | PI 355517, SGC | Турция | Hap14 |
| 16 | T. monococcum | PI 277137, SGC | Греция | Hap9 |
| 17 | T. monococcum | PI 326317, SGC | Азербайджан | Hap9 |
| 18 | T. monococcum | PI 306540, SGC | Румыния | Hap9 |
| 19 | T. monococcum | PI 503874, SGC | Иран | Hap9 |
| 20 | T. monococcum | PI 355537, SGC | Турция | Hap9 |
| 21 | T. monococcum | PI 427959, SGC | Ирак | Hap9 |
| 22 | T. monococcum | PI 427927, SGC | Иран | Hap9 |
| 23 | T. monococcum | PI 393496, SGC | Израиль | Hap9 |
| 24 | T. monococcum | PI 264935, SGC | Греция | Hap9 |
| 25 | T. monococcum | PI 221329, SGC | Сербия | Hap9 |
| 26 | T. monococcum | PI 377662, SGC | Сербия | Hap9 |
| 27 | T. monococcum | PI 355523, SGC | Малая Азия | Hap9 |
| 28 | T. sinskajae | к-48993, ВИР | Турция | Hap9 |
| 29 | T. urartu | IG 110784*, ICARDA | Сирия | Hap15 |
| 30 | T. urartu | IG 116196, ICARDA | Турция | Hap16 |
Примечание. Полужирным шрифтом выделены варианты, впервые описанные в настоящем исследовании. SGC – the Small Grain Collection, Aberdeen, USA. * Короткий фрагмент гена Btr1-A был установлен в работе В. Вавиловой и соавт. [14].
С целью установления последовательностей гена Btr1-А, включая также 5'- и 3'-нетранслируемые районы данного гена, дополнительно к паре праймеров Btr-A1-F и Btr-A1-R (целевой фрагмент ~900 пн) [14] были сконструированы две пары праймеров: Btr-A1-F1 5'-GGTTTCAGAATCCAAGGGG-3' и Btr-A1-R1 5'-TTACCACTTCCTCGTGCG-3' (целевой фрагмент ~700 пн); Btr-A1-F2 5'- GCATCCAGTGACAGGTCTCTT-3' и Btr-A1-R2 5'-GTTCGCTACCACACGTGCAA-3' (целевой фрагмент ~900 пн).
ПЦР-амплификация целевых фрагментов была проведена с использованием образцов тотальной ДНК, выделенной из листьев растений диплоидных пшениц. Секвенирование нуклеотидных последовательностей было проведено в ЦКП “Геномика” СО РАН (http://sequest.niboch.nsc.ru/). Полученные фрагменты были собраны в полноразмерные последовательности гена Btr1-А с использованием алгоритма MUSCLE [15] в программе AliView v. 1.18.1 [16]. Нуклеотидные последовательности новых гаплотипов гена Btr1-А были размещены в базе данных GenBank под номерами MN159595–MN159605.
С использованием алгоритма MUSCLE [15] в программе AliView v. 1.18.1 [16] было проведено сравнение полученных последовательностей с последовательностями генов Btr1-A диплоидных и полиплоидных видов пшениц, представленными в базе данных GenBank [9, 10, 14]. Филогенетический анализ последовательностей был выполнен с использованием программы IQ-TREE версии 1.6.11 [17].
У всех проанализированных образцов диплоидных пшениц не было выявлено гетерозигот по гену Btr1-A. Среди образцов T. boeoticum выявлены четыре гаплотипа из восьми описанных ранее: Hap2, Hap3, Hap7 и Hap8 [9, 10, 14] (табл. 1). Кроме того, у данного вида в настоящем исследовании были установлены три новых гаплотипа, обозначенных нами Нар12, Нар13 и Нар14. Гаплотип Нар12 отличается от гаплотипа Нар1 одной однонуклеотидной заменой в 3'-нетранслируемом районе, отличий между гаплотипами в 5'-нетранслируемом районе и кодирующей последовательности гена Btr1-A не выявлено. Отличие гаплотипа Нар13 от Нар1 заключается в двух однонуклеотидных заменах в 5'-нетранслируемом районе и одной однонуклеотдиной замене в 3'‑нетранслируемом районе гена Btr1-A. Гаплотип Нaр12 был выявлен у образцов, собранных на территории Армении, Турции и Азербайджана (Нахичевань), а гаплотип Нар13 уникален — обнаружен только в одном образце с территории Ирана (рис. 1,а). Гаплотипы Нар12 и Нар13 сформировались в популяциях T. boeoticum, и согласно проведенному филогенетическому анализу предком гаплотипа Нар12, вероятнее всего, является гаплотип Нар2, в то время как гаплотип Нар13, по всей видимости, происходит от гаплотипа Нар3 (рис. 1,б). Гаплотипы Нар1 и Нар4–Нар6, ранее описанные у T. boeoticum [9], среди проанализированных нами образцов не выявлены. У образца T. boeoticum PI 355517, собранного на территории Турции, выявлен гаплотип, отличающийся от Нар1 двумя однонуклеотидными заменами в 5'‑нетранслируемом районе и четырьмя однонуклеотидными заменами в 3'-нетранслируемом районе. Данный гаплотип был обозначен нами как Нар14. Эволюционно он ближе к гаплотипам Нар5, Нар7 и Нар8 вида T. boeoticum (см. рис. 1,б).
Рис. 1.
Гаплотипы Btr1-A у образцов диплоидных видов пшениц. а – географическое распределение гаплотипов на основании результатов настоящего исследования и литературных данных [9]; б – филогенетическое дерево, реконструированное на базе 5'-нетранслируемого района, кодирующей части и 3'-нетранслируемого района гена Btr1-A, с использованием программы IQ-TREE версии 1.6.11 [17]. Коэффициенты поддержки менее 50% не представлены. Полужирным шрифтом выделены гаплотипы, впервые описанные (б) и представленные на территории изучаемых стран (а).
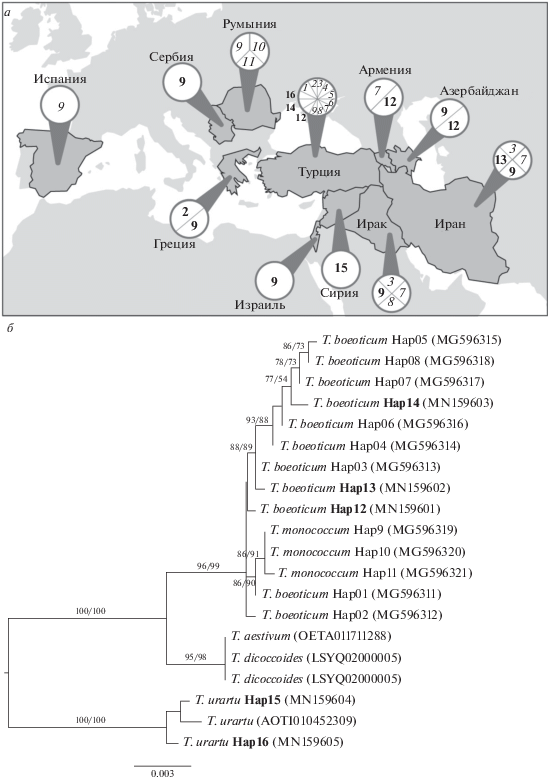
У всех 12 изученных нами образцов T. monococcum был идентифицирован ранее описанный гаплотип Нар9. Он характеризуется заменой нуклеотида G на А, что приводит к несинонимичной замене в позиции 355 (аллель btr1-Am) и обеспечивает у данного вида формирование почти неломкого колоса [9]. Гаплотип Нар9 широко представлен среди образцов T. monococcum и география его распространения включает Испанию, Грецию, Азербайджан, Румынию, Иран, Турцию, Ирак, Израиль, Сербию и Малую Азию (рис. 1) [9]. По всей видимости, он сформировался из гаплотипа Нар1 T. boeoticum. Гаплотипы T. monococcum Нар10 и Нар11 согласно филогенетическому анализу, представленному на рис. 1,б, произошли от гаплотипа Нар9.
Ген Btr1-A неломкоколосого образца к-48993 T. sinskajae был проанализирован впервые и был идентичен последовательности гаплотипа Нар9 T. monococcum (табл. 1), что дополнительно подтверждает происхождение T. sinskajae от T. monococcum. Данный результат также позволяет сделать вывод о том, что у T. sinskajae отсутствие ломкости колоса контролируется иным геном.
В настоящем исследовании для T. urartu были выявлены два новых варианта гаплотипов: Нар15 и Нар16 (табл. 1). Ранее для данного вида был описан только один вариант Нар1 с G в позиции 355 гена Btr1-A [10]. Нар15 отличается от Нар1 46 однонуклеотидными заменами и двумя однонуклеотидными делециями. Данный гаплотип был выявлен у образца T. urartu (IG 110784), собранного на территории Сирии. Гаплотип Нар16 (T. urartu IG 116196) отличается от Нар1 двумя однонуклеотидными делециями, идентичными таковым гаплотипа Нар15, и 46 однонуклеотидными заменами, отличными от замен, показанных для Нар15. Для Нар16 в кодирующем районе Btr1-A выявлены четыре однонуклеотидные замены: две синонимичные, а также две несинонимичные, приводящие к замене аргинина на глутамин в позиции 124 и аланина на пролин в позиции 149. У гаплотипа Нар15 в дополнение к вышеописанным заменам в позиции 42 установлена замена лейцина на глутамин. Гаплотипы, выявленные у образцов T. urartu, содержат нуклеотид G в позиции 355, что характерно для ломкоколосых диплоидных пшениц, и не содержат делецию длиной 2 пн, описанную ранее у полиплоидных видов рода Triticum [10].
Таким образом, в рамках настоящего исследования были установлены и проанализированы последовательности 5'-нетранслируемого, кодирующего и 3'-нетранслируемого районов гена Btr1-A у четырех диплоидных видов пшениц из Южной Европы, Закавказья и Малой Азии. Всего было установлено 11 различных гаплотипов этого гена, причем пять из них обнаружены впервые: два у T. urartu (Нap15 и Нap16) и три у T. boeoticum (Нap12–Нap14). В 355-й позиции гена Btr1-A у всех образцов с ломким колосом был выявлен нуклеотид G, а у образцов с неломким колосом – нуклеотид А, что полностью согласуется с данными, представленными в литературе. Заметим, что остальные из выявленных нами замен не изменяют функции проанализированного гена. Сравнительный анализ показал, что Btr1-A полиплоидных видов обладает более высоким сходством с последовательностями диплоидных видов пшениц T. monococcum и T. boeoticum, чем с последовательностями трех изученных образцов T. urartu (см. рис. 1).
Исследование выполнено за счет гранта Российского научного фонда (№ 16-16-10021).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Kimber G., Feldman M. Wild Wheat: An Introduction. College of Agriculture, Univ. Missouri-Columbia, 1987. Special reports № 363. 146 p.
Watanabe N., Sugiyama K., Yamagishi Y., Sakata Y. Comparative telosomic mapping of homoeologous genes for brittle rachis in tetraploid and hexaploid wheats // Hereditas. 2002. V. 137. P. 180–185. https://doi.org/10.1034/j.1601-5223.2002.01609.x
Li W., Gill B.S. Multiple genetic pathways for seed shattering in the grasses // Funct. Integr. Genomics. 2006. V. 6. P. 300–309. https://doi.org/10.1007/s10142-005-0015-y
Дорофеев В.Ф., Филатенко А.А., Мигушова Э.Ф. и др. Пшеница. Л.: Колос, 1979. 348 с.
Zhang Z., Belcram H., Gornicki P. et al. Duplication and partitioning in evolution and function of homoeologous Q loci governing domestication characters in polyploid wheat // Proc. Natl Acad. Sci. USA. 2011. V. 108. P. 18737–18742. https://doi.org/10.1073/pnas.1110552108
Simons K.J., Fellers J.P., Trick H.N. et al. Molecular characterization of the major wheat domestication gene Q // Genetics. 2006. V. 172. P. 547–555. https://doi.org/10.1534/genetics.105.044727
Sormacheva I., Golovnina K., Vavilova V. et al. Q gene variability in wheat species with different spike morphology // Genet. Resour. Crop Evol. 2015. V. 62. P. 837–852. https://doi.org/10.1007/s10722-014-0195-1
Sharma J.S., Running K.L.D., Xu S.S. et al. Genetic analysis of threshability and other spike traits in the evolution of cultivated emmer to fully domesticated durum wheat // Mol. Genet. Genomics. 2019. V. 294. P. 757–771. https://doi.org/10.1007/s00438-019-01544-0
Pourkheirandish M., Dai F., Sakuma S. et al. On the origin of the non-brittle rachis trait of domesticated einkorn wheat // Front. Plant Sci. 2018. V. 8. P. 2031. eCollection 2017https://doi.org/10.3389/fpls.2017.02031
Zhao Y., Xie P., Guan P. et al. Btr1-A induces grain shattering and affects spike morphology and yield-related traits in wheat // Plant Cell Physiol. 2019. V. 60. № 6. P. 1342–1353. https://doi.org/10.1093/pcp/pcz050
Sood S., Kuraparthy V., Bai G., Gill B.S. The major threshability genes soft glume (sog) and tenacious glume (Tg), of diploid and polyploid wheat, trace their origin to independent mutations at non-orthologous loci // Theor. Appl. Genet. 2009. V. 119. P. 341–351. https://doi.org/10.1007/s00122-009-1043-0
Nalam V., Vales M.I., Watson C.J.W. et al. Map based analysis of genetic loci on chromo-some 2D that affect glume tenacity and threshability, components of the free-threshing habit in common wheat (Triticum aestivum L.) // Theor. Appl. Genet. 2007. V. 116. P. 135–145. https://doi.org/10.1007/s00122-007-0653-7
Гончаров Н.П., Кондратенко Е.Я., Банникова С.В. и др. Сравнительно-генетический анализ голозерной диплоидной пшеницы Triticum sinskajae и ее исходной формы T. monococcum // Генетика. 2007. Т. 43. № 11. С. 1491–1500.
Vavilova V., Konopatskaia I., Goncharov N.P. Non-brittle rachis 1-A (Btr1-A) gene in di- and hexaploid wheat species // Current Challenges in Plant Genetics, Genomics, Bioinformatics, and Biotechnology. Proc. Fifth Intern. Sci. Conf. Plant Gen. 2019. P. 202–214. https://doi.org/10.18699/ICG-PlantGen2019-65
Edgar R.C. MUSCLE: multiple sequence alignment with high accuracy and high throughput // Nucl. Acids Res. 2004. V. 32. P. 1792–1797. https://doi.org/10.1093/nar/gkh340
Larsson A. AliView: a fast and lightweight alignment viewer and editor for large datasets // Bioinformatics. 2014. V. 30. № 22. P. 3276–3278. https://doi.org/10.1093/bioinformatics/btu531
Nguyen L.T., Schmidt H.A., von Haeseler A., Minh B.Q. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies // Mol. Biol. Evol. 2015. V. 32. № 1. P. 268–274. https://doi.org/10.1093/molbev/msu300
Дополнительные материалы отсутствуют.


