Генетика, 2020, T. 56, № 6, стр. 732-738
Анализ родоплеменной структуры тувинцев по маркерам Y-хромосомы
М. К. Жабагин 1, *, Л. Д. Дамба 2, 3, Н. А. Короткова 3, 4, Д. Н. Чернышенко 3, 5, С. А. Утриван 3, 5, В. Ю. Пылёв 3, М. В. Олькова 3, Е. В. Балановская 3, 4, Н. К. Янковский 5, О. П. Балановский 3, 4, 5
1 Национальный центр биотехнологии Комитета науки Министерства образования и науки
Республики Казахстан
010000 Нур-Султан, Казахстан
2 Научно-исследовательский институт медико-социальных проблем и управления
Республики Тыва
667003 Кызыл, Россия
3 Медико-генетический научный центр им. академика Н.П. Бочкова
115478 Москва, Россия
4 Биобанк Северной Евразии
115201 Москва, Россия
5 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
* E-mail: mzhabagin@gmail.com
Поступила в редакцию 23.05.2019
После доработки 17.07.2019
Принята к публикации 23.07.2019
Аннотация
По данным о полиморфизме Y-хромосомы дана оценка влияния на структуру генофонда тувинцев двух факторов: родоплеменной (роды́) и административно-территориальной (кожууны) структуры. В этот анализ включены десять наиболее распространенных родóв тувинцев (ак, бараан, иргит, кол, кыргыс, монгуш, ооржак, оюн, хертек, чооду), охватывающих две трети общей выборки (N = 545) тувинцев из шести кожуунов (Барун-Хемчикского, Тандынского, Тере-Хольского, Тоджинского, Чаа-Хольского, Эрзинского). Генетические портреты родóв, созданные по 52 SNP-маркерам Y-хромосомы, выявили эффект основателя для всех родóв, за исключением двух наиболее крупных – кыргыс и монгуш, охватывающих более трети выборки и являющихся конгломератами. Анализ молекулярной дисперсии (AMOVA) указывает на равную степень генетической дифференциации родóв (5.2%) и кожуунов (6.8%). Но тест Мантеля обнаруживает высокую достоверную корреляцию (r = 0.50) между генетической и родовой структурами на фоне недостоверной (r = 0.25) корреляции между генетическими и географическими расстояниями. Причина различий между результатами тестов AMOVA и Мантеля в том, что четыре наиболее многочисленных кожууна являются “моноклановыми” (в их населении превалирует один род), и лишь в двух кожуунах (Тоджинском и Тере-Хольском) представители трех разных родóв распределены равномерно. Выделение на графике многомерного шкалирования западного и восточного кластеров хорошо согласуется с данными антропологии (саянский и катангский антропологические типы), но не подтверждает ни одну из этнографических версий этногенеза тувинцев (“самодийскую”, “монгольскую”, “тюркскую”). Совокупность результатов показывает, что архитектонику генофонда тувинцев наиболее полно отражает их родоплеменная структура, которую необходимо учитывать в популяционно-генетических исследованиях.
Горные хребты Алтая и Саян являются географической и генетической границей между популяциями сибирской тайги и центральноазиатских степей [1]. В то же время горная котловина Саяно-Алтая в разные времена была местом, где происходило взаимодействие племен и народов, говоривших на языках самодийской, енисейской, тюркской, монгольской и тунгусской лингвистических групп [2]. Представителями генетических наследников этих племен являются современные тувинцы и тоджинцы, изолированно проживающие в горных Саянах и в верховьях Енисея на границе с Монголией.
Отличительной чертой тувинцев (чья численность составляет более 260 тыс. человек, согласно http://www.gks.ru/) по сравнению со многими другими коренными народами Сибири является высокая брачная этническая ассортативность [3]. Однако при высоком уровне инбридинга, рассчитанного изонимным методом, его неслучайная компонента в популяциях с большим накоплением общих фамилий может указывать на избегание тувинцами эндогамных браков. Эту гипотезу подтверждают высокие значения генетического разнообразия по классическим маркерам [4], аутосомным микросателлитным и Alu-повторам [5], мтДНК [6] и Y-хромосоме [7].
Как и у большинства тюркоязычных популяций Евразии, родоплеменная система тувинцев состоит из патрилокальных групп (родóв, кланов), традиционно отслеживавших патрилинейность и практиковавших экзогамные браки. Устойчивость родоплеменной системы в качестве социального института отражена в этнографической истории тувинцев [2], однако сложная политическая история тувинцев внесла значительные изменения в традиционный уклад их жизни. В течение продолжительного периода своей истории (1757–1912 гг.) тувинцы находились в составе Цинской империи, военно-административные единицы которой включали множество родóв. В ХХ в. (1912–1944 гг.) тувинцы отделились от Китая в составе Монголии, сохранив основу родовой организации [8]. При паспортизации населения после вступления Тувы в состав СССР (1944 г.) в качестве фамилии использовалось имя человека, а в качестве имени – его родоплеменное название. Поэтому стандартная процедура определения родовой принадлежности по фамилии для тувинцев малопригодна и реконструируется с помощью генеалогии и информантов. Родоплеменная структура эффективно отражает архитектонику генофонда и демографическую историю населения, что было неоднократно и независимо показано для разных регионов Северной Евразии: Средней Азии [9, 10], Урала [11] и Сибири [7, 12 ]. В основе ее эффективности лежит принцип наследования “от отца к сыну” и “принадлежности к роду”, что соответствует Y-хромосомному типу наследования.
Полиморфизм Y-хромосомы тувинцев ранее изучался [7, 13 ] без учета родоплеменной структуры. Генетические исследования полиморфизма Y-хромосомы были проведены лишь для ограниченного количества тувинских родóв (монгуш, ооржак, кол, оюн и хертек) [14, 15], и существовала необходимость более глубокого генетического изучения тувинских родóв. По этой причине целью настоящего исследования является анализ родоплеменной структуры тувинцев по маркерам Y-хромосомы и решение вопроса: насколько генетическая архитектоника генофонда тувинцев структурирована родоплеменной (роды́) или же административно-территориальной (кожууны) организацией.
Материалом для исследования послужили 545 образцов венозной крови тувинцев (включая тоджинцев) из шести кожуунов Тувы (рис. 1): Барун-Хемчикского (N = 89), Тандынского (N = 90), Тере-Хольского (N = 90), Тоджинского (N = 90), Чаа-Хольского (N = 94), Эрзинского (N = 92). Образцы собраны согласно принципам организации Биобанка Северной Евразии [16] под контролем Этической комиссии Медико-генетического научного центра. Геномная ДНК выделена методом фенол-хлороформной экстракции. Все образцы генотипированы по единой панели 52 SNP-маркеров Y-хромосомы (M130, M217, F2613, CTS4021, M407, Z12266, F2386, F1788, F3918, F3830, F3791, M48, M86, M174, M69, M170, M253, M223, P37, M304, M267, M172, M47, M67, M92, M12, M9, M20, M231, LLY22g, M178, L708, L666, VL29, F4205, P31, M119, M122, M242, M120, M378, M207, M198, M458, M343, L23, L51, Z2103, M73, M269, M124, M70) методом ПЦР в реальном времени с использованием TaqMan зондов. Номенклатура гаплогрупп дана согласно ISOGG (https://isogg.org), для субветвей гаплогруппы N – согласно [17]. Для сравнительного анализа привлечены данные [7, 14, 15]. По частотам гаплогрупп рассчитаны генетические расстояния Нея [18] с помощью программы DJgenetic (www.genofond.ru). Визуализация итогов проведена методом многомерного шкалирования в программе Statistica 6.0 (StatSoft Inc., 2001). Генетическая дифференциация популяций оценена методом AMOVA и тестом Мантеля в программе Arlequin 3.5.1.3 [19].
Рис. 1.
Положение тувинских родóв и географических популяций в пространстве многомерного шкалирования на основе матриц генетических расстояний по частотам гаплогрупп Y-хромосомы. Величина стресса – 0.079, алиенация – 0.098. Названия родóв даны курсивом и выделены ромбиками. Треугольниками показаны популяции из работы В.Н. Харьков и др. [7].
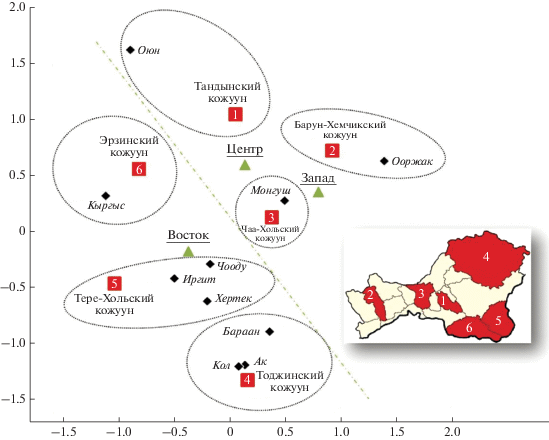
Среди обследованных тувинцев (N = 545) было обнаружено 32 рода, из которых большинство (22) очень редки: каждый из них занимает менее 3% выборки, по этой причине из дальнейшего генетического анализа они были исключены. Треть родов (N = 10) охватывает три четверти (74%) общей выборки тувинцев, в табл. 1 даны генетические портреты этих десяти родов по SNP-маркерам Y-хромосомы. Однако около 40% тувинцев из десяти наиболее частых родóв относится к двум родáм – кыргыс (20%) и монгуш (18%), а представители каждого из остальных восьми родóв (ак, бараан, иргит, кол, ооржак, оюн, хертек, чооду) встречаются в популяции с частотой 4–6%.
Таблица 1.
Частоты гаплогрупп Y-хромосомы в тувинских родáх
| Гаплогруппа Y-хромосомы | Тувинцы | Родоплеменные группы тувинцев | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| ак | бараан | иргит | кол | кыргыс | монгуш | ооржак | оюн | хертек | чооду | ||
| N = 405 | N = 23 | N = 21 | N = 20 | N = 25 | N = 109 | N = 98 | N = 28 | N = 29 | N = 30 | N = 22 | |
| C2*-M217 | 0 | 0 | 0 | 0 | 0 | 0.01 | 0 | 0 | 0 | 0 | 0 |
| C2a1a1b1a-F3830 | 0 | 0 | 0 | 0 | 0 | 0.02 | 0 | 0 | 0 | 0 | 0 |
| C2a1a2a-M86 | 0.06 | 0.04 | 0.05 | 0 | 0 | 0.05 | 0.14 | 0.04 | 0 | 0 | 0.05 |
| C2a1a3-F3791 | 0.05 | 0 | 0 | 0.15 | 0 | 0.11 | 0 | 0.07 | 0.03 | 0.03 | 0.09 |
| C2b1a*-CTS4021(xM407) | 0 | 0 | 0 | 0 | 0 | 0.01 | 0 | 0 | 0 | 0 | 0 |
| C2b1a1a1a-M407 | 0.05 | 0 | 0 | 0.05 | 0 | 0.17 | 0 | 0 | 0 | 0.03 | 0.09 |
| C2b1b-F845 | 0 | 0 | 0 | 0 | 0 | 0.02 | 0 | 0 | 0 | 0 | 0 |
| D-M174 | 0.01 | 0 | 0 | 0 | 0 | 0.03 | 0 | 0 | 0 | 0 | 0 |
| I1-M253 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0.03 | 0 |
| J2*-M172(xM12,M47,M67) | 0.01 | 0 | 0 | 0 | 0 | 0.01 | 0 | 0 | 0.07 | 0.03 | 0 |
| N2a-L666 | 0.20 | 0.22 | 0.24 | 0.20 | 0.24 | 0.16 | 0.21 | 0.14 | 0.24 | 0.20 | 0.32 |
| N3*-M178(L708) | 0 | 0 | 0 | 0 | 0 | 0.01 | 0.01 | 0 | 0 | 0 | 0 |
| N3a*-L708(xF4205,VL29) | 0.05 | 0 | 0 | 0 | 0 | 0.01 | 0.09 | 0.32 | 0 | 0 | 0 |
| N3a5-F4205 | 0.09 | 0.13 | 0.14 | 0.10 | 0.08 | 0.04 | 0.12 | 0.14 | 0 | 0.10 | 0.09 |
| N3a3-VL29 | 0 | 0 | 0.05 | 0 | 0.04 | 0 | 0 | 0 | 0 | 0 | 0 |
| O1a-M119 | 0.03 | 0 | 0 | 0.05 | 0 | 0.06 | 0 | 0 | 0 | 0.10 | 0.05 |
| O2-M122 | 0.02 | 0 | 0 | 0 | 0.04 | 0.02 | 0.03 | 0.04 | 0.03 | 0.03 | 0.05 |
| Q-M242 | 0.21 | 0.52 | 0.43 | 0.30 | 0.52 | 0.11 | 0.16 | 0.07 | 0.03 | 0.30 | 0.23 |
| R1a1a*-M198(xM458) | 0.16 | 0.04 | 0.05 | 0.15 | 0.04 | 0.17 | 0.18 | 0.14 | 0.48 | 0.13 | 0.05 |
| R1b*-M343(xM269) | 0.01 | 0 | 0 | 0 | 0 | 0 | 0.03 | 0.04 | 0.07 | 0 | 0 |
| R1b1a1b1*-L23(xL51,Z2103) | 0 | 0 | 0.05 | 0 | 0.04 | 0 | 0 | 0 | 0 | 0 | 0 |
| R1b1a1b1a-L51 | 0 | 0 | 0 | 0 | 0 | 0.01 | 0 | 0 | 0 | 0 | 0 |
| R1b1a1b1b-Z2103 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0.03 | 0 | 0 |
| R2a-M124 | 0 | 0 | 0 | 0 | 0 | 0 | 0.01 | 0 | 0 | 0 | 0 |
| T1a-M70 | 0 | 0.04 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Разнообразие (HD) | 0.87 ± 0.01 | 0.69 ± 0.08 | 0.77 ± 0.07 | 0.85 ± 0.05 | 0.69 ± 0.08 | 0.89 ± 0.01 | 0.86 ± 0.01 | 0.85 ± 0.04 | 0.72 ± 0.07 | 0.85 ± 0.04 | 0.85 ± 0.05 |
Основная часть генофонда тувинцев (87%) представлена восемью Y-гаплогруппами с частотами более 5%: Q (21%), N2a (20%), R1a1a* (16%), N3a5 (9%), C2a1a2a (6%), C2a1a3 (5%), C2b1a1a1a (5%), N3a* (5%). Однако при учете родоплеменной структуры обнаруживается дифференциация: только три наиболее частые гаплогруппы (Q, N2a, R1a1a*) встречаются во всех десяти родáх. Важно, что частота гаплогруппы N2a во всех родáх составляет не менее 10%, что может указывать на сохранение во всех десяти родáх субстрата самодийских племен. В целом, несмотря на высокое генетическое разнообразие тувинцев (HD = 0.87), генетические портреты большинства родóв свидетельствуют о наличии эффекта основателя. Мажорные гаплогруппы охватывают не менее трети Y-генофонда каждого рода: гаплогруппа Q – у родóв ак (52%), кол (53%), бараан (43%), иргит (30%), хертек (30%); гаплогруппа R1a1a* – у оюн (48%); гаплогруппа N2a – у чооду (32%); гаплогруппа N3a* – у ооржак (32%). Исключением являются только два самых многочисленных рода – кыргыс (HD = 0.86) и монгуш (HD = 0.89): их огромная гетерогенность указывает на то, что они являются конгломератами более мелких родóв. В целом, генетические портреты родов указывают на то, что родоплеменная структура формирует архитектонику генофонда тувинцев.
Для решения вопроса о соотношении ролей родоплеменной и административно-территориальной организации в архитектонике генофонда тувинцев проведен анализ молекулярной дисперсии (AMOVA). Для этого одни и те же образцы были сгруппированы двумя способами: по их принадлежности к десяти наиболее крупным родáм и по месту их рождения – географическим популяциям (шесть кожуунов). Метод AMOVA указывает на практически равную роль обоих факторов: принадлежности к роду (5.2%) и принадлежности к кожууну (6.8%).
Однако корреляционный тест Мантеля (табл. 2) четко указывает, что роль родоплеменной структуры является ключевой. В этом виде анализа были рассчитаны три матрицы: (1) матрица генетических расстояний между кожуунами (территориальными единицами Тувы); (2) матрица географических расстояний (в километрах) между кожуунами; (3) матрица квазигенетических расстояний (родоплеменной структуры). При расчете матрицы квазигенетических расстояний каждый род традиционно рассматривается как аналог одного аллеля нейтрального мультиаллельного локуса (квазигенетического маркера) [20]. Корреляционный анализ этих трех матриц убедительно показал, что корреляция между генетикой и родоплеменной структурой велика и достоверна (r = 0.50; P < 0.05), в то время как корреляция между генетикой и географией в два раза меньше и недостоверна (средняя r = 0.25; P > 0.05).
Таблица 2.
Корреляции между матрицами генетических расстояний, географических расстояний и расстояний по частотам квазигенетических маркеров между популяциями тувинцев
| Матрицы расстояний | Коэффициент корреляции | P-значение |
|---|---|---|
| Генетика и география* | 0.33 | 0.12 |
| Генетика и родоплеменная структура* | 0.54 | 0.04 |
| Генетика и география (без влияния родоплеменной структуры)** | 0.24 | 0.21 |
| Генетика и родоплеменная структура (без влияния географии)** | 0.50 | 0.04 |
Различие между результатами AMOVA и теста Мантеля становится понятным при рассмотрении положения тувинских родóв и кожуунов в пространстве многомерного шкалирования (рис. 1). Мы видим, что четыре наиболее многочисленных кожууна являются “моноклановыми” кластерами, в их населении превалирует один род. И лишь два кожууна (Тоджинский и Тере-Хольский) в равном соотношении включают представителей трех разных родóв. Роды́ кол и ак являются генетически самыми близкими друг к другу среди всех прочих, что указывает на их схожий генезис. Каждому кожууну соответствуют генетически близкие роды́, которые преимущественно проживают в данном кожууне.
Выделение на графике многомерного шкалирования Западного (включающего Запад и Центр) и Восточного кластеров показывает хорошее согласование данных генетики и антропологии: для западных тувинцев характерен саянский вариант центральноазиатского типа североазиатской расы; для восточных тувинцев, отличающихся большей изолированностью, – катангский вариант байкальского типа североазиатской расы [21].
Совокупность же гуманитарных данных – этнографических, исторических и лингвистических – не позволяет сформировать единое представление о генезисе родоплеменных групп [22]. График многомерного шкалирования (рис. 1) не согласуется ни с одной из наиболее распространенных гипотез этногенеза тувинцев – “самодийской”, “монгольской” и “тюркской” [2]. Поэтому для решения вопросов происхождения тувинских родóв необходимы дальнейшие междисциплинарные исследования, в которых генетики и представители гуманитарных наук должны работать совместно.
Молекулярная генеалогия Y-хромосомы для родоплеменных групп довольно успешно применяется при решении этой задачи, как это было сделано ранее для тувинских родóв – монгуш, ооржак, кол, оюн и хертек [14, 15]. В дальнейших исследованиях тувинских родóв необходимо оценить, насколько в них выражен эффект основателя, и рассчитать генетическую близость между членами рода с использованием STR-маркеров и полных сиквенсов Y-хромосомы, сопоставляя генеалогические, этнографические, исторические и лингвистические данные. Но вся совокупность полученных нами результатов однозначно указывает на то, что дальнейшее изучение генофонда тувинцев необходимо проводить с обязательным учетом их родоплеменной структуры.
Работа выполнена при финансовой поддержке РНФ № 17-1401345; в разделе формирования ДНК-коллекций – в рамках государственного задания Министерства науки и высшего образования России для ИОГен РАН (тема 0112-2019-0001) и для Медико-генетического научного центра.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартами институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Choongwon J., Balanovsky O., Lukianova E. et al. The genetic history of admixture across inner Eurasia // Nat. Ecol. Evol. 2019. https://doi.org/10.1038/s41559-019-0878-2
Сердобов Н.А. История формирования тувинской нации. Кызыл: Тув. кн. изд-во, 1971. 482 с.
Пузырев В.П., Эрдыниева Л.С., Кучер А.Н. Генетико-эпидемиологическое исследование населения Тувы. Томск: STT, 1999. 256 с.
Кучер А.Н., Ондар Э.А., Степанов В.А и др. Тувинцы: гены, демография, здоровье. Томск: Печатная мануфактура, 2003. 232 с.
Степанов В.А. Этногеномика населения Северной Евразии. Томск: Печатная мануфактура, 2002. 244 с.
Голубенко М.В., Еремина Е.Р., Тадинова В.Н. и др. Распространенность европеоидных и монголоидных гаплогрупп митохондриальной ДНК у народов Сибири и Средней Азии // Генофонд населения Сибири / Под ред. Коненкова В.И., Пузырева В.П., Воеводы М.И. Новосибирск: Изд-во Ин-та археологии и этнографии СО РАН, 2003. С. 9–13.
Харьков В.Н., Хамина К.В., Медведева О.Ф. и др. Структура генофонда тувинцев по маркерам Y‑хромосомы // Генетика. 2013. Т. 49. № 12. С. 1418–1420. https://doi.org/10.7868/S0016675813120035
Маннай-оол М.Х. Тувинцы. Происхождение и формирование этноса. Новосибирск: Наука, 2004. С. 99–166.
Жабагин М.К., Сабитов Ж.М., Агджоян А.А. и др. Генезис крупнейшей родоплеменной группы казахов – аргынов – в контексте популяционной генетики // Вестн. Моск. ун-та. Сер. ХХIII. Антропология. 2016. № 4. С. 59–68.
Схаляхо Р.А., Жабагин М.К., Юсупов Ю.М. и др. Генофонд туркмен Каракалпакстана в контексте популяций Центральной Азии (полиморфизм Y-хромосомы) // Вестн. Моск. ун-та. Сер. XXIII. Антропология. 2016. № 3. С. 86–96.
Юсупов Ю.М., Балановская Е.В., Жабагин М.К. и др. Генофонд юго-западных башкир по маркерам Y‑хромосомы: опыт междисциплинарного анализа // Генетика. 2018. Т. 54. ПРИЛОЖЕНИЕ. С. 95–98. https://doi.org/10.1134/S001667518130222
Богунов Ю.В., Жабагин М.К., Богунова А.А. и др. Генофонд коренных народов Дальнего Востока: генетическая реконструкция происхождения нанайских родов (бельды и самар) // Генетика. 2018. Т. 54. ПРИЛОЖЕНИЕ. С. 99–102. https://doi.org/10.1134/S0016675818130052
Derenko M., Malyarchuk B., Denisova G. et al. Contrasting patterns of Y-chromosome variation in South Siberian population from Baikal and Altai Sayan regions // Hum. Genet. 2006. V. 118. P. 591–604. https://doi.org/10.1007/s00439-005-0076-y
Дамба Л.Д., Балановская Е.В., Жабагин М.К. и др. Оценка вклада монгольской экспансии в генофонд тувинцев // Вавиловский журн. генет. и селекции. 2018. Т. 22. № 5. С. 611–619. https://doi.org/10.18699/VJ18.402
Дамба Л.Д., Балановская Е.В., Агджоян А.Т. и др. Генофонд трех восточных родов тувинцев по данным полиморфизма Y-хромосомы // Вестн. Моск. ун-та. Сер. XXIII. Антропология. 2019. № 1. С. 74–85.https://doi.org/10.32521/2074-8132.2019.1.074-085
Балановская Е.В., Жабагин М.К., Агджоян А.Т. и др. Популяционные биобанки: принципы организации и перспективы применения в геногеографии и персонализированной медицине // Генетика. 2016. Т. 52. № 12. С. 1371–1387. https://doi.org/10.7868/S001667581612002X
Ilumäe A.M., Reidla M., Chukhryaeva M. et al. Human Y chromosome haplogroup N: A non-trivial time-resolved phylogeography that cuts across language families // Am. J. Hum. Genet. 2016. V. 99. P. 163–173. https://doi.org/10.1016/j.ajhg.2016.05.025
Nei M. Molecular Population Genetics and Evolution. Amsterdam: North-Holland Publ. Co., 1975. P. 288.
Excoffier L., Lischer H.E. Arlequin suite ver. 3.5: a new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Жабагин М.К., Балановский О.П., Сабитов Ж.М. и др. Реконструкция структуры генофонда казахов по данным об их родорасселении // Вавиловский журн. генет. и селекции. 2018. Т. 22. № 7. С. 895–904. https://doi.org/10.18699/VJ18.431
Аксянова Г.А. Основные результаты расогенетических исследований в Туве в XX столетии // Археология, этнография и антропология Евразии. 2009. Т. 4. № 40. С. 137–144.
Дамба Л.Д., Айыжы Е.В., Монгуш Б.Б. и др. Комплексный подход в изучении родовой структуры тувинцев на примере родов ооржак и монгуш // Вестн. Тув. гос. ун-та. 2018. № 2. С. 37–44.
Дополнительные материалы отсутствуют.


