Генетика, 2021, T. 57, № 1, стр. 44-55
Ингибитор амилаз SbAI видов картофеля: вариабельность структуры и профиля экспрессии
Е. А. Дьяченко 1, *, А. В. Кулакова 1, А. А. Мелешин 2, А. В. Щенникова 1, Е. З. Кочиева 1
1 Институт биоинженерии, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” Российской академии наук
119071 Москва,, Россия
2 Всероссийский научно-исследовательский институт картофельного хозяйства им. А.Г. Лорха
140051 Московская область, Люберецкий район, п. Красково, Россия
* E-mail: dyachenko-el@yandex.ru
Поступила в редакцию 20.02.2020
После доработки 27.04.2020
Принята к публикации 08.07.2020
Аннотация
Деградация крахмала в растениях опосредуется амилазами, активность которых посттрансляционно регулируется ингибиторами. Предполагается, что работа гена ингибитора амилаз SbAI связана с устойчивостью картофеля к холодовому стрессу. Изучение возможных корреляций вариабельности гена SbAI с холодостойкостью видов картофеля позволит повысить степень понимания регуляции метаболизма крахмала в процессе развития растения и его реакции на стресс. В настоящем исследовании идентифицированы полногеномные последовательности гомологов гена SbAI у 12 видов картофеля секции Petota. Выявлено 36 аллельных вариантов гена и 31 вариант белка. Сопоставление с данными по сортам Solanum tuberosum показало, что сортовой полиморфизм в 1.4–1.7 раза превышает межвидовой, однако видам соответствует большее число аллельных вариантов. Филогенетический анализ показал, что аллельные варианты SbAI видов S. demissum, S. acaule и S. stoloniferum разнесены по разным кладам. Профиль экспрессии гена SbAI определен в различных органах видов S. tuberosum (сорт Надежда), S. rybinii, S. chacoense и S. kurtzianum. В листе S. chacoense уровень экспрессии гена в 13, 50 и 250 раз выше, чем у S. tuberosum, S. kurtzianum и S. rybinii соответственно. Различный уровень экспрессии SbAI в листьях видов может быть следствием разного исходного содержания в листьях как синтезируемого крахмала, так и редуцирующих сахаров. В клубне S. tuberosum уровень выше в 5.2, 8.6 и 430 раз, чем у S. kurtzianum, S. chacoense и S. rybinii. В корне S. rybinii уровень SbAI в 2.4 раза выше, чем у S. chacoense, и в 42.3 раза выше, чем у S. kurtzianum и S. tuberosum. Показано отсутствие корреляции уровня экспрессии SbAI с холодостойкостью видов. Полученные данные профиля экспрессии позволяют предположить, что разные виды и сорта картофеля могут по-разному регулировать содержание крахмала в клубнях.
Низкие температуры являются одним из наиболее распространенных факторов, запускающих механизмы адаптации или толерантности растений и определяющих географическое распределение и расхождение видов. В процессе холодовой акклиматизации в растении значительно изменяются транскриптомные данные и включается биосинтез и накопление различных криопротекторов с одновременным преобразованием состава клеточных мембран [1, 2]. Одна из его самых первых значительных метаболических реакций на холод – накопление растворимых сахаров за счет деградации крахмала [3]. Сначала происходит фосфорилирование амилопектиновых полимеров на поверхности крахмальных гранул, что повышает их гидрофильность и доступность для гидролитических ферментов – α-амилаз (AMY, EC 3.2.1.1), β-амилаз (BAM, EC 3.2.1.2) и изоамилаз (EC 3.2.1.68) [3].
Такая важная сельскохозяйственная культура как картофель (Solanum tuberosum) является чувствительным к холоду растением: температура ниже нуля может привести к повреждению надземной биомассы и задержке развития. Между метаболизмом крахмала в гетеротрофных клубнях и фотосинтезирующих листьях картофеля есть глубокие различия. В клубнях крахмал накапливается по мере развития и хранится в течение длительного времени, поддерживая энергетические потребности, а после пробуждения клубня активно разлагается, подпитывая рост побегов [4]. В листьях же крахмал синтезируется днем и разлагается ночью, поддерживая энергоснабжение биологических процессов [4]. В клубнях деградация крахмала опосредуется амилазами: StBAM9 связывается с крахмальным зерном и высвобождает растворимые глюканы, которые гидролизуются в амилопластах с помощью StBAM1 и затем в цитозоле при участии StAmy23 [5–7]. В листьях, в ночной деградации транзитного крахмала активное участие принадлежит β-амилазам и изоамилазам [8]. Несмотря на различия, в регуляции метаболизма крахмала в клубнях и листьях существует много параллелей [4].
Активность амилаз регулируется белковыми ингибиторами на посттрансляционном уровне. Известно, как минимум, шесть классов растительных ингибиторов α-амилаз белковой природы с разной третичной структурой, которые способны связываться с каталитическим центром α-амилазы с помощью водородных связей, блокируя его или изменяя конформацию фермента [9].
Один из классов ингибиторов амилаз – семейство AAI_LTSS (Alpha-Amylase Inhibitors, Lipid Transfer and Seed Storage), играющее важную роль в защите растений от насекомых и патогенов, в транспорте липидов между внутриклеточными мембранами и хранении питательных веществ. Представителем семейства является белок Amylase Inhibitor Solanum berthaultii (SbAI, другое название classical arabinogalatan protein (AGP) 4-like) [6], пространственная структура которого (с характерными восемью остатками цистеина, образующими четыре дисульфидных мостика) консервативна для различных видов растений [10] (https://www.ncbi.nlm.nih.gov/Structure/cdd/cddsrv.cgi?uid=352835). В базе данных NCBI имеется множество гомологов SbAI, однако информация об их функции крайне ограничена. Для двух отдаленных гомологов показана связь с устойчивостью к тле (PERK10, XP_009766757.1 [11]) и с формированием семян (JAGGER, AGP4, NP_196605.1 [12]). Несмотря на низкий уровень гомологии между SbAI и описанными гомологами, становится понятна важность роли SbAI в развитии растения.
Впервые ген SbAI, как следует из его названия, клонирован и охарактеризован у клубнеобразующего вида картофеля S. berthaultii [6]. Интересной особенностью белка SbAI оказалась его способность подавлять активность не только α-амилаз, но и β-амилаз: было подтверждено экспериментально, что SbAI взаимодействует с амилазами StAmy23, StBAM1 и StBAM9 [6]. Кроме того, удалось обнаружить положительную корреляцию уровня экспрессии гена SbAI в клубнях картофеля с устойчивостью клубней к холодовому осахариванию (cold-induced sweetening, CIS) [6]. Интересно, что имеется также обратная корреляция между уровнем экспрессии SbAI и генов амилаз StAmy23, StBAM1 и StBAM9, несмотря на то что взаимодействие ингибитора с амилазами происходит на белковом уровне [6].
Повышение содержания редуцирующих сахаров является сигналом к росту активности SbAI и подавлению активности амилаз. Ответ SbAI на сахара, а также на низкие температуры в значительной степени зависит от генотипа картофеля. Кроме S. berthauiltii, имеется огромное разнообразие видов и сортов картофеля, различающихся по степени холодо- и CIS-устойчивости и особенностям метаболизма крахмала. Однако информации об ортологах SbAI крайне мало.
Поэтому целью данной работы стала оценка вариабельности ингибитора амилаз SbAI у клубнеобразующих видов картофеля (род Solanum, секция Petota) и поиск возможных корреляций полиморфизма и профиля экспрессии гена с характеристиками вида. Для этого были впервые идентифицированы полногеномные последовательности гомологов гена SbAI дикорастущих и примитивных культурных видов картофеля, охарактеризованы их структура, филогения, аллельная вариабельность и аминокислотный полиморфизм кодируемых белков, определены профили экспрессии гена в различных органах анализируемых образцов картофеля.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал. Образцы 12 дикорастущих клубнеобразующих видов картофеля секции Petota и сорта Надежда S. tuberosum (табл. 1) выращивали в 2019 г. при нормальных температурных условиях (23/25°С, 16/8 ч – день/ночь, теплица). Из молодых листьев выделяли геномную ДНК калий-ацетатным методом [13] для идентификации полногеномных последовательностей гомологов гена SbAI. В сентябре 2019 г. были собраны ткани (лист, стебель, бутон, цветок, столон, корень, клубень) для выделения РНК и анализа профиля экспрессии идентифицированных гомологов гена SbAI.
Таблица 1.
Характеристики гомологов SbAI у видов картофеля и томата
| Вид | Каталожный номер/ NCBI ID | Плоидность; W/C*, ** | CR* | Аллели – ген/белок | Ген, пн | кДНК, пн | Белок, а.о. | Замещения/индели, а.о. | |
|---|---|---|---|---|---|---|---|---|---|
| список | количество | ||||||||
| Solanum acaule Bitter | ВИР 9784/ MT074624–MT074626 |
4Х/6X; W (clades 4, 3) | ++ | 1/1 | 1817 | 630 | 209 | S94P; A133T; A136P; E163A; A205V | 5/0 |
| 2/2 | 1868 | 618 | 205 | V15G; P130S; E163V; F192L/T135_P138del | 4/1 | ||||
| 3/3 | 1868 | 618 | 205 | V15G; G67A; E163V; F192L/T135_P138del | 4/1 | ||||
| 31-2012/ MT074627–MT074629 |
4/4 | 1868 | 618 | 205 | V15G; R63I; E163V; F192L/T135_P138del | 4/1 | |||
| 5/5 | 1818 | 630 | 209 | A133T; A136P; E163A; N172S; A205V | 5/0 | ||||
| 6/6 | 1867 | 618 | 205 | M149V; E163V; F192L/T135_P138del | 3/1 | ||||
| Solanum rybinii Juz & Bukasov (=Solanum tuberosum subsp. andigenum) | 9342/ MT074630–MT074632 |
4X; С (4 cultivated) | + | 7/7 | 1763 | 618 | 205 | S78T; E163V/T135_P138del | 2/1 |
| 8/8 | 1763 | 618 | 205 | S78T; E163V; A196V/T135_P138del | 3/1 | ||||
| 9/9 | 1807 | 630 | 209 | A136P; E163V | 2/0 | ||||
| Solanum berthaultii Hawkes | ВИР 24267/ MT074633–MT074636 |
2Х; W (4 south; 4B) | – | 10/10 | 1803 | 627 | 208 | A136P; E163V/S137del | 2/1 |
| 11/11 | 1811 | 630 | 209 | T123R; A136P; E163V | 3/0 | ||||
| 12/12 | 1801 | 627 | 208 | V15G; A136P; E163V/S137del | 3/1 | ||||
| 13/13 | 1802 | 630 | 209 | A136P; E163V; M207I | 3/0 | ||||
| Solanum chacoense Bitter | 3678/ MT074637–MT074638 |
2Х; W (4 south) | + | 14/14 | 1781 | 621 | 206 | A136P; A152T; E163V; P179S; A196V; F197L; M207I/G202_I204del | 7/1 |
| 15/15 | 1781 | 621 | 206 | P86A; A136P; A152T; E163V; P179S; A196V; F197L; M207I/G202_I204del | 8/1 | ||||
| Solanum demissum Lindl. | 15176/34/ MT074639–MT074640 |
6Х; W (4 south, 3) | + | 16/16 | 1822 | 630 | 209 | A133T; A136P; E163A; A205V | 4/0 |
| 17/17 | 1858 | 609 | 202 | V15G; E163V/T135_P138del; G202_I204del | 2/2 | ||||
| Solanum gourlay (=Solanum brevicaule Bitter) | GB 18038/ MT074641–MT074642 |
2Х/4Х; W (4 south) | + | 18/9 | 1796 | 630 | 209 | A136P; E163V | 2/0 |
| 19/18 | 1869 | 630 | 209 | A136P; E163V; P179S | 3/0 | ||||
| Solanum jamesii Torr. | ВИР 15203/ MT074643–MT074644 |
2Х; W (1 + 2) | – | 20/19 | 1808 | 618 | 205 | A17V; T53A; E163V; P179S; T181I/T135_P138del | 5/1 |
| 21/19 | 1813 | 618 | 205 | A17V; T53A; E163V; P179S; T181I/T135_P138del | 5/1 | ||||
| Solanum kurtzianum Bitter & Wittm. | ВИР 11969/ MT074645–MT074646 |
2Х; W (4 south) | + | 22/18 | 1788 | 630 | 209 | A136P; E163V; P179S | 3/0 |
| 23/20 | 1788 | 630 | 209 | T64A; A136P; E163V; P179S; G189S | 5/0 | ||||
| ВИР 12483/ MT074647—MT074648 |
24/21 | 1795 | 630 | 209 | A136P; E163V; P179S; F201V | 4/0 | |||
| 25/18 | 1795 | 630 | 209 | A136P; E163V; P179S | 3/0 | ||||
| Solanum stoloniferum Schltdl. & Bouchet | ВИР 21618/ MT074649–MT074651 |
4Х; W (1 + 2, 3, 4 south) | – | 26/22 | 1857 | 642 | 213 | T53A; E124K; T135S; E163V; P179S/P138_S139insTSAP | 5/1 |
| 27/23 | 1805 | 630 | 209 | A136P; E163V | 2/0 | ||||
| 28/24 | 1858 | 642 | 213 | T53A; T135S; E163V; P179S/P138_S139insTSAP | 4/1 | ||||
| Solanum sucrense Hawkes | ВИР 23598/ MT074652–MT074653 |
4Х; W (4 B) | – | 29/25 | 1797 | 621 | 206 | L35Q; A136P; A152T; E163V; P179S; A196V; F197L; M207I/G202_I204del | 8/1 |
| 30/26 | 1797 | 621 | 206 | A2D; Q84H; A136P; A152T; E163V; P179S; A196V; F197L; M207I/G202_I204del | 9/1 | ||||
| Solanum vernei Bitter & Wittm. | ВИР 20332/ MT074654–MT074656 |
2Х; W (4 south; 4B) | ++ | 31/27 | 1779 | 621 | 206 | A136P; A152T; E163V; P179S; A196V; F197L; M207I / G202_I204del | 7/1 |
| 32/28 | 1781 | 621 | 206 | H4R; S46T; C74W; T92A; A136P; A152T; E163V; P179S; N180Y; M207I/G202_I204del | 10/1 | ||||
| 33/29 | 1777 | 621 | 206 | S46T; G57D; A136P; A152T; E163V; P179S; A196V; F197L; M207I/G202_I204del | 9/1 | ||||
| Solanum verrucosum Schltdl. | ВИР 24467/ MT074657–MT074659 |
2Х; W (4 south; 4B) | + | 34/30 | 1805 | 630 | 209 | A136P; E163V; F201L | 3/0 |
| 35/31 | 1805 | 630 | 209 | T27A; A136P; E163V; A185T; F201L | 5/0 | ||||
| 36/30 | 1805 | 630 | 209 | A136P; E163V; F201L | 3/0 | ||||
| Solanum tuberosum | GeneID: 102591697 NW_006239139.1 (302246..304341)*** | cv. DM 1-3 516 R44 | Нет данных | – | 1848 | 630 | 209 | – | |
| Solanum lycopersicum | Gene ID: 101255064 NC_015440.3 (122217..124105)*** |
cv. Heinz 1706 | Нет данных | – | 1670 | 627 | 208 | T53A; S78T; M93L; T112S; P113V; S132P; A133S; T135S; A136P; A145P; A152T; E163V; F192L; I198S; F201V; A205V; F206V; M207L; L208V/Y209del | 19/1 |
| Solanum pennellii | Gene ID: 107012074 NC_028639.1 (120187..122094)*** |
LA0716 | Нет данных | – | 1706 | 627 | 208 | T53A; S78T; M93L; T112S; P113A; S132P; T135S; A136P; A145P; A152T; E163V; F192L; A205V; F206V; M207L; L208V/Y209del | 16/1 |
Идентификация гомологов гена SbAI. Геномную ДНК растений использовали для амплификации полногеномных последовательностей гомологов гена SbAI с помощью ПЦР с праймерами SbaI_F/SbaI_R (табл. 2; [14]). Фрагменты ожидаемой длины (около 2 тыс. пн) вырезали из геля (Zymoclean™ Gel DNA Recovery Kit, Zymo Research, США), клонировали (Quick-TA Kit; Евроген, РФ) и секвенировали (по 3–5 клонов каждого вида) с дополнительными праймерами (табл. 2; [14]) на автоматическом секвенаторе ABI 310 Сapillary DNA Analyzer (Applied Biosystems, США; ЦКП “Биоинженерия” РАН).
Таблица 2.
Список праймеров, использованных в работе
| Название | Последовательность (5' → 3') | Назначение |
|---|---|---|
| SbAIrtF | TTGTAACATGGCTCGCGTTC | РВ-ПЦР (SbAI) [18] |
| SbAIrtR | TGTTGGTGAAGCACTTGGAG | |
| ef1αF | ATTGGAAACGGATATGCTCCA | РВ-ПЦР (ef1α) [19] |
| ef1αR | TCCTTACCTGAACGCCTGTCA | |
| sec3F | GCTTGCACACGCCATATCAAT | РВ-ПЦР (sec3) [20] |
| sec3R | TGGATTTTACCACCTTCCGCA | |
| SbaI_F | ACTATGGCTTTTCATTACTCTA | Амплификация полногеномной последовательности гомологов SbAI; секвенирование [14] |
| SbaI_R | TTACATCAAAGAATAGTTGTATAAC | |
| SbaI_in1R | TCGTGAGAATAGTCTCTTGC | Секвенирование гомологов SbAI [14] |
| SbaI_ex1F | GTAACATGGCTCGCGTTC | |
| SbaI_ex3F | AACAGAGGCTCCAAGTGC | |
| SbaI_in3R | GGATAGTTTGAGCAACATAACTT |
Структура и филогения гомологов гена SbAI и кодируемых ими белков. Структуру, вариабельность и филогению полученных последовательностей анализировали с помощью пакета программ MEGA 7.0 [15]. Возможное влияние замещений аминокислотных остатков на структуру гомологов SbAI оценивали с использованием программы PROVEAN (http://provean.jcvi.org/index.php) [16]. Пространственную структуру белков-гомологов SbAI предсказывали с помощью Phyre2 [17].
Профили экспрессии гомологов SbAI. Препараты суммарной РНК выделяли (RNeasy Plant Mini Kit; Qiagen, Германия) из листа, стебля, бутона, цветка, столона, корня и клубня образцов четырех видов картофеля (включая сорт Надежда S. tuberosum) и использовали для синтеза кДНК (GoScript; Promega, США). Анализ профиля экспрессии гомологов SbAI проводили методом количественной ПЦР в реальном времени (РВ-ПЦР) в трех технических повторах. Для этого применяли ранее разработанные праймеры SbAIrtF/SbAIrtR (табл. 2; [18]), набор “Реакционная смесь для проведения РВ-ПЦР в присутствии SYBR GreenI и ROX” (ЗАО Синтол, Россия) и следующие условия: 95°C – 5 мин; 40 циклов (95°C – 15 с, 60°C – 50 с). Для нормализации экспрессии использовали референсные гены EF1α и SEC3 и праймеры к ним (табл. 2; [19, 20]). Статистическую обработку полученных данных проводили с помощью программного обеспечения GraphPadPrism v. 7.02 (https://www.graphpad.com).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Идентификация и структурный анализ генов-гомологов SbAI у видов картофеля
Были идентифицированы (амплифицированы, клонированы, секвенированы) полногеномные последовательности гомологов гена SbAI у образцов 12 видов картофеля (табл. 1). Гомологи имели характерную для SbAI четырехэкзонную структуру, однако различались по общей длине (от 1763 пн у S. tuberosum ssp. andigenum до 1869 пн у S. gourlay) и по размеру кДНК (от 609 пн у S. demissum до 642 пн у S. stoloniferum) (табл. 1). По сравнению со SbAI S. tuberosum cv. DM 1–3 516 R44 (Gene ID: 102591697; далее использовался как референс при сравнениях) вариабельность полногеномных последовательностей идентифицированных гомологов составила 19.3% (381 SNPs), а кДНК–12.6% (82 SNPs) (табл. 1).
Кодируемые белки SbAI характеризовались наличием консервативных для AAI_LTSS-семейства доменов LTP_2 (в позиции 16–102 а.о. – у всех) и PRK14971 superfamily (в позиции 77–189 а.о. по SbAI S. tuberosum cv.DM 1-3 516 R44; 77–196 а.о. по выравниванию). Около 13–14% последовательности каждого белка составляли остатки пролина, что подтверждает принадлежность гомологов к AGP-семейству Pro-обогащенных белков. Также это свидетельствует о высокой способности SbAI взаимодействовать с другими белками, как это было показано ранее для Pro-обогащенных мотивов [21]. Также все исследуемые гомологи содержали консенсусные для AAI_LTSS-белков восемь остатков цистеина, формирующих дисульфидные мостики, в позициях 30, 41, 58, 59, 72, 74, 99 и 108 (соответствует домену LTP_2).
Третичные структуры всех анализируемых гомологов SbAI были предсказаны с помощью Phyre2 на основе известных матриц (PDB ID c2rknA_ “x‑ray structure of the self-defense and signaling protein DIR1 from Arabidopsis thaliana” и c2ljoA “3d solution structure of lipid transfer protein lc-ltp2”). С достоверностью >90% были моделированы 38–40% аминокислотных остатков (~30–107 а.о., что соответствует домену LTP_2), остальные – ab initio. Около 60% последовательности, куда полностью входит домен PRK14971, имеет неупорядоченную структуру, вследствие чего достоверные предсказания вторичной структуры данной области отсутствуют. Домен LTP_2 укладывается в пучок из четырех–пяти спиралей, стабилизированных четырьмя дисульфидными мостиками. Сравнение анализируемых белков с известной структурой мембранных белков (PDBID c6ithA_, “membrane protein, syndecan-2”) позволило предсказать наличие на C-конце всех гомологов SbAI трансмембранной спирали (~189–207 а.о.), важной для передачи сигнала через клеточную мембрану. Показанное структурное соответствие анализируемых гомологов SbAI указывает на высокую вероятность сходства их функций у разных видов картофеля.
Гомологи SbAI 12 анализируемых видов картофеля содержали 41 замещение аминокислотных остатков (а.о.; 19.25% от выравненной длины 213 а.о.), одну нейтральную вставку (P138_S139insTSAP) и три нейтральные делеции (T135_P138del, S137del и G202_I204del) (табл. 1). Радикальных замещений а.о. было найдено всего семь, и все они (а также вставка и первые две делеции) локализовались в консервативных доменах. Больше всего замещений (включая радикальные) содержали гомологи SbAI образцов видов S. acaule, S. sucrense, S. vernei и S. chacoense. По одному радикальному замещению имели также некоторые варианты SbAI S. berthaultii и S. demissum. Интересно, что гомологи SbAI видов томата имели в 2–10 раз больше замещений а.о. в сравнении с S. tuberosum, однако все они носили нейтральный характер (табл. 1).
Всего было выявлено 36 аллельных вариантов гена и 31 вариант белка (табл. 1). Наибольшее число аллельных вариантов соответствовало видам S. acaule, S. berthaultii и S. kurtzianum. Какая-либо корреляция количества вариантов с плоидностью анализируемых видов отсутствовала. Так, у гексаплоида S. demissum вариантов оказалось меньше, чем у диплоида S. berthaultii (табл. 1).
Мы сравнили полученные результаты с данными по ранее идентифицированным и охарактеризованным последовательностям гомологов гена SbAI у отечественных и зарубежных сортов и линий картофеля S. tuberosum [14]. Оказалось, что общий и экзонный полиморфизм сортовых последовательностей (27.0 и 21.3% соответственно) [14] в 1.4–1.7 раза превышает выявленную в настоящем исследовании межвидовую вариабельность. Однако для видов было идентифицировано большее число (~1.5 раза) аллельных вариантов, чем для сортов (36 сортам соответствовали 70 аллельных вариантов гена и 69 – белка [14]).
Дикорастущий картофель достаточно давно используется в селекционных программах, и в родословных большинства сортов можно найти, как минимум, один из шести видов (S. demissum, S. acaule, S. chacoense, S. stoloniferum, S. vernei и S. spegazzinii), которые являются донорами признаков устойчивости к различным биотическим и абиотическим стрессовым факторам; прочие виды картофеля (S. verrucosum и другие) в сортах встречаются реже [22]. Большая вариабельность SbAI у сортов, чем у видов, может быть связана с тем, что родословная сорта включает больше одного, а часто и несколько видов картофеля.
Филогения полногеномных последовательностей гомологов SbAI видов картофеля
Чтобы оценить степень родства видов и сортов картофеля, был проведен филогенетический анализ полногеномных последовательностей соответствующих гомологов SbAI; в качестве внешней группы использовали SbAI видов томата S. lycopersicum и S. pennellii (рис. 1).
Рис. 1.
Филогенетическая дендрограмма видов и сортов картофеля, основанная на полногеномной последовательности гомологов гена SbAI. Построена в MEGA 7.0, с использованием метода максимального правдоподобия (Maximum Likelihood), модели Tamura 3 и 1000 бутстрэп-реплик. Гомологи SbAI Solanum lycopersicum и Solanum pennellii использованы в качестве внешней группы. Использованы аллельные варианты 10 сортов S. tuberosum (Голубизна, Крепыш, Браво, Фиолетовый, Ирбитский, Фаворит, Старт, Горняк, Арлекин и Кузнечанка; на дендрограмме названия даны в латинской транслитерации) и линия 3-43-6. “А” после названия вида/сорта обозначает аллельный вариант. Значения бутстрэп указаны в узлах.
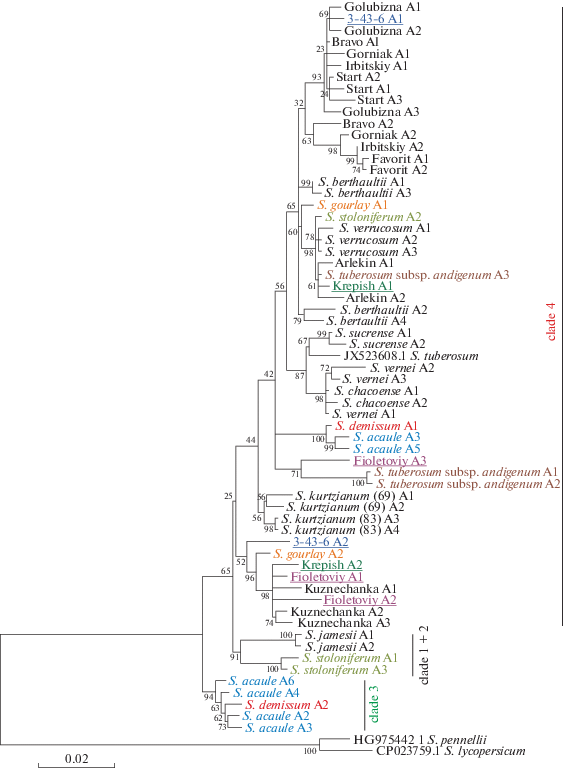
На дендрограмме произошло разделение сортов и видов картофеля на несколько клад. Выделилась группа из четырех аллельных вариантов S. acaule и одного S. demissum, тогда как оставшиеся аллельные варианты данных видов объединились в другую группу внутри соседнего большого кластера (рис. 1). Кроме этой группы кластер содержит клады, состоящие: 1) из трех подкластеров, объединяющих аллели нескольких сортов/линий (Голубизна, Браво, Горняк, Старт, Ирбитский, Фаворит, Арлекин, Крепыш и 3-43-6) и видов (S. berthaultii, S. gourlay, S. tuberosum subsp. andigenum, S. stoloniferum и S. verrucosum); 2) из аллелей одних только видов (S. sucrense, S. vernei, S. tuberosum и S. chacoense); 3) из четырех аллелей S. kurtzianum; 4) из двух аллелей subsp. andigenum и аллеля сорта Фиолетовый; 5) из аллелей трех видов (S. gourlay, S. jamesii и S. stoloniferum) и сортов/линий (3-43-6, Крепыш, Фиолетовый и Кузнечанка) (рис. 1).
Таким образом, выявленные аллельные варианты пяти видов (S. acaule, S. demissum, subsp. andigenum, S. gourlay и S. stoloniferum), а также аллели одной линии (3-43-6) и двух сортов (Фиолетовый и Крепыш) оказались разнесены по разным кладам. Варианты SbAI семи из 11 взятых в анализ сортов составляют единый подкластер, тогда как остальные сорта попадают в кластеры, образованные аллельными вариантами видов.
Ранее было показано, что филогенетически виды картофеля делятся на три клады (“clade 1 + 2”, “clade 3” и “clade 4”), где “clade 4” делится на подклады “4 south”, “4 north” и “4 cultivated”, или же на “4А” и “4В” [23–25]. При этом аллельные варианты одного вида могут попадать в разные клады [23]. Обнаруженный в нашем исследовании разброс аллельных вариантов S. acaule и S. demissum по разным кладам подтверждает показанный ранее сложный состав аллелей этих двух видов. Считается, что аллели S. acaule и S. demissum в основном принадлежат кладе 4, однако оба вида имеют хотя бы по одному аллелю клады 3 [23]. В нашем же случае два аллельных варианта S. demissum распределились поровну между кладами 3 и 4, а вот из шести аллелей S. acaule четыре попали в кладу 3 и только два в кладу 4 (рис. 1). У еще одного вида со сложным составом аллелей – S. stoloniferum один из трех аллелей SbAI оказался в кладе 4, а два других объединились в одну группу с аллелями S. jamesii (рис. 1), который принадлежит кладе 1 + 2. Это подтверждает выявленное ранее присутствие у S. stoloniferum аллелей клады 1 + 2 [23]. Показанное распределение аллельных вариантов линии 3-43-6 и сортов Фиолетовый и Крепыш по отдаленным кластерам дендрограммы может отражать комплексную родословную образцов, связанную со сложными межвидовыми скрещиваниями.
Вариабельность профиля экспрессии гомологов гена SbAI видов картофеля
Для сравнительного анализа экспрессии гена SbAI были выбраны образцы клубнеобразующих видов секции Petota – S. tuberosum (сорт Надежда в качестве контроля), S. rybinii (=S. tuberosum subsp. andigenum, клада 4В) и двух дикорастущих видов S. chacoense и S. kurtzianum (клада 4 south) [22].
Предварительно профиль экспрессии гена SbAI был определен в ряде органов S. tuberosum. В не запасающих вегетативных органах (стебле, листе, бутоне, цветке и корне) наблюдался низкий уровень транскрипции гена, тогда как в клубне транскрипция резко усиливалась (рис. 2). Значительный рост экспрессии гена в запасающих органах в сравнении с другими вегетативными тканями свидетельствует об участии SbAI в предохранении резервного крахмала клубня от гидролиза.
Рис. 2.
Профиль экспрессии гена ингибитора амилаз SbAI в различных органах (стебель – St, лист – L, бутон – B, цветок – F, корень – R, столон – S, клубень – T) образцов четырех видов картофеля: Solanum tuberosum (сорт Надежда), Solanum kurtzianum, Solanum chacoense, Solanum rybinii. Буквами над столбиками указаны достоверные различия (P < < 0.01) в уровне экспрессии SbAI между отдельными органами внутри одного вида. Каждому органу присвоено свое буквенное обозначение (a–g). Например, для S. tuberosum экспрессия в цветке (e) достоверно отличается от экспреcсии в листе (a), корне (b), клубне (d) и стебле (g), соответственно над столбиком abdg.
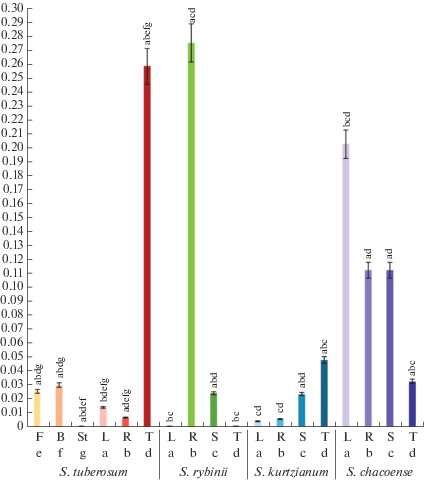
С учетом полученных результатов экспрессию SbAI у трех выбранных видов S. rybinii, S. chacoense и S. kurtzianum определяли в листе (фотосинтезирующий орган), корне и столоне (подземные не запасающие органы), а также в клубне (подземный запасающий орган). Транскрипты гена были выявлены во всех анализируемых органах всех образцов с относительным уровнем транскрипции в интервале 0.00025–0.3 (рис. 2), что соответствовало ранее определенному профилю экспрессии SbAI у двух сортов картофеля [5].
Транскрипция SbAI S. kurtzianum повторяла профиль SbAI S. tuberosum, за исключением менее выраженного роста уровня транскрипции SbAI в клубнях, в то время как S. rybinii и S. chacoense имели другие профили экспрессии гена (рис. 2).
Согласно суточной динамике метаболизма крахмала в листьях, днем (когда собирали листья образцов на анализ) должен происходить интенсивный синтез и накопление крахмала [26], и в данной ткани ожидался повышенный уровень экспрессии SbAI. Однако в листьях анализируемых образцов транскрипция носила следовый характер, за исключением S. chacoense, где уровень экспрессии гена был выше в 13, 50 и 250 раз, чем у S. tuberosum, S. kurtzianum и S. rybinii соответственно (рис. 2). Известно, что виды S. rybinii, S. chacoense, S. kurtzianum, а также сорт Надежда (взятый как представитель S. tuberosum) обладают хорошей устойчивостью к низким температурам [22]. Поэтому корреляции уровня экспрессии SbAI в листе с холодостойкостью образцов не отмечено.
В этой связи интересны данные о разной реакции двух сортов картофеля на холодовой стресс: в листьях Desiree экспрессия генов деградации крахмала росла с одновременным накоплением редуцирующих сахаров, в то время как у RussetBurbank данные характеристики почти не менялись [3]. Это позволило заключить, что активация гидролиза крахмала зависит не только от понижения температуры, но и от содержания редуцирующих сахаров при нормальных условиях: в листьях Russet Burbank моносахаридов исходно было достаточно, чтобы, не повышая их концентрации, перенести охлаждение [3]. Более того, на примере пяти видов картофеля была показана корреляция между исходным количеством крахмала и степенью роста экспрессии SbAI в листе в ответ на холодовой стресс [18].
Поэтому различный уровень экспрессии гена ингибитора амилаз SbAI в листьях анализируемых видов может быть следствием разного исходного содержания в листьях как синтезируемого крахмала, так и редуцирующих сахаров.
Интересно, что при нормальных условиях уровень экспрессии SbAI в листьях никак не коррелирует со степенью устойчивости клубней к холодовому осахариванию. Так, ранее было показано, что в листьях чувствительного и устойчивого к CIS сортов картофеля ген ингибитора амилаз экспрессируется на одинаковом уровне [5]. Однако в клубнях SbAI транскрибируется высоко у CIS-чувствительного сорта и низко у CIS-устойчивого [5].
В настоящей работе самая высокая экспрессия SbAI в клубне была у S. tuberosum – выше в 5.2, 8.6 и 430 раз, чем у S. kurtzianum, S. chacoense и S. rybinii соответственно (рис. 2). Это может свидетельствовать о меньшей подверженности клубней образцов S. tuberosum и S. kurtzianum холодовому осахариванию.
В случае корня и столона ситуация различалась. Так, S. rybinii с минимальной экспрессией SbAI в клубне показал самый высокий уровень транскрипции в корне – в 2.4 раза выше, чем у S. chacoense, и в целых 42.3 раза выше, чем у S. kurtzianum и S. tuberosum (рис. 2).
Полученные данные профиля экспрессии “корень–столон–клубень” позволяют предположить, что разные виды и сорта картофеля могут по-разному регулировать содержание крахмала в клубнях: одни предупреждают его распад, ингибируя активность амилаз уже в корнях и столонах; другие – останавливают деградацию крахмала непосредственно в запасающих органах.
Представленные результаты свидетельствуют о том, что говорить о корреляции непосредственно между уровнем экспрессии гена ингибитора амилаз и холодоустойчивостью или CIS-устойчивостью растения картофеля пока преждевременно. Для корректных заключений необходимо учитывать исходные (предстрессовые) транскриптомные, протеомные и биохимические данные каждого конкретного генотипа в нормальных условиях развития.
Работа выполнена при поддержке РФФИ (грант 17-29-08017) и ФНТП развития сельского хозяйства РФ на 2017–2025 гг. (подпрограмма “Развитие селекции и семеноводства картофеля в Российской Федерации”). Растения выращивали с использованием экспериментальной установки искусственного климата ЭУИК (Институт биоинженерии ФИЦ Биотехнологии РАН).
Список литературы
Dametto A., Sperotto R.A., Adamski J.M. et al. Cold tolerance in rice germinating seeds revealed by deep RNAseq analysis of contrasting indica genotypes // Plant Sci. 2015. V. 238. P. 1–12. https://doi.org/10.1016/j.plantsci.2015.05.009
Leuendorf J.E., Frank M., Schmülling T. Acclimation, priming and memory in the response of Arabidopsis thaliana seedlings to cold stress // Sci. Rep. 2020. V. 10. № 1. Article 689. https://doi.org/10.1038/s41598-019-56797-x
Sitnicka D., Orzechowski S. Cold-induced starch degradation in potato leaves – intercultivar differences in the gene expression and activity of key enzymes // Biol. Plantarum. 2014. V. 58. № 4. P. 659–666. https://doi.org/10.1007/s10535-014-0453-2
Van Harsselaar J.K., Lorenz J., Senning M. et al. Genome-wide analysis of starch metabolism genes in potato (Solanum tuberosum L.) // BMC Genomics. 2017. V. 18. № 1. Article 37. https://doi.org/10.1186/s12864-016-3381-z
Zhang H., Hou J., Liu J. et al. Amylase analysis in potato starch degradation during cold storage and sprouting // Potato Res. 2014. V. 57. P. 47–58. https://doi.org/10.1016/j.foodchem.2019.124991
Zhang H., Liu J., Hou J. et al. The potato amylase inhibitor gene SbAI regulates cold-induced sweetening in potato tubers by modulating amylase activity // Plant Biotech. J. 2014. V. 12. № 7. P. 984–993. https://doi.org/10.1111/pbi.12221
Hou J., Zhang H., Liu J. et al. Amylases StAmy23, StBAM1 and StBAM9 regulate cold-induced sweetening of potato tubers in distinct ways // J. Exp. Bot. 2017. V. 68. № 9. P. 2317–2331. https://doi.org/10.1093/jxb/erx076
Møller M.S., Svensson B. Structural biology of starch-degrading enzymes and their regulation // Curr. Opin. Struct. Biol. 2016. V. 40. P. 33–42. https://doi.org/10.1016/j.sbi.2016.07.006
Wang Z., Chen M., Zhang Y. et al. A cupin domain is involved in α-amylase inhibitory activity // Plant Sci. 2018. V. 277. P. 285–295. https://doi.org/10.1016/j.plantsci.2018.10.001
Lay F., Anderson M. Defensins-components of the innate immune system in plants // Curr. Protein Pept. Sci. 2005. V. 6. P. 85–101. https://doi.org/10.2174/1389203053027575
Liang D., Chen M., Qi X. et al. QTL mapping by SLAF-seq and expression analysis of candidate genes for aphid resistance in cucumber // Front. Plant Sci. 2016. V. 7. Article 1000. https://doi.org/10.3389/fpls.2016.01000
Pereira A.M., Nobre M.S., Pinto S.C. et al. “Love is strong, and you’re so sweet”: JAGGER is essential for persistent synergiddegeneration and polytubeyblock in Arabidopsis thaliana // Mol. Plant. 2016. V. 9. № 4. P. 601–614. https://doi.org/10.1016/j.molp.2016.01.002
Дьяченко Е.А., Слугина М.А. Внутривидовой полиморфизм гена сахарозосинтазы Sus1 у образцов Pisum sativum L. // Вавиловский журн. генетики и селекции. 2018. Т. 22. № 1. С. 108–114. https://doi.org/10.18699/VJ18.338
Дьяченко Е.А., Кулакова А.В., Мелешин А.А., Кочиева Е.З. Аллельная вариабельность гена ингибитора амилаз AI у сортов и линий картофеля // С.-х. биология. 2019. Т. 54. № 5. С. 970–977. https://doi.org/10.15389/agrobiology.2019.5.970rus
Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Choi Y., Sims G.E., Murphy S. et al. Predicting the functional effect of amino acid substitutions and indels // PLoS One. 2012. V. 7. № 10. Article e46688. https://doi.org/10.1371/journal.pone.0046688
Kelley L.A., Mezulis S., Yates C.M. et al. The Phyre2 web portal for protein modeling, prediction and analysis // Nat. Prot. 2015. V. 10. P. 845–858.https://doi.org/10.1038/nprot.2015.053
Слугина М.А., Филюшин М.А., Мелешин А.А. и др. Различия в экспрессии гена ингибитора амилаз SbAI при длительном низкотемпературном хранении клубней и кратковременном холодовом стрессе у картофеля // Генетика. 2020. Т. 56. № 3. С. 361–365. https://doi.org/10.1134/S1022795420030163
Lopez-Pardo R., de Galarreta J.I.R., Ritter E. Selection of housekeeping genes for qRT-PCR analysis in potato tubers under cold stress // Mol. Breed. 2013. V. 31. № 1. P. 39–45. https://doi.org/10.1007/s11032-012-9766-z
Tang X., Zhang N., Si H., Calderón-Urrea A. Selection and validation of reference genes for RT-qPCR analysis in potato under abiotic stress // Plant Methods. 2017. V. 13. Article 85. https://doi.org/10.1186/s13007-017-0238-7
Williamson M.P. The structure and function of proline-rich regions in proteins // Biochem. J. 1994. V. 297. P. 249–260. https://doi.org/10.1042/bj2970249
Machida-Hirano R. Diversity of potato genetic resources // Breed Sci. 2015. V. 65. № 1. P. 26–40. https://doi.org/10.1270/jsbbs.65.26
Cai D., Rodríguez F., Teng Y. et al. Single copy nuclear gene analysis of polyploidy in wild potatoes (Solanum section Petota) // BMC Evol. Biol. 2012. V. 12. Article 70. https://doi.org/10.1186/1471-2148-12-70
Huang B., Ruess H., Liang Q. et al. Analyses of 202 plastid genomes elucidate the phylogeny of Solanum section Petota // Sci. Rep. 2019. V. 9. № 1. Article 4454. https://doi.org/10.1038/s41598-019-40790-5
Rodríguez F., Ghislain M., Clausen A.M. et al. Hybrid origins of cultivated potatoes // Theor. Appl. Genet. 2010. V. 121. № 6. P. 1187–1198. https://doi.org/10.1007/s00122-010-1422-6
Zeeman S.C., Smith M.C., Smith A.M. The diurnal metabolism of leaf starch // Biochem. J. 2007. V. 401. P. 13–28. https://doi.org/10.1042/BJ20061393
Дополнительные материалы отсутствуют.


