Генетика, 2021, T. 57, № 11, стр. 1246-1251
Изменчивость митохондриальной ДНК указывает на американское происхождение современных пихт
В. Л. Семериков 1, *, С. А. Семерикова 1, Ю. А. Путинцева 2, **
1 Институт экологии растений и животных, Уральское отделение
Российской академии наук
620144 Екатеринбург, Россия
2 Cибирский федеральный университет
660041 Красноярск, Россия
* E-mail: semerikov@ipae.uran.ru
** E-mail: yputintseva@sfu-kras.ru
Поступила в редакцию 20.12.2020
После доработки 05.02.2021
Принята к публикации 09.02.2021
Аннотация
Приводятся результаты филогенетического анализа 15 видов пихт, представляющих все основные эволюционные линии рода Abies, и Keteleeria davidiana, использованной в качестве внешней группы. Данные включают нуклеотидные последовательности митохондриальной ДНК длиной около 28000 пн, полученные путем частичного ресеквенирования сборки митохондриального генома пихты сибирской A. sibirica. Установлено базальное положение линий мтДНК некоторых американских пихт, что подтверждает американское происхождение современных Abies. Митотипы большинства евроазиатских видов образуют кладу, дочернюю по отношению к американским пихтам, указывая на ее происхождение в результате одной миграции из Америки в Евразию. В то же время ранее полученные данные по ядерной и хлоропластной ДНК свидетельствуют о неоднократных миграциях пихт из Америки в Евразию. Этот конфликт митохондриальных и ядерных данных можно объяснить, предполагая гибридный захват митохондриальной ДНК аборигенных евроазиатских видов видами-мигрантами.
Миграции между Северной Америкой и Евразией оказали решающий вклад в формирование разнообразия многих таксономических групп умеренной и бореальной флор северного полушария (например [1, 2]). В ходе трансконтинентальных миграций переселенцы могут вытеснять родственные аборигенные виды, а при отсутствии репродуктивных барьеров – формировать зоны интрогрессивной гибридизации. В последнем случае распространяемый с пыльцой генетический поток со стороны более адаптивного вида “разбавляет” гены менее адаптивного вида, которым обычно является аборигенный вид, вплоть до их полной замены. В то же время менее подвижные гены, передающиеся по материнской линии и не распространяющиеся с пыльцой (например хлоропластные и митохондриальные у цветковых растений), наследуются гибридом от аборигенного вида, что проявляется в виде “гибридного захвата” цитоплазматических геномов. Древние события “гибридного захвата” проявляются в конфликте между филогенетическими деревьями, основанными на маркерах с разным типом наследования. В частности, такие конфликты характерны для родов сем. Pinaceae (сосны, ели) [3–7], у которых маркеры ядерного, хлоропластного (хлДНК) и митохондриального (мтДНК) геномов имеют двуродительское, отцовское и материнское наследование, соответственно [8]. В современную эпоху локальные процессы “гибридного захвата”, хотя и не связанные с трансконтинентальными миграциями, так же широко распространены [9, 10].
Согласно последнему биогеографическому исследованию рода Abies [7], наиболее базальными и удаленными от остальных видов по ядерной и хлоропластной ДНК являются некоторые американские пихты, что указывает на американское происхождение современных Abies. В то же время евроазиатские виды входят в несколько клад, часть которых содержат также и американские виды, поддерживая предположение о множественных миграциях из Америки в Евразию. Однако дерево мтДНК [7] демонстрирует разделение гаплотипов на два кластера – кластер американских видов вместе с несколькими островными азиатскими видами и кластер большинства евроазиатских видов, что, в свою очередь, соответствует однократной миграции мтДНК. Одновременный анализ ядерных, хлоропластных и митохондриальных данных позволил обосновать гипотезу о нескольких волнах миграции пихт из Северной Америки в Евразию [7], которые сопровождались гибридным замещением ядерного и хлоропластного геномов аборигенных форм – продуктов предыдущих волн миграции, геномами новых мигрантов, что возможно ввиду относительной легкости межвидовых скрещиваний у пихт. При этом, благодаря гибридному захвату виды-мигранты приобретали митохондриальные геномы видов-аборигенов. Однако разрешение полученного мтДНК дерева [7] было невысоким. Не было установлено положение внешней группы и, как следствие, не были выявлены наиболее дивергентные клады и не были определены филогенетические отношения евроазиатской и американской групп: являются ли они сестринскими, или одна из групп является частью другой. Данное филогенетическое исследование направлено на разрешение этих неопределенностей. Для этого была использована выборка видов, относящихся ко всем основным группам, выявляемым согласно ядерному дереву видов Abies [7]. Нуклеотидные последовательности включали 27 регионов митохондриального генома.
МАТЕРИАЛЫ И МЕТОДЫ
В филогенетическое исследование были включены 15 видов Abies, представляющих все основные клады дерева видов Abies, выделенные ранее на основании изменчивости ядерной и хлоропластной ДНК [7], и все основные секции рода [11], а также Keteleeria davidiana в качестве внешней группы (табл. 1).
Таблица 1.
Использованный материал
| Таксон | Характеристика образца | Естественное распространение | Секция |
|---|---|---|---|
| Abies sibirica Ledeb. | 51°48′/87°15′ E Is24-2007 |
Сибирь, Восточная Европа | Balsamea |
| A. sachalinensis (F. Schmidt) Mast. |
46°40′/141°50′ E Is41-2008 |
Хоккайдо, Сахалин и Курилы | Balsamea |
| A. nephrolepis (Trautv. ex Maxim.) Maxim. | 49°00′/131°05′ E Is01-2008 |
Северо-Восточная Азия | Balsamea |
| A. alba Mill. | 47°48′/13°02′ E Is6-2010 |
Европа | Abies |
| A. balsamea (L.) Mill. | MBG, voucher А0718 | Восток Сев. Америки | Balsamea |
| A. recurvata Mast. | Kew, voucher 36850 | Юго-Зап. Китай | Momi |
| A. mariesii Mast. | 35°46′/137°48′ Is5n-2015 |
Япония | Amabilis |
| A. firma Siebold & Zucc. | 36°13′/140°06′ E Is7n-2015 | Япония | Momi |
| A. fargesii Franch. | M, v. K09-13-062Is28-2014 | Центральный Китай | Pseudopicea |
| A. amabilis Douglas ex J. Forbes | M, Is19-2014 | Запад Сев. Америки | Amabilis |
| A. bracteata (D. Don) A. Poit | NBG, Is12-2011 | Запад Сев. Америки | Bracteata |
| A. koreana E.H. Wilson | MBG, voucher А048Is48-2013 | Корея | Balsamea |
| A. pindrow (Royle ex D. Don) Royle | 34°03′/74°24′ E Is31-2013 |
Зап. Гималаи | Momi |
| A. concolor (Gordon) Lindl. ex Hildebr. | 37°45′/119°30′ W Is13-2012 | Запад Сев. Америки, Центральная Америка | Grandis |
| A. magnifica A. Murray | 37°45′/119°30′ W Is16-2012 | Запад Сев. Америки | Nobilis |
| Keteleeria davidiana v. formosana(Hayata) Hayata | Kew, voucher 40615 | Китай |
Примечание. Характеристика образца: указан источник согласно [7] и Приложения, табл. S1 , координаты природных популяций (с.ш./долгота), MBG – Московский ботанический сад, M – ботанический сад г. Мустила, Финляндия, Kew – ботанический сад Кью, NBG – Никитский ботанический сад. Секции указаны согласно [11].
Для исследования филогении мтДНК рода Abies было использовано ресеквенирование сборки митохондриального генома пихты сибирской A. sibirica, полученной на основе полногеномного NGS-секвенирования [12]. Для этого в контигах данной сборки были случайно выбраны 42 фрагмента. Для их амплификации и секвенированияс помощью программы Primer3 [13] были разработаны 67 пар ПЦР-праймеров. Фрагменты имели длину 700–1500 пн. Условия ПЦР и секвенирования соответствовали описанным в работе [12]. Для дальнейшего использования были отобраны 27 пар праймеров, дававших амплификацию не менее чем у 13 видов Abies из 15.
Редактирование сиквенсных данных и выравнивание последовательностей осуществлялось с помощью программ CodonCodeAligner v. 3.7.1 (CodonCodeCorporation) и BioEdit v. 7.2.5 [14]. Для филогенетической реконструкции нуклеотидные последовательности отдельных фрагментов мтДНК были конкатенированы в объединенный алайнмент. Анализ проводился с использованием байесовского метода (BI) и метода максимальной экономии (MP). Все признаки были равновзвешены. Инделы и инверсии рассматривались как отдельные события и были закодированы как (1/0). BI выполняли с использованием MrBAYES v. 3.1.2 [15] с использованием модели нуклеотидных замен GTR + G + I. Были сгенерированы две цепи MCMC по 10 млн поколений каждая и с сохранением текущих данных каждые 1000 поколений. Для контроля сходимости цепей учитывали их стандартное отклонение. Когда последнее упало ниже 0.01, уже сгенерированные 10 млн деревьев были отброшены как “burn-in”, а последующие 10 млн деревьев были использованы для построения консенсусного дерева по принципу “majority rule”. MP-анализ был выполнен с помощью PAUP * v. 4.0b10 [16] с использованием эвристического поиска, случайного порядка выбора с алгоритмом TBR, опцией COLLAPSE, MaxTrees = 500 и опцией MulTrees. Поддержка ветвей была оценена путем “bootstap” с использованием 1000 повторов.
РЕЗУЛЬТАТЫ
В результате секвенирования 27 фрагментов мтДНК были получены выровненные последовательности (алайнмент) общей длиной 29 968 пн (Приложение, табл. S1 ). Без делеций и отсутствующих данных длина алайнмента у Abies составляла 14 015 пн, из которых изменчивыми являлись 153 признака. Нуклеотидное разнообразие π = 0.002.
Байесовское филогенетическое дерево имело существенно лучшее разрешение, чем дерево, построенное на основе метода максимальной парсимонии, при этом они не противоречили друг другу (рис. 1). Евроазиатские виды A. sibirica, A. nephrolepis, A. koreana, A. fargesii, A. recurvata, A. firma, A. alba и A. pindrow образовали поддержанную кладу (апостериорная вероятность 1). В нее не вошли митотипы восточноазиатских видов недавнего американского происхождения A. sachalinensis и A. mariesii [7]. Ближайшими родственниками пихты сибирской по мтДНК являются A. fargesii (Китай) и A. koreana (Корея). Среди евроазиатских пихт гималайский вид A. pindrow оказался генетически более обособленным, чем европейско-средиземноморский вид A. alba.
Рис. 1.
Филогенетическое дерево 15 видов пихт, построенное на основе нуклеотидных последовательностей мтДНК. Статистическая поддержка полученных клад (BI апостериорная вероятность/MP бутстрэп) приведена в их узлах. Группы выделены на основе ядерной ДНК [7], их цветовое обозначение указано в соответствии с работой [7]. Названия таксонов из Северной Америки затенены.
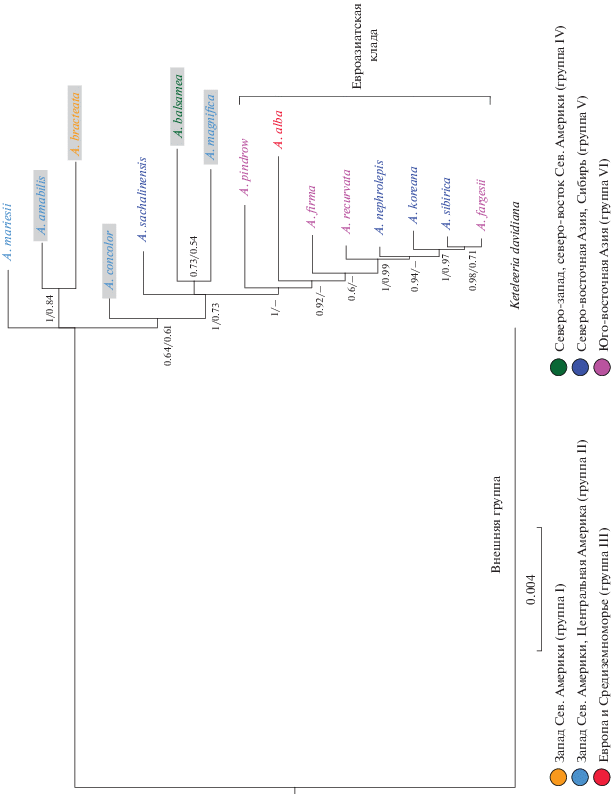
Митотипы американских видов не образовали монофилетичную кладу и разделились на несколько дивергентных линий. Клада евроазиатских видов находится внутри клады нескольких американских видов плюс A. sachalinensis (1/0.73 апостериорная вероятность/поддержка бутстрэп). A. bracteata и A. amabilis (западно-североамериканские виды) образовали кладу с поддержкой 1/0.84. Также весьма дивергентным оказался митотип азиатской пихты A. mariesii (о. Хонсю), который занял неопределенное филогенетическое положение в основании дерева.
ОБСУЖДЕНИЕ
Мы получили на настоящий момент наиболее поддержанное филогенетическое дерево мтДНК Abies. Дерево в основном не противоречит построенному ранее на основе трех фрагментов мтДНК [7]. Евроазиатские виды пихт разделились сходным образом: митотипы большинства из них формируют евроазиатскую кладу (рис. 1), при этом гаплотипы части островных восточноазиатских видов (в настоящем исследовании это A. sachalinensis и A. mariesii) не входят в данную кладу и более близки американским гаплотипам, что соответствует их положению в работе [7]. Поддержка евроазиатской клады (рис. 1), согласно полученным данным, оказалась существенно выше (100), чем в предыдущем исследовании (57/54) [7]. Впервые показано, что митотипы большинства евроазиатских видов образуют кладу, дочернюю по отношению к американским пихтам. Это поддерживает гипотезу об американском происхождении современных евроазиатских пихт и о происхождении мтДНК основной части евроазиатских пихт от одной, первоначальной, миграции из Америки в Евразию [7]. Вероятно, последующие волны миграции сопровождались вытеснением видов предыдущих миграции, но при этом митохондриальные геномы первой волны наследовались видами более поздних волн. Филогенетические деревья мтДНК в обоих исследованиях существенно противоречили деревьям ядерной и хлДНК, в том числе в пределах Евразии. Например, митотипы родственных по ядерной ДНК A. sibirica, A. koreana и A. nephrolepis образуют кладу с митотипами A. fargesii и A. recurvata, хотя по ядерной ДНК они входят в две обособленные ветви – северо-восточноазиатскую и юго-восточноазиатскую. Несмотря на то, что по ядерной ДНК гималайский вид A. pindrow и является более близким родственником восточноазиатских видов, чем средиземноморский вид A. alba, но по мтДНК – наоборот, A. alba более близка к восточноазиатским видам. Такие противоречия могут быть результатом нескольких причин, включая неполный сортинг линий, гибридный захват, рекомбинацию мтДНК и взаимодействие этих факторов.
Несмотря на значительный объем данных секвенирования, многие узлы дерева оказались слабо поддержаны. Очевидно это может быть связано с низкой скоростью мутирования и, соответственно, с низкой изменчивостью мтДНК. Кроме того, филогенетический сигнал может значительно деградировать ввиду рекомбинации мтДНК. Благодаря этому при гибридном контакте филогенетически удаленных таксонов, возможно формирование химерных митотипов, как, например, у дальневосточных видов пихт [7]. В частности это может быть причиной значительного отклонения положения A. mariesii на дереве мтДНК от положения, реконструированного ранее, при использовании меньшего объема сиквенсных данных, когда митотип A. mariesii оказался родственным митотипам A. balsamea и A. lasiocarpa [7]. Мы ожидаем, что включение в анализ большей части видов Abies с использованием нескольких образцов на вид, увеличение количества и длины проанализированных фрагментов мтДНК заметно увеличат информативность филогенетического анализа для реконструкции истории пихт. Решение проблемы низкой изменчивости мтДНК видится в использовании NGS-секвенирования, которое позволяет получить значительно больший объем данных. Благодаря существенно большему числу копий на клетку мтДНК, по сравнению с ядерной ДНК, митохондриальные геномы могут быть собраны из прочтений (ридов) при относительно низкой глубине полногеномного секвенирования. Также может быть использовано обогащение геномных библиотек митохондриальной фракцией с помощью гибридизации с митохондриальными зондами “байтами” [17].
Работа выполнена в рамках государственного задания Института экологии растений и животных УрО РАН и при финансовой поддержке РФФИ, проект № 19-04-00795A.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объектов животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объектов людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Tiffney B.H. Perspectives on the origin of the floristic similarity between eastern Asia and eastern North America // J. Arnold Arboretum. 1985. V. 66. P. 73–94. https://doi.org/10.5962/bhl.part.13179
Wen J., Nie Z.L., Ickert-Bond S.M. Intercontinental disjunctions between eastern Asia and western North America in vascular plants highlight the biogeographic importance of the Bering land bridge from late Cretaceous to Neogene // J. Syst. Evol. 2016. V. 54. № 5. P. 469–490. https://doi.org/10.1111/jse.12222
Tsutsui K., Suwa A., Sawada K. et al. Incongruence among mitochondrial, chloroplast and nuclear gene trees in Pinus subgenus Strobus (Pinaceae) // J. Plant Res. 2009. V. 122. P. 509–521. https://doi.org/10.1007/s10265-009-0246-4
Wang B.S., Wang X.R. Mitochondrial DNA capture and divergence in Pinus provide new insights into the evolution of the genus // Mol. Phylogenet. Evol. 2014. V. 80. P. 20–30. https://doi.org/10.1016/j.ympev.2014.07.014
Hao Z.Z., Liu Y.Y., Nazaire M. et al. Molecular phylogenetics and evolutionary history of sect. Quinquefoliae (Pinus): Implications for Northern Hemisphere biogeography // Mol. Phylogenet. Evol. 2015. V. 87. P. 65–79. https://doi.org/10.1016/j.ympev.2015.03.013
Ran J.H., Shen T.T., Liu W.J. et al. Mitochondrial introgression and complex biogeographic history of the genus Picea // Mol. Phylogenet. Evol. 2015. V. 93. P. 63–76. https://doi.org/10.1016/j.ympev.2015.07.020
Semerikova S.A., Khrunyk Y.Y., Lascoux M., Semerikov V.L. From America to Eurasia: a multigenomes history of the genus Abies // Mol. Phylogenet. Evol. 2018. V. 125. P. 14–28. https://doi.org/10.1016/j.ympev.2018.03.009
Neale D.B., Sederoff R.R. Paternal inheritance of chloroplast DNA and maternal inheritance of mitochondrial DNA in loblolly pine // Theor. Appl. Genet. 1989. V. 77. P. 212–216. https://doi.org/10.1007/BF00266189
Watano Y., Kanai I., Tani N. Genetic structure of hybrid zones between Pinuspumila and P. parviflora var. pentaphylla (Pinaceae) revealed by molecular hybrid index analysis // Am. J. Bot. 2004. V. 91. № 1. P. 65–72. https://doi.org/10.3732/ajb.91.1.65
Semerikov V.L., Semerikova S.A., Polezhaeva M.A. et al. Southern montane populations did not contribute to the recolonization of West Siberian Plain by Siberian larch (Larixs ibirica): A range-wide analysis of cytoplasmic markers // Mol. Ecol. 2013. V. 22. № 19. P. 4958–4971. https://doi.org/10.1111/mec.12433
Farjon A., Rushforth K.D.A classification of Abies Miller. (Pinaceae) // Not. Roy. Bot. Garden. Edinburg. 1989. V. 46. P. 59–79.
Semerikov V.L., Semerikova S.A., Putintseva Y.A. et al. Mitochondrial DNA in Siberian conifers indicates multiple postglacial colonization centers // Canad. J. For. Res. 2019. V. 49. № 8. P. 875–883. https://doi.org/10.1139/cjfr-2018-0498
Untergasser A., Cutcutache I., Koressaar T. et al. Primer3 – new capabilities and interfaces // Nucl. Ac. Res. 2012. V. 40. № 15. e115. https://doi.org/10.1093/nar/gks596
Hall T.A. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Ac. Symp. Series. 1999. V. 41. P. 95–98.
Ronquist F., Huelsenbeck J.P. MrBAYES 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. № 12. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
Swofford D.L. PAUP*. Phylogenetic analysis using parsimony (* and other methods). Version 4.0 beta10. Sunderland: Sinauer Associates, Massachusetts, 2002.
Mamanova L., Coffey A.J., Scott C.E. et al. Target-enrichment strategies for next-generation sequencing // Nat. Methods. 2010. V. 7. № 2. P. 111–118. https://doi.org/10.1038/nmeth.1419
Дополнительные материалы
- скачать TableS1.xlsx
- Таблица S1.


