Генетика, 2021, T. 57, № 7, стр. 850-854
Оценка числа генов, полиморфизм которых влияет на уровень нейротизма
Т. И. Аксенович 1, 2, *, Н. М. Белоногова 1, И. В. Зоркольцева 1, Я. А. Цепилов 1, 2
1 Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
2 Новосибирский государственный университет
630090 Новосибирск, Россия
* E-mail: aks@bionet.nsc.ru
Поступила в редакцию 12.08.2020
После доработки 21.10.2020
Принята к публикации 27.10.2020
Аннотация
Нейротизм – одна из основных личностных характеристик человека, являющаяся важным фактором риска развития психических заболеваний. Интенсивные генетические исследования последних лет позволили сформировать список из примерно 600 генов-кандидатов нейротизма. В этот список вошли как гены, полиморфизм которых непосредственно меняет уровень нейротизма, так и гены, экспрессия которых контролируется генетическими вариантами, лежащими вне гена. Первая группа генов кажется наиболее интересной, поскольку внутригенные сигналы ассоциации легче интерпретировать и для них меньше вероятность ложноположительного результата. В данной работе мы оценили число таких генов в списке из почти 600 известных генов-кандидатов нейротизма, проведя сравнительный анализ результатов двух опубликованных исследований. Мы показали, что 98 из списка известных генов-кандидатов ассоциированы с нейротизмом благодаря своему внутригенному полиморфизу, а 134 – благодаря генетическим вариантам, лежащим вне гена. Для остальных генов мы оценили шанс оказаться в первой группе генов. В итоге, согласно нашим оценкам, только от 153 до 198 генов из почти 600 известных генов-кандидатов нейротизма могут влиять на признак благодаря своему внутригенному полиморфизму. Таким образом, на примере нейротизма мы впервые показали, что только для 25–33% генов, идентифицированных общепринятыми методами, контроль признака обеспечивается внутригенным полиморфизмом. Эта оценка позволяет предсказать эффективность полноэкзомного секвенирования для увеличения мощности анализа ассоциаций.
Нейротизм является одной из основных личностных характеристик человека [1]. Люди с высокими показателями нейротизма характеризуются эмоциональной нестабильностью, возбудимостью, тревожностью, неуверенностью в себе. Генетика нейротизма уже давно привлекает внимание исследователей, поскольку этот признак относительно стабилен [2], обладает высокой наследуемостью [3] и является важным фактором риска развития психических расстройств [4, 5]. Высокая генетическая корреляция между уровнем нейротизма и психическим здоровьем [6–8] позволяет предположить, что изучение генетического контроля нейротизма может помочь понять генетическую архитектуру психических расстройств [9].
До недавнего времени было известно всего несколько потенциальных генов-кандидатов нейротизма. Однако в последние годы был достигнут огромный прогресс в этой области. В наиболее масштабной работе путем метаанализа трех выборок (UK Biobank study, n = 372 903; 23andMe, n = 59 206 и Genetics of Personality Consortium, n = 17 375), включающих в сумме 449 484 человека, было идентифицировано 170 независимых однонуклеотидных полиморфных вариантов (ОНП), ассоциированных с нейротизмом, и предложен список из 599 генов, предположительно влияющих на нейротизм [10]. Этот список был сформирован с помощью нескольких методов и в него вошли гены, которые могли участвовать в контроле признака благодаря внутригенному полиморфизму (первая категория), или ОНП, лежащим вне гена и меняющим его экспрессию (вторая категория). Первая категория генов является более интересной. Внутригенный полиморфизм может модифицировать структуру кодируемых белков благодаря замене аминокислот или альтернативному сплайсингу. Полиморфизм некодирующих внутригенных регионов может регулировать транскрипцию и трансляцию этих генов, формирование белковых комплексов или посттрансляционные модификации [11, 12]. В итоге, внутригенные сигналы ассоциации более интерпретабельны, чем межгенные. В целом, около 85% известных клинически значимых генетических вариантов большого эффекта находятся именно в кодирующих последовательностях или сайтах сплайсинга – c этим связано появление и популярность полноэкзомного секвенирования, в особенности для поиска редких генетических вариантов [13, 14]. Известно также, что вероятность получения ложноположительного результата в поиске ассоциаций существенно зависит от априорной вероятности того, что тестируемый генетический вариант действительно влияет на признак [15]. Судя по имеющимся данным [13, 14], для внутригенных вариантов априорная вероятность выше, и, следовательно, шанс получить ложно-положительный результат значительно ниже, чем при анализе межгенных вариантов.
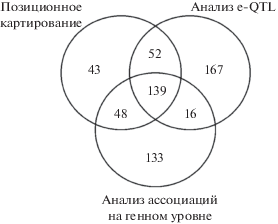
Подавляющее большинство (598 из 599) генов нейротизма, предложенных в работе [10], было идентифицировано с помощью трех методов (рис. 1), каждый из которых не позволяет однозначно отнести найденный ген к одной из двух перечисленных категорий. Позиционное картирование и анализ e-QTL базируются на результатах полногеномного анализа ассоциаций (ПГАА), с помощью которого можно идентифицировать ОНП, независимо ассоциированные с признаком. При позиционном картировании ОНП приписывается к гену, если он лежит внутри него или отстоит от него не далее, чем на 10 тысяч пар оснований [10]. Очевидно, что из-за неравновесия по сцеплению нельзя однозначно отнести найденный таким образом ген к одной из категорий, руководствуясь только положением ОНП относительно гена. При анализе e-QTL проверяется, есть ли разница в экспрессии какого-либо гена у носителей разных аллелей ОНП. В большинстве случаев такой ОНП лежит вне гена, однако иногда он находится внутри него. Наиболее адекватным методом поиска ассоциаций признака с внутригенными вариантами является полногеномный анализ ассоциаций на генном уровне (gene-based association analysis). Суть этого метода заключается в одновременном анализе всех генетических вариантов, лежащих внутри гена [16]. Благодаря этому в анализ вовлекаются редкие генетические варианты, потенциально обладающие большим эффектом на признак. Преимуществами этого метода являются не только увеличение мощности, но и прямое указание на ген, ассоциированный с признаком. Однако недавно нами было показано, что для примерно половины таких генов ассоциация с признаком объясняется неравновесием по сцеплению с ОНП вне гена, дающими сильные сигналы ассоциации [17]. В этой работе был предложен метод, названный polygene pruning, который позволил разделить сигналы, полученные с помощью анализа ассоциаций на генном уровне на те, которые обусловлены внутригенным полиморфизмом, и те, которые наведены ОНП, лежащими вне гена и находящимися в неравновесии по сцеплению с внутригенными ОНП.
Рис. 1.
Диаграмма, иллюстрирующая комбинацию методов, с помощью которых был сформирован список из 598 генов-кандидатов нейротизма в работе [10].
К сожалению предложенный метод нельзя применить ко всем генам из списка 599 генов нейротизма, так как индивидуальные данные о фенотипах и генотипах членов выборки не находятся в открытом доступе, так же как и результаты метаанализа трех выборок, поскольку он использует материал базы данных 23andMe, для которого запрещена публикация результатов ПГАА. Однако результаты ПГАА, полученные в работе [10] на выборке из UK Biobank study (n = 380 506), были опубликованы и позволили нам повторно провести анализ ассоциаций на генном уровне, используя специально разработанные методы [18], и протестировать, обусловлен ли сигнал ассоциации внутригенным полиморфизмом [17].
В данной работе мы сопоставили результаты, полученные в двух исследованиях [10] и [17], для того, чтобы оценить долю генов, ассоциированных с нейротизмом благодаря собственному полиморфизму, среди 598 генов нейротизма, предложенных в работе [10].
Материалом для данного исследования послужил список 598 генов с указанием методов их идентификации и p-value, полученных при анализе ассоциаций на генном уровне (дополнительная табл. 15 из работы [10]), а также списки генов, полученные в работе [17] с использованием всех ОНП внутри гена до и после процедуры polygene pruning (https://zenodo.org/record/3888340#.X3fX7u1S_IU) и дополнительная табл. 1 из работы [17]). Рассматривали только гены с p-value <2.7 × 10–6.
Таблица 1.
Анализ групп генов, включенных в список генов-кандидатов нейротизма благодаря результатам, полученным различными методами
| GB + PM + e-QTL* | GB + PM | GB + e-QTL | GB | PM + e-QTL | PM | e-QTL | Всего | |
|---|---|---|---|---|---|---|---|---|
| Первая категория** | 48 | 23 | 1 | 18 | 5 | 3 | 0 | 98 |
| Вторая категория | 63 | 15 | 4 | 23 | 17 | 8 | 4 | 134 |
| Доля первой категории (m ± se) | 0.43 ± 0.08 | 0.61 ± 0.14 | 0.2 ± 0.2 | 0.44 ± 0.08 | 0.23 ± 0.11 | 0.27 ± 0.13 | 0 ± 0.01 | |
| Прочие | 28 | 10 | 11 | 92 | 30 | 32 | 163 | 366 |
| Всего | 139 | 48 | 16 | 133 | 52 | 43 | 167 | 598 |
Примечание. * – GB (gene-based association analysis) – анализ ассоциаций на генном уровне, PM (positional mapping) – позиционное картирование, e-QTL – анализ e-QTL; ** – первая категория включает гены, полиморфизм которых ассоциирован с нейротизмом, вторая категория включает гены, ассоциированные с нейротизмом благодаря неравновесию по сцеплению с ОНП вне гена.
Прежде всего мы сравнили результаты полногеномного анализа ассоциаций на генном уровне, полученные для одних и тех же генов в работах [10] и [17] и показали, что коэффициент корреляции между логарифмами p-value достаточно высок (r = 0.78, n = 225). Это позволяет нам использовать результаты, полученные в работе [17] для детализации результатов, описанных в статье [10].
Затем мы сравнили списки генов из работ [10] и [17]. Оказалось, что 98 генов, успешно прошедших процедуру polygene pruning в работе [17] – присутствуют в списке из 598 известных генов-кандидатов. Эти гены продемонстрировали ассоциацию с нейротизмом благодаря внутригенному полиморфизму, то есть относятся к первой категории. Еще 134 гена, не прошедшие процедуру polygene pruning в работе [17], также оказались в списке из 598 известных генов-кандидатов. Ассоциация этих генов с нейротизмом была обусловлена ОНП, лежащими за пределами гена. Эти гены относятся ко второй категории. Списки генов, отнесенных нами к первой и второй категориям, свободно доступны по адресу (https://zenodo.org/record/4063652#.X3cEuWlS_IU).
Для остальных 366 генов-кандидатов, идентифицированных в работе [10], мы не располагали достаточной информацией для отнесения их к какой-либо категории. Для этих генов мы попытались оценить шансы попасть в первую категорию, используя информацию о том, с помощью каких методов были идентифицированы эти гены. Мы разбили полный список из 598 генов-кандидатов нейротизма, предложенный в работе [10] на семь частей в зависимости от комбинации методов, используемых для составления этого списка, в соответствии с рис. 1. Для каждой части мы подсчитали число генов, отнесенных нами к первой и второй категориям. В качестве оценки шанса генов попасть в первую категорию мы использовали отношение числа генов первой категории к сумме генов первой и второй категорий. Полученные результаты приведены в табл. 1.
Как видно, наибольший шанс попасть в первую категорию наблюдается у генов, включенных в список генов-кандидатов по результатам анализа ассоциаций на генном уровне. В среднем он составляет 0.46. Для генов, не показавших сигнала ассоциации на генном уровне, но включенных в список по результатам позиционного картирования, шанс попасть в первую категорию ниже, в среднем он составляет 0.24. Самый низкий шанс у генов, попавших в список только благодаря анализу e-QTL.
Используя полученные оценки, мы подсчитали, что среднее число генов, предположительно относящихся к первой категории, среди генов, пока не отнесенных ни к одной из категорий, равно 76.3. С учетом 95%-го доверительного интервала минимальная оценка числа таких генов равна 55.01, а максимальная – 99.47. Учитывая, что 98 генов из списка генов-кандидатов уже были включены в первую категорию, можно заключить, что только от 153 (25%) до 198 (33%) генов из 598 генов-кандидатов нейротизма могут влиять на признак благодаря своему внутригенному полиморфизму.
Таким образом, мы впервые оценили долю генов, идентифицированных общепринятыми методами, для которых контроль признака обеспечивается внутригенным полиморфизмом. Для нейротизма она составила от 25 до 33%. Это означает, что мощность анализа ассоциаций значительной части генов не увеличится при использовании результатов секвенирования экзома. Кроме того, мы составили список из 98 генов-кандидатов нейротизма, внутригенный полиморфизм которых наиболее вероятно контролирует уровень этого признака. Таким образом, нам удалось конкретизировать накопленную информацию о генетике нейротизма, что может способствовать лучшему пониманию генетической архитектуры этого признака.
Работа поддержана грантом РФФИ (20-04-00464), бюджетным проектом Института цитологии и генетики (0324-2019-0040-C-01/АААА-А17-117092070032-4) и программой “5-100 лучших университетов” Министерства науки и высшего образования Российской Федерации.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Widiger T.A. Neuroticism // Handbook of Individual Differences in Social Behavior. N.Y.: Guilford Press, 2009. P. 129–146.
Matthews G., Deary I., Whiteman M. Personality Traits. Cambridge: Cambridge Univ. Press, 2009. 602 p.
Vukasovic T., Bratko D. Heritability of personality: A meta-analysis of behavior genetic studies // Psychol. Bull. 2015. V. 141. № 4. P. 769–785. https://doi.org/10.1037/bul0000017
Hettema J.M., Neale M.C., Myers J.M. et al. A population-based twin study of the relationship between neuroticism and internalizing disorders // Am. J. Psychiatry. 2006. V. 163. № 5. P. 857–864. https://doi.org/10.1176/ajp.2006.163.5.857
Kendler K.S., Myers J. The genetic and environmental relationship between major depression and the five-factor model of personality // Psychol. Med. 2010. V. 40. № 5. P. 801–806. https://doi.org/10.1017/S0033291709991140
Adams M.J., Howard D.M., Luciano M. et al. Genetic stratification of depression by neuroticism: revisiting a diagnostic tradition // Psychol. Med. 2019. V. 50. № 15. P. 2526–2535. https://doi.org/10.1017/S0033291719002629
Luciano M., Hagenaars S.P., Davies G. et al. Association analysis in over 329 000 individuals identifies 116 independent variants influencing neuroticism // Nat. Genet. 2018. V. 50. № 1. P. 6–11. https://doi.org/10.1038/s41588-017-0013-8
Ohi K., Otowa T., Shimada M. et al. Shared genetic etiology between anxiety disorders and psychiatric and related intermediate phenotypes // Psychol. Med. 2020. V. 50. № 4. P. 692–704. https://doi.org/10.1017/S003329171900059X
Goldstein B.L., Klein D.N. A review of selected candidate endophenotypes for depression // Clin. Psychol. Rev. 2014. V. 34. № 5. P. 417–427. https://doi.org/10.1016/j.cpr.2014.06.003
Nagel M., Jansen P. R., Stringer S. et al. Meta-analysis of genome-wide association studies for neuroticism in 449 484 individuals identifies novel genetic loci and pathways // Nat. Genet. 2018. V. 50. № 7. P. 920–927. https://doi.org/10.1038/s41588-018-0151-7
Leppek K., Das R., Barna M. Functional 5' UTR mRNA structures in eukaryotic translation regulation and how to find them // Nat. Rev. Mol. Cell Biol. 2018. V. 19. № 3. P. 158–174. https://doi.org/10.1038/nrm.2017.103
Mayr C. What are 3' UTRs doing? // Cold Spring Harb. Perspect Biol. 2019. V. 11. № 10. P. a034728. https://doi.org/10.1101/cshperspect.a034728
Choi M., Scholl U.I., Ji W. et al. Genetic diagnosis by whole exome capture and massively parallel DNA sequencing // PNAS. 2009. V. 106. № 45. P. 19096–19101. https://doi.org/10.1073/pnas.0910672106
LaDuca H., Farwell K.D., Vuong H. et al. Exome sequencing covers >98% of mutations identified on targeted next generation sequencing panels // PLoS One. 2017. V. 12. № 2. P. e0170843. https://doi.org/10.1371/journal.pone.0170843
Wacholder S., Chanock S., Garcia-Closas M. et al. Assessing the probability that a positive report is false: An approach for molecular epidemiology studies // J. Natl. Cancer Inst. 2004. V. 96. № 6. P. 434–442. https://doi.org/10.1093/jnci/djh075
Li B., Leal S.M. Methods for detecting associations with rare variants for common diseases: Application to analysis of sequence data // Am. J. Hum. Genet. 2008. V. 83. № 3. P. 311–321. https://doi.org/10.1016/j.ajhg.2008.06.024
Belonogova N.M., Zorkoltseva I.V., Tsepilov Y.A., Axenovich T.I. Gene-based association analysis identified 190 genes with polymorphisms affecting neuroticism // Sci. Rep. 2021. V. 11. № 1. P. 2484. https://doi.org/10.1038/s41598-021-82123-5
Svishcheva G.R., Belonogova N.M., Zorkoltseva I.V. et al. Gene-based association tests using GWAS summary statistics // Bioinformatics. 2019. V. 35. № 19. P. 3701–3708. https://doi.org/10.1093/bioinformatics/btz172
Дополнительные материалы отсутствуют.


