Генетика, 2021, T. 57, № 8, стр. 871-886
Генетические факторы риска развития коронавирусной инфекции COVID-19
О. С. Глотов 1, 2, *, А. Н. Чернов 1, С. Г. Щербак 1, 3, В. С. Баранов 2
1 Городская больница № 40 Курортного административного р-на Сестрорецк
197706 Санкт-Петербург, Россия
2 Научно-исследовательский институт акушерства,
гинекологии и репродуктологии им. Д.О. Отта
199034 Санкт-Петербург, Россия
3 Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: olglotov@mail.ru
Поступила в редакцию 14.09.2020
После доработки 28.12.2020
Принята к публикации 18.01.2021
Аннотация
Пандемия коронавирусной инфекции COVID-19 распространилась на 215 стран мира и вызвала десятки миллионов случаев инфицирования и более миллиона летальных исходов во всем мире. В разгар COVID-19-инфекции крайне актуально для борьбы с данной пандемией выявление новых белковых и генных мишеней, которые могут оказаться высокочувствительными диагностическими и прогностическими маркерами тяжести и исхода заболевания. Выявление индивидуальной генетической предрасположенности позволяет персонифицировать программы медицинской реабилитации и терапии. Сейчас уже показано, что на трансмиссивность и тяжесть COVID-19-инфекции могут влиять варианты как в генах организма человека ACE2, HLA-B*4601, FcγRIIA, MBL, TMPRSS2, TNF-α, IL6, антигена А группы крови и др., так и самого вируса (в РНК-полимеразе – ORF8, РНК-праймазе – ORF6, S-, N-, E-белках). Наличие мутаций в белках вируса может изменять аффинность и специфичность связывания с ними таргентных препаратов, являясь молекулярной основой индивидуальных различий в реакции организма человека на противовирусные препараты и/или вакцины. В обзоре обобщены данные о вариантах геномов коронавируса и человека, ассоциированных с индивидуальной предрасположенностью к повышенному или пониженному риску передачи, тяжести и исхода COVID-19-инфекции. Кратко рассматриваются таргетные лекарственные препараты и вакцины, создаваемые для терапии инфекции COVID-19.
Коронавирусы (CoV) являются возбудителями острого тяжелого респираторного синдрома (SARS-CoV), впервые вызвавшего вспышку глобальной эпидемии в 2002 г., когда было инфицировано 8000 человек, 10% из которых завершились летальным исходом [1]. Позднее, в 2012 г. произошла вспышка ближневосточного респираторного синдрома (MERS-CoV), когда эпидемия охватила 26 стран [2]. В декабре 2019 г. в Китае была зафиксирована новая форма коронавируса (SARS-CoV-2), которая стала причиной глобальной пандемии коронавирусной болезни (COVID-19) в 2020 г. [3]. За короткое время COVID-19-инфекция распространилась на 215 стран мира, в том числе и на Россию. В мире и в России количество новых случаев инфицирования и летальности от коронавируса с каждым днем продолжает увеличиваться и на сегодняшний день достигло более 45 миллионов [4].
При инфицировании SARS-CoV-2 у пациента по истечению 2–14 суток инкубационного периода характерно проявление симптомов острого респираторного вирусного заболевания: повышенной температуры тела (90%); кашля (в 80% случаев), одышки (в 30% случаев), утомляемости (в 40% случаев), заложенности в грудной клетке (в 20%), боли в горле, насморка, снижения обоняния и вкуса, конъюнктивита. Данные симптомы могут свидетельствовать о развитии пневмонии без дыхательной недостаточности, острого респираторного дистресс-синдрома – ОРДС (пневмония с острой дыхательной недостаточностью), сепсиса, септического шока или полиорганной недостаточности [5]. Эти патологические состояния наиболее часто приводят к летальности пациентов трудоспособного возраста (59.7 ± 13.3 года) с хроническими заболеваниями: артериальной гипертензией (23.7–30%), сахарным диабетом (16.2%), метаболическим синдромом, ишемической болезнью сердца (5.8%), хронической обструктивной болезнью легких (ХОБЛ), никотиновой зависимостью, воспалительными заболеваниями кишечника и онкологической патологией [6–8]. Кроме того, группы риска инфицирования COVID-19 могут составлять пациенты с генетическими заболеваниями. Например, в исследовании, проведенном учеными из университетского медицинского центра Утрехта (Нидерланды), 180 (45.6%) из 395 пациентов с синдромом Дауна заболели тяжелой респираторно-синцитиальной вирусной инфекцией [9]. Клиническая картина у пациентов, входящих в группы риска, характеризуется развитием “синдрома взаимного отягощения”, сопровождающегося прогрессирующей дыхательной и сердечной недостаточностью, что в конечном итоге утяжеляет их состояние и приводит к трудовым потерям, ранней инвалидности и высокой летальности. В связи с этим инфицированные COVID-19 пациенты с хроническими и генетическими заболеваниями особенно остро нуждаются в немедленной диагностике и реабилитации.
В разгар инфекции COVID-19 крайне актуальным для борьбы с данной пандемией является изучение патогенеза данного заболевания, выявление новых белковых и генных мишеней, которые могут оказаться высокочувствительными и специфическими диагностическими и прогностическими маркерами тяжести и исхода заболевания. Единственным точным методом, который позволяет четко верифицировать COVID-19-инфекцию, диагностировать генетические заболевания, сформировать новые группы риска на основе генетической предрасположенности среди пациентов с хроническими заболеваниями или по восприимчивости к инфицированию COVID-19, а также персонифицировать программы медицинской реабилитации, является генетический анализ.
В силу того, что патогенетические механизмы инфекции COVID-19 носят множественный характер, в обзоре рассматриваются генетические аспекты этого процесса на уровне геномов вируса и человека с акцентом на выявление ассоциаций потенциальных прогностических генетических маркеров с тяжестью и исходом заболевания и генетических мишеней для воздействия таргетных фармпрепаратов.
КОРОНАВИРУС SARS-CoV-2: СТРУКТУРА, ГЕНОМ, МУТАЦИИ И ВАРИАНТЫ, АССОЦИИРУЕМЫЕ С РАЗВИТИЕМ ИНФЕКЦИИ
Коронавирусы представляют собой крупные сферические частицы (диаметр 120 нм), состоящие из двухслойной липидной оболочки, включающей четыре белка: мембранный (M, E), шипиковый (S) и гемагглютининовую эстеразу (HE) вокруг нуклеокапсида (N), образованного множеством копий этого белка, связанных с одноцепочечной РНК [10] (рис. 1).
Рис. 1.
Микрофотография (а) и схематическая структура коронавируса SARS-CoV-2 (б). Spike Glycoprotein (S) – S-белок (шипиковый белок); M-Protein – мембранный М-белок; Hemagglutinin-esterase dimer (HE) – димер гемагглютинина и эстеразы; Envelope – оболочка; RNA and N-protein – РНК и N-белок нуклеокапсида, E-Protein – Е-белок оболочки.
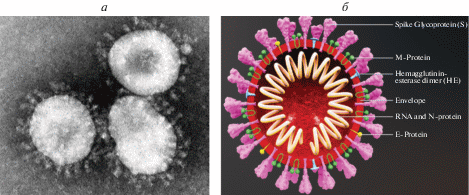
S-белки образуют выросты на оболочке вируса, создавая вид “короны”, благодаря которой вирус и получил свое название [11]. С помощью этих шипов вирусы прикрепляются к белкам-рецепторам клеток хозяина, которые обеспечивают слияние вирусной и клеточной мембран и проникновение вирусной РНК в клетку. S-белки содержат рецептор-связывающий домен (RBD, аминокислоты N318-T509), обеспечивающий взаимодействие с рецептор-связывающим мотивом (RBM, аминокислоты S432-T486) ангиотензин-превращающего фермента 2 (ACE2) – клеточным рецептором для SARS-CoV-2 [12, 13]. Кроме того, S-гликопротеин содержит фурин-подобный сайт рестрикции [14], который необходим для распознавания при пиролизе и, следовательно, способствует зоонозной инфекции вируса.
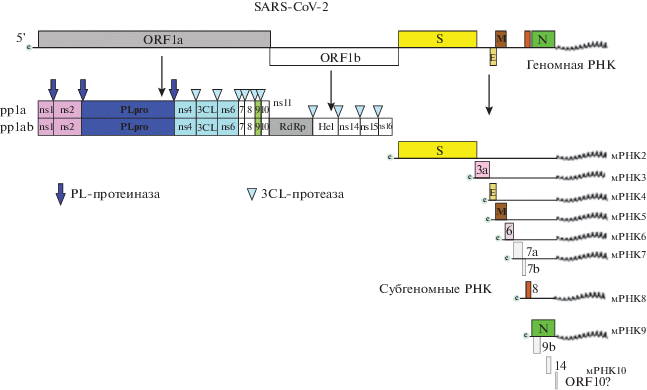
Геном SARS-CoV-2 представлен одноцепочечной РНК длиной около 30 тыс. пн, содержащей на 5'-конце cap-область и на 3'-конце poly-A-последовательность, которые позволяют вирусной РНК транслироваться на рибосомах клеток хозяина. Вирусная РНК включает регуляторные последовательности, в которых происходит терминация транскрипции и 10 открытых рамок считывания (ORF), которые транскрибируются с образованием мРНК (рис. 2).
При трансляции вирусной РНК в клетке хозяина синтезируется полипротеин 1a/1ab (pp1a/pp1ab). Гены ORF1a и ORF1b кодируют продукцию полипротеина репликазы/транскриптазы pp1a/pp1ab, который затем расщепляется вирусной химо-трипсиноподобной (3CLpro) основной (Mpro) или двумя папаиноподобными протеазами с образованием 16 неструктурных белков (Nsps). Другие ORF кодируют структурные протеины: S-белок, мембранный, белок оболочки и нуклеокапсида, а также вспомогательные белки, которые обладают более высокой иммуногенностью, чем неструктурные белки [10, 16]. Во время сборки вириона нуклеопротеин (ORF9a) упаковывает вирусную РНК в спиральный рибонуклеокапсид (РНП) посредством ее взаимодействия с вирусным геномом и мембранным белком М [17]. Нуклеопротеин участвует в повышении эффективности транскрипции субгеномной вирусной РНК и в репликации вируса. В образовании мультипротеинового репликационно-транскрипционного комплекса (RTC) также участвуют неструктурные белки. Основным белком этого комплекса является РНК-зависимая РНК-полимераза (RdRp), которая участвует в репликации и транскрипции вирусной РНК. Другие неструктурные белки комплекса выполняют вспомогательные функции при репликации и транскрипции. Например, белок экзорибонуклеаза, обладая корректирующей активностью, обеспечивает дополнительную точность репликации, отсутствующую у РНК-зависимой РНК-полимеразы.
Мутации в генах коронавируса могут фенотипически проявляться в изменении аминокислот в кодируемых белках, а, следовательно, влиять на функции этих белков и процессы с их участием. X. Tang. с соавт. [18] провели популяционный анализ 103 геномов SARS-CoV-2 у больных из разных регионов мира. На основании различий штаммов (r2 = 0.954) по двум SNP в точках 8782 (ORFLab, T8517C, синонимичная) и 28144 (ORF8, C251T, S84L) выделено два генетических типа вируса – L (частота 71%) и S (частота 29%). Вирусы L-типа, производные вирусов S-типа, более агрессивны и вирулентны вследствие более высоких скоростей передачи генетической информации и репликации [18]. В 103 секвенированных геномах SARS-CoV-2 идентифицированы мутации в 149 участках, причем родительские штаммы содержали 43 синонимичные, 83 несинонимичные и две стоп-мутации. Среди этих мутаций наиболее частыми были синглтоны (65.1%, 28/43) синонимичных и 84.3% (70/83) несинонимичных мутаций, что указывает на их недавнее происхождение или рост числа случаев. Однако 16.3% (7 из 43) синонимичных мутаций и одна несинонимичная (ORF8, L84S, 28144) мутация имели частоту ≥70% среди штаммов SARS-CoV-2. Другие несинонимичные мутации, имеющие аллели более чем в двух штаммах SARS-CoV-2, затрагивали шесть белков: Orf1ab (A117T, I1607V, L3606F, I6075T), S (H49Y, V367F), Orf3a (G251V), Orfa (P34S), Orf8 (V62L, S84L) и N (S194L, S202N, P344S) [18] (табл. 1).
Таблица 1.
Мутации и функции белков SARS-CoV-2
| Мутация, ген | Белок | Функция |
|---|---|---|
| 241C>T, лидерная последовательность | Необходима для трансляции вирусной мРНК на рибосомах в клетках человека | |
| A117T, I1607V, L3606F, I6075T, ORFLab | PL-протеиназа | Протеиназа PL в Nsp3 расщепляет Nsps 1–3 и блокирует врожденный иммунный ответ хозяина, способствуя экспрессии цитокинов |
| H49Y, V367F, 23403A>G (D614G) |
S-белок | Белок S представляет собой сильно гликозилированный белок, который образует шипы на поверхности вириона и обеспечивает проникновение вируса в клетки хозяина. Белок обладает высокой иммуногенностью по отношению к ответу Т-клеток |
| 26144G>T (G251V), 3037C>T (F105F), ORF3a | Nsp3 | Nsp3 – наиболее важный и самый крупный компонент комплекса репликации и транскрипции |
| 8782C>T(S75S), ORF4 | Nsp4 | Белок Nsp4 кооперируется с Nsp3 и Nsp6 для индукции двух мембранных везикул (DMV) и мембранного комплекса, служащего платформой для репликации и сборки РНК |
| 11083G>T (L37F), ORF6 | Nsp6 | |
| P34S, ORF7a |
Nsp7 | Nsp7 и Nsp8 необходимы для репликации и транскрипции SARS-CoV-2. Комплекс Nsp7 и Nsp8 представляет собой мультимерную РНК-полимеразу, участвующую в инициации de novo репликации и элонгации праймера |
| V62L, 28144T>C (S84L), ORF8, 28881G>A (R203K), 28882G>A (R202R), 28883G>C (G204R) |
Nsp8, РНК-праймаза | РНК-зависимые РНК-полимеразы (RNApol). В отличие от Nsp12, Nsp8 обладает способностью инициации репликации de novo без праймеров |
| 14408C>T (P323L) | Nsp12, РНК-полимераза | РНК-зависимая РНК-полимераза |
| 17747C>T (P504L), 17858A>G (Y541C) |
Геликаза | Участвует в создании репликационного комплекса |
| S194L, S202N, P344S | N-белок | Нуклеопротеидный N-белок располагается в ядре и взаимодействует с вирусной РНК, образуя нуклеокапсид |
| 27046C>T | М-белок (мембранный белок) | Входит в состав липидной мембраны вируса |
Другие авторы (O.A. MacLean et al. [19]) подвергали критике утверждение о том, что обнаружение большинства образцов с определенной мутацией не является доказательством более высокой скорости распространения вирусов с этой мутацией. Для такого утверждения необходимо сравнить результаты (числа инфицированных случаев) при нулевом распределении и равных скоростях передачи. Поскольку этого не было сделано, авторы считают, что нет достаточных доказательств о наличии разницы в скоростях передачи между разными генетическими типами SARS-CoV-2. Различия в наблюдаемом количестве образцов, содержащих и не содержащих эти мутации, скорее всего, могли быть результатом случайных эпидемиологических факторов [19].
В исследовании Уотсонского центра (Нью-Йорк, США) [20] просеквенировано 48 геномов SARS-CoV-2 и обнаружено 129 мутаций, в том числе 80 уникальных, 43 миссенс, 21 синонимичная, три делеции, 11 некодирующих мутаций и две делеции в некодирующей ДНК. Наиболее распространенные варианты 8782C>T (ORF1ab) и 28144T>C (ORF8) были найдены в 13 образцах, а вариант 29095C>T (N) – в пяти образцах. Варианты 8782C>T и 29095C>T являются синонимичными, тогда как 28144T>C вызывает замену аминокислоты L84S в Orf8. 12 из 13 образцов с этими вариантами были выявлены за пределами Ухани. Из 43 миссенс-вариантов 30 обнаружены в локусе ORF1ab. Интересно, что число мутаций в Nsp3-домене больше, чем в других доменах. Все три обнаруженные делеции локализованы в домене Nsp1 ORF1ab, а две делеции в некодирующей ДНК – в 3'-UTR и 5'-UTR областях, которые не влияют на функции белка [20].
Интерес вызывает исследование C. Yin (кафедра математики, статистики и информатики Иллинойского университета, Чикаго, США) [21]. При генотипировании 442 штаммов SARS-CoV-2 из мировой базы данных GISAID, в геномах SARS-CoV-2 обнаружено несколько частых мутаций (вариантов) и проведен сравнительный анализ их частот в образцах эпидемиологического региона. Эти SNP, по-видимому, могут влиять на трансмиссивность и патогенность SARS-CoV-2 (табл. 1).
Первая частая мутация (SNP, 241C>T) в геноме SARS-CoV-2 локализована в лидерной последовательности – важном сайте генома для прерывистой субгеномной репликации. Эта мутация ассоциирована с тремя мутациями: 3037C>T, 14408C>T и 23403A>G, вызывающими замены аминокислот в Nsp3 (синонимичная мутация), РНК-полимеразе (P323L) и S-белке (D614G) соответственно. Поскольку эти три мутации (241C>T, 14408C>T и 23403A>G) находятся в белках, участвующих в репликации: в РНК (241C>T, 14408C>T) и в связывании с рецептором ACE2: в S-белке (23403A>G) и распространены в изолятах вируса из Европы, где наблюдалось большое число и более тяжелых случаев инфицирования COVID-19, чем в других географических регионах, то эти варианты могут способствовать повышенной трансмиссивности вируса [21]. Второй вариант (28144T>C) локализуется в белке Nsp8, вызывая замену аминокислоты лейцин (L) на серин (S). Белок Nsp8 инициирует de novo репликацию РНК в отсутствие праймеров [22]. Этот результат согласуется с данными исследования, выполненного на 103 геномах SARS-CoV-2, в котором были выделены S- и L-типы вируса по двум ко-мутациям (8782C>T и 28144T>C) [18]. Интересно, что праймер-независимая РНК-полимераза (Nsp8) содержит больше мутаций, чем любые другие белки (28144T>C, 28881G>A, 28882G>A и 28883G>C), которые могут способствовать устойчивости к мутагенным воздействиям благодаря повышенной точности репликации. Об этом свидетельствует исследование, в котором мутация в РНК-полимеразе повышала точность репликации в РНК-вирусе [23]. Третий SNP (26144G>T) присутствует в неструктурном белке 3 (Nsp3: G251V), который кооперируется с Nsp4 и Nsp6 для индукции двух мембранных везикул (DMV) и мембранного комплекса, служащего платформой для репликации и сборки РНК [24]. Мутация D614G локализована в области связывания S1–S2 вблизи фуринового сайта распознавания (R667), участвующего в расщеплении S-белка, которое необходимо для проникновения вириона в клетки и их персистенции в них [25]. Функциональное значение SNP (23403A>G), вызывающего аминокислотную замену в S-белке (D614G), неясно. Если мутация летальна или снижает способность вируса к распространению, то функциональное значение такой мутации не может быть установлено. Только мутации в S-белке, которые повышают аффинность к клеточным рецепторам ACE2 и снижают иммунный ответ, имеют шансы на существование. По всей вероятности, данные варианты являются результатом естественного отбора и эволюции вируса [21].
Анализ SNP в образцах из эндемического региона позволил выявить два SNP (8782C>T, 28144Tdel>C), распространенных в Китае, Европе и США. Позже один из этих штаммов мутировал (8782C>T, 28144T>C, 18060C>T), что привело к возникновению четырех генотипов SARS-CoV-2: генотип I (11083G>T), генотип II (26144G>T), генотип III (8782C>T, 28144T>C), генотип IV (241C>T, 3037C>Т, 14408С>Т, 23403А>G). Штаммы в пределах одного генотипа происходят от одного предка [21]. Предполагается существование восьми генетических штаммов SARS-CoV-2 [26].
Штаммы вируса с функционально значимыми SNP (241C>T, 3037C>T, 23403A>G) встречались преимущественно в изолятах SARS-CoV-2 из стран Европы. В этих штаммах выявлены дополнительные мутации в положениях (14408C>T, 23403A>G), которые затрагивали РНК-полимеразу (Nsp8), РНК-праймазу (Nsp12) и S-белок. Эти мутации, вероятно, могут быть ассоциированы с тяжестью инфекции COVID-19 в странах Европы. Анализируя профили SNP вирусов из изолятов мировой коллекции разных генераций вируса, установлено, что одна мутация может возникать в одном поколении [21].
Полногеномное секвенирование образцов мировой коллекции вируса SARS-CoV-2 (хранилище NCBI) позволило выявить 47 распространенных SNP, затрагивающих основные вирусные белки: S-белок, Nsp1, RdRp и Orf8. Белки сильно мутировали. Мутации возникали при распространении вируса от человека к человеку за последние 3 мес. (март–май) пандемии. Наличие этих мутаций в ключевых белках вируса может быть причиной его различной вирулентности, особенностей реакции человека на противовирусные препараты и различной летальности от вирусной инфекции [27]. Китайскими учеными обнаружено 14 новых SNP (9 из 14 несинонимичных) в кодирующей области генома СOVID-19 с частотами от 10 до 50%, которые объединены в четыре группы LG_1–LG_4 [28]. Показано, что при мутационном отборе штаммов из Европы возникли мутанты LG_1-группы, тогда как в результате отбора штаммов из Америки образовались мутанты LG_2 и LG_3 групп. Таким образом, увеличение числа новых аллелей COVID-19 обусловлено генетической дифференциацией штаммов Европы и Америки. Установлены положительная и отрицательная корреляции между инфицированностью пациентов группами мутантов LG_1 и LG_2 и LG_3 соответственно и показателем летальности от COVID-19. Эти наблюдения позволяют предположить, что по сравнению со штаммами LG_2 и LG_3 групп штаммы вируса LG_1 более вирулентны, что отчасти может объяснять более высокие показатели летальности от инфекции COVID-19 в странах Европы, чем в США [28].
Напротив, мутации в Orf1a/b могут ослаблять вирулентность коронавируса. Так, мутация Y6398H в белке Orf1a/b (p59/Nsp14/ExoN) ослабляет вирулентность мышиного коронавируса (MHV-A59), что было связано с уменьшением его репликации в течение пяти суток после заражения [29]. Мутация T8517C в Orf1ab SARS-CoV-2 не изменяет последовательности белка (замена кодона AGT (Ser) на AGC (Ser)), но, скорее всего, влияет на трансляцию белка Orf1ab [18].
ВАРИАНТЫ ГЕНОМА ЧЕЛОВЕКА, АССОЦИИРОВАННЫЕ С РАЗВИТИЕМ ИНФЕКЦИИ COVID-19
ACE2
Генетические факторы организма-хозяина могут способствовать развитию и определять тяжесть инфекции SARS-Cov-2. Установлено, что клеточным рецептором SARS-Cov-2 является ангиотензин-превращающий фермент-2 (ACE2) [30, 31]. В организме человека этот белок кодируется геном ACE2. Полиморфизмы ACE2 могут влиять на аффинность и специфичность связывания S-белка с ACE2, а, следовательно, определять наследственную предрасположенность к риску инфицирования и летальности SARS-CoV-2, которые ассоциированы с развитием артериальной гипертензии, сахарного диабета, церебрального инсульта, составляющих группу высокого риска инфицирования и летальности инфекции COVID-19. Примечательно, что экспрессия ACE2 существенно повышена у пациентов с диабетом 1-го и 2-го типов, при терапии которых используются ингибиторы ACE2. В свою очередь лечение ингибиторами ACE повышает экспрессию ACE2. Следовательно, увеличенная экспрессия ACE2 может способствовать повышению риска инфицирования и летальности от COVID-19. А восприимчивость человека к SARS-Cov-2 может быть результатом сочетанного влияния терапии и особенностей полиморфизма гена ACE2 [32].
С другой стороны, популяционные частоты 15 SNP гена ACE2 в различных регионах мира, согласно китайским исследованиям, показали, что наибольшей восприимчивостью к SARS-Cov-2 обладает население Китая, Юго-Восточной Азии и Северной Америки, тогда как европейцы имеют наименьшие частоты этих SNP. Например, rs4646127, локализованныйв интроне ACE2, имеет самые высокие частоты (AF) в популяциях Китая (0.997), Юго-Восточной Азии (0.994) и низкие AF в популяциях Европы (0.651) и Америки (0.754). Причем 11 SNP (AF > 0.05) и один редкий вариант (rs143695310) были ассоциированы с высокой экспрессией ACE2 в тканях [33] (табл. 2).
Таблица 2.
Полиморфизм генов человека, ассоциированный с развитием острой респираторной коронавирусной инфекции
| Генетические варианты, ассоциированные с повышенным риском инфекции | Генетические варианты, ассоциированные с пониженным риском инфекции |
|---|---|
| ACE2 (rs200180615, C>T, c.2002G>A, c.1669G>A, p.Glu668Lys, p.Glu557Lys, T = 0.00053) 34; | AHSG (rs2248690, T>A, g.186612299T>A, AA, T = 0.239816) 62; |
| ACE2 (rs140473595, С>T, c.1501G>A, p.Ala501Thr, T = 0.000265) 34; | CD209 (rs4804803, A>G, c.–336A>G, G = = 0.233427, AG/GG – лучший прогноз, устойчивость к инфекции) 62; |
| ACE2 (rs2285666, C>T, g.8790G>A, T = 0.350199) 34; | Mx1 (rs17000900, C>A, c.–123C>A, A=0.171925) 94; |
| ACE2 (rs4646127, A>G, g.15597330A>G, A = 0.190728) 13; | OAS1 (3'-нетранслируемая область, 3'-UTR, AG, GGvsAA – низкий риск) 95 |
| AHSG (rs4917, T>C, c.743T>C, с.746T>C, c.740T>C, p.Met248Thr, p.Met247Thr, p.Met249Thr, T = 0.264577) 61; | |
| ALOXE3 (rs147149459, G>A, c.1889C>T, c.2285C>T, c.1886C>T, p.Pro630Leu, p.Pro762Leu, p.Pro629Leu, A = 0.000399) 98; | |
| ALOXE3 (rs151256885, C>G,T, g.8109899C>G, T = 0.020367) 98; | |
| ANPEP (rs141945020, G>A,C,T, c.389C>T, c.389C>G, c.389C>A, p.Thr130Ile, p.Thr130Ser, p.Thr130Asn, T = 0.00004 – рецептор короновируса 229E) 96; | |
| BRF2 (rs138763430, C>T, c.25G>A, p.Asp9Asn, T = 0.001597) 98; | |
| CCL2 (rs1024611, A>G, g.34252769A>G, G-2518A, G = 0.363618, GG-генотип) 51; | |
| CD14 (rs2569190, A>G, с-159CC, g.140633331A>G, A = 0.46885, CC-генотип) 59; | |
| CYP4F3A (rs3794987, A>G, g.15640081A>G, G = 0.360823) 61; | |
| ERAP2 (rs150892504, C>G,T, c.2251C>G,T, c.2116C>G,T, c.2182C>G,T, p.Arg751Gly, p.Arg751Cys, p.Arg706Gly, p.Arg706Cys, p.Arg728Gly, p.Arg728Cys, T = 0.002596) 21; | |
| Fcgamma RIIA (RR131) 58; | |
| FGL2 (rs2075761, C>A,T, c.158G>A, c.158G>T, p.Gly53Val, p.Gly53Glu, T = 0.151757) 50; | |
| HLA-B*4601 (OR = 2.08, P = 0.04 – тяжесть заболевания) 57; | |
| HLA-B*5401 (OR = 5.44, P = 0.02 – тяжесть заболевания) 57; | |
| ICAM3 (rs2304237, T>C, c.428A>G, c.443T>C, C = 0.179513, TT, CT, CC) 62; | |
| ILA (rs1800587, с.–889T, TC, OR = 10.2, P = 0.031 – повышенная вирусная нагрузка) 51; | |
| IL4 (rs2070874, С>T, c.–33C>T, T = 0.401158) 43; | |
| IL18 (rs1946518, T>G, c.–607, T = 0.407947, с.607T – повышенная вирусная нагрузка, TT, GT) 51; | |
| MBL (codon 54, A/B, B-аллель) 51,52,58; | |
| Mx1 (rs2071430, G>T, c.–88G>T, T = 0.238219, GT vs GG – высокий риск) 95; | |
| RANTES (rs2107538, C>G, c.–28, T = 0.307708, G-аллель,GG-генотип) 53; | |
| RelB (rs2288918, +23962T, TT, OR = 7.2, P = 0.034) 52; | |
| TMEM181 (rs117665206, C>T, c.874C>T, c.874C>T, c.850C>T, c.1207C>T, c.919C>T, p.Arg292Cys, p.Arg411Cys, p.Arg284Cys, p.Arg307Cys, p.Arg411Cys, T = 0.004593) 98; | |
| TNFA (с.–1031CT/CC, остеонекроз головки бедра при SARS-CoV-инфекции) 98; | |
| TNFA (rs1800630, C>A, c–863A>C, A=0.154153 – при SARS-CoV-инфекции) 97 |
Методом РНК-секвенирования установлено, что азиаты имеют более высокую экспрессию ACE2, чем европейцы и афроамериканцы [34]. Установлен высокий уровень экспрессии ACE2 в клетках легких, печени, почек, миокарда, тонкого кишечника и семенников – органах с наибольшим тропизмом к SARS-CoV-2 [10, 35, 36]. Еще в 2005 г. было показано, что в легких, где реплицируется SARS-CoV, ген ACE2 имеет различные варианты сплайсинга [37]. Установлены корреляции между наиболее часто встречающимися (38) вариантами ACE2 и уровнем экспрессии белка в различных тканях человека [38]. Показано, что возраст, пол, раса и курение достоверно (Р = 0.008) влияют на экспрессию гена ACE2 [38]. Проведенный нами анализ 2754 вариантов ACE2 из базы данных геномов популяции Европы выявил более низкое соотношение миссенс-SNP в популяции Южной Европы по сравнению с другими регионами Европы, что может, отчасти, объяснить более высокий уровень смертности от COVID-19 в Испании и Италии. Сравнив частоты пяти вариантов (rs35803318, rs41303171, rs113691336, rs971249, rs2285666) ACE2 в русской и европейской популяциях, мы обнаружили, что русские похожи на другие европейские популяции, что указывает на схожий уровень инфицирования и тяжести заболевания [39]. Эти исследования отчасти могут объяснять основные симптомы COVID-19-инфекции, ее ассоциацию с тяжестью хронических заболеваний перечисленных выше органов. Анализ экспрессии гена ACE2 в 119 типах клеток, 13 тканях человека, а также спектр коэкспрессии ACE2 с 51 корецептором РНК-вируса и 400 мембранными белками жителей Китая, подтвердил экспрессию ACE2 в легких, холангиоцитах печени, клетках толстой кишки, пищевода, эпителиоцитах подвздошной, прямой кишки, желудка и проксимальных канальцах почек. Потенциальными корецепторами SARS-CoV-2 с наиболее сходными паттернами экспрессии с ACE2 в 13 тканях человека могут быть пептидазы ANPEP, DPP4 и ENPEP [40]. Интерес вызывает работа итальянских ученых, проведших полноэкзомное (3984 пациента) и полногеномное (3284 пациента) секвенирование для поиска генетических факторов тяжести инфекции COVID-19. Установлено, что уровни экспрессии трансмембранной сериновой протеазы 2 (TMPRSS2) и частоты ее четырех SNP (rs2298659 (p.Gly259Gly); rs17854725 (p.Ile256Ile), rs12329760 (p.Val160Met), rs3787950 (p.Thr75Thr)) достоверно (P < 2.2 × 10–16) отличались у итальянцев по сравнению с восточноазиатскими и европейскими популяциями. Поскольку уровень летальности от COVID-19 в Италии один из наиболее высоких среди иных популяций, варианты TMPRSS2 могут служить возможными модуляторами тяжести заболевания у итальянцев [41]. Уровни экспрессии ACE2 и TMPRSS2 также были изучены в CD11b-экспрессируемых в эпителиальных клетках кишечника и Т-клетках у пациентов с воспалительным заболеванием кишечника. Экспрессия ACE2 и TMPRSS2 не была увеличена в образцах от пациентов по сравнению с группой контроля. Применение антител к TNF-α – ведолизумаба, устекинумаба и стероидов достоверно снижает экспрессию ACE2 в CD11b-клетках [42].
IL-4, IL-6, IL-18 и TNF-α
Международным коллективом авторов из Великобритании, Нидерландов и Хорватии проведен метаанализ, включавший 386 исследований в отношении поиска SNP, ассоциированных с развитием туберкулеза, гриппа, респираторно-синцитиального вируса, коронавируса и пневмонии. Один SNP (rs2070874) гена IL4 был достоверно (OR = 1.66, 95% CI 1.29–2.14) ассоциирован с риском респираторных инфекций [43] (табл. 2). Повышенная вирусная нагрузка SARS-CoV была ассоциирована с TT-, GT-генотипами rs1946518 (T>G, c.–607IL1, OR = 10.6, 95% CI 2.03–55.0, P = = 0.014), TC-генотипом rs1800587 (с.–889T, OR = 10.2, 95% CI 1.82–56.8, P = 0.031) IL1A и TT-генотипом rs2288918 (+23962T, OR = 7.2, 95% CI 1.47–35.3, P = 0.034) RelB у 94 пациентов Тайваня, инфицированных SARS-CoV. Также обнаружено, что вирусная нагрузка была выше у мужчин (P = 0.0014) и пожилых (старше 65 лет) пациентов (P = 0.015). При COVID-19 наблюдается повышение уровня интерлейкина-6 (IL-6), фактора некроза опухолей-α (TNF-α) и интерферона-γ [44]. Такой “цитокиновый шторм” вызывает тяжелую воспалительную реакцию, наблюдающуюся при COVID-19-инфекции. В ряде исследований и метаанализе, включающем девять исследований и 1426 пациентов с инфекцией, показано, что ее тяжесть (30.66, 95% CI 7.53–53.78, P = 0.009) и процент летальных исходов (41.32, 95% CI 28.15–54.49, P < 0.001) положительно коррелируют с уровнем IL-6 у пациентов [45–48]. Однако дискуссионным остается вопрос об ассоциации вариантов гена TNFА с развитием инфекции SARS-CoV. Одни исследования не обнаружили ассоциации генотипов TT, CT локуса с.–204 TNFА (OR = 0.95, 95% CI 0.90–0.99) у 75 пациентов с интерстициальным фиброзом легких при развитии SARS-CoV-инфекции [49]. Напротив, M. Feldmann et al. полагают, что терапия инфликсимабом и адалимумабом – препаратами против TNF-α – способна уменьшать интенсивность “цитокинового шторма” и воспаления, тем самым может оказаться более эффективной при тяжелой форме COVID-19 [44]. Следовательно, вирусная нагрузка SARS-CoV, являясь предиктором клинических исходов у пациентов, ассоциирована с полиморфизмом в провоспалительных генах, участвующих во врожденном иммунитете, зависимом от возраста и пола [50].
CCL2, RANTES и MBL
Хемокины участвуют в воспалении и противовирусном иммунитете. Установлена ассоциация вариантов rs1024611 (A>G, g.34252769A>G, G-2518A) в гене хемокинового лиганда-2 (CCL2) и лектина, связывающего маннозу (MBL), с восприимчивостью к SARS-CoV у 932 пациентов и у 982 лиц, составляющих группу контроля (табл. 2). Генотип GG rs1024611 связан с высоким уровнем белка CCL2, а B-аллель MBL – с низким уровнем Mbl, оба ассоциированы с повышенным риском инфицирования SARS-CoV (P = 1.6 × 10–4 и 4.9 × × 10–8 для CCL2 и MBL соответственно). При этом сочетание вариантов продемонстрировало (P = 1.3 × × 10–10) кумулятивный эффект риска инфицирования SARS-CoV. Не обнаружено ассоциации между полиморфизмом и тяжестью заболевания [51]. Полиморфизм rs1800450 A/B (230G>A) в промоторе 54-го кодона гена MBL выявлен у 123 (36.0%) из 352 пациентов и достоверно (P = 0.00086) ассоциирован (OR = 1.73, 95% CI 1.25–2.39) с их восприимчивостью к коронавирусной инфекции. При этом носители данного варианта имели умеренную или низкую экспрессию белка Mbl [52]. Поскольку Mbl узнает остатки маннозы в составе углеводородов и глипопротеинов оболочек многих бактерий и вирусов, стимулируя селектиновый путь активации комплемента, уменьшение клеточной экспрессии Mbl будет способствовать снижению врожденного иммунитета и риску инфицирования SARS-CoV-2. В другом исследовании установлена ассоциация CG- (P < 0.0001, OR = 3.28, 95% CI 2.32–4.64) и GG-генотипов (P < 0.0001, OR = 3.06, 95% CI 1.47–6.39) rs2107538 (C>G, c.–28) RANTES с предрасположенностью к инфицированию SARS-CoV у 495 гонконгских пациентов с SARS-CoV и у 578 лиц контрольной группы (табл. 2). У 28 пациентов G-аллель rs2107538 и CG- (OR = = 2.12, 95% CI 1.11–4.06) и GG-генотипы также ассоциированы с повышенной смертностью (OR = = 4.01, 95% CI 1.30–12.4). Таким образом, RANTES участвует в патогенезе SARS-CoV инфекции [53].
АВО и HLA
Коронавирус может реплицироваться в клетках, экспрессирующих антигены группы АBО. Эпидемиологический анализ показал, что носители I (О) группы крови имели низкий риск (OR = = 0.699, 95% CI 0.635–0.770, P < 0.001) инфицирования коронавирусом (SARS-CoV) [54]. Французские исследователи использовали клеточную модель CHO, чтобы изучить, могут ли антитела к антигенам АВО блокировать взаимодействие S-белка SARS-CoV с ACE2. С этой целью EGFP-меченный S-белок экспрессировали в клетках яичника китайского хомячка, трансфицированных α-1,2-фукозилтрансферазой и α-трансферазой. Моноклональные или анти-А антитела человека ингибировали адгезию клеток, экспрессирующих S-белок, с клетками, экспрессирующими ACE2. Эти данные показывают, что анти-А антитела блокируют взаимодействие между вирусом и его рецептором [55]. На 65 пациентах с коронавирусной инфекцией установлено, что повышенный риск инфицирования ассоциирован с аллелями HLA-B*4601 (OR = 2.08, P = 0.04) и HLA-B*5401 (OR = 5.44, P = 0.02), а тяжесть заболевания ассоциирована с аллелем HLA-B*4601 (P = 0.0008) [56]. Интересно, что в семи итальянских и испанских центрах пандемии COVID-19 провели полногеномное секвенирование и проанализировали 8 582 968 SNP у 835 итальянцев и 775 испанцев, инфицированных SARS-CoV-2. Обнаружена ассоциация (OR = 1.77, 95% CI 1.48–2.11, P = 1.15 × 10–10; OR = 1.32, 95% CI 1.20–1.47, P = 4.95 × 10–8) соответственно между сайтами rs11385942 (del>A, g.45834969dup) в гене транскрипционного фактора LZTFL1 (локус 3p21.31) и rs657152 (A>C,T, g.133263862A>C) ABO (локус 9q34.2). В локусе 3p21.31 ассоциация затрагивает гены LZTFL1, SLC6A20, CCR9, FYCO1, CXCR6 и XCR1. Анализ групп крови показал, что более высокий риск инфицирования SARS-CoV-2 связан с группой крови II (А) (OR = 1.45, 95% CI 1.20–1.75, Р = 1.48 × 10–4), тогда как группа крови I (О) по сравнению с другими группами крови оказывает защитный эффект (OR = 0.65, 95% CI 0.53–0.79, Р = 1.06 × 10–5) [57].
FcγRIIA и CD14
Исследователями из Сиднея (Австралия) обнаружена ассоциация (P = 0.03, OR = 3.2, 95% CI 1.1–9.1) гомозиготного H/H131 генотипа Fc-γ-рецептора IIA (FcγRIIA) человека с тяжестью и исходом коронавирусной инфекции у 180 пациентов из Гонконга, инфицированных SARS-CoV [58]. В более поздней работена 152 пациентах установлена ассоциация СС-генотипа rs2569190 (A>G, с.–159CC g.140633331A>G) гена CD14, кодирующего мембранный белок макрофагов с тяжестью коронавирусной инфекции (P = 0.029, OR = 2.74, 95% CI 1.15–6.57) [59]. Эти данные согласуются с тем, что избыточный уровень секреторного белка sCD14 в крови связан с воспалением и грамотрицательным септическим шоком при инфекционном процессе.
AHSG
Известно, что высокий уровень фетуина-A (AHSG) наблюдается при ожирении и ассоциирован с резистентностью к инсулину [60]. В жировой ткани фетуин-A ингибирует экспрессию адипонектина, усиливает воспаление и инактивирует макрофаги [60]. Ген семейства 3A цитохрома P450 (CYP4F3A) кодирует ω-оксидазу, которая инактивирует лейкотриен B4 (LTB4) в печени. Установлена ассоциация трех SNP: rs2248690 (OR = 2.42, 95% CI 1.30–4.51), rs4917 (ОR = 1.84, 95% CI 1.02–3.34) в гене AHSG и rs3794987 (A>G, g.15640081A>G) CYP4F3 с восприимчивостью к SARS-CoV у 624 пациентов из провинции Гуанчжоу (табл. 2). Кроме того, rs2248690 оказывает влияние на транскрипционную активность промотора AHSG, регулирующего сывороточный уровень AHSG [61].
ICAM3
Полиморфизм rs2304237 (c.428A>G) гена, кодирующего молекулу межклеточной адгезии-3 был исследован на восприимчивость носителей полиморфизма к заболеванию и исходу SARS-CoV инфекции у 817 пациентов с тяжелым острым респираторным синдромом. Не обнаружено ассоциации полиморфизма ICAM3 с восприимчивостью организма к коронавирусу. Тем не менее у пациентов с SARS-CoV, гомозиготных по Gly143 гена ICAM3, были определены высокий уровень лактатдегидрогеназы (P = 0.0067, OR = 4.31, 95% CI 1.37–13.56) и низкое количество лейкоцитов (P = 0.022, OR = 0.30, 95% CI 0.10–0.89), что подтверждает роль ICAM3 в патогенезе коронавирусной инфекции [62].
Таким образом, полиморфизмы ACE2 ассоциированы с динамикой и распространенностью, лейкоцитарного антигена – с восприимчивостью и тяжестью SARS-СoV, а гены, регулирующие сигнальные пути через Toll-подобные рецепторы, – с тяжестью инфекции COVID-19 [63].
ТАРГЕТНЫЕ ЛЕКАРСТВЕННЫЕ ПРЕПАРАТЫ И ВАКЦИНЫ
Основные белки: S-гликопротеин, РНК-полимераза, РНК-праймаза, основная протеаза (Mpro, 3CLpro) SARS-CoV-2 рассматриваются в качестве привлекательных и эффективных мишеней для разработки противовирусных препаратов [21].
Ингибиторы 3CLpro
3C-подобная протеаза (3CLpro) участвует в процессинге полипротеинов, транслируемых с вирусной РНК, и необходима для репликации вируса. Например, ученые из Любекского университета (Любек, Германия) на основе рентгеноструктурного анализа 3CLpro SARS-CoV-2 разработали α-кетоамидные ингибиторы с P3-P2 амидной связью в пиридоновом кольце для увеличения периода их полураспада в плазме. Оценив фармакокинетические характеристики ингибитора, исследователи установили его выраженный тропизм к ткани легких и безопасность для ингаляционного введения [64]. Поскольку 3CLpro расщепляет коронавирусные полипротеины по высококонсервативным сайтам рестрикции, разработана in silico трехмерная модель 3CLpro SARS-CoV-2 на основе кристаллической структуры очень схожего ортолога SARS-CoV. С помощью этой модели проведен виртуальный скрининг на применяемые противовирусные препараты и найдены 16 кандидатов в качестве эффективных ингибиторов. Среди них ледипасвир и велпатасвир оказались наиболее привлекательными для терапии COVID-19-инфекции и имели минимальные побочные эффекты в виде усталости и головной боли (табл. 3).
Таблица 3.
Препараты, таргетированные к вирусным и клеточным белкам (по [28])
| Вирусный белок | Название | Препарат | Литературный источник |
|---|---|---|---|
| 3CLpro | Протеаза 3CLpro | Лопинавир | [73, 74] |
| PLpro | Папаиноподобная протеаза PLpro | Лопинавир | [73, 74] |
| RdRp | РНК-зависимая РНК-полимераза | Ремдесивир, рибавирин | [73, 75, 76] |
| S-белок | S-белок | Арбидол | [77, 78] |
| TMPRSS2 | Трансмембранная сериновая протеаза-2 | Камостат мезилат | [79] |
| ACE2 | Ангиотензинпревращающий фермент 2 | Арбидол | [77, 78] |
| AT2 | AT2-рецептор ангиотензина | L-163491 | [80] |
Такие препараты как эпклус (велпатасвир/софосбувир) и ледипасвир (софосбувир) оказались особенно эффективными благодаря их двойному ингибирующему действию на оба вирусных фермента – РНК-зависимую РНК-полимеразу (RdRp) или 3CLpro, вызывая легкие побочные реакции. Тенипозид и этопозид (и его фосфат) были созданы на основе модели их химического связывания с 3CLpro. Однако эти препараты имеют много побочных эффектов и должны применяться внутривенно. Одобренный к применению препарат венетоклакса хорошо связывается с 3CLpro, но имеет побочные действия, включая инфекции верхних дыхательных путей [65]. Другие ингибиторы протеаз – лопинавир и ритонавир, применяемые при терапии ВИЧ-инфекции, могут снижать вирусную нагрузку и улучшать исход COVID-19-инфекции [66].
Препараты, нацеленные на S-белок
В S1 домене S-гликопротеина SARS-CoV-2 содержатся уникальные N- и O-связанные сайты гликозилирования, которые взаимодействуют с CD26 человека, маскируя SARS-CoV-2 от иммунного ответа хозяина и развития вирулентности [67]. Также S-гликопротеин содержит сайт связывания с ACE2, так называемый рецептор-связывающий мотив (RBM, аминокислоты S432-T486), который является основной антигенной детерминантой, способной вызывать выработку нейтрализующих антител, например, моноклональных антител человека mAb80R, конкурирующих с ACE2 за связывание. Скрининг библиотек пептидов RBM позволил получить короткие аминокислотные последовательности (около 40 аминокислот), которые связываются с ACE2 и нейтрализующими антителами mAb80R [1]. В более ранних исследованиях (2009) показано, что RBD формирует комплекс с Fab-доменом нейтрализующего моноклонального антитела F26G19, полученного иммунизацией мышей инактивированным SARS-CoV. Кристаллическая структура комплекса показывает, что поверхность RBD, распознаваемая F26G19, перекрывается молекулярной структурой белка ACE2. Предполагается, что антитела F26G19 нейтрализуют SARS-CoV, блокируя взаимодействие вируса с клетками организма хозяина [68]. Белок S содержит фуриновый сайт рестрикции на границе между S1 и S2 субъединицами, который отличает SARS-CoV-2 от других SARS-CoV вирусов. Американские ученые показали, что мышиные поликлональные антитела против S-белка SARS-CoV-2 эффективно ингибируют проникновение вируса в клетки [69]. Приводятся данные, что блокаторы ангиотензинового рецептора могут оказаться эффективными при терапии коронавирусной инфекции. Например, ирбесартан, одобренный Управлением по санитарному надзору за качеством пищевых продуктов и медикаментов (FDA) для лечения гипертонии и диабетической нефропатии, ингибирует SLC10A1 – котранспортер натрия и желчной кислоты (NTCP), который взаимодействует с транскрипционным репрессором C11orf74, препятствуя образованию Nsp10 SARS-CoV [70, 71]. В июле–августе 2020 г. биотехнологическая компания Regeneron Pharmaceuticals (Нью-Йорк, США) выступила спонсором клинических испытаний по эффективности и безопасности применения двойных моноклональных антител REGN-COV-2, нацеленных на связывание с двумя сайтами S-белка SARS-CoV-2 у 2000 бессимптомных взрослых носителей COVID-19. Эффективность REGN-COV-2 антител оценивается по предотвращению развития инфекции или симптомов у инфицированных пациентов в течение месяца после введения REGN-COV-2 или плацебо. Все участники испытания будут контролироваться на предмет безопасности в течение семи месяцев после окончания испытания [72].
Препараты к липидам и холестерину
Еще одной стратегией терапии коронавирусной инфекции может быть использование препаратов, связывающихся с липидной оболочкой вирусов. Например, циклодекстрин и стеролы снижают инфекционность коронавирусов путем ингибирования вирус-липид-зависимого прикрепления к клеткам-мишеням организма-хозяина [81].
Препараты к рецептору эстрогена
Установлено, что сверхэкспрессия рецептора эстрогена ESR1 ингибирует репликацию вируса [82]. Нестероидные ингибиторы эстрогеновых рецепторов (торемифен) могут эффективно блокировать вирусные инфекции, в том числе COVID-19 посредством дестабилизации гликопротеина вирусной мембраны и ингибирования связи вирусной оболочки с эндосомальной мембраной и, в конечном итоге, подавлять репликации вируса [83]. Торемифен также регулирует экспрессию RPL19, HNRNPA1, NPM1, EIF3I, EIF3F и EIF3E белков, участвующих в COVID-19-инфекции [84]. Следовательно, торемифен может быть потенциальным лекарственным средством для терапии COVID-19-инфекции [66].
Вакцины на основе вирусных белков
Эффективное сдерживание эпидемий коронавируса у сельскохозяйственных животных с помощью вакцин указывает на потенциальный успех вакцинации. S-белок рассматривается в качестве наиболее многообещающей мишени вакцин против коронавируса, а также как интраназальная вакцина против MERS-CoV [85]. Началу таких исследований способствовала разработка моделей на мелких животных, которые эффективно воспроизводят передачу MERS-CoV и симптомы заболевания человека [88]. В настоящее время разрабатываются разные варианты вакцин против SARS-CoV: векторные ДНК-вакцины, живые аттенуированные вакцины и вакцины против субъединицы S-белка MERS-CoV коронавируса человека [86]. Аденовирусная вакцина может эффективно индуцировать иммунный ответ на SARS-CoV-инфицированные Т-клетки и на вирус-нейтрализующие антитела [87]. Оба иммунных ответа обеспечивают длительную защиту организма от вирусной инфекции. У выздоровевших пациентов с коронавирусной инфекцией ответы вирус-нейтрализующих антител снижались примерно через 6 лет, тогда как иммунные ответы на SARS-CoV-инфицированные Т-клетки сохранялись, что свидетельствует о том, что последние необходимы для длительного иммунитета [88, 89]. Кембриджская и Массачусетская биотехнологические компании ModernaInc (США) совместно с Национальным институтом аллергии и инфекционных заболеваний (NIAID) Национального института здравоохранения США разработали вакцину мРНК-1273 против COVID-19. Вакцина предназначена для доставки мРНК, кодирующей S-белок SARS-CoV-2 (S-2P), и вызывает сильный иммунный ответ. На третьей фазе рандомизированного, плацебо-контролируемого исследования на 30 000 взрослых добровольцах в 89 клинических центрах США показано, что данная вакцина безопасна и иммуногенна, т.е. способна индуцировать выработку высокого титра антител, нейтрализующих вирус. Предполагается развитие иммунного ответа и предотвращение симптоматических проявлений COVID-19 после двукратной вакцинации [90, 91]. В России на базе Первого Московского государственного медицинского университета им. И.М. Сеченова и Главного военного клинического госпиталя им. Н.Н. Бурденко на 76 добровольцах проводятся клинические испытания безопасности и переносимости двух вакцин – соответственно “Гам-КОВИД-Вак” и “Гам-КОВИД-Вак Лио”. Сообщается, что российская вакцина против SARS-CoV-2 вызывает иммунный ответ у 100% добровольцев и не обладает побочным действием [92]. Национальным исследовательским центром эпидемиологии и микробиологии им. Н.Ф. Гамалеи (НИЦЭМ) создана вакцина “Спутник-V”, о чем 11 августа 2020 г. объявил Президент России В.В. Путин. В настоящее время завершаются ее клинические испытания на 40 000 добровольцах. Центром “Вектор” создана вакцина “ЭпиВакКорона”, а в октябре 2020 г. она зарегистрирована в Минздраве России. Сейчас обе вакцины проходят пострегистрационные испытания. Центр исследований и разработки иммунобиологических препаратов им. М.П. Чумакова РАН разрабатывает собственную вакцину против SARS-CoV-2, которая в данное время находится на стадии клинических испытаний [93].
ЗАКЛЮЧЕНИЕ
Тяжесть инфекции COVID-19 зависит от полиморфизма генома вируса и некоторых аллелей генома человека. Мутации в вирусных РНК-полимеразе (ORF8, 241C>T, S84L, 14408C>T, C251T), РНК-праймазе (P323L), S-белке (23403A>G, D614G), а также в структурных белках (N, E) повышают иммуногенность белков в отношении иммунного T-клеточного ответа, что может быть ассоциировано с повышенной трансмиссивностью и тяжестью COVID-19-инфекции в странах Европы. Мутации в генах ключевых вирусных белков меняют аффинность и специфичность таргентных препаратов, являясь молекулярной основой различий заболеваемости и смертности, а также реакции организма на противовирусные препараты или вакцины. Установлено, что вирус SARS-CoV-2 проникает в клетку с помощью трансмембранного рецептора, которым является белок ACE2. В свою очередь экспрессия гена ACE2 варьирует в зависимости от возраста, пола, этнической расы пациента и привычки курения. Корецепторами ACE2 для SARS-CoV-2 в тканях человека могут служить пептидазы ANPEP, DPP4 и ENPEP. Особенности экспрессии гена ACE2 отчасти могут объяснять основные симптомы COVID-19-инфекции и ее ассоциацию с хроническими заболеваниями. Установлена ассоциация тяжести COVID-19-инфекции с локусами (HLA-B*4601), генами (FcγRIIA, MBL, TMPRSS2, TNF-α, IL-6 и др.) и антигеном А групп крови человека. Таким образом, проведение генетического тестирования позволяет не только выявить COVID-19-инфекцию у пациентов, но и сформировать новые группы риска на основе как наличия тех или иных хронических заболеваний, так и генетической предрасположенности или по восприимчивости к инфицированию SARS-CoV-2, а также персонифицировать программы медицинской реабилитации.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Freund N.T., Roitburd-Berman A., Sui J. et al. Reconstitution of the receptor-binding motif of the SARS coronavirus // Protein Eng. Des. Sel. 2015. V. 28. № 12. P. 567–575. https://doi.org/10.1093/protein/gzv052
Mackay I.M., Arden K.E. MERS coronavirus: diagnostics, epidemiology and transmission // Virol J. 2015. V. 12. P. 222. https://doi.org/10.1186/s12985-015-0439-5
Liu Z., Xiao X., Wei X. et al. Composition and divergence of coronavirus spike proteins and host ACE2 receptors predict potential intermediate hosts of SARS-CoV-2 // J. Med. Virol. 2020. V. 92. № 6. P. 595–601. https://doi.org/10.1002/jmv.25726
https://www.worldometers.info/coronavirus/#countries.
Временные методические рекомендации: профилактика, диагностика и лечение новой коронавирусной инфекции (COVID-19). Версия 7, М. 03.06.2020. М., 2020. 166 с.
Yang X., Yu Y., Xu J. et al. Clinical course and outcomes of critically ill patients with SARS-CoV-2 pneumonia in Wuhan, China: a single-centered, retrospective, observational study // Lancet Respir. Med. 2020. V. 8. № 5. P. 475–481. https://doi.org/10.1016/S2213-2600(20)30079-5
Fang L., Karakiulakis G., Roth M. Are patients with hypertension and diabetes mellitus at increased risk for COVID-19 infection? // Lancet Respir. Med. 2020. V. 8. № 4: e21. https://doi.org/10.1016/S2213-2600(20)30116-8
Shitao R.A.O., Alexandria L.A.U., Hon-Cheong S.O. Exploring diseases/traits and blood proteins causally related to expression of ACE2, the putative receptor of 2019-nCov: A Mendelian Randomization analysis // Diabetes Care. 2020. V. 43. № 7. P. 1416–1426. https://doi.org/10.2337/dc20-0643
Beatrijs L.P. Bloemers, van Furth A.M., Weijerman M.E. et al. Strengers and Louis J. Bont. Down Syndrome: A Novel Risk Factor for Respiratory Syncytial Virus Bronchiolitis – A Prospective Birth-Cohort Study // Pediatrics. 2007. V. 120. № 4. P. e1076–e1081. https://doi.org/10.1542/peds.2007-0788
Cascella M., Rajnik M., Cuomo A. et al. Features, evaluation and treatment coronavirus (COVID-19) // Stat- Pearls. 2021. Apr 20. PMID: 32150360.
Ashour M.H., Elkhatib W.F., Rahman M.d.M., Elshabrawy H.A. Insights into the Recent 2019 Novel Coronavirus (SARS-CoV-2) in Light of Past Human Coronavirus Outbreaks // Pathogens. 2020. V. 9. № 3. P. 186. https://doi.org/10.3390/pathogens9030186
Li F., Li W., Farzan M., Harrison S.C. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor // Science. 2005. V. 309. P. 1864–1868. https://doi.org/10.1126/science.1116480
Chen Y., Guo Y., Pan Y., Zhao Z.J. Structure analysis of the receptor binding of 2019-nCoV // Biochem. Biophys. Res. Commun. 2020. V. 525. № 1. P. 135–140. https://doi.org/10.1016/j.bbrc.2020.02.071
Coutard B., Valle C., de Lamballerie X. et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade // Antiviral. Res. 2020. V. 176. P. 104742. https://doi.org/10.1016/j.antiviral.2020.104742
Schidtke P. SARS-CoV-2 – part 2 – From the viral genome to protein structures. MARCH 27, 2020. https://www.discngine.com/blog?author=52850d39e-4b0b817d0c61ff9
Li C.K., Wu H., Yan H. et al. T cell responses to whole SARS coronavirus in humans // J. Immunol. 2008. V. 181. № 8. P. 5490–5500. https://doi.org/10.4049/jimmunol.181.8.5490
He R., Leeson A., Ballantine M. et al. Characterization of protein–protein interactions between the nucleocapsid protein and membrane protein of the SARS coronavirus // Virus Res. 2004. V. 105. № 2. P. 121–125. https://doi.org/10.1016/j.virusres.2004.05.002
Tang X., Wu C., Li X. et al. On the origin and continuing evolution of SARS-CoV-2 // Nat. Science Rev. 2020. V. 7. № 6. P. 1012–1023. https://doi.org/10.1093/nsr/nwaa036
MacLean O.A., Orton R., Singer J.B., Robertson D.L. Response to “On the origin and continuing evolution of SARS-CoV-2”. https://virological.org/t/response-to-on-the-origin-and-continuing-evolution-of-sars-cov-2/418
Koyama T., Parida L., Platt D.E. Variant analysis of COVID-19 genomes // Bull. World Health Organ. 2020. V. 98. № 7. P. 495–504. https://doi.org/10.2471/BLT.20.253591
Yin C. Genotyping coronavirus SARS-CoV-2: methods and implications // Genomics. 2020. V. 112. № 5. P. 3588–3596. https://doi.org/10.1016/j.ygeno.2020.04.016
teVelthuis A.J.W., van den Worm S.H.E., Snijder E.J. The SARS-coronavirus nsp7+ nsp8 complex is a unique multimeric RNA polymerase capable of both de novo initiation and primer extension // Nucl. Acids Res. 2012. V. 40. № 4. P. 1737–1747. https://doi.org/10.1093/nar/gkr89
Pfeiffer J.K., Kirkegaard K. A single mutation in poliovirus RNA-dependent RNA polymerase confers resistance to mutagenic nucleotide analogs via increased fidelity // Proc. Natl Acad. Sci. USA. 2003. V. 100. № 12. P. 7289–7294. https://doi.org/10.1073/pnas.1232294100
Angelini M.M., Akhlaghpour M., Neuman B.W., Buchmeier M.J. Severe acute respiratory syndrome coronavirus nonstructural proteins 3, 4, and 6 induce double-membrane vesicles // mBio. 2013. V. 4. № 4. P. e00524-13. https://doi.org/10.1128/mBio.00524-13
Follis K.E., York J., Nunberg J.H. Furin cleavage of the SARS coronavirus spike glycoprotein enhances cell–cell fusion but does not affect virion entry // Virology. 2006. V. 350. № 2. P. 358–369. https://doi.org/10.1016/j.virol.2006.02.003
Weise E. 8 strains of the coronavirus are circling the globe. Here’s what clues they’re giving scientists. USA TODAY. https://www.usatoday.com/story/news/nation/2020/03/27/scientists-track-coronavirus-strains-mutation/5080571002/.
Vankadari N. Overwhelming mutations or SNPs of SARS-CoV-2: A point of caution // Gene. 2020. V. 752. P. 144792. https://doi.org/10.1016/j.gene.2020.144792
Zhu Z., Liu G., Meng K. et al. Rapid spread of mutant alleles in worldwide COVID-19 strains revealed by genome-wide SNP analysis // Virology Bioinformatics. 2021. V. 13. No 2 :evab015. https://doi.org/10.21203/rs.3.rs-23205/v1
Denison M. The attenuated MHV virus exhibited reduced replication in mice at day five following intracerebral inoculation // Patent US20060039926. 2006.
Li F., Li W., Farzan M., Harrison S.C. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor // Science. 2005. V. 309. P. 1864–1868. https://doi.org/10.1126/science.1116480
Yan R., Zhang Y., Li Y. et al. Structural basis for the recognition of the SARS-CoV-2 by full-length human ACE2 // Science. 2020. V. 367. P. 1444–1448. https://doi.org/10.1126/science.abb2762
Miller L.E., Bhattacharyya R., Milleret A.L. Diabetes mellitus increases the risk of hospital mortality in patients with Covid-19: Systematic review with meta-analysis // Medicine (Baltimore). 2020. V. 99. № 40: e22439. https://doi.org/10.1097/MD.0000000000022439
Cao Y., Li L., Feng Z. et al. Comparative genetic analysis of the novel coronavirus (2019-nCoV/SARS-CoV-2) receptor ACE2 in different populations // Cell Discov. 2020. V. 6:11. https://doi.org/10.1038/s41421-020-0147-1
Zhao Y., Zhao Z., Wang Y. et al. Single-cell RNA expression profiling of ACE2, the putative receptor of Wuhan 2019-nCoV // bioRxiv. 2020. https://doi.org/10.1101/2020.01.26.919985
Li W., Zhang C., Sui J. et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2 // EMBO J. 2005. V. 24. № 8. P. 1634–1643. https://doi.org/10.1038/sj.emboj.7600640
Feng Z., Wang Y., Qi W. The small intestine, an underestimated site of SARS-CoV-2 infection: from Red Queen effect to probiotics // Preprints. Posted. 10 March 2020. https://doi.org/10.20944/preprints202003.0161.v1
Itoyama S., Keicho N., Hijikata M. et al. Identification of an alternative 5'-untranslated exon and new polymorphisms of angiotensin-converting enzyme 2 gene: lack of association with SARS in the Vietnamese population // Am. J. Med. Genet. A. 2005. V. 136. № 1. P. 52–57. https://doi.org/10.1002/ajmg.a.30779
Chen J., Jiang Q., Xia X. et al. Individual variation of the SARS-CoV2 receptor ACE2 gene expression and regulation // Aging Cell. 2020. V. 19. No 7: e13168. https://doi.org/10.1111/acel.13168
Shikov A.E., Barbitoff Y.A., Glotov A.S. et al. Analysis of the spectrum of rare and common variants on susceptibility to COVID-19 and severity of outcome // Front. Genet. 2020. V. 11:551220. https://doi.org/10.3389/fgene.2020.551220
Qi F., Qian S., Zhang S., Zhang Z. Single cell RNA sequencing of 13 human tissues identify cell types and receptors of human coronaviruses // Biochem. Biophys. Res. Commun. 2020. V. 526. № 1. P. 135–140. https://doi.org/10.1016/j.bbrc.2020.03.044
Asselta R., Paraboschi E.M., Mantovani A., Duga S. ACE2 and TMPRSS2 variants and expression as candidates to sex and country differences in COVID-19 severity in Italy // Aging (Albany NY). 2020. V. 12. № 11. P. 10087–10098. https://doi.org/10.18632/aging.103415
Burgueño J.F., Reich A., Hazime H. et al. Expression of SARS-CoV-2 entry molecules ACE2 and TMPRSS2 in the gut of patients with IBD // Inflamm. Bowel. Dis. 2020. V. 26. № 6. P. 797–808. https://doi.org/10.1093/ibd/izaa085
Patarčić I., Gelemanović A., Kirin M. et al. The role of host genetic factors in respiratory tract infectious diseases: systematic review, meta-analyses and field synopsis // Sci. Rep. 2015. V. 5: 16119. https://doi.org/10.1038/srep16119
Feldmann M., Maini R.N., Woody J.N. et al. Trials of anti-tumour necrosis factor therapy for COVID-19 are urgently needed // The Lancet. 2020. V. 395. P. 1407–1409. https://doi.org/10.1016/S0140-6736(20)30858-8
Ulhaq Z.S., Soraya G.V. Interleukin-6 as a potential biomarker of COVID-19 progression // Med. Mal. Infect. 2020. V.50. № 4. P. 382–383. https://doi.org/10.101
Aziz M., Fatima R. Elevated interleukin-6 and severe COVID-19: a meta-analysis // J. Med. Virol. 2020. V. 92. No 11. P. 2283–2285. https://doi.org/10.1002/jmv.25948
Zhu J., Pang J., Ji P. et al. Elevated interleukin-6 is associated with severity of COVID-19: a meta-analysis // J. Med. Virol. 2021. V. 93. No 1. P. 35–37. https://doi.org/10.1002/jmv.26085
Chen X., Zhao B., Qu Y. et al. Detectable serum SARS-CoV-2 viral load (RNAaemia) is closely correlated with drastically elevated interleukin 6 (IL-6) level in critically ill COVID-19 patients // Clin. Infect. Dis. 2020. V. 71. No 8. P. 1937–1942. https://doi.org/10.1093/cid/ciaa449
Wang S., Wei M., Han Y. et al. Roles of TNF-alpha gene polymorphisms in the occurrence and progress of SARS-Cov infection: a case-control study // BMC Infect. Dis. 2008. V. 29. № 8:27. https://doi.org/10.1186/1471-2334-8-27
Chen W.J., Yang J.Y., Lin J.H. et al. Nasopharyngeal shedding of severe acute respiratory syndrome-associated coronavirus is associated with genetic polymorphisms // Clin. Infect. Dis. 2006. V. 42. № 11. P. 1561–1569.
Tu X., Chong W.P., Zhai Y. et al. Functional polymorphisms of the CCL2 and MBL genes cumulatively increase susceptibility to severe acute respiratory syndrome coronavirus infection // J. Infect. 2015. V. 71. № 1. P. 101–109. https://doi.org/10.1016/j.jinf.2015.03.006
Zhang H., Zhou G., Zhi L. et al. Association between mannose-binding lectin gene polymorphisms and susceptibility to severe acute respiratory syndrome coronavirus infection // J. Infect. Dis. 2005. V. 192. № 8. P. 1355–1361. https://doi.org/10.1086/491479
Ng M.W., Zhou G., Chong W.P. et al. The association of RANTES polymorphism with severe acute respiratory syndrome in Hong Kong and Beijing Chinese // BMC Infect. Dis. 2007. V. 7: 50.
Wu B.-B., Gu D.-Z., Yu J.-N. et al. Association between ABO blood groups and COVID-19 infection, severity and demise: A systematic review and meta-analysis // Infect. Genet. Evol. 2020. V. 84:104485. https://doi.org/10.1016/j.meegid.2020.104485
Guillon P., Clément M., Sébille V. et al. Inhibition of the interaction between the SARS-CoV spike protein and its cellular receptor by anti-histo-blood group antibodies // Glycobiology. 2008. V.18. № 12. P. 1085–1093. https://doi.org/10.1093/glycob/cwn093
Lin M., Tseng H.K., Trejaut J.A. et al. Association of HLA class I with severe acute respiratory syndrome coronavirus infection // BMC Med. Genet. 2003. V. 4:9.
Ellinghaus D., Degenhardt F., Bujanda L. et al. The severe Covid-19 GWAS group. Genomewide association study of severe Covid-19 with respiratory failure // The New England J. Med. 2020. https://doi.org/10.1056/NEJMoa2020283
Yuan F.F., Tanner J., Chan P.K. et al. Influence of FcgammaRIIA and MBL polymorphisms on severe acute respiratory syndrome // Tissue Antigens. 2005. V. 66. № 4. P. 291–296. https://doi.org/10.1111/j.1399-0039.2005.00476.x
Yuan F.F., Boehm I., Chan P.K. et al. High prevalence of the CD14-159CC genotype in patients infected with severe acute respiratory syndrome-associated coronavirus // Clin. Vaccine Immunol. 2007. V. 14. № 12. P. 1644–1645. https://doi.org/10.1128/CVI.00100-07
Bourebaba L., Marycz K. Pathophysiological implication of fetuin-A glycoprotein in the development of metabolic disorders: a concise review // J. Clin. Med. 2019. V. 8. № 12. P. 2033. https://doi.org/10.3390/jcm8122033
Zhu X., Wang Y., Zhang H. et al. Genetic variation of the human α-2-Heremans-Schmid glycoprotein (AHSG) gene associated with the risk of SARS-CoV infection // PLoS One. 2011. V. 6. № 8: e23730. https://doi.org/10.1371/journal.pone.0023730
Chan K.Y., Ching J.C., Xu M.S. et al. Association of ICAM3 genetic variant with severe acute respiratory syndrome // J. Infect. Dis. 2007. V. 196. № 2. P. 271–280.
Debnath M., Banerjee M., Berk M. Genetic gateways to COVID-19 infection: Implications for risk, severity, and outcomes // FASEB J. 2020. V. 34. No 7. P. 8787–8795. https://doi.org/10.1096/fj.202001115R
Zhang L., Lin D., Sun X. et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors // Science. 2020. V. 368. P. 409–412. https://doi.org/10.1126/science.abb3405
Chen Y.W., Yiu C.B., Wong K.Y. Prediction of the SARS-CoV-2 (2019-nCoV) 3C-like protease (3CL pro) structure: Virtual screening reveals velpatasvir, ledipasvir, and other drug repurposing candidates // F1000Res. 2020. V. 9:129. https://doi.org/10.12688/f1000research.22457.1
Zhou J., Fang L., Yang Z. et al. Identification of novel proteolytically inactive mutations in coronavirus 3C-like protease using a combined approach // FASEB J. 2019. V. 33. № 12. P. 14575–14587. https://doi.org/10.1096/fj.201901624RR
Vankadari N., Wilce J.A. Emerging WuHan (COVID-19) coronavirus: Glycan shield and structure prediction of spike glycoprotein and its interaction with human CD26 // Emerg. Microbes Infect. 2020. V. 9. № 1. P. 601–604. https://doi.org/10.1080/22221751.2020.1739565
Pak J.E., Sharon C., Satkunarajah M. et al. Structural insights into immune recognition of the severe acute respiratory syndrome coronavirus S protein receptor binding domain // J. Mol. Biol. 2009. V. 388. № 4. P. 815–823. https://doi.org/10.1016/j.jmb.2009.03.042
Walls A.C., Park Y.J., Tortorici M.A. et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein // Cell. 2020. V. 181. № 2. P. 281–292. https://doi.org/10.1016/j.cell.2020.02.058
Zhou Y., Hou Y., Shen J. et al. Network-based drug repurposing for novel coronavirus 2019-nCoV/SARS-CoV-2 // Cell Discov. 2020. V. 6:14. https://doi.org/10.1038/s41421-020-0153-3
Hong M., Li W., Wang L. et al. Identification of a novel transcriptional repressor (HEPIS) that interacts with nsp-10 of SARS coronavirus // Viral. Immunol. 2008. V. 21. № 2. P. 153–162. https://doi.org/10.1089/vim.2007.0108
Study assessing the efficacy and safety of anti-spike SARS CoV-2 monoclonal antibodies for prevention of SARS CoV-2 infection asymptomatic in healthy adults who are household contacts to an individual with a positive SARS-CoV-2 RT-PCR assay. https://clinicaltrials.gov/ct2/show/NCT04452318. Data assess: 19.08.2020.
Sheahan T.P., Sims A.C., Leist S.R. et al. Comparative therapeutic efficacy of remdesivir and combination lopinavir, ritonavir, and interferon beta against MERS-CoV // Nat. Commun. 2020. V. 11: 222. https://doi.org/10.1038/s41467-019-13940-6
Maxmen A. More than 80 clinical trials launch to test coronavirus treatments // Nature. 2020. V. 578. P. 347–348. https://doi.org/10.1038/d41586-020-00444-3
Guo D. Old weapon for new enemy: drug repurposing for treatment of newly emerging viral diseases // Virol. Sin. 2020. V. 35. № 3. P. 253–255. https://doi.org/10.1007/s12250-020-00204-7
Wang M., Cao R., Zhang L. et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro // Cell Res. 2020. V. 30. № 3. P. 269–271. https://doi.org/10.1038/s41422-020-0282-0
Kadam R.U., Wilson I.A. Structural basis of influenza virus fusion inhibition by the antiviral drug Arbidol // Proc. Natl Acad. Sci. USA. 2017. V. 114. № 2. P. 206–214. https://doi.org/10.1073/pnas.1617020114
The Efficacy of Lopinavir Plus Ritonavir and Arbidol against Novel Coronavirus Infection (ELACOI). https://clinicaltrials.gov/ct2/show/NCT04252885.
Hoffmann M., Kleine-Weber H., Schroeder S. et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor // Cell. V. 181. No 2. P. 271–280.e8. https://doi.org/10.1016/j.cell.2020.02.052
De Witt B.J., Garrison E.A., Champion H.C., Kadowitz P.J. L-163,491 is a partial angiotensin AT(1) receptor agonist in the hindquarters vascular bed of the cat // Eur. J. Pharmacol. 2000. V. 404. № 1–2. P. 213–219. https://doi.org/10.1016/S0014-2999(00)00612-9
Baglivo M., Baronio M., Natalini G. et al. Natural small molecules as inhibitors of coronavirus lipid-dependent attachment to host cells: A possible strategy for reducing SARS-COV-2 infectivity? // Acta Biomed. 2020. V. 91. № 1. P. 161–164. https://doi.org/10.23750/abm.v91i1.9402
Lasso G., Mayer S.V., Winkelmann E.R. et al. A structure-informed atlas of human-virus interactions // Cell. 2019. V. 178. № 6. P. 1526–1541.e16. https://doi.org/10.1016/j.cell.2019.08.005
Zhao Y., Ren J., Harlos K. et al. Toremifene interacts with and destabilizes the Ebola virus glycoprotein // Nature. 2016. V. 535. P. 169–172. https://doi.org/10.1038/nature18615
V’kovski P., Gerber M., Kelly J. et al. Determination of host proteins composing the microenvironment of coronavirus replicase complexes by proximity-labeling // Elife. 2019. V. 8:e42037. https://doi.org/10.7554/eLife.42037
Ma C., Wang L., Tao X. et al. Searching for an ideal vaccine candidate among different MERS coronavirus receptor-binding fragments–The importance of immunofocusing in subunit vaccine design // Vaccine. 2014. V. 32. № 46. P. 6170–6176. https://doi.org/10.1016/j.vaccine.2014.08.086
Schindewolf C., Menachery V.D. Middle east respiratory syndrome vaccine candidates: cautious optimism // Viruses. 2019. V. 11. № 1: 74. https://doi.org/10.3390/v11010074
Gao W., Tamin A., Soloff A. et al. Effects of a SARS-associated coronavirus vaccine in monkeys // Lancet. 2003. V. 362. P. 1895–1896. https://doi.org/10.1016/S0140-6736(03)14962-8
Zumla A., Azhar E.I., Arabi Y. et al. Host-directed therapies for improving poor treatment outcomes associated with the middle east respiratory syndrome coronavirus infections // Int. J. Infect. Dis. 2015. V. 40. P. 71–74. https://doi.org/10.1016/j.ijid.2015.09.005
Prajapat M., Sarma P., Shekhar N. et al. Drug targets for corona virus: A systematic review // Indian J. Pharmacol. 2020. V. 52. № 1. P. 56–65. https://doi.org/10.4103/ijp.IJP_115_20
Phase 3 clinical trial of investigational vaccine for COVID-19 begins. Multi-site trial to test candidate developed by Moderna and NIH. https://www.nih.gov/news-events/news-releases/phase-3-clinical-trial-investigational-vaccine-covid-19-begins.
Jackson L.A., Anderson E.J., Rouphael N.G. et al. An mRNA vaccine against SARS-CoV-2 – preliminary report // New Engl. J. Med. 2020. NEJMoa2022483. https://doi.org/10.1056/NEJMoa2022483
РБК: https://www.rbc.ru/rbcfreenews/5f07c7e49a794-74e5e57c745. Дата обращения 31.07.2020.
РБК: https://www.rbc.ru/society/14/10/2020/ 5f86e-e589a79471be7f80074. Дата обращения 03.11.2020.
Ching J.C., Chan K.Y., Lee E.H. et al. Significance of the myxovirus resistance A (MxA) gene –123C>a single-nucleotide polymorphism in suppressed interferon beta induction of severe acute respiratory syndrome coronavirus infection // J. Infect. Dis. 2010. V. 201. № 12. P. 1899–1908. https://doi.org/10.1086/652799
He J., Feng D., de Vlas S.J. et al. Association of SARS susceptibility with single nucleic acid polymorphisms of OAS1 and MxA genes: a case-control study // BMC Infect. Dis. 2006. V. 6. № 6:106. https://doi.org/10.1186/1471-2334-6-106
Vijgen L., Keyaerts E., Zlateva K., Van Ranst M. Identification of six new polymorphisms in the human coronavirus 229E receptor gene (aminopeptidase N/CD13) // Int. J. Infect. Dis. 2004. V. 8. № 4. P. 217–222. https://doi.org/10.1016/j.ijid.2004.03.004
Wang S., Wei M., Han Y. et al. Roles of TNF-alpha gene polymorphisms in the occurrence and progress of SARS-Cov infection: a case-control study // BMC Infect. Dis. 2008. V. 8:27. https://doi.org/10.1186/1471-2334-8-27
Lu C., Gam R., Pandurangan A.P., Gough J. Genetic risk factors for death with SARS-CoV-2 from the UK Biobank // MedRxiv. 2020. https://doi.org/10.1101/2020.07.01.20144592
Дополнительные материалы отсутствуют.