Генетика, 2022, T. 58, № 1, стр. 116-122
Маркеры хлоропластной ДНК для идентификации близкородственных редких видов рододендронов Восточной Азии
М. А. Полежаева 1, Д. Р. Юнусова 1, *, Н. А. Тихонова 2, А. Н. Полежаев 3, М. Н. Колдаева 4
1 Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
2 Институт леса им. В.Н. Сукачева Сибирского отделения Российской академии наук
660036 Красноярск, Россия
3 Институт биологических проблем Севера Дальневосточного отделения
Российской академии наук
685000 Магадан, Россия
4 Ботанический сад-институт Дальневосточного отделения Российской академии наук
690024 Владивосток, Россия
* E-mail: dianaiunusova@mail.ru
Поступила в редакцию 02.04.2021
После доработки 27.05.2021
Принята к публикации 28.05.2021
- EDN: XUXCCT
- DOI: 10.31857/S0016675822010088
Аннотация
Проведен анализ изменчивости двух фрагментов хлоропластной ДНК (trnH-psbA, TabCD) в шести выборках Rhododendron adamsii с территории Магаданской обл., о. Сахалина, Якутии, Бурятии, Западного Саяна и в двух выборках Rhododendron parvifolium из Хабаровского края и Бурятии. Выявлено шесть гаплотипов: у R. parvifolium изменчивость отсутствует, присутствует один специфический гаплотип; у R. adamsii распространение гаплотипов структурировано географически – образцы из Бурятии образуют гомогенный кластер, в дальневосточной части ареала выявлен полиморфизм. Анализ молекулярной дисперсии (AMOVA) в пределах R. аdamsii показал, что около 98% генетического разнообразия приходится на межпопуляционные различия (FST = 0.98, p < 0.001). Проведено сравнение с близкородственными видами по последовательностям из Генбанка. Данные фрагменты можно рекомендовать для видовой идентификациии изучения популяционной структуры краснокнижного вида R. adamsii.
Динамика состава растительных сообществ и ареалов отдельных видов растений обусловлена различными факторами, включающими хозяйственную деятельность человека, климатические флуктуации, специфические требования видов к условиям местообитаний. Все это отражается на генетическом разнообразии популяций – основе их эволюционного потенциала и устойчивости. Таким образом, эффективные стратегии оценки биологического разнообразия и сохранения редких видов должны основываться на знаниях об их генетической структуре. Особенно это актуально в отношении лекарственных растений. Даже будучи внесенными в реестр Красной книги, они оказываются под угрозой снижения численности из-за бессистемной заготовки сырья. Разработка видоспецифичных генетических маркеров – инструмент, позволяющий не только идентифицировать растение, определить место сбора редкого вида в случае незаконной заготовки, но и изучить его генетическую структуру на всем ареале.
Rhododendron adamsii Rehd. – низкорослый вечнозеленый кустарник, образующий заросли в альпийском и субальпийском поясах на высоте 1200–2500 м над уровнем моря в горах Восточной Сибири и Дальнего Востока, а также Монголии [1]. Вид занесен в красные книги во всех регионах произрастания [2]. Это растение широко используется в индо-тибетской, монгольской и бурятской медицине. В настоящее время R. adamsii не включен в официальный список фармакопейных растений России, однако вид считается перспективным для внедрения в медицинскую практику и активно исследуется с биохимической точки зрения [3–5]. Неконтролируемый сбор в качестве пищевого и лекарственного сырья, приуроченность к особым условиям произрастания (факультативная кальцефилия) и медленный рост [1] – все это ставит под угрозу существование некоторых популяций вида. Особенный антропогенный пресс оказывается в прибайкальских районах, где заготовки осуществляются в коммерческих целях.
Морфологической изменчивости вида посвящено мало работ [3, 6], а популяционно-генетические исследования вовсе не проводились. Очевидно, что необходима разработка маркеров для целей природоохранного мониторинга, а также позволяющих эффективно проводить идентификацию сырья: отличать его от близкородственного схожего морфологически вида R. parvifolium, с которым он нередко сосуществует в одних местообитаниях и даже образует гибриды (×R. burjaticum) [1, 7]. Кроме практического применения, знания о генетической структуре вида представляют фундаментальный интерес, направленный на выявление центральноазиатских флористических связей высокогорной флоры Восточной Сибири. Близкородственные виды R. adamsii произрастают в Гималаях (R. аnthopogon D. Don), Афганистане (R. сolletianum Aith. Et Hemsl.), в Центральном Китае (R. аnthopogonoides Maxim., R. сephalanthum Franch.) [1].
Цель настоящей работы – изучение уровня генетического разнообразия и популяционно-генетической структуры R. adamsii, а также выявление маркера, пригодного для идентификации сырья.
Проанализировано 50 растений рододендрона Адамса из природных популяций с территории Западного Саяна, Бурятии, Якутии, Магаданской обл. и о. Сахалин, а также 23 растения рододендрона мелколистного из Бурятии и Хабаровского края (табл. 1). ДНК выделяли по стандартному протоколу для растительных тканей (СТАВ-метод) [8] из высушенных в силикагеле листьев. Выборка из Магаданской обл. выделена из гербарных образцов ИБПС ДВО РАН (индекс гербария MAG, номера: 0009893, 0009891, 0009902, 0009897, 0009898, 0009909, 0009903, 0009904, 0009905, 009910, 0009915, 0009920). На первом этапе на нескольких образцах была проведена амплификация с универсальными праймерами для девяти маркерных хлоропластных фрагментов. Согласно протоколу и температурному профилю ПЦР, рекомендованными авторами, проведена амплификация части фрагментов trnK–matK [9] и psbC–trnS [10]; трех участков trnT–trnF (TabAB, TabCD, TabEF) [11]; trnF-TrnV [12]; trnH-psbA, trnS-trnG [13] и atpH-atpI [14]. Для фрагментов со стабильной амплификацией методом прямого секвенирования на генетическом анализаторе ABI 3130 (Applied Biosystems, USA) были определены последовательности и выявлены вариабельные сайты. Для анализа всего материала были отобраны маркерные фрагменты, в которых обнаружено больше трех вариабельных сайтов: trnH-psbA и TabCD. Выравнивание последовательностей проводили вручную в программе BioEdit [15]. Расчет уровня дифференциации и распределения генетической изменчивости внутри и между популяциями (анализ молекулярной дисперсии, AMOVA) проводили в программе Arlequin v. 3.5.1.2 [16]. Дерево отношений гаплотипов хпДНК построено в программе Network v. 4.6.1.2 [17]. Каждая мутация (мононуклеотидная замена или индел, независимо от размера), кодировалась как единичное мутационное событие. Филогенетическое дерево для всех образцов построено с использованием байесовского анализа в программе MrBayes v. 3.1.2 [18] на основе модели нуклеотидных замен GTR + G + I. При построении деревьев включены данные из генбанка для близкородственных видов: по фрагменту trnH-psbA для R. anthopogon (номера в Генбанке – KM605665.1, KM605664.1, KM605603.1) и R. cephalantum (номера в Генбанке – JN046777.1, JN046776.1, KM605536.1, JN046775.1); по фрагменту TabCD для R. lapponicum (номера в Генбанке – GQ245396.1, DQ860601.1). В качестве внешней группы использовали образец подрода Terorhodion – Rhododednron camtschaticum.
Таблица 1.
Географические координаты мест сбора и выявленные гаплотипы хпДНК в изученных выборках R. adamsii и R. parvifolium
| Популяции | Коорд., с.ш./в.д. | Число особей | Гаплотип (N) | |
|---|---|---|---|---|
| 1 | R. adamsii. Магаданская обл. (образцы из гербария с индексом MAG) | – | 12 | h1 (11) h3 (1) |
| 2 | R. adamsii. Сахалинская обл., Смирныховский р-н, гора Вайда | 49°52′/143°27′ | 10 | h4 (10) |
| 3 | R. adamsii. Респ. Саха (Якутия), Усть-Майский р-н, пос. Усть-Юдома, гора Иченэ | 59°24′/135°06′ | 9 | h1 (8) h2 (1) |
| 4 | R. adamsii. Бурятия, Тункинский р-н, окр. пос. Аршан | 51°55′/102°24′ | 4 | h5 (4) |
| 5 | R. adamsii. Бурятия, Тункинский р-н, окр. пос. Монды | 51°44′/101°04′ | 9 | h5 (9) |
| 6 | R. adamsii. Красноярский край, Ермаковский р-н, гора Тушканчик (Западный Саян) | 52°49′/93°23′ | 6 | h5 (6) |
| 7 | R. parvifolium. Хабаровский край, Нанайский р-н, южный склон Тордоки-Яни | 48°52′/138°02′ | 11 | h6 (11) |
| 8 | R. parvifolium. Бурятия, Тункинский р-н, окр. пос. Монды | 51°44′/101°04′ | 12 | h6 (12) |
Длина объединенной последовательности по двум фрагментам составила 755 пн (TabCD 1–387; trnH-psbA 388–755) . Выявлено десять вариабельных сайтов (табл. 2), из них во фрагменте TabCD – три замены и два микросателлита во фрагменте; trnH-psbA – четыре мононуклеотидных замены и один индел; изменчивость сгруппировалась в шесть гаплотипов (h1–h6). Последовательности фрагментов для каждого гаплотипа размещены в Генбанк (GenBank) под номерами доступа MW888399–MW888409. Образцы из Генбанка закодированы как гаплотип h8 для R. lapponicum и h9–h11 для R. cephalantum и R. anthopogon.
Таблица 2.
Сегрегирующие сайты для семи гаплотипов хпДНК. Референсный гаплотип h1
| Гаплотип | Позиция нуклеотида | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| trnL-trnF (TabCD) | trnH-psbA | |||||||||
| 54 | 68 | 80 | 151 | 258 | 515 | 548 | 542–557 | 622 | 702 | |
| h1 | T | A | A | C | – | A | T | – | T | T |
| h2 | T | A | A | C | – | A | G | – | T | T |
| h3 | G | – | A | C | – | A | G | – | T | T |
| h4 | T | A | A | C | – | A | T | – | T | C |
| h5 | G | – | A | C | T | A | G | – | T | T |
| h6 | T | – | G | T | – | C | – | a | A | T |
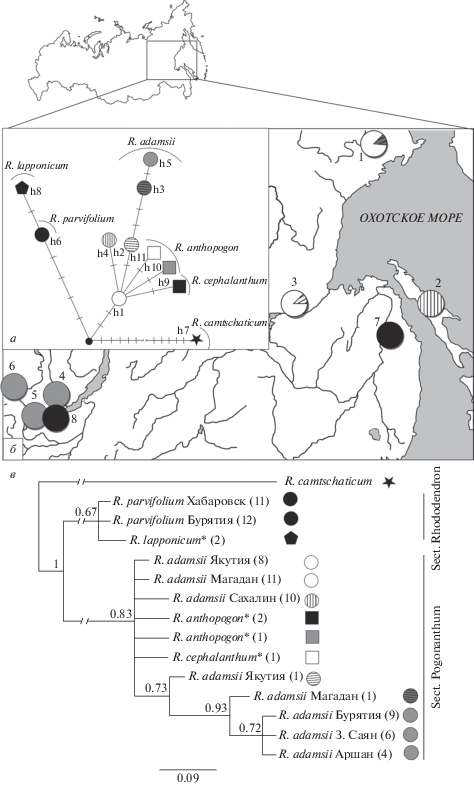
Распределение гаплотипов в изученных образцах отображено на рис. 1 ,б. В выборках R. parvifolium присутствует один видоспецифичный гаплотип h6. У R. adamsii обнаружено пять гаплотипов, причем их распределение структурировано географически. Гаплотип h1 распространен на востоке ареала – в выборках из Магаданской обл. и Якутии. Здесь также выявлены редкие гаплотипы h2 и h3. В выборке из центральной части Сахалина фиксирован гаплотип h4. Гаплотип h5 фиксирован в западной части ареала на территории Бурятии. Для родственных реконструкций изученные образцы были сгруппированы с образцами из Генбанка. Медианная сеть генеалогических связей между гаплотипами (рис. 1 ,а) выявила две гаплогруппы, соответствующие двум секциям, к которым относятся изученные виды: R. parvifolium и R. lapponicum (h6, h8) к секции Rhododendron; R. adamsii, R. anthopogon, R. cephalantum (h1–h5, h9–h11) к секции Pogonanthum [19]. На филогенетическом древе, построенном байесовским методом (рис. 1 ,в), образцы разных популяций сгруппированы в соответствии с секционной таксономической принадлежностью с высокой статистической поддержкой (PP-value = 1.00). В пределах клады секции Pogonathum, подклады выделены с меньшими поддержками (PP-value = 0.72–0.93), однако с четкой структурой по регионам в пределах R. adamsii. По результатам AMOVA коэффициент дифференциации между выборками двух видов – R. parvifolium и R. adamsii, составил FST = 0.985, (P < 0.0001); между выборками в пределах R. adamsii FST = 0.923 (P < 0.0001), еще выше с учетом родства гаплотипов, FST = 0.961 (P < 0.0001).
Таким образом, дифференциация по данным изменчивости хпДНК соответствует двум описанным видам. Отметим, что ранее для R. parvifolium и R. adamsii были определены идентичные последовательности по ядерному маркеру ITS (internal transcribed spaser) [20], который широко используется для видовой идентификации. Использованные нами маркеры хлоропластной ДНК показали большую эффективность. Любой из фрагментов может быть успешно применен на практике для определения вида – R. parvifolium или R. adamsii – методом ПЦР-диагностики. Хотя эти виды имеют отчетливые морфологические признаки (наиболее яркий из которых – соцветия) – из 5–10 воронковидно-колокольчатых цветков с ярко выраженной трубчатой частью у R. adamsii и из 2–5 широко-колокольчатых цветков у R. parvifolium – окрас венчика у обоих видов варьирует от белого до розового и, в целом, они имеют схожий облик [1, 3, 21, 22]. Морфологическая схожесть, способность к скрещиванию, а также генетическая близость по ITS-маркеру ставила под сомнение даже правомерность разнесение данных видов по разным подсекциям [23]. При видовой идентификации в отсутствии генеративных органов, как например высушенных листьев, представляющих пищевое сырье, или в гербарии без цветов, их часто можно спутать. Так, известна работа по определению подлинности сырья на основе анатомических признаков листьев и гистохимической реакции на локализацию действующих веществ [3]. Диагностика с помощью генетического маркера – наиболее простое и оптимальное решение проблемы видового определения.
Использование обоих фрагментов позволяет с большой вероятностью уточнить район произрастания R. adamsii. По нашим данным популяционно-генетическая структура этого вида представляет два кластера (рис. 1 ): мономорфный западный и более разнообразный восточный. Различия между западной и восточной частями ареала также наблюдаются по данным морфологической изменчивости. Линейные параметры листовых пластинок и черешков у тувинской популяции (западная часть ареала) отличались низким уровнем изменчивости по сравнению с якутской популяцией (восточная часть ареала), образцы из этих популяций имели четкое разделение не только по линейным размерам листа, но и по окраске венчика [21].
Особого обсуждения заслуживает сравнение изученных нами видов с близкородственными видами по данным из Генбанка. R. parvifolium и R. lapponicum часто указываются в качестве синонимов [1, 24]. Эти виды слабо различимы морфологически и, скорее, выделяются исключительно за счет географической отдаленности. В широком смысле рододендрон мелколистный имеет обширный ареал, захватывающий кроме Сибири и Дальнего Востока, Скандинавию, Гренландию и Северную Америку [24]. Очевидно, что на таком обширном ареале он вряд ли будет однородным, и вопрос о его генетической структуре и таксономическом делении остается открытым. В нашем исследовании не было выявлено различий между выборками из Бурятии и Хабаровского края, два образца из Генбанка происходящие из Норвегии, также не отличались между собой, но отличались от наших образцов на одну мутацию во фрагменте TabCD.
За схожую морфологию образцы R. adamsii из Сибири были изначально отнесены к виду R. anthopogon, произрастающему в Гималаях, что позже было признано ошибочным [26]. Сравнение наших образцов с несколькими последовательностями trnH-psbA двух близкородственных видов – R. anthopogon и R. cephalantum – указывает на их тесное родство. Ранее сравнение этих видов генетическими маркерами не проводилось. В нашем древе (рис. 1 ,в) они образуют ветви с поддержками на уровне внутривидовой изменчивости. С одной стороны, это может быть связано с неравномерными выборками, так как даже если несколько образцов из Генбанка имеют разные гаплотипы, то сравнение полноразмерных выборок с большой вероятностью позволило бы выявить более четкую структуру. Другая причина может быть в том, что данный тип маркера обладает недостаточной разрешающей способностью именно в пределах секции Pogonathum.
Полученные молекулярно-генетические данные могут быть использованы для разработки программ мониторинга и сохранения этих редких видов, а также для разработки мероприятий по рациональному природопользованию. Фрагменты хлоропластной ДНК trnH-psbA, TabCD подходят для различения видов R. adamsii и R. parvifolium в случае, когда это затруднительно сделать по морфологическим признакам. Для выявления наиболее четкой генетической структуры в пределах западной части ареала R. adamsii, где выражен антропогенный пресс, необходимы дальнейшие исследования с применением мультилокусного генетического анализа. Подтверждено родство R. adamsiiс другими видами (R. anthopogon и R. cephalantum) секции Pogonanthum.
Авторы выражают благодарность Н.К. Бадмаевой, Т.А. Поляковой, А.П. Ефимовой, Е.А. Мудрик, И.В. Фролову за помощь по сбору образцов для исследования, а также М.Г. Хоревой за помощь в работе с гербарием ИБПС ДВО РАН (MAG).
Работа (сбор образцов) выполнена в рамках государственного задания Института экологии растений и животных УрО РАН, проведение молекулярно-генетического анализа поддержано грантом РФФИ № 20-04-00417.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Рис. 1.
а – местоположение изученных выборок, распределение обнаруженных гаплотипов хпДНК. б – дерево гаплотипов хпДНК (h1–h11). Поперечные штрихи на ветвях дерева – мутационные события. Гаплотип h7 составлен по последовательностям соответствующих межгенных спейсеров для образца внешней группы R. camtschaticum; гаплотипы h8–h11 – по данным из Генбанка для видов R. lapponicum, R. anthopogon, R. cephalanthum. в – филогенетическое дерево изученных образцов. (*) отмечены образцы, последовательности для которых взяты из Генбанка, в скобках указано количество особей, у которых встретился данный гаплотип.
Список литературы
Александрова М.С. Рододендроны природной флоры СССР. М.: Наука, 1975. 112 с.
Плантариум: открытый онлайн атлас-определитель растений и лишайников России и сопредельных стран. 2007–2021. https://www.plantarium.ru/page/view/item/31845.html
Мирович В.М., Левента А.И., Федосеева Г.М., Усов Л.А. К морфолого-анатомическому изучению рододендрона Адамса – перспективного растения Восточной Сибири // Сибир. мед. журн. 1997. № 4. С. 34–37.
Мирович В.М. Изучение химического состава надземных органов рододендрона Адамса методом ВЭЖХ // Бюлл. ВСНЦ СО РАМН. 2005. № 7. С. 164–167.
Razgonova M., Zakharenko A., Ercisli S. et al. Comparative analysis of Far East Sikhotinsky rhododendron (Rh. sichotense) and East Siberian rhododendron (Rh. adamsii) using supercritical CO2-extraction and HPLC-ESI-MS/MS spectrometry // Molecules. 2020. V. 25(17). P. 3774. https://doi.org/10.3390/molecules25173774
Каракулов А.В. Межпопуляционная изменчивость морфологических признаков у Rhododendron adamsii Rehd. (Ericaceae DC) // Мат. VIII междунар. форума “Охрана и рациональное использование лесных ресурсов”. Благовещенск: ДГАУ, 2015. С. 71–74.
Малышев Л.И. О новых и редких видах с Восточного Саяна // Ботанические материалы гербария ботанического ин-та АН СССР. Т. 21. М.-Л.: АН СССР, 1961. С. 451–467.
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinusradiata based on RFLP, RAPD and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. P. 673–679. https://doi.org/10.1007/BF00226088
Johnson L.A., Soltis D.E. Phylogenetic inference in Saxifragace aesensustricto and Gilia (Polemoniaceae) using matK sequences // Ann. Mol. Bot. Gard. 1995. V. 82. P. 149–175. https://doi.org/10.2307/2399875
Demesure B., Sodzi N., Petit R.J. A set of universal primers for amplification of polymorphic non-coding regions of mitochondrial and chloroplast DNA in plants // Mol. Ecol. 1995. V. 4. P. 129–134. https://doi.org/10.1111/j.1365-294X.1995.tb00201.x
Taberlet P.T., Geilly L., Patou G., Bouvet J. Universal primers for amplification of three noncoding regions of chloroplast DNA // Pl Mol. Biol. 1991. V. 17. P. 1105–1109. https://doi.org/10.1007/BF00037152
Dumolin-Lapegue S., Pemonge M.-H., Petit R. J. An enlarged set of consensus primers for the study of organelle DNA in plants // Mol. Ecol. 1997. V. 6. P. 393–397. https://doi.org/10.1046/j.1365-294x.1997.00193.x
Hamilton M.B. Four primer pairs for the amplification of chloroplast intergenic regions with intraspecific variation // Mol. Ecol. 1999. V. 8. P. 521–523.
Grivet D., Heinze B., Vendramin G.G., Petit R.J. Genome walking with consensus primers: Application to the large single copy region of chloroplast DNA // Mol. Ecol. Not. 2001. V. 1. P. 345–349. https://doi.org/10.1046/j.1471-8278.2001.00107.x
Hall T.A. Bioedit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Ac. Symp. Ser. 1999. V. 41. P. 95–98. https://doi.org/10.1111/jbi.12867
Excoffier L., Lischer H. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Bandelt H.J., Forster P., Rohl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16 P. 37–48. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Ronquist F., Huelsenbeck J.P. MrBAYES 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. № 12. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
Chamberlain D.F., Hyam R., Argent G., Fairweather G., Walter K.S. The genus Rhododendron, its classification and synonymy. Oxford: Royal Botanic Garden Edinburgh, 1996. 181 p.
Баранова Т.В., Календарь Р.Н., Калаев В.Н. К вопросу филогении видов рода Rhododendron L. на основании исследований последовательности спейсеров ITS1–ITS2 // Сибир. лесной журн. 2014. № 6. С. 29–45.
Каракулов А.В. Межпопуляционная изменчивость морфологических признаков у Rhododendron adamsii Rehd. (Ericaceae DC) // Мат. VIII междунар. форума “Охрана и рациональное использование лесных ресурсов”. Благовещенск: ДГАУ, 2015. С. 71–74.
Каракулов А.В., Карпова Е.А., Васильев В.Г. Эколого-географическая изменчивость морфометрических показателей и состава флавоноидов Rhododendron parvifolium // Turczaninowia, 2018. Т. 21. № 2. С. 133–144. https://doi.org/10.14258/turczaninowia.21.2.14
Куцев М.Г., Каракулов А.В. Реконструкция филогении рода Rhododendron L. (Ericaceae) флоры России на основе последовательности спейсеров ITS1–ITS2 // Turczaninowia. 2010. Т. 13. № 3. С. 59–62.
Сосудистые растения советского Дальнего Востока. Т. 5 / Отв. ред. Харкевич С.С. СПб.: Наука,1991. 390 с.
Флора Сибири и Дальнего Востока. Вып. 2. Двудольные. Ericaceae (в обработке E. Буш). СПб.: Гос. типография, 1915. 364 с.
Wilson E.H. The rhododendrons of Northeastern Asia exclusive of those belonging to the subgenus Anthodendron // J. Arnold Arboretum. 1923. V. 4. № 1. P. 33–56.
Дополнительные материалы отсутствуют.


