Генетика, 2022, T. 58, № 10, стр. 1185-1196
ADAMTS1 дифференциально экспрессируется в лимфоцитах индивидов с различным уровнем эндогенных фокусов γH2AX и частотой радиационно-индуцированных микроядер
С. А. Васильев 1, *, Р. Р. Савченко 1, А. А. Беленко 1, Н. А. Скрябин 1, А. А. Слепцов 1, В. С. Фишман 2, А. А. Мурашкина 3, О. В. Грибова 4, Ж. А. Старцева 4, Е. С. Сухих 5, 6, А. В. Вертинский 5, 6, Л. Г. Сухих 6, О. Л. Серов 2, И. Н. Лебедев 1
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
2 Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
3 Национальный исследовательский Томский государственный университет
634050 Томск, Россия
4 Научно-исследовательский институт онкологии, Томский национальный исследовательский медицинский центр Российской академии наук
634009 Томск, Россия
5 Томский областной онкологический диспансер
634063 Томск, Россия
6 Национальный исследовательский Томский политехнический университет
634050 Томск, Россия
* E-mail: stanislav.vasilyev@medgenetics.ru
Поступила в редакцию 04.04.2022
После доработки 06.05.2022
Принята к публикации 12.05.2022
- EDN: RUPFWK
- DOI: 10.31857/S0016675822100125
Аннотация
Уровень спонтанных и радиационно-индуцированных повреждений ДНК в соматических клетках человека варьирует в зависимости от генетических и средовых факторов. Такая вариация может быть ассоциирована с транскрипционными изменениями в клетках, позволяя использовать уровень экспрессии генов в качестве маркеров индивидуальной чувствительности к мутагенному воздействию. Данное исследование было направлено на выявление и характеристику дифференциально экспрессирующихся генов (ДЭГ) в лимфоцитах индивидов с различным уровнем эндогенных фокусов белков репарации ДНК γH2AX и частотой радиационно-индуцированных микроядер (n = 37). Группа с низким уровнем фокусов характеризовалась 0.18 ± 0.02 фокусов γH2AX на клетку и частотой радиационно-индуцированных микроядер 155.78 ± 47.19‰. Группа с высоким уровнем фокусов характеризовалась 0.49 ± 0.07 фокусов γH2AX на клетку и частотой микроядер 78.44 ± 33.21‰. С помощью полнотранскриптомного анализа профиля экспрессии генов было выделено семь ДЭГ (ENST00000424415, CRNDE, ADAMTS1, ENST00000424084, EIF2A, PNPLA5, FRG2C) (FDR < 0.2). Поскольку металлопротеиназа межклеточного матрикса ADAMTS1 может активировать латентную форму TGFβ, а TGFβ вовлечен в радиационно-индуцированный клеточный ответ, в модельной системе на основании клеточной линии HeLa было изучено влияние дифференциальной экспрессии данного гена на транскрипционный профиль. Показано, что 29 из 160 выявленных ДЭГ вовлечены в апоптоз, репарацию ДНК, переход клеточного цикла из стадии G2 в M и сигнальный путь TGFβ. Таким образом, ADAMTS1 может быть рассмотрен в качестве потенциальной мишени для противоопухолевой терапии.
Популяция человека характеризуется широкой вариабельностью индивидуальной чувствительности к воздействию ионизирующего излучения (ИИ). С одной стороны, радиочувствительность определяется индивидуальной генетической конституцией, с другой – на радиочувствительность могут влиять многие факторы окружающей среды, включая предыдущее воздействие ИИ, связанное с формированием адаптивного ответа [1]. Известно, что повышенная радиочувствительность может являться следствием мутаций в генах, вовлеченных в ответ на повреждение ДНК, как, например, при ряде тяжелых наследственных синдромов, включая атаксию–телеангиэктазию и синдром Ниймеген [2]. Однако эти редкие наследственные состояния объясняют только малую часть популяционной вариабельности индивидуальной радиочувствительности, поэтому наиболее вероятно, что данный феномен определяется генетическим вкладом многих аллелей с низкой пенетрантностью [3]. В последние годы значительные успехи широкогеномных ассоциативных исследований в области радиогеномики позволили выявить SNPs и паттерны экспрессии генов, ассоциированные с повышенным риском развития побочных эффектов лучевой терапии [4–7]. Помимо экспрессии генов, для прогнозирования радиочувствительности могут быть использованы такие функциональные маркеры, как апоптотический индекс [8, 9], частота хромосомных аберраций [10–12] и уровень фокусов белков репарации двунитевых разрывов ДНК [3, 13, 14].
Радиационно-индуцированные фокусы γH2AX и 53BP1 являются хорошо известными маркерами двухцепочечных разрывов ДНК [15], в то время как природа эндогенных фокусов этих белков изучена недостаточно. Эндогенные фокусы γH2AX могут маркировать нерепарированные двухцепочечные разрывы ДНК, вызванные окислительными агентами или другими факторами [16]. Более того, данные фокусы можно увидеть в местах локальных изменений в структуре хроматина после завершения репарации двунитевых разрывов ДНК [17, 18] или на укороченных теломерах [19, 20], которые являются маркером клеточного старения [21].
Фокусы белков репарации ДНК – это молекулярные машины, состоящие из множества белков, вовлеченных в ответ на повреждение ДНК [22], которые потенциально могут влиять на транскрипционный профиль генов. Однако остается неясным, отличается ли транскрипционный профиль в клетках индивидов с различным уровнем эндогенных фокусов белков репарации двунитевых разрывов ДНК. Поэтому цель данного исследования заключалась в выявлении и описании дифференциально-экспрессирующихся генов в клетках индивидов с различным уровнем фокусов белков репарации двухцепочечных разрывов ДНК.
МАТЕРИАЛЫ И МЕТОДЫ
Материалы
Цельная кровь была получена от 37 здоровых добровольцев мужского пола, проживающих в г. Томск и г. Северск (РФ), не подвергавшихся профессиональному воздействию ИИ в возрасте 24–72 лет (38.9 ± 13.9 лет). 16 индивидов были курильщиками. От каждого участника исследования было получено добровольное информированное согласие. Исследование было одобрено локальным этическим комитетом НИИ медицинской генетики Томского НИМЦ (№ 4 от 21.11.2017).
Модельная система с нокаутом и транзиентной экспрессией гена ADAMTS1 была создана на основании клеточной линии HeLa (карцинома шейки матки), любезно предоставленной к. б. н. М.А. Булдаковым (НИИ онкологии Томского НИМЦ).
Забор материала и культивирование
Забор цельной крови осуществлялся в вакуумные пробирки с гепарином натрия (Greiner, Германия). Для анализа частоты микроядер в двухъядерных клетках 1.5 мл цельной крови после воздействия ИИ культивировали в 6 мл среды RPMI 1640 (ПанЭко, Россия), содержащей 10%-ную эмбриональную телячью сыворотку (ЭТС) (HyClone, США) и 20 мкг/мл фитогемагглютинина. Через 44 ч в культуру были добавлены 6 мкг/мл цитохалазина В (Sigma, США), а через 72 ч культивирования была проведена фиксация раствором метанол–уксусная кислота 3 : 1.
Мононуклеары периферической крови (МПК) для оценки уровня фокусов γH2AX и 53BP1 и выделения РНК для анализа экспрессии генов были выделены из необлученных образцов цельной крови с помощью центрифугирования в градиенте фиколла (ПанЭко) с последующей отмывкой в однократном фосфатно-солевом буфере (ПанЭко).
Клеточные линии культивировали в среде DMEM (ПанЭко) с добавлением 10% ЭТС и 0.02 мг/мл эритромицина (Синтез, Россия) при 37°С и 5% СО2.
Иммуноокрашивание и анализ уровня фокусов γH2AX и 53BP1
Процедура иммуноокрашивания была проведена по протоколу, описанному в [23], с использованием первичных моноклональных антител мыши к белку γH2AX (Novus Biologicals) и поликлональных антител кролика к белку 53BP1 (Novus Biologicals) в разведении 1 : 400. В качестве вторичных антител были использованы антитела козы к антителам мыши AF 488 (Thermo, США) и антитела козы к антителам кролика AF 546 (Thermo) в разведении 1 : 400.
Микрофотографии были получены с помощью микроскопа AxioImager Z2 (Carl Zeiss, Германия) с автоматической системой анализа препаратов Metafer (MetaSystems, Германия) с использованием фильтров для DAPI, родамина и FITC. Результирующие изображения получали путем слияния снимков 15 фокальных плоскостей с шагом 0.5 мкм. Фокусы белков репарации ДНК анализировали визуально в 150–200 клетках. Распределение числа фокусов γH2AX и 53BP1 аппроксимировали с помощью распределения Пуассона, как описано в [24], и в последующем анализе вместо среднего числа фокусов была использована λ из распределения Пуассона.
Воздействие ионизирующим излучением
Образцы цельной крови и клеточные линии HeLa с дифференциальной экспрессией ADAMTS1 подвергали воздействию γ-излучения 60Co в дозе 2 Гр с использованием гамма-терапевтического аппарата Theratron Equinox 100 (Best Theratronics, Канада). Образцы цельной крови были облучены в НИИ онкологии Томского НИМЦ. Клетки HeLa были облучены в Томском областном онкологическом диспансере. Все экспериментальные образцы транспортировались на льду.
Микроядерный тест
Микроядерный тест в лимфоцитах периферической крови был проведен в сочетании с флуоресцентной in situ гибридизацией (FISH) с панцентромерными ДНК-зондами, полученными с помощью ПЦР как описано ранее [25], и мечеными TAMRA-dUTP (БиоСан, Россия) в ходе ник-трансляции. FISH была проведена по протоколу, описанному в [26]. Частота центромеро-положительных (MnC+) и центромеро-отрицательных (MnC–) микроядер была оценена по наличию флуоресцентного сигнала от панцентромерного ДНК-зонда с помощью микроскопа AxioImager Z2 (Carl Zeiss, Германия) с фильтрами DAPI и TAMRA путем подсчета 2000 двухъядерных клеток.
Анализ экспрессии генов
РНК из МПК была выделена с помощью набора RNeasy Micro Kit (Qiagen, США) по протоколу производителя. Качество образцов оценивали по показателю целостности РНК (RIN 8.0–10.0) с использованием Agilent 2200 TapeStation (Agilent Technologies, США). Мечение и гибридизацию проводили с использованием микрочипов SurePrint G3 Human GE v2, 8×60K (Agilent Technologies), которые сканировали с помощью сканера Agilent SureScan (Agilent Technologies) в соответствии с рекомендациями производителя.
Клеточные линии HeLa подвергали воздействию γ-излучения в дозе 2 Гр и помещали в СО2-инкубатор на 30 мин при 37°С. Затем клетки промывали 1× PBS и лизировали для выделения РНК с использованием реактива Лира (Биолабмикс, Россия) в соответствии с протоколом производителя. Выделенные образцы РНК очищали с использованием мини-набора RNeasy (Qiagen, США) в соответствии с протоколом производителя. Качество РНК оценивали по числу целостности РНК (RIN 9.0–10.0) с использованием системы Agilent 2100 (Agilent Technologies). Полногеномный анализ экспрессии генов проводили с использованием микрочипов SurePrint G3 Human Gene Expression v2 8×60K (Agilent Technologies) в соответствии с протоколом производителя (версия 6.9.1 от декабря 2015 г.).
Для оценки экспрессии ADAMTS1 и ДЭГ в клеточной линии HeLa была проведена ПЦР в реальном времени. РНК для ПЦР-анализа была выделена через 30 мин после облучения, как было описано ранее. Полученные образцы были обработаны ДНКазой I (Thermo, США) и использованы для синтеза кДНК с помощью набора реагентов RТ-M-MuLV-RH (Biolabmix, Россия) в соответствии с протоколом производителя. ПЦР в реальном времени проводили с использованием BioMaster HS-qPCR SYBR Blue (Biolabmix, Россия). Праймеры для ADAMTS1, SEPP1, LXN, ALPK2 и RPS18 (референс) были разработаны с использованием инструмента для создания праймеров NCB IPrimer Blast (табл. 1).
Таблица 1.
Праймеры для ПЦР в реальном времени
| Олигонуклеотид | Последовательность |
|---|---|
| ADAMTS1_exp_F | 5'-ACATGATGGCGTCAATGC-3' |
| ADAMTS1_exp_R | 5'-ATCAAACATTCCCCATGACC-3' |
| ALPK2_exp_F | 5'-CCATAGCCCAAGTGCAGAGA-3' |
| ALPK2_exp_R | 5'-TTCCGTAGCTGCTGTTCTTGATGC-3' |
| LXN_exp_F | 5'-GAGGATATTCCAGGAAGAGGACA-3' |
| LXN_exp_R | 5'-TTCTTGTCCCGTTGAAGGGT-3' |
| RPS18_exp_F | 5'-GAGGATGAGGTGGAACGTGT-3' |
| RPS18_exp_R | 5'-GGCTAGGACCTGGCTGTATTT-3' |
| SEPP1_exp_F | 5'-ACTGCTCTCTCACGACTCTC-3' |
| SEPP1_exp_R | 5'-TGGTAATGAGGCGATGGAGT-3' |
Создание клеточных линий с нокаутом и сверхэкспрессией гена ADAMTS1
Клеточная линия HeLa с нокаутом гена ADAMTS1 была получена путем внесения мутации сдвига рамки считывания во второй экзон гена с помощью технологии редактирования генома CRISPR/Cas9, как описано в [27]. Сверхэкспрессия ADAMTS1 в HeLa была обеспечена с помощью транзиентной трансфекции плазмиды ADAMTS1_pCSdest (Addgene, 53807) [28], содержащей кодирующую последовательность гена ADAMTS1, как описано в [27].
Статистический анализ данных
Статистический анализ профилей экспрессии генов проводили в среде R на log2-трансформированных данных, после фоновой коррекции и межчиповой квантильной нормализации. Для межгрупповых сравнений использовали модифицированный t-критерий с учетом доли ложноположительных результатов. Данные, обсуждаемые в настоящей публикации, были депонированы в Gene Expression Omnibus и доступны через номер доступа серии GEO GSE97000 [29]. Все остальные статистические анализы выполнялись с помощью рангового критерия Манна–Уитни для групповых сравнений и критерия Спирмена для корреляционного анализа с использованием STATISTICA 8.0 (Statsoft, США).
Для аннотирования ДЭГ использовали базу данных DAVID и инструмент STRING для анализа предсказанных функциональных взаимодействий на уровне белков. Статистический анализ данных, полученных в ходе ПЦР в реальном времени, проводили с использованием t-критерия Стьюдента в STATISTICA 8.0 (Statsoft). Различия считались статистически значимыми при FDR < 0.2 для анализа экспрессии генов после применения поправки на множественность сравнения и p < 0.05 для других анализов. Численные данные представлены в виде среднего значения ± стандартное отклонение (SD).
РЕЗУЛЬТАТЫ
Фокусы белков репарации ДНК и частота микроядер в МПК
В необлученных МПК частота эндогенных фокусов белков репарации составила 0.30 ± 0.19 фокусов/клетку для γH2AX и 1.23 ± 0.81 для 53BP1, а частота γH2AX коррелировала с частотой фокусов 53BP1 (R = 0.48, р = 0.006) (рис. 1,а). Однако только 12.3% фокусов γH2AX были колокализованы с 53BP1. Частота микроядер статистически значимо увеличилась с 5.9 ± 2.2‰ (MnC–: 3.8 ± 1.8, MnC+: 2.1 ± 1.7) в необлученных лимфоцитах до 111.7 ± 43.1‰ (MnC–: 105.6 ± 41.9, MnC+: 6.1 ± 6.5) после облучения в дозе 2 Гр (р < < 0.001) (рис. 1,б). Возраст и статус курения не оказывали существенного влияния на уровень фокусов белков репарации ДНК или микроядер (данные не представлены).
Рис. 1.
Уровень эндогенных фокусов γH2AX и 53BP1 и частоты эндогенных и радиационно-индуцированных микроядер в МПК здоровых индивидов (n = 37). а – корреляция между фокусами γH2AX и 53BP1 в МПК у одних и тех же индивидов; б – частоты MnC– и MnC+ в необлученных лимфоцитах и в лимфоцитах через 72 ч после облучения γ‑излучением в дозе 2 Гр; в – корреляция эндогенного уровня фокусов γH2AX в МПК с частотой эндогенных и радиационно-индуцированных MnC-микроядер через 72 ч после облучения; г – отсутствие корреляции между уровнями эндогенных фокусов 53BP1 в МПК и частотами эндогенных и радиационно-индуцированных MnC-микроядер через 72 ч после облучения.
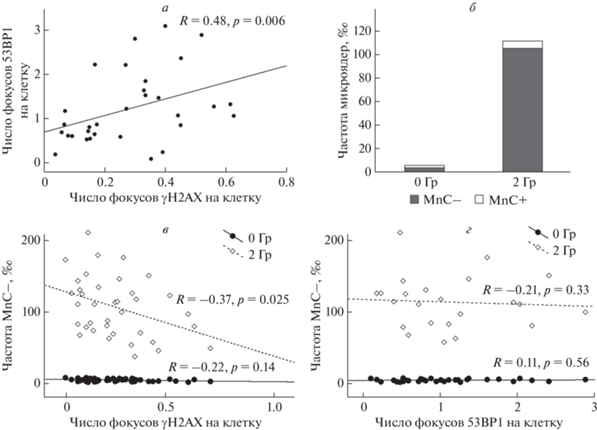
Была показана отрицательная корреляция между уровнем эндогенных фокусов γH2AX в МПК и частотой радиационно-индуцированных центромеро-негативных микроядер (R = –0.37, p = = 0.025) (рис. 1,в). Однако уровень эндогенных фокусов 53BP1 не коррелировал ни с частотой эндогенных, ни с частотой радиационно-индуцированных центромеро-негативных микроядер (рис. 1,г). Возможно, наблюдаемый эффект мог быть результатом потенциального влияния эндогенных фокусов γH2AX на активацию и арест клеточного цикла. Однако доля двухъядерных клеток в облученных клетках обратно коррелировала с частотой центромеро-негативных микроядер после облучения (R = –0.36, p = 0.041), но не с частотой фокусов γH2AX и 53BP1.
Дифференциально-экспрессирующиеся гены в МПК
Полнотранскриптомные профили экспрессии генов были проанализированы в двух группах МПК: 1) с высокой частотой радиационно-индуцированных центромеро-негативных микроядер и низким уровнем эндогенных фокусов γH2AX (n = 3) (группа с низким уровнем фокусов – НФ); 2) с низкой частотой радиационно-индуцированных центромеро-негативных микроядер и высоким уровнем эндогенных фокусов γH2AX (n = 3) (группа с высоким уровнем фокусов – ВФ) (табл. 2).
Таблица 2.
Характеристика здоровых индивидов с различным уровнем фокусов белков репарации ДНК и частотой микроядер
| Доза | Признак | НФ | ВФ | p |
|---|---|---|---|---|
| Количество индивидов | 3 | 3 | ||
| Пол | Мужской | Мужской | ||
| Курильщики | 1 | 1 | ||
| Возраст, лет | 37 ± 14 | 36 ± 8 | ||
| 0 Гр | γH2AX, фокус/клетку | 0.18 ± 0.02 | 0.49 ± 0.07 | 0.001* |
| 53BP1, фокус/клетку | 0.24 ± 0.36 | 1.57 ± 0.7 | 0.042* | |
| Колокализация γH2AX и 53BP1, фокус/клетку | 0.03 ± 0.03 | 0.11 ± 0.08 | 0.167 | |
| Частота MnC–, ‰ | 2.83 ± 1.77 | 3.16 ± 1.35 | 0.808 | |
| Частота MnC+, ‰ | 2.16 ± 1.25 | 1.19 ± 0.36 | 0.269 | |
| Частота Mn, ‰ | 4.99 ± 3.01 | 4.36 ± 0.99 | 0.746 | |
| 2 Гр | Частота MnC–, ‰ | 155.78 ± 47.19 | 78.44 ± 33.21 | 0.081 |
| Частота MnC+, ‰ | 0.67 ± 1.15 | 5.11 ± 6.19 | 0.288 | |
| Частота Mn, ‰ | 156.44 ± 46.53 | 83.56 ± 29.52 | 0.083 |
Анализ результатов микрочипового исследования с использованием t-критерия Стьюдента с поправкой на множественность сравнения показал статистически значимые различия в экспрессии генов FDXR, EDA2R, ENST00000424084, TNFRSF10B, ENST00000452402, RPS27L, FRG2C, BBC3, PVT1, PHLDA3. Воздействие радиации повышало экспрессию шести генов в обеих группах лиц: FDXR, EDA2R, TNFRSF10B, BBC3, RPS27L и PHLDA3. Согласно данным литературы, экспрессия FDXR и EDA2R активируется в ответ на облучение, и эти гены используются в качестве биодозиметров [30].
Некоторые гены дифференциально экспрессировались у индивидов с различным уровнем фокусов белков репарации ДНК и частотой микроядер. Так, в необлученных лимфоцитах экспрессия гена ADAMTS1 и двух генов длинных некодирующих РНК (ENST00000424415 и XLOC_011950 (CRNDE)) была статистически значимо выше в группе индивидов с высоким уровнем фокусов белков репарации и низкой частотой микроядер (табл. 3). После облучения наблюдалось возрастание уровня экспрессии генов ENST00000424415, XLOC_011950 (CRNDE) и ADAMTS1 в группе ВФ и снижение экспрессии EIF2A, PNPLA5 и FRG2C и транскрипта ENST00000424084 по сравнению с группой НФ (табл. 3).
Таблица 3.
ДЭГ в лимфоцитах индивидов с различным уровнем фокусов γH2AX и частотой радиационно-индуцированных микроядер с учетом поправки на множественность сравнения
| Доза | ДЭГ | FC ВФ/НФ |
p (FDR) |
|---|---|---|---|
| 0 Гр | ENST00000424415 | 24.1 | 0.003 |
| CRNDE | 2.9 | 0.107 | |
| ADAMTS1 | 3.3 | 0.114 | |
| 2 Гр | ENST00000424415 | 24.2 | 0.003 |
| CRNDE | 2.9 | 0.052 | |
| ADAMTS1 | 4.2 | 0.023 | |
| ENST00000424084 | 0.4 | 0.005 | |
| EIF2A | 0.2 | 0.133 | |
| PNPLA5 | 0.2 | 0.158 | |
| FRG2C | 0.3 | 0.158 |
Принимая во внимание, что ADAMTS1 был единственным белок-кодирующим геном со значимо отличающейся экспрессией при FDR < 0.05, эффекты дифференциальной экспрессии ADAMTS1 были изучены в модельной системе с нокаутом и сверхэкспрессией ADAMTS1 на основании клеточной линии HeLa.
ДЭГ в клеточной линии HeLa на фоне нокаута или сверхэкспрессии ADAMTS1
Нокаут и сверхэкспрессия гена ADAMTS1 приводили к значимому снижению и повышению экспрессии гена ADAMTS1 в клеточной линии HeLa соответственно (рис. 2,a), что технически верифицирует использованную модельную систему.
Рис. 2.
Верификация экспрессии генов, выявленных с помощью полнотранскриптомного анализа в клеточной линии HeLa с нокаутом ADAMTS1 методом ПЦР в реальном времени. Экспрессия генов ADAMTS1 (а), ALPK2 (б), LXN (в), SEPP1 (г) в клеточной линии HeLa на фоне нокаута и сверхэкспрессии ADAMTS1. ADAMTS1 ko – клеточная линия HeLa с нокаутом ADAMTS1, ADAMTS1 ov – клеточная линия HeLa со сверхэкспрессией ADAMTS1, ADAMTS1 ko-ov – клеточная линия HeLa с нокаутом гена ADAMTS1 после трансфекции плазмидой ADAMTS1_pCSdest. * – статистически значимые различия по сравнению с интактной клеточной линией HeLa; # – статистически значимые различия по сравнению с клеточной линией HeLa с нокаутом ADAMTS1 (p < 0.05).
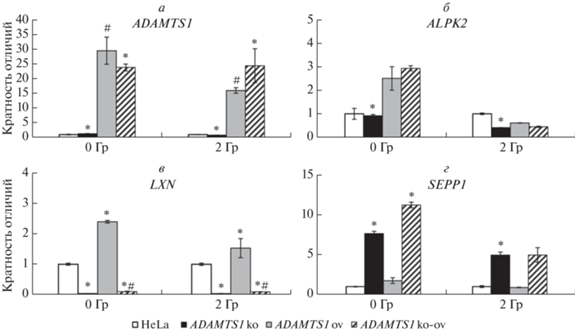
Полнотранскриптомный анализ профиля экспрессии генов в клеточной линии HeLa с нокаутом ADAMTS1 выявил 57 ДЭГ (33 с повышенным и 24 с пониженным уровнем экспрессии, p < 0.05) в клетках до облучения и 103 ДЭГ (56 с повышенным и 47 с пониженным уровнем экспрессии, p ≤ 0.05) после облучения в дозе 2 Гр. Некоторые ДЭГ были связаны с опухолевой резистентностью к γ-облучению и терапии таксолом (GAGE7) [31], с апоптозом (GAGE7, POU3F2) [31, 32], частотой и репарацией радиационно-индуцированных повреждений ДНК (LXN, POU3F2 и RUNX3) [33–35], пролиферацией, миграцией и апоптозом опухолевых клеток (ALPK2) [35], защитой клеток от радиационно-индуцированной токсичности (SEPP1) [36].
После применения поправки на множественность сравнения число ДЭГ уменьшилось до 12 (7 с повышенным и 5 с пониженным уровнем экспрессии, FDR ≤ 0.05) в клетках до облучения и 16 генов (9 с повышенным и 7 с пониженным уровнем экспрессии, FDR ≤ 0.05) в клетках после облучения (табл. 4). Экспрессия некоторых из этих генов (ALPK2, LXN и SEPP1) была выборочно верифицирована в клеточной линии HeLa с нокаутом ADAMTS1 и оценена в клетках со сверхэкспрессией ADAMTS1 с использованием ПЦР в реальном времени (рис. 2). Выявленные отличия в уровне экспрессии были подтверждены для генов ALPK2 и SEPP1 (рис. 2,б–г). Примечательно, что сверхэкспрессия ADAMTS1 в клетках HeLa оказывала противоположный эффект на экспрессию гена LXN. Более того, восстановление экспрессии ADAMTS1 в клеточной линии HeLa с нокаутом ADAMTS1 привело к увеличению экспрессии LXN как в необлученных, так и в облученных клетках (рис. 2,в).
Таблица 4.
ДЭГ в клеточной линии HeLa с нокаутом гена ADAMTS1
| Доза | ДЭГ | FC | p (FDR) |
|---|---|---|---|
| 0 Гр | ↑ PAGE1 | 22.5 | 0.008 |
| ↑ FGF13 | 20.1 | 6.82E-07 | |
| ↑ SEPP1 | 5.8 | 0.004 | |
| ↑ MDFI | 3.8 | 0.028 | |
| ↑ BCHE | 2.6 | 3.93E-05 | |
| ↑ TUSC3 | 2.24 | 0.001 | |
| ↑ POPDC3 | 1.9 | 0.001 | |
| ↓ DLG5 | 2.0 | 0.027 | |
| ↓ ENST00000549261 | 2.0 | 0.001 | |
| ↓ GAGE7 | 3.8 | 2.40E-06 | |
| ↓ POU3F2 | 5.8 | 0.028 | |
| ↓ LXN | 14.2 | 0.004 | |
| 2 Гр | ↑ FGF13 | 21.1 | 5.27E-07 |
| ↑ PAGE1 | 20.5 | 0.011 | |
| ↑ MDFI | 4.6 | 0.009 | |
| ↑ SEPP1 | 4.2 | 0.027 | |
| ↑ SULT4A1 | 4.0 | 0.011 | |
| ↑ SMIM3 | 2.7 | 0.057 | |
| ↑ BCHE | 2.2 | 0.001 | |
| ↑ TUSC3 | 2.1 | 0.002 | |
| ↑ POPDC3 | 1.54 | 0.053 | |
| ↓ DLG5 | 2.0 | 0.023 | |
| ↓ ENST00000549261 | 2.3 | 5.00E-05 | |
| ↓ OLFML1 | 2.6 | 0.022 | |
| ↓ GAGE7 | 4.5 | 0.005 | |
| ↓ POU3F2 | 6.7 | 0.011 | |
| ↓ LXN | 17.3 | 0.002 | |
| ↓ ALPK2 | 2.3 | 0.093 |
Анализ обогащения с использованием инструмента DAVID Bioinformatics Resources не выявил статистически значимо обогащенных групп генов. Однако анализ предсказанных функциональных взаимодействий на уровне белков с помощью инструмента STRING позволил выявить статистически значимые взаимодействия между списками ДЭГ (57 генов до облучения и 103 гена после облучения), выявленных в клеточной линии HeLa с нокаутом ADAMTS1, и наборами генов сигнального пути апоптоза, индуцированного внутриклеточными (GO: 0072332, 75 генов) и внеклеточными (GO: 0097191, 217 генов) сигналами, представленными в базе данных Molecular Signatures Database. Было показано, что 7 из 57 ДЭГ до облучения и 6 из 103 ДЭГ после облучения имеют функциональные взаимодействия с генами, участвующими в сигнальном пути апоптоза, индуцированном внутриклеточными сигналами (табл. 5). Другая часть генов (12 из 57 ДЭГ до облучения и 8 из 103 ДЭГ после радиационного воздействия) имела функциональные взаимодействия на уровне белков с генами, вовлеченными в сигнальный путь апоптоза, индуцируемый внеклеточными молекулярными сигналами (табл. 5).
Таблица 5.
ДЭГ, идентифицированные в клеточной линии HeLa с нокаутом ADAMTS1, вовлеченные в сигнальные пути апоптоза, репарации двунитевых разрывов ДНК, перехода клеточного цикла из G2 в M и сигнальный путь TGFβ
| Доза | Сигнальные пути апоптоза (GO: 0072332; 0097191) |
Репарация двухцепочечных разрывов ДНК (GO: 0006302) |
G2/M переход клеточного цикла (GO: 0044839) |
Сигнальный путь TGFβ (KEGG M2642) |
|---|---|---|---|---|
| 0 Гр | HBS1L, RPL21, PRICKLE1, COPS7A, MYLIP, SQSTM1, HLA-DPA1, FGF13, IL11, ESR2, COL18A1, STK3, POT1, NKX2-1 | HLA-DPA1, POT1, PRICKLE1, MYLIP, COPS7A | CDK14, COPS7A, PRICKLE1, MYLIP, POT1, RPL21, HBS1L, ESR2, HLA-DPA1, OPTN, SQSTM1 | PRICKLE1, SQSTM1, RPL21, COPS7A, ESR2, MYLIP |
| 2 Гр | RFC3, MSH2, MSH6, ARID3A, HLA-DPA1, COL18A1, FGF13, IER3, NMU, TRPV2, SPON2, PRICKLE1 | HLA-DPA1, PRICKLE1, SETD2, ARID3A, MSH2, MSH6, RFC3 | RAP2C, HLA-DPA1, COG5, CDK14, MSH6, RFC3, PRICKLE1, ARID3A, NMU, IER3 | PRICKLE1, SRGAP2, IER3, RFC3, SPON2 |
Более того, ДЭГ, идентифицированные в клеточной линии HeLa с нокаутом ADAMTS1, по-видимому, имеют функциональные белковые взаимодействия с генами, вовлеченными в репарацию двухцепочечных разрывов ДНК (GO: 0006302, 268 генов), переход клеточного цикла из стадии G2 в M (GO: 0044839, 276 генов) и сигнальный путь TGFβ (KEGG M2642, 86 генов) (табл. 5).
Поскольку было обнаружено, что ADAMTS1 дифференциально экспрессируется в МПК индивидов с различным уровнем эндогенных фокусов γH2AX и радиационно-индуцированных микроядер, был проведен анализ с целью выявления общих ДЭГ в МПК здоровых индивидов и в клеточной линии HeLa с нокаутом ADAMTS1. Однако сравнение списков ДЭГ не выявило каких-либо генов, общих для этих групп.
ОБСУЖДЕНИЕ
В настоящем исследовании были проанализированы полнотранскриптомные профили экспрессии генов в МПК здоровых индивидов с различным уровнем эндогенных фокусов γH2AX и частотой радиационно-индуцированных микроядер до облучения и после воздействия γ-излучением. Было показано, что у индивидов с высоким уровнем фокусов белков репарации двунитевых разрывов ДНК наблюдалась повышенная экспрессия генов ENST00000424415, CRNDE и ADAMTS1 как до облучения, так и в облученных мононуклеарах периферической крови.
Функциональная связь выявленных дифференциально-экспрессирующихся генов с процессами репарации ДНК не описана в литературе и может быть связана с их участием в различных сигнальных путях. Повышенная экспрессия гена длинной некодирующей РНК, CRNDE, наблюдалась при многих типах солидных опухолей и при лейкемии [37–40]. Кроме того, с данным геном связывают устойчивость опухолевых клеток к 5-фтор-урацилу [41] и паклитакселу [42]. Повышенная экспрессия CRNDE в клетках лиц с высоким уровнем спонтанных фокусов γH2AX, наблюдаемая в настоящем исследовании, может быть связана с активацией Wnt/β-катенина или инсулин/IGF-зависимой передачи сигналов, что способствует активации апоптоза, устранению поврежденных клеток и снижению частоты микроядер [41, 43]. Данные о возможной функции длинной некодирующей РНК ENST00000424415 отсутствуют.
ADAMTS1 представляет собой ген, кодирующий металлопротеиназу внеклеточного матрикса, имеющую мембранную локализацию. В настоящем исследовании установлено, что экспрессия гена ADAMTS1 снижена в лимфоцитах лиц с низким уровнем эндогенных фокусов γH2AX и высокой частотой радиационно-индуцированных микроядер. В недавнем биоинформатическом анализе ADAMTS1 был идентифицирован как ключевой ген в развитии опухолей молочной железы [44]. По-видимому, ADAMTS1 может активировать TGFβ во внеклеточном матриксе, расщепляя его неактивную форму. Это особенно важно, учитывая, что TGFβ является важным компонентом клеточного ответа на повреждение ДНК, за счет регуляции киназной активности ATM. Известно, что нокаут TGFβ в мышиных клетках или ингибирование передачи сигналов TGFβ в клетках человека снижает активность ATM и аутофосфорилирование этой киназы, приводя к деактивации контрольных точек клеточного цикла и повышенной радиочувствительности клеток. Следовательно, высокая частота радиационно-индуцированных микроядер в лимфоцитах индивидов с низким уровнем экспрессии ADAMTS1 может быть связана с ингибированием передачи сигналов TGFβ, что в свою очередь повышает радиочувствительность клеток [45–47].
Учитывая отсутствие в литературе данных об участии выявленных дифференциально-экспрессирующихся генов в ответе клеток на повреждение ДНК, был проведен анализ влияния нокаута гена ADAMTS1 на транскрипционный профиль в модельной клеточной линии. Было показано, что нокаут ADAMTS1 приводит к дифференциальной экспрессии ряда генов, некоторые из которых функционально связаны с апоптозом, репарацией двунитевых разрывов ДНК, переходом клеточного цикла из стадии G2 в M и в сигнальном пути TGFβ. Эта гипотеза согласуется с нашими предыдущими результатами. Ранее нами сообщалось, что нокаут ADAMTS1 в клеточной линии HeLa приводил к 1.9-кратному снижению клональной выживаемости после облучения в дозе 2 Гр и повышению частоты радиационно-индуцированных микроядер с 36.0 ± 7.2 до 55.3 ± 8.3‰ (p < 0.05) по сравнению с контрольной линией клеток HeLa. Более того, мы также обнаружили, что трансфекция плазмиды ADAMTS1_pCSdest в клеточную линию HeLa с нокаутом гена ADAMTS1 приводила к снижению частоты радиационно-индуцированных микроядер с 55.3 ± 8.3 до 28.7 ± 10.3‰ (p < 0.05), что сопоставимо с частотой микроядер в контрольной клеточной линии HeLa после облучения (36.0 ± 7.2‰) [27]. Таким образом, нарушение экспрессии гена ADAMTS1 связано с нарушением транскрипционного профиля генов, связанных с ответом на повреждение ДНК и регуляцию процесса апоптоза.
Полученные результаты указывают на то, что различный уровень спонтанных и радиационно-индуцированных повреждений ДНК в клетках разных индивидов ассоциирован с дифференциальной экспрессией отдельных генов в необлученных клетках, включая ген ADAMTS1. Поэтому анализ экспрессии гена ADAMTS1 может быть интересен с точки зрения использования в качестве маркера индивидуальной радиочувствительности человека. Показанное ранее влияние нокаута ADAMTS1 на выживаемость клеток и выявленное в настоящей работе изменение транскрипционного профиля генов, участвующих в регуляции апоптоза, указывают на необходимость изучения роли ADAMTS1 в радиационно-индуцированном ответе клеток и возможности его использования в качестве мишени противоопухолевой терапии.
Исследование выполнено при финансовой поддержке РФФИ в рамках научных проектов № 14-04-31867 (эксперименты, выполненные на МПК здоровых индивидов) и 19-34-90143 (эксперименты, выполненные на клеточных линиях).
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Sokolov M., Neumann R. Global gene expression alterations as a crucial constituent of human cell response to low doses of ionizing radiation exposure // Int. J. Mol. Sci. 2015. V. 17. № 1. P. 55. https://doi.org/10.3390/ijms17010055
Foray N., Bourguignon M., Hamada N. Individual response to ionizing radiation // Mutat. Res. – Rev. Mutat. Res. 2016. V. 770. P. 369–386. https://doi.org/10.1016/j.mrrev.2016.09.001
Rajaraman P., Hauptmann M., Bouffler S., Wojcik A. Human individual radiation sensitivity and prospects for prediction // Ann. ICRP. 2018. V. 47. № 3–4. P. 126–141. https://doi.org/10.1177/0146645318764091
Andreassen C.N., Schack L.M.H., Laursen L.V., Alsner J. Radiogenomics – current status, challenges and future directions // Cancer Lett. 2016. V. 382. № 1. P. 127–136. https://doi.org/10.1016/j.canlet.2016.01.035
Wang T.M., Shen G.P., Chen M.Y. et al. Genome-wide association study of susceptibility loci for radiation-induced brain injury // J. Natl Cancer Inst. 2019. V. 111. № 6. P. 620–628. https://doi.org/10.1093/jnci/djy150
Yang D.W., Wang T.M., Zhang J.B. et al. Genome-wide association study identifies genetic susceptibility loci and pathways of radiation-induced acute oral mucositis // J. Transl. Med. 2020. V. 18. № 1. P. 1–12. https://doi.org/10.1186/s12967-020-02390-0
Zheng S., Tao W. Identification of novel transcriptome signature as a potential prognostic biomarker for anti-angiogenic therapy in glioblastoma multiforme // Cancers. 2021. V. 13. № 5. P. 1013. https://doi.org/10.3390/cancers13051013
Bhosle S.M., Huilgol N.G., Mishra K.P. Apoptotic index as predictive marker for radiosensitivity of cervical carcinoma: Evaluation of membrane fluidity, biochemical parameters and apoptosis after the first dose of fractionated radiotherapy to patients // Cancer Detect. Prev. 2005. V. 29. № 4. P. 369–375. https://doi.org/10.1016/j.cdp.2005.05.002
Azria D., Riou O., Castan F. et al. Radiation-induced CD8 T-lymphocyte apoptosis as a predictor of breast fibrosis after radiotherapy: Results of the prospective multicenter French Trial // EBioMedicine. 2015. V. 2. № 13. P. 1965–1973. https://doi.org/10.1016/j.ebiom.2015.10.024
Anderson R.M. Cytogenetic biomarkers of radiation exposure // Clin. Oncol. 2019. V. 31. № 5. P. 311–318. https://doi.org/10.1016/j.clon.2019.02.009
Bucher M., Endesfelder D., Roessler U. et al. Analysis of chromosomal aberrations and γH2A.X foci to identify radiation-sensitive ataxia-telangiectasia patients // Mutat. Res. – Genet. Toxicol. Environ. Mutagen. 2021. V. 861. P. 503301. https://doi.org/10.1016/j.mrgentox.2020.503301
Guogytė K., Plieskienė A., Ladygienė R. et al. Assessment of correlation between chromosomal radiosensitivity of peripheral blood lymphocytes after in vitro irradiation and normal tissue side effects for cancer patients undergoing radiotherapy // Genome Integr. 2017. V. 8. P. 1. https://doi.org/10.4103/2041-9414.198907
Redon C.E., Dickey J.S., Bonner W.M., Sedelnikova O.A. γ‑H2AX as a biomarker of DNA damage induced by ionizing radiation in human peripheral blood lymphocytes and artificial skin // Adv. Space Res. 2009. V. 43. № 8. P. 1171–1178. https://doi.org/10.1016/j.asr.2008.10.011
Markova E., Vasilyev S., Belyaev I. 53BP1 foci as a marker of tumor cell radiosensitivity // Neoplasma. 2015. V. 62. № 5. P. 770–776. https://doi.org/10.4149/neo_2015_092
Belyaev I.Y. Radiation-induced DNA repair foci: Spatio-temporal aspects of formation, application for assessment of radio-sensitivity and biological dosimetry // Mutat. Res. – Rev. Mutat. Res. 2010. V. 704. № 1–3. P. 132–141. https://doi.org/10.1016/j.mrrev.2010.01.011
Sedelnikova O.A., Horikawa I., Zimonjic D.B. et al. Senescing human cells and ageing mice accumulate DNA lesions with unrepairable double-strand breaks // Nat. Cell Biol. 2004. V. 6. № 2. P. 168–170. https://doi.org/10.1038/ncb1095
Han J., Hendzel M.J., Allalunis-Turner J. Quantitative analysis reveals asynchronous and more than DSB-associated histone H2AX phosphorylation after exposure to ionizing radiation // Radiat. Res. 2006. V. 165. № 3. P. 283–292. https://doi.org/10.1667/rr3516.1
Kato T.A., Okayasu R., Bedford J.S. Comparison of the induction and disappearance of DNA double strand breaks and γ-H2AX foci after irradiation of chromosomes in G1-phase or in condensed metaphase cells // Mutat. Res. – Fundam. Mol. Mech. Mutagen. 2008. V. 639. № 1–2. P. 108–112. https://doi.org/10.1016/j.mrfmmm.2007.11.006
Nakamura A.J., Redon C.E., Bonner W.M., Sedelnikova O.A. Telomere-dependent and telomere-independent origins of endogenous DNA damage in tumor cells // Aging. 2009. V. 1. P. 212–218. https://doi.org/10.18632/aging.100019
Fumagalli M., Rossiello F., Clerici M. et al. Telomeric DNA damage is irreparable and causes persistent DNA-damage-response activation // Nat. Cell Biol. 2012. V. 14. № 4. P. 355–365. https://doi.org/10.1038/ncb2466
Bernadotte A., Mikhelson V.M., Spivak I.M. Markers of cellular senescence. Telomere shortening as a marker of cellular senescence // Aging. 2016. V. 8. № 1. P. 3–11. https://doi.org/10.18632/aging.100871
Georgoulis A., Vorgias C.E., Chrousos G.P., Rogakou E.P. Genome instability and γH2AX // Int. J. Mol. Sci. 2017. № 9. V. 18. P. 1979–1989. https://doi.org/10.3390/ijms18091979
Васильев С.А., Величевская А.И., Вишневская Т.В. и др. Фоновое количество фокусов gH2AX в клетках человека как фактор индивидуальной радиочувствительности // Радиац. биология и радиоэкология. 2015. Т. 55. №. 4. С. 402–410. https://doi.org/10.7868/S0869803115040128
Martin O.A., Ivashkevich A., Choo S. et al. Statistical analysis of kinetics, distribution and co-localisation of DNA repair foci in irradiated cells: Cell cycle effect and implications for prediction of radiosensitivity // DNA Repair. 2013. V. 12. № 10. P. 844–855. https://doi.org/10.1016/j.dnarep.2013.07.002
Muller S., Neusser M., Kohler D., Cremer M. Preparation of complex DNA probe sets for 3D FISH with up to six different fluorochromes // Cold Spring Harb. Protoc. 2007. V. 2007. № 5. pdb.prot4730.https://doi.org/10.1101/pdb.prot4730
Мельников А.А., Васильев С.А., Мусабаева Л.И. и др. Индикация цитогенетических нарушений в лимфоцитах периферической крови больных злокачественными новообразованиями при лечении быстрыми нейтронами // Тюменский мед. журн. 2012. № 4. С. 76–78.
Savchenko R.R., Murashkina A.A., Fishman V.S. et al. Effect of ADAMTS1 differential expression on the radiation-induced response of HeLa cell line // Rus. J. Genet. 2021. V. 57. № 7. P. 856–862. https://doi.org/10.1134/S1022795421070127
Edie S., Zaghloul N.A., Leitch C.C. et al. Survey of human chromosome 21 gene expression effects on early development in Danio rerio // G3 (Bethesda, Md.). 2018. V. 8. № 7. P. 2215–2223. https://doi.org/10.1534/g3.118.200144
GEO accession viewer. Available online: https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE97000 (accessed on 19 May 2021).
Tilton S.C., Markillie L.M., Hays S. et al. Identification of differential gene expression patterns after acute exposure to high and low doses of low-LET ionizing radiation in a reconstituted human skin tissue // Radiat. Res. 2016. V. 186. № 5. P. 531–538. https://doi.org/10.1667/rr14471.1
Cilensek Z.M., Yehiely F., Kular R.K., Deiss L.P. A member of the GAGE family of tumor antigens is an anti-apoptotic gene that confers resistance to Fas/CD95/APO-1, interferon-gamma, taxol and gamma-irradiation // Cancer Biol. Ther. 2002. V. 1. № 4. P. 379–386. https://doi.org/10.4161/cbt.1.4.11
Herbert K., Binet R., Lambert J.P. et al. BRN2 suppresses apoptosis, reprograms DNA damage repair, and is associated with a high somatic mutation burden in melanoma // Genes Dev. 2019. V. 33. № 5–6. P. 310–332. https://doi.org/10.1101/gad.314633.118
You Y., Wen R., Pathak R. et al. Latexin sensitizes leukemogenic cells to gamma-irradiation-induced cell-cycle arrest and cell death through Rps3 pathway // Cell Death Dis. 2014. V. 5. № 10. e1493. https://doi.org/10.1038/cddis.2014.443
Tanaka Y., Imamura J., Kanai F. et al. Runx3 interacts with DNA repair protein Ku70 // Exp. Cell Res. 2007. V. 313. № 15. P. 3251–3260. https://doi.org/10.1016/j.yexcr.2007.06.012
Jiang J., Han P., Qian J. et al. Knockdown of ALPK2 blocks development and progression of renal cell carcinoma // Exp. Cell Res. 2020. V. 392. № 2. P. 112029. https://doi.org/10.1016/j.yexcr.2020.112029
Eckers J.C., Kalen A.L., Xiao W. et al. Selenoprotein P inhibits radiation-induced late reactive oxygen species accumulation and normal cell injury // Int. J. Radiat. Oncol. Biol. Phys. 2013. V. 87. № 3. P. 619–625. https://doi.org/10.1016/j.ijrobp.2013.06.2063
Graham L.D., Pedersen S.K., Brown G.S. et al. Colorectal neoplasia differentially expressed (CRNDE), a novel gene with elevated expression in colorectal adenomas and adenocarcinomas // Genes Cancer. 2011. V. 2. № 8. P. 829–840. https://doi.org/10.1177/1947601911431081
Ellis B.C., Molloy P.L., Graham L.D. CRNDE: A long non-coding RNA involved in CanceR, neurobiology, and development // Front. Genet. 2012. V. 3. P. 270. https://doi.org/10.3389/fgene.2012.00270
Zhang X., Sun S., Pu J.K.S. et al. Long non-coding RNA expression profiles predict clinical phenotypes in glioma // Neurobiol. Dis. 2012. V. 48. № 1. P. 1–8. https://doi.org/10.1016/j.nbd.2012.06.004
Szafron L.M., Balcerak A., Grzybowska E.A. et al. The novel gene CRNDE encodes a nuclear peptide (CRNDEP) which is overexpressed in highly proliferating tissues // PLoS One. 2015. V. 10. № 5. P. e0127475. https://doi.org/10.1371/journal.pone.0127475
Han P., Li J.W., Zhang B.M. et al. The lncRNA CRNDE promotes colorectal cancer cell proliferation and chemoresistance via miR-181a-5p-mediated regulation of Wnt/β-catenin signaling // Mol. Cancer. 2017. V. 16. № 1. P. 1–13. https://doi.org/10.1186/s12943-017-0583-1
Liu C., Hou J., Shan F. et al. Long non-coding RNA CRNDE promotes colorectal carcinoma cell pro-gression and paclitaxel resistance by regulating miR-126-5p/ATAD2 axis // OncoTargets Ther. 2020. V. 13. P. 4931–4942. https://doi.org/10.2147/OTT.S237580
Ellis B.C., Graham L.D., Molloy P.L. CRNDE, a long non-coding RNA responsive to insulin/IGF signaling, regulates genes involved in central metabolism // Biochim. Biophys. Acta (BBA) – Mol. Cell Res. 2014. V. 1843. № 2. P. 372–386. https://doi.org/10.1016/j.bbamcr.2013.10.016
Wu D., Han B., Guo L., Fan Z. Molecular mechanisms associated with breast cancer based on integrated gene expression profiling by bioinformatics analysis // J. Obstet. Gynaecol. 2016. V. 36. № 5. P. 615–621. https://doi.org/10.3109/01443615.2015.1127902
Ewan K.B., Henshall-Powell R.L., Ravani S.A. et al. Transforming growth factor-B1 mediates cellular response to DNA damage in situ // Cancer Res. 2002. V. 62. № 20. P. 5627–5631.
Kirshner J., Jobling M.F., Pajares M.J. et al. Inhibition of transforming growth factor-β1 signaling attenuates ataxia telangiectasia mutated activity in response to genotoxic stress // Cancer Res. 2006. V. 66. № 22. P. 10861–10869. https://doi.org/10.1158/0008-5472.can-06-2565
Wiegman E.M., Blaese M.A., Loeffler H. et al. TGFβ-1 dependent fast stimulation of ATM and p53 phosphorylation following exposure to ionizing radiation does not involve TGFβ-receptor I signaling // Radiother. Oncol. 2007. V. 83. № 3. P. 289–295. https://doi.org/10.1016/j.radonc.2007.05.013
Дополнительные материалы отсутствуют.


