Генетика, 2022, T. 58, № 12, стр. 1400-1417
Анализ генов, определяющих компактную форму растений арбуза Citrullus lanatus (Thunb.) Matsum. & Nakai, у образцов из коллекции ВИР
К. В. Стрыгина 1, *, А. Г. Елацкова 1, Ю. А. Елацков 1, Г. А. Теханович 1, Е. К. Хлесткина 1
1 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
* E-mail: k.strygina@vir.nw.ru
Поступила в редакцию 04.12.2021
После доработки 15.03.2022
Принята к публикации 12.04.2022
- EDN: IVWFXO
- DOI: 10.31857/S001667582212013X
Аннотация
Компактная форма растений арбуза, определяемая признаками кустовости и короткоплетистости, имеет важное экономическое значение, определяя рациональное использование посевных площадей за счeт высокой плотности посадки и возможность механизированной обработки и уборки. В связи с этим изучение генов, определяющих малый габитус растений арбуза, является важной задачей для ускорения селекции этой культуры. Цель данной работы – изучение известных и поиск новых генов компактности растений арбуза Citrullus lanatus (Thunb.) Matsum. & Nakai на основе анализа уникальной коллекции бахчевых культур ВИР им. Н.И. Вавилова и селекционных форм селекции Кубанской опытной станции ВИР. В результате у всех кустовых и ультракустовых генотипов, кроме двух образцов из Азербайджана, выявлена известная мутация гена dw-1 (кодирует ABC-транспортeр). Компактная форма этих двух образцов, как удалось установить, не связана и с другими известными генами, dsh (кодирует гиббереллин-20-оксидазу) и df (кодирует гиббереллин-3β-гидроксилазу), или их копиями, df2 и dsh2, выявленными в настоящем исследовании. Таким образом, в коллекции бахчевых культур ВИР содержатся источники потенциально новых генов, определяющих кустовую форму арбуза.
Бахчевые культуры (семейство Cucurbitaceae Juss.) известны своими крупными, отличающимися по окраске и морфологии плодами. Наиболее экономически важные культуры данного семейства – кабачок, тыква (Cucurbita L.), огурец, дыня (Cucumis L.) и арбуз (Citrullus Schrad. ex Eckl. & Zeyh.). Представители пяти дополнительных родов (Momordica L., Lagenaria Ser., Benincasa Savi, Luffa Mill. и Trichosanthes L.) являются второстепенными культурами с глобальной точки зрения, важными для мелкого фермерского производства в Восточной, Южной и Юго-Восточной Азии.
Компактность растений бахчевых культур так же как и короткостебельность многих других культур (зерновых и др.) – важные признаки, благодаря которым можно повышать урожайность на единицу площади. Компактные кустовые или короткоплетистые растения обеспечивают более высокую плотность посадки [1]. Короткостебельные формы зерновых культур обеспечивают более высокую урожайность за счет устойчивости к полеганию благодаря так называемым генам “зеленой революции”: semi-dwarf 1 (sd1) у риса Oryza sativa L. и Reduced height-1 (Rht-D1 и Rht-B1) у пшеницы Triticum aestivum L. [2, 3]. Короткостебельность/короткоплетистость связана с мутациями в генах биосинтеза растительных гормонов (или с генами, связанными с ответом на них), которые регулируют удлинение и/или деление клеток [3–7].
К сегодняшнему моменту в семействе Cucurbitaceae идентифицирован ряд генов, обусловливающих компактную форму растений. Некоторые из этих генов были картированы и расшифрованы. У тыквы мускатной C. moschata Duchesne и тыквы твердокорой C. pepo L. кустовость контролируется неохарактеризованными генами Bu [8–12]. У тыквы крупноплодной C. maxima Duchesne ex Lam. известны три QTL в группах сцепления 1, 3 и 4, отвечающие за формирование компактного растения. В основном из этих трех локусов, qCmB2, в качестве гена-кандидата предложен ген, кодирующий гиббереллин-20-оксидазу, Cma_004516 [13].
У дыни C. melo L. известно четыре основных гена, определяющих компактную форму: si-1, si-2, si-3 и mdw1 (slb) [11, 14–17]. Длина междоузлий у растений si-2 и si-3 короче, чем у мутантов si-1, но растения менее компактны. У растений si-2 короткими являются только первые междоузлия. Длина междоузлий у мутантов si-3 короче, чем у растений дикого типа, на всех стадиях развития. Гены si пока еще не картированы, их продукты не описаны. Ген mdw1 (slb) картирован в хромосоме 7 в участке, содержащем ряд генов, включая ген цитокининоксидазы (CKX) – возможный кандидат для mdw1 [14].
У огурца C. sativus L. известно шесть генов, отвечающих за компактную форму взрослого растения: dwarf (Csdw), compact (cp), cp-1, supercompact-1 (scp-1), scp-2 и short internode (si) [18–24]. Ген сp был картирован в дистальной части хромосомы 4, в которой содержится ген цитокининоксидазы CKX, являющийся кандидатом. Ген Cullin 1 является кандидатом для cp-1, а scp-1 и scp-2, как выяснилось, кодируют цитохром P450 (CsCYP85A1) и стероид 5-альфа-редуктазу (CsDET2); эти три гена участвуют в пути биосинтеза брассиностероидов [19, 20, 25, 26]. Кроме того, было установлено, что усеченный белок гена CsaVBF1 (семейства F-box) связан с мутацией si огурца, а CLAVATA1-подобный ген, CsCLAVATA1, считается кандидатом для локуса Csdw [23, 24].
В геноме арбуза C. lanatus (Thunb.) Matsum. & Nakai известно пять генов dwarf (dw), из которых гены dw-1 и dw-1s являются аллельными вариантами, а dw-2, dw-3 и предположительно dw-4 – независимыми [27–30]. Отмечается, что короткоплетистость у арбуза наследуется как моногенный рецессивный признак и контролируется геном short vine (shv) [29]. В 2018 г. благодаря естественной мутации в линии “I911” у арбуза был выявлен еще один ген dsh, который проявился в образце с мелкими плодами и короткими междоузлиями. Ген dsh был картирован в хромосоме 7, в качестве кандидата был обозначен ген Cla010726, кодирующий продукт, сходный с ферментом гиббереллин-20-оксидазой [31, 32]. В 2019 г. ген dw-1 был картирован в хромосоме 9. Среди шести генов, находящихся в кандидатной области, один ген Cla010337, кодирующий АТФ-связывающий кассетный транспортер, был обозначен как ген-кандидат [33, 34]. Кроме того, изучение 165 контрастных по архитектонике форм показало, что ген dw-1 являлся основной причиной компактности растений у образцов арбуза разного происхождения [33]. Компактность связана с однонуклеотидной делецией, поэтому ген dw-1 – хорошая мишень для геномного редактирования с целью получения компактных форм арбуза. Также независимыми группами исследователей в хромосоме 9 был выявлен ген Cla015407, обозначенный как df (Cldf), кодирующий гиббереллин-3β-гидроксилазу [35–38]. Влияние данных мутаций на урожайность сортов арбуза и на качество продукции не исследовалось. Таким образом, благодаря успехам в секвенировании генома арбуза можно ожидать открытий новых генов, обусловливающих изменение растения с плетистой формы на кустовую, кроме упомянутых трех.
Цель настоящей работы – анализ известных генов компактности растений арбуза C. lanatus dw-1, df и dsh и поиск новых на основе анализа уникальной коллекции бахчевых культур ВИР им. Н.И. Вавилова и селекционных форм селекции Кубанской опытной станции ВИР.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал, выделение ДНК и РНК, синтез кДНК
Объектом исследования были растения C. lanatus коллекции бахчевых культур ВИР им. Н.И. Вавилова и гибриды, полученные на Кубанской ОС ВИР (табл. 1). Выращивание растений для выделения ДНК проводилось в полевых условиях Кубанской ОС ВИР в 2020 г. Суммарная ДНК выделялась из зеленых листьев взрослых растений с использованием набора DNeasy Plant Mini Kit (QIAGEN) согласно методике производителя. Для выделения РНК семена арбуза проращивались при 12-часовом световом дне при 20°C на светоустановке. Выделение РНК из стеблей проводилось через 63 дня после посева семян в грунт в трех биологических повторностях (по два растения на повторность). РНК была выделена с использованием набора RNeasy Mini Kit (QIAGEN) и обработана RNase-free DNase set (QIAGEN). По матрице РНК была синтезирована одноцепочечная кДНК с использованием ОТ-M-MuLV-RH (Биолабмикс) в 20 мкл реакционной смеси, содержащей 1 мг РНК-матрицы. Все процедуры выполнялись согласно протоколам производителей используемых наборов.
Таблица 1.
Список образцов арбуза коллекции ВИР, использованных в настоящем исследовании. Образцы, выделенные серым фоном, использовались для изучения уровня экспрессии генов
| № п/п | Каталожный номер | Происхождение | Название образца | Фенотип | |
|---|---|---|---|---|---|
| стебель | цвет листьев | ||||
| 1 | K-4128 | США | Charleston Gray 133 | Плетистый | Зеленый |
| 2 | K-5382 | Россия | Ранний Кубани | Короткоплетистый | » |
| 3 | K-2578 | » | Красавчик | Плетистый | » |
| 4 | K-5090 | » | Ольгинский | » | » |
| 5 | K-5426 | » | Благодатный | » | » |
| 6 | K-5429 | » | Сюрприз | Короткоплетистый | » |
| 7 | – | » | Лидер | » | » |
| 8 | K-5427 | » | Любимчик | Плетистый | » |
| 9 | K-5428 | » | Святослав | Кустовой | » |
| 10 | K-5430 | » | Родник | Плетистый | » |
| 11 | K-5402 | » | Подарок Солнца | Короткоплетистый | Желто-зеленый мозаичный |
| 12 | K-5425 | » | Черный Принц | Плетистый | Зеленый |
| 13 | – | » | Адам | » | » |
| 14 | – | » | Солярис | » | Желто-зеленый мозаичный |
| 15 | K-3414 | Украина | Мелитопольский 60 | » | Зеленый |
| 16 | K-5136 | США | Royal Jubely | » | Желто-зеленый мозаичный |
| 17 | K-4857 | КОС ВИР | Целебный | » | Зеленый |
| 18 | K-137 | США | Klekley’s Sweet | » | » |
| 19 | – | КОС ВИР | ЦЛ 552 | » | » |
| 20 | – | » | ЦЛ 752 | » | » |
| 21 | – | » | КРЛ 694 | Кустовой | » |
| 22 | – | » | ЦЛ 638/14 | Плетистый | » |
| 23 | – | » | КРЛ 730 | Короткоплетистый | Желто-зеленый мозаичный |
| 24 | – | » | F12 ЦЛ 752 × Ольгинский | Кустовой | Зеленый |
| 25 | – | » | » | Кустовой | » |
| 26 | – | » | » | Плетистый | » |
| 27 | – | » | » | » | » |
| 28 | – | » | » | » | » |
| 29 | – | » | » | Короткоплетистый | » |
| 30 | – | » | » | Кустовой | » |
| 31 | – | » | » | » | » |
| 32 | – | » | » | » | » |
| 33 | – | » | F7 ЦЛ 752 × Ольгинский | Плетистый | » |
| 34 | – | » | F9 ЦЛ 752 × Ольгинский | » | » |
| 35 | – | » | КРЛ 694 | Короткоплетистый | » |
| 36 | – | » | F12 ЦЛ 752 × Волжанин | Плетистый | » |
| 37 | – | » | » | » | » |
| 38 | – | » | F9 ЦЛ 752 × Ольгинский | » | Желто-зеленый мозаичный |
| 39 | – | КОС ВИР | F13 Святослав × Подарок Солнца | Кустовой | Желто-зеленыймозаичный |
| 40 | K-3848 | США | Congo | Плетистый | Зеленый |
| 41 | » | » | » | » | » |
| 42 | K-5373 | » | Sweet Treat | Ультракустовой | » |
| 43 | » | » | » | Плетистый | » |
| 44 | » | » | » | Ультракустовой | Зеленоватый |
| 45 | » | » | » | Плетистый | Зеленый |
| 46 | K-4039 | Азербайджан | Местный | Короткоплетистый | Зеленоватый |
| 47 | » | » | » | Кустовой | Зеленый |
| 48 | » | » | » | Короткоплетистый | » |
| 49 | » | » | » | Кустовой | » |
| 50 | K-5131 | США | Bush Charleston Gray | » | Зеленоватый |
| 51 | – | КОС ВИР | F12 ЦЛ 752 × Ольгинский | » | Зеленый |
Идентификация и анализ копий генов dw-1, df, dsh
Идентификация генов, гомологичных известным генам арбуза dw-1 (Cla010337), df (Cla015407) и dsh (Cla010726), проводилась в базе данных геномов Cucurbitaceae (http://cucurbitgenomics.org/) при использовании алгоритма BLASTN. Выявленные гены представлены в табл. 2. Структура неаннотированных генов предсказывалась при использовании программы FGENESH+. Аннотация функциональных доменов осуществлялась с помощью InterPro. Построение модели белка dsh осуществлялось в программе SWISS-MODEL, в качестве модели использовался белок 6lsv.1 (Crystal structure of JOX2 in complex with 2OG, Fe, and JA). Для построения множественного выравнивания использовалась программа Multalin. Филогенетический анализ осуществлялся в программе MEGA X при использовании метода Maximum likelihood на основании кодирующих последовательностей генов с бутстреп-поддержкой 1000; на полученных изображениях показаны данные значений бутстрэпа ≥70%.
Таблица 2.
Список гомологичных последовательностей генов, выявленных в исследовании на основании последовательностей генов dw-1, df и dsh арбуза, в базе данных геномов Cucurbitaceae (http://cucurbitgenomics.org/). Серым фоном обозначены аннотированные в настоящем исследовании последовательности
| Ген C. lanatus | Вид | Название образца | Последовательность | Локализация в геноме | Описание |
|---|---|---|---|---|---|
| dw-1(Cla010337,Cla97C09G179710) | C. lanatus | 97103 (v2) | Cla97C09G179710 | Cla97Chr09:33248469..33254809 (+) | ABC transporter B family member 19 |
| Charleston Gray | ClCG09G018320 | CG_Chr09:35331134..35338351 (+) | » | ||
| L. siceraria | USVL1VR-Ls | Lsi02G014910 | Chr02:20679307..20686179 (+) | Multidrug resistance ABC transporter ATP-binding and permease protein | |
| B. hispida | Wax gourd | Bhi06G000344 | Chr6:8912006..8918888 (+) | ABC transporter B family protein | |
| C. sativus | Chinese Long (v3) | CsaV3_5G037960 | Chr5:30108267..30115095 (–) | » | |
| Gy14 (v2) | CsGy5G028210 | Chr5:31935937..31942837 (–) | ABC transporter B family member 19 | ||
| PI 183967 | CSPI05G28850 | Chr5:27240346..27247058 (–) | Lipid A export ATP-binding/permease protein msba | ||
| C. melo | DHL92 (v3.6.1) | MELO3C021982 | Chr09:2244766..2251632 (+) | ABC transporter B family protein | |
| MELO3C026136 | Chr03:18058108..18058505 (–) | » | |||
| C. maxima | Rimu | CmaCh15G002770 | Cma_Chr15:1315054..1321049 (+) | ATP-binding cassette transporter | |
| CmaCh04G027610 | Cma_Chr04:18563539..18569742 (–) | Lipid A export ATP-binding/permease protein msba | |||
| C. moschata | Rifu | CmoCh04G028900 | Cmo_Chr04:20570027..20575914 (–) | » | |
| CmoCh15G002920 | Cmo_Chr15:1372725..1379019 (+) | Multidrug resistance ABC transporter ATP-binding and permease protein | |||
| C. pepo | Zucchini | Cp4.1LG13g08370 | Cp4.1LG13:7948987..7956776 (–) | Lipid A export ATP-binding/permease protein msba | |
| Cp4.1LG01g22940 | Cp4.1LG01:19956958..19964623 (–) | ABC transporter B family protein | |||
| dw-1(Cla010337,Cla97C09G179710) | C. argyrosperma | Silver-seed gourd | Carg04387 | Cucurbita_argyrosperma_scaffold_007 | ABC transporter B family protein |
| Carg02255 | Cucurbita_argyrosperma_scaffold_032 | ABC transporter B family member 19 | |||
| df(Cla015407,Cla97C09G164590) | C. lanatus | 97103 (v2) | Cla97C09G164590 | Cla97Chr09:2119445..2120701 (+) | Gibberellin 3-beta-dioxygenase 1-like |
| Cla97C09G164580 | Cla97Chr09:2088858..2090591 (+) | » | |||
| Charleston Gray | ClCG09G002420 | CG_Chr09:2217620..2219353 (+) | Gibberellin 3-beta L-hydroxylase | ||
| ClCG09G002450 | CG_Chr09:2251701..2253314 (+) | » | |||
| L. siceraria | USVL1VR-Ls | Lsi02G027550 | Chr02:33766399..33768094 (–) | » | |
| Lsi02G027560 | Chr02:33802265..33805308 (–) | Gibberellin 3-beta-hydroxylase | |||
| B. hispida | Wax gourd | Bhi09G000068 | Chr9:1592538..1594505 (–) | Gibberellin 3-beta-dioxygenase 1-like | |
| Bhi09G000070 | Chr9:1647297..1647887 (–) | Gibberellin 3-beta-dioxygenase 2-1 | |||
| Bhi09G000067 | Chr9:1579477..1580813 (+) | Gibberellin 3-beta-dioxygenase | |||
| Bhi09G000069 (в Bhi09G000068) |
Chr9:1593660..1594004 (+) | Unknown protein | |||
| C. sativus | Chinese Long (v3) | CsaV3_7G032870 (NCBI FR720085) |
Chr7:20688063..20689463 (–) | Gibberellin 3-beta-dioxygenase 1-like | |
| CsaV3_7G032900 (NCBI NM_001280696) |
Chr7:20713927..20715207 (–) | » | |||
| CsaV3_7G032880 (NCBI MK433203) |
Chr7:20705253..20706866 (–) | » | |||
| Gy14 (v2) | CsGy7G019280 | Chr7:21903314..21904735 (–) | » | ||
| CsGy7G019320 | Chr7:21929206..21930486 (–) | » | |||
| CsGy7G019290 | Chr7:21920567..21922180 (–) | » | |||
| df(Cla015407,Cla97C09G164590) | C. sativus | PI 183967(wild cucumber) | CSPI07G21030 | Chr7:18010033..18011300 (–) | Gibberellin 3-beta-hydroxylase |
| CSPI07G21040 | Chr7:18027267..18037094 (–) | » | |||
| C. melo | DHL92 (v3.6.1) | MELO3C023378 | Chr01:34777271..34778731 (–) | Gibberellin 3-beta-dioxygenase 1-like | |
| MELO3C023375 | Chr01:34813832..34815725 (–) | » | |||
| – | Region in chr01 34803145 to 34802534 |
– | |||
| C. maxima | Rimu | CmaCh08G006320 (NCBI AJ302040) |
Cma_Chr08:3573502..3574908 (+) | Gibberellin 3-beta-hydroxylase | |
| CmaCh17G008310 (NCBI AJ006453) |
Cma_Chr17:6527065..6528927 (+) | » | |||
| C. moschata | Rifu | CmoCh08G006170 | Cmo_Chr08:3778589..3779817 (+) | » | |
| CmoCh17G008010 | Cmo_Chr17:7674873..7676851 (+) | » | |||
| C. pepo | Zucchini | Cp4.1LG17g08980 | Cp4.1LG17:4952161..4953544 (–) | » | |
| Cp4.1LG12g07890 | Cp4.1LG12:6749201..6751027 (+) | » | |||
| C. argyrosperma | Silver-seed gourd | Carg23157 | Cucurbita_argyrosperma_scaffold_194:202055..203342 (+) | Gibberellin 3-oxidase | |
| – | Region in Cucurbita_argyrosperma_scaffold_029 833417 to 833954 |
– | |||
| dsh(Cla010726,Cla97C07G143880) | C. lanatus | 97103 (v2) | Cla97C07G143880 | Cla97Chr07:31385311..31387981 (–) | Gibberellin 2-beta-dioxygenase 8 |
| Cla97C09G170450 | Cla97Chr09:6844481..6846371 (+) | » | |||
| Charleston Gray | ClCG07G016880 | CG_Chr07:33412201..33414994 (–) | 2-Oxoglutarate (2OG) and Fe(II)-dependent oxygenase superfamily protein LENGTH=324 | ||
| ClCG09G007850 | CG_Chr09:7173838..7178119 (+) | » | |||
| L. siceraria | USVL1VR-Ls | Lsi07G001050 | Chr07:1191928..1194575 (–) | 2-Oxoglutarate (2OG) and Fe(II)-dependent oxygenase superfamily protein | |
| B. hispida | Wax gourd | Bhi01G002032 | Chr1:64479937..64484521 (–) | Gibberellin 2-beta-dioxygenase | |
| BhiUN281G14 | Contig281:522408..525386 (+) | Gibberellin 3-beta-dioxygenase | |||
| Bhi09G000978 | Chr9:28762801..28764982 (+) | Gibberellin 2-beta-dioxygenase 1 | |||
| Bhi09G000975 | Chr9:28741111..28741777 (+) | » | |||
| C. sativus | Chinese Long (v3) | CsaV3_4G004090 | Chr4:2517519..2519186 (–) | Gibberellin 2-beta-dioxygenase 8 | |
| Gy14 (v2) | CsGy4G003950 | Chr4:2515408..2517091 (–) | Gibberellin 2-beta-dioxygenase 8 | ||
| PI 183967 | CSPI04G04030 | Chr4:2554662..2556152 (–) | 2-Oxoglutarate (2OG) and Fe(II)-dependent oxygenase superfamily protein | ||
| C. melo | DHL92 (v3.6.1) | MELO3C032832 | Chr07:26056883..26058365 (+) | Gibberellin 2-beta-dioxygenase 8 | |
| C. maxima | Rimu | – | – | – | |
| C. moschata | Rifu | – | – | – | |
| C. pepo | Zucchini | Cp4.1LG12g10360 | Cp4.1LG12:9440006..9445293 (–) | 2-Oxoglutarate (2OG) and Fe(II)-dependent oxygenase superfamily protein | |
| C. argyrosperma | Silver-seed gourd | – | – | – |
Дизайн праймеров, ПЦР, количественная ПЦР
Дизайн праймеров был выполнен с использованием Oligo Primer Analysis Software v.7 (табл. 3). Амплификация ДНК проводилась в 20 мкл ПЦР-смеси, содержащей 50 нг ДНК-матрицы, по 10 пг каждого праймера, 0.25 мМ каждого dNTP, 1× реакционного буфера, 15 мМ MgCl2 и 1 U Taq-полимеразы (Синтол), при следующих условиях: 95°С 1.5 мин; 35 циклов 95°С 30 с, 50–60°С 30 с, 72°С 1 мин; 72°С 5 мин. Количественная ПЦР была проведена на кДНК в реакционной смеси объемом 15 мкл при использовании набора реагентов SYBR Green I (Синтол) и системы CFX96 Touch (Bio-Rad) при следующих условиях: 50°С 10 мин; 95°С 5 мин; 50 циклов 95°С 10 с, 60°С 30 с. Показания для построения кривых плавления продуктов ПЦР снимались при 95°С 10 с; 65°С 5 с; 95°С 50 с. Графики зависимости порогового цикла от исходной концентрации матриц строились на основе трех последовательных 3-кратных разведений. Для стандартизации количества кДНК матрицы проводилась количественная ОТ-ПЦР с праймерами к референсному гену C. lanatus Clathrin adaptor complex subunit (ClCAC) [39]. Каждый образец запускался в трех технических повторностях. Различия между генотипами были проверены с помощью T-теста, при котором значение p ≤ 0.05 считалось значимым.
Таблица 3.
Последовательности ПЦР-праймеров, использованных в настоящей работе для анализа генов карликовости арбуза
| Ген | Прямой праймер (5′ → 3′) | Обратный праймер (5′ → 3′) | Цель | Ссылка |
|---|---|---|---|---|
| dw-1 | TGATGCTCGCAGGTAAAATA | ATCCCACTTCATTCCTCAAA | Маркер Indel1 | [34] |
| TATGCTTATACTCTCACTGGAATT | TTAACATTGCAGCCAAAAATAG | Маркер dCAPS3 | [34] | |
| TTGTTGTCTTGGTTGTTACTG | AATTGATTCACCACCTCTAATG | Количественная ОТ-ПЦР | [34] | |
| df | ATGGGAAGCATCAAAATAACCGA | CCTTCGGACCACATACGTTTAG | Секвенирование | – |
| ACCCGATTCTTTCCCTTCTTC | CAGCCTTCCACATAGACTCTTC | » | – | |
| GCCTCACGACTACGTTCAATA | GGTAAAGCGGTGGTTGAGTT | » | – | |
| GAGTCTATGTGGAAGGCTGATG | TTAACCTACTTTAACCTGGCTGT | » | – | |
| GCCTCACGACTACGTTCAATA | CAGCCTTCCACATAGACTCTTC | Количественная ОТ-ПЦР | – | |
| dsh | ATGTCAGAAGAATCATCGATTGA | TAAACCCTAAGTCGGAGGCA | Секвенирование | – |
| CTACCAGTGATGGACATGGATTAT | GTGCTTTCCAAACGCTTCTATT | » | – | |
| CGACTTAGGGTTTACGGAACAA | CCTCGTATTTCCTTCTCCAACA | » | – | |
| CCTCGAAACTCCAACGAAGAA | TCATTCTACAATTCCGGCAAATTC | » | – | |
| CGACTTAGGGTTTACGGAAC | GCTCTCAAAATTATCTCCCA | Количественная ОТ-ПЦР | [32] | |
| df2 | ATGTCATCAAAAGAATGGATAGATG | ACCACTCTACCCTCCAAACT | Секвенирование | – |
| CGACCGACGACTGTTTACTT | ACCACTCTACCCTCCAAACT | » | – | |
| GCCCAAACTTCTCTGCTTCT | CTCCAGTACAACAAATAGGGT | » | – | |
| GGTAGAGTGGTCAGATTGAATTA | ATTGCTCCAACCCTCCATAAA | » | – | |
| GAGGCGAGTGGTTTGATGTA | TTATATTCCAGTAAAATCTTTAAGTGTG | » | – | |
| CCCAACTCAACCACCACTTTA | CTCCAGTACAACAAATAGGGT | Количественная ОТ-ПЦР | – | |
| dsh2 | ATGGCCACCATCCCCAAAATG | CTTCAGACCACATACGCTTAG | Секвенирование | – |
| CTCCACATTCCCGACAACAA | CCTTCCACATAGACTCCTTAAC | » | – | |
| GAGTCTATGTGGAAGGCTGATG | TTAGCCTACTTTCACCTGGCTATG | » | – | |
| GAGACGAAGCGGAAACTGAA | TGAGCAATAGCAGAAGCAGAG | Количественная ОТ-ПЦР | – |
Гидролиз продуктов ПЦР, электрофорез, секвенирование
Гидролиз продуктов ПЦР был проведен эндонуклеазой рестрикции EcoRI (Сибэнзим). Электрофорез проводился в агарозном геле, окрашенном бромистым этидием. Изображение геля получалось с помощью прибора Molecular Imager Gel Doc XR System (BioRad) с использованием УФ-излучения. Выделение ДНК из геля проводили с помощью набора для очистки ДНК diaGene (Диаэм). Выделенные фрагменты ДНК секвенировали с использованием реактива BigDye™ Terminator v3.1 Cycle Sequencing Kit (Thermo Fisher Scientific) и генетического анализатора 3500 (Applied Biosystems). Результаты секвенирования обрабатывались в программе UGENE. Полученные последовательности были размещены в GenBank (NCBI) под номерами OM401277–OM401308.
РЕЗУЛЬТАТЫ
Анализ аллельного состояния генов dw-1, df и dsh
Zhu et al. [33] продемонстрировали, что нефункциональный аллель гена dw-1 является наиболее частой причиной появления карликового фенотипа арбуза. Авторы также предложили два различных типа молекулярных маркеров, направленных на идентификацию мутантного и аллеля дикого типа: 1) маркер Indel1, фланкирующий делецию в интроне гена, ассоциированную с карликовым фенотипом, и 2) маркер dCAPS3, основанный на амплификации и гидролизе эндонуклеазой EcoRI несинонимичной замены в кодирующей части гена [33]. Для анализа аллельного состояния гена dw-1 в сформированной выборке из коллекции ВИР, которая была представлена 12 кустовыми, двумя ультракустовыми, девятью короткоплетистыми и 28 плетистыми образцами (табл. 1), мы использовали оба типа молекулярных маркеров.
В результате нами было замечено, что маркеры Indel1 дали ложноположительный результат с плетистым образцом № 14 (сорт Солярис) (рис. 1). Остальные результаты анализа с обоими маркерами подтверждали друг друга. Мутантный аллель гена dw-1 был выявлен у двух ультракустовых и десяти кустовых фенотипов (рис. 1). Оставшиеся два кустовых фенотипа № 47 и 49, представленных образцом Азербайджан местный (K-4039), вероятно, являются мутантами по другому гену.
Рис. 1.
Анализ аллельного полиморфизма гена dw-1 среди образцов арбуза коллекции ВИР с маркерами Indel1 и dCAPS3. Список образцов приведен в табл. 1. Ожидаемая длина фрагментов при скрининге с маркером Indel1: нормальный аллель 171 пн, мутантный аллель 166 пн. Ожидаемая длина фрагментов при скрининге с маркером dCAPS3: нормальный аллель 121 пн, мутантный аллель 91 + 29 пн. Красным цветом обозначены номера карликовых образцов – мутантов по dw-1, синим цветом обозначен ложноположительный результат при скрининге с маркером Indel1, черными стрелками обозначены ультракустовые образцы, красными стрелками обозначены кустовые образцы – не мутанты по dw-1.

Кустовые фенотипы № 47 и 49 (образец Азербайджан местный, K-4039) и родственные им короткоплетистые № 46 и 48 были взяты для анализа других генов карликовости df и dsh. В данную выборку также попали ультракустовые формы № 42 и 44 и родственные им плетистые № 43 и 45 (сорт Sweet Treat, K-5373), поскольку по нашему предположению данные образцы являются мутантами не только по гену dw-1, но и по другому гену, в результате чего у них проявился такой фенотип (табл. 1).
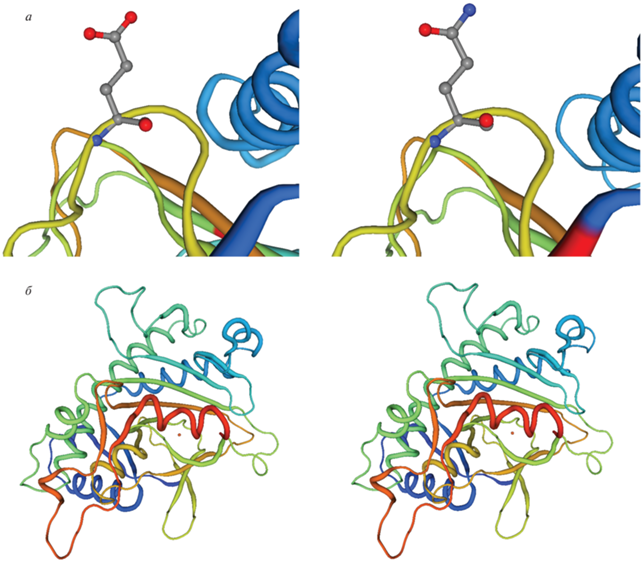
В результате секвенирования кодирующей части гена df и интрона мутаций не было выявлено ни в одном анализируемом образце. В гене dsh нам удалось обнаружить одну несинонимичную замену во втором экзоне во всех образцах по сравнению с референсным сортом Charleston Gray, приводящую к замене глутаминовой кислоты (E) на глутамин (Q) (рис. 2) в положении 223. Однако каких-то существенных изменений в структуре белка эта замена не вызвала. Остальные выявленные мутации являлись однонуклеотидными инсерциями и делециями в интроне гена, не приводящими к изменению предсказанного белкового продукта. Таким образом, необходимо было расширить поиск генов-кандидатов для потенциально нового гена кустовости, характерного для K-4039.
Рис. 2.
Предсказанная структура белка dsh. а – изменение аминокислотного остатка глутаминовая кислота (E) на глутамин (Q) в положении 223 у проанализированных образцов арбуза по сравнению с референсным сортом Charleston Gray, б – сохранение предсказанной структуры белка dsh у проанализированных образцов арбуза по сравнению с референсным сортом Charleston Gray. Список образцов приведен в тексте статьи.
Поиск потенциальных новых генов-кандидатов, отвечающих за формирование кустового фенотипа арбуза
Далее нами был проведен поиск ортологичных и паралогичных копий генов dw-1, df и dsh в геномах представителей семейства Cucurbitaceae, секвенированных к настоящему моменту.
dw-1. В результате проведенного анализа для гена dw-1 было установлено, что в семействе Cucurbitaceae у образцов, принадлежащих к трибе Cucurbiteae (тыква крупноплодная C. maxima, тыква мускатная C. moschata), а также у дыни C. melo имеется дополнительная копия данного гена, а у тыквы обыкновенной C. pepo и тыквы серебристосемянной C. argyrosperma K. Koch – две копии гена dw-1 (табл. 2). На основании выявленных последовательностей был проведен филогенетический анализ, демонстрирующий, что дупликация dw-1 произошла у общего предка Cucurbiteae (рис. 3, синий цвет). Дополнительные копии гена dw-1 в геноме C. melo, C. pepo и C. argyrosperma образовались в результате независимых актов дупликации. У арбуза дополнительная копия гена не была обнаружена.
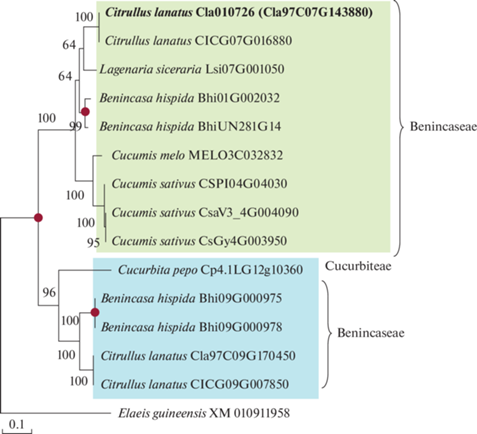
Рис. 3.
Анализ филогенетического сходства генов dw-1 (CDS). Жирным шрифтом выделен ген dw-1 арбуза. Зеленым и синим фоном выделены разные группы генов. Красными кругами обозначены выявленные акты дупликации генов.
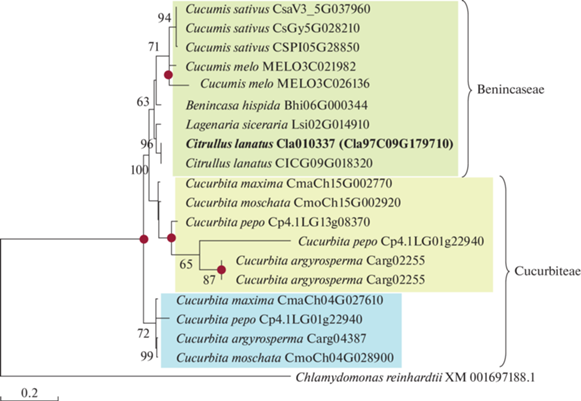
df. Нам удалось выявить в геномах представителей Cucurbitaceae от двух до трех копий гена df, причем данные дупликации происходили на разных этапах эволюции семейства (табл. 2, рис. 4). Группа 1, в которую входит ген df (рис. 4, зеленый фон), представлена одним геном у всех изученных видов, кроме восковой тыквы B. hispida (Thunb.) Cogn. В ее геноме произошла дополнительная дупликация. Кроме того, при поиске гомологичных последовательностей в геноме B. hispida удалось также обнаружить четвертую аннотированную последовательность Bhi09G000068, которая лежит внутри Bhi09G000069 (табл. 2).
Рис. 4.
Анализ филогенетического сходства генов df (CDS). Жирным шрифтом выделен ген df арбуза. Красным, зеленым и синим фоном выделены разные группы генов. Красными кругами обозначены выявленные акты дупликации генов.

Вторая группа генов, обозначенная нами как df-2 (рис. 4, красный фон), образовалась в результате дупликации df-1 у предка Benincaseae. При этом в роду Cucumis произошла дополнительная дупликация гена, которая сохранилась у всех проанализированных генотипов (включая неаннотированную последовательность в хромосоме 1 в геноме C. melo), кроме C. sativus PI 183967 (табл. 2).
К третьей группе генов, обозначенной как df-3 (рис. 4, синий фон), принадлежат последовательности, обнаруженные только в роду Cucurbita, включая неаннотированную последовательность в скаффолде 029 C. argyrosperma. Данная клада, вероятно, образовалась в результате дупликации у предка Cucurbiteae.
Таким образом, в результате поиска по гомологии и анализу последовательностей нам удалось выявить дополнительную копию гена df арбуза, которую мы обозначили df2.
dsh. В результате поиска по гомологии нам удалось выявить дополнительные копии гена dsh только у тыквы обыкновенной C. pepo, арбуза C. lanatus и восковой тыквы B. hispida. Причем у B. hispida данный ген дуплицировался дважды в обеих кладах. Вероятнее всего, дупликация данного гена возникла на ранних этапах эволюции семейства Cucurbitaceae, однако не сохранилась в геномах остальных представителей семейства (рис. 5). Таким образом, нам удалось выявить в геноме арбуза дополнительную копию гена dsh, которую мы обозначили как dsh2.
Анализ аллельного состояния и уровня экспрессии выявленных генов
Выявленные гены df2 (гиббереллин-2β-диоксигеназа) и dsh2 (гиббереллин-3β-гидроксилаза) обладают двухэкзонной структурой, как и их гомологичные копии df и dsh; в каждом из них сохранены ключевые домены PF03171 (2OG-Fe(II) oxygenase superfamily) и PF14226 (non-haem dioxygenase in morphine synthesis N-terminal). С помощью разработанных праймеров было осуществлено секвенирование последовательностей данных генов в геномах образцов Азербайджан местный и Sweet Treat, однако каких-либо изменений в кодирующих частях генов выявлено не было. Вероятно, данные гены также не являются причиной проявления кустового фенотипа растений.
На следующем этапе был проведен анализ относительного уровня экспрессии данных генов в сравнении с dw-1, df и dsh в стеблях и листьях следующих образцов: Charleston Gray 133 (плетистый), Родник (плетистый), Азербайджан местный (короткоплетистый) и Святослав (кустовой) (табл. 1, серый фон).
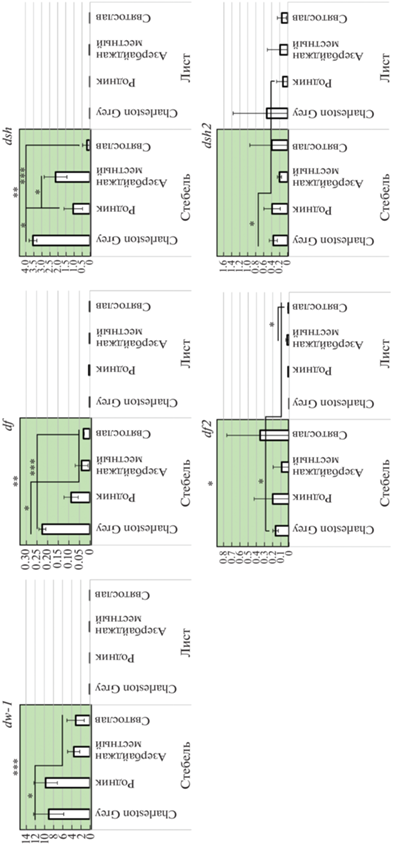
Было установлено, что экспрессия dw-1, df, dsh и df2 значимо выше в стеблях растений, чем в листьях (рис. 6). Активность генов dw-1 и df оказалась значимо ниже в стеблях образцов Азербайджан местный (короткоплетистый) и Святослав (кустовой) по сравнению с плетистыми сортами Charleston Gray 133 и Родник. Ген dsh экспрессировался слабее всего в кустовом сорте Святослав. Также его экспрессия была значимо ниже в сорте Родник. Экспрессия генов df2 и dsh2 находится на низком уровне во всех проанализированных образцах, экспрессия в стеблях у разных форм не различается (рис. 6). Таким образом, гены df2 и dsh2, вероятно, являются функционально активными, однако мы предполагаем, что они не вносят вклад в изменчивость по такому признаку, как длина стебля.
Рис. 6.
Относительный уровень экспрессии генов dw-1, df, dsh и их копий df2 и dsh2 в стеблях и листьях образцов арбуза коллекции ВИР. Планки погрешности отражают стандартную ошибку. Статистическая значимость определялась T-тестом. Различия статистически значимы между образцами: * при p ≤ 0.05, ** при p ≤ 0.01, *** при p ≤ 0.001.
ОБСУЖДЕНИЕ
В настоящее время селекция бахчевых культур является одним из важных направлений развития сельского хозяйства. При этом наиболее удобными для механизированных обработок и уборки плодов являются сорта бахчевых культур с малым габитусом растения: кустовые и плетистые [40].
Несмотря на то что геном арбуза C. lanatus секвенирован [41, 42], к настоящему моменту известны функции только небольшого числа генов, контролирующих хозяйственно ценные признаки [42–49]. Таким образом, в настоящее время актуальной задачей генетики арбуза является выявлениe эффективных генетических источников и доноров для создания новых сортов, а также идентификация в геноме данных доноров ценных для селекции генов-кандидатов.
К сегодняшнему моменту известны последовательности только трех генов компактности растений арбуза, различающихся по фенотипическому проявлению: dw-1, df и dsh [31, 33, 35, 36]. В настоящей работе был проведен скрининг выборки образцов арбуза из уникальной коллекции ВИР для выявления аллельного состояния этих известных генов. Нам удалось при помощи диагностических маркеров подтвердить, что все кустовые (и ультракустовые), но не короткоплетистые образцы арбуза коллекции ВИР и селекционные образцы Кубанской опытной станции ВИР, изученные в настоящей работе, являются носителями мутантного (нефункционального) аллеля гена dw-1 (рис. 1). Ранее при использовании данных источников признака кустовости отбор велся только по фенотипическим маркерам [1, 50]. В отличие от мутантов по другим генам карликовости у мутантов по гену dw-1 наблюдается неравномерное укорочение междоузлий и гофрированность листа. В дальнейшем для отбора селекционного материала на более ранних стадиях развития из двух известных маркеров [33] можно рекомендовать маркер dCAPS3 как наиболее подходящий, так как другой маркер, Indel1, давал ложноположительный результат.
Тем не менее в коллекции нам удалось обнаружить кустовые образцы, в которых гены dw-1, df и dsh, а также выявленные в настоящей работе копии данных генов df2 и dsh2, не утратили свою функциональность, что было подтверждено анализом структуры данных генов и их относительного уровня экспрессии. Это свидетельствует о том, что в коллекции бахчевых культур ВИР содержатся источники нового гена карликовости.
Интересным является фенотипическое проявление мутантов по новому гену карликовости арбуза в популяции Азербайджан местный (K-4039). У кустовых фенотипов изменяется не только габитус, но и окраска листьев – с зеленой на зеленоватую. При этом такая окраска не характерна для плетистых и короткоплетистых форм в данной популяции. Возможно, ген карликовости является сцепленным с геном зеленоватой окраски листьев, поскольку схожий цвет листьев был замечен нами также у другого образца Bush Charleston Gray (K-5131), являющегося мутантом по гену dw-1 (рис. 1).
В настоящей работе мы также обнаружили два ультракустовых фенотипа в популяции Sweet Treat (K-5373) (табл. 1). Данные растения отличаются нехарактерным для dw-1 ультракомпактным габитусом и ультрагофрированным листом с зеленоватой окраской. Мы предположили, что данные растения могут являться мутантами не только по гену dw-1, но и по новому гену карликовости и гену зеленоватой окраски листьев.
Подобный фенотип известен для дыни C. melo L., у которой ген желтовато-зеленоватой окраски листьев yv сцеплен с геном компактности si-1, в результате чего мутанты по yv и si-1 отличаются суперкомпактной формой с измененной окраской листьев [17].
Кроме того, у огурца C. sativus известны гены v-1 и v-2 (Virescent Leaf). Ген v-1 был картирован в хромосоме 6, где ген CsaCNGCs, который кодирует управляемый циклическими нуклеотидами ионный канал, является единственным геном-кандидатом [51]. Ген v-2 идентифицирован в линии 104Y, в которой семядольные и пять настоящих листьев были желтовато-зеленоватого цвета [52]. Также данная линия характеризовалась замедленным ростом, задержкой цветения и карликовостью. Однако в отличие от выявленных в настоящем исследовании мутантов арбуза желтовато-зеленоватые настоящие листья постепенно становились зелеными с повышенным содержанием хлорофилла. В качестве наиболее вероятного гена-кандидата желтовато-зеленоватого листа у огурца был предложен ген в хромосоме 3, кодирующий белок F-box, вовлеченный в биосинтез ауксина, что, в свою очередь, приводит к нарушению процесса регуляции биосинтеза хлорофилла [52].
Таким образом, выявление в коллекции ВИР новых источников кустовости дает начало исследованиям, заключающимся в идентификации и анализе генов-кандидатов карликовости и зеленоватой окраски листьев. Одновременно представляет интерес природа короткоплетистости. Такие формы не имеют мутации loss-of-function в кодирующей части гена dw-1 в отличие от кустовых форм, но так же, как и у кустовых форм, экспрессия гена dw-1 в стебле у короткоплетистых снижена в сравнении с плетистыми формами. То же самое наблюдается для гена df, но не dsh. У последнего экспрессия снижена только в стебле кустовых форм. Эти сведения намечают поиск гена-кандидата для короткоплетистости в последовательностях, связанных с регуляцией dw-1 и df, но не dsh.
В настоящей работе нами был проведен скрининг выборки образцов арбуза коллекции бахчевых культур ВИР им. Н.И. Вавилова для выявления аллельного состояния известных генов, определяющих компактную форму растений арбуза, dw-1, df и dsh. Все кустовые сорта и линии арбуза коллекции ВИР, кроме двух кустовых образцов популяции Азербайджан местный, являются носителями нефункционального аллеля гена dw-1. В геномах данных образцов в генах dw-1, df и dsh и их копиях, как и dw-1, не были выявлены значимые мутации, которые могли бы отразиться на функциональности генов. Кроме того, мы обнаружили два ультракустовых мутанта по dw-1 в популяции Sweet Treat, которые отличаются нехарактерным ультракомпактным габитусом, ультрагофрированным листом и зеленоватой окраской. Это свидетельствует о том, что в коллекции бахчевых культур ВИР содержатся источники новых генов, определяющих компактную форму.
Коллектив благодарит Анастасию Янышевскую (студент ЛГУ) за техническую помощь.
Определение доноров гена dw-1 в коллекции арбуза ВИР с помощью ДНК-маркеров выполнено при поддержке Министерства науки и высшего образования России в рамках соглашения № 075-15-2020-911 от 16.11.2020 о предоставлении гранта в форме субсидий из федерального бюджета на осуществление государственной поддержки создания и развития научного центра мирового уровня “Агротехнологии будущего”. Поиск новых генов кустовости арбуза выполнен в рамках темы НИР 0481-2019-0001/0481-2022-0007.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Tekhanovich G.A., Elatskova A.G., Elatskova Y.A. Genetic sources for breeding bushy and short-vine watermelon cultivars // Proc. Appl. Bot. Genet. Breed. 2019. V. 180. № 2. P. 89–94. https://doi.org/10.30901/2227-8834-2019-2-89-94
Peng J., Richards D.E., Hartley N.M. et al. ‘Green revolution’ genes encode mutant gibberellin response modulators // Nature. 1999. V. 400. № 6741. P. 256–261. https://doi.org/10.1038/22307
Spielmeyer W., Ellis M.H., Chandler P.M. Semidwarf (sd-1), “green revolution” rice, contains a defective gibberellin 20-oxidase gene // Proc. Natl Acad. Sci. 2002. V. 99. № 13. P. 9043–9048. https://doi.org/10.1073/pnas.132266399
Multani D.S., Briggs S.P., Chamberlin M.A. et al. Loss of an MDR transporter in compact stalks of maize br2 and sorghum dw3 mutants // Science. 2003. V. 302. № 5642. P. 81–84. https://doi.org/10.1126/science.1086072
Nomura T., Jager C.E., Kitasaka Y. et al. Brassinosteroid deficiency due to truncated steroid 5 α-reductase causes dwarfism in the lk mutant of pea // Plant Physiol. 2004. V. 135. № 4. P. 2220–2229. https://doi.org/10.1104/pp.104.043786
Pearce S., Saville R., Vaughan S.P. et al. Molecular characterization of Rht-1 dwarfing genes in hexaploid wheat // Plant Physiol. 2011. V. 157. № 4. P. 1820–1831. https://doi.org/10.1104/pp.111.183657
Pearce S., Saville R., Vaughan S.P. et al. A cytochrome P450, OsDSS1, is involved in growth and drought stress responses in rice (Oryza sativa L.) // Plant Mol. Biol. 2015. V. 88. № 1–2. P. 85–99. https://doi.org/10.1007/s11103-015-0310-5
Shifriss O. Developmental reversal of dominance in Cucurbita pepo // Proc. Amer. Soc. Hort. Sci. 1947. V. 50. P. 330–346.
Denna D.W., Munger H.M. Morphology of the bush and vine habits and the allelism of the bush genes in Cucurbita maxima and C. pepo squash // Proc. Amer. Soc. Hort. 1963. V. 82. P. 370–377.
Robinson R.W., Munger H.M., Whitaker T.W., Bohn G.W. Genes of the Cucurbitaceae // Hortscience. 1976. V. 11. № 6. P. 554–568.
Paris H.S., Brown R.N. The genes of pumpkin and squash // HortScience. 2005. V. 40. № 6. P. 1620–1630. https://doi.org/10.21273/hortsci.40.6.1620
Wang S., Li H., Zhang Z. et al. Comparative mapping of the dwarf gene Bu from tropical pumpkin (Cucurbita moschata Duchesne) // Hortic. Sin. 2011. V. 38. № 1. P. 95–100.
Zhang G., Ren Y., Sun H. et al. A high-density genetic map for anchoring genome sequences and identifying QTLs associated with dwarf vine in pumpkin (Cucurbita maxima Duch.) // BMC Genomics. 2015. V. 16. № 1. P. 1101. https://doi.org/10.1186/s12864-015-2312-8
Hwang J., Oh J., Kim Z. et al. Fine genetic mapping of a locus controlling short internode length in melon (Cucumis melo L.) // Mol. Breed. 2014. V. 34. № 3. P. 949–961. https://doi.org/10.1007/s11032-014-0088-1
Knavel D.E. Inheritance of a short-internode mutant of ‘Mainstream’ muskmelon // HortScience. 1990. V. 25. № 10. P. 1274–1275. https://doi.org/10.21273/HORTSCI.25.10.1274
Fukino N., Ohara T., Sugiyama M. et al. Mapping of a gene that confers short lateral branching (slb) in melon (Cucumis melo L.) // Euphytica. 2012. V. 187. № 1. P. 133–143. https://doi.org/10.1007/S10681-012-0667-3/FIGURES/4
Pitrat M. Linkage Groups in Cucumis melo L. // J. Hered. 1991. V. 82. № 5. P. 406–411. https://doi.org/10.1093/oxfordjournals.jhered.a111112
Xin M., Qin Z., Wang L. et al. Genetic identification of a dwarf mutant in cucumber (Cucumis sativus L.) // Afr. J. Biotechnol. 2012. V. 11. № 20. P. 4493–4498. https://doi.org/10.5897/AJB11.968
Hou S., Niu H., Tao Q. et al. A mutant in the CsDET2 gene leads to a systemic brassinosteriod deficiency and super compact phenotype in cucumber (Cucumis sativus L.) // Theor. Appl. Genet. 2017. V. 130. № 8. P. 1693–1703. https://doi.org/10.1007/s00122-017-2919-z
Wang H., Li W., Qin Y. et al. The cytochrome P450 gene CsCYP85A1 is a putative candidate for super compact-1 (Scp-1) plant architecture mutation in cucumber (Cucumis sativus L.) // Front. Plant Sci. 2017. V. 8. P. 1–13. https://doi.org/10.3389/fpls.2017.00266
Kauffman C.S., Lower R.L. Inheritance of an extreme dwarf plant type in the cucumber // J. Am. Soc. Hortic. Sci. 1976. V. 101(2). P. 150–151.
Kubicki B., Soltysiak U., Korzeniewska A. Induced mutation in cucumber (Cucumis sativus L.) V. Compact type of growth // Genet. Pol. 1986. V. 27. P. 3–4.
Lin T., Wang S., Zhong Y. et al. A truncated F-box protein confers the dwarfism in cucumber // J. Genet. Genomics. 2016. V. 43. № 4. P. 223–226. https://doi.org/10.1016/j.jgg.2016.01.007
Xu L., Wang C., Cao W. et al. CLAVATA1-type receptor-like kinase CsCLAVATA1 is a putative candidate gene for dwarf mutation in cucumber // Mol. Genet. Genomics. 2018. V. 293. № 6. P. 1393–1405. https://doi.org/10.1007/s00438-018-1467-9
Li Y., Yang L., Pathak M. et al. Fine genetic mapping of cp: a recessive gene for compact (dwarf) plant architecture in cucumber, Cucumis sativus L. // Theor. Appl. Genet. 2011. V. 123. № 6. P. 973–983. https://doi.org/10.1007/s00122-011-1640-6
Van der Linden L. Marker for compact growth in cucumber. Patent WO/2017/042272. 2018.
Liu P.B.W., Loy J.B. Inheritance and morphology of two dwarf mutants in watermelon // Amer. Soc. Hort. Sci. J. 1972. P. 745–748.
Mohr H.C., Sandhu M.S. Inheritance and morphological traits of a double recessive dwarf in watermelon, Citrullus lanatus (Thunb.) Mansf. // J. Am. Soc. Hortic. Sci. 1975. V. 100(2). P. 135–137.
Dyutin K.E., Afanasyeva E.A. Inheritance of the short vine trait in watermelon // Cytol. Genet. 1987. V. 21. № 3. P. 227–229.
Hexun H., Xiaoqi Z., Zhencheng W. et al. Inheritance of male-sterility and dwarfism in watermelon [Citrullus lanatus (Thunb.) Matsum. and Nakai] // Sci. Hortic (Amsterdam). 1998. V. 74. № 3. P. 175–181. https://doi.org/10.1016/S0304-4238(97)00102-7
Dong W., Wu D., Li G. et al. Next-generation sequencing from bulked segregant analysis identifies a dwarfism gene in watermelon // Sci. Rep. 2018. V. 8. № 1. P. 2908. https://doi.org/10.1038/s41598-018-21293-1
Dong W., Wu D., Wang C. et al. Characterization of the molecular mechanism underlying the dwarfism of dsh mutant watermelon plants // Plant Sci. 2021. V. 313. P. 111074. https://doi.org/10.1016/j.plantsci.2021.111074
Zhu H., Zhang M., Sun S. et al. A single nucleotide deletion in an ABC transporter gene leads to a dwarf phenotype in watermelon // Front. Plant Sci. 2019. V. 10. P. 1–13. https://doi.org/10.3389/fpls.2019.01399
Cho Y., Lee S., Park J. et al. Identification of a candidate gene controlling semi-dwarfism in watermelon, Citrullus lanatus, using a combination of genetic linkage mapping and QTL-seq // Hortic. Environ. Biotechnol. 2021. V. 62. № 3. P. 447–459. https://doi.org/10.1007/S13580-020-00330-X/TABLES/3
Gebremeskel H., Dou J., Li B. et al. Molecular mapping and candidate gene analysis for GA3 responsive short internode in watermelon (Citrullus lanatus) // Int. J. Mol. Sci. 2019. V. 21. № 1. P. 290. https://doi.org/10.3390/ijms21010290
Wei C., Zhu C., Yang L. et al. A point mutation resulting in a 13 bp deletion in the coding sequence of Cldf leads to a GA-deficient dwarf phenotype in watermelon // Hortic. Res. 2019. V. 6. № 1. P. 132. https://doi.org/10.1038/s41438-019-0213-8
Zhang T., Liu J., Amanullah S. et al. Fine mapping of Cla015407 controlling plant height in watermelon // J. Am. Soc. Hortic. Sci. 2021. V. 146. № 3. P. 196–205. https://doi.org/10.21273/JASHS04934-20
Sun Y., Zhang H., Fan M. et al. A mutation in the intron splice acceptor site of a GA3ox gene confers dwarf architecture in watermelon (Citrullus lanatus L.) // Sci. Rep. 2020. V. 10. № 1. P. 14915. https://doi.org/10.1038/s41598-020-71861-7
Kong Q., Yuan J., Gao L. et al. Evaluation of appropriate reference genes for gene expression normalization during watermelon fruit development // PLoS One. 2015. V. 10. № 6. P. e0130865. https://doi.org/10.1371/journal.pone.0130865
Теханович Г.А., Елацкова А.Г., Елацков Ю.А. Роль мировой коллекции бахчевых культур ВИР в селекции // Тр. по прикладной ботанике, генетике и селекции. 2012. Т. 169. С. 289–294.
Guo S., Zhang J., Sun H. et al. The draft genome of watermelon (Citrullus lanatus) and resequencing of 20 diverse accessions // Nat. Genet. 2013. V. 45. № 1. P. 51–58. https://doi.org/10.1038/ng.2470
Guo S., Zhao S., Sun H. et al. Resequencing of 414 cultivated and wild watermelon accessions identifies selection for fruit quality traits // Nat. Genet. 2019. V. 51. № 11. P. 1616–1623. https://doi.org/10.1038/s41588-019-0518-4
Dou J., Zhao S., Lu X. et al. Genetic mapping reveals a candidate gene (ClFS1) for fruit shape in watermelon (Citrullus lanatus L.) // Theor. Appl. Genet. 2018. V. 131. № 4. P. 947–958. https://doi.org/10.1007/s00122-018-3050-5
Legendre R., Kuzy J., McGregor C. Markers for selection of three alleles of ClSUN25-26-27a (Cla011257) associated with fruit shape in watermelon // Mol. Breed. 2020. V. 40. № 2. P. 19. https://doi.org/10.1007/s11032-020-1104-2
Tian S., Jiang L., Gao Q. et al. Efficient CRISPR/ Cas9-based gene knockout in watermelon // Plant Cell Rep. 2017. V. 36. № 3. P. 399–406. https://doi.org/10.1007/s00299-016-2089-5
Wang Y., Wang J., Guo S. et al. CRISPR/Cas9-mediated mutagenesis of ClBG1 decreased seed size and promoted seed germination in watermelon // Hortic. Res. 2021. V. 8. № 1. P. 70. https://doi.org/10.1038/s41438-021-00506-1
Tian S., Jiang L., Cui X. et al. Engineering herbicide-resistant watermelon variety through CRISPR/Cas9-mediated base-editing // Plant Cell Rep. 2018. V. 37. № 9. P. 1353–1356. https://doi.org/10.1007/s00299-018-2299-0
Zhang J., Guo S., Ji G. et al. A unique chromosome translocation disrupting ClWIP1 leads to gynoecy in watermelon // Plant J. 2020. V. 101. № 2. P. 265–277. https://doi.org/10.1111/tpj.14537
Zhang M., Liu Q., Yang X. et al. CRISPR/Cas9-mediated mutagenesis of Clpsk1 in watermelon to confer resistance to Fusarium oxysporum f.sp. niveum // Plant Cell Rep. 2020. V. 39. № 5. P. 589–595. https://doi.org/10.1007/s00299-020-02516-0
Теханович Г.А., Елацкова А.Г., Елацков Ю.А. Новые источники генетической коллекции бахчевых культур // Науч. обеспечение производства сельскохозяйственных культур в современных условиях. 2016. P. 198–203.
Miao H., Zhang S., Wang M. et al. Fine mapping of virescent leaf gene v-1 in cucumber (Cucumis sativus L.) // Int. J. Mol. Sci. 2016. V. 17. № 10. P. 1602. https://doi.org/10.3390/ijms17101602
Zhang K., Li Y., Zhu W. et al. Fine mapping and transcriptome analysis of virescent leaf gene v-2 in cucumber (Cucumis sativus L.) // Front. Plant Sci. 2020. V. 11. P. 1458. https://doi.org/10.3389/FPLS.2020.570817/BIBTEX
Дополнительные материалы отсутствуют.