Генетика, 2022, T. 58, № 12, стр. 1418-1430
Гомологи генов устойчивости к фитофторозу у представителей клубнеобразующих видов рода Solanum L.
А. А. Гурина 1, *, Н. В. Алпатьева 1, Н. А. Чалая 1, Н. В. Мироненко 2, А. В. Хютти 2, Е. В. Рогозина 1
1 Всероссийский институт генетических ресурсов растений им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
2 Всероссийский научно-исследовательский институт защиты растений
196608 Пушкин, Санкт-Петербург, Россия
* E-mail: a.gurina@vir.nw.ru
Поступила в редакцию 16.06.2022
После доработки 27.06.2022
Принята к публикации 28.06.2022
- EDN: USPQYT
- DOI: 10.31857/S0016675822120049
Аннотация
Возбудитель фитофтороза, оомицет Phytophthora infestans Mont de Bary, обладает высокой степенью изменчивости, в результате чего новые расы патогена способны преодолеть устойчивость длительное время возделываемых сортов картофеля. Примитивные культурные виды картофеля относятся к первичному генофонду, представители которого легко скрещиваются с Solanum tuberosum L., и их использование для селекции перспективно. Цель исследования – идентификация генотипов диких и примитивных культурных видов картофеля ВИР, несущих Rpi-гены. Впервые образцы примитивных культурных и диких видов картофеля (105 генотипов из коллекции ВИР) проанализированы на устойчивость к фитофторозу и наличие у них SCAR-маркеров Rpi-генов (RB/blb1, Rpi-blb2, R2-like, Rpi-vnt1.3). У культурного вида S. stenotomum subsp. stenotomum обнаружена высокая (0.71) частота одного из двух маркерных фрагментов гена RB/blb1 (Rpi-sto1), исходно охарактеризованного у дикого североамериканского вида S. bulbocastanum, который относится к третичному генофонду видов картофеля. У видов S. phureja и S. stenotomum subsp. goniocalyx обнаружена высокая (0.71–0.88) частота маркера гена Rpi-vnt1.3, исходно охарактеризованного у дикого южноамериканского вида S. venturii. Впервые у примитивных видов картофеля охарактеризованы последовательности фрагментов – предполагаемых гомологов генов Rpi-vnt1 и RB/blb1. У представителей S. ajanhuiri, S. stenotomum и S. phureja обнаружены три варианта нуклеотидных последовательностей, гомологичных Rpi-vnt1.3. Сделано предположение о возможной роли обнаруженного полиморфизма маркерных фрагментов Rpi-vnt1.3 в обеспечении устойчивости примитивных культурных видов к фитофторозу.
Фитофтороз (возбудитель Phytophthora infestans (Mont.) de Bary) – одно из наиболее вредоносных заболеваний картофеля. Вспышка этого заболевания была причиной Ирландского голода в середине XIX в. [1]. Наиболее эффективным методом борьбы с этим заболеванием является возделывание устойчивых сортов. Такие сорта создаются в результате интрогрессии генов устойчивости (Rpi) путем межвидовой гибридизации и последующего отбора. К настоящему времени у картофеля известно более 20 генов устойчивости к фитофторозу. В практической селекции в основном используют образцы с Rpi-генами, источниками которых являются дикие виды картофеля из Северной и Центральной Америки – S. bulbocastanum Dunal, S. demissum Lindl., S. stoloniferum Schltdl. [2]. Гены устойчивости также обнаружены и в других диких видах: североамериканских S. cardiophyllum Lindl. и S. pinnatisectum Dunal и южноамериканских S. berthaultii Hawkes, S. mochiquense Ochoa и S. venturii Hawkes & Hjert. и др. [3]. Для P. infestans характерна высокая внутрипопуляционная генетическая вариабельность изолятов [4], которая фенотипически проявляется в виде образования новых (более вирулентных, агрессивных) рас патогена. По причине интенсивного расообразовательного процесса невозможно получить стабильно устойчивые сорта картофеля, и для эффективного проведения селекции по этому признаку требуются новые, ранее не используемые гены устойчивости. Источниками новых генов устойчивости могут стать устойчивые примитивные культурные виды, являющиеся представителями первичного генофонда, что значительно облегчает процесс интрогрессии генов, ввиду отсутствия биологических барьеров для скрещивания [5].
Геном возбудителя фитофтороза секвенирован [6, 7]. C помощью молекулярно-генетических подходов достигнут заметный прогресс в изучении популяций P. infestans, молекулярных механизмов взаимодействия хозяина и патогена, генетического контроля вирулентности возбудителя и устойчивости хозяина [8]. Однако до настоящего времени нет единого мнения о том, где находится центр происхождения возбудителя фитофтороза картофеля: в центральной Мексике или южноамериканских Андах [9]. Примечательно, что оба возможных центра происхождения P. infestans расположены в пределах территорий, обозначенных как центры разнообразия клубнеобразующих видов секции Petota Dumort. рода Solanum L. – родичей возделываемого картофеля. В Южной Америке наибольшее видовое разнообразие диких и культурных картофелей выявлено на территории Перу, вторичный центр биоразнообразия картофелей находится на территории Мексики [10].
Клубнеобразующие виды рода Solanum занимают протяженный ареал, простирающийся от 38° с.ш. до 41° ю.ш., произрастают в широком диапазоне вертикальной зональности – от 0 до 5000 м над уровнем моря [10]. В пределах этой территории определены четыре центра возникновения фитофтороустойчивых форм у диких и культурных видов картофеля [11]. Изучение генетического контроля признака устойчивости к фитофторозу у видов картофеля, сформировавшихся в разных частях общего ареала секции Petota, является важным этапом на пути познания их филогении, установления их родственных связей. Сравнительный анализ Rpi-генов, выявление степени их структурного сходства позволит приблизиться к пониманию путей эволюции генетических детерминант устойчивости растений к патогену P. infestans. Секвенирование генома удвоенного моноплоида – клона S. phureja DM1-3 516 R44 [12] и его изучение активизировало интерес к группе культурных видов картофеля (ландрасам, возделываемым коренным населением в различных районах Анд). В референсной последовательности генома в результате биоинформатического поиска обнаружено 435 NBS-LRR генов (Nucleotide binding site, leucine rich repeats). К этому семейству относятся все основные R-гены растений, являющиеся ключевыми в обеспечении устойчивости растений к патогенам разной природы [13]. Методами классической генетики у межвидового гибрида S. stenotomum × S. phureja выявлен ген Rpi-phu1, локализованный на хромосоме 9. Ген Rpi-phu1 отличается широким спектром действия, обеспечивая устойчивость как листьев, так и клубней к фитофторозу [14, 15]. У дикого картофеля из Аргентины S. venturii в том же локусе хромосомы 9 идентифицирован ген устойчивости к фитофторозу Rpi-vnt1.1 (и аллельные варианты Rpi-vnt1.2 и Rpi-vnt1.3), который является гомологом гена Tm-22, обеспечивающего устойчивость томата (S. lycopersicum L.) к вирусу мозаики томата [16]. Белки, кодируемые Rpi-vnt1.1 и Rpi-vnt1.3, имеют 73% идентичности аминокислотной последовательности с белком гена Tm-22 [17]. Устойчивость к широкому спектру штаммов P. infestans обеспечивают гены дикого мексиканского вида S. bulbocastanum: RB/blb1 [18], Rpi-blb2 [19, 20] и Rpi-blb3 [21]. В референсной последовательности DM1-3 516 R44 показана кластерная организация генов устойчивости: в том числе гомологи гена Rpi-vnt1 на хромосоме 9, гена Rpi-blb2 на хромосоме 6 и наиболее представительный (55 R-генов) кластер на хромосоме 4 [13]. Большое семейство генов, расположенных на хромосоме 4 и обеспечивающих защиту от фитофтороза, включает R2, R2-like, Rpi-abpt, Rpi-blb3, Rpi-edn1.1, Rpi-hjt1.1, Rpi-hjt1.2, Rpi-hjt1.3, Rpi-snk1.1, Rpi-snk1.2 [22].
Разнообразие примитивных культурных видов картофеля, обусловленное их возделыванием в сильно различающихся климатических условиях [23], позволяет предположить наличие других, ранее неизвестных генов устойчивости к различным заболеваниям, в том числе к фитофторозу. В частности, по исследованиям Gabriel с соавт. у S. phureja фитофтороустойчивые образцы встречаются довольно часто, что подводит исследователей к необходимости более широкого изучения этой группы [24].
Цель данного исследования ‒ идентификация генотипов, несущих гены устойчивости к фитофторозу (Rpi-гены), среди примитивных культурных видов, а также ранее не охарактеризованных образцов диких видов картофеля в коллекции ВИР. Поскольку спектр известных Rpi-генов чрезвычайно широк, для исследований были выбраны внутригенные SCAR-маркеры тех генов, гомологи которых ранее были обнаружены среди примитивных культурных видов и наиболее близких к культурным, диких видов картофелях [14, 25]. В первую очередь речь идет о генах RB/blb1, Rpi-blb2 и Rpi-vnt1, у которых при биоинформатическом анализе были выявлены гомологичные последовательности референсного генома картофеля [13, 26].
Впервые проведен скрининг представительной выборки культурных и диких клубнеобразующих видов рода Solanum L. из южноамериканского и североамериканского центров биоразнообразия. Среди группы примитивных культурных видов картофеля было показано наличие маркеров всех исследованных генов устойчивости, кроме R2-like. Сопоставление данных о присутствии SCAR-маркеров и полиморфизме их последовательностей с результатами лабораторного заражения картофеля фитофторозом позволило предположить какие из изученных генов могут принимать участие в формировании устойчивости и выявить образцы, устойчивость которых не обусловлена ни одним из исследованных генов.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследования послужили образцы диплоидных примитивных культурных видов: S. ajanhuiri Juz. & Bukasov, S. stenotomum subsp. goniocalyx (Juz. & Bukasov) Hawkes, S. stenotomum subsp. stenotomum и S. phureja, а также образцы трех североамериканских и восьми южноамериканских диких видов картофеля из коллекции ВИР (Приложение 1 ). Для удобства сравнения с литературными источниками видовая принадлежность коллекционных образцов представлена в соответствии с наиболее распространенной систематикой секции Petota Dumort. рода Solanum L., принадлежащей J. Hawkes [27]. Всего исследованы 105 генотипов клубнеобразующих видов рода Solanum. Большинство (75 генотипов) относятся к группе примитивных культурных видов, 13 относятся к трем североамериканским диким видам (S. brachystotrichum, S. lesteri и S. bulbocastanum), а оставшиеся 17 – представители восьми диких южноамериканских видов картофеля. Исследованные генотипы дикорастущих видов получены из семян коллекционных образцов и сохраняются в виде клонов путем получения клубней у оранжерейных растений. Культурные виды поддерживаем путем получения клубневой репродукции в поле.
Культурные виды впервые проанализированы на устойчивость к фитофторозу и наличие ДНК-маркеров R-генов. Включенные в опыт образцы диких видов ранее не были оценены на устойчивость к фитофторозу, но были задействованы в других исследованиях, в частности скрининге на устойчивость к золотистой картофельной нематоде [28].
Лабораторная оценка устойчивости к фитофторозу
Растения выращивали в теплице в пластиковых горшках объемом 500 см3 (по одному клубню в каждый горшок). Для оценки взято по 5 долей листьев срединной формации от растений в возрасте более 60 дней после посадки в двукратной биологической повторности.
Лабораторный скрининг образцов картофеля на устойчивость к фитофторозу проводили по стандартной методике [29]. Для заражения использовался инокулюм на основе изолята MP1841, полученного из Института селекции и акклиматизации растений, Млохов, Польша (IHAR-Mlochow). Изолят содержит все 11 генов вирулентности (1.2.3.4.5.6.7.8.9.10.11). Инокулюм выдерживался в течение 30 мин при температуре 10–12°С для стимуляции выхода зооспор. Концентрация спорангиев в инокулюме соответствовала 50 000 ед./мл. Листья выкладывались на увлажненную фильтровальную бумагу абаксиальной стороной вниз, между центральной и боковыми жилками наносилось по 30 мкл инокулюма. Через сутки после инокуляции листья переворачивали абаксиальной стороной вверх. В течение всего периода инокуляции поддерживались постоянные условия в климатическом боксе: 16°C [30]. Степень поражения оценивалась на шестые сутки после заражения по 9-балльной шкале [31]: образцы с оценкой от 1 до 3 баллов (поражение более 25% площади поверхности зараженного листа) считались восприимчивыми (S) к фитофторозу, от 4 до 6 (от 5 до 25%) – среднеустойчивыми (MR) (умеренно восприимчивыми), а от 7 до 9 (менее 5%) – устойчивыми (R). Опыт проводили в двукратной повторности и устойчивость оценивали по усредненным значениям в обеих повторностях. Контролем в эксперименте служили сорта Невский (восприимчивый) и Сударыня (устойчивый).
Скрининг примитивных и диких видов картофеля с помощью SCAR-маркеров генов устойчивости RB/blb1, Rpi- blb2, R2-like и Rpi-vnt1.3
ДНК выделяли из молодых листьев картофеля в двукратной повторности с использованием СТАВ-буфера по протоколу Гавриленко и соавт. [32]. Фрагменты предполагаемых гомологов генов устойчивости у примитивных видов амплифицировали с помощью специфичных для SCAR-маркеров праймеров Rpi-blb1, Rpi-sto1, Rpi-blb2, R2-like и Rpi-vnt1.3 по протоколам, предложенным авторами (табл. 1) с использованием Taq-полимеразы (Диалат, Москва).
Таблица 1.
Используемые в исследовании SCAR-маркеры генов Rpi
| Ген | Маркер | Длина фрагмента, н | Последовательность праймеров | t°С отжига | Источник |
|---|---|---|---|---|---|
| RB/blb1 | Rpi-sto1 | 890 | F-accaaggccacaagattctc R-cctgcggttcggttaataca |
65 | [33] |
| Rpi-blb1 | 820 | F-aacctgtatggcagtggcatg R-gtcagaaaagggcactcgtg |
62 | [34] | |
| Rpi-blb2 | Rpi-blb2 | 976 | F-ggactgggtaacgacaatcc R-atttatggctgcagaggacc |
55 | [34] |
| R2-like | R2 area 1/2 | 1137 | F-aagatcaagtggtaaaggctgatg R-atctttctagcttccaaagatcacg |
60 | [35] |
| Rpi-vnt1.3 | Rpi-vnt1.3 | 611 | F-ccttcctcatcctcacatttag R-gcatgccaactattgaaacaac |
58 | [17] |
Продукты ПЦР визуализировали в 1.7%-ном агарозном геле, окрашивали бромистым этидием и документировали в системе BioDocII (Biometra GmbH, Германия).
Секвенирование фрагментов предполагаемых гомологов Rpi-vnt1 и RB/blb1 у примитивных видов картофеля
У семи образцов примитивных культурных видов (к-9911, к-3558, к-9345, к-8873, к-17618, к-9301 и к-1120), контрастных по устойчивости к патогену, секвенировали фрагменты, полученные с помощью праймеров Rpi-vnt1.3. У двух образцов S. stenotomum subsp. stenotomum к-7366 и к-10478, восприимчивых к фитофторе, секвенировали фрагменты, полученные с помощью праймеров Rpi-sto1 и Rpi-blb1. Предварительно ампликоны обоих типов выделяли из ПЦР-смеси с помощью набора Cleanup Standard, затем лигировали в pAL-TA вектор согласно протоколу фирмы “Евроген” (http://evrogen.ru/kit-user-manuals/pAL-TA.pdf). Для трансформации использовали штамм DH5α E. coli. Подробно протокол представлен в методических указаниях ВИР [36]. Два фрагмента каждого образца секвенировали в двух направлениях с использованием оборудования ЦКП “Геномные технологии, протеомика и клеточная биология” ФГБНУ ВНИИСХМ на приборе ABI 3500xl Genetic Analyzer (Applied Biosystems, США). Выравнивание полученных последовательностей и их анализ проводили с помощью программы MEGA version 11 [37]. Идентифицировали фрагменты по степени сходства с последовательностями, депонированными в международной базе нуклеотидных последовательностей NCBI GenBank и в поисковой системе BLAST (https:// blast.ncbi.nlm.nih.gov/Blast.cgi). Нуклеотидные последовательности были депонированы в базу данных GenBank под номерами: для последовательностей гена Rpi-vnt1.3 ‒ ON322726–ON322739, для последовательностей гена RB/blb1 ‒ ON515750.
РЕЗУЛЬТАТЫ
Среди образцов каждого культурного вида картофеля были обнаружены устойчивые (среднеустойчивые) к фитофторозу генотипы (рис. 1), что подтверждает перспективность изучения этой группы генофонда Solanum. Среди изученных образцов североамериканских диких видов у представителей S. brachystotrichum (Bitt.) Rydb. не было генотипов, устойчивых к фитофторозу, среди других видов устойчивые формы были обнаружены (рис. 2). Среди южноамериканских диких видов лишь некоторые генотипы S. doddsii Corell и S. leptophyes Bitter были среднеустойчивыми, изученные генотипы всех остальных видов восприимчивы к фитофторозу.
Рис. 1.
Схема, демонстрирующая распределение маркеров среди различающихся по уровню устойчивости образцов примитивных культурных видов картофеля.

Рис. 2.
Схема, демонстрирующая распределение маркеров среди различающихся по уровню устойчивости образцов диких видов картофеля.

Встречаемость SCAR-маркеров генов RB/blb1, Rpi-blb2, R2-like и Rpi-vnt1.3 у культурных и диких видов картофеля оказалась различной (рис. 1, 2). Наиболее заметны различия в распределении маркеров у североамериканских диких и примитивных культурных видов картофеля. В связи с небольшим числом образцов, изученных у южноамериканских диких видов картофеля, они представлены на рис. 2 в виде объединенной группы. Выявлена специфичность распределения SCAR-маркеров у представителей разных видов картофеля. Маркер R2 area 1/2 гена R2-like есть у всех изученных североамериканских диких видов, тогда как среди изученных южноамериканских видов он выявлен только у двух образцов дикого вида S. doddsii, а у культурных видов и вовсе отсутствовал. Противоположная картина характерна для маркера Rpi-blb2: он обнаружен у представителей южноамериканских диких и культурных видов (S. alandiae, S. doddsii, S. kurtzianum, S. sparsipilum, S. yungasense, S. ajanhuirii и S. stenotomum subsp. goniocalyx) и отсутствовал у исследованных образцов из Северной Америки (рис. 1, 2). Маркер Rpi-vnt1.3 обнаружен у южноамериканских видов картофеля: с высокой частотой (0.71–0.88) у культурных видов S. phureja и S. stenotomum subsp. goniocalyx, обнаружен также у диких видов S. doddsii, S. kurtzianum, S. neocardenasii, S. spegazzinii, но найден только у единственного представителя североамериканских видов – образца S. brachystotrichum. Маркер Rpi-sto1 встречается у всех примитивных культурных видов картофеля, в том числе с высокой частотой (0.55–0.71) у образцов двух подвидов S. stenotomum, но из всех исследованных диких видов найден только у единственного образца S. bulbocastanum.
В пределах группы примитивных культурных видов наблюдаются значительные отличия по частоте двух маркеров гена RB/blb1. Маркер Rpi-sto1, обнаруженный у всех образцов примитивных видов, наиболее часто встречается у S. stenotomum subsp. stenotomum и крайне редко у S. phureja; второй маркер – Rpi-blb1 обнаружен только у двух генотипов S. stenotomum subsp. stenotomum (рис. 1). Маркер Rpi-blb2 обнаружен у представителей двух других видов – S. ajanhuirii и S. stenotomum subsp. goniocalyx (рис. 1).
У S. phureja обнаружены два устойчивых (кр8873, к-17618) и пять среднеустойчивых (к-9345, к-11547, к-16896, к-19321, к-23516) к фитофторозу генотипов (табл. 2). Оба устойчивых образца имеют перуанское происхождение, четыре из пяти среднеустойчивых – колумбийское, еще один среднеустойчивый образец получен в Англии путем скрещивания образцов из Боливии и Колумбии. Образцы S. phureja из Боливии и Эквадора оказались неустойчивыми к фитофторозу. Корреляции между наличием маркеров и показателями устойчивости исследованных образцов S. phureja не обнаружено. Фрагмент Rpi-vnt1.3 был амплифицирован у образцов устойчивых, среднеустойчивых и восприимчивых к фитофторозу (рис. 1). У двух среднеустойчивых образцов S. phureja (к‑11547, к-23516) также обнаружен один из двух маркеров гена RB/blb1 – Rpi-sto1.
Таблица 2.
Характеристика устойчивых и среднеустойчивых образцов
| Вид | Происхождение образца | Номер коллекционного образца | Фенотип по устойчивости к фитофторозу | SCAR-маркеры |
|---|---|---|---|---|
| Примитивные культурные виды | ||||
| S. ajanhuirii | Боливия | к-9900 | MR | Rpi-sto1, Rpi-blb2 |
| S. ajanhuirii | Боливия | к-9911-2 | R | Rpi-blb2, Rpi-vnt1.3 |
| S. stenotomum subsp. goniocalyx | Боливия | к-9922 | MR | Rpi-sto1, Rpi-vnt1.3 |
| S. phureja | Великобритания (Bol × Col) | к-9345 | MR | Rpi-vnt1.3 |
| S. phureja | Колумбия | к-11547 | MR | Rpi-sto1, Rpi-vnt1.3 |
| S. phureja | Колумбия | к-16896 | MR | Rpi-vnt1.3 |
| S. phureja | Колумбия | к-19321 | MR | Rpi-vnt1.3 |
| S. phureja | Колумбия | к-23516 | MR | Rpi-sto1, Rpi-vnt1.3 |
| S. phureja | Перу | к-8873 | R | Rpi-vnt1.3 |
| S. phureja | Перу | к-17618 | R | Rpi-vnt1.3 |
| S. stenotomum subsp. stenotomum | Великобритания (Bol × Bol) | к-9278 | MR | Не обнаружено |
| S. stenotomum subsp. stenotomum | Перу | к-8354 | MR | Rpi-sto1 |
| S. stenotomum subsp. stenotomum | Перу | к-11020 | R | Rpi-vnt1.3 |
| S. stenotomum subsp. stenotomum | Перу | к-17486 | MR | Не обнаружено |
| Южноамериканские дикие виды картофеля | ||||
| S. doddsii | Боливия | к-19817 | MR | Rpi-blb2 |
| S. leptophyes | Великобритания | к-5764 | MR | Не обнаружено |
| Североамериканские дикие виды картофеля | ||||
| S. lesteri | Мексика | к-24475-gt1 | MR | Не обнаружено |
| S. lesteri | Мексика | к-24475-gt5 | R | R2-area 1/2 |
| S. bulbocastanum | Гватемала | к-24866 | R | R2-area 1/2, Rpi-sto1, Rpi-blb1 |
У S. stenotomum subsp. stenotomum выявлен один устойчивый (к-11020) и три среднеустойчивых (к‑8354; к-9278; к-17486) генотипа. Три из них имеют перуанское происхождение, один среднеустойчивый (к-9278) получен в Англии путем скрещивания двух образцов из Боливии. У двух среднеустойчивых образцов этого вида не обнаружено ни одного из использованных маркеров. Единственный устойчивый генотип S. stenotomum subsp. stenotomum (к-11020) так же, как и образцы S. phureja, обладает маркером Rpi-vnt1.3. У среднеустойчивого образца к-8354 обнаружен маркерный фрагмент Rpi-sto1, разработанный на coiled-coil домен (СС) гена RB/blb1, но отсутствует Rpi-blb1 – специфичный маркер для LRR-домена (leucine-rich repeat) того же гена [2]. В то же время два восприимчивых генотипа S. stenotomum subsp. stenotomum (к-7366 и к-10478) имеют оба маркера гена RB/blb1 и маркер Rpi-vnt1.3 (рис. 1).
Изученные генотипы S. ajanhuiri различаются по реакции на заражение патогеном, но у каждого обнаружен маркер Rpi-blb2. Кроме того, у устойчивого S. ajanhuiri к-9900 обнаружен SCAR-маркер Rpi-vnt1.3, а у среднеустойчивого S. ajanhuiri к-9911 − Rpi-sto1 (рис. 1). Среди изученных образцов S. stenotomum subsp. goniocalyx выявлен только один среднеустойчивый генотип (к-9922), у которого обнаружены маркеры – Rpi-sto1 и Rpi-vnt1.3.
Среди представителей диких южноамериканских видов картофеля выявлены два среднеустойчивых генотипа – S. doddsii (к-19817) и S. leptophyes (к-5764). У S. doddsii присутствует лишь один SCAR-маркер – Rpi-blb2, а у S. leptophyes маркеры не обнаружены.
Среди образцов диких североамериканских видов картофеля S. bulbocastanum и S. lesteri обнаружены один среднеустойчивый и два устойчивых генотипа. Все образцы S. brachystotrichum были восприимчивы к фитофторозу. У двух генотипов S. bulbocastanum к-24868 и трех генотипов S. lesteri к-24475 (в том числе одного среднеустойчивого) не обнаружено ни одного из исследованных маркеров. У устойчивого образца S. bulbocastanum обнаружены оба маркера гена RB/blb1, а также маркер гена R2-like. Можно предположить, что именно ген RB/blb1 обеспечивает устойчивость этого генотипа S. bulbocastanum к фитофторозу, поскольку среди всех диких видов это единственный образец, обладающий обоими маркерными фрагментами гена.
У контрастных по устойчивости образцов примитивных культурных видов были изучены нуклеотидные последовательности ампликонов, полученных с использованием праймеров Rpi-sto1, Rpi-blb1 и Rpi-vnt1.3.
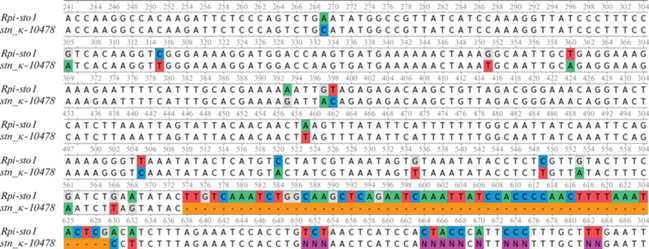
Нуклеотидная последовательность фрагмента Rpi-sto1 у образца S. stenotomum subsp. stenotomum к-10478 (GenBank: ON515750) в значительной степени сходна с референсной последовательностью СС-домена (coiled-coil) гена RB/blb1 у S. stoloni-ferum (GenBank: EU884421.1), представленной в информационно-поисковой базе BLAST. Обнаружены восемь SNP в области экзона и достаточно крупная делеция (в районе между позициями 574 и 629 референсного гена) в некодирующей области (рис. 3). В то же время гомологии между нуклеотидной последовательностью другого ампликона, полученного с использованием праймеров Rpi-blb1, и референсной последовательностью LRR-домена гена RB/blb1 (GenBank: EU884421.1) не обнаружено.
Рис. 3.
Выравнивание нуклеотидной последовательности фрагмента Rpi-sto1 у образца S. stenotomum subsp. stenotomum к-10478 (GenBank: ON515750) и фрагмента СС-домена референсной последовательности гена RB/blb1 у S. stoloniferum (GenBank: EU884421.1). Сокращенные названия видов для рис. 3–5 приведены согласно Z. Huaman, R. Ross, 1985 [38].
С использованием пары праймеров Rpi-vnt1.3 у большинства образцов примитивных видов были амплифицированы фрагменты ожидаемой длины около 600 пн. Выборочно фрагменты, амплифицированные у семи генотипов культурных видов, контрастных по устойчивости к фитофторозу, были выделены, клонированы и секвенированы (см. Материалы и методы). Полученные нами фрагменты представлены на рис. 4.
Рис. 4.
Выравнивание нуклеотидных последовательностей фрагмента СС-домена гомологов гена устойчивости Rpi-vnt1.3 у клонов культурных видов: S. ajanhuiri (к-9911) (GenBank: ON322726, ON322727), S. stenotomum subsp. goniocalyx (к-3558) (GenBank: ON322728, ON322729), S. phureja (к-9345 (GenBank: ON322730, ON322731), к-8873 (GenBank: ON322732, ON322733), к-17618 (GenBank: ON322734, ON322735)) и S. stenotomum subsp. stenotomum (к-9301 (GenBank: ON322736, ON322737), к-11020 (GenBank: ON322738, ON322739)). Нумерация нуклеотидов соответствует референсной последовательности гена Rpi-vnt1.3 (GenBank: FJ423046.1).

Полученные последовательности имеют сходство с фрагментами генов Rpi-vnt1.1 (GenBank: FJ423044), Rpi-vnt1.2 (GenBank: FJ423045) и Rpi-vnt1.3 (GenBank: FJ423046.1) вида S. venturii [16, 17] и псевдогенами типа Rpi-vnt1 у S. phureja (GU338337.1) и S. stenotomum (GU338321.1, GU338322.1 и GU338323.1) [39]. Также выявлено сходство с фрагментом гена, кодирующего RPP13-подобный белок в полногеномной последовательности удвоенного моноплоида DM1–3 516 R44 (GenBank: XM_015315064.1).
Всего было обнаружено три варианта последовательностей, степень сходства которых с референсной последовательностью гена Rpi-vnt1.3 (FJ423046.1) составляет 90.2, 97.7 и 94.9%. Первый вариант найден только у устойчивых к фитофторозу образцов: к-9911 S. ajanhuiri (GenBank: ON322726, ON322727), к-9345 (GenBank: ON322730, ON322731), к-8873 (GenBank: ON322733) и к‑17618 (GenBank: ON322735) S. phureja, к-11020 (GenBank: ON322739) S. stenotomum subsp. stenotomum. По сравнению с референсной последовательностью гена Rpi-vnt1.3 в исследуемом фрагменте обнаружено 52 SNP, среди которых 22 транзиции: двенадцать G ↔ A и десять Т ↔ С, а также 30 трансверсий: одиннадцать A ↔ T, четыре T ↔ G, семь G ↔ С и восемь А ↔ С. 28 нуклеотидных замен привели к замене аминокислот. Второй вариант последовательности фрагмента гена Rpi-vnt1.1 найден только у одного ампликона устойчивого образца к-11020 S. stenotomum subsp. stenotomum (GenBank: ON322738) и он отличался от аналогичного фрагмента в последовательности FJ423046.1 21 SNP, среди которых 10 транзиций (пять G ↔ A и пять Т ↔ С) и 11 трансверсий (три A ↔ T, две T ↔ G, пять G ↔ С и одна А ↔ С). 14 нуклеотидных замен оказались смысловыми. Несмотря на существенные различия, оба найденных нами варианта имеют традиционную для СС-домена структуру, в значительной степени сходную с доменом гена Rpi-vnt1.3. Известно, что аминокислотные последовательности СС-доменов включают повторы из семи аминокислот (гептады), причем в положениях 1 и 4 располагаются гидрофобные, а в положениях 5 и 7 – полярные аминокислотные остатки [40]. Предполагаемые аминокислотные последовательности, найденные у устойчивых к патогену образцов, имеют ту же структуру. Смысловых замен в ключевых точках гептад не обнаружено (рис. 5).
Рис. 5.
Выравнивание предполагаемых белковых последовательностей фрагментов coiled-coil (СС) домена Rpi-vnt1.3 (ACJ66594.1) и предполагаемых гомологичных белковых последовательностей изученных образцов видов S. ajanhuiri (к-9911) (GenBank: ON322726, ON322727), S. phureja (к-8873 (GenBank: ON322733), к-9345 (GenBank: ON3227230, ON322731) и к-17618 (GenBank: ON322735)) и S. stenotomum subsp. stenotomum (к-11020 (GenBank: ON322739, ON322738). Предполагаемые coiled-coil домены выделены рамками. Нумерация аминокислот соответствует последовательности Rpi-vnt1.3 (ACJ66596.1).

Третий вариант фрагмента гена Rpi-vnt1.1 встречался как у устойчивых, так и восприимчивых образцов и является фрагментом псевдогена, так как имеет делецию размером в 5 нуклеотидов в СС-домене, что приводит к сдвигу рамки считывания и, следовательно, к образованию стоп-кодона (рис. 4).
ОБСУЖДЕНИЕ
По результатам фитопатологического скрининга 71 коллекционного образца примитивных клубнеобразующих видов S. ajanhuiri, S. stenotomum subsp. goniocalyx, S. stenotomum subsp. stenotomum и S. phureja с использованием в качестве инокулюма изолята P. infestans MP1841 выделено 14 устойчивых и среднеустойчивых образцов. В целом частота устойчивых и среднеустойчивых генотипов в выборке соответствует опубликованным данным об устойчивости к фитофторозу образцов S. phureja [24] и других примитивных видов [11].
При помощи SCAR-маркеров Rpi-генов, гомологи которых были ранее идентифицированы у удвоенного моноплоида, клона S. phureja DM1-3 516 R44 [13, 26], мы проанализировали присутствие маркерных фрагментов этих генов и их изменчивость у образцов S. phureja и близкородственных культурных видов картофеля. Проанализирована связь устойчивости к фитофторозу представителей диких и культурных видов картофеля с наличием фрагментов генов RB/blb1, Rpi-blb2 и Rpi-vnt1.3, обеспечивающих защиту от фитофтороза у S. bulbocastanum и S. venturii [15–20]. Какой-либо связи между присутствием/отсутствием ампликонов SCAR-маркеров Rpi-генов и устойчивостью культурных видов картофеля к патогену P. infestans не выявлено.
Маркерный фрагмент гена R2-like не амплифицировался ни у одного из образцов примитивных видов, в то время как среди образцов североамериканских диких видов картофеля он был найден в большинстве образцов. Изначально этот ген был обнаружен у культурного картофеля (клон SW93-1015), в который он предположительно был интрогрессирован из S. demissum [41], ареал произрастания которого лежит в Северной и Центральной Америке. При сравнении последовательности R2-like (GenBank: FJ536323.1) с гомологами гена в полногеномной нуклеотидной последовательности референсного генома картофеля (NW_006239540.1) [42] были обнаружены существенные различия в нуклеотидных последовательностях. В частности, в области отжига праймеров маркера R2 area 1/2 находились несколько SNP (от трех до восьми в разных гомологах). В любом случае корреляции между наличием этого маркера и устойчивостью не обнаружено ни в одной из изученных групп.
Оба маркера другого эффективного гена устойчивости к фитофторозу RB/blb1 (Rpi-sto1 и Rpi-blb1) найдены только у одного устойчивого генотипа S. bulbocastanum и у двух восприимчивых S. stenotomum subsp. stenotomum, не выявлены ни у одного из исследованных образцов южноамериканских диких видов. В то же время фрагмент гена RB/blb1 (маркер Rpi-sto1) встречался часто у примитивных культурных видов. Ранее при исследовании семейства NBS-LRR, к которым относится большинство известных генов устойчивости к разным заболеваниям растений, включая фитофтороз, в референсном геноме картофеля (S. phureja DM1-3 516 R44) был обнаружен целый ряд последовательностей схожих по отдельным участкам с RB/blb1 [13]. Как и для гена R2-like, последовательности гена RB/blb1 и его гомологов в референсном геноме удвоенного моноплоида DM1–3 516 R44 имеют ряд существенных отличий. Предсказуемо самые значительные отличия были найдены в области отжига праймеров маркера Rpi-blb1 – до 15 нуклеотидных замен. С другой стороны, наличие обоих маркеров гена RB/blb1 у некоторых образцов S. bulbocastanum и S. stenotomum subsp. stenotomum свидетельствует о сходстве областей праймирования у этих образцов.
Маркерный фрагмент другого гена, изначально обнаруженного у S. bulbocastanum – Rpi-blb2, не найден в образцах диких североамериканских видов и обнаружен лишь у единичных примитивных культурных видов картофеля, что также можно объяснить нуклеотидным полиморфизмом между последовательностью гена S. bulbocastanum (GenBank: DQ122125.1) и его гомологами у примитивных видов, особенно в областях отжига праймеров. Степень сходства Rpi-blb2 и сходных последовательностей у DM1–3 516 R44 не превышает 90%.
Результаты исследований по поиску гомологов генов RB/blb1 и Rpi-blb2 у диких видов картофеля согласуются с опубликованными данными: гомологи гена Rpi-blb2 обнаружены у южноамериканских видов S. alandiae и S. okadae [2] и отсутствуют у североамериканских видов S. сardiophyllum, S. jamesii, S. lesteri, S. pinnatisectum, S. polyadenium, S. polytrichon, S. stoloniferum, S. trifidum и S. verrucosum [43]. Для дальнейшего изучения гомологов гена RB/blb1 у представителей клубнеобразующих видов рода Solanum следует использовать расширенную выборку образцов южноамериканских диких видов. В настоящем исследовании маркеры Rpi-sto1 и Rpi-blb1 не обнаружены у S. alandiae, что согласуется с данными Муратовой и соавт. [2], а также не найдены у образцов S. doddsii, S. kurtzianum, S. leptophyes, S. neocardenasii, S. sparsipilum, S. spegazzinii и S. yungasense. В то же время маркер RGA1F/R, амплифицируемый с другой парой праймеров, найден у южноамериканских видов S. chacoense и S. huancabambense, а также у североамериканских S. cardiophyllum, S. jamesii, S. lesteri, S. pinnatisectum, S. polyadenium, S. polytrichon, S. stoloniferum, S. trifidum и S. verrucosum [43].
С помощью праймеров, разработанных для маркирования эффективного гена устойчивости Rpi-vnt1, впервые обнаруженного у дикорастущего вида S. venturii, и его гомолога – гена Rpi-phu1 [16], почти у всех исследуемых образцов примитивных видов картофеля были получены маркерные фрагменты. Однако корреляции между наличием маркера и устойчивостью не наблюдалось. Авторы, ранее изучавшие гены устойчивости к фитофторозу у S. venturii, выявили целую серию близких по последовательностям генов, расположенных в едином кластере на хромосоме 9 [17]. Близко к этому кластеру также расположен известный ген устойчивости томата Tm-22 к вирусу мозаики, а также ген устойчивости к фитофторозу у S. phureja – Rpi-phu1. Высокая степень сходства Rpi-vnt1.1, Rpi-vnt1.3 и относительная их схожесть с Tm-22 позволила предполагать их общее происхождение. В настоящем исследовании мы проанализировали N-концевые последовательности coiled-coil (CC) домена у примитивных культурных видов картофеля и выявили несколько вариаций. Один из вариантов является псевдогеном, так как несет делецию в 5 нуклеотидов, что ведет к значительным изменениям в структуре белка. У устойчивых образцов S. ajanhuiri, S. phureja и S. stenotomum subsp. stenotomum найдены другие варианты последовательностей, не имеющие стоп-кодонов в данном участке. Предполагаемые аминокислотные последовательности этих фрагментов имеют характерную для СС-домена структуру и, возможно, являются частью функциональных генов. Одновременное присутствие нескольких вариантов сходных последовательностей в одном генотипе вполне объяснимо сложным строением локусов генов устойчивости типа CC-NBS-LRR с тандемно расположенными сходными последовательностями. На основе выявленных SNP у найденных вариантов в перспективе может быть разработан ПЦР-маркер для скрининга расширенной выборки примитивных видов картофеля из коллекции ВИР и оценки корреляции вариантных последовательностей подобных Rpi-vnt1 с устойчивостью к фитофторе.
Устойчивость к фитофторозу примитивных культурных видов S. ajanhuiri, S. stenotomum, S. phureja и диких видов картофеля, по-видимому, обусловлена разной генетической детерминацией. В настоящей работе для большинства устойчивых и среднеустойчивых образцов примитивных культурных видов обнаружена связь устойчивости к патогену с наличием одной из аллельных вариаций Rpi-vnt1. Биоинформационный поиск гомологов других известных генов устойчивости RB/blb1, Rpi-blb2, R2-like, изучение их полиморфизма и возможных связей с признаком у примитивных культурных видов картофеля является перспективным. Наиболее интересны для дальнейшего изучения образцы S. stenotomum subsp. stenotomum, устойчивые к фитофторозу, но не обладающие ни одним из исследованных SCAR-маркеров Rpi-генов. Они могут послужить источниками новых, ранее неизвестных генов устойчивости.
Результаты секвенирования фрагментов Rpi-генов получены с использованием оборудования ЦКП “Геномные технологии, протеомика и клеточная биология” ФГБНУ ВНИИСХМ.
Работа выполнена при поддержке Российского научного фонда, проект № 22-26-00111 “Гены устойчивости картофеля к фитофторозу в контексте эволюции культурных и диких клубненосных видов Solanum L.”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Ristaino J.B. Tracking historic migrations of the Irish potato famine pathogen, Phytophthora infestans // Microbes Infect. 2002. V. 4. № 13. P. 1369–1377. https://doi.org/10.1016/s1286-4579(02)00010-2
Муратова (Фадина) О.А., Бекетова М.П., Кузнецова М.А. и др. Южноамериканские виды Solanum alandiae Card. и S. okadae Hawkes et Hjerting как потенциальные источники генов устойчивости к фитофторозу картофеля // Труды по прикладной ботанике, генетике и селекции. 2020. Т. 181. № 1. С. 7–83. https://doi.org/10.30901/2227-8834-2020-1-73-83
Kim H.J., Lee H.R., Jo K.R. et al. Broad spectrum late blight resistance in potato differential set plants MaR8 and MaR9 is conferred by multiple stacked R genes // Theor. Appl. Genet. 2012. V. 124. P. 923–935. https://doi.org/10.1007/s00122-011-1757-7
Samen F.M., Secor G.A., Gudmestad N.C. Variability in virulence among asexual progenies of Phytophthora infestans // Phytopathology. 2003. V. 93. № 3. P. 293–304. https://doi.org/10.1094/PHYTO.2003.93.3.293
Bradeen J.M., Haynes K.G., Kole C. Introduction to potato // Genetics, Genomics and Breeding of Potatoes. Enfield, NH: Sci. Publ., 2011. P. 1–19.
Haas B., Kamoun S., Zody M. et al. Genome sequence and analysis of the Irish potato famine pathogen Phytophthora infestans // Nature. 2009. V. 461. P. 393–398. https://doi.org/10.1038/nature08358
Lee Y., Cho K.S., Seo J.H. et al. Improved genome sequence and gene annotation resource for the potato late blight pathogen Phytophthora infestans // Mol. Plant Microbe Interact. 2020. V 33. № 8. P. 1025–1028. https://doi.org/10.1094/MPMI-02-20-0023-A
Khavkin E.E. Plant–pathogen molecular dialogue: evolution, mechanisms and agricultural implementation // Rus. J. Plant. Physiol. 2021. V. 68. P. 197–211. https://doi.org/10.1134/S1021443721020072
Martin M.D., Vieira F.G., Ho S.Y.W. et al. Genomic characterization of a South American Phytophthora hybrid mandates reassessment of the geographic origins of Phytophthora infestans // Mol. Biol. Evol. 2016. V. 33. P. 478–491. https://doi.org/10.1093/molbev/msv241
Spooner D.M., Ghislain M., Simon R. et al. Systematics, diversity, genetics, and evolution of wild and cultivated potatoes // Bot. Rev. 2014. V. 80. P. 283–383. https://doi.org/10.1007/s12229-014-9146-y
Budin K.Z. Genetic foci of Solanum species, Petota Dumort, resistant to Phytophthora infestans (Mont.) De Bary // Gen. Resurces and Crop Evolution. 2002. V. 49. P. 229–235. https://doi.org/10.1023/A:1015549214779
Potato Genome Sequencing Consortium. Genome sequence and analysis of the tuber crop potato // Nature. 2011. V. 475. P. 189–195. https://doi.org/0.1038/nature10158
Lozano R., Ponce O., Ramirez M. et al. Genome-wide identification and mapping of nbs-encoding resistance genes in Solanum tuberosum group phureja // PLoS One. 2012, V. 7. № 4. https://doi.org/10.1371/journal.pone.0034775
Sliwka J., Jakuczun H., Lebecka R. et al. The novel, major locus Rpi-phu1 for late blight resistance maps to potato chromosome IX and is not correlated with long vegetation period // Theor. Appl. Genet., 2006. V. 113. № 4. P. 685–695. https://doi.org/10.1007/s00122-006-0336-9
Śliwka J., Jakuczun H., Kamiński P. et al. Marker-assisted selection of diploid and tetraploid potatoes carrying Rpi-phu1, a major gene for resistance to Phytophthora infestans // J. Appl. Genet. 2010. V. 51. P. 133–140. https://doi.org/10.1007/BF03195721
Foster S., Park T.H., Pel M. et al. Rpi-vnt1.1, a Tm-22 homolog from Solanum venturii, confers resistance to potato late blight // MPMI. 2009. V. 22. P. 589–600. https://doi.org/10.1094/MPMI-22-5-0589
Pel M.A., Foster S.J., Park T.H. et al. Mapping and cloning of late blight resistance genes from Solanum venturii using an interspecific candidate gene approach // MPMI. 2009. V. 22. P. 601–615. https://doi.org/10.1094/MPMI-22-5-0601
Song J., Bradeen J.M., Naess S.K. et al. Gene RB cloned from Solanum bulbocastanum confers broad spectrum resistance to potato late blight // Proc. Natl Acad. Sci. USA. 2003. V. 100. № 16. P. 9128–9133. https://doi.org/10.1073/pnas.1533501100
van der Vossen E.A., Gros J., Sikkema A. et al. The Rpi-blb2 gene from Solanum bulbocastanum is an Mi-1 gene homolog conferring broad-spectrum late blight resistance in potato // The Plant J. 2005. V. 44. P. 208–222. https://doi.org/10.1111/j.1365-313X.2005.02527.x
Orbegozo J., Roman M.L., Rivera C. et al. Rpi-blb2 gene from Solanum bulbocastanum confers extreme resistance to late blight disease in potato // Plant Cell Tiss. Organ. Cult. 2016. V. 125. P. 269–281. https://doi.org/10.1007/s11240-016-0947-z
Zhu S., Li Y., Vossen J.H. et al. Functional stacking of three resistance genes against Phytophthora infestans in potato // Transgenic Res. 2012. V. 21. № 1. P. 89–99. https://doi.org/10.1007/s11248-011-9510-1
Aguilera-Galvez C., Champouret N., Rietman H. et al. Two different R gene loci co-evolved with Avr2 of Phytophthora infestans and confer distinct resistance specificities in potato // Stud. Mycol. 2018. V. 89. P. 105–115. https://doi.org/10.1016/j.simyco.2018.01.002
Рогозина Е.В., Гурина А.А. Состав коллекции примитивных культурных видов секции Petota Dumort. рода Solanum L. и актуальные направления их исследования // Труды по прикладной ботанике, генетике и селекции. 2020. Т. 181. № 3. С. 190–202. https://doi.org/10.30901/2227-8834-2020-3-190-202
Gabriel J., Plata G., Cadima X., Franco J. Solanum phureja Juz et Buk.: Valuable source of genetic resistance to potato late blight [Phytophthora infestans (Mont.) de Bary] // Revista Latinoamericana de la Papa. 2013. V. 17. P. 131–142.
Blossei J., Uptmoor R., Thieme R. et al. Insights into the genetic basis of the pre-breeding potato clones developed at the Julius Kühn Institute for high and durable late blight resistance // Plant Genetic Resources: Characterization and Utilization. 2021. V. 1. № 4. https://doi.org/10.1017/S1479262121000447
Jupe F., Pritchard L., Etherington G.J. et al. Identification and localisation of the NB-LRR gene family within the potato genome // BMC Genomics. 2012. V. 13. № 75. https://doi.org/10.1186/1471-2164-13-75
Hawkes J.G. The Potato: Evolution, Biodiversity and Genetic Resources. London: Belhaven Press, 1990. 259 p.
Мироненко Н.В., Рогозина Е.В., Гурина А.А. и др. Дикие родичи и межвидовые гибриды картофеля – исходный материал для селекции на устойчивость к золотистой нематоде // Труды по прикладной ботанике, генетике и селекции. 2020. Т. 181. № 4. С. 173–184. https://doi.org/10.30901/2227-8834-2020-4-173-184
Brylińska M., Śliwka J. Laboratory assessment of potato resistance to Phytophthora infestans // Plant Breed. Seed Sci. 2017. V. 76. P. 17–23. https://doi.org/10.1515/plass-2017-00016
Хютти А.В., Рыбаков Д.А., Гавриленко Т.А., Афанасенко О.С. Устойчивость к возбудителям фитофтороза и глободероза современного сортимента семенного картофеля и его фитосанитарное состояние в различных агроклиматических зонах европейской части России // Вавил. журн. генетики и селекции. 2020. Т. 24. № 4. С. 363–375. https://doi.org/10.18699/VJ20.629
Vleeshouwers V.G., Van Dooijeweert W., Keizer L.C. et al. A laboratory assay for Phytophthora infestans resistance in various Solanum species reflects the field situation // Eur. J. Plant Pathol. 1999. V. 105. P. 241–250. https://doi.org/10.1023/A:1008710700363
Gavrilenko T., Antonova O., Shuvalova A. et al. Genetic diversity and origin of cultivated potatoes based on plastid microsatellite polymorphism // Genet. Resour. Crop Evol. 2013. V. 60. № 7. P. 1997–2015. https://doi.org/10.1007/s10722013-9968-1
Haesaert G., Vossen J.H., Custers R. et al. Transformation of the potato variety Desiree with single or multiple resistance genes increases resistance to late blight under field conditions // CROP PROTECTION. 2015. V. 77. P. 163–175. https://doi.org/10.1016/J.CROPRO.2015.07.018
Wang M., Allefs S., van den Berg R.G. et al. Allele mining in Solanum: Conserved homologues of Rpi-blb1 are identified in Solanum stoloniferum // Theor. Appl. Genet. 2008. V. 116. № 7. P. 933–943. https://doi.org/10.1007/s00122-008-0725-3
Lenman M., Ali A., Mühlenbock P. et al. Effector-driven marker development and cloning of resistance genes against Phytophthora infestans in potato breeding clone SW93-1015 // Theor. Appl. Genet. 2016. V. 129. № 1. P. 105–115. https://doi.org/10.1007/s00122-015-2613-y
Алпатьева Н.В., Антонова О.Ю., Радченко Е.Е. и др. ПЦР-диагностика вредных организмов гуара: (методические указания). Санкт-Петербург: ВИР, 2019. 36 с. https://doi.org/10.30901/978-5-907145-44-3
Tamura K., Stecher G., Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis version 11 // Mol. Biol. Evol. 2021. V. 38. № 7. P. 3022–3027. https://doi.org/10.1093/molbev/msab120
Huaman Z., Ross R. Updated listing of potato species names, abbreviations and taxonomic status // Am. Potato J. 1985. V. 62. № 11. P. 629–641. https://doi.org/10.1007/BF02854438
Pel M.A., Jacobsen E., Van der Vossen E.A.G et al. Chapter 4: The potato late blight resistance alleles Rpi-vnt1.1, Rpi-vnt1.2 and Rpi-vnt1.3 from S. venturii are not widely spread across Solanum section Petota and have evolved most probably by Illegitimate recombination // Mapping, Isolation and Characterization of Genes Responsible for Late Blight Resistance in Potato: PhD Thesis. The Netherlands: Wageningen Univ., 2010.
Mason J.M., Arndt K.M. Coiled coil domains: stability, specificity, and biological implications // ChemBioChem. 2004. V. 5. P. 170–176. https://doi.org/10.1002/cbic.200300781
Plich J., Tatarowska B., Lebecka R. et al. R2-like gene contributes to resistance to Phytophthora infestans in Polish potato cultivar Bzura // Am. J. Potato Res. 2015. V. 92. P. 350–358. https://doi.org/10.1007/s12230-015-9437-9
Veilleux R.E. Genetic stocks used for potato genome sequencing // The Potato Genome Compendium of Plant Genomes. Springer, 2017. https://doi.org/10.1007/978-3-319-66135-3_4
Tiwari J.K., Devi S., Sharma S. et al. Allele mining in Solanum germplasm: Cloning and characterization of RB-homologous gene fragments from late blight resistant wild potato species // Plant Mol. Biol. Reporter. 2015. V. 33. № 5. P. 1584–1598. https://doi.org/10.1007/s11105-015-0859-9
Дополнительные материалы
- скачать ESM.docx
- Приложение 1.
Таблица 1. Коллекционные образцы картофеля, использованные в работе.


