Генетика, 2022, T. 58, № 2, стр. 137-147
Zvezda – новое подсемейство Tc1-подобных транспозонов в геномах Asterozoa
Л. В. Пузакова 1, *, М. В. Пузаков 1, 2
1 Федеральный исследовательский центр Институт биологии южных морей им. А.О. Ковалевского
Российской академии наук
299011 Севастополь, Россия
2 Севастопольский государственный университет
299053 Севастополь, Россия
* E-mail: kvluda@yandex.ru
Поступила в редакцию 29.04.2021
После доработки 02.06.2021
Принята к публикации 23.06.2021
- EDN: DDPPWA
- DOI: 10.31857/S001667582201009X
Аннотация
Мобильные генетические элементы оказывают существенное влияние на эволюцию геномов живых организмов. Эукариотические МГЭ подразделяют на два класса – ретротранспозоны и ДНК-транспозоны. ITm-транспозоны являются одной из широко распространенных групп ДНК-транспозонов и обнаруживаются практически у всех организмов. В настоящей работе мы исследовали распространенность, структуру и эволюцию элементов с необычно длинным для ITm-транспозонов каталитическим доменом DD46E. Данные элементы были обнаружены только в подтипе Asterozoa. Была изучена их копийность, структура, возможная функциональность транспозазы, а также филогенетические взаимоотношения с другими представителями ITm-транспозонов. Показано, что обнаруженная нами группа транспозонов, которую мы назвали Zvezda, является подсемейством Tc1-подобных транспозонов.
Мобильные генетические элементы (МГЭ) являются неотъемлемой частью геномов как эукариотических, так и прокариотических организмов. Они представляют собой фрагменты ДНК, способные к перемещениям внутри хозяйского генома. В результате транспозиций МГЭ могут увеличивать число собственных копий, а также оказывать существенное влияние на структуру и функционирование всего генетического аппарата [1]. Более поздние исследования свидетельствуют о том, что мобильные генетические элементы могут служить источником новых генов [1].
Неоднократно показано, что активность МГЭ может возрастать в ответ на воздействие физических, химических и биологических факторов окружающей среды [2–4]. Активизация МГЭ в ответ на стресс вызывает дестабилизацию генома и всевозможные мутации, которые могут стать “сырьем” для действия движущего отбора. Таким образом, МГЭ играют немаловажную роль в адаптивных и эволюционных процессах [2–4].
В основе классификации МГЭ лежат различия в их структурно-функциональных особенностях. На сегодняшний день все эукариотические МГЭ подразделяются на два класса – ретротранспозоны и ДНК-транспозоны [1, 5]. Ретротранспозоны, или класс I, кодируют обратную транскриптазу и перемещаются посредством создания собственной копии и еe вставки в какой-либо участок генома. Такой тип перемещения называется “копирование–вставка” и он позволяет ретротранспозонам быстро увеличивать число копий. ДНК-транспозоны, или класс II, кодируют транспозазу. Этот тип элементов перемещается путем вырезания собственной копии и ее последующей вставки в какой-либо другой участок генома. Такой тип перемещения называется “вырезание–вставка”. Несмотря на то что механизм увеличения количества копий транспозонов не описан, все же ДНК-транспозоны способны довольно эффективно “оккупировать” хозяйские геномы.
Так, например, одна из распространенных групп ДНК-транспозонов – IS630/Tc1/mariner (ITm) насчитывает в геномах до нескольких сотен и даже тысяч копий [6–8]. Представители ITm-транспозонов присутствуют практически во всех живых организмах [9]. Длина элементов колеблется от 1 до 3 тыс. пар нуклеотидов (тпн), но может достигать и 6000 пн. ITm-элементы ограничены концевыми инвертированными повторами (КИП), протяженность которых очень вариабельна и колеблется от 20 до 1900 пн. У некоторых ITm-транспозонов имеются еще и субконцевые инвертированные повторы (СИП), длиной от 175 до 1403 пн [10]. Неразрывная открытая рамка считывания (ОРС) кодирует фермент транспозазу, протяженность которого варьирует в среднем от 350 до 650 аминокислотных остатков (а.о.). ITm-транспозаза имеет ДНК-связывающийся домен (PAIRED) в N-концевой части и каталитический (DDE/D) домен в С-концевой части [11]. Домен PAIRED состоит из шести α-спиралей. Первая триада α-спиралей носит название PAI-субдомена, вторая триада – RED-субдомена. Домен PAIRED обеспечивает сайт-специфическое связывание с ДНК-мищенью и с КИП. Между PAI- и RED-субдоменами расположен GRPR-подобный мотив, функцией которого, как предполагается, является связывание PAIRED-домена с малой бороздкой ДНК дуплицируемого сайта встраивания ТА [12]. DDE/D-домен обладает эндонуклеазной и лигирующей активностью и обеспечивает вырезание и вставку МГЭ. Также для ITm-транспозазы характерно наличие сигнала ядерной локализации (NLS-сигнала), который, как предполагается, обеспечивает проникновение транспозазы из цитоплазмы в ядро [13, 14].
Классификация ITm-транспозонов достаточно сложная, в некоторых моментах противоречивая, и в последние годы претерпела значительные дополнения и изменения [15, 16]. Обобщая последние данные, можно выделить несколько основных групп: Tc1-подобные элементы (TLE/DD34-38E), mariner-подобные элементы (MLE/DD34D), maT/ DD37D, Visitor/DD41D, Guest/DD39D, mosquito/ DD37E, pogo/DDxD и IS630/DDxE [8, 10, 11, 16–18]. Группу элементов pogo/DDxD определяют как отдельное надсемейство [16]. Оставшиеся перечисленные группы (за исключением бактериальных транспозонов IS630/DDxE) объединяют в надсемейство Tc1/mariner. Еще выделяют четыре малых семейства: Tec/DD34E, HvSm/DD34E, L31/DD37E и TBE/DD34E, которые не входят в вышеперечисленные группы [15, 19].
В настоящей работе мы изучали распространенность, структуру и эволюцию ДНК-транспозонов надсемейства Tc1/mariner с необычно длинным каталитическим доменом DD46E, которые были названы нами Zvezda.
МАТЕРИАЛЫ И МЕТОДЫ
Поиск DD46E-транспозонов
Для поиска ДНК-транспозонов с каталитическим доменом DD46E был использован tBLASTn со стандартными настройками (https://blast.ncbi.nlm.nih.gov/Blast.cgi). В качестве матрицы для поиска были взяты аминокислотные последовательности транспозаз Mariner-18_CGi_p (Repbase) и Zvezda-1_ARub/CABPRM03. Полногеномные последовательности ДНК иглокожих были взяты из базы данных NCBI (https://www.ncbi.nlm.nih.gov) (табл. 1). Для того чтобы выявить полные нуклеотидные последовательности МГЭ, гомологичные транспозазе последовательности с наивысшей идентичностью к матрице были взяты из соответствующих скаффолдов вместе с фланкирующими областями протяженностью 3000 пн. Полноразмерная последовательность каждого выявленного элемента использовалась для подсчета присутствующих в геноме копий. Копии протяженностью менее 10% от длины полноразмерного МГЭ не учитывали при подсчете. Копии протяженностью от 95 до 100% от длины полноразмерного МГЭ считались полноразмерными. Копии протяженностью от 10 до 100% от длины полноразмерного МГЭ подсчитывались как общее количество копий.
Таблица 1.
Характеристика сборок геномных последовательностей, использованных для поиска транспозонов Zvezda
| Класс | Вид | Место получения образца |
Дата получения образца | Название геномного проекта | Идентификатор сборки | Уровень сборки | N50 | Покрытие генома | Размер сборки, пн |
|---|---|---|---|---|---|---|---|---|---|
| Asteroidea | Asterias rubens | – | 2018-02-23 | CABPRM03 | GCA_902459465 | Хромосомы | 20 558 067 | 103× | 417 601 740 |
| – | 2018-02-23 | CABPRO01 | GCA_902459445 | Скаффолды | 161 267 | 103× | 390 768 305 | ||
| Pisaster ochraceus | USA: California | 2016-11-12 | JAAFGO01 | GCA_010994315 | Хромосомы | 20 188 303 | 212.8× | 401 943 971 | |
| Patiria miniata | USA: California | 2003 | AKZP01 | GCA_000285935 | Скаффолды | 52 614 | 15.0× 454; 70× Illumina | 811 028 858 | |
| USA: California | 2019-09 | JADOBP01 | GCA_015706575 | Скаффолды | 23 093 800 | 150.0× | 608 344 308 | ||
| Acanthaster planci | Japan: Okinawa, Motobu | 2013-05-28 | BDGF01 | GCA_001949145 | Скаффолды | 1 521 119 | 46× | 383 860 178 | |
| Australia: Great Barrier Reef | 2013-02-04 | BDGH01 | GCA_001949165 | Скаффолды | 916 880 | 40× | 383 525 304 | ||
| Australia: Great Barrier Reef | 2013-02-04 | BBNW01* | GCA_000950615 | Скаффолды | 1 389 596 | 50× | 2 584 918 | ||
| Patiriella regularis | – | – | CYSQ01 | GCA_900067625 | Скаффолды | 557 | 23× | 949 333 185 | |
| Ophiuroidea | Ophionereis fasciata | – | – | CZLG01 | GCA_900067615 | Скаффолды | 484 | 13× | 1 184 528 790 |
| Ophiothrix spiculata | USA: California | – | JXSR01 | GCA_000969725 | Скаффолды | 72 780 | 275.0× | 2 764 315 159 |
Анализ последовательностей
Границы предполагаемых ОРС определяли с помощью ORF Finder (https://www.ncbi.nlm.nih.gov/ orffinder/) и далее уточняли визуально. Последовательность сигнала ядерной локализации (NLS) выявляли с помощью PSORT (https://www.genscript.com). ДНК-связывающий мотив PAIRED определяли, анализируя вторичную структуру, предсказанную с помощью PSIPRED v4.0 [20]. Мотив GRPR-типа, а также DDE/D-домен идентифицировали визуально.
Филогенетический анализ
Для филогенетического анализа были взяты аминокислотные последовательности транспозаз, относящиеся к разным группам ITm-транспозонов (табл. 2), и последовательности, принадлежащие транспозонам Zvezda (табл. 3). Множественное выравнивание аминокислотных последовательностей было выполнено с помощью MUSCLE [21] с использованием стандартных настроек. Филогенетический анализ проводили с использованием пакета программ MEGA X [21].
Таблица 2.
ДНК-транспозоны IS630/Tc1/mariner, используемые в филогенетическом анализе
| Семейство | Элемент | Источник | Семейство | Элемент | Источник |
|---|---|---|---|---|---|
| Guest DD39D |
Soymar1 Br-oleracea Ca-sativa Phyllostachys edulis Pisum sativum |
AF078934 XP013589454 XP010462775 ADP24264 AAX51974 |
Visitor DD41D |
Crmar2.5 Apismar4.1 Mariner-12_CGi Lsra_Ap rosa_Ae |
AAK61417 ABLF02014333 Repbase [11] [11] |
| maT DD37D |
Bmmar1 Bmmar6 Cemar6 CbmaT4 |
U47917 AF461149 LK928390 AC084524 |
Mosquito DD37E |
Ae-atropalpus1 An-gambiae1 PrDD37E1 |
AF377999 AF378002 DQ138288 |
| Traveler DD35E | TR-Habu TR-Xetr TR-Tafu TR-Onmy TR-Xihe |
[27] [27] [27] [27] [27] |
L18 DD37E |
Mariner-18_CGi L18-NVec L18-APar L18-HMag L18-AOce |
Repbase NW_001834331 JXUT01105677 EQ256867 NXFZ01003443 |
| Intruder DD38E |
IT_Cf IT_Sl IT_Sp IT_Rm IT_At |
[29] [29] [29] [29] [29] |
Incomer DD36E |
In-MHra In-Sale In-MLuc In-CVar In-RMar |
[28] [28] [28] [28] [28] |
| Tc1 DD34E |
Tc1 Passport Quetzal Frog_Prince Sleeping_Beauty |
X01005 CAB5137 AAB02109 AAP49009 AFR53956 |
TRT DD37E |
DrTRT CmpTRT TrTRT SsTRT HbTRT |
[30] [30] [30] [30] [30] |
| ctmTLE DD34E |
Mariner-5_CGi Mariner-23_CGi Mariner-8_CGi Mariner-14_CGi |
Repbase Repbase Repbase Repbase |
TLEWI DD36E |
TLEWI-1_CGi TLEWI-2_CGi TLEWI-2_MPh TLEWI-4_MYe TLEWI-1_BPl TLEWI-1_NSu |
[26] [26] [26] [26] [26] [26] |
| MLE DD34D |
Dmmar1 Hsmar1 Famar1 Bytmar1 Tvmar1 |
AAA28678 AAC52010 AAO12863 CAD45367 AAP45328 |
IS630 DDxE |
RS(alfa) IS630Ss IS630Se |
X02581 X05955 NP_073225 |
Таблица 3.
ДНК-транспозоны Zvezda у Asterozoa
| Вид | Название транспозона | Длина элемента, пн | Длина КИП | Длина СИП | Транспозаза, а.о. | Общее число копий | Число полноразмерных копий | Число потенциально функциональных копий |
|---|---|---|---|---|---|---|---|---|
| Patiria miniata | Zvezda-1_PMin/AKZP01 | 3742 | 12/12 | н/о | 340 | 59 | 1 | 1 |
| Zvezda-1_PMin/JADOBP01 | 757 | н/о | н/о | 248 | 12 | 0 | 0 | |
| Pisaster ochraceus | Zvezda-1_POch | 3977 | 17/17 | н/о | 339 | 18 | 1 | 0 |
| Acanthaster planci | Zvezda-1_APla/BDGF01 | 1056 | н/о | н/о | 334 | 32 | 0 | 0 |
| Zvezda-1_APla/BDGH01 | 1785 | 38/38 | н/о | 355 | 32 | 1 | 0 | |
| Patiriella regularis | Zvezda-1_PReg | 1811 | 26/26 | 52/52 | 353 | 15 | 1 | 1 |
| Asterias rubens | Zvezda-1_ARub/CABPRM03 | 1777 | 31/31 | 46/46 | 327 | 3 | 1 | 1 |
| Zvezda-1_ARub/CABPRO01 | 1772 | 31/31 | 46/46 | 289 | 3 | 2 | 0 | |
| Ophionereisfasciata | Zvezda-1_OFas | 1086 | н/о | н/о | 361 | 2 | 0 | 0 |
| Ophiothrix spiculata |
Zvezda-1.1_OSpi | 1542 | н/о | 156/– | 366 | 493 | 1 | 0 |
| Zvezda-1.2_OSpi | 3445 | 36/36 | 156/159 | 358 |
Выявление случаев горизонтального переноса
Для выявления случаев горизонтального переноса у многоклеточных была использована аминокислотная последовательность Zvezda-1_ARub/ CABPRM03. Для поиска использовали программу BLASTn (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Поскольку никаких соответствий с транспозонами Zvezda найдено не было, то и дальнейший поиск в соответствии с рекомендованным алгоритмом [22] не осуществлялся.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Распространенность транспозона Zvezda у Metazoa
При исследовании разнообразия ITm-транспозонов у Asterias rubens нами был обнаружен МГЭ с необычно длинным каталитическим доменом – DD46E. Для того чтобы выяснить, единичный ли это мутантный вариант или представитель уникальной эволюционной группы ITm-транспозонов, мы изучили распространенность данного МГЭ. ITm-транспозоны с DD46E-доменом (названные нами Zvezda) обнаружились у семи организмов (т.е. у всех, имеющих в NCBI полногеномные последовательности ДНК), принадлежащих к двум классам – морским звездам (Asteroidea) и офиурам (Ophiuroidea) (табл. 3). Эти два таксона объединены в подтип Asterozoa, который отделился от остальных иглокожих примерно 541 млн лет назад (по разным оценкам 458–625 млн лет назад, http://www.timetree.org/) [23]. При исследовании всех Metazoa на предмет гомологии к DD46E-транспозону A. rubens ни у кого, кроме Asterozoa, подобный транспозон обнаружен не был. Для объяснения данного феномена можно предложить две версии – либо ДНК-транспозон Zvezda возник непосредственно в геноме предка Asterozoa, либо встроился в геном предка Asterozoa в результате горизонтального переноса от неизвестного нам организма, который на сегодняшний день либо потерял его в процессе эволюции, либо вымер, либо его полная геномная последовательность ДНК еще не определена. Так или иначе, но ДНК-транспозон Zvezda не получил распространения по древу жизни и присутствует лишь в узкой филогенетической группе, внутри которой распространился, по всей видимости, в результате дивергенции видов. Отсутствие ДНК-транспозона Zvezda у каких-либо групп организмов, кроме Asterozoa, свидетельствует об отсутствии случаев горизонтального переноса.
Анализ числа копий ДНК-транспозона Zvezda у Asterozoa показал значительную неоднородность среди изученных организмов (табл. 3). У двух видов число копий крайне низкое (2–3 копии). Четыре вида имели от 15 до 60 копий. И только один вид – Ophiothrix spiculata – имеет большое число копий – 493, при этом подавляющая часть этих копий сохранила КИП при делетированной центральной части. Согласно модели жизненного цикла, увеличение числа копий происходит на стадии амплификации в жизненном цикле МГЭ [24]. Однако мы не обнаружили в геноме O. spiculata ни одной потенциально функциональной копии элемента Zvezda, следовательно в настоящее время данный транспозон не может находиться на стадии амплификации. Очевидно, транспозон Zvezda был активен у предка Asterozoa, поэтому все Asterozoa в настоящий момент имеют малое число копий, большинство из которых повреждены и нефункциональны вследствие постепенной деградации. Вероятно, O. spiculata имеет на сегодняшний день так много копий по причине еще одного всплеска активности, произошедшего позже. Причиной второй волны активности мог быть рестарт жизненного цикла – одна из копий могла вновь стать активной вследствие случайных мутаций и вызвать “взрыв” перемещений. Также нельзя исключить и возможность кросс-индукции. Транспозаза какого-либо активного МГЭ, проникшего в геном O. spiculata, могла воздействовать и на копии элемента Zvezda, индуцируя их активность. Явление кросс-мобилизации уже было описано ранее [25]. Было обнаружено, что hobo-транспозаза способна взаимодействовать с терминальными последовательностями не только элемента hobo, но и Hermes. Хотя у ITm-транспозонов явление кросс-мобилизации не известно, такую возможность нельзя исключить, тем более что КИП, необходимые для перемещения, присутствуют практически во всех копиях.
Zvezda – новое подсемейство Tc1-подобных транспозонов
Для того чтобы определить эволюционные отношения элементов Zvezda/DD46E и ITm-транспозонов мы провели филогенетический анализ, в который были включены 11 транспозаз Zvezda/ DD46E (с сохранившимся DDE-доменом) и 45 МГЭ, представляющих различные группы ITm-транспозонов (табл. 2). В результате было установлено, что элементы Zvezda/DD46E формируют отдельную ветвь, которая располагается в одной кладе с Tc1-подобными элементами (рис. 1). Таким образом, элементы Zvezda/DD46E являются новым подсемейством Tc1-подобных элементов.
Рис. 1.
Эволюционные взаимоотношения транспозонов Zvezda и ДНК-транспозонов ITm. Филогенетический анализ выполнен в программе MEGA X c помощью метода максимального правдоподобия. Используемая модель WAG + + G + I + F. Бутстрэп 1000.
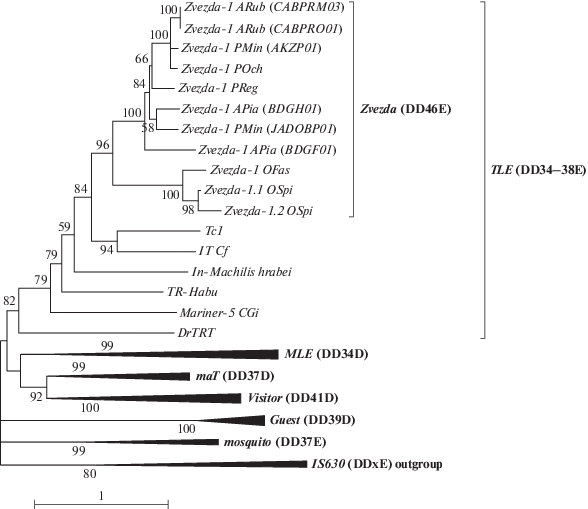
На филогенетическом дереве видно, что транспозоны Zvezda объединяются в одну монофилетическую группу с высокой бутстрэп-поддержкой. В последние годы семейство Tc1-подобных элементов хорошо изучено и в нем найден ряд семейств, которые имеют домен, отличающийся от классического домена DD34E. Это, например, TLEWI/DD36E, Traveler/DD35E, Incomer/DD36E, Intruder/DD38E, TRT/DD37E [26–30]. Все они имеют не столь существенные отличия в размере домена от классического DD34E, в то время как у ДНК-транспозона Zvezda протяженность последовательности между вторым D и E сотавляет 46 а.о. (DD46E). Филогенетический анализ, в который были включены все известные на данный момент Tc1-подобные элементы, подтвердил принадлежность транспозонов Zvezda к отдельному подсемейству (рис. 2).
Рис. 2.
Эволюционные взаимоотношения транспозонов Zvezda и Tc1-подобных транспозонов. Транспозоны морских звезд отмечены черным кругом, транспозоны офиур – черным квадратом. Филогенетический анализ выполнен в программе MEGA X c помощью метода максимального правдоподобия. Используемая модель LG + G + I. Бутстрэп 1000.

Структурные особенности ДНК-транспозона Zvezda
У большинства видов полноразмерные копии элемента Zvezda были обнаружены в единственном числе (табл. 3). Полноразмерными мы считали копии, обладающие обоими КИП и неповрежденной транспозазой. У A. rubens в геномной сборке CABPRO01 было обнаружено два полноразмерных элемента. У Ophionereis fasciata и Ophiothrix spiculata полноразмерных копий не было обнаружено.
В среднем длина полноразмерных элементов Zvezda колеблется от 1700 до 1800 пн (табл. 3), что характерно для Tc1-подобных элементов. Однако есть случаи, выбивающиеся из общей тенденции. У O. spiculata длина элемента – 3445 пн, у Patiria miniata (AKZP01) – 3742 пн, у Pisaster ochraceus – 3977 пн. В последних двух случаях КИП находятся далеко от гена транспозазы, вследствие этого общая длина элемента сильно увеличена. У O. spiculata внутри гена транспозазы присутствуют вставки, увеличивающие длину элемента, а также один из КИП находится значительно дальше от гена транспозазы, чем обычно (табл. 3).
КИП были найдены у шести видов Asterozoa, СИП – у трех видов (табл. 3). Длина КИП колебалась от 26 до 38 пн. У Patiria miniate (AKZP01) и Pisaster ochraceus КИП были нетипично короткие, 12/12 и 17/17 пн соответственно. При этом они отстояли от гена транспозазы довольно далеко, в связи с чем общая длина элемента у этих видов превышает среднюю. СИП имели длину 46/46, 52/52, а в случае O. spiculata СИП были необычно длинные – 156/159 пн. Оба исследованных вида офиур имеют длинные СИП. У некоторых видов морских звезд также есть СИП, но значительно короче. Возможно, морские звезды потеряли СИП в процессе эволюции.
Длина полноразмерных транспозаз оказалась обычная для этого семейства транспозонов, а именно от 327 до 366 а.о. (табл. 3). Неповрежденные транспозазы имеются у четырех видов. У трех видов транспозазы имеют повреждения – стоп-кодоны и делеции.
Чтобы лучше понять, являются ли транспозазы потенциально функциональными, мы изучили такие их особенности, как PAIRED-домен, AT-крюк, каталитический домен DDE/D и NLS (рис. 3). Во всех транспозазах был обнаружен PAIRED-домен. Однако у МГЭ Zvezda-1_APla/ BDGF01 и Zvezda-1_POch первая альфа-спираль PAI-субдомена отсутствует. Также у Zvezda-1_APla/ BDGF01 третья альфа-спираль RED-домена отсутствует. ARub/CABPRO01 и Zvezda-1_PMin/JADOBP01 вообще не имеют PAI-субдомена.
Рис. 3.
Множественное выравнивание транспозаз элемента Zvezda. Серым цветом выделен PAIRED-домен, состоящий из шести α-спиралей, жирным шрифтом с подчеркиванием выделен GRPR-подобный мотив, белым шрифтом на черном фоне – DDE-домен.
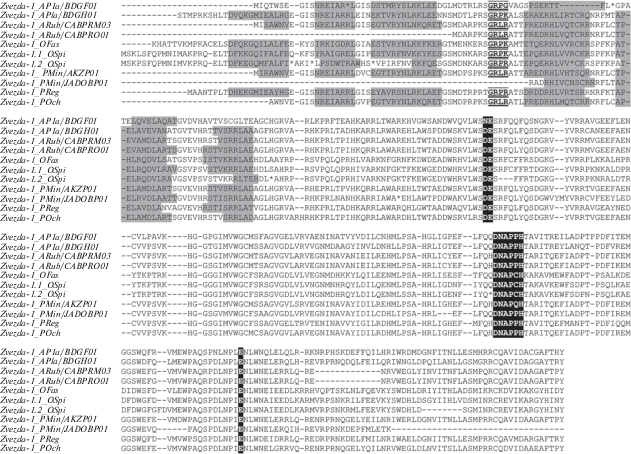
Последовательность NLS не была обнаружена нами ни в одной из транспозаз Zvezda, что вполне закономерно, учитывая работы коллег, в которых показана редкая встречаемость NLS в ITm-транспозонах.
АТ-крюк обнаружен у всех исследованных видов, за исключением Zvezda-1_PMin/JADOBP01. Он расположен на своем месте, между PAI- и RED-субдоменами.
Каталитический домен DD46E был обнаружен во всех транспозазах, но в аминокислотной последовательности транспозазы элемента Zvezda-1_ APla/BDGF01 первый аспартат DDE/D-домена был замещен на аспарагин (N); кроме того, промежуток между вторым D (аспартат) и Е (глутамат) составляет 47 аминокислотных остатков, а не 46, как во всех остальных транспозазах. Кроме того, данный транспозон имеет стоп-кодоны и сдвиги рамки считывания. Так же у Zvezda-1_ PMin/JADOBP01 каталитический домен был изменен (DD42E). Однако в других сборках этих же видов у мобильных элементов Zvezda-1_APla/ BDGH01 и Zvezda-1_PMin/AKZP01 присутствует классический DD46E-домен.
Подытоживая результаты проведенного анализа, потенциально функциональными можно назвать только три элемента – Zvezda-1_PMin/AKZP01, Zvezda-1_PReg и Zvezda-1_ARub/CABPRM03 (рис. 3). Сравнивая полученные данные с имеющимися данными по другим ITm-транспозонам, можно сделать вывод, что только незначительное их количество имеют функциональную транспозазу и обладают транспозиционной активностью [31–34]. Этот факт связывают с тем, что пик активности ITm-транспозонов был в далеком прошлом, а следовательно “жизненный цикл” элементов проходит свою завершающую стадию [24]. По этой причине у большинства ITm-транспозонов последовательности, кодирующие транспозазу, нарушены делециями, стоп-кодонами и сдвигами ОРС. Наличие одиночных полноразмерных/потенциально функциональных копий элемента Zvezda при наличии делетированных копий в геномах морских звезд и офиур может свидетельствовать о древности его происхождения.
Внутровидовое разнообразие
Были исследованы семь видов подтипа Asterozoa, полногеномные последовательности которых на момент исследования были представлены в NCBI (табл. 3). У всех семи видов был обнаружен мобильный элемент с каталитическим доменом DD46E. У Acanthaster planci имелось три сборки, одна из которых (GCA_000950615.1) не имела сходства с матрицей, в качестве которой мы использовали аминокислотную последовательность элемента Mariner-18_CGi, обнаруженного у Crassostrea gigas [19]. У Asterias rubens и Patiria miniatа имелось по две сборки.
Имея в доступе по две сборки видов Acanthaster planci, Patiria miniata и Asterias rubens, мы получили интересную возможность провести небольшое сравнение внутривидового разнообразия, хоть и на столь малом количестве особей. Мы сравнивали общее число транспозонов и их структуру.
Так, у Asterias rubens в двух разных сборках оказались удивительно воспроизводимые результаты – общее число транспозонов в сборке CABPRM03 и сборке CABPRO01 оказалось 3, полноразмерных копий 1 и 2 соответственно, терминальные и субтерминальные повторы оказались в обеих сборках абсолютно идентичными по длине и составили 31 и 46 пн соответственно; общая длина элементов отличалась несущественно – 1777 и 1772 пн, соответственно. Поскольку дата получения обоих образцов указана одна и та же, по всей видимости образцы были взяты в одном и том же месте, из одной популяции (табл. 1). Вероятно, данная популяция довольно гомогенна по количеству и структурным особенностям элемента Zvezda, что свидетельствует о том, что в данный момент этот транспозон не активен. Это подтверждается и малым количеством копий транспозона Zvezda в геноме Asterias rubens.
У Acanthaster planci в обеих сборках оказалось по 32 копии транспозона Zvezda. Образцы были взяты из разных регионов – A. planci (BDGF01) собран в Японии, а A. planci (BDGH01) в Австралии в одном и том же году. Равное число копий можно считать как совпадением, так и признаком однородности популяций. Сравнить строение транспозона Zvezda у A. planci оказалось невозможным, так как в одной из сборок (BDGF01) не было полноразмерных копий транспозона Zvezda (табл. 3). В базе данных присутствует и третья сборка A. planci, но результат поиска транспозона Zvezda в программе BLAST отрицательный, по всей видимости из-за частичной представленности генома (табл. 1).
Сходная ситуация и у Patiria miniatа, также представленной двумя сборками, – в одной из двух сборок ОРС транспозона Zvezda также отсутствовали полноразмерные копии, следовательно структуру транспозонов сравнить нам не удалось. Количество копий Zvezda в этих двух геномах P. miniatа – 59 и 12 (табл. 3). Образцы были взяты с промежутком в 16 лет, местом сбора указана Калифорния. Разное число копий в геномах образцов P. miniatа может быть связано с внутрипопуляционным разнообразием, а также и с тем, что сборка генома Patiria miniatа/JADOBP01 неполная, поскольку общий размер сборки Patiria miniatа/JADOBP01 на 25% меньше, чем у Patiria miniatа/AKZP01.
Изучение МГЭ Zvezda проведено в рамках Государственного задания ФИЦ ИнБЮМ “Функциональные, метаболические и токсикологические аспекты существования гидробионтов и их популяций в биотопах с различным физико-химическим режимом”, номер гос. регистрации 121041400077-1.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Bourque G., Burns K.H., Gehring M. et al. Ten things you should know about transposable elements // Genome Biol. 2018. V. 19. P. 199. https://doi.org/10.1186/s13059-018-1577-z
Юрченко Н.Н., Коваленко Л.В., Захаров И.К. Мобильные генетические элементы: нестабильность генов и геномов // Вавилов. журн. генетики и селекции. 2011. Т. 15. № 2. С. 261–270.
Piacentini L., Fanti L., Specchia V. et al. Transposons, environmental changes, and heritable induced phenotypic variability // Chromosoma. 2014. V. 123. P. 345–354.
Auvinet J., Graça P., Belkadi L. et al. Mobilization of retrotransposons as a cause of chromosomal diversification and rapid speciation: The case for the Antarctic teleost genus Trematomus // BMC Genom. 2018. V. 19. P. 339.
Kojima K.K. Structural and sequence diversity of eukaryotic transposable elements // Genes Genet Syst. 2020. V. 94. P. 233–252. Epub 2018 Nov 9. PMID: 30416149.https://doi.org/10.1266/ggs.18-00024
Lee C.C., Wang J. Rapid expansion of a highly germline-expressed Mariner element acquired by horizontal transfer in the fire ant genome // Genome Biol. Evol. 2018. V. 10. P. 3262–3278. https://doi.org/10.1093/gbe/evy220
Xie L.-Q., Wang P.-L., Jiang S.-H. et al. Genome-wide identification and evolution of TC1/Mariner in the silkworm (Bombyx mori) genome // Genes Genomics. 2018. V. 40. P. 485–495. https://doi.org/10.1007/s13258-018-0648-6
Shen D., Gao B., Miskey C. et al. Multiple invasions of Visitor, a DD41D family of Tc1/mariner transposons, throughout the evolution of vertebrates // Genome Biol. Evol. 2020. V. 12. P. 1060–1073. https://doi.org/10.1093/gbe/evaa135
Yuan Y.W., Wessler S.R. The catalytic domain of all eukaryotic cut-and-paste transposase superfamilies // Proc. Natl Acad. Sci. USA. 2011. V. 108. P. 7884–7889. https://doi.org/10.1073/pnas.1104208108
Zhang H.H., Shen Y.H., Xiong X.M. et al. Identification and evolutionary history of the DD41D transposons in insects // Genes Genom. 2016. V. 38. P. 109–117.https://doi.org/10.1007/s13258-015-0356-4
Tellier M., Claeys Bouuaert C., Chalmers R. Mariner and the ITm superfamily of transposons // Microbiol. Spectrum. 2015. V. 3. https://doi.org/10.1128/microbiolspec.MDNA3-0033-2014
Ivics Z., Izsvák Z. Sleeping beauty transposition // Microbiol. Spectrum. 2015. V 3. https://doi.org/10.1128/microbiolspec.MDNA3-0042-2014
Ivics Z., Hackett P.B., Plasterk R.H., Izsvak Z. Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells // Cell. 1997. V. 91. P. 501–510.
Plasterk R.H., Izsvák Z., Ivics Z. Resident aliens the Tc1/mariner superfamily of transposable elements // Trends in Genetics. 1999. V. 15. P. 527–538.
Dupeyron M., Baril T., Bass C., Hayward A. Phylogenetic analysis of the Tc1/mariner superfamily reveals the unexplored diversity of pogo-like elements // Mobile DNA. 2020. V. 11. P. 21. https://doi.org/10.1186/s13100-020-00212-0
Gao B., Wang Y., Diaby M. Evolution of pogo, a separate superfamily of IS630-Tc1-mariner transposons, revealing recurrent domestication events in vertebrates // Mobile DNA. 2020. V. 11. P. 25. https://doi.org/10.1186/s13100-020-00220-0
Shao H., Tu Z. Expanding the diversity of the IS630-Tc1-mariner superfamily: discovery of a unique DD37E transposon and reclassification of the DD37D and DD39D transposons // Genetics. 2001. V. 159. P. 1103–1115.
Wang S., Diaby M., Puzakov M. et al. Divergent evolution profiles of DD37D and DD39D families of Tc1/mariner transposons in eukaryotes // Mol. Phylogenet. Evol. 2021. V. 161. A. 107143.https://doi.org/10.1016/j.ympev.2021.107143
Puzakov M.V., Puzakova L.V., Cheresiz S.V. An analysis of IS630/Tc1/mariner transposons in the genome of a Pacific oyster, Crassostrea gigas // J. Mol. Evol. 2018. V. 86. P. 566–580. https://doi.org/10.1007/s00239-018-9868-2
Buchan D.W.A., Jones D.T. The PSIPRED protein analysis workbench: 20 years on // Nucl. Acids Res. 2019. V. 47. P. 402–407. https://doi.org/10.1093/nar/gkz297
Kumar S., Stecher G., Li M. et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evol. 2018. V. 35. P. 1547–1549.
Filée J., Rouault J.D., Harry M., Hua-Van A. Mariner transposons are sailing in the genome of the blood sucking bug Rhodnius prolixus // BMC Genomics. 2015. V. 16. P. 1061. https://doi.org/10.1186/s12864-015-2060-9
Kumar S., Stecher G., Suleski M., Hedges S.B. TimeTree: a resource for timelines, timetrees, and divergence times // Mol. Biol. Evol. 2017. V. 34. P. 1812–1819. https://doi.org/10.1093/molbev/msx116
Schaack S., Gilbert C., Feschotte C. Promiscuous DNA: Horizontal transfer of transposable elements and why it matters for eukaryotic evolution // Trends Ecol. Evol. 2010. V. 25. P. 537–546.
Sundararajan P., Atkinson P.W., O’Brochta D.A. Transposable element interactions in insects: crossmobilization of hobo and Hermes // Insect Mol. Biol. 1999. V. 8. № 3. P. 359–368.
Puzakov M.V., Puzakova L.V., Cheresiz S.V. The Tc1-like elements with the spliceosomal introns in mollusk genomes // Mol. Genet. Genomics. 2020. V. 295. P. 621–633.
Zong W., Gao B., Diaby M. et al. Traveler, a new DD35E family of Tc1/mariner transposons, invaded vertebrates very recently // Genome Biol. Evol. 2020. V. 12. P. 66–76.
Sang Y., Gao B., Diaby M. et al. Incomer, a DD36E family of Tc1/mariner transposons newly discovered in animals //Mobile DNA. 2019. V. 10. P. 45.
Gao B., Zong W., Miskey C. et al. Intruder (DD38E), a recently evolved sibling family of DD34E/Tc1 transposons in animals // Mobile DNA. 2020. V. 11. P. 32.
Zhang H.H., Li G.Y., Xiong X.M. et al. TRT, a vertebrate and protozoan Tc1-like transposon: current activity and horizontal transfer // Genome Biol. Evol. 2016. V. 8. P. 2994–3005.
Emmons S.W., Yesner L., Ruan K., Katzenberg D. Evidence for a transposon in Caenorhabditis elegans //Cell. 1983. V. 32. P. 55–65.
Franz G., Savakis C. Minos, a new transposable element from Drosophila hydei, is a member of the Tc1-like family of transposons // Nucl. Acids Res. 1991. V. 19. P. 6646.
Langin T., Capy P., Daboussi M.J. The transposable element impala, a fungal member of the Tc1-mariner superfamily // Mol. Gen. Genet. 1995. V. 246. P. 19–28.
Clark K.J., Carlson D.F., Leaver M.J. et al. Passport, anative Tc1 transposon from flatfish, is functionally active in vertebrate cells // Nucl. Acids Res. 2009. V. 37. P. 1239–1247.
Дополнительные материалы отсутствуют.


