Генетика, 2022, T. 58, № 2, стр. 163-168
Распространение ассоциированных с болезнью хронического изнурения аллелей гена PRNP у диких и домашних северных оленей Rangifer tarandus на территории России
К. А. Курбаков 1, 2, *, Е. А. Коноров 1, 2, М. Т. Семина 1, Ю. А. Столповский 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Федеральный научный центр пищевых систем им. В.М. Горбатова Российской академии наук
109316 Москва, Россия
* E-mail: homo_ludens@vniimp.ru
Поступила в редакцию 09.04.2021
После доработки 30.09.2021
Принята к публикации 01.10.2021
- EDN: GWUUZS
- DOI: 10.31857/S0016675822020102
Аннотация
Болезнь хронического изнурения – это прионное заболевание, встречающееся у представителей семейства оленьих. Аминокислотные замены Ser138, Ser225 и 2M-129S-169M гена прионного белка PRNP увеличивают риск заболевания. В работе мы приводим данные о вариабельности участка гена PRNP с 108-ого по 240-ой кодон у Rangifer tarandus на территории азиатской части России и Ненецкого АО. Обнаружено семь полиморфных сайтов с несинонимичными заменами, пять из которых наблюдались ранее у оленей из Европы. Более резистентный к болезни хронического изнурения вариант Asn138 встречается примерно в четверти гаплотипов домашних оленей, другой устойчивый вариант Tyr225 встретился лишь у небольшой доли оленей с Чукотки. Дикие особи отличались от домашних по частотам аллелей с заменами Val169 и Gly129, аллель предрасположенности 2M-129S-169M встречался, в основном, у одомашненных особей. Из-за большой изменчивости гена PRNP у северного оленя России в случае возникновения у него болезни хронического изнурения возможно повторение ситуации, наблюдающейся в Норвегии – появление разных штаммов с отличающимися клиническими симптомами и проявлением болезни.
Ген PRNP кодирует прионный белок PrP, который имеет две конформационные изоформы. Инфекционная изоформа способна менять конформацию нормальной клеточной, накапливаясь в виде резистентного для протеаз агрегата в нервной ткани [1]. У разных видов млекопитающих данный процесс приводит к развитию трансмиссивных губчатых энцефалопатий, таких как болезнь Крейтцфельдта–Якоба у человека, коровье бешенство или губчатая энцефалопатия КРС, скрепи у овец и коз, болезнь хронического изнурения у оленьих. Передача инфекционной формы может происходить даже между разными видами животных, например, возможна передача коровьего бешенства от КРС человеку при употреблении в пищу зараженных животных. Случаи передачи болезни хронического изнурения и скрепи от животных к человеку не были отмечены, скорее всего, из-за особенностей последовательности и структуры PrP человека, затрудняющие конформационный переход [2, 3]. Таким образом, риск передачи болезни хронического изнурения от оленьих к человеку довольно низок, хотя нельзя исключать возможность адаптации прионов к новому хозяину или обнаружения гаплотипов PRNP, имеющих большую способность к трансмиссии [4].
Распространение и изменчивость PRNP изучена для разных представителей семейства Cervidae: для европейского лося, косули, лани [5], белохвостого [6, 7], благородного, пятнистого [7] и северного оленей [5, 8]. Для северного оленя прослежена также ассоциация отдельных гаплотипов с частотой болезни хронического изнурения [9], например, распространенный аллель Ser225 и делеция Trp84_Gly91del увеличивает риск развития болезни в пять раз в гетерозиготе и в более чем в 40 раз, находясь в гомозиготном состоянии. Гомозиготы Tyr225, наоборот, были обнаружены только у здоровых животных. Поиск ассоциаций был проведен также для белохвостого оленя Odocoileus virginianus, где эффект от замены Ser225 не был статистически значимым, однако обнаружена повышенная устойчивость к болезни при наличии несинонимической замены Ser95 [10]. Замена на аспарагин в 138-ом кодоне также предположительно связана с резистентностью [11].
По вышеперечисленным позициям в природных и одомашненных популяциях наблюдается изменчивость, а в некоторых случаях дифференциация. В Северной Америке отдельные локальные популяции северных оленей демонстрируют избыток гетерозиготи гомозигот c Asn138, что может быть результатом действия балансирующего отбора или генетического дрейфа [12, 13]. На севере европейской части России было описано распространение вариантов гена PRNP у диких и одомашненных популяций северного оленя, при этом отмечалось более высокая доля аллелей с заменой Asn138 у домашних оленей по сравнению с дикими [8].
В настоящей работе мы приводим данные о вариабельности участка гена PRNP у Rangifer tarandus с 108-ого по 240-ой кодон на территории азиатской части России и Ненецкого АО. На данном участке расположены основные нуклеотидные замены, ассоциированные с риском болезни хронического изнурения. Также мы провели поиск отличий между дикими и домашними оленями.
МАТЕРИАЛЫ И МЕТОДЫ
Всего исследовано 89 оленей, из них 72 особи: ненецкой, эвенкийской, чукотской пород и 17 диких оленей (п-ов Чукотка) (табл. 1). Образцы дикой популяции были представлены мышечной тканью, домашних животных – ушным выщипом. ДНК выделялось с использованием набора QIAmp DNA mini Kit (QIAGEN, Хильден, Германия). Амплификация гена PRNP происходила с помощью qPCRmix-HS (Евроген, Москва, Россия) и праймеров RangPrP-F AGTCAGTGGAACAAGCCCAG и RangPrP-R TGAGGAGGATCACAGGAGGG, подобранных с помощью Primer-BLAST [14] и OligoAnalyzer v. 3 (http://www.idtdna.com/analyzer/applications/oligoanalyzer/). Длина амплифицируемого фрагмента составила 442 пар оснований (участок с 298 по 739 нуклеотид гена PRNP, GenBank – ID MK097270). Режим реакции ПЦР: предварительная денатурация при 95°C, 5 мин и 45 циклов амплификации (95°C, 15 с, 60°C, 20 с и 72°C 15 с). Секвенирование по Сенгеру полученных апликонов проводили в ЗАО “Евроген” (Евроген, Москва, Россия). Секвенирование проводили с использованием как форвард-, так и реверс-праймера. Анализ полученных хроматограмм проводили с использованием программы UGENE [15]. Обнаруженные аллельные варианты были депонированы в базе данных GenBank под идентификационными номерами MZ442704–MZ442716.
Таблица 1.
Объемы выборок, места сбора и показатели генетического разнообразия
| Выборка | Объем выборки | Место сбора материала | π × 103 | D Таджимы |
|---|---|---|---|---|
| Чукотские, дикая популяция | 17 | О-в Айон, Певек | 4.26 | –0.66 |
| Эвенкийские (тоджинские) | 18 | Тува, Тоджинский р-н | 3.68 | 0.023 |
| Чукотка, домашняя порода | 16 | П-ов Камчатка | 4.54 | 0.66 |
| Эвенкийские | 20 | Амурская обл., Зейский р-он | 3.65 | –0.29 |
| Ненецкие | 18 | СПК Коопхоз “ЕРВ” (племрепродуктор) НАО, западная часть Большеземельной тундры Ненецкого АО | 3.14 | –0.33 |
Выравнивание последовательностей гена PRNP с помощью Muscle [16] и тест Таджимы на нейтральность были проведены в пакете MEGAX [15]. TCS-сеть аллелей гена PRNP была построена в программе Popart [17]. Анализ главных компонент производился с помощью предустановленной в язык R функции prcomp.
РЕЗУЛЬТАТЫ
В процессе исследования было получено 89 последовательностей гена PRNP северного оленя длиной 399 пн, начиная с 108-ого кодона и заканчивая 240-ым. Согласно показателям генетического разнообразия и D Таджимы, выборка домашних чукотских оленей единственная из изученных нами демонстрирует тенденцию на увеличение гетерозиготности (табл. 1, рис. 1).
Рис. 1.
Частоты генотипов для наиболее вариабельных кодонов гена PRNP у особей R. tarandus, изученных в данной работе.
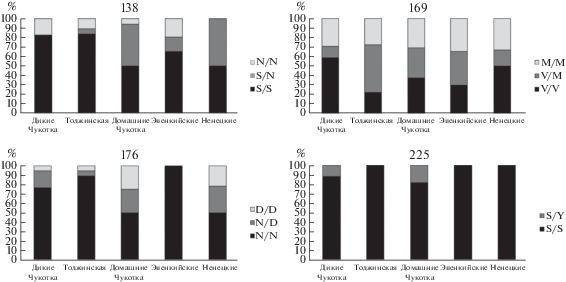
Обнаружено семь полиморфных сайтов, все они несинонимичные. В кодоне 135 найдена новая для северных оленей замена Arg135, встретившаяся у двух диких особей чукотского оленя. Ранее не встречавшаяся замена Ser184 наблюдается в гетерозиготном состоянии в эвенкийской и ненецкой популяциях, по одной особи в каждой. Для остальных полиморфных сайтов наблюдалось большее генетическое разнообразие (рис. 1, табл. 2). Связанный с резистентностью к болезни хронического изнурения вариант Asn138 встречается примерно в четверти гаплотипов чукотских, эвенкийских и ненецких пород оленей, в два раза меньше его у оленей тоджинской породы. У диких оленей из Чукотки Asn138 обнаружен только в гомозиготном состоянии, как и у популяций диких оленей с Европейского Севера России [8]. Доля резистентного варианта ниже по сравнению с выборками домашних оленей, исключая тоджинских.
Таблица 2.
Частоты аминокислотных замен в наиболее вариабельных кодонах гена PRNP у R. tarandus на территории России
| Выборка | 129 | 138 | 169 | 176 | 225 | ||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| G | S | S | N | V | M | N | D | S | Y | ||
| Тоджинский | 0.35 | 0.65 | 0.86 | 0.14 | 0.47 | 0.53 | 0.92 | 0.08 | 1.00 | 0.00 | |
| Чукотка, домашние | 0.63 | 0.38 | 0.72 | 0.28 | 0.53 | 0.47 | 0.63 | 0.38 | 0.91 | 0.09 | |
| Эвенкийские | 0.33 | 0.67 | 0.73 | 0.28 | 0.48 | 0.53 | 1.00 | 0.00 | 1.00 | 0.00 | |
| Ненецкие | 0.44 | 0.56 | 0.75 | 0.25 | 0.58 | 0.42 | 0.64 | 0.36 | 1.00 | 0.00 | |
| Республика Коми | 0.53 | 0.47 | 0.72 | 0.28 | 0.78 | 0.22 | 0.94 | 0.06 | 1.00 | 0.00 | * |
| О-в Колгуев | 0.53 | 0.47 | 0.63 | 0.37 | 0.45 | 0.55 | 0.76 | 0.24 | 1.00 | 0.00 | * |
| Новая Земля | 0.85 | 0.15 | 0.91 | 0.09 | 0.87 | 0.13 | 0.89 | 0.11 | 0.98 | 0.02 | * |
| Кольский п-ов | 0.89 | 0.11 | 0.89 | 0.11 | 0.93 | 0.07 | 1.00 | 0.00 | 0.86 | 0.14 | * |
| Дикие: Архангельск, Республика Коми | 1.00 | 0.00 | 0.82 | 0.18 | 1.00 | 0.00 | 1.00 | 0.00 | 0.86 | 0.14 | * |
| Чукотка, дикие | 0.63 | 0.38 | 0.82 | 0.18 | 0.65 | 0.35 | 0.85 | 0.15 | 0.94 | 0.06 | |
Примечание. Верхняя строка в шапке – позиция кодона, * – данные для выборок, взяты из исследования [8] для сравнения.
У диких северных оленей с Чукотки и домашних оленей чукотской породы присутствуют аллели с Tyr225 (аллель B и аллель Asp176_Tyr225), у остальных изученных в настоящей работе выборок подобный вариант отсутствует даже в гетерозиготном состоянии. У оленей с Европейского Севера данные аллели наблюдались почти исключительно у диких, но не домашних особей [8].
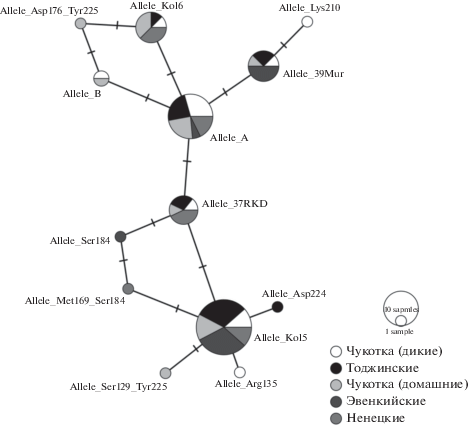
На рис. 2 приведены взаимоотношения между найденными в ходе исследования аллелями в виде TCS-сети. Наиболее распространенными аллелями в изученной выборке являются аллель, идентичный на рассмотренном участке аллелю A (Ser225, SH-SY5Y rePrPwt, [9]) и аллель Kol5 (MSSM, DQ154295, 2M-129S-169M). Реже встречались (у восьми–девяти особей) аллели 39Mur (VGNV, DQ154292), RKD37 (VSSV, DQ154294) и Kol6 (176D, JQ290075).
Анализ главных компонент на основании частот аллелей гена PRNP в разных выборках оленей с территории России выявил дифференциацию диких особей с европейской части России от всех домашних оленей (рис. 3). Основным источником данной дифференциации являются аллели Val169 и Gly129, которые у диких оленей представлены в большей концентрации. Исключением является выборка диких оленей с п-ова Чукотка, которая близка по частотам Gly129 к оленям чукотской породы и не является статистическим выбросом из выборок домашних оленей по частоте Val169 (согласно тесту Диксона).
Рис. 3.
Результаты анализа главных компонент (оси абсцисс и ординат) на основании частот аллелей гена PRNP в разных выборках северного оленя на территории России.

Выборки домашних оленей при этом также разбиваются на две группы: 1) с полным преобладанием Asn176 – тоджинская, эвенкийская породы и олени из выборки Коми; 2) со значительной долей гомо- и гетерозигот (до 50%) по замене Asp176 – олени ненецкой и чукотской пород и популяции с о-ва Колгуев.
ОБСУЖДЕНИЕ
Случаи болезни хронического изнурения северного оленя наблюдаются в соседних с Россией странах – Норвегии и Финляндии, с 2016 г. Как и в случае с другими прионными болезнями, в большинстве случаев предотвратить распространение инфекции возможно лишь с помощью уничтожения целых локальных популяций зараженных животных [19]. Болезнь трудно диагностировать у животных из-за неспецифичных симптомов, выражающихся в изменении поведения и потере веса, развивающихся за период от нескольких недель до месяцев [20]. На территории США болезнь распространялась со скоростью большей, чем почти все остальные известные прионные заболевания. На территории Норвегии, Швеции и Финляндии распространение было медленнее, но полностью остановить его не получилось несмотря на жесткие меры [21]. Появление болезни на территории России потребует срочных мер, внедрить которые будет сложно из-за большой территории и традиционного образа оленеводства различных народов Европейского Севера, Сибири и Дальнего Востока.
У исследованных на данный момент популяций одомашненного и дикого оленей, обитающих на территории России распространены аллели, ассоциированные ранее с предрасположенностью к болезни хронического изнурения. У всех особей, генотипированных в ходе данного исследования, наблюдается замена Ser225, лишь у домашних и диких оленей из Чукотки обнаружен более резистентный вариант Tyr225 в гетерозиготном состоянии. Аллель Kol5 (2M-129S-169M), для которого в ходе анализа ассоциации также была обнаружена предрасположенность к болезни [9], встречается в основном у одомашненных оленей, как из европейской, так и азиатской части России, практически отсутствует у диких оленей Европейского Севера России.
В более старом очаге болезни хронического изнурения оленьих – Северной Америке – предрасположенность связывали в основном с заменой Ser138 [12, 13], полиморфизма в 129-ом и 225-ом кодонах не наблюдалось. В то же время там, где болезнь впервые появилась в Европе, в Норвегии, нет полиморфизма в 138-ом кодоне, а ассоциация наблюдается с заменами Ser225 и Ser129 [9]. У северного оленя на территории России наблюдаются выявленные как европейскими, так и североамериканскими авторами факторы предрасположенности к болезни, такие как Ser138, так и Ser225 с Ser129. В ходе сравнения течения болезней у европейских и североамериканских оленей недавние исследования показали, что из-за разной конформации инфекционной изоформы белка PrP, кодируемого геном PRNP, штаммы прионов из Норвегии и Северной Америки обладают разными трансмиссионными и нейропатологическими паттернами; причем внутри норвежских штаммов также существует вариативность в патологической картине [22]. Из-за большой изменчивости гена PRNP у северного оленя России в случае возникновения у него болезни хронического изнурения возможна ситуация, повторяющая таковую в Норвегии: большое разнообразие клинических картин болезни или появление нового, специфичного для российских популяций штамма, что затруднит использование накопленного опыта борьбы с болезнью хронического изнурения.
Работа выполнена при поддержке гранта РФФИ № 17-29-08003.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Houston F., Andréoletti O. Animal prion diseases: the risks to human health // Brain Pathology. 2019. V. 29. № 2. P. 248–262. https://doi.org/10.1111/bpa.12696
Kurt T.D., Jiang L., Fernández-Borges N. et al. Human prion protein sequence elements impede cross-species chronic wasting disease transmission // J. Clin. Investigation. 2015. V. 125. № 4. P. 1485–1496. https://doi.org/10.1172/JCI79408
Concepcion G.P., David M.P. C., Padlan E.A. Why don’t humans get scrapie from eating sheep? A possible explanation based on secondary structure predictions // Med. Hypotheses. 2005. V. 64. № 5. P. 919–924. https://doi.org/10.1016/j.mehy.2004.11.021
Hannaoui S., Schatzl H.M., Gilch S. Chronic wasting disease: Emerging prions and their potential risk // PLoS Pathogens. 2017. V. 13. № 11. P. e1006619. https://doi.org/10.1371/journal.ppat.1006619
Wik L., Mikko S., Klingeborn M. et al. Polymorphisms and variants in the prion protein sequence of European moose (Alces alces), reindeer (Rangifer tarandus), roe deer (Capreolus capreolus) and fallow deer (Dama dama) in Scandinavia // Prion. 2012. V. 6. № 3. P. 256–260. https://doi.org/10.4161/pri.19641
Chafin T.K., Douglas M.R., Martin B.T. et al. Age structuring and spatial heterogeneity in prion protein gene (PRNP) polymorphism in white-tailed deer // Prion. 2020. V. 14. № 1. P. 238–248. https://doi.org/10.1080/19336896.2020.1832947
Robinson S.J., Samuel M.D., O’Rourke K.I., Johnson C.J. The role of genetics in chronic wasting disease of North American cervids // Prion. 2012. V. 6. № 2. P. 153–162. https://doi.org/10.4161/pri.19640
Kholodova M.V., Baranova A.I., Mizin I.A. et al. A genetic predisposition to chronic wasting disease in the reindeer Rangifer tarandus in the Northern European Part of Russia // Biol. Bulletin. 2019. V. 46. № 6. P. 555–561. https://doi.org/10.1134/S1062359019060074
Güere M.E., Våge J., Tharaldsen H. et al. Chronic wasting disease associated with prion protein gene (PRNP) variation in Norwegian wild reindeer (Rangifer tarandus) // Prion. 2020. V. 14. № 1. P. 1–10. https://doi.org/10.1080/19336896.2019.1702446
Rhyan J.C., Miller M.W., Spraker T.R. et al. Failure of fallow deer (Dama dama) to develop chronic wasting disease when exposed to a contaminated environment and infected mule deer (Odocoileus hemionus) // J. Wildlife Diseases. 2011. V. 47. № 3. P. 739–744. https://doi.org/10.7589/0090-3558-47.3.739
Mitchell G.B., Sigurdson C.J., O’Rourke K.I. et al. Experimental oral transmission of chronic wasting disease to reindeer (Rangifer tarandus tarandus) // PLoS One. 2012. V. 7. № 6. P. e39055. https://doi.org/10.1371/journal.pone.0039055
Cheng Y.C., Musiani M., Cavedon M., Gilch S. High prevalence of prion protein genotype associated with resistance to chronic wasting disease in one Alberta woodland caribou population // Prion. 2017. V. 11. № 2. P. 136–142. https://doi.org/10.1080/19336896.2017.1300741
Happ G.M., Huson H.J., Beckmen K.B., Kennedy L.J. Prion protein genes in caribou from Alaska // J. Wildlife Diseases. 2007. V. 43. № 2. P. 224–228. https://doi.org/10.7589/0090-3558-43.2.224
Ye J., Coulouris G., Zaretskaya I. et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction // BMC Bioinformatics. 2012. V. 13. № 1. P. 1–11. https://doi.org/10.1186/1471-2105-13-134
Okonechnikov K., Golosova O., Fursov M. Ugene Team. Unipro UGENE: A unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. № 8. P. 1166–1167. https://doi.org/10.1093/bioinformatics/bts091
Edgar R.C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput // Nucl. Ac. Res. 2004. V. 32. № 5. P. 1792–1797. https://doi.org/10.1093/nar/gkh340
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. and Evol. 2018. V. 35. № 6. P. 1547–1549. https://doi.org/10.1093/molbev/msy096
Leigh J.W., Bryant D. Popart: full-feature software for haplotype network construction // Met. in Ecol. and Evol. 2015. V. 6. № 9. P. 1110–1116. https://doi.org/10.1111/2041-210X.12410
Mysterud A., Rolandsen C.M. A reindeer cull to prevent chronic wasting disease in Europe // Nat. Ecol. & Evol. 2018. V. 2. № 9. P. 1343–1345. https://doi.org/10.1038/s41559-018-0616-1
Benestad S.L., Telling G.C. Chronic wasting disease: An evolving prion disease of cervids // Handbook of Clin. Neurology. 2018. V. 153. P. 135–151. https://doi.org/10.1016/B978-0-444-63945-5.00008-8
Maraud S., Roturier S. Chronic wasting disease (CWD) in sami reindeer herding: The socio-political dimension of an epizootic in an indigenous context // Animals. 2021. V. 11. № 2. P. 297. https://doi.org/10.3390/ani11020297
Nonno R., Di Bari M.A., Pirisinu L. et al. Studies in bank voles reveal strain differences between chronic wasting disease prions from Norway and North America // PNAS. 2020. V. 117. № 49. P. 31417–31426. https://doi.org/10.1073/pnas.2013237117
Дополнительные материалы отсутствуют.