Генетика, 2022, T. 58, № 2, стр. 169-176
Генетические маркеры овец карачаевской породы, выявленные методом полногеномного поиска ассоциаций
А. Ю. Криворучко 1, О. А. Яцык 1, А. В. Скокова 1, *, А. А. Каниболоцкая 1
1 Северо-Кавказский федеральный научный аграрный центр
356241 Ставропольский край, Михайловск, Россия
* E-mail: antoninaskokova@mail.ru
Поступила в редакцию 14.05.2021
После доработки 15.07.2021
Принята к публикации 20.07.2021
- EDN: AXVCYD
- DOI: 10.31857/S0016675822020096
Аннотация
Проведен полногеномный поиск ассоциаций (GWAS) у овец карачаевской породы с использованием ДНК-биочипов Ovine Infinium HD BeadChip 600K для выявления вариантов однонуклеотидного полиморфизма (SNP), связанных с породной принадлежностью. На основании проведенного анализа было выявлено более 100 SNP, имеющих высокодостоверные различия в частоте встречаемости у животных карачаевской породы; для последующей оценки было отобрано по пять единичных SNP с наивысшими показателями на хромосомах 12, 13 и 14. Три замены находятся в экзонах генов, одна расположена в интроне, остальные – на разном удалении от генов. Самую высокую достоверность в ассоциациях с породной принадлежностью показали замены rs408511664, rs429132161 и rs409651063 (р = 4.637 × 10–84), находящиеся на 14-й хромосоме и встречающиеся только у овец карачаевской породы. Большинство обнаруженных SNP локализованы рядом с генами, играющими немаловажную роль в процессе развития фенотипических признаков (TUBB3, MC1R, BMP2, LYPLAL1‑201, PAPPA2). Проведенные исследования позволяют использовать совокупность выявленных SNP в качестве информативных молекулярных маркеров при идентификации породной принадлежности карачаевских овец.
Определение породной принадлежности овец является основой для селекционной работы. В настоящее время оно выполняется с использованием показателей фенотипа и данных родословной с подтверждением достоверности происхождения [1, 2]. Однако этой информации на современном уровне развития овцеводства уже недостаточно. Новые методы оценки породной принадлежности необходимы для выявления различий у близких по происхождению пород, для регистрации новой породы, определения происхождения гибридов, при выполнении экспертизы мясной продукции и целом ряде других мероприятий [3, 4]. Особенно это актуально в странах с большим количеством используемых пород. К таким относится и Россия, где официально зарегистрировано более 40 пород овец.
Наиболее перспективным для оценки породной принадлежности является применение современных достижений генетики. Методы кластеризации овец на отдельные породы по молекулярным ДНК-маркерам используются достаточно давно. Для этого чаще всего применяют генотипирование по микросателлитным локусам [5]. Их полиморфизма достаточно для разделения животных на группы, соответствующие породе, по совокупности встречающихся с разной частотой аллелей [6].
Между тем обратный процесс, т.е. определение породной принадлежности конкретного животного по полиморфизму микросателлитных локусов, не может быть выполнен с высокой достоверностью. Это связано с тем, что уникальных аллелей микросателлитных локусов, встречающихся только у одной породы, не обнаружено. Поэтому оценка носит вероятностный характер и не достаточна для точной идентификации породы. Тем не менее при проведении генетической паспортизации или экспертизы продукции достоверность определения породы по результатам исследования ДНК должна приближаться к 100 процентам [7, 8].
Более точной оценку породной принадлежности можно сделать при использовании в качестве маркеров вариантов однонуклеотидного полиморфизма (Single Nucleotide Polymorphism, SNP). Их количество в геноме значительно превышает число микросателлитных локусов, а число аллелей, как правило, не более двух. Используя уникальную комбинацию из нескольких SNP, встречающихся только у исследуемой породы, вполне можно достичь требуемой точности определения породной принадлежности [9]. Для обнаружения этих SNP можно использовать проведение полногеномного поиска ассоциаций с помощью ДНК-биочипов высокой плотности [10]. Выполнение исследования целесообразно по принципу “случай–контроль”. При этом “случай” – животные исследуемой породы, а “контроль” – особи других пород. Это позволит выявить однонуклеотидные варианты полиморфизма, достоверно отличающие исследуемую породу на уровне генома. Обнаруженные SNP могут быть использованы как молекулярные маркеры породы [11]. Расположенные рядом с ними гены можно рассматривать как гены-кандидаты, их полиморфизм обеспечивает проявление фенотипических признаков, на основании которых животные были выделены в отдельную породу [12].
Интересным представляется поиск молекулярных маркеров породной принадлежности среди аборигенных пород овец, длительно существующих в условиях относительной изоляции. Одной из таких пород является карачаевская, разводимая в условиях Северного Кавказа на протяжении двухсот лет. По продуктивности она относится к грубошерстным, однако используется как универсальная. От овец получают шерсть (настриг у баранов до 3 кг, у ярок до 2 кг), мясо (вес баранов доходит до 70 кг) и молоко, выход которого составляет 50 кг в год. Несмотря на то что животные эти некрупные, порода является скороспелой, а мясо обладает высокими вкусовыми качествами. Овцы отличаются высокой устойчивостью к воздействию климатических факторов, так как разводятся в горной местности, а также резистентны ко многим инфекционным заболеваниям [13, 14].
Целью нашей работы стал поиск новых молекулярных маркеров карачаевской породы методом полногеномного поиска ассоциаций.
МАТЕРИАЛЫ И МЕТОДЫ
Исследования проводились на базе лабораторий Всероссийского научно-исследовательского института овцеводства и козоводства (ВНИИОК) – филиала ФГБНУ “Северо-Кавказский федеральный научный аграрный центр”, Сколковского института науки и технологий “Сколтех”, Научно-диагностического и лечебного ветеринарного центра ФГБОУ ВО “Ставропольский государственный аграрный университет”, племенного завода “Восток” Ставропольского края.
Исследование было организовано по принципу “случай–контроль”, где “случай” соответствовал баранчикам карачаевской породы, а “контроль” – баранчикам других пород. В группу “случай” по итогам комплексной оценки были отобраны 55 баранчиков карачаевской породы (из 417 баранчиков, полученных от маток селекционного ядра) в возрасте 12 мес. Критерии комплексной оценки (бонитировки) баранчиков – густота, длина, тонина шерсти, ее настриг в чистом волокне, качество жиропота, экстерьер, живая масса. По результатам проведенной бонитировки все 55 баранчиков были отнесены к классу “элита” и имели наиболее ярко выраженные признаки карачаевской породы. Группа “контроль” состояла из 220 животных (по 55 особей каждой породы) северокавказской мясо-шерстной породы, романовской породы, породы джалгинский меринос и российский мясной меринос. Они также отбирались из представителей селекционного ядра по тем же критериям комплексной оценки, как имеющие наиболее ярко выраженные признаки соответствующей породы. Все баранчики были клинически здоровыми.
Генотипирование
Геномную ДНК выделяли из образцов цельной крови, взятой в асептических условиях из яремной вены, с использованием набора Pure Link Genomic DNA MiniKit (Invitrogen Life Technologies, США) в соответствии с протоколом производителя. Генотипирование животных выполнялось с использованием Ovine Infinium HD BeadChip 600K (Illumina, США) согласно протоколу производителя. Первичную обработку результатов генотипирования выполняли с использованием программного обеспечения Genome Studio 2.0 (Illumina).
Контроль качества генотипирования
Контроль качества генотипирования проводили с использованием программного обеспечения PLINK V.1.07 [15]. В обработку данных были включены образцы с показателем количества выявленных SNP (Call Rate) больше 0.95. Из анализа были исключены SNP с частотой минорных аллелей (MAF – Minor Allele Frequency) меньше 0.01, частотой потерянных генотипов (missing genotype) больше 0.1. В качестве порогового значения по критерию Харди–Вайнберга (Hardy–Weinberg equilibrium) методом Фишера использовалось значение p = 0.00001. С положительным результатом контроль качества генотипирования прошли все 275 образцов. Из 606 006 SNP для дальнейшего анализа были использованы 550 991 полиморфизм.
Генетический и статистический анализы
Полногеномный поиск ассоциаций выполняли с использованием программного обеспечения PLINK V.1.07 [15] на основе оценки значимости влияния SNP на породную принадлежность особей. Для подтверждения достоверности различий при множественных сравнениях использовали оценку р с поправкой Бонферрони. Визуализацию и построение графиков проводили с применением пакета “QQman” на языке программирования R. Поиск генов-кандидатов выполнялся в области 500 000 пн вокруг SNP (половина сантиморганиды), показавших достоверные различия по встречаемости среди животных исследуемых групп. Для картирования использовалась сборка генома Oar_rambouillet_v1.0. Аннотирование генов выполнялось с использованием геномных браузеров UCSC (www.genome.ucsc.edu) и Ensemble (www.ensembl.org).
РЕЗУЛЬТАТЫ
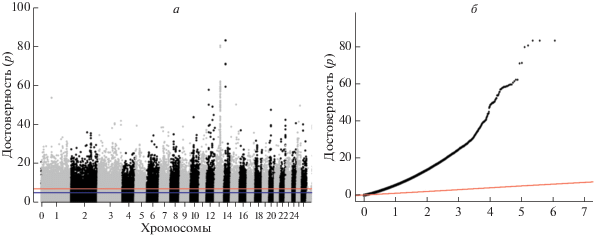
Полногеномный поиск ассоциаций частоты встречаемости вариантов однонуклеотидного полиморфизма с фенотипическими признаками карачаевской породы овец показал наличие очень большого количества локусов, преодолевших порог достоверности, установленный в соответствии с поправкой Бонферрони на уровне ‒log10(p) = 7 (рис. 1,а). Квантиль-квантиль график (рис. 1,б) показывает, что абсолютное большинство исследуемых SNP имеет показатели, отличающиеся от таковых при подтверждении нулевой гипотезы (отсутствие достоверных различий). Отклонение от ожидаемых значений наблюдается начиная от величины –log10(p) > 0.5.
Рис. 1.
Результаты полногеномного поиска ассоциаций у овец карачаевской породы. а – Манхэттенский график результатов GWAS с набором значений –log10(p) для исследуемых SNP. Нижняя линия обозначает порог различий с ожидаемой достоверностью различий при значении –log10(p) = 5, верхней линией указан порог высокой достоверности различий при значении –log10(p) = 7: б – Q-Q-график для вероятностей распределения достоверности оценок SNP по всему геному. Точками обозначены значения –log10(p) для отдельных SNP. Линия обозначает ожидаемые значения при подтверждении нулевой гипотезы об отсутствии ассоциаций.
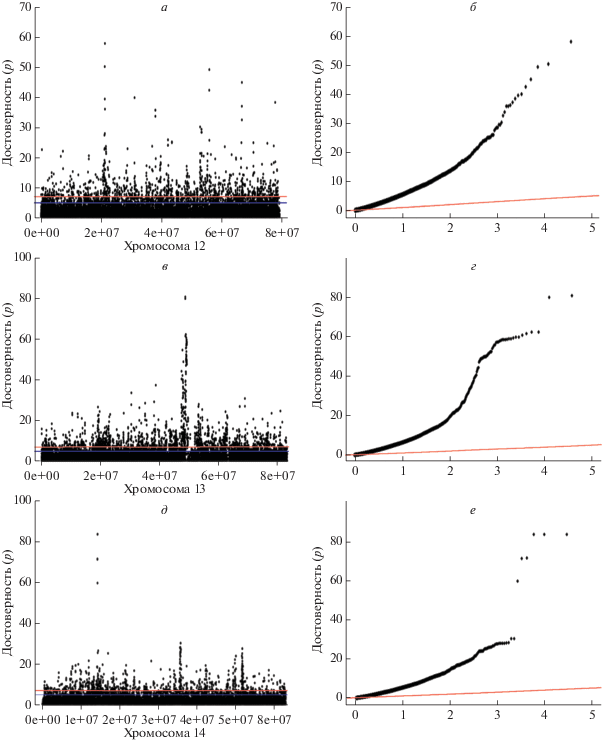
Наиболее высокие показатели достоверности выявлены для SNP, расположенных на хромосомах 12, 13 и 14 (рис. 2). На 12-й хромосоме полиморфизмы, связанные с породной принадлежностью, находятся в локусах по всей длине хроматид. Максимальные отличия отмечены для rs424156280, находящейся в позиции 73451671. Хромосома 13 имеет целый пул SNP с высокими показателями достоверности, локализованный вблизи ее центральной области. На 14-й хромосоме нами были выявлены SNP с наибольшими показателями достоверности ассоциаций с породной принадлежностью, однако их количество существенно уступает хромосомам 12 и 13.
Рис. 2.
Результаты поиска ассоциаций по хромосомам у овец карачаевской породы. а, в, д – Манхэттенский график результатов GWAS с набором значений –log10(p) исследуемых SNP в отдельных локусах хромосом 12, 13 и 14. Нижняя линия обозначает порог различий с ожидаемой достоверностью различий при значении –log10(p) = 5, верхней линией указан порог высокой достоверности различий при значении –log10(p) = 7; б, г, е – Q-Q-график для вероятностей распределения достоверности оценок SNP, локализованных на хромосомах 12, 13 и 14. Точками обозначены значения ‒log10(p) для отдельных SNP. Линия обозначает ожидаемые значения при подтверждении нулевой гипотезы об отсутствии ассоциаций.
В связи с большим количеством SNP, достоверность связи которых с породной принадлежностью животных превышает установленный порог, для дальнейшего анализа их расположения в геноме относительно аннотированных генов были отобраны по пять SNP с максимальными показателями на хромосомах 12, 13 и 14 (табл. 1).
Таблица 1.
Характеристика SNP с наибольшими показателями достоверности ассоциации с породной принадлежностью животных при проведении GWAS
| № | SNP | Хромосома/ позиция | A1 | A2 | F_A | F_U | p | Ген/расстояние до гена |
|---|---|---|---|---|---|---|---|---|
| 1 | rs408511664 | 14/15 483 844 | A | G | 0.75 | 0 | 4.637e-84 | TUBB3/перед геном 6999 пн |
| 2 | rs429132161 | 14/15 489 751 | A | G | 0.75 | 0 | 4.637e-84 | TUBB3/перед геном 1092 пн |
| 3 | rs409651063 (Oar_v3.1) |
14/14 231 948 | A | G | 0.75 | 0 | 4.637e-84 | MC1R/экзон 3, 105 aa, D/N (миссенс-мутация) |
| 4 | rs413629721 | 14/15 451 699 | С | А | 0.75 | 0.02 | 4.861e-72 | LncRNA/экзон 10 (некодирующий транскрипт) |
| 5 | rs412344125 | 14/15 448 879 | А | С | 0.74 | 0.02 | 8.764e-72 | LncRNA/экзон 4 (некодирующий транскрипт) |
| 6 | rs427301675 | 13/51 330 485 | A | G | 0.80 | 0.02 | 1.966e-81 | BMP2/129 839 пн |
| 7 | rs411672645 | 13/51 327 453 | A | G | 0.80 | 0.02 | 1.402e-80 | BMP2/126 807 пн |
| 8 | rs412370368 | 13/51 502 951 | A | G | 0.79 | 0.06 | 5.580e-63 | BMP2/302 305 пн |
| 9 | rs401025140 | 13/51 506 415 | G | A | 0.79 | 0.06 | 5.580e-63 | BMP2/305 769 пн |
| 10 | rs425709044 | 13/51 358 671 | G | A | 0.81 | 0.07 | 3.400e-62 | BMP2/158 025 пн |
| 11 | rs426400216 | 12/25 137 722 | A | G | 0.68 | 0.03 | 1.133e-58 | LYPLAL1-201/377 126 пн |
| 12 | rs415954733 | 12/25 076 577 | A | G | 0.72 | 0.07 | 5.472e-51 | LYPLAL1-201/315 981 пн |
| 13 | rs419909433 | 12/61 840 136 | A | G | 0.75 | 0.09 | 5.502e-50 | PAPPA2/перед геном 1724 пн |
| 14 | rs424156280 | 12/73 451 671 | G | А | 0.52 | 0.02 | 1.016e-45 | KCNK2/интрон 3 |
| 15 | rs406878732 | 12/61 816 311 | A | G | 0.75 | 0.11 | 3.803e-43 | PAPPA2/25 549 пн |
Из 15 рассматриваемых замен три находятся в экзонах генов, одна в интроне, а остальные расположены на разном удалении от кодирующих генов. Ближе всего к границам генов находятся замены rs408511664 и rs429132161 в 14-й хромосоме. Замена rs415954733 в хромосоме 12 располагается дальше всех рассматриваемых от ближайшего гена, однако это расстояние не превышает половины сантиморганиды. На основе сборки генома Oar_rambouillet_v1.0. нам не удалось картировать замену rs409651063, поэтому ее параметры определены на основе сборки Oar_v3.1, согласно которой она попадает в экзон гена. Пять замен, локализованных на 13-й хромосоме, расположены в одном межгенном пространстве, на различном расстоянии от гена BMP2 (Bone Morphogenetic Protein-2, BMP2). Две пары замен на 12-й хромосоме также находятся достаточно близко друг от друга. Гены, расположенные в области выявленных SNP, можно считать генами-кандидатами с возможным влиянием на фенотипические признаки породной принадлежности овец карачаевской породы.
ОБСУЖДЕНИЕ
Проведенное исследование подтвердило наличие генетических основ внутривидовой (породной) изменчивости у овец. Выявлено достаточно большое количество SNP, достоверно различающихся по частоте встречаемости у овец карачаевской породы и других пород овец. При этом три замены на 14-й хромосоме были обнаружены только у карачаевских овец и не встречались ни у одной особи остальных пород. Две из них, rs408511664 и rs429132161, расположены рядом с геном тубулина бета 3 (Tubulin Beta 3 Class III, TUBB3). Кодируемый им белок вместе с тубулином альфа образуют структурную основу микротрубочек цитоскелета [16]. Кроме чисто механической поддержки формы клетки и расположения органелл, микротрубочки играют важную роль в процессах транспорта внутриклеточных компонентов и отдельных молекул. Также они обеспечивают подвижность клеток, организацию работы нейронов, секрецию везикул и многие другие функции клеток [17]. Учитывая участие белка тубулина в таком количестве жизненно важных процессов, мы считаем, что его ген может обеспечивать фенотипические особенности карачаевских овец.
Замена rs409651063 находится в экзоне гена, кодирующего рецептор меланокортина 1 (Melanocortin 1 Receptor, MC1R). Так же как и две описанные выше замены она встречается только у карачаевских овец и не обнаруживается в группе контроля. Рецептор меланокортина участвует в регуляции синтеза меланина и влияет на цвет шерсти и волос у млекопитающих [18]. Наличие обнаруженной нами замены уже рассматривается как маркер темного цвета шерсти у некоторых видов овец [19, 20]. Стоит отметить, что карачаевская порода характеризуется шерстью черного цвета, а животные в группе контроля имеют белый цвет шерсти. Таким образом, наши исследования подтверждают результаты, полученные другими учеными, и позволяют считать ген MC1R связанным с породной характеристикой карачаевских овец.
Еще два SNP на 14-й хромосоме, rs413629721 и rs412344125, располагаются в экзонах гена длинной некодирующей РНК. Функции некодирующих РНК до конца не изучены. Однако уже известно, что подобные РНК участвуют в регуляции многих внутриклеточных процессов [21]. Это не только влияние на трансляцию и транскрипцию ДНК, но и прямое воздействие на работу молекулярных комплексов в цитоплазме [22]. Эти замены обнаружены нами всего у 2% животных группы контроля, а у карачаевских овец носителями являются 75% выборки. Исходя из приведенных данных, ген длинной некодирующей РНК может быть связан с породной принадлежностью изучаемых овец.
На 13-й хромосоме все пять отобранных нами замен располагались на разном удалении от одного и того же гена, кодирующего костный морфогенетический белок типа 2 (Bone Morphogenetic Protein-2, BMP2). Вместе с другими белками этой группы он регулирует развитие костной и хрящевой тканей, стимулируя остеогенную дифференцировку клеток [23]. Было обнаружено, что полиморфизм гена BMP2 связан с размерами тела и развитием мышц у крупного рогатого скота [24]. У овец выявлена связь этого гена с фенотипом жирнохвостых овец [25], к которым относится и карачаевская порода. Поэтому мы считаем возможным связывать наличие обнаруженных нами замен с породными особенностями карачаевских овец.
Обнаруженные нами замены rs426400216 и rs415954733 локализуются в области гена лизофосфолипазы I (Lysophospholipase-like protein 1, LYPLAL1). Несмотря на то что дистанция до гена достаточно велика, она укладывается в половину сантиморганиды. Это значит, что исследуемый локус с большой вероятностью наследуется вместе с указанным геном. Лизофосфолипаза принимает участие в метаболизме фосфолипидов клеточной стенки и органелл. Однако, как и у других фосфолипаз, у нее имеются и регуляторные функции за счет включения в процессы синтеза эйкозаноидов и других медиаторов межклеточной коммуникации [26]. Исходя из этого, мы считаем ген LYPLAL1 возможным кандидатом, влияющим на фенотип породы у овец.
Еще две замены на 12-й хромосоме, rs419909433 и rs406878732, находятся очень близко к гену паппализина 2 (Рappalysin 2, PAPPA2). Эта металлопротеиназа специфически разрезает протеин, связывающийся с инсулиноподобным фактором роста 5, важным стимулятором роста костей. Для гена паппализина 2 была установлена связь с размерами тела у мышей [27], что позволяет считать этот ген каким-то образом связанным с реализацией фенотипических признаков карачаевской породы.
В интроне гена, кодирующего один из белков калиевого канала 2 типа (Potassium two pore domain channel subfamily K member 2, KCNK2), нами выявлена замена rs424156280. Этот тип калиевых каналов широко распространен в клетках организма, особенно в нейронах, и относится к классу липидно-управляемых. Обеспечивая поддержание трансмембранного потенциала покоя, каналы этого типа участвуют во всех клеточных процессах, связанных с изменениями электрических параметров мембраны [28]. Важность этих функций указывает на то, что ген KCNK2 вполне может оказывать влияние на параметры фенотипа как через связь с процессами трансмембранного транспорта, так и посредством регуляции вегетативной нервной системой.
Выявленные нами замены, достоверно различающиеся по частоте встречаемости у овец карачаевской породы, по сравнению с другими породами, располагаются рядом с генами, выполняющими важные функции в развитии фенотипических признаков. Для некоторых из них уже имеются данные о связи с внешними параметрами животных. Мы считаем возможным использовать совокупность выявленных замен в качестве молекулярно-генетических маркеров для подтверждения породной принадлежности карачаевских овец. Предложенные гены-кандидаты, которые могут быть напрямую связаны с фенотипом животных, требуют дальнейшего изучения для поиска полиморфизмов, обеспечивающих реализацию признаков.
Работа выполнена при поддержке Минобрнауки РФ в рамках Государственного задания Федерального государственного бюджетного научного учреждения “Северо-Кавказский федеральный научный аграрный центр” АААА-А19-119072690006-3 (тема № 0513-2019-0002).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Yurchenko A., Yudin N., Aitnazarov R. et al. Genome-wide genotyping uncovers genetic profiles and history of the Russian cattle breeds // Heredity. 2018. V. 120(2). P. 125–137. https://doi.org/10.1038/s41437-017-0024-3
Eriksson C., Petitt A. Designing cattle: The social practice of constructing breeds // Anthrozoös. 2020. V. 33(2). P. 175–190. https://doi.org/10.1080/08927936.2020.1719758
Li D., Sun G., Zhang M. et al. Breeding history and candidate genes responsible for black skin of Xichuan black-bone chicken // BMC Genomics. 2020. V. 21(1). P. 1–15. https://doi.org/10.1186/s12864-020-06900-8
Barcaccia G., Lucchin M., Cassandro M. DNA barcoding as a molecular tool to track down mislabeling and food piracy // Diversity. 2016. V. 8(1). P. 2. https://doi.org/10.3390/d8010002
Широкова Н.В., Гетманцева Л.В., Колосов Ю.А. и др. Полиморфизм микросателлитных локусов OarCP549, CSRD247, FCB20 и MAF65 у овец // Аграрная наука Евро-Северо-Востока. 2017. № 5. С. 57–62.
Арнаутовский И.Д., Шарвадзе Р.Л., Гогулов В.А. и др. Племенному животноводству – инновационные, молекулярно-генетические, биотехнические технологии и современные кадры // Дальневосточный аграрный вестник. 2017. T. 3(43). С. 75–83.
McClure M.C., McCarthy J., Flynn P. et al. SNP data quality control in a national beef and dairy cattle system and highly accurate SNP based parentage verification and identification // Frontiers Genet. 2018. № 9. P. 84. https://doi.org/10.3389/fgene.2018.00084
Muñoz M., García-Casco J.M., Alves E. et al. Development of a 64 SNV panel for breed authentication in Iberian pigs and their derived meat products // Meat Sci. 2020. V. 167. P. 108152. https://doi.org/10.1016/j.meatsci.2020.108152
Pasupa K., Rathasamuth W., Tongsima S. Discovery of significant porcine SNPs for swine breed identification by a hybrid of information gain, genetic algorithm, and frequency feature selection technique // BMC Bioinformatics. 2020. V. 21. P. 1–28.https://doi.org/10.1186/s12859-020-3471-4
Денискова Т.Е., Петров С.Н., Доцев А.В. и др. Поиск QTL и генов-кандидатов, ассоциированных с интенсивностью роста овец, на основе GWAS // Биотехнология в растениеводстве, животноводстве и с.-х. микробиологии. 2020. С. 95–97.
Gebrehiwot N.Z., Strucken E.M., Marshall K. et al. SNP panels for the estimation of dairy breed proportion and parentage assignment in African crossbred dairy cattle // Genet. Selection Evol. 2021. № 53(1). P. 1–18. https://doi.org/10.1186/s12711-021-00615-4
Tinarelli S., Ribani A., Utzeri V. et al. Redefinition of the mora romagnola pig breed herd book standard based on DNA markers useful to authenticate its “Mono-Breed” products: An example of sustainable conservation of a livestock genetic Resource // Animals. 2021. V. 11(2). P. 526. https://doi.org/10.3390/ani11020526
Шевхужева Л.А., Арова О.З., Шевхужева С.А. О мерах по дальнейшему расширению производства мяса в Карачаево-Черкесской Республике // Изв. ОГАУ. 2012. № 1-1. С. 166–169.
Болатчиев А.Т., Гогуев Э.Х., Шаманова О.А. и др. Молочная продуктивность и качество молока овцематок карачаевской породы при разных условиях содержания // Овцы, козы, шерстяное дело. 2020. № 1. С. 19–20.
Purcell S., Neale B., Todd-Brown K. et al. PLINK: A tool set for whole-genome association and population-based linkage analyses // Am. J. Hum. Genet. 2007. V. 81. № 3. P. 559–575. https://doi.org/10.1086/519795
Lee Y.H., Park N.H. A Complex cortical malformation caused by a mutation in the tubulin-encoding TUBB3 gene // J. Korean Soc. Radiol. 2020. V. 81(5). P. 1246–1249. https://doi.org/10.3348/jksr.2019.0175
Mutch C.A., Poduri A., Sahin M. et al. Disorders of microtubule function in neurons: imaging correlates // Am. J. Neuroradiology. 2016. V. 37(3). P. 528–535. https://doi.org/10.3174/ajnr.A4552
Goud T.S., Upadhyay R.C., Onteru S.K. et al. Identification and sequence characterization of melanocortin 1 receptor gene (MC1R) in Bos indicus versus (Bos taurus × Bos indicus) // Animal Biotechnology. 2020. V. 31(4). P. 283–294. https://doi.org/10.1080/10495398.2019.1585866
Hepp D., Gonçalves G., Moreira G. et al. Identification of the e allele at the Extension locus (MC1R) in Brazilian Creole sheep and its role in wool color variation // Genet. Mol. Res. 2012. V. 11(11). P. 2997–3006. https://doi.org/10.4238/2012.May.22.5
Koseniuk A., Ropka-Molik K., Rubiś D. et al. Genetic background of coat colour in sheep // Archives Animal Breeding. 2018. V. 61(2). P. 173–178. https://doi.org/10.5194/aab-61-173-2018
Мустафин Р.Н., Хуснутдинова Э.К. Некодирующие части генома как основа эпигенетической наследственности // Вавиловский журн. генетики и селекции. 2017. Т. 21(6). С. 742–749.
Рябых Г.К., Жарикова А.А., Миронов А.А. Анализ данных РНК-ДНК взаимодействий: Сб. трудов 42-й междисциплинарной школы-конф. ИППИ РАН “Информационные технологии и системы 2018”. 2018. С. 659–661.
Lu Z., Liu J., Han J. et al. Association between BMP2 functional polymorphisms and sheep tail type // Animals. 2020. V. 10(4). P. 739. https://doi.org/10.3390/ani10040739
Ma T., Liu G., Lin D. et al. BMP2 gene is a direct target gene of miR-378 in cattle // Indian J. Animal Sci. 2017. V. 87(6). P. 768–769. http://www.icar.org.in/.
Baazaoui I., Bedhiaf-Romdhani S., Mastrangelo S. et al. Genome-wide analyses reveal population structure and identify candidate genes associated with tail fatness in local sheep from a semi-arid area // Animal. 2021. 100193. https://doi.org/10.1016/j.animal.2021.100193
Wepy J.A., Galligan J.J., Kingsley P.J. et al. Lysophospholipases cooperate to mediate lipid homeostasis and lysophospholipid signaling // J. Lipid Res. 2019. V. 60(2). P. 360–374. https://doi.org/10.1194/jlr.M087890
Christians J.K., Hoeflich A., Keightley P.D. PAPPA2, an enzyme that cleaves an insulin-like growth-factor-binding protein, is a candidate gene for a quantitative trait locus affecting body size in mice // Genetics. 2006. V. 173(3). P. 1547–1553. https://doi.org/10.1534/genetics.106.057513
Es-Salah-Lamoureux Z., Steele D.F., Fedida D. Research into the therapeutic roles of two-pore-domain potassium channels // Trends in Pharmacological Sci. 2010. V. 31(12). P. 587–595. https://doi.org/10.1016/j.tips.2010.09.001
Дополнительные материалы отсутствуют.


