Генетика, 2022, T. 58, № 7, стр. 754-764
Идентификация генов монодегидроаскорбатредуктаз (MDHAR) чеснока (Allium sativum L.) и их участие в ответе на заражение Fusarium proliferatum
О. К. Анисимова 1, А. В. Щенникова 1, Е. З. Кочиева 1, М. А. Филюшин 1, *
1 Институт биоинженерии, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” Российской академии наук
119071 Москва, Россия
* E-mail: michel7753@mail.ru
Поступила в редакцию 26.11.2021
После доработки 22.12.2021
Принята к публикации 03.02.2022
- EDN: IOZFZQ
- DOI: 10.31857/S0016675822070037
Аннотация
Монодегидроаскорбатредуктазы (MDHAR) восстанавливают образующуюся в результате нейтрализации активных форм кислорода монодегидроаскорбиновую кислоту до аскорбиновой кислоты, что является одним из основных механизмов поддержания внутриклеточного окислительно-восстановительного статуса. В геноме чеснока (Allium sativum L.) идентифицированы последовательности трех генов MDHAR, кодирующих монодегидроаскорбатредуктазы различной клеточной локализации. В аминокислотных последовательностях AsMDHAR1, AsMDHAR4 и AsMDHAR5 были идентифицированы домен Pyr_redox_2, домены связывания с кофакторами FAD и NAD(P)H и мотив уникальной длинной петли, необходимый для формирования активного сайта фермента. Определены профили экспрессии генов MDHAR1, MDHAR4 и MDHAR5 в различных органах растения чеснока, а также исследовано изменение уровней транскрипции генов AsMDHARs в ответ на заражение патогеном Fusarium proliferatum. Показано, что максимальные уровни транскрипции генов AsMDHARs характерны для листьев, ложного стебля и в случае AsMDHAR1 – корней. В ответ на заражение F. proliferatum выявлено увеличение транскрипции всех трех генов AsMDHAR в корнях растений чеснока, при этом активация генов была сходной и не зависела от степени устойчивости сорта к фузариозной гнили.
Образующиеся в процессе метаболизма в клетках растений активные формы кислорода (АФК) вызывают окисление компонентов клеточных мембран и деградацию нуклеиновых кислот, белков и пигментов, что в конечном итоге может приводить к гибели клеток [1]. Важным компонентом антиоксидантной системы растений является L-аскорбиновая кислота (АК, аскорбат, витамин С), которая нивелирует действие некоторых АФК, напрямую связываясь с ними [2, 3]. Также АК является субстратом для аскорбатпероксидаз (APX; EC 1.11.1.11) и аскорбатоксидаз (АО; ЕС 1.10.3.3), ключевых ферментов окислительно-восстановительной системы растений [3, 4].
В результате реакций нейтрализации АФК и деятельности ферментов APX и AO образуется окисленная форма АК – монодегидроаскорбиновая кислота (MDHA), которая затем может самопроизвольно диспропорционировать с образованием дегидроаскорбиновой кислоты (DHA) и молекулы аскорбата [3]. Образующиеся окисленные формы АК (MDHA и DHA) способны восстанавливаться до аскорбата специфичными ферментами – монодегидроаскорбатредуктазой (MDHAR; EC 1.6.5.4) и дегидроаскорбатредуктазой (DHAR; EC 1.8.5.1) соответственно [3].
Монодегидроаскорбатредуктазы в клетках растений локализуются в хлоропластах, митохондриях, пероксисомах и цитозоле [5, 6]. К настоящему времени гены MDHAR идентифицированы у многих видов растений [5–10]. Показано существенное изменение уровней транскрипции генов MDHAR и активности кодируемых ферментов в ответ на различные абиотические стрессы [1, 5, 9–12]. Так, сверхэкспрессия MDHAR повышает устойчивость к солевому стрессу у растений табака [13] и риса [14]. У лука репчатого Allium cepa в ответ на солевой стресс выявлено увеличение активности MDHAR в симпласте корня и листа [15]. Исследований изменения экспрессии/активности MDHAR в ответ на биотические стрессы крайне мало. Например, у пшеницы Triticum aestivum в ответ на заражение различными расами грибного патогена Puccinia striiformis выявлено увеличение транскрипции гена TaMDHAR4 через 48 ч после заражения, при этом замалчивание гена TaMDHAR4 повышает устойчивость пшеницы к P. striiformis [9]. В зерне кукурузы Zea mays после инокуляции грибами Fusarium proliferatum, F. subglutinans и Aspergillus flavus наблюдается значительное снижение активности MDHAR [10]. Для некоторых видов растений была изучена роль MDHAR в росте и развитии растений, а также в регуляции накопления сахаров и аскорбата в плодах и листьях. Например, у томата снижение активности MDHAR подавляет рост растений, а также снижает содержание сахарозы в плодах и урожайность [16], однако специфичное отсутствие экспрессии MDHAR только в плодах томата на продуктивность не влияет [17]. Сверхэкспрессия MDHAR значительно снижает содержание АК в спелых плодах томата [18]. У лука-порея Allium porrum были определены профили экспрессии трех генов MDHAR, выявлена положительная корреляция транскрипции гена MDHAR4 с содержанием АК в белой части и зеленых листьях растений [19]. Таким образом, на различных видах растений показаны участие MDHAR в определении стрессоустойчивости, а также взаимосвязь активности MDHAR и содержания АК.
Чеснок (Allium sativum L.) является экономически значимой мировой овощной культурой с ежегодным производством более 50 млн т (из них более 200 тыс. т в РФ) (FAO 2019; http://www.fao.org). В процессе роста и хранения чеснок подвержен воздействию различных абиотических и биотических стрессов. Значительные потери урожая чеснока связаны с грибными патогенами, наиболее вредоносными из которых являются грибы рода Fusarium Link, вызывающие гниль луковиц и/или увядание листьев чеснока [20–23]. Гены метаболизма АК у чеснока к настоящему времени не охарактеризованы, их участие в ответе на стрессы ранее не исследовалось. В настоящей работе были проведены идентификация и характеристика генов монодегидроаскорбатредуктаз в геноме чеснока A. sativum, определен профиль их экспрессии в различных органах растения, в том числе в ответ на заражение патогеном Fusarium proliferatum.
МАТЕРИАЛЫ И МЕТОДЫ
Идентификация и структурная характеристика генов MDHARs. Поиск последовательностей генов монодегидроаскорбатредуктаз проводили в геноме и транскриптоме чеснока A. sativum cv. Ershuizao, доступных в базе данных NCBI (PRJNA606385, сборка Garlic.V2.fa; http://www.ncbi.nlm.nih.gov) [24]. В качестве референса использовали последовательности мРНК трех генов MDHAR спаржи Asparagus officinalis (AoMDHAR1 (XP_020260315.1), AoMDHAR4 (XP_020251572.1), AoMDHAR5 (XP_020241046.1)).
Выравнивание и анализ последовательностей MDHARs проводили в программе MEGA 7.0 (https://www.megasoftware.net/). Консервативные домены и мотивы белков MDHARs определяли с помощью программ NCBI-CDD (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) и MEME 5.1.1 (http://meme-suite.org/tools/meme) в сопоставлении с литературными данными. Молекулярный вес, изоэлектрическую точку (pI) и значение индекса гидропатичности (GRAVY) рассчитывали с помощью ExPASy (https://web.expasy.org/protparam/). Прогнозирование образования дисульфидных связей выполняли с помощью сервиса DiANNA 1.1 (http://clavius.bc.edu/~clotelab/DiANNA/). Для поиска трансмембранных спиралей использовали сервис TMHMM server 2.0 (http://www.cbs.dtu.dk/ services/TMHMM/), для поиска доменов связывания с кофакторами – Cofactory-1.0 (https://services.healthtech.dtu.dk/service.php?Cofactory-1.0), для поиска транспортных и сигнальных пептидов – TargetP-2.0 (http://www.cbs.dtu.dk/services/TargetP/). Внутриклеточную локализацию белков MDHARs предсказывали с помощью WoLF PSORT (https:// wolfpsort.hgc.jp/).
Растительный материал. В работе были использованы растения чеснока сортов Сармат и Стрелец. Луковицы сортов Сармат и Стрелец были предоставлены Федеральным научным центром овощеводства (ФНЦО, Московская обл.).
Растения чеснока сорта Сармат были выращены в 2021 г. в условиях экспериментальной установки искусственного климата (ЭУИК, ФИЦ Биотехнологии РАН; день/ночь – 16/8 ч, 22/16°С; освещенность 190 мкМ/(м2/с)). Через 90 дней различные органы (корни, донце, луковица, листья, цветонос, цветоложе и воздушные луковицы) растений были собраны и использованы для анализа тканеспецифичной экспрессии генов MDHARs.
Зубки чеснока сортов Сармат и Стрелец были использованы для исследования изменения уровней транскрипции генов MDHARs в ответ на инфицирование патогеном F. proliferatum. По данным полевых наблюдений ФНЦО (2015–2020 гг.) сорт Сармат устойчив к фузариозной гнили, а сорт Стрелец – восприимчив (в отдельные годы наблюдается гибель 70–90% растений). По 12 зубков каждого сорта чеснока стерилизовали в 70%-ном этаноле (3 мин), промывали стерильной дистиллированной водой, помещали на чашки Петри с влажной фильтровальной бумагой и инкубировали при 25°С в темноте. Через 72 ч, когда наблюдался активный рост корней, половину зубков каждого сорта заражали грибом F. proliferatum изолят Стрелец [23] путем вымачивания в суспензии конидий (~106 конидий/мл) в течение 5 мин. После этого зубки помещали на чашки Петри и инкубировали при 25°С в темноте. Оставшиеся зубки использовали в качестве контроля: вымачивали в дистиллированной воде в течение 5 мин, помещали на чашки Петри и далее инкубировали при 25°С в темноте. Через 24 и 96 ч после заражения визуально оценивали состояние зараженных и контрольных зубков чеснока и отбирали корни (по два биологических повтора для каждого сорта) для анализа экспрессии генов MDHARs.
Выделение РНК, синтез кДНК. Собранный растительный материал растирали в жидком азоте и использовали для выделения РНК с последующей очисткой от примесей ДНК (наборы RNeasy Plant Mini Kit и RNase free DNasy set; QIAGEN, Германия). На основе полученных препаратов РНК синтезировали кДНК (набор GoScriptтм Reverse Transcription System, Promega, США).
Определение профиля экспрессии генов MDHARs. Профиль экспрессии генов MDHARs определяли методом ПЦР в реальном времени (РВ-ПЦР). Для этого на основе идентифицированных кодирующих последовательностей генов MDHARs были разработаны специфичные праймеры (табл. 1). Относительный уровень экспрессии исследуемых генов MDHAR оценивали, используя референсные гены GAPDH [25] и UBQ [26]. Для РВ-ПЦР использовали набор “Реакционная смесь для проведения РВ-ПЦР в присутствии SYBR GreenI и ROX” (ООО “Синтол”, Россия) и термоциклер CFX96 Real-Time PCR Detection System (Bio-Rad Laboratories, США). Реакции проводили в двух биологических и трех технических повторах в следующих условиях: 95°C – 5 мин; 40 циклов (95°C – 15 с, 62°C – 50 с). Для визуализации данных и статистической обработки результатов использовали программу GraphPad Prism v 8 (https://www.graphpad.com).
Таблица 1.
Последовательности праймеров для проведения РВ-ПЦР
| Ген | Последовательности праймеров (5'–3') |
|---|---|
| MDHAR1 | TTTGAACCCTGGCGAGCTTG CTGGCAGTAAGCGTTCTCCA |
| MDHAR4 | CGCAGGTTATGCAGCTCTTG CGCCTACGCAAGTATGAAATGC |
| MDHAR5 | GGGGCTCGCATAGATAAGTTGA TCCCACGGACTTATTCAGCC |
| GAPDH | CCATGTTTGTTGTTGGTGTGAATGAG TGGTGCAGCTAGCGTTGGAGAC |
| UBQ | AAGCCAAGATACAGGACAAG GCATACCACCTCTCAATCTC |
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Идентификация и анализ последовательностей MDHARs чеснока
В результате проведенного поиска в геноме чеснока Allium sativum cv. Ershuizao (PRJNA606385) [24] были идентифицированы три последовательности, гомологичные генам AoMDHAR1, AoMDHAR4 и AoMDHAR5 спаржи As. officinalis (табл. 2). Найденные гены были обозначены как AsMDHAR1, AsMDHAR4 и AsMDHAR5. В версии 01 генома чеснока (сборка ASM1415589v1) ген AsMDHAR1 локализован на хромосоме 5, ген AsMDHAR4 – на хромосоме 8, ген AsMDHAR5 – на хромосоме 6. Однако в версии 02 генома чеснока (сборка Garlic.V2.fa) ген AsMDHAR1 локализован на хромосоме 7, а хромосомная локализация генов AsMDHAR4 и AsMDHAR5 не выяснена (табл. 2), что можно объяснить тем, что взаимосвязь между хромосомами версии 01 (ASM1415589v1) и версии 02 (Garlic.V2.fa) окончательно не определена.
Таблица 2.
Характеристики генов AsMDHARs и кодируемых ими монодегидроаскорбатредуктаз Allium sativum cv. Ershuizao (PRJNA606385)
| Ген | Ген ID [19] | Локализация в геноме | Длина гена, пн | Число экзонов | Размер кДНК, пн | Белок, ао | MW, кДа | pI |
|---|---|---|---|---|---|---|---|---|
| AsMDHAR1 | Asa7G02482.1 | chr5:1670600630–1670609439*; chr7:671746557–671755367** | 8810 | 10 | 1308 | 435 | 47.1 | 5.81 |
| AsMDHAR4 | Asa0G05211.1 | chr8:228808527–228829319*; scaffold6471:143589–164381** | 20 793 | 7 | 1425 | 474 | 52.1 | 9.03 |
| AsMDHAR5 | Asa5G00445.1 | chr6:153009626–153033684*; – | 24 059 | 17 | 1458 | 485 | 52.9 | 6.54 |
Проведенный структурный анализ полногеномных последовательностей AsMDHARs позволил определить размеры и экзон-интронную структуру генов (табл. 2). Экзон-интронная структура полностью соответствовала структуре гомологов AsMDHARs у спаржи As. officinalis (AoMDHAR1, AoMDHAR4 и AoMDHAR5) и Arabidopsis thaliana (AtMDHAR1, AtMDHAR4 и AtMDHAR6).
Аминокислотные последовательности, кодируемые идентифицированными генами монодегидроаскорбатредуктаз чеснока, были высоко гомологичны последовательностям MDHARs As. officinalis (сходство 83% (MDHAR5), 84% (MDHAR4) и 86% (MDHAR1)). Сходство последовательностей AsMDHARs и AtMDHARs составило 70–72%.
С помощью ресурса NCBI-CDD в аминокислотных последовательностях AsMDHARs был идентифицирован консервативный домен Pyr_redox_2 (pfam07992) (рис. 1). Домен Pyr_redox_2 специфичен для семейства пиридиннуклеотиддисульфидоксидоредуктаз (PNDR) и содержит консервативные мотивы связывания с кофакторами FAD/NAD(P)H [11]. С помощью программы Cofactory-1.0 данные мотивы были идентифицированы в белках AsMDHARs (рис. 1). Было показано, что AsMDHAR1 и AsMDHAR4 имеют в своем составе по два мотива (в положениях 5–47 и 163–203 ао (AsMDHAR1), 4–47 и 161–200 ао (AsMDHAR4)), а AsMDHAR5 – три мотива (48–89, 204–243 и 272–310 ао). Характерный для FAD/NAD(P)H-связывающих доменов консенсус GxGxxG [27] был выявлен в обоих FAD/NAD(P)H-связывающих мотивах белков AsMDHAR1 и AsMDHAR4 и в первых двух мотивах белков AsMDHAR5 (рис. 1). Также у всех трех AsMDHARs была обнаружена последовательность уникальной длинной петли (unique long loop; AsMDHAR1 – RLPGFHVCVGSGGERLLP, AsMDHAR4 – RLPAFHTCVGANEDRLTP, AsMDHAR5 – RLPGFHTCVGSGGERQTP), необходимой для формирования активного сайта фермента [11, 28].
Рис. 1.
Выравнивание аминокислотных последовательностей белков MDHARs чеснока A. sativum (As; синий шрифт), спаржи As. officinalis (Ao; зеленый шрифт) и A. thaliana (At; красный шрифт). Домен Pyr_redox_2 (pfam07992) подчеркнут фиолетовым. Черными рамками выделены найденные с помощью программы Cofactory-1.0 мотивы связывания с FAD/NAD(P)H, синим подчеркиванием обозначены консенсусные мотивы GxGxxG/GxGxxxG. Красной рамкой выделен C‑концевой мотив, специфичный для пероксисомальных монодегидроаскорбатредуктаз.

Важную роль в определении функций и поддержании пространственной структуры белка, а также устойчивости к действию денатурирующих агентов и протеолитических ферментов играют дисульфидные связи, формируемые остатками цистеина [11, 29]. Образование дисульфидной связи защищает молекулу фермента от окислительного повреждения, что продемонстрировано на примере глутатионтрансферазы A. thaliana [30]. Нами было обнаружено, что последовательность белка AsMDHAR1 содержит три остатка цистеина в положениях 70, 179 и 199, при этом два из них, Cys179 и Cys199, согласно предсказанию сервиса DiANNA 1.1, могут формировать дисульфидную связь Cys179–Cys199 (YIGLECAAALK–YPEPWCMPRLF). Для шести остатков цистеина AsMDHAR4 (положения 34, 68, 142, 160, 177 и 197) самая высокая вероятность (score 0.99) образования дисульфидной связи была предсказана для Cys68–Cys160 (PAFHTCVGANE–DVMKSCNGGNA), Cys68–Cys197 (PAFHTCVGANE–FPEAHCMARLF) и Cys142–Cys197 (DAENVCYLRNL–FPEAHCMARLF). В последовательности AsMDHAR5 было выявлено пять остатков цистеина в положениях 34, 79, 115, 170 и 368, из которых дисульфидную связь могут образовывать Cys34–Cys170 (MQRVPCRRSFV–IVSTGCESARL) и Cys79–Cys115 (ADGKLCIVTKE–PGFHTCVGSGG). Предсказанные дисульфидные мостики у AsMDHARs могут оказывать стабилизирующее влияние на структуру данных белков в процессе их функционирования в окислительно-восстановительной системе растения чеснока.
Внутриклеточная локализация ферментов связана с той функцией, которую выполняет конкретный компартмент клетки. Растения могут генерировать большое количество АФК через хлоропластные, пероксисомные или митохондриальные пути [31]. При этом ответственными за улавливание АФК посредством ферментативных процессов являются, прежде всего, цитоплазма и хлоропласты [32]. Мы использовали программу WoLF PSORT для предсказания возможной внутриклеточной локализации белков AsMDHAR. Было показано, что активность анализируемых ферментов может быть ассоциирована с АФК-связанными процессами в цитоплазме и органеллах: AsMDHAR1 – в цитоплазме, AsMDHAR5 – в митохондриях и хлоропластах, а AsMDHAR4 – в пероксисомах. Дополнительным свидетельством локализации AsMDHAR4 явилось присутствие на его С-конце мотива WYGRRRRRW (рис. 1), характерного для пероксисомных монодегидроаскорбатредуктаз [11]. Наличие трансмембранной спирали (VYNWHAAAGIAVSVSIAAFAYWY) в положении 445–467 ао, предсказанное с помощью TMHMM server 2.0, определило AsMDHAR4 как интегральный мембранный белок. Подтверждением локализации AsMDHAR5 в митохондриях и хлоропластах стал предсказанный с помощью TargetP-2.0 N-концевой митохондриальный транспортный пептид (1–39 ао).
Филогенетический анализ аминокислотных последовательностей AsMDHARs и их гомологов у Allium cepa, Arabidopsis thaliana и Aporagus officinalis показал четкое разделение на три группы, соответствующие предполагаемой внутриклеточной локализации белков (рис. 2,а). В проводимых ранее исследованиях также наблюдалось разделение MDHARs высших растений на три группы [6, 33]. В этой связи интересно, что все три изоформы фермента, обнаруженные у мха Physcomitrella patens, PpMDHAR1, PpMDHAR2 и PpMDHAR3, напоминали исключительно цитозольную форму монодегидроаскорбатредуктазы, сохранившуюся у всех наземных растений [33]. Это может свидетельствовать о цитозольной форме MDHAR как основной, в результате эволюции которой возникли сначала пероксисомная, а затем хлоропластная/митохондриальная изоформы фермента (рис. 2,а).
Рис. 2.
а – филогенетическое дерево на основе последовательностей белков MDHARs чеснока A. sativum (AsMDHARs, полужирным шрифтом), лука репчатого A. cepa (AcMDHAR1 (GEOY01092914.1), AcMDHAR4 (GFAK01049639.1), AcMDHAR5 (GFAK01071117.1)), спаржи As. officinalis (AoMDHAR1 (XP_020260315.1), AoMDHAR4 (XP_020251572.1), AoMDHAR5 (XP_020241046.1)), A. thaliana (AtMDHAR1 (At3g52880, NP_190856), AtMDHAR2 (At5g03630, NP_568125.1), AtMDHAR3 (At3g09940, NP_566361.1), AtMDHAR4 (At3g27820, NP_189420.1), AtMDHAR6 (At1g63940, NP_849839.1)) и P. patens (PpMDHAR1 (ABA47446.1), PpMDHAR2 (ABA47447.1) и PpMDHAR3 (ABA47448.1)). Дендрограмма построена с помощью программы MEGA 7.0 (метод Maximum Likelihood, 1000 бутстреп-реплик). Предсказание локализации белков сделано с помощью программы WoLF PSORT, а также UniProtKB (для AtMDHARs); б – идентифицированные с помощью MEME 5.1.1 консервативные мотивы в белках MDHARs A. sativum (As), A. cepa (Ac), As. officinalis (Ao) и A. thaliana (At).
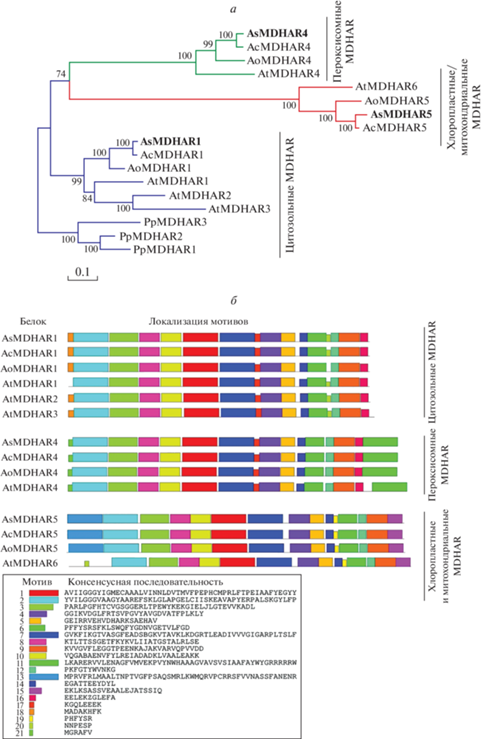
В дополнение к результатам филогенетического анализа была проведена сравнительная характеристика консервативных мотивов (MEME 5.1.1) в последовательностях белков MDHARs A. sativum, A. cepa, As. officinalis и A. thaliana. Всего был идентифицирован 21 консервативный мотив, 12 из которых были общими для всех трех изоформ фермента (рис. 2,б). Цитозольные и пероксисомные MDHARs отличались только консенсусом коротких N-концевых мотивов 18 и 21 и присутствием у пероксисомной формы С-концевого мотива 11 (рис. 2,б), который включал консенсус WYGRRRRRW, специфичный для данного типа монодегидроаскорбатредуктаз [11]. Цитозольные MDHARs (кроме AoMDHAR1) выделялись также наличием мотива 20 (NNPESP) (рис. 2,б). Хлоропластные/ митохондриальные MDHARs отличались от остальных изоформ присутствием С-концевого мотива 15, мотива 19, а также N-концевого мотива 13, соответствующего транспортному митохондриальному пептиду, предсказанному в последовательности AcMDHAR5 и AoMDHAR5 (рис. 2,б). Таким образом, пероксисомная изоформа могла произойти в результате дупликации и модификации гена цитозольной изоформы, сопровождающейся изменением N-области и увеличением размера кодирующей последовательности на С-конце, что предполагает участие данных консенсусов в обеспечении пероксисомной локализации белка. Дальнейшие модификации N- и С-концевых участков (мотивы 13, 15, 19), по всей вероятности, способствовали возникновению хлоропластной/митохондриальной изоформы MDHAR.
Профиль экспрессии генов AsMDHARs в различных органах растения чеснока
Транскриптомные данные A. sativum cv. Ershuizao (PRJNA607255, GSE145455) [24] были использованы для in silico оценки уровней экспрессии генов AsMDHARs в различных тканях растения чеснока (рис. 3). Наибольшая транскрипция гена AsMDHAR1 была обнаружена в корнях, листьях, ложном стебле и на некоторых стадиях развития луковицы. Максимальная экспрессия гена AsMDHAR4 наблюдалась в зеленых фотосинтезирующих тканях – листьях, ложном стебле и проростках. Высокие уровни транскрипции AsMDHAR5 выявлены в процессе формирования луковицы (стадии 1–5), листьях, бутонах и проростках.
Рис. 3.
Тепловая карта экспрессии генов AsMDHAR1, AsMDHAR4 и AsMDHAR5 у A. sativum cv. Ershuizao в корнях, луковицах (1, 2, 3, 4, 5, 6, 7 и 8 соответствуют 192, 197, 202, 207, 212, 217, 222 и 227 дням развития луковицы), листьях, ложном стебле, бутонах, цветках и проростках. Цвета от красного к зеленому показывают градиент экспрессии гена от низкого к высокому.

Методом РВ-ПЦР был определен профиль экспрессии генов AsMDHAR1, AsMDHAR4 и AsMDHAR5 в корне, луковице, донце, псевдостебле, листе, цветоносе, цветоложе и воздушной луковице растения чеснока сорта Сармат (рис. 4,а). В целом было показано, что уровень транскрипции AsMDHAR1 значительно превышает экспрессию двух других генов. К примеру, экспрессия в корнях AsMDHAR1 была выше таковой AsMDHAR5 и AsMDHAR4 почти в 6 и 60 раз соответственно (рис. 4,а). Транскрипты всех трех генов были выявлены во всех анализируемых тканях. Уровни транскрипции AsMDHAR1 в корнях, ложном стебле и листьях были сопоставимо высокими. В донце, цветоносе, цветоложе и воздушных луковицах уровень экспрессии AsMDHAR1 был одинаков и минимален. Максимальный уровень экспрессии гена AsMDHAR4 наблюдался в листьях, а минимальный – в корнях и донце. В луковице и воздушных луковицах уровни транскрипции AsMDHAR4 были сходными. Максимальные уровни экспрессии гена AsMDHAR5 выявлены в ложном стебле и листьях, тогда как в корнях, цветоносе, цветоложе и воздушных луковицах уровни транскрипции гена были в 2 раза ниже (рис. 4,а). Полученные результаты в целом согласуются с данными экспрессионного in silico анализа A. sativum cv. Ershuizao (рис. 3) – максимальные уровни транскрипции генов AsMDHARs характерны для листьев, ложного стебля и, в случае AsMDHAR1 – корней (рис. 4,а).
Рис. 4.
а – профиль экспрессии генов AsMDHAR1, AsMDHAR4 и AsMDHAR5 в различных органах чеснока (сорт Сармат); б – изменение уровней транскрипции генов AsMDHARs в корнях чеснока сортов Сармат (устойчивый) и Стрелец (восприимчивый) через 24 и 96 ч после заражения F. proliferatum. Данные нормализовали по уровням транскрипции генов GAPDH и UBQ и представляли как кратное изменение (среднее ± стандартная ошибка) от контроля (24 ч) сорта Сармат, принятого за 1; * p < 0.01 по сравнению с неинфицированным контролем. hpi – часов после заражения.

С целью определить, участвуют ли гены AsMDHARs в ответе на стрессы, нами было исследовано изменение уровней транскрипции генов AsMDHARs в корнях чеснока в ответ на заражение патогеном F. proliferatum (рис. 4,б) через 24 и 96 ч после заражения зубков сортов Сармат (устойчив к фузариозной гнили) и Стрелец (восприимчив к фузариозной гнили). Признаки фузариозной гнили (развитие белого пушистого мицелия на корнях и неровных светло-коричневых пятен на зубках) появились через 96 ч после заражения только у растений восприимчивого сорта Стрелец.
В целом у анализируемых образцов чеснока, контрастных по устойчивости к фузариозной гнили, наблюдалась сходная динамика экспрессии генов AsMDHARs в ответ на заражение F. proliferatum. В результате анализа у обоих сортов чеснока была выявлена значительная активация транскрипции всех трех генов AsMDHAR1, AsMDHAR4 и AsMDHAR5 в ответ на заражение F. proliferatum, при этом пик увеличения экспрессии приходился на 24 ч после заражения (рис. 4,б). В случае незараженного контроля, в точке (24 ч) уровни транскрипции генов AsMDHARs были в 1.4–2.3 раза выше у восприимчивого сорта Стрелец, чем у устойчивого сорта Сармат; в точке (96 ч) уровни экспрессии генов AsMDHARs у обоих сортов чеснока снизились в 1.3–2.7 раза по сравнению с точкой (24 ч) (рис. 4,б).
Известно, что при воздействии различных абиотических и биотических стрессов в тканях растений увеличивается производство АФК, вызывающих неконтролируемое окисление компонентов клеток [2, 34, 35]. Антиоксидантная система растений, важным компонентом которой является АК и аскорбат-зависимые ферменты, предотвращает накопление АФК и минимизирует негативные последствия их перепроизводства [1, 36].
Ранее сообщалось об увеличении активности MDHAR у растений в ответ на различные абиотические стрессы, такие как засоление, повышенная освещенность, УФ-излучение, низкая температура, засуха, тяжелые металлы, гормоны и др. [12, 35, 37]. Исследования реакции генов MDHAR или активности кодируемых ими ферментов на биотические стрессы ограничиваются несколькими работами. У кукурузы Z. mays в ответ на заражение грибами F. proliferatum, F. subglutinans и A. flavus наблюдалось значительное снижение ферментативной активности MDHAR в зерне [10]. В листьях томата S. lycopersicum, начиная со второго дня грибной инфекции Botrytis cinerea, снижались антиоксидантные свойства митохондрий за счет снижения активности MDHAR [38]. В ответ на заражение тлей в листьях кукурузы было выявлено повышение транскрипции генов MDHAR [39].
С учетом вышесказанного можно предположить, что активация транскрипции генов AsMDHARs в корнях чеснока в ответ на заражение F. proliferatum связана с необходимостью восстановления образующихся при нейтрализации АФК окисленных форм АК для поддержания окислительно-восстановительного баланса. На это также указывает повышенная (в сравнении с другими тканями растения) экспрессия генов MDHAR в листьях и корнях растений – органах, первыми получающих стрессовые сигналы от окружающей среды.
Работа выполнена при финансовой поддержке гранта РНФ (№ 21-76-00007) и Министерства науки и высшего образования РФ.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Gill S.S., Tuteja N. Reactive oxygen species and antioxidant machinery in abiotic stress tolerance in crop plants // Plant Physiol. Biochem. 2010. V. 48. P. 909–930. https://doi.org/10.1016/j.plaphy.2010.08.016
Mittler R. Oxidative stress, antioxidants and stress tolerance // Trends Plant Sci. 2002. V. 7. P. 405–410.
Smirnoff N. Ascorbic acid metabolism and functions: A comparison of plants and mammals // Free Radical Biology and Medicine. 2018. V. 22. P. 116–129. https://doi.org/10.1016/j.freeradbiomed.2018.03.033
De Tullio M.C., Guether M., Balestrini R. Ascorbate oxidase is the potential conductor of a symphony of signaling pathways // Plant Signal. Behav. 2013. V. 8. Article e23213. https://doi.org/10.4161/psb.23213
Leterrier M., Corpas F.J., Barroso J.B. et al. Peroxisomal monodehydroascorbate reductase, genomic clone characterization and functional analysis under environmental stress conditions // Plant Physiol. 2005. V. 138. P. 2111–2123. https://doi.org/10.1104/pp.105.066225
Sano S., Tao S., Endo Y. et al. Purification and cDNA cloning of chloroplastic monodehydroascorbate reductase from spinach // Biosc. Biotechnol. Biochem. 2005. V. 69. P. 762–772.
Chew O., Whelan J., Millar A.H. Molecular definition of the ascorbate-glutathione cycle in Arabidopsis mitochondria reveals dual targeting of antioxidant defenses in plants // J. Biol. Chem. 2003. V. 278. P. 46869–46877.
Zhou F., Zheng B., Wang F. et al. Genome-wide analysis of MDHAR gene family in four cotton species provides insights into fiber development via regulating AsA redox homeostasis // Plants (Basel). 2021. V. 10(2). Article 227. https://doi.org/10.3390/plants10020227
Feng H., Liu W., Zhang Q. et al. TaMDHAR4, a monodehydroascorbate reductase gene participates in the interactions between wheat and Puccinia striiformis f. sp. tritici // Plant Physiol. Biochem. 2014. V. 76. P. 7–16. https://doi.org/10.1016/j.plaphy.2013.12.015
Lanubile A., Maschietto V., De Leonardis S. et al. Defense responses to mycotoxin-producing fungi Fusarium proliferatum, F. subglutinans, and Aspergillus flavus in kernels of susceptible and resistant maize genotypes // Mol. Plant Microbe Interact. 2015. V. 28(5). P. 546–557. https://doi.org/10.1094/MPMI-09-14-0269-R
Negi B., Salvi P., Bhatt D. et al. Molecular cloning, in-silico characterization and functional validation of monodehydroascorbate reductase gene in Eleusine coracana // PLoS One. 2017. V. 12. Article e0187793. https://doi.org/10.1371/journal.pone.0187793
Zhang Y., Li Z., Peng Y. et al. Clones of FeSOD, MDHAR, DHAR genes from white clover and gene expression analysis of ROS-scavenging enzymes during abiotic stress and hormone treatments // Molecules. 2015. V. 20. P. 20939–20954. https://doi.org/10.3390/molecules201119741
Qi Q., Yanyan D., Yuanlin L. et al. Overexpression of SlMDHAR in transgenic tobacco increased salt stress tolerance involving S-nitrosylation regulation // Plant Sci. 2020. V. 299. Article 110609. https://doi.org/10.1016/j.plantsci.2020.110609
Sultana S., Khew C.Y., Morshed M.M. et al. Overexpression of monodehydroascorbate reductase from a mangrove plant (AeMDHAR) confers salt tolerance on rice // J. Plant Physiol. 2012. V. 169. P. 311–318. https://doi.org/10.1016/j.jplph.2011.09.004
García G., Clemente-Moreno M.J., Díaz-Vivancos P. et al. The apoplastic and symplastic antioxidant system in onion: response to long-term salt stress // Antioxidants (Basel). 2020. V. 12. Article 67. https://doi.org/10.3390/antiox9010067
Truffault V., Gest N., Garchery C. et al. Reduction of MDHAR activity in cherry tomato suppresses growth and yield and MDHAR activity is correlated with sugar levels under high light // Plant Cell Environ. 2016. V. 39. P. 1279–1292. https://doi.org/10.1111/pce.12663
Truffault V., Riqueau G., Garchery C. et al. Is monodehydroascorbate reductase activity in leaf tissue critical for the maintenance of yield in tomato? // J. Plant Physiol. 2018. V. 222. P. 1–8. https://doi.org/10.1016/j.jplph.2017.12.012
Haroldsen V.M., Chi-Ham C.L., Kulkarni S. et al. Constitutively expressed DHAR and MDHAR influence fruit, but not foliar ascorbate levels in tomato // Plant Physiol. Biochem. 2011. V. 49. P. 1244–1249. https://doi.org/10.1016/j.plaphy.2011.08.003
Филюшин М.А., Анисимова О.К., Кочиева Е.З., Щенникова А.В. Корреляция содержания аскорбиновой кислоты и профиля экспрессии генов монодегидроаскорбатредуктаз (MDHARs) у лука-порея (Allium porrum L.) // Физиол. растений. 2021. Т. 68(5). С. 501–508. https://doi.org/10.31857/S0015330321050031
Gálvez L., Urbaniak M., Waśkiewicz A. et al. Fusarium proliferatum – causal agent of garlic bulb rot in Spain: genetic variability and mycotoxin production // Food Microbiology. 2017. V. 67. P. 41–48. https://doi.org/10.1016/j.fm.2017.05.006
Filyushin M.A., Anisimova O.K., Kochieva E.Z., Shchennikova A.V. Genome-wide identification and expression of chitinase class I genes in garlic (Allium sativum L.) cultivars resistant and susceptible to Fusarium proliferatum // Plants. 2021. V. 10. Article 720. https://doi.org/10.3390/plants10040720
Anisimova O.K., Shchennikova A.V., Kochieva E.Z., Filyushin M.A. Pathogenesis-related genes of PR1, PR2, PR4 and PR5 families are involved in the response to Fusarium Infection in garlic (Allium sativum L.) // Int. J. Mol. Sci. 2021. V. 22. Article 6688. https://doi.org/10.3390/ijms22136688
Anisimova O.K., Seredin T.M., Danilova O.A., Filyushin M. First report of Fusarium proliferatum causing garlic clove rot in Russian Federation // Plant Dis. 2021. https://doi.org/10.1094/PDIS-12-20-2743-PDN
Sun X., Zhu S., Li N. et al. A chromosome-level genome assembly of garlic (Allium sativum) provides insights into genome evolution and allicin biosynthesis // Mol. Plant. 2020. V. 13. P. 1328–1339. https://doi.org/10.1016/j.molp.2020.07.019
Liu M., Wu Z., Jiang F. Selection and validation of garlic reference genes for quantitative real-time PCR normalization // Plant Cell Tiss. Organ Cult. 2015. V. 122. P. 435–444. https://doi.org/10.1007/s11240-015-0780-9
Schwinn K.E., Ngo H., Kenel F. et al. The onion (Allium cepa L.) R2R3-MYB gene MYB1 regulates anthocyanin biosynthesis // Front. Plant. Sci. 2016. V. 7. Article 1865. https://doi.org/10.3389/fpls.2016.01865
Yoon H.S., Lee H., Lee I.A. et al. Molecular cloning of the monodehydroascorbate reductase gene from Brassica campestris and analysis of its mRNA level in response to oxidative stress // Biochim. Biophys. Acta. 2004. V. 1658. P. 181–186. https://doi.org/10.1016/j.bbabio.2004.05.013
Park A.K., Kim I.S., Do H. et al. Structure and catalytic mechanism of monodehydroascorbate reductase, MDHAR, from Oryza sativa L. japonica // Sci. Rep. 2016. V. 6. Article 33903. https://doi.org/10.1038/srep33903
Ferrè F., Clote P. DiANNA 1.1: an extension of the DiANNA web server for ternary cysteine classification // Nucl. Acids Res. 2006.V. 34. P. 182–185. https://doi.org/10.1093/nar/gkl189
Tossounian M.A., Van Molle I., Wahni K. et al. Disulfide bond formation protects Arabidopsis thaliana glutathione transferase tau 23 from oxidative damage // Biochim. Biophys. Acta Gen. Subj. 2018. V. 1862. P. 775–789. https://doi.org/10.1016/j.bbagen.2017.10.007
Mignolet-Spruyt L., Xu E., Idänheimo N. et al. Spreading the news: subcellular and organellar reactive oxygen species production and signaling // J. Exp. Bot. 2016. V. 67. P. 3831–3844. https://doi.org/10.1093/jxb/erw080
Fan W.J., Feng Y.X., Li Y.H. et al. Unraveling genes promoting ROS metabolism in subcellular organelles of Oryza sativa in response to trivalent and hexavalent chromium // Sci. Total Environ. 2020. V. 744. Article 140951. https://doi.org/10.1016/j.scitotenv.2020.140951
Lunde C., Baumann U., Shirley N.J. et al. Gene structure and expression pattern analysis of three monodehydroascorbate reductase (Mdhar) genes in Physcomitrella patens: implications for the evolution of the MDHAR family in plants // Plant Mol. Biol. 2006. V. 60. P. 259–275. https://doi.org/10.1007/s11103-005-3881-8
García-Limones C., Hervás A., Navas-Cortés J.A. et al. Induction of an antioxidant enzyme system and other oxidative stress markers associated with compatible and incompatible interactions between chickpea // Physiol. Mol. Plant Pathol. 2002. V. 61. P. 325–337.
Pandey P., Singh J., Achary V.M.M., Reddy M.K. Redox homeostasis via gene families of ascorbate-glutathione pathway // Front. Environ. Sci. 2015. V. 3. Article 25. https://doi.org/10.3389/fenvs.2015.00025
Anjum N.A., Gill S.S., Gill R. et al. Metal/metalloid stress tolerance in plants: role of ascorbate, its redox couple, and associated enzymes // Protoplasma. 2014. V. 251. P. 1265–1283. https://doi.org/10.1007/s00709-014-0636-x
Mittova V., Tal M., Volokita M., Guy M. Up-regulation of the leaf mitochondrial and peroxisomal antioxidative systems in response to salt-induced oxidative stress in the wild salt-tolerant tomato species Lycopersicon pennellii // Plant Cell Environ. 2003. V. 26. P. 845–856. https://doi.org/10.1046/j.1365-3040.2003.01016.x
Kuzniak E., Skłodowska M. The effect of Botrytis cinerea infection on the antioxidant profile of mitochondria from tomato leaves // J. Exp. Bot. 2004. V. 55. P. 605–612. https://doi.org/10.1093/jxb/erh076
Sytykiewicz H. Expression patterns of genes involved in ascorbate-glutathione cycle in aphid-infested maize (Zea mays L.) seedlings // Int. J. Mol. Sci. 2016. V. 17. Article 268. https://doi.org/10.3390/ijms17030268
Дополнительные материалы отсутствуют.


