Генетика, 2022, T. 58, № 9, стр. 979-998
Методологии выделения древней ДНК из костной ткани для геномного анализа: подходы и практические рекомендации
Т. В. Андреева 1, 2, 3, *, А. Б. Малярчук 1, А. Д. Сошкина 1, Н. А. Дудко 1, 3, М. Ю. Плотникова 3, Е. И. Рогаев 3, 4, 5, **
1 Центр генетики и генетических технологий, Московский государственный университет
им. М.В. Ломоносова
119234 Москва, Россия
2 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
3 Центр генетики и наук о жизни, Университет “Сириус”
354340 Сочи, Россия
4 Московский государственный университет им. М.В. Ломоносова
119234 Москва, Россия
5 Медицинская школа Чан Массачусетского университета, департамент психиатрии
01545 Шрусбери, США
* E-mail: andreeva@rogaevlab.ru
** E-mail: rogaev@vigg.ru
Поступила в редакцию 01.04.2022
После доработки 28.04.2022
Принята к публикации 29.04.2022
- EDN: KYZRUL
- DOI: 10.31857/S001667582209003X
Аннотация
Появление и развитие методов масштабного параллельного секвенирования открыло новые перспективы в области изучения древних, в том числе вымерших организмов. Многочисленные костные останки, представленные в археологических и музейных коллекциях, зачастую являются единственными источниками информации о древних видах и популяциях. В обзоре обсуждаются особенности костной ткани человека и связанные с ними преимущества и недостатки использования костного материала как источника ДНК для проведения геномного анализа древних людей. Представлены новые методологические подходы к выделению ДНК из древних костных останков человека и ее подготовке к масштабному параллельному секвенированию, а также перспективы и направления дальнейших исследований в новой междисциплинарной области – палеогеномике.
Выделение и анализ ДНК из древних археологических и палеонтологических находок стало возможно не так давно, первые эксперименты были проведены в 80-х гг. XX в., а первая работа по выделению древней ДНК из бедренной кости, датированной XVII в. [1], открыла невероятные перспективы в новой области исследований – палеогенетике. Древняя ДНК является уникальным источником новых знаний об эволюции, популяционной истории, палеопатологии древних видов, популяций и народов. По проведенным ранее оценкам ДНК, пригодная для анализа, не может сохраняться в древних останках более 10–100 тыс. лет [2, 3], при этом ядерная ДНК деградирует примерно в 2 раза быстрее, чем митохондриальная [4]. Эти расчеты были сделаны для ДНК размером 400–800 пн в начале 90-х гг. прошлого века, когда анализ древней ДНК проводили с использованием полимеразной цепной реакции (ПЦР) и секвенирования по методу Сенгера, для применения которых требовались достаточно длинные фрагменты ДНК. Более короткие фрагменты, очевидно, могут сохраняться дольше, но только недавно, с появлением новых технологий секвенирования открылась возможность исследовать такие очень короткие молекулы ДНК (от 20–25 пн), а также были разработаны подходы, позволяющие реконструировать и анализировать молекулы ДНК, представленные в биологических материалах в виде одноцепочечных фрагментов, в том числе претерпевших различные химические модификации, включая постмортальные.
В настоящее время возраст самого древнего палеонтологического образца – шерстистого мамонта, из бивня которого была успешно выделена и проанализирована геномная ДНК, оценивают в более чем один миллион лет [5]. Следует, однако, учитывать, что рассчитанный молекулярный возраст животного (1.65 млн лет) может не соответствовать действительному возрасту останков. Калибровку молекулярного возраста авторы этой работы проводили относительно образцов мамонтов, возраст которых был определен радиоуглеродным методом. Поскольку в настоящее время максимальный абсолютный возраст, который может быть определен с помощью радиоуглеродного метода, составляет не более 55 тыс. лет [6], погрешность молекулярных датировок для всех более древних объектов может быть достаточно высокой.
Значительная часть публикаций по древней ДНК посвящена исследованию останков человека, поэтому в настоящем обзоре сделан акцент на результаты исследования костных останков человека и рассмотрены методы анализа, применимые к ним. Изучение древней ДНК человека, а не животных, связано с одной критически важной проблемой – исследователи должны быть уверены в аутентичности древней ДНК, т.е. в том, что ДНК получена непосредственно из древнего образца, а не выделена из материала, попавшего в образец в результате контаминации (загрязнения), которая может произойти во время раскопок, при хранении археологических образцов и музейных экспонатов, а также при выделении и анализе ДНК. Поэтому при работе с древней ДНК человека необходимо учитывать высокий риск такой контаминации и предпринимать все возможные меры, в том числе представленные в настоящем обзоре, для ее минимизации, своевременного выявления и учета при проведении генетического анализа.
Костная ткань (кости и зубы) – важный источник биологического материала для генетических исследований в криминалистике и в палеогенетике. Благодаря физико-химическим особенностям костной ткани клетки и содержащаяся в них ДНК защищены от разрушающего влияния окружающей среды, а фрагменты костей и зубов часто являются единственным источником древнего биологического материала, доступным для исследования. Возможность выделения и анализа ДНК из древних костных останков была показана более 30 лет назад [1, 7]. С тех пор произошел значительный прогресс в этой новой области исследования, были разработаны многочисленные методы и подходы для работы с древней ДНК, исследование которой существенно обогатило наши знания о процессах, происходивших в прошлом как в жизни людей, так и всего живого мира.
ОСОБЕННОСТИ ДРЕВНЕЙ ДНК
Древняя ДНК, извлеченная из живых организмов, живших много лет назад, имеет ряд специфических особенностей. В живой клетке спонтанные повреждения ДНК исправляются (репарируются) специальными ферментативными системами. После смерти организма клеточные механизмы репарации перестают работать, тогда как химические изменения ДНК продолжают происходить в результате воздействия факторов окружающей среды и компонентов самой клетки. Это ведет к накоплению химических модификаций и постепенному расщеплению молекул ДНК. Для древней ДНК характерны несколько типов постмортальных изменений, среди них наиболее значимыми являются фрагментация и укорочение молекул ДНК, химические модификации нуклеотидов и образование сшивок между молекулами ДНК и с другими соединениями.
Самая частая причина нарушения в структуре ДНК, не только древней, – гидролиз азотистых оснований. Легче всего происходит гидролиз пуриновых оснований – апуринизация (рис. 1,а). Реакция бета-элиминации, которая следует за гидролизом, приводит к одноцепочечным разрывам в молекуле ДНК. Наиболее часто именно пурины представлены в начале последовательности ДНК при геномном секвенировании, что подтверждает разрыв фрагментов ДНК непосредственно по сайтам апуринизации [8]. Гидролитическое дезаминирование азотистых оснований – еще одна химическая модификация ДНК. Цитозин в результате потери аминогруппы превращается в урацил, метил-цитозин – в тимин (рис. 1,б). Таким образом, при анализе участка ДНК с дезаминированным цитозином с использованием полимеразной реакции в комплементарную цепь встраивается аденин, и новый синтезированный фрагмент ДНК содержит мутацию C>T по одной цепи и G>A по комплементарной. Примечательно, что уровень дезаминирования цитозина в двуцепочечной ДНК существенно ниже (в 140 раз), чем в одноцепочечной ДНК [9], таким образом, одноцепочечные разрывы и фрагментация древней ДНК способствуют повышению уровня замен C>T в древней ДНК в целом. Более того, это соответствует наблюдаемому увеличению числа замен C>T на концах ДНК-фрагментов [8] – они часто представлены одноцепочечными участками (так называемыми “липкими” концами).
Рис. 1.
Химические модификации древней ДНК. а – разрыв N-гликозидной связи между сахаром сахаро-фосфатного остова и азотистым основанием приводит к образованию апуринового или апиримидинового сайта, и далее разрыв цепи ДНК в результате реакции β-элиминации; б – дезаминирование цитозина и метилцитозина приводит к образованию урацила и тимина соответственно. ДНК-полимераза при синтезе второй цепи встраивает аденин комплементарно урацилу и тимину, что приводит к возникновению специфичных для древней ДНК мутаций C>T (или G>A в комплементарной цепи ДНК); в – образование тиминовых димеров в результате реакции алкилирования двух тиминов, которые могут располагаться как на одной цепи ДНК, так и на разных. Сшивки между цепями ДНК препятствуют продвижению ДНК-полимеразы по матрице, что затрудняет проведение анализа с использованием реакций амплификации и секвенирования; г – модификации нуклеотидов, блокирующие работу полимеразы, присутствуют в древней ДНК в минорных количествах.
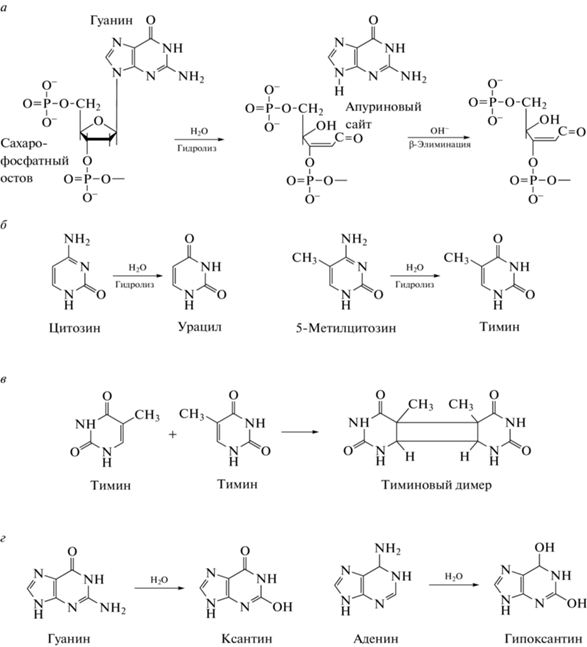
Некоторые модификации древней ДНК блокируют движение ДНК-полимеразы по матрице и соответственно делают невозможным ее амплификацию и секвенирование. К таким модификациям в первую очередь относятся внутри- и межмолекулярные сшивки между цепями ДНК, возникающие в результате реакции алкилирования (рис. 1,в), а также сшивки ДНК с белками в результате реакции Майяра (реакция конденсации между сахаром сахарофосфатного остова ДНК и аминогруппой азотистого основания или аминокислоты). К группе модификаций, затрудняющих анализ древней ДНК в связи с блокировкой движения полимеразы, также относится ряд химических модификаций нуклеотидов [10] (рис. 1,г). Следует отметить, что хотя такие модификации, препятствующие работе ДНК-полимеразы, были выявлены в древней ДНК, экспериментальные исследования показали, что они составляют небольшую долю от всех модификаций древней ДНК [11], а наиболее распространенными изменениями ДНК, которые играют ключевую роль в исследовании древней ДНК, являются дезаминирование цитозина, приводящее к заменам C>T, и высокая степень фрагментации ДНК – средняя длина фрагментов древней ДНК, доступных для анализа, как правило составляет менее 100 пн.
СТРУКТУРА КОСТИ И СОХРАННОСТЬ ДНК
Костные клетки (остеобласты, остеоциты и остеокласты), являющиеся источником ДНК в костях, составляют всего лишь 2% костной ткани, тогда как значительная часть кости образована межклеточным матриксом, в котором 70% и более представлено неорганическими соединениями, преимущественно нерастворимыми кристаллами гидроксиапатитов кальция (общая формула Ca10(PO4)6(OH)2), фосфатом кальция Са3(РО4)2 и в небольшом количестве карбонатом кальция (СаСО3). Основной органический компонент межклеточного матрикса – коллаген. Несмотря на крайне малое содержание клеточных компонентов, а следовательно и ДНК в костной ткани, зачастую только костные останки, которые сохраняются длительное время, могут быть доступны для палеогенетических и геномных исследований.
Деградация костных тканей после смерти организма (диагенез) обусловлена двумя ключевыми процессами. Первый происходит с участием микроорганизмов [12, 13] и протекает относительно быстро, особенно если останки находятся в аэробной среде, где оказываются легко доступны для разложения с участием микроорганизмов (бактерий и грибов). Второй процесс связан с химической деградацией компонентов кости и протекает значительно медленнее. Химическая деградация ДНК в клетках костной ткани в процессе диагенеза костей преимущественно связана с гидролизом пуринов (апуринизацией), а наиболее важными факторами, влияющими на скорость апуринизации, являются температура, влажность и pH. Постмортальные модификации ДНК накапливаются с возрастом археологического биологического образца, однако скорость их появления находится в прямой зависимости от условий, в которых находится соответствующий образец. В результате этого сохранность древнего костного материала может быть лучше, чем у более молодых объектов, а значит качество ДНК, которая может быть выделена из археологичсекого материала, не всегда напрямую связано с его возрастом. Чем больше воды может проникнуть внутрь костей через поры, присутствующие в костях или образующиеся в процессе диагенеза, тем выше скорость деградации как самой кости, так и содержащейся в ней ДНК. Таким образом, размер пор, имеющихся в костях или возникающих в процессе их деградации, оказывает существенное влияние на проникновение воды внутрь костной ткани [14], а следовательно на сохранность в ней ДНК: пористая структура губчатых костей способствует быстрой деградации ДНК, плотные непористые кости менее подвержены деградации благодаря тому, что вода плохо в них проникает. Кроме того, в плотной костной ткани содержится больше кристаллов гидроксиапатита, чем в губчатой. Связанная с гидроксиапатитом ДНК в 1000 раз более устойчива к расщеплению ДНКазами, чем свободная [15]; также было показано, что уровень апуринизации ДНК, связанной с гидроксиапатитом, ниже, чем свободной ДНК [2]. Гидроксиапатит связывает не только ДНК, но и ДНКазы, снижая таким образом их экзонуклеазную активность [16, 17]. Все вместе это дополнительно способствует лучшей сохранности ДНК в плотных костных тканях, чем в пористых губчатых.
Степень сохранности ДНК в кости является ключевым параметром при исследовании древнего костного материала. Зачастую при исследовании особо ценных археологических или музейных образцов важно до выделения ДНК и соответствующего разрушения кости или зуба определить возможное наличие в образце ДНК, предсказать ее количество и оценить перспективность ее использования для генетического анализа. Ряд работ посвящен попыткам исследовать связь структуры и степени сохранности костного материала с содержанием в нем ДНК (например, [18]), а также разработке простых методов такой оценки, аналогичных, например, подходу, предложенному для анализа мягких тканей [19]. Так, в качестве маркеров сохранности ДНК в кости рассматривают степень диагенеза кости, характеризующуюся изменением соотношения органической и неорганической фракций [20, 21] и сохранностью белков [18, 22], а также гистологическое состояние кости [23]. Также показано, что ДНК лучше сохраняется внутри коллагеновых фибрилл, а учитывая тот факт, что в целом ДНК является менее стабильным органическим соединением, чем белки, было высказано предположение, что костные останки, в которых не сохранилась белковая фракция, являются плохим источником древней ДНК [18]. Это предположение, однако, остается спорным – при исследовании серии костей из вечной мерзлоты не было выявлено связи между содержанием в них ДНК и коллагена [21]. В целом в настоящее время отсутствуют эффективные общепринятые методы предварительной оценки сохранности ДНК в костном материале, в большинстве случаев такая оценка может быть сделана только по результатам уже проведенного генетического анализа.
Была показана значимая корреляция сохранности костей и ДНК и температурного режима, в котором находились костные останки, – ДНК практически не удается выделить из древних образцов, найденных в теплых регионах [24], исключением являются египетские мумии, прошедшие специальную обработку, замедляющую процесс разложения тканей [25–27]. Температура оказывает прямое влияние на скорость апуринизации ДНК и, таким образом, является ключевым фактором деградации ДНК (помимо расщепления ферментами клетки и микроорганизмами). Согласно эмпирическим оценкам, при температуре –5°С ДНК может сохраняться до 6.8 млн лет, а ожидаемая длина фрагментов ДНК через 10 тыс. лет будет составлять более 650 пн. При температуре 25°С максимальный срок присутствия следов ДНК в образце составляет 22 тыс. лет, при этом уже спустя 10 тыс лет средняя длина сохранившихся фрагментов оценивается в 2 пн [4]. Удивительно, но в целом ряде работ было показано, что непродолжительное воздействие экстремально высоких температур (например, в результате сжигания останков или тепловой обработки для приема в пищу) не только не приводит к полной деградации ДНК [28–30], но может даже способствовать лучшему сохранению ДНК, предположительно за счет инактивации клеточных нуклеаз при повышении температуры [31]. Более того, некоторые авторы предлагают нагревать кости перед процедурой выделения ДНК [32, 33].
Отдельно следует отметить, что количество ДНК и ее сохранность зависят не только от условий, в которых находился биологический объект после своей смерти. Процедуры извлечения костного материала из археологического памятника, его последующая обработка и хранение в составе археологических и музейных коллекций вносят значительный вклад в качество ДНК, которая может быть извлечена из костного фрагмента. Было показано, что в ряде случаев потери ДНК за короткий период хранения костей в составе таких коллекций могут быть сопоставимы с потерями за время, прошедшее с момента смерти индивида (несколько тысяч лет) [34]. Эти данные показывают необходимость разработки и внедрения в практику проведения полевых археологических исследований новых правил и требований, препятствующих потерям ДНК.
В целом пористые губчатые кости оказываются наиболее разрушенными со временем под воздействием внешних факторов и соответственно практически непригодными для выделения ДНК и проведения генетического анализа (за рядом исключений, см. ниже). Наибольший выход эндогенной ДНК может быть получен при использовании наиболее плотных костей организма человека.
КАКИЕ КОСТИ СКЕЛЕТА ЧЕЛОВЕКА НАИБОЛЕЕ ПРИГОДНЫ ДЛЯ ВЫДЕЛЕНИЯ ДРЕВНЕЙ ДНК?
Зубы являются одним из самых распространенных объектов для исследований древней ДНК благодаря устойчивости их тканей к воздействиям окружающей среды. Основную массу зуба составляет дентин – разновидность костной ткани, главным компонентом которой (60%) является гидроксиапатит [35]. Верхняя часть зуба покрыта защитным слоем эмали, нижняя – цементом. Эмаль, покрывающая зуб, считается самым твердым веществом в человеческом организме, это ее свойство обеспечивает дополнительную защиту ДНК внутри зуба от воздействия внешней среды [36].
Внутренняя часть зуба – пульпа – представляет собой рыхлую волокнистую соединительную ткань и содержит наибольшее число клеток по сравнению с другими тканями зуба, следовательно она является наилучшим источником ДНК [37]. Однако таким свойством обладает только пульпа живого здорового зуба. В процессе жизни индивида, в том числе при поражении зубов болезнями, а также в результате постмортальной деградации зуба, состояние пульпы ухудшается [38, 39]. В некоторых случаях пульпа может подвергаться постмортальной мумификации и длительное время сохраняться в таком состоянии, являясь хорошим источником ДНК, но в большинстве случаев происходит ее полная деградация [39]. Тем не менее благодаря тому, что пульпа зуба включает в себя нервные клетки, а также кровеносные и лимфатические сосуды, по которым патогенные микроорганизмы могут попадать в зуб и быть там обнаружены, пульпа может быть использована как источник ДНК древних патогенов, например возбудителя лепры Mycobacterium leprae [40].
Твердые ткани зуба (дентин, эмаль и цемент) менее подвержены деградации со временем, поэтому ДНК в них сохраняется лучше. Эмаль состоит преимущественно из неорганических соединений (до 97%) и содержит относительно небольшое количество ДНК, что делает ее не самым лучшим объектом для выделения древней ДНК [41]. Основную часть зуба составляет дентин, который считается одним из лучших источников древней ДНК. Было показано, что содержание ядерной ДНК во внутренних частях дентина уменьшается с возрастом индивида [38], что, вероятно, связано с деградацией прилежащей к этим слоям пульпы, однако дентин все равно наиболее часто используется для проведения генетического анализа древних костных останков (например, [42–45], в том числе останков архаичных людей – денисовцев [46]). Содержание мтДНК еще в одной ткани зуба – цементе – в несколько раз выше, чем в дентине [47]. В отличие от дентина количество ядерных клеток в апикальном слое цемента не уменьшается с возрастом [41], а ДНК, выделенная из цемента зуба, характеризуется более высоким содержанием эндогенной ядерной ДНК по сравнению с дентином [48]. Таким образом, цемент, по-видимому, является лучшим источником ДНК из древних зубов, чем дентин. Тем не менее его использование в палеогеномных исследованиях может быть затруднено – несмотря на высокое содержание ДНК, количество цемента в одном зубе крайне мало, толщина цементного слоя зуба составляет всего лишь 20–50 мкм, достигая 150–200 мкм в апексе корня [49]. К другим проблемам, связанным с использованием цемента, можно отнести высокий уровень поверхностного загрязнения, а также сложности с отделением цементной ткани от зуба. Так, например, наиболее эффективный способ получения цемента предполагает удаление коронки зуба с последующей очисткой внутренней части зуба от пульпы и дентина [48].
Среди других костей тела человека наилучшим источником древней ДНК является каменистая кость – часть височной кости (рис. 2,в), которая считается самой плотной костью у млекопитающих [50]. Сравнительный анализ показал, что количество эндогенной ДНК в цементе зуба сопоставимо с ее количеством в каменистой кости, а в каменистой кости плохо сохранившихся древних образцов оно даже выше, чем в цементе [51]. Следует отметить, что наблюдаются существенные различия в количестве ДНК, которая выделяется из разных частей каменистой кости. Так, количество эндогенной ДНК в плотной костной ткани внутри слуховой капсулы в десятки и даже сотни раз выше, чем в плотной части, окружающей внутреннее ухо, и губчатой части височной кости [52]. Плотные участки височной кости, включая улитку, рассматриваются в настоящее время как наиболее перспективный источник древней ДНК, в связи с чем даже разработаны стандартизованные методы извлечения этих участков из височных костей скелета человека [53].
Рис. 2.
Примеры костных объектов, характеризующихся наибольшим количеством древней ДНК: зуб из средневекового захоронения с территории Самарской области (а); слуховые косточки (молоточек, стремечко, наковальня) из захоронения скифского времени с территории Воронежской области (б); фрагмент височной кости с наружным слуховым проходом из захоронения черняховской культуры с территории Одесской области Украины – плотная каменистая кость в области внутреннего уха является одним из лучших источников древней эндогенной ДНК (в).

Недавно в качестве альтернативного источника эндогенной древней ДНК были предложены слуховые косточки (молоточек, стремечко и наковальня, рис. 2,б). Было показано, в том числе в наших собственных исследованиях, что их использование позволяет получить древнюю ДНК сопоставимого с участками каменистой кости качества [54]. Способы извлечения слуховых косточек из черепа представляют собой менее разрушительные для скелета процедуры, чем выпиливание каменистой кости, а структура внутреннего уха может служить источником важной морфологической информации (см, например, [54, 55]); таким образом, использование плотных фрагментов височной кости, извлечение которых требует значительных вмешательств в структуру черепа и изменений его целостности, может быть недопустимо при исследовании уникальных особо ценных древних антропологических объектов.
Помимо наиболее плотных тканей скелета человека в качестве источника древней ДНК используют целый ряд других костей, включая ребра и ключицу, трубчатые кости (плюсны, пястные кости, дистальные фаланги кисти рук, бедренную кость), седалищный бугорок седалищной кости, таранную кость (см. обзор [56]), плечевую кость [57], фрагменты ребер [58], угол нижней челюсти (результаты генетической экспертизы останков императора Николая II Романова [59]). Следует отдельно отметить, что древние костные останки являются перспективным материалом не только для исследования эндогенной ДНК, но и для изучения древних патогенов. Такая ДНК лучше всего сохраняется в васкуляризированных участках костей, именно они с большей вероятностью подвергаются воздействию патогенов, которые переносятся током крови при жизни человека [60]. Поэтому именно такие костные ткани (включая пульпу зуба, ключицу, таранную кость) являются лучшим объектом для поиска и исследования древних патогенов человека, что следует учитывать при планировании экспериментов.
В пилотных работах, связанных с исследованием древних костей, ДНК извлекали из достаточно большого количества кости – до нескольких граммов [1]. По мере совершенствования методов выделения ДНК и последующего генетического анализа все меньшее количество костного материала требуется для проведения работ. В среднем 50–300 мг очищенной от загрязнения кости достаточно для выделения ДНК и проведения геномного анализа. Минимальное количество костной ткани, которое к настоящему времени было использовано для выделения древней ДНК, составляет 9.4 мг [61]. Однако ожидаемое геномное покрытие, которое потенциально может быть достигнуто при использовании такого количества костного порошка (в данном случае оно было оценено авторами в ×2.39), недостаточно для реконструкции полной геномной последовательности и проведения полногеномного анализа не только из-за малого количества ДНК в столь малом костном образце, но и в связи с потенциально высоким уровнем контаминации любых древних образцов, оценить который можно только по результатам проведенного генетического анализа. Поэтому минимизация размера костных фрагментов, используемых для выделения ДНК, всегда должна коррелировать с возможностью получения достаточного для анализа количества эндогенной ДНК. Также необходимо отметить, что исследования древней ДНК требуют проведения нескольких повторных независимых экспериментов, в том числе независимых экстракций ДНК из одного и того же биологического образца, что также следует учитывать при выборе количества материала для анализа ДНК [62].
Кости и зубы из археологических памятников представляют собой ограниченный ресурс и являются частью культурного наследия человечества, поэтому каждый эксперимент должен быть тщательно спланирован, в том числе с этической точки зрения [63]. Целесообразность разрушающего анализа, а также выбор костного материала для него должны проводиться в рамках совместных междисциплинарных исследований генетиков, археологов и антропологов.
МЕТОДИЧЕСКИЕ ПОДХОДЫ ДЛЯ ВЫДЕЛЕНИЯ ДНК ИЗ КОСТЕЙ
Процесс выделения ДНК из любого костного материала, как правило, включает в себя три основных этапа: измельчение кости, растворение ткани с высвобождением ДНК в раствор и, наконец, очистку ДНК от белков и других компонентов клетки (табл. 1). При работе с древней ДНК важным этапом является очистка (деконтаминация) археологического образца от внешних загрязнений. Основными задачами, которые стоят перед исследователями при выделении ДНК из древних костных останков, являются получение максимального количества ДНК из небольшого доступного для анализа фрагмента кости, минимизация повреждений ДНК в процессе выделения и удаление возможных ингибиторов, способных помешать последующим ферментативным реакциям, в которых будет использоваться выделенная ДНК. Следует отметить, что ранние исследования древней ДНК, в рамках которых и были разработаны базовые методические подходы, представленные ниже, были ориентированы преимущественно на получение большого количества ДНК с длиной фрагментов, пригодной для ПЦР-амплификации и последующего секвенирования по методу Сенгера и/или проведения анализа STR-маркеров. В связи с развитием в последние годы методологии масштабного параллельного секвенирования требования к качеству и количеству ДНК, необходимой для геномного анализа, претерпели существенные изменения – появилась возможность использовать для анализа малые количества очень коротких фрагментов ДНК, поэтому разработанные ранее методы выделения ДНК в настоящее время корректируются в соответствии с новыми методами геномного анализа.
Таблица 1.
Обзор методов и растворов, используемых для выделения ДНК из древних костных останков
| Реагент или способ | Механизм действия | Примечание, ссылка на примеры использования |
|---|---|---|
| 1-й этап – деконтаминация | ||
| Стерильная вода | Очистка поверхности костного материала от остатков мягких тканей и внешних загрязнений | |
| Удаление поверхностного слоя кости | Альтернативный способ – высверливание костного порошка из внутренних незагрязненных участков кости | |
| Гипохлорит натрия (NaClO) | [64] | |
| Щелочь (0.1 Н NaOH) | [7] | |
| Уксусная кислота | [65] | |
| Соляная кислота | [58] | |
| Этиловый спирт (70%) | [58] | |
| Фосфатный буфер (50 мM NaH2PO4, pH 7.5, 25 мM Tris, pH 8.0, 50 мM NaCl) |
[58] | |
| Перекись водорода | [66] | |
| УФ-облучение | [67] | |
| 2-й этап – измельчение костной ткани | ||
| Измельчение в жидком азоте | Увеличение поверхности соприкосновения растворов с костной тканью | [1, 68] |
| Высверливание с помощью дрели и сверла | [43] | |
| Измельчение в шаровой мельнице или ее аналогах | [69] | |
| Экстракция ДНК без разрушения кости | Сохранение внешней целостности костных останков | [70–72] |
| 3-й этап – декальцинирование и лизис костной ткани | ||
| ЭДТА (0.45–0.5 М раствор) |
Растворение солей кальция, хелатирование ионов кальция и магния для инактивации клеточных ферментов (нуклеаз) | [73] |
| ЭГТА (0.5 М раствор) | Растворение солей кальция, хелатирование ионов кальция | [45] |
| Фосфатный буфер (50 мM NaH2PO4, pH 7.5, 25 мM Tris, pH 8.0, 50 мM NaCl) |
Фосфат вытесняет ДНК из минерального матрикса кости | [74] |
| Натрий-солевой буфер (100 мM Tris, pH 8.0, 500 мM NaCl, 10 мM EDTA, pH 8.0) |
[74] | |
| Соляная кислота (1 H раствор) | Растворение минеральных компонентов клетки | [7] |
| Буфер на основе гуанидинтиоционата натрия (GuSCN), например, 5 M GuSCN, 50 мM Tris, pH 8.0, 25 мM NaCl, 20 мM EDTA | Вытесняет ДНК из минерального матрикса кости | [70, 75, 76] |
| Дополнительные компоненты в составе лизирующих и декальцинирующих растворов | ||
| Протеиназа К (20 мг/мл) | Расщепление белкового компонента кости | Рекомендуется в большинстве протоколов выделения ДНК |
| Поверхностно-активные вещества (например, лаурилсаркозил натрия, додецилсульфат натрия, Тритон Х-100, поливинилпирролидон, Твин-20 и др.) | Диссоциация клеток и их компонентов | [74–78] |
| Дитиотриетол (DTT) | Удаление дисульфидных мостиков для высвобождения ДНК из белковой фракции | [79] |
| N-Фенацилтиазолиум бромид (PTB) | Разрушение связей между сахарами ДНК и аминокислотами белковой фракции кости | [70] |
| 4-й этап – очистка ДНК | ||
| Фенол-хлороформный метод | Классический метод очистки ДНК от белковой фракции | [80] |
| C использованием силики (в растворе или на колонках) | Связывание молекул ДНК | [74, 81, 82] |
| Преципитация спиртами (изопропанолом) | Осаждение ДНК из раствора | [83, 84] |
Деконтаминация
Одна из главных проблем, с которыми приходится сталкиваться при анализе древней эндогенной ДНК, – ее контаминация (загрязнение) чужеродными фрагментами ДНК, преимущественно из окружающей среды (экзогенной ДНК), содержание которых в древних образцах может значительно превышать содержание эндогенных фрагментов. В ряде случаев эндогенная ДНК может составлять менее 1% от всей получаемой из образца ДНК. Для снижения риска загрязнения современной ДНК все работы с древними образцами проводят в специальных помещениях, изолированных от современного биологического материала, с соблюдением целого ряда условий и правил [62]. Кроме того, перед непосредственной процедурой выделения ДНК из кости проводят деконтаминацию, которая заключается в первую очередь в очистке костных останков от загрязнений, попавших из окружающей среды, и поэтому сконцентрированных преимущественно на поверхности кости. После первоначальной отмывки кости или зуба в стерильной воде и удаления остатков мягких тканей и загрязнений дальнейший процесс деконтаминации проводят различными способами (табл. 1). Наиболее эффективным является механическое удаление верхнего самого загрязненного слоя кости, например, путем шлифовки [85–87] или взятие для анализа только материала из внутренних частей кости или зуба [88]. Кроме механической очистки кости предлагают обрабатывать различными деконтаминирующими растворами. Наиболее широко используемый и эффективный среди них – раствор гипохлорита натрия [64]. Также в литературе представлены успешные результаты выделения ДНК из костей после их очистки с помощью 30%-ной уксусной кислоты [65], 15%-ной соляной кислоты, 70%-ного этилового спирта [58], перекиси водорода [66]. УФ-облучение кости или костного порошка тоже может применяться для деконтаминации [67].
Наряду с описанными выше стандартными методами очистки костных образцов недавно были предложены дополнительные подходы, позволяющие получить значительно более высокий выход эндогенной ДНК. Они основаны на обработке уже измельченной кости или зуба (костного порошка) растворами ЭДТА (этилендиаминтетраацетата натрия), фосфатным буфером или раствором гипохлорита натрия [89] (табл. 1). Фосфатный буфер способствует высвобождению ДНК, связанной на поверхности кости, в раствор. Исследования показали, что удаление ДНК с поверхности костей фосфатным буфером может уменьшить количество бактериальных фрагментов в выделенной ДНК в среднем на 64% [90]. В то же время он малоэффективен для очистки от загрязнения древнего костного образца человеком, в первую очередь современным, в таких случаях рекомендуют использовать обработку костного порошка гипохлоритом натрия. Раствор гипохлорита натрия окисляет органические компоненты кости, приводя к образованию неорганических кристаллических агрегатов, в которых лучше сохраняется эндогенная ДНК [91, 92]. Для предотвращения еще большей деградации ДНК, которая может происходить при взаимодействии ее с гипохлоритом натрия, требуется подбор оптимальной концентрации гипохлорита натрия и времени его воздействия на костный порошок [93, 94].
Подготовка образца к лизису – измельчение костной ткани
Для эффективного проникновения декальцинирующего лизирующего раствора в костную ткань требуется измельчение кости до состояния костной муки (костного порошка). Стандартными методами получения костного порошка являются растирание кости в жидком азоте [1, 68], измельчение с помощью шаровой мельницы или высверливание материала с помощью дрели и сверла [69]. Сравнительный анализ показал, что использование дрели в 30 раз снижает выход мтДНК по сравнению с другими способами измельчения кости. Такой эффект, вероятно, связан с температурной деградацией ДНК, которая происходит за счет высокой скорости вращения сверла и связанного с этим повышения температуры образца [43]. В то же время использование сверла является менее разрушающим методом извлечения материала и позволяет сохранить значительную часть кости в первоначальном виде. Использование шаровой мельницы требует разрушения костного фрагмента большего размера, при этом измельчение происходит в более щадящих условиях, чем с использованием дрели, – без нагрева образца и соответствующей деградации ДНК.
Следует отдельно отметить, что материал археологических памятников, используемый для проведения генетического анализа, является, как правило, уникальным и может представлять археологическую, культурную или даже художественную ценность. В связи с этим актуальным является вопрос о разработке методов анализа древней ДНК без разрушения археологического объекта (кости). Некоторыми исследователями предложен ряд способов такого выделения. Так, например, сравнительный анализ трех различных буферов (основанных на растворах GuSCN, Tris/NaCl и фосфатного буфера) для экстракции ДНК из костей и зубов без их разрушения показал возможность получения митохондриальной ДНК при использовании всех трех растворов. При этом только при использовании буфера с GuSCN авторам работы [70] удалось амплифицировать фрагменты не только митохондриальной ДНК, но и ядерной. Также было показано, что использование 0.5 М раствора ЭДТА с протеиназой К без предварительного измельчения костей и зубов дает возможность анализировать не только митохондриальную, но и геномную ДНК [71]. Примечательно, что для выделения ДНК не обязательно инкубировать весь зуб в соответствующем растворе. Экстракция ДНК путем погружения в раствор корня зуба (при этом возможна защита парафильмом других корней зуба от растворения) может быть перспективным неразрушающим кость методом выделения ДНК. По некоторым оценкам содержание эндогенных молекул ДНК и степень контаминации ДНК, выделенной таким способом из цемента зуба, могут быть сопоставимы с образцами, выделенными из каменистой кости [72]. Некоторые авторы отмечают, что даже повторное проведение неразрушающей процедуры экстракции древней ДНК (до 5 раз) из одного и того же зуба позволяет получить пригодную для генетического анализа ДНК [70, 71]. Очевидно, что при этом происходят химические модификации тканей зуба, однако сохраняется возможность проводить на том же образце морфологические и биохимические исследования, например радиоуглеродную датировку [61].
При использовании подобных методов выделения ДНК без разрушения и измельчения костного образца следует ожидать высокий уровень контаминации эндогенной ДНК в связи с тем, что используемые для инкубации костей растворы способствуют экстракции ДНК преимущественно из поверхностных, наиболее загрязненных другими организмами и бактериями участков ДНК. Удивительно, но это предположение не подтвердилось при выделении ДНК из цемента зуба – предположительно плотная структура цемента защищает его от проникновения внутрь ткани внешних загрязнений [70, 71]. Таким образом, подобные неразрушающие методы могут быть полезны при исследовании уникальных объектов, например из музейных коллекций, разрушение которых недопустимо.
Лизис костной ткани
Следующим этапом выделения ДНК из костей является инкубация костного порошка в различных растворах для лизиса костной ткани и высвобождения ДНК (табл. 1). Наиболее часто для декальцинирования кости используют раствор 0.45–0.5 M ЭДТА, а расщепление белковой фракции костной ткани проводят, как правило, с использованием протеиназы К. В качестве дополнительных компонентов, способствующих лучшему высвобождению ДНК из кости в раствор, успешно применяют различные поверхностно-активные вещества, например лаурилсаркозил натрия [77], додецилсульфат натрия [74], Тритон Х-100 [75, 76], поливинилпирролидон [76, 78], Твин-20 [95] и другие. Для удаления дисульфидных мостиков может использоваться диэтилтиотриетол (DTT) [79], для удаления сшивок ДНК с белками, возникших в результате реакций гликозилирования, применяют PTB (N-phenacylthiazolium bromide) [70]. Лизис костной ткани, как правило, проводят в течение ночи при температуре 55–56°С, однако предлагаются и более длительные инкубации (до нескольких суток). Следует заметить, что длительная инкубация при высокой температуре может приводить к еще большей фрагментации древней ДНК, поэтому при проведении долгой инкубации следует использовать более низкую температуру – 37°С или даже комнатную.
ЭДТА, входящая в состав большинства растворов для лизиса костной ткани, выполняет две ключевые задачи: во-первых, переводит кальций, который входит в состав кости, в растворимое состояние, а во-вторых, являясь хелатирующим агентом, инактивирует ДНКазы, связывая катионы магния и кальция, необходимые для их работы. До сих пор среди исследователей нет единого мнения о необходимости полного растворения кальция на этапе обработки костного порошка ЭДТА. С одной стороны, предлагается проведение полной деминерализации кости перед экстракцией ДНК [73], с другой – целый ряд работ свидетельствует о том, что возможно эффективное выделение древней ДНК из кости без полного растворения костного материала. В нескольких работах было показано, что из нерастворившегося костного порошка может быть выделена дополнительная ДНК после повторной инкубации осадка с новой порцией ЭДТА [96–98].
Интересные результаты были получены в экспериментах по исследованию процессов взаимодействия ДНК с гидроксиапатитом – основным компонентом кости. Было показано, что максимальное количество связанной с гидроксиапатитом ДНК высвобождается после кратковременной (от 15 мин до 2 ч) обработки материала раствором ЭДТА, инкубация длительностью более 4 ч позволяет получить значительно меньшее количество ДНК, а спустя 7 ч от начала инкубации ДНК совсем не детектируется в растворе. Предполагают, что при длительной инкубации происходит повторное связывание ДНК с гидроксиапатитом после насыщения раствора комплексами Ca–ЭДТА [17].
ЭДТА – не единственный агент, который может быть использован для декальцинирования костного материала. В одной из первых работ по выделению ДНК из древних костей их декальцинирование проводили с помощью концентрированной соляной кислоты [7]. Для этих же целей потенциально могут быть использованы ЭГТА (EGTA, ethylene-glycol-ether-diaminetetra-acetic acid), CDTA (1,2 cyclohexane-diaminetetra-acetic acid), лимонная кислота [99]. Эксперименты, проведенные на зубах, показали, что ЭГТА эффективнее, чем ЭДТА, растворяет кальций из дентина при терапии зубных каналов в стоматологии за счет более быстрого по сравнению с ЭДТА связывания им кальция [100, 101]. В недавней работе по исследованию древней ДНК раствор, содержащий ЭГТА, был успешно использован для выделения ДНК из зуба [45], что также подтверждает перспективность его использования в палеогенетических исследованиях.
Среди альтернативных растворов для лизиса костной ткани, наиболее часто используемых для выделения ДНК из древних костей, – растворы, содержащие гуанидинтиоционат натрия (GuSCN) или трис-буферный солевой раствор (табл. 1). В случае последнего для успешного лизиса, так же как и при использовании, например, фосфатного буфера, требуется добавление протеиназы К, необходимой для расщепления белков и высвобождения связанной с ними ДНК [70, 83]. Гуанидинтиоционат натрия является сильным денатурирующим белок агентом, способным денатурировать не только растворенные белки, но и белки, связанные с гидроксиапатитом кости. Предполагается также, что он, как и фосфат, может напрямую вытеснять ДНК из гидроксиапатитного матрикса кости [70]. Благодаря этим его свойствам растворы на основе гуанидинтиоционата достаточно широко применяются в палеогеномных исследованиях [75, 76].
В целом, учитывая тот факт, что ДНК в древних костях связана как с минеральным компонентом кости, так и с белковым, наиболее эффективными являются растворы, способствующие высвобождению ДНК из обеих фракций. Несмотря на то что была показана возможность получения ДНК путем полной деминерализации кости [73], максимальный выход древней ДНК при ее выделении из костных фрагментов возможен только при комбинации в составе раствора для лизиса двух компонентов: одного – для растворения кальция и второго – для расщепления белков. При этом условия проведения лизиса костного материала (в первую очередь время и температура) должны быть подобраны таким образом, чтобы минимизировать фрагментацию ДНК во время проведении этой процедуры.
Очистка ДНК
После экстракции ДНК из кости в раствор ее дальнейшую очистку проводят преимущественно двумя основными способами: с помощью классического фенол-хлороформного метода или с использованием силики (silica, диоксид кремния, SiO2). Силика обладает способностью эффективно связывать на своей поверхности молекулы ДНК, которые далее в связанном состоянии удобно очищать как от компонентов лизирующего раствора, так и от других соединений, которые могут ингибировать последующие ферментативные реакции [102]. В случае использования больших объемов лизирующего раствора, необходимых для успешной деминерализации кости, перед дальнейшей очисткой проводят концентрирование раствора ДНК, например с помощью концентрирующих колонок Amicon-30 [28, 79, 103] или преципитируют ДНК изопропанолом [84]. Необходимо заметить, что при выборе концентрирующих колонок важное значение имеет размер фрагментов ДНК, который ожидается получить и который необходим для проведения дальнейшего анализа. Например, при исследовании останков гейдельбергского человека (Sima de los Huesos) длина фрагментов эндогенной ДНК не превышала 45 пн [98]. Фрагменты ДНК такого размера могут быть полностью потеряны, если проводить выделение ДНК с использованием, например, концентрирующих колонок Amicon-30, размер пор которых может быть слишком большим для коротких фрагментов. В случае подобной сильно фрагментированной древней ДНК необходимо либо использовать мембраны с меньшим размером пор, либо, как было сделано авторами работы [98], использовать другой метод очистки ДНК.
Преципитация 2-изопропанолом – один из наиболее дешевых и простых способов получения ДНК из лизирующего раствора [83, 84], позволяющий быстро очистить ДНК от ингибиторов для последующих реакций. Однако из-за потенциально малого количества ДНК, которое, как правило, удается выделить из костных останков, существует вероятность потери ДНК во время осаждения. Проблема может быть частично решена, например, добавлением в раствор РНКового носителя, который может позволить получить визуализируемый осадок ДНК после преципитации, однако не увеличивает общий выход ДНК [104].
Силика (SiO2) эффективно связывает все формы ДНК (двуцепочечная, одноцепочечная, кольцевая) даже в минимальных количествах. Первоначально метод выделения ДНК с применением силики был основан на связывании ДНК с силикой в растворе [81]. Позже многие коммерческие фирмы разработали так называемые “центрифужные колоноки”, в которых силика нанесена на специальную мембрану [74, 82]. Первое успешное выделение ДНК из костной ткани с использованием колонок фирмы “Qiagen” с силикой было проведено в 1998 г. в рамках генетической экспертизы предполагаемых останков царской семьи Романовых [105]. Использование таких колонок значительно удобнее, чем применение растворов, содержащих силику, а специальные буферные составы, предлагаемые исследователями и фирмами-производителями колонок, позволяют проводить очистку и даже отбор фрагментов ДНК нужного размера. Размер фрагментов ДНК, которые могут быть связаны соответствующими колонками, является критически важным параметром, который необходимо учитывать при выборе колонок с силикой для очистки коротких фрагментов древней ДНК. Многочисленные модификации метода, в основе которого лежит использование силики, предлагают различные составы растворов для связывания ДНК с силикой и для последующей ее отмывки и элюции [42, 74, 75, 106–109], в том числе готовые решения для автоматизации процесса очистки [110].
На данный момент нет универсального способа эффективного выделения древней ДНК. Целая серия недавних работ посвящена сравнению различных методов выделения ДНК из костных останков. Для сравнения используют не только лабораторные протоколы, но и готовые коммерческие наборы, в том числе рекомендованные для использования в криминалистике. Так, например, сравнительные исследования показали большую эффективность выделения ДНК с использованием силики (silica-based метод) на колонках по сравнению со стандартной фенол-хлороформной очисткой [111]. Тем не менее выделение ДНК с использованием фенол-хлороформа может быть успешным в случае использования костей хорошей сохранности, а также для мумифицированных и мягких тканей [19, 73]. Обработка костного порошка гипохлоритом натрия позволяет эффективно удалить контаминацию и получить доступ к ДНК, находящейся в кристаллических агрегатах, не подверженных воздействию гипохлорита натрия [112]. Использование лизирующего буфера PrepFiler BTA из коммерческого набора производства фирмы Thermo Fischer Scientific дает возможность значительно снизить время лизиса костной ткани: 2 ч вместо более чем 12 ч, рекомендуемых при использовании ЭДТА [113].
В настоящее время многие коммерческие фирмы предлагают свои решения для выделения ДНК из костного материала, которые могут быть использованы как для анализа древней ДНК, так и в области криминалистики, а существующее оборудование позволяет автоматизировать процесс выделения ДНК из костей. Одним из перспективных и широко используемых в криминалистике коммерческих наборов для выделения ДНК из костей является PrepFiler™ BTA Forensic DNA Extraction Kit (Thermo Fisher Scientific). После быстрого лизиса со специальным буфером ДНК, высвобожденную из кости в раствор, связывают с магнитными частицами, покрытыми силикой, что позволяет минимизировать потери ДНК. Важным преимуществом данного набора является то, что эффективность связывания с магнитными частицами не зависит от размера фрагментов ДНК, таким образом удается избежать потерь очень коротких фрагментов, характерных для древней ДНК. Набор реагентов DNA Investigator Kit (Qiagen), а также другие колонки этого производителя давно успешно используются в палеогенетических лабораториях благодаря своем удобству – очиcтка ДНК происходит на силике, связанной с мембраной колонки [107]. Следует, однако, заметить, что эффективность связывания ДНК с колонками Qiagen (как и в целом с силикой) напрямую зависит от состава и уровня pH связывающего раствора, поэтому крайне важно при работе с древним костным материалом контролировать эти параметры, в частности использовать специальные растворы, рекомендованные фирмой для очистки очень коротких фрагментов ДНК (менее 70 пн). Кроме того, необходимо минимизировать возможные потери коротких фрагментов ДНК при центрифугировании, проводя связывание ДНК с мембраной и ее отмывку на невысоких скоростях центрифугирования. Недавно на рынке появился набор FSC Casework kit (Promega), позволяющий проводить эффективный лизис костного порошка всего за два часа и в полуавтоматическом режиме выделять ДНК из костей [114], что может быть востребовано при проведении масштабных популяционных палеогенетических исследований.
ГЕНОМНОЕ СЕКВЕНИРОВАНИЕ ДРЕВНЕЙ ДНК
Анализ выделенной ДНК
Проведение любых экспериментов с выделенной ДНК обычно требует проведения ее качественной и количественной оценки, чтобы определить необходимый для той или иной реакции объем полученного препарата ДНК. В ранних работах, когда для выделения ДНК использовали достаточно крупные фрагменты костей или зубов (до нескольких грамм), такая оценка была возможна электрофоретическими или спектрофлуориметрическими методами. В последние годы новые технологии выделения ДНК позволяют проводить экстракцию ДНК из минимальных количеств костных останков – от 9 мг [61]. Выделенную из таких количеств костного материала ДНК зачастую невозможно оценить стандартными количественными методами. В этом случае возможна качественная оценка ДНК, например с использованием высокочувствительных капиллярных анализаторов (рис. 3). Некоторые исследователи предлагают не проводить никаких оценок ДНК, выделенной из 10–50 мг ткани, а напрямую использовать ее, например для приготовления геномных библиотек для секвенирования [115], а оценку аутентичности полученной ДНК проводить уже по результатам секвенирования по наличию “древних усов” (рис. 4) – характерной для древней ДНК картине деградации ДНК, обусловленной заменами цитозина на тимин преимущественно на концах фрагментов древней ДНК [116].
Рис. 3.
Профиль длин фрагментов, характерный для древней ДНК, оцененный с использованием Bioanalyzer 2100 (Agilent), собственные данные. а – ДНК, выделенная из каменистой кости (II–V в. н.э.), содержит преимущественно короткие фрагменты древней эндогенной ДНК длиной менее 200 пн; б – ДНК, выделенная из зуба скифа (III–IV в. до н.э.), содержит только очень короткие фрагменты ДНК менее 100 пн; в – ДНК, выделенная из височной кости скифа (IV в. до н.э.), наряду с короткими фрагментами древней эндогенной ДНК (менее 100 пн) содержит высокомолекулярные (более 1000 пн) фрагменты, наиболее вероятно соответствующие бактериальному загрязнению образца; г – негативный контроль выделения ДНК, должен использоваться при каждой экстракции ДНК. Фрагменты размером 35 и 10 380 пн соответствуют маркеру длин ДНК.
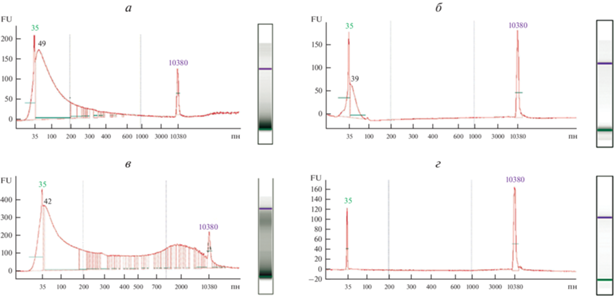
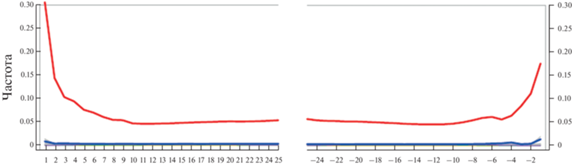
Рис. 4.
Повышение частоты замен C>T (красная кривая на рисунке) к концам фрагментов ДНК, характерное для древней ДНК. Результаты получены с помощью программного пакета MapDamage2 [116] с использованием данных геномного секвенирования ДНК из костных останков скифского захоронения IV в. до н.э.
ДНК-модификации и репарация древней ДНК
Присутствие характерных для древней ДНК модификаций может быть использовано не только для проверки аутентичности выделенной ДНК (рис. 4). В случае высокого уровня контаминации древнего образца современными молекулами ДНК наличие специфичных C>T замен в анализируемых фрагментах является маркером того, что данные фрагменты принадлежат древнему образцу, а не являются современной контаминацией. Такой подход был успешно использован, например, при исследовании геномов неандертальцев – перед проведением геномного анализа из всех полученных в результате секвенирования и картирования на референсный геном коротких последовательностей ДНК отбирали только те, которые содержат на концах “древние” замены C>T [117–119]. С другой стороны, такой тип модификаций древней ДНК может потенциально приводить к ошибкам при непосредственном определении последовательности древней ДНК и анализе мутаций [120]. Чтобы уменьшить число таких ошибок, было предложено использовать специальные ферменты, узнающие урацилы, которые появляются в древней ДНК в результате дезаминирования цитозина. Применение фермента урацил-ДНК-гликозилазы (UDG) позволяет удалить модифицированные основания из молекулы ДНК, а последующее использование эндонуклеазы VIII (Endo VIII) приводит к разрезанию молекулы ДНК в сайте, где был удален нуклеотид [121, 122], альтернативный результат может быть получен при использовании готовой смеси двух ферментов, например смеси USER (New England Biolabs). Таким образом удается понизить число потенциальных ложноположительных замен C>T в последовательности древней ДНК. Главным недостатком такого способа устранения ошибок секвенирования является то, что эндонуклеаза способствует еще большей фрагментации исходно коротких фрагментов древней ДНК. Кроме того, удаление этих “древних” меток делает практически невозможным анализ аутентичности древней ДНК – после обработки ДНК значительно уменьшается размер “древних усов” в анализируемых фрагментах ДНК. Для решения такой проблемы при анализе древних образцов следует проводить анализ как нативной ДНК, содержащей “древние” замены C>T, – для подтверждения ее аутентичности, так и ДНК, обработанной урацил-ДНК-гликозилазой – для снижения вероятности ошибки при определении последовательности ДНК. Протокол частичного удаления урацилов [122], включающий использование ингибитора урацил-ДНК-гликозилазы и позволяющий сохранить “древние” замены только на концах последовательностей, также может быть эффективно использован при анализе древней ДНК. В последние годы все более широкое применение для репарации древней ДНК находят смеси ферментов, которые позволяют проводить не только репарацию дезаминированных цитозинов в древней ДНК, но и удалять ряд других модификаций ДНК, которые потенциально могут вносить ошибки в результаты геномного анализа, например смеси ферментов FFPE DNA Repair Mix [123, 124] и PreCR MIX [125, 126] производства New England Biolabs (табл. 2).
Таблица 2.
Ферменты и смеси ферментов, используемые для репарации древней ДНК
| Наименование фермента или смеси ферментов | Повреждения древней ДНК, которые могут быть репарированы | Ссылка |
|---|---|---|
| Урацил-ДНК-гликозилазы (UDG) | Дезаминирование цитозина | [121] |
| Эндонуклеаза VIII | Апуриновые и апиримидиновые сайты | [121] |
| USER (New England Biolabs) | Дезаминирование цитозина, апуриновые и апиримидиновые сайты | [121] |
| FFPE DNA Repair Mix (New England Biolabs) | Дезаминирование цитозина, апуриновые и апиримидиновые сайты, одноцепочечные разрывы ДНК, модификации 3'-концов фрагментов ДНК, продукты окисления гуанина и пиримидинов | [123, 124] |
| PreCR® Repair Mix (New England Biolabs) | Дезаминирование цитозина, апуриновые и апиримидиновые сайты, одноцепочечные разрывы ДНК, модификации 3'-концов фрагментов ДНК, продукты окисления гуанина и пиримидинов | [125, 126] |
Приготовление фрагментных библиотек и секвенирование
Технология масштабного параллельного секвенирования имеет несколько преимуществ по сравнению с другими методами секвенирования применительно к анализу древней ДНК. Во-первых, в рамках одного эксперимента возможно определение последовательности нуклеотидов всех фрагментов ДНК, которые были выделены из древнего (в том числе костного) материала. Это дает возможность анализировать даже молекулы ДНК, присутствующие в минорном количестве в полученном препарате древней ДНК. Во-вторых, в отличие от классических технологий секвенирования масштабное параллельное секвенирование позволяет определять последовательности экстремально коротких фрагментов ДНК – длиной всего несколько нуклеотидов. Это дает возможность проведения геномного анализа для образцов, ДНК в которых сильно фрагментирована, и секвенирование которых было практически невозможно методом Сенгера.
Основной целью при подготовке ДНК для глубокого секвенирования является добавление к концам фрагментов ДНК специальных адаптерных последовательностей, необходимых для отжига праймера, с которого будет происходить амплификация ДНК при секвенировании. В последние годы был разработан целый ряд подходов для приготовления геномных библиотек из древней ДНК [127]. Среди них наиболее эффективным является метод, основанный на использовании одноцепочечных фрагментов ДНК [115, 128]. В общих чертах он заключается в том, что перед приготовлением геномных библиотек ДНК денатурируют нагреванием для получения одноцепочечных фрагментов, и далее адаптерные последовательности лигируют к каждой одноцепочечной молекуле. Такой подход позволяет значительно повысить количество информативных последовательностей ДНК, поскольку в случае повреждения одной из цепей, что характерно для древней ДНК, комплементарная ей цепь остается пригодна для анализа (значительная часть фрагментов древней ДНК, содержащих одноцепочечные разрывы, при использовании двуцепочечной ДНК для приготовления геномных будет потеряна) [115, 128]. Модификация данного протокола, основанного на одноцепочечной ДНК, также дает возможность на этапе приготовления библиотек селективно отбирать из тотального препарата ДНК только древние фрагменты, в которых присутствуют специфичные для древней ДНК модификации [129].
ЗАКЛЮЧЕНИЕ
Таким образом, за более чем 30 лет исследования ДНК из древних костных останков был наработан огромный методологический материал, позволяющий успешно извлекать ДНК и исследовать уникальный археологический материал, геномный анализ которого дает новую, недоступную ранее информацию об истории человеческих популяций, их возникновении, исчезновении и миграциях. Палеогеномные исследования требуют аккуратного и скрупулезного планирования всех этапов работы, от выбора образцов для анализа (в том числе при проведении археологических раскопок) до интерпретации полученных результатов исследования. Нами рассмотрены существующие методы выделения древней ДНК, используемые при исследовании костных останков человека, предложены направления их модификации и адаптации для использования в полногеномном анализе, а также сделан обзор подходов для проведения геномного анализа этой ДНК с помощью масштабного параллельного секвенирования. Большинство методов может быть успешно адаптировано для анализа других археологических находок, с учетом специфики и анатомии. По мере совершенствования методологий все более древние образцы становятся доступными для проведения генетических исследований, а новые данные в области исследования древней ДНК обязательно внесут свой вклад в понимание исторических и эволюционных процессов прошлого.
Авторы работы выражают благодарность М.В. Добровольской, Н.А. Макарову (Институт археологии РАН) и А.П. Бужиловой (НИИ и Музей антропологии МГУ) за предоставленные образцы костного материала.
Работа выполнена при финансовой поддержке проекта Минобрнауки России, системный номер 075-10-2020-116 (номер гранта 13.1902.21.0023).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Hagelberg E., Sykes B., Hedges R. Ancient bone DNA amplified [5] // Nature. 1989. V. 342. № 6249. P. 485. https://doi.org/10.1038/342485a0
Lindahl T. Instability and decay of the primary structure of DNA // Nature. 1993. V. 362. № 6422. P. 709–715. https://doi.org/10.1038/362709a0
Pääbo S., Wilson A.C. Miocene DNA sequences – a dream come true? // Curr. Biol. 1991. V. 1. № 1. P. 45–46. https://doi.org/10.1016/0960-9822(91)90125-G
Allentoft M.E., Collins M., Harker D. et al. The half-life of DNA in bone: Measuring decay kinetics in 158 dated fossils // Proc. R. Soc. B Biol. Sci. 2012. V. 279. № 1748. P. 4724–4733. https://doi.org/10.1098/rspb.2012.1745
van der Valk T., Pečnerová P., Díez-del-Molino D. et al. Million-year-old DNA sheds light on the genomic history of mammoths // Nature. 2021. V. 591. № 7849. P. 265–269. https://doi.org/10.1038/s41586-021-03224-9
Van Der Plicht J., Bronk Ramsey C., Heaton T.J. et al. Recent developments in calibration for archaeological and environmental samples // Radiocarbon. 2020. V. 62. № 4. P. 1095–1117. https://doi.org/10.1017/RDC.2020.22
Horai S., Hayasaka K., Murayama K. et al. DNA amplification from ancient human skeletal remains and their sequence analysis. // Proc. Japan Acad. Ser. B Phys. Biol. Sci. 1989. V. 65. № 10. P. 229–233. https://doi.org/10.2183/pjab.65.229
Briggs A.W., Stenzel U., Johnson P.L.F. et al. Patterns of damage in genomic DNA sequences from a Neandertal // Proc. Natl Acad. Sci. USA. 2007. V. 104. № 37. P. 14616–14621. https://doi.org/10.1073/pnas.0704665104
Frederico L.A., Shaw B.R., Kunkel T.A. A sensitive genetic assay for the detection of cytosine deamination: determination of rate constants and the activation energy // Biochemistry. 1990. V. 29. № 10. P. 2532–2537. https://doi.org/10.1021/bi00462a015
Dabney J., Meyer M., Pääbo S. Ancient DNA damage // Cold Spring Harb. Perspect. Biol. 2013. V. 5. № 7. https://doi.org/10.1101/cshperspect.a012567
Heyn P., Stenzel U., Briggs A.W. et al. Road blocks on paleogenomes-polymerase extension profiling reveals the frequency of blocking lesions in ancient DNA // Nucl. Acids Res. 2010. V. 38. № 16. https://doi.org/10.1093/nar/gkq572
Trueman C.N., Martill D.M. The long-term survival of bone: The role of bioerosion // Archaeometry. 2002. V. 44. № 3. P. 371–382. https://doi.org/10.1111/1475-4754.t01-1-00070
Bell L.S., Skinner M.F., Jones S.J. The speed of post mortem change to the human skeleton and its taphonomic significance // Forensic Sci. Int. 1996. V. 82. № 2. P. 129–140. https://doi.org/10.1016/0379-0738(96)01984-6
Turner-Walker G., Nielsen-Marsh C.M., Syversen U. et al. Sub-micron spongiform porosity is the major ultra-structural alteration occurring in archaeological bone // Int. J. Osteoarchaeol. 2002. V. 12. № 6. P. 407–414. https://doi.org/10.1002/oa.642
Romanowski G., Lorenz M.G., Wackernagel W. Adsorption of plasmid DNA to mineral surfaces and protection against DNase I // Appl. Environ. Microbiol. 1991. V. 57. № 4. P. 1057–1061. https://doi.org/10.1128/aem.57.4.1057-1061.1991
Demanèche S., Jocteur-Monrozier L., Quiquampoix H., Simonet P. Evaluation of biological and physical protection against nuclease degradation of clay-bound plasmid DNA // Appl. Environ. Microbiol. 2001. V. 67. № 1. P. 293–299. https://doi.org/10.1128/AEM.67.1.293-299.2001
Brundin M., Figdor D., Sundqvist G., Sjögren U. DNA binding to hydroxyapatite: A potential mechanism for preservation of microbial DNA // J. Endod. 2013. V. 39. № 2. P. 211–216. https://doi.org/10.1016/j.joen.2012.09.013
Sosa C., Vispe E., Núñez C. et al. Association between ancient bone preservation and DNA yield: A multidisciplinary approach // Am. J. Phys. Anthropol. 2013. V. 151. № 1. P. 102–109. https://doi.org/10.1002/ajpa.22262
Rogaev E.I., Moliaka Y.K., Malyarchuk B.A. et al. Complete mitochondrial genome and phylogeny of pleistocene mammoth Mammuthus primigenius // PLoS Biol. 2006. V. 4. № 3. P. 0403–0410. https://doi.org/10.1371/journal.pbio.0040073
Götherström A., Collins M.J., Angerbjörn A., Lidén K. Bone preservation and DNA amplification // Archaeometry. 2002. V. 44. № 3. P. 395–404. https://doi.org/10.1111/1475-4754.00072
Schwarz C., Debruyne R., Kuch M. et al. New insights from old bones: DNA preservation and degradation in permafrost preserved mammoth remains // Nucl. Acids Res. 2009. V. 37. № 10. P. 3215–3229. https://doi.org/10.1093/nar/gkp159
Poinar H.N., Artur Stankiewicz B. Protein preservation and DNA retrieval from ancient tissues // Proc. Natl Acad. Sci. USA. 1999. V. 96. № 15. P. 8426–8431. https://doi.org/10.1073/pnas.96.15.8426
Colson I.B., Bailey J.F., Vercauteren M. et al. The preservation of ancient DNA and bone diagenesis // Anc. Biomol. 1997. V. 1. № 2. P. 109–117.
Smith C.I., Chamberlain A.T., Riley M.S. et al. The thermal history of human fossils and the likelihood of successful DNA amplification // J. Hum. EV. 2003. V. 45. № 3. P. 203–217. https://doi.org/10.1016/S0047-2484(03)00106-4
Pääbo S. Molecular cloning of Ancient Egyptian mummy DNA // Nature. 1985. V. 314. № 6012. P. 644–645. https://doi.org/10.1038/314644a0
Schuenemann V.J., Peltzer A., Welte B. et al. Ancient Egyptian mummy genomes suggest an increase of Sub-Saharan African ancestry in post-Roman periods // Nat. Commun. 2017. V. 8. https://doi.org/10.1038/ncomms15694
Gad Y.Z., Abu-Mandil Hassan N., Mousa D.M. et al. Insights from ancient dna analysis of egyptian human mummies: Clues to disease and kinship // Hum. Mol. Genet. 2021. V. 30. № 2. P. R24–R28. https://doi.org/10.1093/hmg/ddaa223
Rogaev E.I., Grigorenko A.P., Moliaka Y.K. et al. Genomic identification in the historical case of the Nicholas II royal family // Proc. Natl Acad. Sci. USA. 2009. V. 106. № 13. P. 5258–5263. https://doi.org/10.1073/pnas.0811190106
Grela M., Jakubczak A., Kowalczyk M. et al. Effectiveness of various methods of DNA isolation from bones and teeth of animals exposed to high temperature // J. Forensic Leg. Med. 2021. V. 78. https://doi.org/10.1016/j.jflm.2021.102131
Emery M.V., Bolhofner K., Winingear S. et al. Reconstructing full and partial STR profiles from severely burned human remains using comparative ancient and forensic DNA extraction techniques // Forensic Sci. Int. Genet. 2020. V. 46. https://doi.org/10.1016/j.fsigen.2020.102272
Ottoni C., Koon H.E.C., Collins M.J. et al. Preservation of ancient DNA in thermally damaged archaeological bone // Naturwissenschaften. 2009. V. 96. № 2. P. 267–278. https://doi.org/10.1007/s00114-008-0478-5
Latham K.E., Miller J.J. DNA recovery and analysis from skeletal material in modern forensic contexts // Forensic Sci. Research. 2019. V. 4. № 1. P. 51–59. https://doi.org/10.1080/20961790.2018.1515594
Prado M., Franco C.M., Fente C.A. et al. Comparison of extraction methods for the recovery, amplification and species-specific analysis of DNA from bone and bone meals // Electrophoresis. 2002. V. 23. № 7–8. P. 1005–1012. https://doi.org/10.1002/1522-2683(200204)23:7/8<1-005::AID-ELPS1005>3.0.CO;2-1
Pruvost M., Schwarz R., Correia V.B. et al. Freshly excavated fossil bones are best for amplification of ancient DNA // Proc. Natl Acad. Sci. USA. 2007. V. 104. № 3. P. 739–744. https://doi.org/10.1073/pnas.0610257104
Bowes J.H., Murray M.M. The chemical composition of teeth // Biochem. J. 1935. V. 29. № 12. P. 2721–2727. https://doi.org/10.1042/bj0292721
Beniash E., Stifler C.A., Sun C.Y. et al. The hidden structure of human enamel // Nat. Commun. 2019. V. 10. № 1. https://doi.org/10.1038/s41467-019-12185-7
Malaver P.C., Yunis J.J. Different dental tissues as source of DNA for human identification in forensic cases // Croat. Med. J. 2003. V. 44. № 3. P. 306–309.
Trivedi R., Chattopadhyay P., Kashyap V.K. A new improved method for extraction of DNA from teeth for the analysis of hypervariable loci // Am. J. Forensic Med. Pathol. 2002. V. 23. № 2. P. 191–196. https://doi.org/10.1097/00000433-200206000-00016
Pötsch L., Meyer U., Rothschild S. et al. Application of DNA techniques for identification using human dental pulp as a source of DNA // Int. J. Legal Med. 1992. V. 105. № 3. P. 139–143. https://doi.org/10.1007/BF01625165
Schuenemann V.J., Singh P., Mendum T.A. et al. Genome-wide comparison of medieval and modern Mycobacterium leprae // Science. 2013. V. 341. № 6142. P. 179–183. https://doi.org/10.1126/SCIENCE.1238286
Higgins D., Austin J.J. Teeth as a source of DNA for forensic identification of human remains: A Review // Science and Justice. 2013. V. 53. № 4. P. 433–441. https://doi.org/10.1016/j.scijus.2013.06.001
Dabney J., Meyer M. Extraction of highly degraded DNA from ancient bones and teeth // Methods Mol. Biol. 2019. V. 1963. P. 25–29. https://doi.org/10.1007/978-1-4939-9176-1_4
Adler C.J., Haak W., Donlon D., Cooper A. Survival and recovery of DNA from ancient teeth and bones // J. Archaeol. Sci. 2011. V. 38. № 5. P. 956–964. https://doi.org/10.1016/j.jas.2010.11.010
Campos P.F., Craig O.E., Turner-Walker G. et al. DNA in ancient bone – Where is it located and how should we extract it? // Ann. Anat. 2012. V. 194. № 1. P. 7–16. https://doi.org/10.1016/j.aanat.2011.07.003
Андреева Т.В., Малярчук А.Б., Григоренко А.П. и др. Археогенетический анализ индивида из захоронения с территории древнего Ярославского Кремля // Краткие сообщ. Института археологии. 2021. Т. 265. С. 209–308.
Sawyer S., Renaud G., Viola B. et al. Nuclear and mitochondrial DNA sequences from two Denisovan individuals // Proc. Natl Acad. Sci. USA. 2015. V. 112. № 51. P. 15696–15700. https://doi.org/10.1073/pnas.1519905112
Adler C.J., Haak W., Donlon D., Cooper A. Survival and recovery of DNA from ancient teeth and bones // J. Archaeol. Sci. 2011. V. 38. № 5. P. 956–964. https://doi.org/10.1016/j.jas.2010.11.010
Damgaard P.B., Margaryan A., Schroeder H. et al. Improving access to endogenous DNA in ancient bones and teeth // Sci. Rep. 2015. V. 5. № 1. P. 11184. https://doi.org/10.1038/srep11184
Freeman E. Periodontium // Oral Histology: Development, Structure, and Function / Ed. Ten Cate A.R. Mosby, St. Louis, 1994. P. 276–312.
Lam Y.M., Chen X., Pearson O.M. Intertaxonomic variability in patterns of bone density and the differential representation of bovid, cervid, and equid elements in the archaeological record // Am. Antiq. 1999. V. 64. № 2. P. 343–362. https://doi.org/10.2307/2694283
Hansen H.B., Damgaard P.B., Margaryan A. et al. Comparing ancient DNA preservation in petrous bone and tooth cementum // PLoS One. 2017. V. 12. № 1. https://doi.org/10.1371/journal.pone.0170940
Pinhasi R., Fernandes D., Sirak K. et al. Optimal ancient DNA yields from the inner ear part of the human petrous bone // PLoS One. 2015. V. 10. № 6. https://doi.org/10.1371/journal.pone.0129102
Pinhasi R., Fernandes D.M., Sirak K., Cheronet O. Isolating the human cochlea to generate bone powder for ancient DNA analysis // Nat. Protoc. 2019. V. 14. № 4. P. 1194–1205. https://doi.org/10.1038/s41596-019-0137-7
Sirak K., Fernandes D., Cheronet O. et al. Human auditory ossicles as an alternative optimal source of ancient DNA // Genome Res. 2020. V. 30. № 3. P. 427–436. https://doi.org/10.1101/gr.260141.119
Ponce de León M.S., Koesbardiati T., Weissmann J.D. et al. Human bony labyrinth is an indicator of population history and dispersal from Africa // Proc. Natl Acad. Sci. USA. 2018. V. 115. № 16. P. 4128–4133. https://doi.org/10.1073/pnas.1717873115
Parker C., Rohrlach A.B., Friederich S. et al. A systematic investigation of human DNA preservation in medieval skeletons // Sci. Rep. 2020. V. 10. № 1. https://doi.org/10.1038/s41598-020-75163-w
Krings M., Stone A., Schmitz R.W. et al. Neandertal DNA sequences and the origin of modern humans // Cell. 1997. V. 90. № 1. P. 19–30. https://doi.org/10.1016/S0092-8674(00)80310-4
Lalueza C., Pérez-Pérez A., Prats E. et al. Lack of founding Amerindian mitochondrial DNA lineages in extinct Aborigines from Tierra del Fuego-Patagonia // Hum. Mol. Genet. 1997. V. 6. № 1. P. 41–46. https://doi.org/10.1093/hmg/6.1.41
Преступление века: Материалы следствия. Документально-архивная хронология событий, связанных с гибелью Российского императора Николая II, его семьи и их приближенных: в 3 т. М.: Следственный комитет РФ, 2021. Т. 2.
Margaryan A., Hansen H.B., Rasmussen S. et al. Ancient pathogen DNA in human teeth and petrous bones // Ecol. EV. 2018. V. 8. № 6. P. 3534–3542. https://doi.org/10.1002/ece3.3924
Hajdinjak M., Fu Q., Hübner A. et al. Reconstructing the genetic history of late Neanderthals // Nature. 2018. V. 555. № 7698. P. 652–656. https://doi.org/10.1038/nature26151
Григоренко А.П., Боринская С.А., Янковский Н.К. Рогаев Е.И. Достижения и особенности в работе с древней ДНК и ДНК из сложных криминалистических образцов // Acta Naturae (русскоязычная версия). 2009. Т. 1. № 3.
Pálsdóttir A.H., Bläuer A., Rannamäe E. et al. Not a limitless resource: Ethics and guidelines for destructive sampling of archaeofaunal remains // R. Soc. Open Sci. 2019. V. 6. № 10. https://doi.org/10.1098/rsos.191059
Kemp B.M., Winters M., Monroe C., Barta J.L. How much DNA is lost? Measuring DNA loss of short-tandem-repeat length fragments targeted by the PowerPlex 16® system using the Qiagen MinElute Purification Kit // Hum. Biol. 2014. V. 86. № 4. P. 313–329. https://doi.org/10.13110/humanbiology.86.4.0313
Montiel R., Malgosa A., Francalacci P. Authenticating ancient human mitochondrial DNA // Hum. Biol. 2001. V. 73. № 5. P. 689–713. https://doi.org/10.1353/hub.2001.0069
Ginther C., Issel-Tarver L., King M.C. Identifying individuals by sequencing mitochondrial DNA from teeth // Nat. Genet. 1992. V. 2. № 2. P. 135–138. https://doi.org/10.1038/ng1092-135
Champlot S., Berthelot C., Pruvost M. et al. An efficient multistrategy DNA decontamination procedure of PCR reagents for hypersensitive PCR applications // PLoS One. 2010. V. 5. № 9. https://doi.org/10.1371/journal.pone.0013042
Morales Colón E., Hernández M., Candelario M. et al. Evaluation of a freezer mill for bone pulverization prior to DNA extraction: an improved workflow for STR analysis // J. Forensic Sci. 2018. V. 63. № 2. P. 530–535. https://doi.org/10.1111/1556-4029.13551
Stone A.C., Milner G.R., Paäbo S., Stoneking M. Sex determination of ancient human skeletons using DNA // Am. J. Phys. Anthropol. 1996. V. 99. № 2. P. 231–238. https://doi.org/10.1002/(SICI)1096-8644(199602)9-9:2<231::AID-AJPA1>3.0.CO;2-1
Rohland N., Siedel H., Hofreiter M. Nondestructive DNA extraction method for mitochondrial DNA analyses of museum specimens // Biotechniques. 2004. V. 36. № 5. P. 814–821. https://doi.org/10.2144/04365st05
Bolnick D.A., Bonine H.M., Mata-Míguez J. et al. Nondestructive sampling of human skeletal remains yields ancient nuclear and mitochondrial DNA // Am. J. Phys. Anthropol. 2012. V. 147. № 2. P. 293–300. https://doi.org/10.1002/ajpa.21647
Harney É., Cheronet O., Fernandes D.M. et al. A minimally destructive protocol for DNA extraction from ancient teeth // Genome Res. 2021. V. 31. № 3. P. 472–483. https://doi.org/10.1101/GR.267534.120
Loreille O.M., Diegoli T.M., Irwin J.A. et al. High efficiency DNA extraction from bone by total demineralization // Forensic Sci. Int. Genet. 2007. V. 1. № 2. P. 191–195. https://doi.org/10.1016/j.fsigen.2007.02.006
Yang D.Y., Eng B., Waye J.S. et al. Technical note: Improved DNA extraction from ancient bones using silica- based spin columns // Am. J. Phys. Anthropol. 1998. V. 105. № 4. P. 539–543. https://doi.org/10.1002/(SICI)1096-8644(199804)1-05:4<539::AID-AJPA10>3.0.CO;2-1
Höss M., Pääbo S. DNA extraction from pleistocene bones by a silica-based purification method // Nucleic Acids Res. 1993. V. 21. № 16. P. 3913–3914. https://doi.org/10.1093/nar/21.16.3913
Voong C.P., Spencer P.S., Navarrete C. V. et al. HLA-DR genotyping and mitochondrial DNA analysis reveal the presence of family burials in a fourth century Romano-British Christian cemetery // Front. Genet. 2017. V. 8. № DEC. https://doi.org/10.3389/fgene.2017.00182
Kalmár T., Bachrati C.Z., Marcsik A., Raskó I. A simple and efficient method for PCR amplifiable DNA extraction from ancient bones // Nucl. Acids Res. 2000. V. 28. № 12. P. 67. https://doi.org/10.1093/nar/28.12.e67
Hofreiter M., Rabeder G., Jaenicke-Després V. et al. Evidence for reproductive isolation between Cave Bear populations // Curr. Biol. 2004. V. 14. № 1. P. 40–43. https://doi.org/10.1016/j.cub.2003.12.035
Leonard J.A., Wayne R.K., Cooper A. Population genetics of Ice Age brown bears // Proc. Natl Acad. Sci. USA. 2000. V. 97. № 4. P. 1651–1654. https://doi.org/10.1073/pnas.040453097
Scheible M., Loreille O., Just R., Irwin J. Short tandem repeat typing on the 454 platform: Strategies and considerations for targeted sequencing of common forensic markers // Forensic Sci. Int. Genet. 2014. V. 12. P. 107–119. https://doi.org/10.1016/j.fsigen.2014.04.010
Boom R., Sol C.J.A., Salimans M.M.M. et al. Rapid and simple method for purification of nucleic acids // J. Clin. Microbiol. 1990. V. 28. № 3. P. 495–503. https://doi.org/10.1128/jcm.28.3.495-503.1990
Gamba C., Hanghøj K., Gaunitz C. et al. Comparing the performance of three ancient DNA extraction methods for high-throughput sequencing // Mol. Ecol. Resour. 2016. V. 16. № 2. P. 459–469. https://doi.org/10.1111/1755-0998.12470
Vigilant L., Hofreiter M., Siedel H., Boesch C. Paternity and relatedness in wild chimpanzee communities // Proc. Natl Acad. Sci. USA. 2001. V. 98. № 23. P. 12890–12895. https://doi.org/10.1073/pnas.231320498
Hänni C., Brousseau T., Laudet V., Stehelin D. Isopropanol precipitation removes PCR inhibitors from ancient bone extracts // Nucl. Acids Res. 1995. V. 23. № 5. P. 881–882. https://doi.org/10.1093/nar/23.5.881
Richards M.B., Sykes B.C., Hedges R.E.M. Authenticating DNA extracted from ancient skeletal remains // J. Archaeol. Sci. 1995. V. 22. № 2. P. 291–299. https://doi.org/10.1006/jasc.1995.0031
Keyser-Tracqui C., Crubézy E., Ludes B. Nuclear and mitochondrial DNA analysis of a 2,000-year-old necropolis in the Egyin Gol valley of Mongolia // Am. J. Hum. Genet. 2003. V. 73. № 2. P. 247–260. https://doi.org/10.1086/377005
Lalueza-Fox C., CalderÓn F.L., Calafell F. et al. MtDNA from extinct Tainos and the peopling of the Caribbean // Ann. Hum. Genet. 2001. V. 65. № 2. P. 137–151. https://doi.org/10.1046/j.1469-1809.2001.6520137.x
Palmirotta R., Verginelli F., Di Tota G. et al. Use of a multiplex polymerase chain reaction assay in the sex typing of DNA extracted from archaeological bone // Int. J. Osteoarchaeol. 1997. V. 7. № 6. P. 605–609. https://doi.org/10.1002/(sici)1099-1212(199711/12)7: 6<605::aid-oa365>3.0.co;2-r
Korlević P., Meyer M. Pretreatment: Removing DNA contamination from ancient bones and teeth using sodium hypochlorite and phosphate // Methods Mol. Biol. 2019. V. 1963. P. 15–19. https://doi.org/10.1007/978-1-4939-9176-1_2
Korlević P., Gerber T., Gansauge M.-T. et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth // Biotechniques. 2015. V. 59. № 2. P. 87–93. https://doi.org/10.2144/000114320
Weiner S., Price P.A. Disaggregation of bone into crystals // Calcif. Tissue Int. 1986. V. 39. № 6. P. 365–375. https://doi.org/10.1007/BF02555173
Salamon M., Tuross N., Arensburg B., Weiner S. Relatively well preserved DNA is present in the crystal aggregates of fossil bones // Proc. Natl Acad. Sci. USA. 2005. V. 102. № 39. P. 13783–13788. https://doi.org/10.1073/pnas.0503718102
Hayatsu H., Pan S. kung, Ukita T. Reaction of sodium hypochlorite with nucleic acids and their constituents // Chem. Pharm. Bull. 1971. V. 19. № 10. P. 2189–2192. https://doi.org/10.1248/cpb.19.2189
Prince A.M., Andrus L. PCR: How to kill unwanted DNA // Biotechniques. 1992. V. 12. № 3.
Rohland N., Glocke I., Aximu-Petri A., Meyer M. Extraction of highly degraded DNA from ancient bones, teeth and sediments for high-throughput sequencing // Nat. Protoc. 2018. V. 13. № 11. P. 2447–2461. https://doi.org/10.1038/s41596-018-0050-5
Ginolhac A., Vilstrup J., Stenderup J. et al. Improving the performance of true single molecule sequencing for ancient DNA // BMC Genomics. 2012. V. 13. № 1. https://doi.org/10.1186/1471-2164-13-177
Orlando L., Ginolhac A., Raghavan M. et al. True single-molecule DNA sequencing of a pleistocene horse bone // Genome Res. 2011. V. 21. № 10. P. 1705–1719. https://doi.org/10.1101/gr.122747.111
Meyer M., Fu Q., Aximu-Petri A. et al. A mitochondrial genome sequence of a hominin from Sima de los Huesos // Nature. 2014. V. 505. № 7483. P. 403–406. https://doi.org/10.1038/nature12788
de Sousa S.M.G., Silva T.L. Efeito do EDTA, EGTA, CDTA e ácido cítrico na desmineralização da dentina radicular: Estudo comparativo // Braz. Oral Res. Sociedade Brasileira de Pesquisa Odontológica – SBPqO. 2005. V. 19. № 3. P. 188–192. https://doi.org/10.1590/S1806-83242005000300006
Medina Cardenas M.E., Calvo Pérez V., Sánchez Planells U. Comparison of the chelating capacity of EDTA and EGTA, a new demineralized agent, on molars in vitro // Rev. Dent. Chile. 1989. V. 80. № 1. P. 4–10.
Tripodi D., D’Ercole S., De Fazio P., Spoto G. Demineralizing action of EGTA in endodontics. // Int. J. Immunopathol. Pharmacol. 2007. V. 20. № 1. Suppl. 1. P. 93–96. https://doi.org/10.1177/039463200702001s18
Simpson T.A., Smith R.J.H. Amplification of mitochondrial DNA from archival temporal bone specimens // Laryngoscope. 1995. V. 105. № 1. P. 28–34. https://doi.org/10.1288/00005537-199501000-00009
Hagelberg E., Clegg J.B. Isolation and characterization of DNA from archaeological bone // Proc. R. Soc. B Biol. Sci. 1991. V. 244. № 1309. P. 45–50. https://doi.org/10.1098/rspb.1991.0049
Higgins D., Kaidonis J., Townsend G., Austin J.J. Evaluation of carrier RNA and low volume demineralization for recovery of nuclear DNA from human teeth // Forensic Sci. Med. Pathol. 2014. V. 10. № 1. P. 56–61. https://doi.org/10.1007/s12024-013-9519-2
Аксючиц Виктор. Покаяние: Материалы правительственной Комиссии по изучению вопросов, связанных с исследованием и перезахоронением останков Российского Императора Николая II и членов его семьи. М.: Выбор, 1998. 288 с.
Katevatis C., Fan A., Klapperich C.M. Low concentration DNA extraction and recovery using a silica solid phase // PLoS One. 2017. V. 12. № 5. https://doi.org/10.1371/journal.pone.0176848
Bouwman A.S., Brown T.A. Comparison between silica-based methods for the extraction of DNA from human bones from 18th to mid-19th century London // Anc. Biomol. 2002. V. 4. № 4. P. 173–178. https://doi.org/10.1080/1358612021000028470
Rohland N., Siedel H., Hofreiter M. A rapid column-based ancient DNA extraction method for increased sample throughput // Mol. Ecol. Resour. 2010. V. 10. № 4. P. 677–683. https://doi.org/10.1111/j.1755-0998.2009.02824.x
Dabney J., Knapp M., Glocke I. et al. Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments // Proc. Natl Acad. Sci. USA. 2013. V. 110. № 39. P. 15758–15763. https://doi.org/10.1073/pnas.1314445110
Amory S., Huel R., Bilić A. et al. Automatable full demineralization DNA extraction procedure from degraded skeletal remains // Forensic Sci. Int. Genet. 2012. V. 6. № 3. P. 398–406. https://doi.org/10.1016/j.fsigen.2011.08.004
Marshall P.L., Stoljarova M., Schmedes S.E. et al. A high volume extraction and purification method for recovering DNA from human bone // Forensic Sci. Int. Genet. 2014. V. 12. P. 155–160. https://doi.org/10.1016/j.fsigen.2014.06.011
Jakubowska J., Maciejewska A., Pawłowski R. Comparison of three methods of DNA extraction from human bones with different degrees of degradation // Int. J. Legal Med. 2012. V. 126. № 1. P. 173–178. https://doi.org/10.1007/s00414-011-0590-5
Hasap L., Chotigeat W., Pradutkanchana J. et al. A novel, 4-h DNA extraction method for STR typing of casework bone samples // Int. J. Legal Med. 2020. https://doi.org/10.1007/s00414-019-02232-9
Duijs F.E., Sijen T. A rapid and efficient method for DNA extraction from bone powder // Forensic Sci. Int. Reports. 2020. V. 2. P. 100099. https://doi.org/10.1016/j.fsir.2020.100099
Gansauge M.T., Meyer M. Single-stranded DNA library preparation for the sequencing of ancient or damaged DNA // Nat. Protoc. 2013. V. 8. № 4. P. 737–748. https://doi.org/10.1038/nprot.2013.038
Jónsson H., Ginolhac A., Schubert M. et al. mapDamage2.0: fast approximate Bayesian estimates of ancient DNA damage parameters. // Bioinformatics. 2013. V. 29. № 13. P. 1682–1684. https://doi.org/10.1093/bioinformatics/btt193
Zavala E.I., Jacobs Z., Vernot B. et al. Pleistocene sediment DNA reveals hominin and faunal turnovers at Denisova Cave // Nature. 2021. https://doi.org/10.1038/s41586-021-03675-0
Vernot B., Zavala E.I., Gómez-Olivencia A. et al. Unearthing Neanderthal population history using nuclear and mitochondrial DNA from cave sediments // Science. 2021. V. 372. № 6542. https://doi.org/10.1126/science.abf1667
Bokelmann L., Hajdinjak M., Peyrégne S. et al. A genetic analysis of the Gibraltar Neanderthals // Proc. Natl Acad. Sci. USA. 2019. V. 116. № 31. P. 15610–15615. https://doi.org/10.1073/pnas.1903984116
Axelsson E., Willerslev E., Gilbert M.T.P., Nielsen R. The effect of ancient DNA damage on inferences of demographic histories // Mol. Biol. EV. 2008. V. 25. № 10. P. 2181–2187. https://doi.org/10.1093/molbev/msn163
Briggs A.W., Stenzel U., Meyer M. et al. Removal of deaminated cytosines and detection of in vivo methylation in ancient DNA // Nucl. Acids Res. 2009. V. 38. № 6. https://doi.org/10.1093/nar/gkp1163
Rohland N., Harney E., Mallick S. et al. Partial uracil-DNA-glycosylase treatment for screening of ancient DNA // Philos. Trans. R. Soc. B Biol. Sci. 2015. V. 370. № 1660. https://doi.org/10.1098/rstb.2013.0624
Gorden E.M., Sturk-Andreaggi K., Marshall C. Repair of DNA damage caused by cytosine deamination in mitochondrial DNA of forensic case samples // Forensic Sci. Int. Genet. 2018. V. 34. P. 257–264. https://doi.org/10.1016/j.fsigen.2018.02.015
Segawa T., Yonezawa T., Mori H. et al. Ancient DNA reveals multiple origins and migration waves of extinct Japanese brown bear lineages // R. Soc. Open Sci. 2021. V. 8. № 8. https://doi.org/10.1098/rsos.210518
Mouttham N., Klunk J., Kuch M. et al. Surveying the repair of ancient dna from bones via high-throughput sequencing // Biotechniques. 2015. V. 59. № 1. P. 19–25. https://doi.org/10.2144/000114307
Andreeva T., Manakhov A., Kunizheva S., Rogaev E.I. Genetic evidence of authenticity of a hair shaft relic from the portrait of Tsesarevich Alexei, the son of the last Russian Emperor // Biochem. 2021. V. 86. № 12.
Orlando L., Gilbert M.T.P., Willerslev E. Reconstructing ancient genomes and epigenomes // Nat. Rev. Genet. 2015. V. 16. № 7. P. 395–408. https://doi.org/10.1038/nrg3935
Gansauge M.-T., Gerber T., Glocke I. et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase // Nucl. Acids Res. 2017. V. 45. № 10. P. e79. https://doi.org/10.1093/nar/gkx033
Gansauge M.-T., Meyer M. Selective enrichment of damaged DNA molecules for ancient genome sequencing // Genome Res. 2014. V. 24. № 9. P. 1543–1549. https://doi.org/10.1101/gr.174201.114
Дополнительные материалы отсутствуют.


