Генетика, 2022, T. 58, № 9, стр. 999-1007
Гены MSMEG_1963 и MSMEG_5597, но не inhA, регулируют устойчивость Mycobacterium smegmatis к триптантринам
С. Г. Фролова 1, 2, В. Н. Даниленко 1, Д. А. Маслов 1, *
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Московский физико-технический институт (Национальный исследовательский университет)
141701 Долгопрудный, Московская обл., Россия
* E-mail: maslov_da@vigg.ru
Поступила в редакцию 29.12.2021
После доработки 08.02.2022
Принята к публикации 15.02.2022
- EDN: JHZTAD
- DOI: 10.31857/S0016675822090089
Аннотация
Распространение лекарственно-устойчивых штаммов Mycobacterium tuberculosis является одной из главных угроз мировому контролю за туберкулезом, требующей постоянной разработки новых противотуберкулезных препаратов (ПТП). Триптантрины – удобные соединения для разработки кандидатов в ПТП, отличающиеся простотой синтеза, низкой токсичностью и активностью как в отношении лекарственно-чувствительных, так и лекарственно-устойчивых штаммов M. tuberculosis. Ранее еноил-(ацил-переносящий белок)-редуктаза InhA была in silico предсказана в качестве возможной биомишени триптантринов, а у спонтанных мутантов M. smegmatis, устойчивых к триптантринам, были обнаружены мутации в генах MSMEG_1963, MSMEG_4427 и MSMEG_5597. В данной работе методами обратной генетики мы показали, что именно мутации в генах MSMEG_1963 и MSMEG_5597 приводят к устойчивости к триптантринам за счет нарушения функции репрессии транскрипции кодируемых ими белков. Установлено, что мутации в генах MSMEG_1963 и MSMEG_5597 приводят к сверхэкспрессии генов MSMEG_1964 и MSMEG_5596 соответственно, кодирующих ферменты, вероятно принимающие участие в окислительно-восстановительной инактивации триптантринов. Также установлено, что InhA не является мишенью триптантринов, поскольку его сверхэспрессия не влияет на чувствительность микобактерий к ним.
Mycobacterium tuberculosis – инфекционный агент, вызывающий самое смертоносное бактериальное заболевание, ежегодно уносящее около 1.4 млн жизней – туберкулез [1].
Стрептомицин – природный антибиотик, производимый бактерией Streptomyces griseus, открытый в 1944 г., стал первым противотуберкулезным препаратом (ПТП) и положил начало терапии туберкулеза [2]. После стрептомицина были открыты и другие эффективные ПТП, что в итоге привело к разработке шестимесячного курса терапии лекарственно-чувствительного туберкулеза с использованием комбинаций нескольких антибиотиков [3]. Однако нарушения режима приема препаратов в конечном счете приводят к возникновению и распространению штаммов M. tuberculosis с множественной (МЛУ) и широкой (ШЛУ) лекарственной устойчивостью. Так, проблема лекарственной устойчивости M. tuberculosis делает необходимым проводить поиск антибиотиков с принципиально новыми механизмами действия. Лекарственная устойчивость M. tuberculosis обусловлена, как правило, возникновением мутаций в генах, кодирующих мишени ПТП, активаторы пролекарств, белки транскрипционные регуляторы эффлюкса и генов резистома [4, 5]. В постгеномную эру обязательным этапом поиска новых ПТП является установление биомишеней и механизмов действия кандидатных молекул во избежание развития перекрестной устойчивости. Сам же поиск при этом может идти как от лекарства к мишени, так и от мишени к лекарству [6].
Привлекательным кандидатом для разработки новых ПТП является триптантрин (рис. 1, 1a), обладающий высокой активностью in vitro в отношении M. tuberculosis, которая также сохраняется в отношении штаммов с МЛУ (МИК = 1 мкг/мл) [7]. Триптантрин – природный алкалоид, принадлежащий к хиназолиновым антибиотикам, впервые был выделен из растения Strobilanthes cusia, Kuntze [8]. Триптантрин обладает широким спектром ингибирующей активности против различных патогенных микроорганизмов, таких как Leishmania donovani [9], Plasmodium falciparum [10], Escherlichia coli [11], Helicobacter pylori [12], а также противогрибковой активностью в отношении родов Trichophyton, Microsporum и Epidermophyron [8, 13]. Ранее исследования с помощью молекулярного докинга показали потенциально высокую аффинность молекулы триптантрина к ферменту еноил-(ацил-переносящий белок)-редуктазе (InhA) M. tuberculosis [14]. InhA играет ключевую роль в биосинтезе миколовых кислот, важных компонент клеточной стенки микобактерий, а также является биомишенью ПТП изониазида [15]. Однако, несмотря на исследования in silico, биомишень и механизм действия триптантрина еще не были установлены in vitro.
Ранее мы показали участие эффлюксной помпы MmpS5-MmpL5 в формировании базового уровня устойчивости Mycobacterium smegmatis к триптантрину и 8-фтортритантрину (рис. 1, 1b) [16]. Также с использованием штаммов M. smegmatis mc2 155 (штамм дикого типа) и M. smegmatis Δmmp5 (штамм с делецией оперона mmpS5-mmpL5 [17]) нами получены две группы спонтанных мутантов, устойчивых к соединению 1b. Мутанты также имели перекрестную устойчивость к 1a, поэтому мы сделали вывод об одинаковом механизме устойчивости M. smegmatis к обоим триптантринам 1a и 1b. Сравнительный геномный анализ устойчивых мутантов выявил три варианта однонуклеотидных замен в гене MSMEG_1963 (R893C, R523C, R523L), один вариант мутации в MSMEG_4427 (L236R), а также инсерцию (Ins50TG), замену (*207R) и инсерцию транспозона в гене MSMEG_5597 [16]. В данной работе описано исследование участия вышеуказанных генов в формировании механизма устойчивости M. smegmatis к триптантринам методами обратной генетики. А также впервые in vitro проверена гипотеза о возможной роли InhA в качестве биомишени триптантринов.
МАТЕРИАЛЫ И МЕТОДЫ
Бактериальные штаммы и условия культивирования
Штаммы Escherichia coli DH5α, используемые для отбора и наработки плазмид, культивировали в среде LB (Amresco, США). Для культивации штаммов M. smegmatis использовали жидкую среду Midllebrook 7H9 (Himedia, Индия) с добавлением OADC (Himedia, Индия), глицерина 0.4% и Tween-80 0.1%, а также триптон-соевый агар (M290, Himedia, Индия) в качестве твердой среды. При необходимости в среду добавляли антибиотик: канамицин 50 мкг/мл, гигромицин (250 мкг/мл для E. coli и 50 мкг/мл M. smegmatis) и ангидротетрациклин 10 нг/мл. Инкубировали бактерии в жидкой среде в шейкере-инкубаторе (Infors HT, Базель, Швейцария) при 37°C и 250 об./мин.
Клонирование генов в экспрессионный вектор
Для анализа фенотипа при сверхэкспрессии генов MSMEG_1963, MSMEG_4427, MSMEG_5597 (как дикого типа, так и их аллельных вариантов), а также генов MSMEG_3151 (inhA M. smegmatis) и Rv1484 (inhA M. tuberculosis), данные гены были клонированы в составе экспрессионного вектора pMIND [18]. Амплификацию генов проводили при помощи набора Q5 для высокоточной ПЦР (NEB, США). В качестве ДНК-матрицы для амплификации генов дикого типа использовали геномную ДНК M. smegmatis mc2 155, а также ДНК M. tuberculosis H37Rv, в то время, как для амплификации аллельных вариантов генов MSMEG_1963, MSMEG_4427, MSMEG_5597 – геномную ДНК соответствующих мутантных штаммов M. smegmatis, описанных ранее [16]. В последовательность праймеров были включены сайты рестрикции NdeI и SpeI, а также в последовательность прямого праймера – сайт посадки рибосомы перед старт-кодоном. Последовательности праймеров приведены в табл. 1. Полученные ампликоны, а также вектор обрабатывали соответствующими эндонуклеазами рестрикции (NEB, США), после чего лигировали с использованием T4 ДНК-лигазы (Thermo Fisher Scientific, США). Лигазную смесь использовали для трансформации химически-компетентных клеток E. coli DH5α, отбор целевых клонов проводили ПЦР-скринингом, после чего подтверждали корректность клонированной последовательности секвенированием по Сэнгеру. Итоговые конструкции использовали для электропорации M. smegmatis mc2 155 по методике, описанной ранее [19].
Таблица 1.
Праймеры, использованные в работе
| ДНК-фрагмент | Праймер | Нуклеотидная последовательность | Сайт рестрикции |
|---|---|---|---|
| Праймеры для клонирования в p2NIL | |||
| HR1963 | pN-1963-f3 | TTTTGAATTCTTAATTAAGCCGACATCGTCGAGAGTT | EcoRI |
| pN-1963-r | TTTTAGTACTTTAATTAAGCGGTAAATTCTGCAGGGAG | ScaI | |
| HR5597 | pN-5597-f | TTTTGGATCCGGCTGTCGACTCCAAAGTCA | BamHI |
| pN-5597-r | GCCCTGCAGGAATGCTAGTT | ||
| HR4427 | pN-4427-f3 | TTTTAAGCTTCTCATCGTCCAGTTGGCGG | HindIII |
| pN-4427-r3 | TTTTGAATTCTTAATTAACCGACTGATTCCACTTCCGA | EcoRI | |
| Праймеры для клонирования в pMIND | |||
| MSMEG_3151 | pM-3151-f | TTTTCATATGAGGAGGAATTGTTATGACAGGCCTACTCGAAGGCAA | NdeI |
| pM-3151-r | TTTTACTAGTTCACAACAGCTGCGTGCTGG | SpeI | |
| Rv1484 | pM-tb-inha-f | TTTTCATATGAGGAGGAATTGTTATGACAGGACTGCTGGACG | NdeI |
| pM-tb-inha-r | TTTTACTAGTCTAGAGCAATTGGGTGTGCG | SpeI | |
| MSMEG_1963 | msmeg-1963-f | TTTTCATATGAGGAGGAATTGTTATGCTCGGACCACTGCAG | NdeI |
| msmeg-1963-r | TTTTACTAGTTCAGGACAGTGAACGGGCCT | SpeI | |
| MSMEG_5597 | msmeg-5597-f | TTTTCATATGAGGAGGAATTGTTATGATGGGACGTCATCGCGAGT | NdeI |
| msmeg-5597-mut-r | TTTTACTAGTCTACGAGGGCGTGCGAGGAGGAATTGGCCGCGA | SpeI | |
| msmeg-5597-r | TTTTACTAGTTCAGCGCTTGCGCGTGG | SpeI | |
| MSMEG_4427 | msmeg-4427-f | TTTTCATATGAGGAGGAATTGTTATGGTGAGTTTGCGCAGCG | NdeI |
| msmeg-4427-r | TTTTACTAGTTCACTGCGGAGGTGACTGTTC | SpeI | |
| Праймеры для ПЦР real-time | |||
| q_5597_f | TCATCGCGAGTTCGATGTGGACAAG | ||
| q_5597_r | GGATGAAGTCCATGTAGCGCTCGTA | ||
| q_5596_f | AGGCTCGACATCCTGGTGAACAAC | ||
| q_5596_r | GAAGCCGGTTCCGATGAAGACGAT | ||
| q_1963_f | GTGGTGATCGATGATGTGCAATGGG | ||
| q_1963_r | ATCGGTCGACAACGACGAGAGC | ||
| q_1964_f | ACGACACCATTCTGATCGTCACCTC | ||
| q_1964_r | TACCCGACGACACCCACGTT | ||
| q_sigA_f | CGAGCTTGTTGATCACCTCGACCAT | ||
| q_sigA_r | CTCGACCTCATCCAGGAAGGCAAC | ||
| q_ftsZ_f | AGCAGCTCCTCGATGTCGTCCTT | ||
| q_ftsZ_r | GCCTGAAGGGCGTCGAGTTCAT | ||
Конструирование рекомбинантных штаммов M. smegmatis
Для создания штаммов M. smegmatis, несущих единичные мутации в генах MSMEG_1963, MSMEG_4427 и MSMEG_5597 на основе штамма M. smegmatis mc2 155, применяли метод гомологичной рекомбинации с использованием суицидной системы p2NIL/pGOAL19 [20]. Вкратце, с использованием набора Q5 для высокоточной ПЦР (NEB, США), праймеров, содержащих специфичные сайты рестрикции (табл. 1), и геномной ДНК спонтанных мутантов M. smegmatis, имеющих целевые мутации [16], были наработаны соответствующие фрагменты, содержащие мутации в целевых генах, а также плечи длиной ~1500 пн для гомологичной рекомбинации (фрагменты HR1963, HR4427 и HR5597). После обработки соответствующими эндонуклеазами рестрикции (Thermo Fischer Scientific, США) ампликонов и плазмиды p2NIL, производили лигирование и отбор целевых клонов по методике, описанной выше. Далее в целевые плазмиды по сайту рестрикции PacI клонировали кассету с маркерными генами из плазмиды pGOAL19. Конечными плазмидами электропорировали клетки M. smegmatis mc2 155. Отбор единичных кроссоверов проводили на триптон-соевом агаре, содержащем канамицин 50 мкг/мл, гигромицин 50 мкг/мл и X-Gal (50 мкг/мл): плазмида p2NIL не имеет микобактериального ориджина репликации, таким образом работа маркерных генов возможна только при интеграции плазмиды в хромосому путем гомологичной рекомбинации. Синие колонии единичных кроссоверов растили ночь в жидкой среде Middlebrook 7H9, после чего высевали на триптон-соевый агар, содержащий X-Gal (50 мкг/мл) и 10%-ную сахарозу для отбора двойных кроссоверов, после чего тестировали их чувствительность к канамицину, для подтверждения полного удаления кассеты с маркерными генами из хромосомы. Для конечного отбора рекомбинантов, несущих желаемую однонуклеотидную замену, секвенировали целевую область по Сэнгеру.
Определение минимальной ингибирующей концентрации
Анализ лекарственной чувствительности штаммов микобактерий проводили методом определения минимальных ингибирующих концентраций (МИК) при серийных микроразведениях. Для определения МИК культуры штаммов M. smegmatis инкубировали ночь в жидкой среде Middlebrook 7H9, затем ночные культуры разводили свежей средой 7H9 до OD600 = 0.05. Далее бактериальные культуры по 196 мкл вносили в 96-луночный планшет, содержащий серийные двукратные разведения соединения 1b в DMSO, до конечных концентраций от 0.5 до 32 мкг/мл и максимально возможную концентрацию – 53.2 мкг/мл, и для изониазида от 25 до 1600 мкг/мл. Оценку роста клеточных культур проводили визуально после двух суток инкубации при 37°С и периодического помешивания при 250 об./мин. За МИК принимали концентрацию, при которой не наблюдалось визуального роста культуры.
Выделение РНК микобактерий и постановка ПЦР в реальном времени
Для выделения тотальной РНК, культуры M. smegmatis растили в 10 мл среды Middlebrook 7H9 до оптической плотности OD600 ≈ 1.0, после чего промывали дважды 3 мл реагента RNAprotect Bacteria Reagent (QIAGEN, США). Тотальную РНК микобактерий выделяли при помощи набора RNeasy Plus Mini Kit (QIAGEN, США) по протоколу производителя, с использованием кремниевых шариков Lysing Matrix B (MP Biomedicals, США) для гомогенизации. После выделения и очистки, РНК обрабатывали ДНКазой TURBO DNAse (Thermo Fisher Scientific, США), измеряли концентрацию на приборе Qubit (Invitrogen, США) набором RNA BR Assay Kit (Thermo Fisher Scientific, США), целостность РНК оценивали при помощи электрофореза в агарозном геле. Синтез первой цепи кДНК проводили набором iScript Select cDNA Synthesis Kit (Bio-Rad, США) с использованием 300 нг тотальной РНК.
Для постановки количественной ПЦР в реальном времени использовали набор qPCR-HS SYBR (Евроген, Россия) и 5 нг кДНК на приборе CFX96 Touch (Bio-Rad, США). Праймеры были подобраны в программе Primer-BLAST [21] и приведены в табл. 1. Оценку результатов проводили в программе CFX Manager V 3.1 (Bio-Rad, США): относительная нормализованная экспрессия была посчитана по трем биологическим повторам как ∆∆Cq, с использованием генов sigA и ftsZ в качестве референса.
РЕЗУЛЬТАТЫ
Мутации в генах MSMEG_1963 и MSMEG_5597 приводят к устойчивости к триптантринам
Участие генов MSMEG_1963, MSMEG_4427 и MSMEG_5597 в формировании устойчивости M. smegmatis к триптантринам проверяли двумя подходами: сверхэкспрессией генов дикого типа и их аллельных вариантов, а также конструированием рекомбинантных штаммов, несущих единичные мутации в указанных генах, с последующей оценкой чувствительности штаммов микобактерий к 1b. Всего нами было сконструировано девять плазмид для сверхэкспрессии исследуемых генов, а также три рекомбинантных штамма (табл. 2).
Таблица 2.
Штаммы микобактерий и плазмиды, описанные в работе
| Штамм M. smegmatis | Описание |
|---|---|
| mc2 155 | Дикий тип |
| 1963c | Рекомбинантный штамм, мутация R523L (CGC>CTC) в гене MSMEG_1963 |
| 5597c | Рекомбинантный штамм, мутация *207R (TGA>CGA) в гене MSMEG_5597 |
| 4427c | Рекомбинантный штамм, мутация L236R (CTG>CGG) в гене MSMEG_4427 |
| Δmmp5-1 | Спонтанный мутант M. smegmatis Δmmp5-1, устойчивый к 8-фтортриптантрину, мутация R523C (CGC>TGC) в гене MSMEG_1963 [16] |
| mc2-10 | Спонтанный мутант M. smegmatis mc2 155, устойчивый к 8-фтортриптантрину, мутация R893C (CGT>TGT) в гене MSMEG_1963 [16] |
| Плазмида | Описание |
| pMIND | Пустой экспрессионный вектор |
| pMIND::1963wt | pMIND с геном MSMEG_1963 дикого типа |
| pMIND::1963d1 | pMIND с мутантным MSMEG_1963 (мутация R523C, CGC>TGC) |
| pMIND::1963d6 | pMIND с мутантным MSMEG_1963 (мутация R523L, CGC>CTC) |
| pMIND::1963mc10 | pMIND с мутантным MSMEG_1963 (мутация R893C, CGT>TGT) |
| pMIND::4427wt | pMIND с геном MSMEG_4427 дикого типа |
| pMIND::4427mc10 | pMIND с мутантным MSMEG_4427 (мутация L236R, CTG>CGG) |
| pMIND::5597wt | pMIND с геном MSMEG_5597 дикого типа |
| pMIND::5597d10 | pMIND с мутантным MSMEG_5597 (мутация Stop207R, TGA>CGA) |
| pMIND::5597mc8 | pMIND с мутантным MSMEG_5597 (сдвиг рамки считывания, С49>CTG) |
| pMIND::ms3151 | pMIND с геном MSMEG_3151 (inhAM. smegmatis) |
| pMIND::Rv1484 | pMIND с геном Rv1484 (inhAM. tuberculosis) |
Сверхэкспрессия генов MSMEG_1963, MSMEG_4427 и MSMEG_5597 не влияла на чувствительность M. smegmatis к 1b. Так, МИК 1b для штаммов со сверхэкспрессией данных генов составлял от 4 до 8 мкг/мл, что лишь на ± 1 шаг отличалось от контроля (пустой вектор pMIND, МИК = 8 мкг/мл) и является погрешностью метода (табл. 3). Также не менялась чувствительность штамма M. smegmatis 4427c. При этом для M. smegmatis 1963c и 5597с значения МИК 1b оказались максимально возможными, что подтвердило роль мутаций в этих генах в формировании устойчивости к триптантринам. В то же время, сверхэкспрессия генов MSMEG_1963 и MSMEG_5597 в штаммах M. smegmatis 1963c и 5597с соответственно, восстанавливала чувствительность данных штаммов к триптантринам.
Таблица 3.
Минимальные ингибирующие концентрации (МИК) триптантринов в отношении различных штаммов M. smegmatis
| Штамм | МИК, мкг/мл |
|---|---|
| 1b | |
| M. smegmatis mc2 155 | 4 |
| M. smegmatis 1963c | >53 |
| M. smegmatis 5597c | >53 |
| M. smegmatis 4427c | 4 |
| M. smegmatis mc2 155 pMIND | 8 |
| M. smegmatis mc2 155 pMIND::1963wt | 8 |
| M. smegmatis mc2 155 pMIND::1963d1 | 8 |
| M. smegmatis mc2 155 pMIND::1963d6 | 8 |
| M. smegmatis mc2 155 pMIND::1963mc10 | 4 |
| M. smegmatis mc2 155 pMIND::4427wt | 8 |
| M. smegmatis mc2 155 pMIND::4427mc10 | 8 |
| M. smegmatis mc2 155 pMIND::5597wt | 8 |
| M. smegmatis mc2 155 pMIND::5597d10 | 8 |
| M. smegmatis mc2 155 pMIND::5597mc8 | 4 |
| M. smegmatis 5597c pMIND | >32 |
| M. smegmatis 5597c pMIND::5597wt | 8 |
| M. smegmatis 1963c pMIND | 16 |
| M. smegmatis 1963c pMIND::1963wt | 4 |
Мутации в MSMEG_1963 и MSMEG_5597 приводят к сверхэкспрессии MSMEG_1964 и MSMEG_5596
Гены MSMEG_1963 и MSMEG_5597 аннотированы как транскрипционные репрессоры (семейства EmbR и TetR соответственно). Как правило, у бактерий транскрипционные репрессоры регулируют экспрессию близлежащих генов. Нами было проанализировано генетическое окружение данных генов, в котором были обнаружены гены MSMEG_1964 и MSMEG_5596, кодирующие, соответственно, митомицин радикальную оксидазу и оксидоредуктазу, которые могут являться потенциальными инактиваторами триптантринов за счет окислительно-восстановительной модификации их молекул.
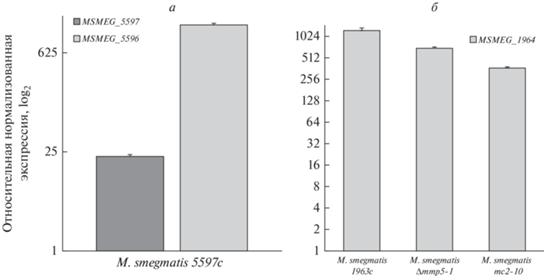
Для анализа влияния мутаций в генах MSMEG_1963 и MSMEG_5597 на изменение экспрессии MSMEG_1964 и MSMEG_5596 проведена количественная ПЦР в реальном времени с использованием кДНК-штаммов M. smegmatis 1963c и 5597c. В штамме M. smegmatis 5597c наблюдалось значительное повышение экспрессии генов MSMEG_5596 и MSMEG_5597 (рис. 2,а), а в штамме M. smegmatis 1963c – лишь гена MSMEG_1964 (рис. 2,б). Нами также была дополнительно исследована экспрессия генов MSMEG_1963 и MSMEG_1964 в двух спонтанных мутантах, устойчивых к 8-фтортриптантрину – M. smegmatis Δmmp5-1 и mc2-10 [16]. В обоих случаях также наблюдалась значительная сверхэкспрессия гена MSMEG_1964, хотя и ниже, чем в случае штамма M. smegmatis 1963c (рис. 2,б), экспрессия гена MSMEG_1963 также не претерпевала значительных изменений в сравнении с контролем. Таким образом, показано, что мутации в генах MSMEG_1963 и MSMEG_5597 приводят к сверхэкспрессии генов MSMEG_1964 и MSMEG_5596 соответственно, кодирующих ферменты, которые потенциально могут инактивировать молекулы триптантринов [22].
InhA не является биомишенью триптантринов
Еноил-(ацил-переносящий белок)-редуктаза InhA – основная мишень изониазида [15], является предсказанной in silico биомишенью триптантринов [14]. Сверхэкспрессия гена, кодирующего биомишень лекарственного препарата, должна приводить к повышению уровня устойчивости бактерии к данному препарату, поскольку равного количества молекул препарата уже не должно хватать для ингибирования увеличенного количества белковых молекул мишени. Таким образом, сверхэкспрессия гена, кодирующего биомишень какого-либо препарата, и исследование чувствительности к нему, является удобным способом проверки гипотезы мишень-специфичности препарата. Чтобы проверить гипотезу о том, является ли InhA биомишенью триптантринов, мы получили генетические конструкции на основе вектора pMIND для сверхэкспрессии генов, кодирующих данный фермент (как M. smegmatis, так и M. tuberculosis), в клетках M. smegmatis mc2 155 (табл. 2).
МИК и 1b в отношении штаммов M. smegmatis pMIND::ms3151 и M. smegmatis pMIND::rv1484 оказались равны 8 мкг/мл и не отличались от МИК 1b для M. smegmatis pMIND, хотя сверхэкспрессия генов MSMEG_3151 и Rv1484 ожидаемо приводила к устойчивости к изониазиду, использованному в качестве контроля (табл. 4). Таким образом, InhA не является биомишенью триптантринов.
ОБСУЖДЕНИЕ
Триптантрины являются перспективными молекулами для разработки на их основе новых ПТП, главная черта которых – сохраняющаяся активность в отношении лекарственно-устойчивых штаммов [7]. Основным направлением поиска производных триптантрина является улучшение биодоступности и активности на моделях in vivo [22]. Однако несмотря на многочисленные исследования, включающие in silico предсказанную биомишень [14], их механизм действия, равно как и механизмы устойчивости микобактерий к ним не подтверждены in vitro. Ранее нам удалось показать участие системы эффлюкса MmpS5-MmpL5 в модуляции устойчивости микобактерий к триптантринам, а также обнаружить мутации в трех генах M. smegmatis (MSMEG_1963, MSMEG_5597 и MSMEG_4427), ассоциированных с устойчивостью к этим соединениям [16], роль каждого из которых предстояло изучить по отдельности.
В данной работе мы показали, что лишь мутации в генах MSMEG_1963 и MSMEG_5597 приводят к устойчивости M. smegmatis к триптантринам, при этом добавление копии гена дикого типа восстанавливало чувствительный фенотип. Известно, что MSMEG_1963 и MSMEG_5597 аннотированы как транскрипционные репрессоры, которые могут регулировать экспрессию близлежащих генов. Мутации в них приводят к нарушению их функции, которая и восстанавливается при гетерологической экспрессии копии данных генов дикого типа. Наши данные подтвердили, что MSMEG_1963 репрессирует экспрессию MSMEG_1964 (кодирует митомицин радикальную оксидазу), а MSMEG_5597 – экспрессию MSMEG_5596 (кодирует оксидоредуктазу). Вероятно, данные ферменты принимают участие в окислительно-восстановительной инактивации триптантринов, приводя к устойчивости при их сверхэкспрессии. Наши данные соответствуют опубликованным ранее [23], согласно которым экспрессия MSMEG_5596 обратно пропорциональна экспрессии MSMEG_5597, однако такой корреляции между экспрессией MSMEG_1963 и MSMEG_1964 ранее не наблюдалось, что может быть обусловлено как структурой промотора, так и недостаточным изменением экспрессии гена MSMEG_1963 в проведенном ранее опыте. В то время как мутации в гене MSMEG_5597 приводили к масштабным изменениям в структуре белка (сдвиг рамки считывания, замена стоп-кода с удлинением полипептидной цепи, инсерции транспозона), в гене MSMEG_1963 наблюдались лишь мутации в двух кодонах (523 и 893), хотя и они приводили к существенной сверхэкспрессии контролируемого им гена. Интересно, что обе эти мутации находятся за пределами аннотированного ДНК-связывающего домена (2-247), а кодон 523 является частью аннотированного АТФазного домена белка. Также, по всей видимости, замены в кодоне 523 критичнее для функционирования белка MSMEG_1963, так как мутации в этой позиции приводили к более высокому уровню экспрессии MSMEG_1964, хотя и не сказывались на уровне устойчивости. Таким образом, более детальное изучение структуры белка MSMEG_1963 может представлять интерес для будущих исследований.
Ген MSMEG_4427, аннотированный как транспортер семейства MFS, не влияет на устойчивость M. smegmatis к триптантринам. Данная мутация была выявлена только у спонтанных мутантов, полученных с использованием штамма дикого типа M. smegmatis mc2 155, помимо этого мутанты также одновременно имели мутации в гене MSMEG_1963 либо в MSMEG_5597 [16]. Поэтому изначально не было ясно, вносит ли какой-то вклад MSMEG_4427 в устойчивость к триптантринам.
Важным результатом является опровержение того, что InhA – возможная мишень триптантринов: сверхэкспрессия генов inhA как M. smegmatis, так и M. tuberculosis не влияла на чувствительность M. smegmatis к триптантринам, хотя повышала устойчивость к изониазиду в случае гена inhA M. smegmatis. Возможно, InhA M. tuberculosis не компенсирует функцию своего гомолога, ингибируемого изониазидом, либо же транскрипция данного гена без адаптации кодонов не столь эффективна.
Авторы выражают благодарность D.B. Salunke (Department of Chemistry and Centre of Advanced Studies in Chemistry, Panjab University, Индия) и P. Kendrekar (Unit for Drug Discovery Research, Department of Health Sciences, Central University of Technology, ЮАР), за предоставление соединений триптантринов для проведения исследований.
Работа поддержана грантом Президента Российской Федерации для молодых российских ученых-кандидатов наук МК-797.2020.4.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
World Health Organization. Global tuberculosis report 2020. Geneva, Switzerland, 2020. 232 p.
Hinshaw H.C., Pyle M.M., Feldman W.H. Streptomycin in tuberculosis // Am. J. Med. 1947. V. 2. № 5. P. 429–435. https://doi.org/10.1016/0002-9343(47)90087-9
British Thoracic Society. A controlled trial of 6 months’ chemotherapy in pulmonary tuberculosis final report: Results during the 36 months after the end of chemotherapy and beyond // Brit. J. Dis. Chest. 1984. V. 78. P. 330–336. https://doi.org/10.1016/0007-0971(84)90165-7
Hameed H.M.A., Islam M.M., Chhotaray C. et al. Molecular targets related drug resistance mechanisms in MDR-, XDR-, and TDR-Mycobacterium tuberculosis strains // Front. Cell. Infect. Microbiol. 2018. V. 8. 114. https://doi.org/10.3389/fcimb.2018.00114
Islam M.M., Hameed H.M.A., Mugweru J. et al. Drug resistance mechanisms and novel drug targets for tuberculosis therapy // J. Genet. Genomics. 2017. V. 44. № 1. P. 21–37. https://doi.org/10.1016/j.jgg.2016.10.002
Coxon G.D., Cooper C.B., Gillespie S.H., McHugh T.D. Strategies and challenges involved in the discovery of new chemical entities during early-stage tuberculosis drug discovery // J. Infect. Dis. 2012. V. 205. Suppl. 2. P. S258–264. https://doi.org/10.1093/infdis/jis191
Mitscher L.A., Baker W. Tuberculosis: A search for novel therapy starting with natural products // Med. Res. Rev. 1998. V. 18. № 6. P. 363–374. https://doi.org/10.1002/(sici)1098-1128(199811)18:6< 363::aid-med1>3.0.co;2-i
Honda G., Tabata M. Isolation of antifungal principle tryptanthrin, from Strobilanthes cusia O. Kuntze // Planta Med. 1979. V. 36. № 05. P. 85–86. https://doi.org/10.1055/s-0028-1097245
Bhattacharjee A.K., Skanchy D.J., Jennings B. et al. Analysis of stereoelectronic properties, mechanism of action and pharmacophore of synthetic indolo[2,1-b]quinazoline-6,12-dione derivatives in relation to antileishmanial activity using quantum chemical, cyclic voltammetry and 3-D-QSAR CATALYST procedures // Bioorgan. Med. Chem. 2002. V. 10. № 6. P. 1979–1989. https://doi.org/10.1016/s0968-0896(02)00013-5
Bhattacharjee A.K., Hartell M.G., Nichols D.A. et al. Structure-activity relationship study of antimalarial indolo[2,1-b]quinazoline-6,12-diones (tryptanthrins). Three dimensional pharmacophore modeling and identification of new antimalarial candidates // Eur. J. Med. Chem. 2004. V. 39. № 1. P. 59–67. https://doi.org/10.1016/j.ejmech.2003.10.004
Bandekar P.P., Roopnarine K.A., Parekh V.J. et al. Antimicrobialactivity of tryptanthrins in Escherichia coli // J. Med. Chem. 2010. V. 53. № 9. P. 3558–3565. https://doi.org/10.1021/jm901847f
Kataoka M., Hirata K., Kunikata T. et al. Antibacterial action of tryptanthrin and kaempferol, isolated from the indigo plant (Polygonum tinctorium Lour.), against Helicobacter pylori-infected mongolian gerbils // J. Gastroenterol. 2001. V. 36. № 1. P. 5–9. https://doi.org/10.1007/s005350170147
Honda G., Tosirisuk V., Tabata M. Isolation of an antidermatophytic, tryptanthrin, from indigo plants, Polygonum tinctorium and Isatis tinctoria // Planta Med. 1980. V. 38. № 03. P. 275–276. https://doi.org/10.1055/s-2008-1074877
Tripathi A., Wadia N., Bindal D., Jana T. Docking studies on novel alkaloid tryptanthrin and its analogues against enoyl-acyl carrier protein reductase (InhA) of Mycobacterium tuberculosis // Indian J. Biochem. Biophys. 2012. V. 49. № 6. P. 435–441.
Banerjee A., Dubnau E., Quemard A. et al. inhA, a gene encoding a target for isoniazid and ethionamide in Mycobacterium tuberculosis // Science. 1994. V. 263. № 5144. P. 227–230.
Frolova S.G., Klimina K.M., Kumar R. et al. Identification of mutations conferring tryptanthrin resistance to Mycobacterium smegmatis // Antibiotics. 2021. V. 10. № 1. P. 6–10. https://doi.org/10.3390/antibiotics10010006
Шур К.В., Фролова С.Г., Акимова Н.И. и др. Тест-система для in vitro скрининга кандидатов в антимикобактериальные препараты на устойчивость, опосредованную MmpS5-MmpL5-транспортерами // Генетика. 2021. Т. 57. № 1. С. 108–111. https://doi.org/10.31857/s001667582101015x
Blokpoel M.C.J. Tetracycline-inducible gene regulation in mycobacteria // Nucl. Ac. Res. 2005. V. 8. № 2. P. e22. https://doi.org/10.1093/nar/gni023
Goude R., Parish T. Electroporation of mycobacteria // Mycobacteria Protocols. Methods in Mol. Biol. (Methods and Protocols) / Eds : Parish T., Brown A. Totowa, NJ: Humana Press, 2009. V. 465. P. 203–215. https://doi.org/10.1007/978-1-59745-207-6_13
Parish T., Stoker N.G. Use of a flexible cassette method to generate a double unmarked Mycobacterium tuberculosis tlyA plcABC mutant by gene replacement // Microbiology (Reading, England). 2000. V. 146 (Pt 8). P. 1969–1975.
Ye J., Coulouris G., Zaretskaya I. et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction // BMC Bioinformatics. 2012. V. 13. № 1. P. 134. https://doi.org/10.1186/1471-2105-13-134
Hwang J.-M., Oh T., Kaneko T. et al. Design, synthesis, and structure-activity relationship studies of tryptanthrins as antitubercular agents // J. Nat. Prod. 2012. V. 76. № 3. P. 354–367. https://doi.org/10.1021/np3007167
Li X., Mei H., Chen F. et al. Transcriptome landscape of Mycobacterium smegmatis // Front. Microbiol. 2017. V. 8. P. 2505. https://doi.org/10.3389/fmicb.2017.02505
Дополнительные материалы отсутствуют.