Генетика, 2023, T. 59, № 1, стр. 50-64
Маркеры хлоропластной ДНК в исследовании филогеографии робуроидных дубов (Quercus L. секции Quercus, Fagaceae) крымско-кавказского региона
С. А. Семерикова *
Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
* E-mail: s.a.semerikova@ipae.uran.ru
Поступила в редакцию 24.02.2022
После доработки 22.06.2022
Принята к публикации 23.06.2022
- EDN: CNHMLD
- DOI: 10.31857/S0016675823010095
Аннотация
Исследована изменчивость пяти участков хлоропластной ДНК (ASq, CDq, TFq, trnH-psbA и trnK-matK) общей длиной более 10 000 пн у трех видов робуроидных дубов крымско-кавказского региона (Q. robur, Q. petraea, Q. pubescens). Всего у 290 особей из 20 популяций обнаружено 11 гаплотипов, 10 из которых отличались от гаплотипов восточноевропейской части ареала. Полученные результаты показывают выраженную структуру географического распределения гаплотипов робуроидных дубов в Крыму и на Кавказе и принадлежность хлоропластных гаплотипов региона к нескольким дивергентным филогенетическим линиям. Проведено сравнение с данными других исследований, с оценкой степени эффективности использования разных хлоропластных регионов и маркеров. Обособленное филогенетическое положение гаплотипов крымско-кавказских популяций и значительное отличие от гаплотипов более северной части ареала позволяет сделать заключение об исторически длительном изолированном существовании робуроидных дубов на Кавказе и в Крыму. В то же время наличие общих гаплотипов указывает на тесные исторические связи крымско-кавказских популяций с Малой Азией и с Балканами. Все гаплотипы выявляются тремя фрагментами (ASq, CDq и TFq), с достаточным уровнем изменчивости и пригодными для решения задач филогеографии дубов в крымско-кавказском регионе. Маркеры trnH-psbA и trnK-matK имели низкий уровень изменчивости и не выявили дополнительных гаплотипов. Предложены оптимизированные варианты генотипирования, включающие как поэтапное секвенирование, так и предполагающие сочетание анализа микросателлитных локусов хлоропластной ДНК (cpSSR), рестриктного анализа (PCR-RFLP) и секвенирования. Для оценки изменчивости маркеров в большем таксономическом диапазоне в анализ были включены представители других секций рода Quercus. Показано, что специфичные для дуба праймеры, предложенные для фрагментов ASq, CDq и TFq, пригодны для видов других секций.
Деревья рода Quercus (Fagaceae) являются важнейшими эдификаторами умеренных и субтропических лесов Северного Полушария [1, 2]. Хотя европейские виды и составляют небольшую долю от мирового разнообразия дубов [1], их экологическое и экономическое значение огромно. Среди них важнейшее место занимают виды робуроидных дубов (Roburoids), под которыми, согласно современной классификации, понимаются евразийские представители секции Quercus [1, 2]. Робуроидные дубы возникли в результате миграции из Сев. Америки видов секции Quercus, с последующей гибридизацией в Евразии с видами секции Ponticae [2]. После разделения в позднем миоцене робуроидных дубов на европейскую и восточноазиатскую группы [3] эволюция европейской группы была связана с горными системами Средиземноморья, среди которых Кавказ и Крым являются древними центрами флористического разнообразия [4–6] и плейстоценовыми рефугиумами лесной, в том числе широколиственной растительности [6–8]. На Кавказе, несмотря на почти непрерывное распространение дубовых лесов и отсутствие существенных географических барьеров, наблюдается сложная картина взаимоотношения видов и внутривидовых форм [9, 10], нуждающаяся во всестороннем исследовании с помощью молекулярных методов.
Большой объем литературы посвящен изучению изменчивости европейских робуроидных дубов, в том числе филогеографии на основе анализа хлоропластной ДНК (хпДНК) [11–21]. К сожалению, восточная часть ареала, включая крымско-кавказский регион, изучена слабо. Исследования на территории Причерноморья, в Малой Азии и на Кавказе проводились в основном с применением метода RFLP (полиморфизм длин рестриктных фрагментов) и ограничивались небольшим числом особей [11–13] или отдельными областями [14–17]. Нерешенными остались вопросы связи Кавказа с прилегающими территориями: с Крымом, Малой Азией, в том числе с северными популяциями Восточной Европы, отделенными от Причерноморского региона степной зоной. Недавние исследования структуры изменчивости хпДНК дуба черешчатого в Восточной Европе показали наличие локальных рефугиумов дуба на востоке Русской равнины и на Урале [20, 21], в то же время в двух кавказских популяциях не были встречены гаплотипы, характерные для основного ареала Восточной Европы [20].
В единственной до настоящего времени работе по кавказскому региону, проведенной с использованием секвенирования [18], исследовалась филогеография дуба скального (Q. petraea ssp. iberica (Steven ex M. Bieb.) Krassiln.) и других видов дуба в пределах Грузии на основе двух межгенных спейсеров хпДНК (trnK-matK, trnH-psbA) [22, 23]. У робуроидных дубов в Закавказье было выявлено пять гаплотипов, H1–H5 (далее в тексте как H1g–H5g), которые подразделялись на две отчетливые географические группы, западную и восточную, в соответствии с флористическими провинциями – Эвксинской и Кавказской [4]. Однако низкая изменчивость использованных фрагментов, изученная в ограниченном районе Закавказья, не позволила детально определить филогеографическую структуру дубов в крымско-кавказском регионе и сопоставить изменчивость с результатами других работ.
Более обширное филогеографическое исследование видов дубов трех секций (Quercus, Cerris, Ilex) было проведено в Турции [19] на основе трех некодирующих участков trnT–L–F региона хпДНК [24]. Внутри секции Quercus (робуроидные дубы) было идентифицировано девять гаплотипов (далее как H1t–H9t), для которых была выявлена филогеографическая структура в Турции. Отсутствие в пределах одного включенного в исследование региона хпДНК достаточного количества информативных сайтов привело к низкому разрешению дерева внутри клады робуроидных дубов. При этом сравнение с другими работами по филогеографии робуроидных дубов не проводилось.
Несмотря на достаточно многочисленные исследования изменчивости хпДНК европейских дубов, существенной проблемой являлось использование разных участков хпДНК и разных методик: RFLP-анализ, секвенирование, cpSSR (хлоропластные микросателлитные локусы) и SNPs (single nucleotide polymorphisms, однонуклеотидные полиморфизмы). Кроме того, очевидно, что для исследования изменчивости на низком таксономическом уровне необходим подбор маркеров, пригодных для проведения сравнений между исследованиями разных авторов и с достаточной изменчивостью внутри рассматриваемого географического региона.
В нашей предыдущей работе [20] по изменчивости дуба черешчатого были использованы три в основном некодирующих участка хпДНК (psaA-trnS, psbC-trnD и trnT-trnF) [24, 25], с применением новых, специфичных для рода праймеров [20]. Результаты секвенирования удалось частично сопоставить с данными RFLP-анализа аналогичных фрагментов у робуроидных дубов в Европе. Была предложена методика генотипирования с помощью cpSSR-маркеров гаплотипов дуба из северо-восточной части ареала. Однако при этом было показано [20], что для разных географических регионов (северная часть ареала – Кавказ) аллельные комбинации cpSSR-локусов могут совпадать между некоторыми гаплотипами, при существенном различии самих гаплотипов между регионами, что требует дальнейшего сопоставления вариабельности cpSSR-маркеров с данными секвенирования в разных регионах.
Цель работы состоит в: 1) изучении изменчивости хлоропластных маркеров в популяциях робуроидных дубов крымско-кавказского региона; 2) оценке степени эффективности использования разных хлоропластных маркеров для решения задач филогеографии дубов в крымско-кавказском регионе; 3) подборе методов, пригодных для широкомасштабного генотипирования большого количества образцов, в том числе, кроме секвенирования, таких анализов, как cpSSR и PCR-RFLP.
Для этого в дополнение к нашим предыдущим исследованиям [20] восточной части ареала дуба черешчатого, которые включали две выборки образцов с Кавказа, был собран материал из 18 популяций трех (Q. robur L., Q. petraea (Matt.) Liebl. и Q. pubescens Willd.) наиболее распространенных представителей робуроидных дубов в Крыму, на Северном Кавказе и в Закавказье [5, 6]. Исследовалось пять участков хпДНК, три из которых использовались в предыдущей работе [20], еще два (trnK-matK, trnH-psbA) были взяты из работ по филогеографии и баркодингу дубов Грузии и Средиземноморья [18, 22, 23]. Проводилось сравнение с данными секвенирования участков trnK-matK, trnH-psbA из Грузии [18] и региона trnT–L–F из Турции [19], а также с результатами филогеографических исследований европейских робуроидных дубов в работах Petit et al. [12, 13], проведенных с использованием RFLP-анализа. Для оценки изменчивости маркеров в большем таксономическом диапазоне в анализ были включены представители других секций рода Quercus.
МАТЕРИАЛЫ И МЕТОДЫ
Было исследовано 20 популяционных выборок (табл. 1, рис. 1) наиболее распространенных в крымско-кавказском регионе таксонов дуба. Для дуба черешчатого (Q. robur L.) всего изучено 159 деревьев из 11 популяций. Для дуба скального (Q. petraea (Matt.) Liebl.) и дуба пушистого (Q. pubescens Willd.) – 131 дерево из девяти популяций. В Западном и Восточном Крыму на северном и южном макросклонах Главной гряды Крымских гор исследовано 105 образцов из девяти выборок. На Кавказе проанализировано 185 деревьев из 11 популяций, расположенных в разных ботанико-географических районах [5], включая Западный, Центральный и Восточный Кавказ, Западное и Центральное Закавказье. Расстояние между изученными деревьями составляло не менее 50 м. Таксономическая принадлежность устанавливалась по описаниям, приведенным в [10]. Ваучерные образцы хранятся в лаб. молекулярной экологии растений ИЭРиЖ УрО РАН. Выделение геномной ДНК проводилось с помощью метода CTAB [26] из высушенных в силикагеле листьев.
Таблица 1.
Характеристика исследованных крымско-кавказских популяций дуба черешчатого (Q. robur), дубов скального Q. petraea (pt) и пушистого Q. pubescens (pb)
| № | Популяции | Координаты | n | N | Гаплотипы (число особей) |
|
|---|---|---|---|---|---|---|
| с.ш. | в.д. | |||||
| Крым,Q. robur | ||||||
| 1 | р. Бельбек | 44°40′43′′ | 33°38′30′′ | 13 | 2 | T (1), K (12) |
| 2 | р. Бодрак | 44°45′59′′ | 34°02′00′′ | 5 | 2 | T (4), K (1) |
| 3 | г. Ак-Кая | 45°06′50′′ | 34°36′33′′ | 15 | 1 | T (15) |
| 4 | г. Агармыш | 45°00′38′′ | 35°01′48′′ | 17 | 1 | Z-I (17) |
| Крым,Q. petraea, Q. pubescens | ||||||
| 5 | Чуфут-Кале (pt) | 44°44′18′′ | 33°55′58′′ | 6 | 1 | K (6) |
| 6 | Изобильное (pt) | 44°41′58′′ | 34°20′34′′ | 9 | 1 | K (9) |
| 7 | р. Биюк-Карасу (pt) | 44°58′49′′ | 34°36′33′′ | 11 | 1 | K (11) |
| 8 | г. Эчки-Даг (pb) | 44°54′19′′ | 35°07′58′′ | 14 | 2 | Z-I (11), E-III (3) |
| 9 | Старый Крым (pb) | 45°08′28′′ | 35°08′04′′ | 15 | 2 | Z-I (14), E-I (1) |
| Кавказ, Q. robur | ||||||
| 10 | Убинская | 44°42′20′′ | 38°31′27′′ | 23 | 1 | Z-I (23) |
| 11 | Куринская | 44°24′20′′ | 39°26′03′′ | 20 | 2 | E-I (9), E-III (11) |
| 12 | Псебай | 44°02′28′′ | 40°45′38′′ | 18 | 2 | E-I (18) |
| 13 | Нальчик | 44°26′41′′ | 43°35′36′′ | 24 | 3 | E-I (18), X-II (5), A-II (1) |
| 14 | Балта | 42°55′12′′ | 44°38′21′′ | 4 | 2 | F (2), E-I (2) |
| 15 | Цхинвал | 42°14′57′′ | 43°59′47′′ | 8 | 1 | X-I (8) |
| 16 | Эрпели | 42°47′21′′ | 46°58′40′′ | 12 | 1 | F (12) |
| Кавказ, Q. petraea | ||||||
| 17 | Новомихайловский (pt) | 44°14′35′′ | 38°50′35′′ | 28 | 3 | Z-I (12), Z-II (12), E-I (4) |
| 18 | Гагра (pt) | 43°16′04′′ | 40°19′40′′ | 29 | 2 | Z-I (12), X-III (17) |
| 19 | Пятигорск (pt) | 44°03′16′′ | 43°06′35′′ | 12 | 1 | E-I (12) |
| 20 | Терменлик (pt) | 42°44′39′′ | 47°00′17′′ | 7 | 1 | F (7) |
| Среднее | 14.5 | 1.6 | ||||
| Всего | 290 | 11 | ||||
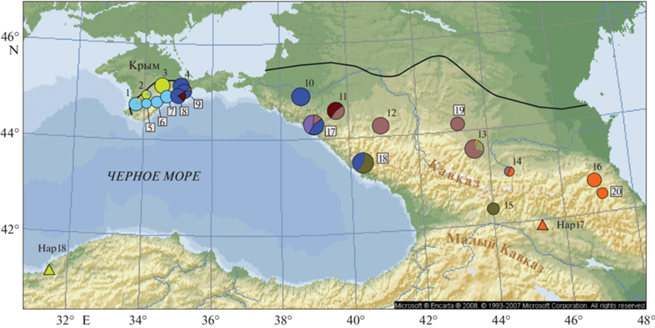
Рис. 1.
Исследованные популяции Quercus robur, Q. petraea, Q. pubescens и географическое распределение хлоропластных гаплотипов дуба в крымско-кавказском регионе. Цифрами обозначены номера популяций из табл. 1. Номера выборок Q. petraea и Q. pubescens приведены в рамках. Цветовые обозначения гаплотипов соответствуют рис. 2. Площадь диаграмм пропорциональна размеру выборки. Частоты гаплотипов в популяциях приведены в табл. 1. Указана северная граница крымско-кавказской части ареала дуба. Треугольниками маркировано происхождение пластомов, полученных из базы данных [31].
Для трех из пяти хлоропластных регионов (далее – фрагментов), psaA-trnS (ASq), psbC-trnD (CDq) и trnT-trnF (TFq), используемые праймеры, условия амплификации и секвенирования описаны ранее [20]. Фрагменты ASq, CDq и TFq были секвенированы у 38 образцов дуба полностью, у 45 образцов – частично для подтверждения гаплотипов. Дополнительно для всех выявленных нами гаплотипов у двух–трех образцов из разных географических регионов были секвенированы фрагменты trnH-psbA и trnK-matK, как описано в [23]. ПРЦ-продукты очищали с помощью набора ExoSAP-IT (Affimetrix). Реакция секвенирования проводилась с помощью набора BigDye v 3.1 (Applied Biosystems), продукты реакции анализировались на автоматическом секвенаторе Нанофор 05 (Институт аналитического приборостроения РАН, Россия) в Центре коллективного пользования “Современные технологии для экологических исследований” ИЭРиЖ УрО РАН. Редактирование и выравнивание полученных последовательностей выполняли вручную в программе BioEdit v 7.2.5 [27].
Филогенетические деревья гаплотипов построены с использованием байесовского подхода (BI) в программе MrBayes v 3.1.2 [28] и метода максимальной экономии (MP) в программном пакете PAUP*4.0b1 [29], параметры BI- и MP-анализов описаны в работе [30]. Нуклеотидные последовательности пяти фрагментов хпДНК были конкатенированы. Учитывались точковые мутации, инделы и инверсии, за исключением мутаций числа микросателлитных повторов ввиду их возможной гомоплазии. В филогенетический анализ, кроме новых гаплотипов из крымско-кавказского региона, были включены гаплотипы, ранее полученные нами [20] из восточной части основного ареала дуба черешчатого, а также нового гаплотипа Y-II из Хорватии (45°16′10′′/14°03′29′′, образец QR15, Q. robur). В анализ не вошли некоторые гаплотипы (R, N, E-II) из предыдущей работы [20], отличавшиеся от родственных гаплотипов только микросателлитными повторами. Для сравнения были взяты участки соответствующих фрагментов полных последовательностей хлоропластных геномов (пластомов) образцов дуба скального, происходящих из восточной части ареала: “Qpetraea_249.35.12.4” (RFLP-гаплотип Hap18 согласно [13]), Турция, Bolu (Ayikayasi) и “Qpetraea_184.43.9.4” (Hap17, Грузия, Telavi) из коллекции пластомов 22 деревьев для Q. robur и Q. petraea [31]. Кроме европейских робуроидных дубов, анализ был дополнен гаплотипом восточноазиатского робуроидного дуба Q. mongolica Fisch. ex Ledeb. (секция Quercus, Roburoids) (пластом из GenBank, номер MK564083). Для определения масштаба различий гаплотипов робуроидных дубов от видов других секций и групп рода Quercus, а также проверки возможности использования анализируемых хлоропластных регионов для европейских дубов других секций, секвенирование всех пяти участков было проведено для двух средиземноморских видов дубов подрода Cerris из разных секций, образцы которых взяты от деревьев, растущих в ООПТ парк Массандра, Ялта: пробковый дуб Q. suber L. (секция Cerris) и каменный дуб Q. ilex L. (секция Ilex). В анализ был включен американский вид Q. lobata Nee (пластом из GenBank, CM012305 [32]) (секция Quercus, Dumosae), поскольку евроазиатские виды секции Quercus монофилетичны по хпДНК по отношению к американским видам той же секции [33]. В качестве внешней группы был использован Trigonobalanus doichangensis (A. Camus) Forman (Fagaceae) (пластом из GenBank, KF990556). Нуклеотидное разнообразие π вычислялось c помощью DnaSP v 10.1 [34].
Одной из целей работы был подбор методики надежного и быстрого генотипирования большого числа образцов. Кроме подбора оптимальных для секвенирования участков с диагностическими для гаплотипов мутациями, была оценена возможность идентификации гаплотипов сочетанием анализа изменчивости хлоропластных микросателлитных локусов (cpSSR) и рестриктного анализа (PCR-RFLP). Ранее для дуба черешчатого [20] был апробирован метод экономичного и быстрого типирования гаплотипов у большого количества образцов, где в качестве маркеров использовались микросателлитные локусы μdt1, μdt3, μdt4, μcd4 и μcd5 [35]. Было установлено соответствие комбинации аллелей cpSSR-локусов с гаплотипами, выявленными в результате секвенирования трех хлоропластных фрагментов ASq, CDq и TFq в восточной части ареала дуба черешчатого, чем была подтверждена возможность типирования маркерами cpSSR хлоропластных гаплотипов дуба в пределах одного региона. Методика исследования изменчивости пяти cpSSR-локусов методом электрофореза в полиакриламидном геле подробно описана в предыдущей работе [20]. Для всех 290 новых образцов крымско-кавказского региона был проведен анализ изменчивости пяти cpSSR-локусов, результаты сопоставлены с данными секвенирования части образцов (3–4) для каждого cpSSR-гаплотипа в каждой популяции. На основе результатов секвенирования подбирались соответствующие рестриктазы, позволяющие подтвердить гаплотип рестриктным анализом. Для всех образцов был проведен анализ RFLP (сочетание фрагмент/рестриктаза, см. Результаты) и установлено соответствие полученных хпДНК гаплотипов с набором cpSSR-аллелей. У части образцов, для которых рестриктаза не была подобрана (образцы с родственными гаплотипами X-I, X-II, X-III и Z-I, Z-II), разделение гаплотипов осуществлялось секвенированием соответствующих фрагментов (см. Результаты).
РЕЗУЛЬТАТЫ
В результате исследования 290 индивидуумов из 20 популяций робуроидных дубов в крымско-кавказском регионе по совокупности результатов секвенирования пяти фрагментов хпДНК обнаружено 10 региональных гаплотипов (табл. 1, рис. 1), которые отличались от гаплотипов основного ареала, выявленных ранее [20]. На Кавказе найдено восемь специфичных гаплотипов (Z-I, Z-II, E-I, E-III, X-I, X-II, X-III, F) и один гаплотип основного ареала (A-II). В Крыму присутствуют пять региональных гаплотипов (K, T, Z-I, E-I, E‑III). Наиболее распространенными в Крыму оказались гаплотипы T (19%), K (37%), Z-I (40%), на Кавказе – гаплотипы Z-I (25%), E-I (34%) и F (11.3%). В большинстве выборок обнаружено один или два гаплотипа, в нескольких – по три гаплотипа. Наиболее частые гаплотипы (Z-I, E-I, F – на Кавказе, Z-I и K – в Крыму) присутствуют в популяциях как Q. robur, так и в выборках Q. petraea или Q. pubescens. Гаплотип T в Крыму отмечен только у Q. robur. Более редкие гаплотипы обычно обнаружены в одной из выборок. На Кавказе гаплотипы X-I и X-II встретились в выборках дуба черешчатого, гаплотипы X-III и Z-II – в выборках дуба скального.
У трех исследованных видов европейских робуроидных дубов в трех географических областях (восточноевропейская часть ареала, Крым, Кавказ) суммарно, c дополнением данных из предыдущей работы [20], обнаружено 19 хпДНК гаплотипов. В пяти секвенированных регионах у 19 гаплотипов присутствуют 49 мутаций, из них 38 точковых замен и 11 инделов и инверсий (табл. 2). При включении в анализ видов других секций (Q. suber, Q. ilex), а также американского вида Q. lobata и азиатского Q. mongolica число изменчивых признаков возрастает в несколько раз. Наибольшее число изменчивых признаков у робуроидных дубов (46 изменчивых сайтов) содержали наиболее длинные фрагменты (ASq, CDq, TFq). Несмотря на то что секвенирование фрагмента CDq выявляет наибольшее число гаплотипов (табл. 2), фрагмент TFq оказался наиболее оптимальным при идентификации гаплотипов секвенированием – у робуроидных дубов в нем присутствовало 14 мутаций (11 замен, 3 индела), из них в Крыму и на Кавказе – шесть мутаций (5 замен, 1 индел). Изменчивые сайты в регионе TFq приведены в табл. 3. Установлено, что секвенированием с праймера TFq(A) определяются почти все крымско-кавказские гаплотипы, кроме некоторых близкородственных в группах X и Z. В восточноевропейской части ареала дуба черешчатого последовательностями TFq также идентифицируется большинство гаплотипов. Разделение близких гаплотипов осуществляется дополнительным секвенированием участков других фрагментов (табл. 3). Кроме указанных в табл. 3, маркирующие гаплотипы диагностические мутации имеются также во фрагменте ASq – для идентификации гаплотипа Z (секвенирование с помощью праймера ASq(A2)), гаплотипа T (праймеры ASq(A2) и ASq(A)), гаплотипа F (праймер ASq(S3)), гаплотипа K (праймер ASq(S)); во фрагменте CDq присутствуют диагностические мутации гаплотипов Z и T (секвенирование c праймера CD(C)).
Таблица 2.
Показатели изменчивости пяти фрагментов хлоропластной ДНК, определенные (I) на основе последовательностей 19 гаплотипов европейских робуроидных дубов в восточной части ареала, (II) при включении в анализ последовательностей видов других групп и секций Quercus
| Фрагмент | (I) 19 гаплотипов европейских робуроидных дубов | (II) Все гаплотипы* | ||||||
|---|---|---|---|---|---|---|---|---|
| AL, пн |
L, мин–макс |
общее число мутаций (инделы, инверсии)** | π | N hap** | AL, пн |
π | общее число мутаций (инделы, инверсии)** | |
| ASq | 3727 | 3663–3717 | 16 (4) | 0.00062 ± 0.00012 | 12 | 3776 | 0.00163 ± 0.00049 | 54 (8) |
| CDq | 3586 | 3554–3577 | 16 (4) | 0.00054 ± 0.00010 | 16 | 3591 | 0.00130 ± 0.00040 | 44 (7) |
| TFq | 1818 | 1804–1812 | 14 (3) | 0.00143 ± 0.00025 | 12 | 1839 | 0.00231 ± 0.00044 | 33 (8) |
| trnK-matK | 602 | 602 | 1 (0) | 0.00082 ± 0.00011 | 2 | 603 | 0.00463 ± 0.00169 | 18 (0) |
| trnH-psbA | 434 | 432–434 | 2 (0) | 0.00160 ± 0.00046 | 4 | 468 | 0.00502 ± 0.00150 | 16 (6) |
| Всего | 10 167 | 49 (11) | 19 | 10 277 | 165 (29) | |||
Примечание. AL – длина выровненных последовательностей; L – длина последовательностей, мин–макс; N hap – число гаплотипов; π – нуклеотидное разнообразие. * – вместе с Q. mongolica, Q. lobata и видами других секций Q. suber и Q. ilex (без внешней группы). ** – не учитывались микросателлитные повторы.
Таблица 3.
Изменчивые сайты в регионе TFq хлоропластной ДНК для 19 гаплотипов робуроидных дубов: Q. robur из основного ареала [20], Q. robur, Q. petraea и Q. pubescens из крымско-кавказского региона. Серым цветом отмечены диагностические признаки для гаплотипов. Последовательность нуклеотидов для инсерций и инверсии см. в Примечании
| Гаплотипы | TFq (0–1806)1 | Фрагмент3 | Праймер для секвенирования3 |
|||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TFq(A)2 | TFq(F)2 | |||||||||||||||
| 341 | 189 | 199 | 203 | 209–232 | 291–292 | 357 | 492 | 695 | 769 | 1007 | 1204 | 1464 | 1528–1529 | |||
| Основной ареал | ||||||||||||||||
| A-I | C | T | A | G | – | – | G | A | A | A | T | T | G | – | ||
| A-II | C | T | A | G | +a | – | G | A | A | A | T | C | G | – | ||
| C | C | T | A | G | – | – | G | A | A | G | T | C | G | +c | ||
| B | C | T | A | T | – | – | G | A | G | A | C | C | G | – | CDq | CDq(D) |
| D | C | T | A | T | – | – | G | A | G | A | C | C | G | – | CDq | CDq(D) |
| S | C | T | A | T | – | – | G | A | G | A | C | C | G | – | CDq | CDq(D) |
| G | C | T | A | T | – | – | G | A | G | A | C | C | G | – | ASq | ASq(A3) |
| Y-I | C | A | G | T | – | – | A | A | A | A | T | C | A | – | ||
| Y-II | C | A | G | T | – | – | G | A | A | A | T | C | A | – | ||
| Крым, Кавказ | G | |||||||||||||||
| T | C | T | A | G | – | – | G | C | A | G | T | C | G | – | ||
| F | C | T | A | G | – | – | G | A | A | A | T | C | G | – | ||
| K | C | T | A | T | – | – | G | A | A | A | T | C | G | – | ||
| E-I | C | T | A | T | – | +b | G | A | G | A | T | C | G | – | CDq | CD(C) |
| E-III | C | T | A | T | – | – | G | A | G | A | T | C | G | – | CDq | CD(C) |
| X-I | C | T | A | T | – | – | G | A | G | A | T | C | G | – | CDq | CDq(D) |
| X-II | C | T | A | T | – | – | G | A | G | A | T | C | G | – | CDq | CDq(D) |
| X-III | C | T | A | T | – | – | G | A | G | A | T | C | G | – | CDq | CDq(D) |
| Z-I | T | T | A | T | – | +b | G | A | G | A | T | C | G | – | CDq | CDq(D) |
| Z-II | T | T | A | T | – | +b | G | A | G | A | T | C | G | – | CDq | CDq(D) |
Примечание. 1 – положение сайтов в последовательности Q. robur, гаплотип A-I (номер GenBank MT180931). 2 – праймер для секвенирования. 3 – дополнительный минимальный набор фрагментов для разделения близкородственных гаплотипов и используемые праймеры. a – инверсия “ATCTAGAATTAGAATAAATAAGTA”. b – инсерция “ATAAT”. c – инсерция “TTACA”.
Инверсия GA, обнаруженная во фрагменте CDq (позиция 2936, положение сайта в последовательности Q. robur, гаплотип A-I (номер GenBank MT180905), является высоко гомопластичной – изменчивость данного признака встречается в разных линиях. Эта инверсия изменчива между такими родственными гаплотипами как Y-I и Y-II, B/D и S/G, F и Hap17. Гаплотипы Z и Z-II, X-I и X-II отличаются друг от друга только этой инверсией. Подобные гомоплаcтичные мутации обычно не учитываются в филогенетическом анализе, однако инверсия, возможно, будет маркировать недавно возникшие “молодые” гаплотипы и их распределение у родственных видов дубов.
В cpSSR-локусах μdt1, μdt3, μdt4, μcd4 и μcd5 было выявлено от двух до четырех аллелей, при этом локус μcd5 был неизменчив (аллель 1). Комбинация четырех вариабельных локусов в крымско-кавказском регионе маркировала гаплотипы K, T, F, Z, Z-II и, не разделяя между собой, родственные гаплотипы E и E-III и близкие X-I, X-II, X-III (табл. 4). Почти для всех гаплотипов Крыма и Кавказа имеются гаплотипы из восточноевропейской части ареала, совпадающие с ними по сочетанию cpSSR-аллелей (табл. 4), но значительно отличающиеся от них по нуклеотидным последовательностям. Для идентификации гаплотипов с одинаковым сочетанием cpSSR-аллелей, кроме секвенирования, использовался рестриктный анализ: комбинацией TFq/HinfI дифференцировались гаплотипы группы E (Крым–Кавказ) от гаплотипа B (Восточная Европа), комбинацией ASq/Tru9 разделялись гаплотипы группы X от гаплотипа D, гаплотип T от A-I, гаплотип K от S, комбинацией CDq/HinfI – гаплотип F от A-II. В результате исследования 290 образцов было установлено, что все гаплотипы являлись регионально-специфичными, за исключением одной особи на Кавказе, в популяции Нальчик, имевшей гаплотип A-II, подтвержденный секвенированием и характерный для востока Русской равнины (см. [20]). Таким образом, в пределах крымско-кавказского региона большинство гаплотипов возможно диагностировать cpSSR-методом (табл. 4). Однако некоторые близкородственные гаплотипы требовали для идентификации дополнительного секвенирования или PCR–RFLP-анализа. Гаплотип E-III дифференцировался секвенированием фрагмента TFq с праймера TFq(A) или рестриктным анализом (TFq/HinfI). Разделение родственных гаплотипов группы X проводилось секвенированием участка фрагмента CDq (табл. 4).
Таблица 4.
Хлоропластные гаплотипы и описание соответствующих им аллелей хлоропластных микросателлитных локусов (cpSSR), используемых для типирования гаплотипов
| Крымско-кавказские гаплотипы |
cpSSR-локус [35] | Восточно-европейские гаплотипы [20]3 | |||
|---|---|---|---|---|---|
| μdt11 | μdt3 | μdt4 | μcd4 | ||
| T | 3 (A)11 | 1 (A)9 | 1 (A)9 | 2 (T)11 | A-I* |
| F | 3 (A)11 | 1 (A)9 | 1 (A)9 | 1 (T)10 | A-II |
| K | 2 (A)10 | 3 (A)11 | 2 (A)10 | 1 (T)10 | S |
| E-I, E-III* | 2 (A)10 | 2 (A)10 | 2 (A)10 | 1 (T)10 | B |
| X, X-II, X-III* | 2 (A)10 | 4 (A)12 | 2 (A)10 | 1 (T)10 | D |
| X-III | 2 (A)10 | 4 (A)12 | 2 (A)10 | 2 (T)11 | |
| Z-I | 2 (A)10 | 2 (A)10 | 2 (A)10 | 2 (T)11 | |
| Z-II | 2 (A)10 | 2 (A)10 | 2 (A)10 | 3 (T)12 | |
| Позиция2 | 35 327 | 34 341 | 35 148 | 31 835 | |
Примечание. 1 – приводятся номера аллелей в порядке убывания подвижности на геле и число повторов в микросателлите по данным секвенирования, см. [20]. 2 – положение в хлоропластном геноме Quercus lobata, CM012305 [32]. 3 – гаплотипы из работы [20], выявленные в популяциях Q. robur основной части ареала, совпадающие по набору cpSSR аллелей с крымско-кавказскими гаплотипами. * – разделение гаплотипов с совпадающими наборами cpSSR-аллелей см. Результаты.
Суммарная длина выровненных последовательностей фрагментов для 19 гаплотипов европейских робуроидных дубов составила 10 167 пар нуклеотидов (пн), с учетом Q. lobata, Q. mongolica и видов других секций – 10 277 пн, с внешней группой – 10 394 пн. На рис. 2 представлено байесовское дерево (BI), с указанием значений статистических поддержек клад обоих деревьев (MP). Гаплотипы робуроидных дубов образуют высокоподдержанную монофилетичную кладу, которая значительно дифференцирована от гаплотипов дубов других секций и американского вида секции Quercus.
Рис. 2.
Филогенетическое дерево хлоропластных гаплотипов робуроидных дубов (Quercus robur, Q. petraea, Q. pubescens и Q. mongolica) и гаплотипов Q. lobata, Q. ilex, Q. suber, построенное с помощью байесовского метода (50%-ный консенсус) на основе нуклеотидных последовательностей пяти фрагментов хпДНК общей длиной ~10100 пн. Над линиями приведена статистическая поддержка байесовским методом (значения апостериорной вероятности, %), под линиями – бутстреп-поддержка (MP, 1000 репликаций). Цветными кругами маркированы полученные в нашей работе крымско-кавказские гаплотипы. Треугольниками обозначены гаплотипы пластомов из базы данных [31]. Обозначения гаплотипов восточноевропейской части ареала и филогенетические линии I и II соответствуют принятым в работе [20]. Шкала отражает число замен на нуклеотидную позицию. Для гаплотипов Q. lobata, Q. mongolica и для внешней группы приводятся номера пластомов из GenBank.
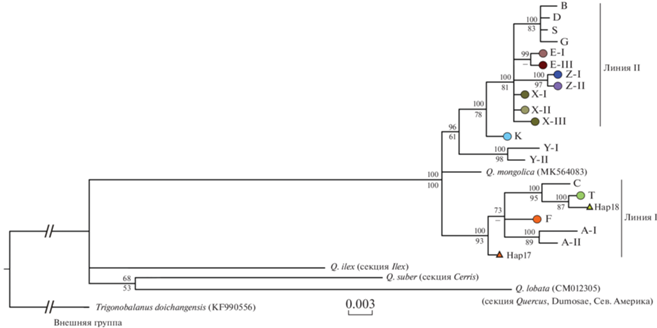
Внутри “робуроидной” клады гаплотипы восточноевропейских и крымско-кавказских дубов распределены на две дивергентные ветви, внутри которых, в свою очередь, идет подразделение еще на несколько субклад. Первая ветвь соответствует выделенной ранее [20] в европейской части ареала линии I, с дополнением крымского гаплотипа T, кавказского гаплотипа F (Дагестан) и RFLP-гаплотипа Hap17 (Восточная Грузия). Крымский гаплотип T почти совпадал по набору признаков с RFLP-гаплотипом Hap18 пластома из Западной Турции [31] и филогенетически является родственным гаплотипу C из Восточной Европы [20]. Вторая ветвь разделяется на две дивергентные линии: одну из них образуют европейские гаплотипы Y-I и Y-II. Гаплотип K является сестринским для линии II, соответствующей выделенной ранее в европейской части ареала дуба черешчатого [20]. Линия II включает несколько крымско-кавказских гаплотипов (группы X, Z, E) и субкладу восточноевропейских гаплотипов (B, S, D, G). Обособленное положение гаплотипа восточноазиатского вида Q. mongolica показывает, что время дивергенции гаплотипа Q. mongolica сравнимо со временем разделения основных линий европейско-западноазиатских хлоропластных гаплотипов робуроидных дубов.
На Северном Кавказе в направлении с запада на восток обнаружена дифференциация популяций по составу гаплотипов (рис. 1). Гаплотип Z встречен только в выборках Западного Кавказа и Западного Закавказья (Гагра), гаплотип F – только в восточных популяциях (Дагестан, Сев. Осетия). В центральной и западной части преобладают родственные гаплотипы E-I и Е-II. Гаплотипы группы X были встречены на северном макросклоне Большого Кавказа в Нальчике (X-II), на южном макросклоне в выборке Цхинвал (X-I) и в выборке Гагра (X-III). На Крымском п-ове в западной и центральной части обнаружены два гаплотипа, T и K. Причем если на северном макросклоне Главной гряды Крымских гор присутствуют оба гаплотипа (T и K), то в выборках южного макросклона, где не встречается дуб черешчатый, гаплотип T не обнаружен. В Восточном Крыму во всех популяциях преобладают кавказские гаплотипы: в основном гаплотип Z-I, в некоторой пропорции встречены E-I и E-III.
По результатам секвенирования межгенных спейсеров trnH-psbA и trnK-matK было проведено сопоставление полученных гаплотипов с данными по дубам в Закавказье (Грузии) [18]. Изменчивость trnH-psbA и trnK-matK была низкой (табл. 2). В пределах крымско-кавказского региона их сочетание определило у наших образцов всего два гаплотипа по точковым заменам и пять гаплотипов добавлением изменчивого микросателлита в trnH-psbA. Гаплотип F по набору признаков, включая микросателлитный повтор, который учитывался авторами работы [18], и по географическому распространению совпал с H1g [18]. Гаплотипы групп E и X совпали с H2g [18]. Последовательности, указанные еще для трех гаплотипов робуроидных дубов Закавказья (H3g, H4g, H5g) [18], в наших данных не обнаружены.
В значительно большей степени удалось провести сравнение по фрагменту trnT-trnF (TFq) полученных нами гаплотипов с данными из Турции [19]. С учетом неполного соответствия между последовательностями, вызванного отсутствием небольших участков фрагмента trnT-trnF в работе [19], большинство изменчивых сайтов совпадает и можно сделать вывод о родстве гаплотипов. Так, при наличии специфических мутаций в TFq обнаружено совпадение гаплотипа T (Крым) с гаплотипом H5t (Турция) [19], гаплотипа С (Восточная Европа) с гаплотипом H4t. По совокупности признаков совпали гаплотипы X (Кавказ) и H7t, гаплотипы F (Кавказ) и H1t. Близкородственными оказались гаплотипы E и H8t, K и H3t. Некоторые гаплотипы: Z (Крым–Кавказ) и большинство гаплотипов из Восточной Европы в Турции отсутствуют. Сравнение полученных нами последовательностей TFq для дубов других секций показало соответствие с видами тех же секций из турецких популяций: гаплотип Q. suber совпал с гаплотипом H14t (MT934505, встречен у видов клады Cerris, Q. cerris и др., Центр. Турция). Гаплотип Q. ilex почти совпал с гаплотипом H27t (MT934553, клада Ilex, Q. ilex, север Турции).
В GenBank помещены последовательности вновь полученных гаплотипов – для фрагментов ASq (ON101338–ON101346), CDq (ON101347–ON101355) и TFq (ON101376–ON101384); для всех гаплотипов, выявленных у изученных робуроидных дубов – последовательности фрагментов trnH-psbA (ON101385–ON101403) и trnK-matK (ON101356–ON101375).
ОБСУЖДЕНИЕ
У трех видов дубов (Q. robur, Q. petraea, Q. pubescens) в трех исследованных областях (восточноевропейская часть ареала, Крым и Кавказ) обнаружено 19 хлоропластных гаплотипов. Из них в крымско-кавказском регионе присутствуют 11 гаплотипов, 10 из которых были специфичны для крымско-кавказской части ареала. Надежная идентификация гаплотипов достигается сочетанием трех фрагментов ASq, CDq и TFq. Для массового генотипирования наиболее эффективным было последовательное секвенирование определенных изменчивых участков, начиная с фрагмента TFq с помощью праймера TFq(A), при добавлении по необходимости секвенирования дополнительных участков (табл. 3). При больших объемах данных может быть применено сочетание cpSSR-маркирования ([20], настоящее исследование), дополненное секвенированием или рестриктным анализом. Показано, что в пределах отдельных регионов (восточноевропейская часть основного ареала дуба черешчатого/крымско-кавказский регион) для генотипирования, как правило, достаточно анализа хлоропластных микросателлитных локусов (табл. 4), что было подтверждено секвенированием (см. Результаты). Кроме того, анализ cpSSR может быть использован для поиска новой изменчивости, так как быстро мутирующие микросателлитные локусы могут маркировать новые гаплотипы (примером является выявление с помощью cpSSR-маркеров гаплотипа Z-II).
Применение маркеров trnH-psbA и trnK-matK в настоящем исследовании не выявило новых гаплотипов, по сравнению с тремя более длинными фрагментами (ASq, CDq и TFq). Наши образцы, представляющие все географические регионы, эти маркеры делят на восемь гаплотипов (c учетом микросателлитного повтора в последовательности trnH-psbA, который авторами работы [18] использовался для учета гаплотипов) или всего на четыре гаплотипа, без учета микросателлита. Использование изменчивости микросателлитного повтора в trnH-psbA [18] при малом числе изменчивых нуклеотидов (табл. 2) может приводить к неверной интерпретации филогенетических отношений гаплотипов. Так, совпадение гаплотипов робуроидных дубов из Грузии c гаплотипами из Кореи, Италии, Болгарии, Украины по маркерам trnH-psbA и trnK-matK, согласно результатам работы [18], может всего лишь означать совпадение последовательностей по двум данным маркерам и требует проверки с применением более изменчивых фрагментов. Например, в настоящей работе сочетание последовательностей trnH-psbA и trnK-matK, включая микросателлитный повтор, совпадает у двух достаточно отличающихся друг от друга по другим фрагментам гаплотипов T (Крым) и Hap17 (Грузия, пластом из [31]).
Сравнением полученных нами последовательностей кавказских гаплотипов с гаплотипами из работы [18] удалось установить соответствие с нашими данными двух гаплотипов из Грузии (H1g и H2g). Еще три родственных им гаплотипа (H3g, H4g, H5g), указанных в работе [18], в исследованных на Кавказе выборках не обнаружены. Возможно, что в Закавказье могут присутствовать дополнительные, по сравнению с Северным Кавказом, гаплотипы. Однако использование маркеров trnH-psbA и trnK-matK с низкой изменчивостью не позволяет разделить некоторые гаплотипы (например, гаплотипы E и X), поэтому для филогеографических исследований робуроидных дубов кавказского региона, очевидно, выбор должен быть сделан в пользу маркеров с большим разрешением.
Лучшую эффективность имело типирование с помощью фрагмента trnT-trnF, которое показало высокую степень совпадения с полученными в работе [19] гаплотипами таксонов секции Quercus в Турции (H1t–H9t). Все они совпали или были близкородственны выявленным в нашем исследовании гаплотипам из Крыма и Кавказа. Использование нами специфических праймеров для региона TFq [20], вместо амплификации и секвенирования трех небольших коротких участков с разными протоколами амплификации [19, 24], позволило получить последовательности большей длины (весь фрагмент trnT-trnF) при меньшем числе ПЦР и меньших затратах. Однако из-за небольшого в целом количества парсимонически информативных сайтов использование только одного фрагмента TFq не позволяет с достаточным разрешением определить филогенетические отношения гаплотипов.
Дополнение нового материала позволило с большей определенностью, по сравнению с нашей предыдущей работой [20], сопоставить полученные филогенетические линии гаплотипов (рис. 1) с RFLP-линиями из работ Petit et al. [12, 13], выделенными для восточной части ареала европейских робуроидных дубов. Клада линии I (рис. 2) определяется как соответствующая зеленой линии “E” из работ [12, 13]. Cубклада восточноевропейских гаплотипов (B, S, D, G) аналогична гаплотипам синей линии “A” [12, 13]. Характер изменчивости последовательностей, в частности, наличие общей для гаплотипов Z и E-I инсерции в TFq позволяет соотнести крымско-кавказские гаплотипы групп E и Z с RFLP-гаплотипами 8 и 9 [13], которые принадлежат крымско-кавказской линии “F” [12]. Гаплотипы Y-I и Y-II (Калининградская обл. и Хорватия) близкородственны RFLP-гаплотипам 1 и 2 из красной линии “C” (Апеннинской) [12, 13]. Дивергентный гаплотип К, распространенный в Крыму, не удалось сопоставить с линиями, определенными в работах Petit et al. [12, 13].
Глубокое разделение представителей разных секций и дифференцирование американского Q. lobata от робуроидных дубов на уровне различий секций Cerris и Ilex соответствует данным последних исследований филогении рода Quercus [3, 36]. Таксономическая принадлежность включенных в анализ образцов дубов других секций (Q. suber и Q. ilex) подтверждается сравнением с последовательностями из работы [19]. Показано, что специфичные для дуба праймеры, предложенные для фрагментов ASq, CDq и TFq [20], пригодны для видов других секций.
Для крымско-кавказского региона характерны значительная дифференциация от восточноевропейской части ареала [20] и отсутствие с последней близкородственных гаплотипов. В исследованных районах наблюдается выраженная пространственная структура распределения гаплотипов. Популяции дуба на Крымском п-ове дифференцированы как от основного ареала, так и от Кавказа. В западной и центральной части там преобладают два гаплотипа, T и K, входящие в разные филогенетические клады. Эти гаплотипы пока не обнаружены на Кавказе. Гаплотип T, при сравнении с данными [19], распространен в Турции и, по-видимому, пришел в Крым из Малой Азии вдоль кавказского побережья или через Балканы. Происхождение гаплотипа K также может быть связано с Малой Азией, где распространены родственные ему гаплотипы [19]. Наличие в лесах Восточного Крыма западно-кавказского гаплотипа Z-I свидетельствует о связях популяций дуба из Восточного Крыма с Западным Кавказом.
На Кавказе, при сравнении наших данных с данными из работы [18], отмечается совпадение нескольких гаплотипов, подтверждающее параллельную дифференциацию с запада на восток на Северном Кавказе и в Закавказье, где по маркерам, использованным в [18], выявлено подразделение на две области. Для Северного Кавказа предварительно предполагается, по крайней мере, три района: западный, центральный и восточный (рис. 1). Возможно, на Кавказе имеется дифференциация и в широтном направлении, так как гаплотипы группы X больше распространены в Закавказье. Отметим также, что гаплотип Z в выборках Грузии не найден, что вероятно означает ограниченность генетического потока между Закавказьем и Западным Кавказом. Более детальное сравнение разнообразия и географического распределения гаплотипов внутри региона и в прилегающих областях поможет объяснить миграционные пути и локализацию рефугиумов. Особое внимание заслуживает увеличение числа выборок и таксонов робуроидных дубов в Закавказье.
В исследованной крымско-кавказской части ареала подавляющее большинство особей (289 из 290) несет гаплотипы, специфичные этому региону. У дуба черешчатого на Кавказе единично встречен гаплотип, характерный для восточной части основного ареала (A-II), и его обнаружение вблизи города Нальчик, возможно, связано с его антропогенным происхождением. Однако факт присутствия “северного” гаплотипа на Кавказе требует расширения исследований популяций дуба в Предкавказье, в областях, близких к северной границе кавказского ареала дуба.
Обособленное филогенетическое положение гаплотипов крымско-кавказских популяций и значительное отличие по хпДНК от восточноевропейской части ареала (рис. 2) позволяет сделать заключение об исторически длительном изолированном существовании робуроидных дубов в Крыму и на Кавказе. При сравнении с данными, полученными с помощью разных хпДНК маркеров [13–16, 19], выявляется историческая связь популяций дуба в крымско-кавказском регионе с Малой Азией и Балканским п-овом в гораздо большей степени, чем с северо-восточной частью ареала. Ранее в нескольких работах, проведенных с помощью RFLP-анализа, были отмечены гаплотипы и группы гаплотипов с дизьюнктивно-широтным распределением, встреченные на Кавказе, в Крыму и на Балканах [11–17]. Наиболее широкое распространение от Италии, через Балканы, до Кавказа отмечается для RFLP-гаплотипа 17 [11–15, 17]. Следует отметить, что этот гаплотип является “комплексным”, т.е. подразделяется на несколько субгаплотипов (17a–17e), дизьюнктивно распределенных на Балканах [12, 14, 15, 17]. В настоящей работе показано близкое, но не полное соответствие RFLP-гаплотипа Hap17 из Грузии и восточно-кавказского гаплотипа F (Дагестан) (рис. 2). Вероятно, гаплотип F совпадает также с турецким гаплотипом H1t из работы [19], который широко распространен в Сев. Турции вплоть до европейской части [19]. Особый интерес представляет обнаружение в Румынии [14, 16] RFLP-гаплотипов (9' и 13), которые указываются в работе Petit et al. на Кавказе [13] и предположительно могли мигрировать из Восточного Причерноморья [14, 16]. Точную степень родства крымско-кавказских гаплотипов с балканскими (такие, как RFLP-гаплотипы 9', 13, 17) можно будет установить только при дальнейшем секвенировании.
В крымско-кавказском регионе наиболее частые гаплотипы присутствуют в популяциях как Q. robur, так и Q. petraea или Q. pubescens (табл. 1, рис. 1). Отдельные, более редкие гаплотипы обнаружены в малом количестве выборок, иногда только у одного из видов. Общность гаплотипов у близких видов робуроидных дубов отмечается во многих работах [11–13], в том числе в Грузии и в Турции [18, 19], и чаще всего связывается с историческим генным потоком, гибридизацией и интрогрессией. Однако информация о локализации редких, более молодых гаплотипов может свидетельствовать об истории конкретной популяции или о разной интенсивности межвидового генного потока. В некоторых работах [19, 37] отмечается разница в составе гаплотипов между симпатрическими популяциями разных видов. Так, в работе [37] гаплотип, преобладающий у Q. robur, отсутствует у других видов в смешанном насаждении, что свидетельствует, по мнению авторов, о поздней миграции этого вида в район исследований. Таким образом, различия в гаплотипическом составе симпатрических популяций может указывать на разные пути колонизации, используемые разными видами. Так, редкий гаплотип E-III на Кавказе был отмечен в выборке дуба черешчатого, в Крыму – в выборке дуба скального. Интересно, что достаточно частый гаплотип T (Крым) был отмечен только в выборках дуба черешчатого. Однако совпадающий с ним по последовательности TFq гаплотип H5t был отмечен в Турции у всех таксонов робуроидных дубов [19]. Для дальнейшего установления соотношений гаплотипов разных видов дубов в Крыму необходимо увеличить число выборок Q. petraea и Q. pubescens на северном макросклоне Главной гряды в Крыму, в том числе в местах их совместного обитания с дубом черешчатым.
В настоящее время все большее значение в популяционных исследованиях приобретают высокопроизводительные (‘high-throughput’) методы генотипирования, позволяющие определять генотип сотен и тысяч индивидуумов по большому количеству маркеров ядерного и цитоплазматического происхождения. Важным этапом разработки соответствующих методик является подбор информативных снипов (SNP) и инделов. В широкомасштабном исследовании с помощью “таргетного секвенирования” 1970 особей в 197 популяциях дубов черешчатого и скального из Восточной и Центральной Европы был использован набор из 381 полиморфных локусов, в том числе 17 хлоропластных и пяти митохондриальных SNP. Было выявлено 13 хлоропластных гаплотипов [21]. При сравнении SNP, использованных при разработке набора, с изменчивостью секвенированных нами фрагментов было обнаружено, что в наших фрагментах CDq и TFq присутствует по одному SNP из набора [38], обе мутации маркируют гаплотипы Y-I и Y-II (Апеннинская линия). Во фрагментах ASq, trnH-psbA и trnK-matK нет ни одного SNP из приведенного набора [38]. При анализе различий между гаплотипами, выявляемыми с помощью SNP из работы [38], очевидно, что большая часть изменчивости приходится на различия между основными линиями хпДНК, выделенными RFLP-анализом в работе Petit et al. [12] (линии B, C, A, E) и, вероятно, маркеры SNP были подобраны с учетом способности дифференциации линий. Но внутри линий (и географических регионов) изменчивость SNP низкая. Например, гаплотипы H_06 и H_07 [21], распространенные в восточной части ареала дуба черешчатого и, несомненно, совпадающие с гаплотипами из нашего предыдущего исследования A-I и A-II [20], различались между собой по SNP только одной мутацией, в то время как по секвенированным фрагментам, использованным в настоящем исследовании, – семью мутациями, что означает необходимость значительного увеличения числа SNP для получения обоснованных филогеографических выводов. В крымско-кавказском регионе, где распространены гаплотипы “восточных” линий из работы Petit et al. [12], также нужны дополнительные маркеры. Выявленная в ходе исследования крымско-кавказских популяций дубов изменчивость пяти участков хпДНК (всего 49 мутаций) может быть использована для разработки новых наборов SNP для высокопроизводительного генотипирования.
Автор благодарит Л.И. Агафонова, И.Ю. Исакова, В.В. Корженевского, В.Л. Семерикова, Е.Г. Филиппова, Х.У. Алиева, И.А. Шанцера, П.А. Волкову за помощь в сборе образцов дуба, К.А. Паниковскую и С.М. Подергину за помощь в лабораторных анализах, рецензента работы за замечания и предложения.
Исследование выполнено за счет гранта Российского научного фонда № 22-24-00667, https://rscf.ru/project/22-24-00667/.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объектов животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объектов людей.
Список литературы
Kremer A., Hipp A.L. Oaks: an evolutionary success story // New Phytologist. 2020. V. 226. № 4. P. 987–1011. https://doi.org/10.1111/nph.16274
Denk T., Grimm G.W., Manos P.S. et al. An updated infrageneric classification of the oaks: Review of previous taxonomic schemes and synthesis of evolutionary patterns // Oaks Physiological Ecology. Exploring the Functional Diversity of Genus Quercus L. / Eds Gil-Pelegrin E., Peguero-Pina J.J., Sancho-Knapik D. Cham, Switzerland: Springer, 2017. V. 7. P. 13–38.
Hipp A.L., Manos P.S., Hahn M. et al. Genomic landscape of the global oak phylogeny // New Phytologist. 2020. V. 226. № 4. P. 1198–1212. https://doi.org/10.1111/nph.16162
Тахтаджян А.Л. Флористические области Земли. Л.: Наука, Ленингр. отд., 1978. 247 с.
Конспект флоры Кавказа: в 3 томах / Под ред. Кудряшовой Г.Л., Татанова И.В. СПб.; М.: Тов-во науч. изданий КМК, 2012. Т. 3. Ч. 2. 623 с.
Ена А.В. Природная флора Крымского полуострова. Симферополь: Н. Орiанда, 2012. 232 с.
Shatilova I., Mchedlishvili N., Rukhadze L., Kvavadze E. The history of the flora and vegetation of Georgia (South Caucasus). Tbilisi: Georgian National Museum, Institute of Paleobiology, 2011. 200 p.
Cordova C.E., Gerasimenko N.P., Lehman P.H., Kliukin A.A. Late Pleistocene and Holocene paleoenvironments of Crimea: pollen, soils, geomorphology, and geoarchaeology // Geology and Geoarchaeology of the Black Sea Region: Beyond the Flood Hypothesis / Eds Buyne-vich I.V., Yanko-Hombach V., Gilbert A.S., Martin R.E. Book series: Geological Society of America Special Paper. 2011. V. 473. P. 133–164. https://doi.org/10.1130/2011.2473(09)
Меницкий Ю.Л. Дубы Азии. Л.: Наука, 1984. 315 с.
Семериков Л.Ф. Популяционная структура древесных растений (на примере видов дуба европейской части СССР и Кавказа). М.: Наука, 1986. 140 с.
Dumolin-Lapegue S., Demesure B., Fineschi S. et al. Phylogeographic structure of white oaks throughout the European continent // Genetics. 1997. V. 146. № 4. P. 1475–1487.
Petit R.J., Csaikl U.M., Bordacs S. et al. Chloroplast DNA variation in European white oaks – phylogeography and patterns of diversity based on data from over 2600 populations // Forest Ecol. Management. 2002. V. 156. № 1–3. P. 5–26. https://doi.org/10.1016/S0378-1127(01)00645-4
Petit R.J., Brewer S., Bordacs S. et al. Identification of refugia and postglacial colonisation routes of European white oaks based on chloroplast DNA and fossil pollen evidence // Forest Ecol. Management. 2002. V. 156. P. 49–74. https://doi.org/10.1016/S0378-1127(01)00634-X
Bordacs S., Popescu F., Slade D. et al. Chloroplast DNA variation of white oaks in northern Balkans and in the Carpathian Basin // Forest Ecol. Management. 2002. V. 156. № 1–3. P. 197–209. https://doi.org/10.1016/S0378-1127(01)00643-0
Slade D., Skvorc Z., Ballian D. et al. The chloroplast DNA polymorphisms of white oaks of section Quercus in the Central Balkans // Silvae Genetica. 2008. V. 57. № 4–5. P. 227–234. https://doi.org/10.1515/sg-2008-0035
Curtu A.L., Sofletea N., Toader A.V., Enescu M.C. Leaf morphological and genetic differentiation between Quercus robur L. and its closest relative, the drought-tolerant Quercus pedunculiflora K. Koch. // Annals Forest Science. 2011. V. 68. № 7. P. 1163–1172. https://doi.org/10.1007/s13595-011-0105-z
Moldovan I.C., Sofletea N., Curtu A.L. et al. Chloroplast DNA diversity of oak species in Eastern Romania // Not. Bot. Hort. Agrobot. Cluj. 2010. V. 38. Iss. 3. P. 302–307.
Ekhvaia J., Simeone M.C., Silakadze N., Abdaladze O. Morphological diversity and phylogeography of the Georgian durmast oak (Q. petraea subsp iberica) and related Caucasian oak species in Georgia (South Caucasus) // Tree Genet. Genom. 2018. V. 14. № 2. https://doi.org/10.1007/s11295-018-1232-6
Tekpinar A.D., Aktas C., Kansu C. et al. Phylogeography and phylogeny of genus Quercus L. (Fagaceae) in Turkey implied by variations of trnT((UGU))-L-(UAA)-F ((GAA)) chloroplast DNA region // Tree Genet. Genom. 2021. V. 17. Iss. 5.https://doi.org/10.1007/s11295-021-01522-x
Семерикова С.А., Исаков И.Ю., Семериков В.Л. Изменчивость хлоропластной ДНК и филогеография дуба черешчатого Quercus robur L. в восточной части ареала // Генетика. 2021. Т. 57. № 1. С. 56–71. https://doi.org/10.31857/S0016675821010136
Degen B., Yanbaev Y., Mader M. et al. Impact of gene flow and introgression on the range wide genetic structure of Quercus robur (L.) in Europe // FORESTS. 2021. V. 12. Iss. 10.https://doi.org/10.3390/f12101425
Simeone M.C., Piredda R., Papini A. et al. Application of plastid and nuclear markers to DNA barcoding of Euro-Mediterranean oaks (Quercus, Fagaceae): problems, prospects and phylogenetic implications // Bot. J. Linn. Soc. 2013. V. 172. Iss. 4. P. 478–499. https://doi.org/10.1111/boj.12059
Piredda R., Simeone M.C., Attimonelli M. et al. Prospects of barcoding the Italian wild dendroflora: oaks reveal severe limitations to tracking species identity // Mol. Ecol. Resources. 2011. V. 11. Iss. 1. P. 72–83. https://doi.org/10.1111/j.1755-0998.2010.02900.x
Taberlet P., Gielly L., Pautou G., Bouvet J. Universal primers for amplification of 3 non-coding regions of chloroplast DNA // Plant Mol. Biology. 1991. V. 17. № 5. P. 1105–1109. https://doi.org/10.1007/BF00037152
Demesure B., Sodzi N., Petit R.J. A set of universal primers for amplification of polymorphic noncoding regions of mitochondrial and chloroplast DNA in plants // Mol. Ecol. 1995. V. 4. № 1. P. 129–131. https://doi.org/10.1111/j.1365-294X.1995.tb00201.x
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinus radiata based on RFLP, RAPD and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. № 6. P. 673–679. https://doi.org/10.1007/BF00226088
Hall T.A. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Acids Symp. Series. 1999. V. 41. P. 95–98.
Ronquist F., Huelsenbeck J.P. MrBAYES 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. № 12. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
Swofford D.L. PAUP*. Phylogenetic analysis using parsimony (* and other methods). Version 4.0 beta10. Sunderland: Sinauer Associates, Massachusetts, 2002.
Семерикова С.А., Исаков И.Ю., Семериков В.Л. Изменчивость хлоропластной ДНК отражает историю Tilia cordata s. l. в восточной части ареала // Генетика. 2020. Т. 56. № 2. С. 188–200. https://doi.org/10.1134/S0016675820020113
https://quercusportal.pierroton.inra.fr/index.php?p= GENOMIC_SEQ
Sork V.L., Fitz-Gibbon S.T., Puiu D. et al. First draft assembly and annotation of the genome of a California endemic oak Quercus lobata Nee (Fagaceae) // G3: Genes Genomes Genetics. 2016. V. 6. № 11. P. 3485–3495. https://doi.org/10.1534/g3.116.030411
Pham K.K., Hipp A.L., Manos P.S., Cronn R.C. A time and a place for everything: Phylogenetic history and geography as joint predictors of oak plastome phylogeny // Genome. 2017. V. 60. № 9. P. 720–732. https://doi.org/10.1139/gen-2016-0191
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btp187
Deguilloux M.F., Dumolin-Lapegue S., Gielly L. et al. A set of primers for the amplification of chloroplast microsatellites in Quercus // Mol. Ecol. Notes. 2003. V. 3. № 1. P. 24–27. https://doi.org/10.1046/j.1471-8286.2003.00339.x
Simeone M.C., Grimm G.W., Papini A. et al. Plastome data reveal multiple geographic origins of Quercus Group Ilex // Peer. J. 2016. V. 4. Article number 40e1897. https://doi.org/10.7717/peerj.1897
Curtu A.L., Gailing O., Finkeldey R. Evidence for hybridization and introgression within a species-rich oak (Quercus spp.) community // BMC Evolutionary Biology. 2007. V. 7. Article number 218. https://doi.org/10.1186/1471-2148-7-218
Degen B., Blanc-Jolivet C., Bakhtina S. et al. Applying targeted genotyping by sequencing with a new set of nuclear and plastid SNP and indel loci for Quercus robur and Quercus petraea // Conserv. Genet. Resour. 2021. V. 13. P. 345–347. https://doi.org/10.1007/s12686-021-01207-6
Дополнительные материалы отсутствуют.


