Генетика, 2023, T. 59, № 1, стр. 39-49
Изучение полиморфизма гена Myb114 у культур вида капуста огородная (Brassica oleracea L.) В связи с регуляцией биосинтеза антоцианов на основе сравнения с MYB-факторами овощных пасленовых культур
Д. А. Фатеев 1, *, Ф. А. Беренсен 1, А. М. Артемьева 1, О. Г. Бабак 2, К. К. Яцевич 2, Е. В. Дрозд 2, А. В. Кильчевский 2
1 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова (ВИР)
190000 Санкт-Петербург, Россия
2 Институт генетики и цитологии Национальной академии наук Беларуси
220072 Минск, Республика Беларусь
* E-mail: fateevdm1@gmail.com
Поступила в редакцию 09.02.2022
После доработки 14.03.2022
Принята к публикации 05.04.2022
- EDN: CLOYTD
- DOI: 10.31857/S0016675823010046
Аннотация
С целью изучения механизма регуляции одного из древнейших у растений процессов биосинтеза антоцианов, на основе сравнения генов R2R3-MYB транскрипционных факторов (ТФ) у пасленовых (S. lycopersicum: An1, An2; S. melongena: Myb1; C. annuum: Myb113-like1 и Myb113-like2) и капустных культур выполнен поиск ортологичных последовательностей у Brassica oleracea. Выявлена наиболее близкая по нуклеотидной структуре к ранее изученным генам у пасленовых последовательность, кодирующая MYB114 ТФ. Подобраны праймеры и выполнен сравнительный анализ последовательностей гена Myb114 у овощных культур B. oleracea (разновидности капуста белокочанная, краснокочанная, листовая, цветная, брокколи, кольраби) с широким внутривидовым разнообразием по накоплению антоцианов в листьях и различных продуктовых органах – кочанах, головках (соцветиях) и стеблеплодах. По результатам выравнивания последовательностей Myb114 выявлен ряд однонуклеотидных замен, тесно коррелирующих с высоким накоплением антоцианов в листьях B. oleracea, включающих 3 SNP в экзонных областях и делецию в первом интроне размером 271 пн. Выявленные SNP ведут к замене двух аминокислот, расположенных в области ДНК-связывающих доменов, что приводит к изменению эффективности связывания данного транскрипционного фактора с промоторами структурных генов биосинтеза и снижению уровня их экспрессии у форм без антоциана в листьях по сравнению с формами с его накоплением в вегетативных частях растений. Предложен молекулярный маркер MYB114.2, который может быть использован для идентификации кочанных и листовых капуст с высоким/низким накоплением антоцианов. По аминокислотной структуре и фенотипическому проявлению (регуляция накопления антоцианов в листьях) Myb114 B. oleracea наиболее близок к аллелям Ant2 томата, Myb2 баклажана и Myb113-like2 перца. Установлена высокая степень консерватизма SANT ДНК-связывающих доменов ТФ MYB114 B. oleracea и R2R3-MYB транскрипционных факторов овощных пасленовых культур.
Антоцианы представляют собой водорастворимые пигменты, синтезируемые в ходе фенилпропаноидного пути, участвующего во вторичном метаболизме растений; антоцианы придают растениям яркую окраску, защищают их от биотических и абиотических стрессов [1, 2]. Как составной элемент продуктов питания антоцианы необходимы для повседневного потребления населением, так как они обладают антиоксидантной активностью, бактерицидным действием, улучшают строение волокон и клеток соединительной ткани, препятствуют повреждению мембран клеток [3, 4].
Механизм биосинтеза и регуляции антоцианов хорошо изучен на модельных растениях, таких как арабидопсис, кукуруза, пшеница, пасленовые [5–7], а также на плодовых яблоне, груше, клубнике и персике [8]. В исследованиях генетических и молекулярных механизмов биосинтеза антоцианов в растениях показано, что данный процесс состоит из трех основных этапов [8].
Первый – метаболизм фенилпропаноидов –предшественников флавоноидов, включая фенилаланинаммиаклиазу (PAL), циннамат-4-гидроксилазу (C4H) и 4-кумарат. Второй, ранний этап включает образование халконсинтазы (CHS), халконизомеразы (CHI), флаванон-3-гидроксилазы (F3H), флаванон-3'-гидроксилазы (F3'H) и флавонолсинтазы (FLS), которые являются предшественниками флавонолов и антоцианов [9]. Третий, поздний этап биосинтеза приводит к синтезу и модификации антоцианов посредством процессинга дигидрофлавонол-4-редуктазы (DFR), антоцианидинсинтазы (ANS), УДФ-глюкозилтрансферазы (UGT) и ацилтрансферазы (AT) [5]. Конкуренция флавонолсинтазы и дигидрофлавонол-4-редуктазы приводит к образованию либо флавонолов, либо антоцианов [2].
Функциональные факторы транскрипции R2R3-MYB (такие как MYB11, MYB12 и MYB111) обычно участвуют в прямой активации генов раннего биосинтеза CHS, CHI, F3H, F3'H и FLS. Поздние гены биосинтеза DFR, ANS, UGTs и ATs активируются тройным комплексом MYB–bHLH–WD40 (MBW), который образован фактором R2R3-MYB, под контролем регуляторного комплекса, называемого MyB–bHLH–WD40 (MBW) [10]. Эти регуляторы или комплексы способны связываться с промоторами генов биосинтеза антоцианов и активировать их экспрессию; регуляторная сеть биосинтеза флавоноидов обычно функционирует как механизм положительной обратной связи с участием и взаимодействием как положительных, так и отрицательных регуляторов, управляя накоплением и восстановлением антоцианина или проантоцианидина [11]. У арабидопсиса в формировании комплекса MBW в основном участвуют следующие позитивные регуляторы: R2R3-MYB AtPAP1, AtPAP2, AtMYB113, AtMYB114 и AtTT2; bHLH AtTT8, AtEGL3 и AtGL3 [10]; комплекс MBW, образованный AtTT2, AtTT8 и AtTTG1, стимулирует выработку проантоцианидина в семенах арабидопсиса [12].
У пасленовых механизм биосинтеза и деградации антоцианов изучен достаточно хорошо. При этом исследований по полиморфизму аллелей, кодирующих транскрипционные факторы, очень мало.
Сравнительный анализ ряда последовательностей генов R2R3-MYB ТФ у пасленовых [13] позволил выявить наиболее близкие к Ant1 Solanum lycopersicum по нуклеотидному составу последовательности Мyb113-like1 и Мyb113-like2 у Capsicum annuum, а также Мyb1 у Solanum melongena. Были установлены SNP данных генов, приводящие к изменению структуры кодируемого белка и нарушению процесса биосинтеза антоцианов в плодах и вегетативных органах. Так, выявлена однонуклеотидная делеция в третьем экзоне фактора Myb113-like1, приводящая к сдвигу рамки считывания с появлением стоп-кодона и синтезу усеченного белка. В гене Myb1 найдены делеция в 26 пн, вызывающая выпадение экзона 2 из зрелой мРНК при ее сплайсинге и сдвиг рамки считывания с образованием стоп-кодона, как следствие–синтез нефункционального белка, а также делеция размером 6 пн, приводящая к выпадению двух аминокислот в белке (сокращению области первого ДНК-связывающего домена SANT). Наряду с мутациями, вызывающими структурные изменения белков, показана связь c отдельными SNP в гене Myb113-like2.
Исследования антоцианов у представителей рода Brassica L. в основном были сосредоточены на идентификации компонентов, картировании и оценке уровня экспрессии генов биосинтеза антоцианов. Картирование показало, что антоциановая окраска является доминирующим признаком; однако локусы различаются у видов и разновидностей Brassica. У фиолетовой цветной капусты B. oleracea L. var. botrytis окраска контролируется BoMYB2 [14]; у краснокочанной капусты B. oleracea L. var. capitata f. rubra – BoMYB2 или BoMYBL2.1 [15, 16]; у кольраби B. oleracea L. var. gongylodes – BoPAP2 и BoTT8 [17]; у пурпурной капусты цикайтай B. rapa L. ssp. chinensis var. purpurea – BrbHLH49, BrEGL3.2 и BrMYBL2.1 [18]; у китайской капусты пак-чой B. rapa L. ssp. chinensis – BrMYB73 [19]; у фиолетовой горчицы сарептской B. juncea – BjP11 [18]; у горчицы пурпурной опухолевидной B. juncea var. tumida Tsen et Lee – BjTT8 [20, 21].
Цель настоящей работы – изучить полиморфизм гена Myb114 у разновидностей капусты огородной (B. oleracea) с различным накоплением антоцианов в продуктовых органах для расширения понимания теоретической основы регуляции процесса биосинтеза антоцианов у капустных культур, а также для разработки молекулярных маркеров, связанных с нарушением/активацией синтеза антоцианов с целью использования в селекции форм с различным содержанием антоцианов; установить степень консерватизма механизма регуляции биосинтеза антоцианов путем сравнения генетических и белковых последовательностей, структуры ДНК-связывающих доменов у двух отдаленных семейств – пасленовые и капустные для получения нового представления о механизме биосинтеза антоцианов.
МАТЕРИАЛЫ И МЕТОДЫ
Объектами исследования служили образцы B. oleracea из коллекции ФГБНУ ФИЦ Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (ВИР). Выборка включала три образца белокочанной капусты: СГВ 0417 F1 (вр.к-2276, Швейцария), Сахарная гора F1 (вр.к-2287, Россия), Derbent F1 (вр.к-2316, Франция), пять образцов краснокочанной капусты: Михневская (к-175, Россия), Pecky F1 (вр.к-198, Швейцария), Pretino F1 (вр.к-200, Япония), Mohrenkopf (вр.к-201, Турция), Прометей F1 (вр.к-202, Россия), два образца листовой капусты: Di Wang (вр.к-265, Китай) с антоциановой окраской верхушечных листьев, Berza alta (вр.к-300, Испания) с зелеными листьями, два образца цветной капусты: Барбара F1 (вр.к-1021, Россия) с белой головкой, Graffiti (к-974, Нидерланды) с ярко-фиолетовой головкой, два образца брокколи: Summer Purple (вр.к-358, Швеция) со светло-фиолетовыми головками и Kuba F1 (вр.к-364, Франция) с зеленой головкой, два образца кольраби: China KRNR1 (к-286, Нидерланды) с бледно-зеленым стеблеплодом и Szentesi folias kek (к-284, Венгрия) с фиолетовым стеблеплодом. Изучаемые образцы характеризовались широким фенотипическим полиморфизмом по признаку накопления антоцианов в листьях и продуктовых органах.
Биохимический анализ осуществляли в отделе биохимии и молекулярной биологии ВИР. Антоцианы извлечены экстракцией раствором 1%-ной соляной кислоты с последующим спектрофотометрированием при длине волны 510 нм, в пересчете на цианидин-3,5-дигликозид (453 нм). Для внесения поправки на содержание зеленых пигментов одновременно определяли оптическую плотность полученных экстрактов при 657 нм. Абсорбция была измерена на спектрофотометре Ultrospec II.
Выделение ДНК проводили при помощи набора Сорб-ГМО-Б (Синтол) из молодых листьев растений согласно рекомендациям производителя (для выделения ДНК используются кремниевый сорбент и ионный детергент СТАВ). Количество ДНК в образце определяли на спектрофотометре SmartSpecplus (Bio-Rad). Нуклеотидные последовательности аллелей гена Myb114 определяли путем секвенирования, выполненного в Институте генетики и цитологии НАН Беларуси. Для этого фрагменты амплификации, полученные на геномной ДНК с подобранными нами геноспецифическими праймерами (табл. 1), разделяли в 1.0%-ном агарозном геле, затем вырезали и очищали с использованием набора DNA ExtractionKit (Thermo Scientific) согласно методике производителя. Секвенирующие реакции выполняли с применением набора BigDye® Terminator v 3.1 Cycle Sequencing Kit (Applied Biosystems) согласно методике производителя. Продукты секвенирующей реакции очищали спиртовым осаждением и растворяли в 20 мкл формамида, денатурировали нагреванием до 95°C в течение 2 мин и далее проводили капиллярный электрофорез с использованием ДНК-секвенатора ABI 3500 Genetic Analyzer (Applied Biosystems).
Таблица 1.
Праймеры, разработанные для секвенирования гена Myb114
| Праймер | Нуклеотидная последовательность |
t отжига, °С | ПЦР-продукт, пн | Примечание |
|---|---|---|---|---|
| BrMYB114.1F | TCAAGGGAGACCAAGAACC | 53.7 | 679 | Часть экзона 1 |
| BrMYB114.1R | CATCGCCTCAAGAGACTATC | 56.0 | ||
| BrMYB114.2F | TCACTGCCTATGTCCCTCAC | 56.6 | 995 | Часть экзона 1, интрон 1, часть экзона 2 |
| BrMYB114.2R | GCCATCTTAGTCTACAACTCTTCC | 57.8 | ||
| BrMYB114.3F | AGGTGTAGGAAGAGTTGTAG | 51.3 | 483 | Часть экзона 2, интрон 2, часть экзона 3 |
| BrMYB114.3R | TGTTGTTGAGTCCAAGGC | 52.1 | ||
| BrMYB114.4F | ACATTCTCTTCTACCACACC | 52.2 | 649 | Часть экзона 3 |
| BrMYB114.4R | AAACAGACGCTCTCACC | 53.1 |
Компьютерную обработку данных, полученных в результате секвенирования, проводили с использованием программы Sequencing Analysis Software v 5.2 (Applied Biosystems). На основе выявленного полиморфизма были подобраны праймеры (табл. 2) и на их основе проведено ДНК-типирование коллекции образцов. ПЦР-реакцию проводили в термоциклере Biometra TProfessional Basic (Германия). Реакционная ПЦР-смесь объемом 15 мкл содержала 60–100 нг геномной ДНК; 2.5 мM dNTP Mix (Thermo Scientific); 1.4 единицы Tornado DNA-полимеразы в инкубационном буфере “F” (Праймтех, Беларусь) и 0.25 пмоль/мл олигонуклеотидных праймеров (Евроген, Россия). Праймеры были разработаны c использованием программы DNAMAN (10.0; Lynnon BioSoft) и синтезированы компанией “Евроген” (Россия). Режим ПЦР был следующим: 95°C – 15 мин, затем следовало 35 циклов, состоящих из инкубаций: 99°C – 1 c, 52–58°C – 30 с и 72°C – 1 мин. Реакцию завершали при 72°C в течение 7 мин. Продукты ПЦР разделяли методом электрофореза в 2%-ном агарозном геле в присутствии бромистого этидия и документировали с помощью системы Bio-Rad GelDoc2000 (США). Размеры амплифицированных фрагментов определяли при использовании в качестве маркера молекулярной массы 100 bp Plus DNA ladder (Thermo Scientific).
Таблица 2.
Идентичность генов MYB-транскрипционных факторов представителей семейств Solanaceae и Myb114 Brassicaсеае по результатам BLAST-анализа
| Ген Myb114 [Brassica oleracea (wild cabbage)] – GenBank: XM_013771565 | ||
|---|---|---|
| Описание видовой принадлежности гена | Область перекрытия, % | Идентичность, % |
| Solanum melongena Myb1, mRNA, complete cds | 29 | 77.66 |
| Solanum melongena cultivar Zi Chang anthocyanin R2R3-MYB transcription factor (MYB2), mRNA, complete cds | 29 | 77.32 |
| PREDICTED: Capsicum annuum transcription factor MYB113-like (LOC107844901), mRNA; Myb113-like1 | 31 | 75.57 |
| Capsicum annuum transcription factor MYB113-like (LOC107844888), mRNA; Myb113-like2 | 30 | 75.00 |
| Solanum lycopersicum cultivar Ailsa Craig anthocyanin 1 (ANT1), mRNA, complete cds | 30 | 81.35 |
| Solanum lycopersicum cultivar Purple Smudge MYB transcription factor AN2 (An2) gene, partial cds | 19 | 75.59 |
Выравнивание нуклеотидных и аминокислотных последовательностей проводили с помощью программы MEGA4 – Molecular Evolutionary Genetics Analysis [22], VectorNTI и Unipro UGENE [23]. Для анализа ассоциаций между однонуклеотидными заменами и содержанием антоцианов использовали программное обеспечение TASSEL 5.0 [24] на основе модели GLM.
РЕЗУЛЬТАТЫ
Проведена работа по поиску ортологов у капустных культур к генам, кодирующим R2R3-MYB ТФ у пасленовых культур. С помощью программы Blast выполнены поиск и сравнительный анализ нуклеотидных последовательностей ранее изученных генов Myb-факторов семейства Solanaceae: S. lycopersicum (MYB R2R3 ТФ: An1, An2), S. melongena (MYB R2R3mel ТФ: Myb1, Myb2), С. annuum (MYB R2R3caps ТФ: Myb113-like1, Myb113-like2) с последовательностями базы данных NCBI у Brassica. В результате был найден ген Myb114 транскрипционного фактора MYB114 (XM_013771565), показывающий максимальную идентичность в области перекрытия у B. oleracea (табл. 2).
К данной последовательности были подобраны праймеры, полностью перекрывающие область экзонов 1–3 (табл. 1). С их помощью методом ПЦР на геномной ДНК у коллекционных образцов капусты с контрастной антоциановой окраской (см. раздел “Материалы и методы”) были получены ампликоны, которые затем секвенированы.
В результате сиквенса ампликонов были получены полные последовательности гена Myb114 у образцов B. oleracea, различающихся окраской продуктовых органов.
Путем выравнивания полученных нуклеотидных последовательностей образцов B. oleracea с контрастным накоплением антоцианов относительно последовательности LOC106333082 выявлены следующие однонуклеотидные замены: SNP в первом экзоне, 2 SNP во втором экзоне и 2 SNP в третьем экзоне аллеля Myb114 у сортов белокочанной капусты (рис. 1).
Рис. 1.
Полиморфизм аллелей гена Myb114 B. oleracea у форм с различным накоплением антоцианов: а – в первом экзоне, б – в первом интроне, в – во втором экзоне, г – в третьем экзоне.
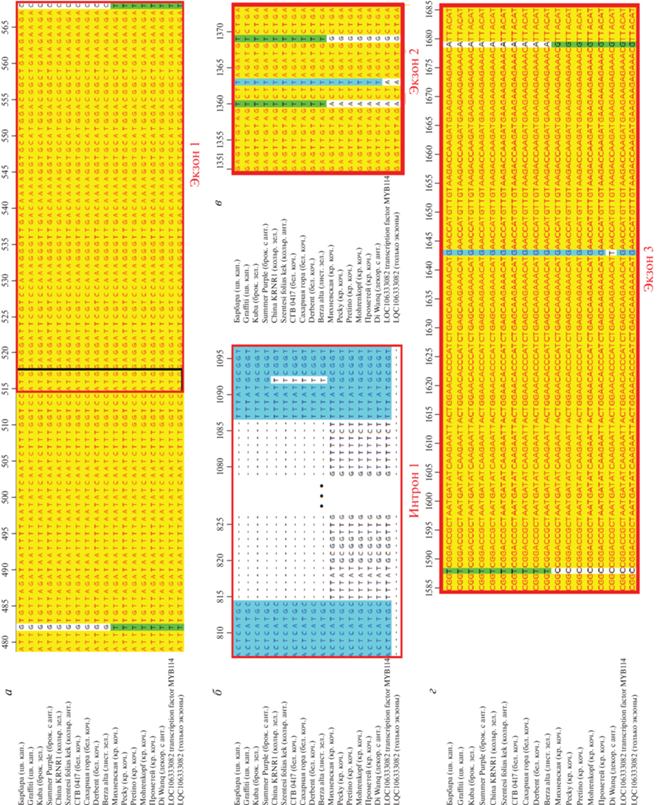
Наряду с SNP в экзонных областях выявлено множество однонуклеотидных замен в интронах и делеция размером в 271 пн в первом интроне у образцов белокочанной и зеленой листовой капусты. Образцы листовой капусты имели несколько уникальных однонуклеотидных замен, которые не встречались у кочанной капусты, хотя паттерн остальных однонуклеотидных замен был схож с таковым у кочанных форм. Так, образец листовой декоративной капусты Di Wang, отличающийся накоплением антоцианов в верхушечных листьях, имел нуклеотидную последовательность, схожую с краснокочанными образцами.
Аллели гена Myb114 у изученных образцов имели разный размер (1502 пн у белокочанной, 1773 пн у краснокочанной) и содержали три экзона. По размерам экзонов аллели были идентичны, но существенно различались по размеру первого интрона.
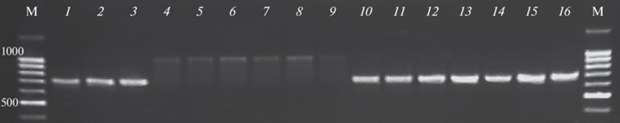
На рис. 2 представлены результаты электрофоретической разгонки ПЦР-продукта с парой праймеров BrMYB114.2.
Рис. 2.
Электрофореграмма продуктов амплификации ДНК образцов B. oleracea с праймерами BrMYB1.2. М – маркер молекулярной массы; 1 – СГВ 0417 F1, 2 – Сахарная гора F1, 3 – Derbent F1, 4 – Михневская, 5 – Pecky F1, 6 – Pretino F1, 7 – Mohrenkopf, 8 – Прометей F1, 9 – Di Wang, 10 – Berza alta, 11 – Барбара F1, 12 – Graffiti, 13 – Summer Purple, 14 – Kuba F1, 15 – China KRNR1, 16 – Szentesi folias kek.
Оценка связи проявления признака “накопление антоцианов в продуктовых органах” с выявленными однонуклеотидными заменами аллелей гена Myb114, для которого были использованы данные о содержании антоцианов у 10 образцов белокочанной, краснокочанной и листовой капусты (табл. 3), выявила высокую степень сопряженности с пятью однонуклеотидными заменами в первом, втором и третьем экзонах и делецией в первом интроне (Р-value 0.0001, R2 0.65). При этом при оценке изучаемой связи только у образцов листовой и кочанной капусты уровень значимости составил 0.001, а коэффициент детерминации 0.76. При этом SNP T → C (1-й экзон) и C → T (3-й экзон) к аминокислотной замене не ведут.
Таблица 3.
Содержание антоцианов в продуктовых органах разновидностей капусты огородной и их связь с выявленными полиморфизмами и делецией гена Myb114
| Наименование образца | SNP, экзон 1 | SNP, экзон 2 | SNP, экзон 3 | Делеция, интрон 1 | Окраска листьев | Антоцианы в прод. органах, мг/100 г cухого вещества | ||
|---|---|---|---|---|---|---|---|---|
| Кочанная, листовая капуста | ||||||||
| СГВ 0417 F1 | С | T | T | T | A | + | Зеленая | 2.16 |
| Сахарная гора F1 | С | T | T | T | A | + | Зеленая | 3.34 |
| Derbent F1 | С | T | T | T | A | + | Зеленая | 4.56 |
| Михневская | Т | A | G | С | G | – | Фиолетовая | 2096.68 |
| Pecky F1 | Т | A | G | С | G | – | Фиолетовая | 3349.65 |
| Pretino F1 | Т | A | G | С | G | – | Фиолетовая | 1181.18 |
| Mohrenkopf | Т | A | G | С | G | – | Фиолетовая | 2586.56 |
| Прометей F1 | Т | A | G | С | G | – | Фиолетовая | 1768.34 |
| Di Wang | Т | A | G | С | G | – | Фиолетовая | 945.73 |
| Berza alta | С | T | T | T | A | + | Зеленая | 13.37 |
| Цветная, брокколи, кольраби | ||||||||
| Барбара F1 | С | T | T | T | A | + | Зеленая | 7.08 |
| Graffiti | С | T | T | T | A | + | Зеленая | 126.82 |
| Summer Purple | С | T | T | T | A | + | Зеленая | 97.55 |
| Kuba F1 | С | T | T | T | A | + | Зеленая | 22.95 |
| China KRNR1 | С | T | T | T | A | + | Зеленая | 9.35 |
| Szentesi folias kek | С | T | T | T | A | + | Фиолетовая | 271.78 |
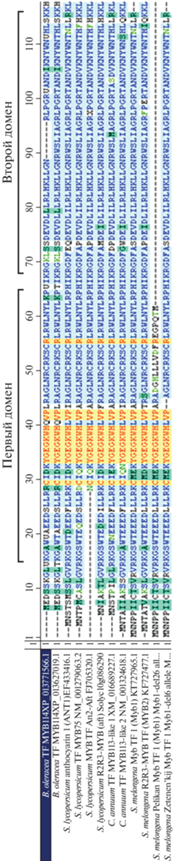
Выполнен анализ влияния генетических полиморфизмов на структуру и функции синтезируемого белка. На основании полученных нуклеотидных последовательностей с помощью программы Unipro UGENE выполнено выравнивание и сравнение аминокислотных последовательностей изученных образцов и референсных последовательностей (рис. 3). Выявлены аминокислотные замены у белокочанной капусты: R → S в позиции 50, W → C в позиции 53 и V → I в позиции 125.
На основании аминокислотных последовательностей MYB114 ТФ (CDSXP 013627020.1, CDSXP 013627019.1) выполнен поиск ДНК-связывающих доменов и их сравнение с доменами R2R3-MYB транскрипционных факторов, кодируемых ранее изученными генами пасленовых культур [13, 25]. Дополнительно показана структура доменов S. melongena с выявленными ранее нарушениями синтеза антоцианов, а также структуры транскрипционных факторов томата и баклажана группы MYB2 из базы NCBI (рис. 4).
ОБСУЖДЕНИЕ
Антоциановая окраска у разновидностей B. ole-racea обычно связана с активацией генов биосинтеза антоцианов и/или их транскрипционных активаторов. Ранее было обнаружено, что фиолетовый цвет листьев у B. oleracea связан с увеличением экспрессии активаторов транскрипции, таких как BoMYB2 и BoTT8 (BobHLH) [14–16], а также BoPAP1 и BoPAP2 [24, 26].
Настоящее исследование показало, что накопление антоцианов в кочанной и листовой капусте также может регулироваться геном транскрипционного фактора MYB114, который на 75–81% идентичен в области перекрытия генам последовательностей R2R3-MYB ТФ у пасленовых культур [13, 25], что позволяет предположить их сходные функции.
С помощью разработанных праймеров были амплифицированы и секвенированы полные последовательности гена Myb114 у разновидностей B. oleracea, различающихся окраской листьев и продуктовых органов. Был выявлен аллельный полиморфизм генов Myb114, который тесно коррелирует с фенотипическим проявлением антоциановой окраски листьев/кочанов (табл. 3). При этом последовательности гена Myb114 изученных образцов с контрастной окраской других органов (генеративных головок/соцветий цветной капусты и брокколи, а также стеблеплодов кольраби) независимо от наличия или отсутствия антоцианов были идентичны последовательностям капусты белокочанной. По всей вероятности, регуляция биосинтеза антоцианов в соцветиях и стеблеплодах у разновидностей B. oleracea контролируется другими генами, в отличие от листьев, что согласуется с другими исследованиями. Только у образца листовой капусты Di Wang (вр. к-265) были идентифицированы SNP в экзонах и инсерции, как у краснокочанной капусты, а также несколько уникальных однонуклеотидных замен, которые не встречались у других образцов. Предположительно, этот образец декоративной капусты представляет собой гибрид листовой и краснокочанной капусты.
Ассоциативный анализ показал наибольшую степень связи пяти SNP в экзонах и делеции в первом интроне у форм (Р-value 0.0001, R2 0.76) с нарушением синтеза антоциана, в результате чего нами предложена пара праймеров (BrMYB114.2) для маркируемой области гена, для разделения листовых и кочанных форм капусты по накоплению антоцианов. Маркер MYB114.2 может быть использован для идентификации кочанных и листовых капуст с высоким/низким накоплением антоцианов.
Согласно выполненному анализу последовательностей ДНК-связывающих доменов в аминокислотной последовательности ранее изученных генов пасленовых показано наличие двух доменов типа SANT в ТФ MYB114 Brassica oleracea и R2R3-MYB ТФ овощных пасленовых, что свидетельствует об их одинаковой структуре и позволяет предполагать выполнение одинаковых функций. Идентичность первого домена у B. oleracea составляет 42 аминокислоты из 50 с доменами ТФ пасленовых ANT1, MYB113-like2, 41 из 50 с доменом ТФ MYB113-like1 и 40 аминокислот из 50 с ТФ MYB1 (84–80%). В структуре второго домена последовательности ТФ MYB114 (NCBICDSXP 013627020.1; XM_013771566.1) важным отличием от доменов всех пасленовых культур является его сокращение на семь аминокислот. Идентичность последовательности B. oleracea без аминокислотной делеции (XP_013627019.1) транскрипционным факторам пасленовых составляет 32 из 41 аминокислоты с доменом ТФ MYB113-like2 и 31 из 41 аминокислоты с доменами ТФ ANT1, MYB113-like1, MYB1. Наибольшее совпадение выявлено в структуре доменов ТФ MYB114 B. oleracea и ТФ MYB113-like2. Несмотря на то что по числу аминокислот оно незначительно, но эти общие аминокислоты, характерные только для двух этих последовательностей, имеют особое расположение: аминокислота аланин (A) в третьем положении первого домена и аминокислота лизин (К) – в конце второго домена. Учитывая, что ранее нами выявлены 2 SNP в структуре 3-го экзона гена Myb113-like2, приводящих к отсутствию синтеза антоцианов в листьях перца и в данном исследовании, установлена связь между рядом SNP в структуре гена Myb114 и нарушением синтеза антоцианов, предполагаем, что по своим функциям эти два гена наиболее близки. Для сравнения с MYB114 ТФ были взяты дополнительно последовательности, кодируемые рядом известных генов R2R3-MYB ТФ базы данных NCBI (RF 7227477.1) S. melongena Myb2 и SlAn2 (FJ705320.1), SlAn2-like (Solyc10g086290), Муb75 (NM_001279063.2) S. lycopersicum, которые оказывают преимущественное влияние на накопление антоцианов в вегетативных органах [27]. Сравнение с этими последовательностями также показывает наличие аминокислоты аланин (A) в третьем положении первого домена и аминокислоты лизин (К) в конце второго домена у группы MYB2 факторов, вместо серина (S) и аргинина (R) у группы MYB1 факторов соответственно, что увеличивает предположение о сходстве функций гена Myb114 и группы генов MYB2 факторов. Аминокислотная последовательность транскрипционного фактора МYB75, который имеет схожее действие с ТФ АNT1 и AN2, имеет третью аминокислоту серин в начале первого домена, как у группы MYB1 факторов, и аминокислоту лизин в конце второго домена, как у группы MYB2 факторов. Данное сравнение структуры SANT доменов и соответствующих фенотипов растений позволяет предположить, что оба домена действуют комплексно и их структура в начале первого и в конце второго оказывает более сильное влияние на выполняемые в растении функции.
Сопоставление нуклеотидной и аминокислотной структуры с выделением областей ДНК-связывающих доменов показывает, что выделенные нами SNP в позициях 50 и 53 (рис. 3) приводят к аминокислотным заменам в конце первого домена, что свидетельствует об их наибольшей связи с регуляцией экспрессии структурных генов. Высокая связь с инсерцией в первом интроне размером в 271 пн, которая встречается у образцов кочанной и листовой капуст только у форм без антоциановой окраски, будет изучена далее. Возможно, по аналогии с последовательностями Myb1-del26 S. melongena (рис. 4), где крупные делеции в интронной области ведут к нарушению сплайсинга, выпадению второго экзона [13, 25], что, в свою очередь, ведет к нарушению структуры первого домена и полному выпадению второго домена, подобное происходит и с геном у B. oleracea. Данное предположение будет проверено путем синтеза РНК изучаемых форм и их секвенирования в дальнейших исследованиях.
Таким образом, в результате сравнения нуклеотидных последовательностей группы MYB транскрипционных факторов овощных пасленовых и капустных культур у капусты огородной B. oleracea выявлен ген Муb114, наиболее близкий по структуре генам R2R3-MYB ТФ пасленовых. Впервые выявлен аллельный полиморфизм Myb114 у форм B. oleracea с широким внутривидовым разнообразием по накоплению антоцианов в листьях. Изучены особенности влияния выявленных SNP и делеции на структуру ДНК-связывающих доменов, кодируемых транскрипционных факторов, а также их связь с накоплением антоцианов в различных продуктовых органах. Предложен молекулярный маркер MYB114.2, который может быть использован для идентификации кочанных и листовых капуст с высоким/низким накоплением антоцианов. Образцы с различным сочетанием аллельного полиморфизма отобраны как модельные для дальнейшего изучения генетической регуляции накопления антоцианов в продуктовых органах и повышения эффективности создания ценных сортов и гибридов для сельского хозяйства.
Работа выполнена при поддержке гранта № 20-516-00017 Бел_а, а также гранта Белорусского республиканского фонда фундаментальных исследований Б20Р-285 “Изучение генетических механизмов регуляции накопления антоцианов и каротиноидов у овощных пасленовых (Solanaceae) и капустных (Brassicaceae) культур”.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Zhai R., Wang Z., Yang C. et al. PbGA2ox8 induces vascular-related anthocyanin accumulation and contributes to red stripe formation on pear fruit // Hortic Res. 2019. № 6. https://doi.org/10.1038/s41438-019-0220-9
Li H., Li Y., Yu J. et al. MdMYB8 is associated with flavonol biosynthesis via the activation of the MdFLS promoter in the fruits of Malus crabapple //Hortic. Res. 2020. № 7. https://doi.org/10.1038/s41438-020-0238-z
Mazewski C., Liang K., Gonzalez de Mejia E. Comparison of the effect of chemical composition of anthocyanin-rich plant extracts on colon cancer cell proliferation and their potential mechanism of action using in vitro, in silico, and biochemical assays // Food Chem. 2018. V. 242. P. 378–388.https://doi.org/10.1016/j.foodchem.2017.09.086
Middleton E.Jr., Kandaswami C., Theoharides T.C. The effects of plant flavonoids on mammalian cells: Implications for inflammation, heart disease, and cancer // Pharmacol. Rev. 2000. V. 52. P. 673–751.
Albert N.W., Davies K.M., Lewis H.D. et al. A conserved network of transcriptional activators and repressors regulates anthocyanin pigmentation in eudicots // Plant Cell. 2014. V. 26. P. 962–980. https://doi.org/10.1105/tpc.113.122069
Хлесткина Е.К., Шоева О.Ю., Гордеева Е.И. Гены биосинтеза флавоноидов пшеницы // Вавилов. журн. генетики и селекции. 2017. Т. 18. № 4/1. С. 784–796.
Liu Y., Tikunov Y., Schouten R.E. et al. Anthocyanin biosynthesis and degradation mechanisms in Solanaceous Vegetables: a review // Frontiers in Chemistry. 2018. V. 6. P. 1–17. https://doi.org/10.3389/fchem.2018.00052
Naing A.H., Kim C.K. Roles of R2R3-MYB transcription factors in transcriptional regulation of anthocyanin biosynthesis in horticultural plants // Plant Mol. Biol. 2018. V. 98. P. 1–18. https://doi.org/10.1007/s11103-018-0771-4
Xie L., Li F., Zhang S. et al. Mining for candidate genes in an introgression line by using RNA Sequencing: The anthocyanin overaccumulation phenotype in Brassica // Front. Plant Sci. 2016. V. 7. P. 1245. https://doi.org/10.3389/fpls.2016.01245
Guo N., Cheng F., Wu J. et al. Anthocyanin biosynthetic genes in Brassica rapa // BMC Genomics. 2014. V. 15. P. 71–81. https://doi.org/10.1186/1471-2164-15-426
Petroni K., Tonelli C. Recent advances on the regulation of anthocyanin synthesis in reproductive organs // Plant Sci. 2011. V. 181. P. 219–229. https://doi.org/10.1016/j.plantsci.2011.05.009
Baudry A., Heim A.M., Dubreucq B. et al. TT2, TT8, and TTG1 synergistically specify the expression of BANYULS and proanthocyanidin biosynthesis in Arabidopsis thaliana // Plant J. 2004. V. 39(3). P. 366–380. https://doi.org/10.1111/j.1365-313X.2004.02138.x
Babak O., Nikitinskaya T., Nekrashevich N. et al. Identification of DNA markers of anthocyanin biosynthesis disorders based on the polymorphism of anthocyanin 1 tomato ortholog genes in pepper and eggplant // Crop. Breed. Genet. Genom. 2020. V. 2. https://doi.org/10.20900/cbgg20200011
Chiu L.W., Zhou X., Burke S. et al. The purple cauliflower arises from activation of a MYB transcription factor // Plant Physiol. 2010. V. 154. P. 1470–1480. https://doi.org/10.1104/pp.110.164160
Yuan Y.X., Chiu L.W., Li L. Transcriptional regulation of anthocyanin biosynthesis in red cabbage // Planta. 2009. V. 230. P. 1141–1153. https://doi.org/10.1007/s00425-009-1013-4
Jin S.W., Rahim M.A., Kim H.T. et al. Molecular analysis of anthocyanin-related genes in ornamental cabbage // Genome. 2018. V. 61. P. 111–120. https://doi.org/10.1139/gen-2017-0098
Rahim M.A., Robin A.H.K., Natarajan S. et al. Identification and characterization of anthocyanin biosynthesis-related genes in Kohlrabi // Appl. Biochem. Biotechnol. 2018. V. 184. P. 1120–1141. https://doi.org/10.1007/s12010-017-2613-2
Zhang X., Zhang K., Wu J. et al. QTL-Seq and sequence assembly rapidly mapped the gene BrMYBL2.1 for the purple trait in Brassica rapa // Sci. Rep. 2020. V. 10. https://doi.org/10.1038/s41598-020-58916-5
Wang J., Su T.B., Yu Y.J. Molecular characterization of BrMYB73: A candidate gene for the purple-leaf trait in Brassica rapa // Int. J. Agric. Biol. 2019. V. 22. P. 122–130. https://doi.org/10.17957/IJAB/15.1041
Zhao Z., Xiao L., Xing X. et al. Fine mapping the BjPl1 gene for purple leaf color in B2 of Brassica juncea L. through comparative mapping and whole-genome re-sequencing // Euphytica. 2017. V. 213. P. 80–90. https://doi.org/10.1007/s10681-017-1868-6
Xie Q., Hu Z., Zhang Y. et al. Accumulation and molecular regulation of anthocyanin in purple tumorous stem mustard (Brassica juncea var. tumida Tsen et Lee) // J. Agric. Food Chem. 2014. V. 62. P. 7813–7821. https://doi.org/10.1021/jf501790a
Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: A unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. P. 1166–1167. https://doi.org/10.1093/bioinformatics/bts091
Bradbury P.J., Zhang Z., Kroon D.E. et al. TASSEL: Software for association mapping of complex traits in diverse samples // Bioinformatics. 2007. V. 23. P. 2633–2635. https://doi.org/10.1093/bioinformatics/btm308
Zhang B., Hu Z., Zhang Y. et al. A putative functional MYB transcription factor induced by low temperature regulates anthocyanin biosynthesis in purple kale (Brassica oleracea var. acephala f. tricolor) // Plant Cell Rep. 2012. V. 31. P. 281–289. https://doi.org/10.1007/s00299-011-1162-3
Бабак О.Г., Некрашевич Н.А., Никитинская Т.В. и др. Изучение полиморфизма генов Myb-факторов на основе сравнительной геномики овощных пасленовых культур (томат, перец, баклажан) для поиска ДНК-маркеров, дифференцирующих образцы по накоплению антоцианов // Докл. Национ. акад. наук Беларуси. 2019. Т. 63. № 6. С. 721–729. https://doi.org/10.29235/1561-8323-2019-63-6-721-729
Zhang Y., Hu Z., Zhu M. et al. Anthocyanin accumulation and molecular analysis of correlated genes in purple kohlrabi (Brassica oleracea var. gongylodes L.) // J. Agr. Food Chem. 2015. V. 63. P. 4160–4169. https://doi.org/10.1021/acs.jafc.5b00473
Kiferle C., Fantini E., Bassolino L. et al. Tomato R2R3-MYB proteins SlANT1 and SlAN2: same protein activity, different roles // PLoS One. 2015. V. 10. https://doi.org/10.1371/journal.pone.0136365
Дополнительные материалы отсутствуют.