Генетика, 2023, T. 59, № 12, стр. 1372-1381
Регуляция одноцепочечных и двуцепочечных кластеров piРНК в герминальных и соматических тканях Drosophila melanogaster зависит от гена rhino
П. А. Миляева 1, 2, А. Р. Лавренов 1, 3, И. В. Кузьмин 1, А. И. Ким 1, 2, Л. Н. Нефедова 1, *
1 Московский государственный университет им. М.В. Ломоносова
119234 Москва, Россия
2 Биологический факультет, университет МГУ-ППИ в Шэньчжэне
518172 Шэньчжэнь, Китай
3 Институт проблем экологии и эволюции им. А.Н. Северцова Российской академии наук
119071 Москва, Россия
* E-mail: nefedova@mail.bio.msu.ru
Поступила в редакцию 05.05.2023
После доработки 17.06.2023
Принята к публикации 29.06.2023
- EDN: QDLMAL
- DOI: 10.31857/S0016675823120056
Аннотация
Drosophila melanogaster – широко используемый объект генетики для изучения механизмов РНК-интерференции и регуляции мобильных генетических элементов (МГЭ), в частности системы piРНК-интерференции. Считается, что у D. melanogaster система piРНК-интерференции работает только в гонадах и существуют два пути биогенеза piРНК – с помощью одноцепочечных и двуцепочечных кластеров, содержащих дефектные копии МГЭ, транскрибируемых с одной или двух цепей соответственно. Предполагается, что функционирование двуцепочечных кластеров зависит от функции гена rhino, в то время как одноцепочечные кластеры транскрибируются и затем сплайсируются независимо от rhino. В настоящей работе мы показали, что ген rhino участвует в регуляции сплайсинга одноцепочечного кластера flamenco. Более того, система piРНК-интерференции необходима для регуляции некоторых ретротранспозонов не только в гонадах, но и в других органах.
У эукариот известны три системы РНК-интерференции, необходимые для подавления транспозиционной активности мобильных генетических элементов (МГЭ), – это системы mi-, si- и piРНК-интерференции [1–3]. Источником miРНК и siРНК могут быть одиночные вставки последовательностей МГЭ и инфицирующих клетку вирусов, в то время как источником piРНК служат как одиночные копии МГЭ, так и кластеры – скопления, как правило, дефектных копий МГЭ [4, 5].
Система piРНК-интерференции, участвующая в подавлении ретротранспозонов, хорошо изучена в тканях яичников Drosophila melanogaster. В клетках зародышевого пути большая часть работающих piРНК-кластеров является двуцепочечной (42AB, 38C, 80EF) [6]. Такие кластеры представлены МГЭ, не имеющими единого направления транскрипции. Их транскрипция запускается с обеих цепей с неканонических сайтов. За счет этого получаются длинные транскрипты, отдельные участки которых являются антисмысловыми по отношению к мРНК МГЭ. Транскрипты двуцепочечных кластеров без сплайсинга отправляются в цитоплазму, где в околоядерной области “Нюаж” вовлекаются в цикл “пинг-понг” с последующим процессингом и преобразованием в piРНК [7]. Блокирование сплайсинга транскриптов таких кластеров необходимо, так как именно в интронах содержатся последовательности, необходимые для получения специфичных piРНК.
Важную роль в транскрипции и блокировании сплайсинга транскриптов двуцепочечных кластеров играет комплекс белков Rhino (Rhi), Deadlock (Del) и Cutoff (Cuff). Rhi, представитель семейства гетерохроматиновых белков Hp1, связывается с кластером, распознавая гистоновые метки H3K9me3. Rhi взаимодействует с Del, который, в свою очередь, связывается с Cuff, и вместе они образуют комплекс RDC. Данный комплекс способствует посадке РНК-полимеразы на неканонические старты транскрипции, а также препятствует кэпированию и полиаденилированию транскриптов таких кластеров. Активный сплайсинг транскриптов двуцепочечных кластеров без работы комплекса RDC, наоборот, приводит к неправильной работе системы piРНК-интерференции и повышению экспрессии некоторых МГЭ [8].
Ген rhino D. melanogaster является настолько быстро эволюционирующим геном, что Rhi самого эволюционно близкого вида, D. simulans, не способен образовывать комплекс с Del D. melanogaster [9]. Стремительная эволюция rhino объясняется высокой скоростью изменчивости МГЭ. В отличие от других белков семейства Hp1 Rhi экспрессируется в основном в гонадах как самцов, так и самок дрозофилы, и отсутствие этого белка ведет к активному сплайсингу транскриптов двуцепочечных кластеров и повышению уровня транспозиции МГЭ [6].
В соматических клетках яичников дрозофилы piРНК образуются из длинных транскриптов одноцепочечных кластеров, таких как flamenco. Длинные транскрипты процессируются путем альтернативного сплайсинга и последующего нарезания на piРНК. Считается, что flamenco транскрибируется независимо от комплекса RDC и его транскрипты перемещаются из ядра в белковый комплекс “Yb-тельце”, локализованный на внешней мембране митохондрий. После процессинга пре-piРНК модифицируются метилтрансферазой Hen1 и транспортируются в ядро в составе комплекса с белком PIWI, где происходит подавление активности ретротранспозонов на уровне транскрипции с привлечением белков для гетерохроматинизации Asterix (Arx), Hp1а и Maelstrom (Mael) [10]. Стоит отметить, что метилтрансфераза Hen1 является неотъемлемым компонентом процессинга любых si- и piРНК как в соматических клетках зародышевой линии, так и в цикле “пинг-понг” в клетках зародышевого пути. Более того, этот фермент выполняет ту же функцию в нервной системе взрослых особей нематоды Caenorhabditis elegans [11]. Таким образом, кластер flamenco участвует исключительно в создании гидовых piРНК и не принимает участия в интерференции. Вероятно, поэтому считается, что кластер в норме подвергается альтернативному сплайсингу и процессируется независимо от rhino. На нервной системе личинок дрозофилы было продемонстрировано, что снижение сплайсинга транскриптов этого кластера приводит к повышению количества пре-piРНК и снижению зрелых piРНК [12], однако эффект от повышения количества сплайсированных форм этого кластера для МГЭ ранее не рассматривался.
Считается, что пути siРНК и piРНК работают в гонадах, в то время как путь siРНК работает еще и в соматических тканях [4]. Однако исследования системы piРНК-интерференции на разных модельных объектах в последнее десятилетие позволили выявить функционирование этой системы не только в клетках зародышевого пути, но и в нервной системе. Так, нормальный процессинг транскриптов piРНК-кластеров необходим для обеспечения двигательной активности как личинок, так и взрослых мух D. melanogaster. Основными участниками контроля piРНК в нервной системе, согласно исследованию [12], являются гены aubergine (aub), cabeza (caz) и rhino, а также одноцепочечный кластер flamenco. У позвоночных и беспозвоночных piРНК принимают участие в ответе на повреждения периферической нервной системы: у нематоды piРНК участвуют в подавлении роста аксонов в ответ на повреждение, а у мыши экспрессия piРНК повышается при повреждении нервов, и одна из piРНК отвечает за миграцию шванновских клеток [13].
Ранее было показано, что МГЭ gypsy повышает экспрессию в тканях головы D. melanogaster при мутациях в генах aub и argonaute 3 (ago3) [14]. Ген rhino комплементарно взаимодействует с этими генами благодаря участию в транскрипции и сплайсинге кластеров piРНК. Таким образом, для отдельных компонентов системы piРНК было показано, что они вносят вклад в подавление экспрессии отдельных МГЭ в соматических тканях с помощью цикла “пинг-понг”.
Ранее нами была показана корреляция между снижением экспрессии rhino и повышением экспрессии сплайсированных форм flamenco у линии с фенотипом flamenco, SS7K, характеризующейся нарушением контроля транспозиции МГЭ gypsy [15]. В настоящей работе мы провели нокдаун гена rhino у D. melanogaster и измерили экспрессию наиболее изученных кластеров piРНК и МГЭ в тканях яичников, головы, корпуса (тканей, оставшихся после отделения головы и яичников) у семидневных самок, чтобы оценить роль комплекса RDC в регуляции системы piРНК-интерференции за пределами гонад, а также установить роль Rhi в регуляции синтеза piРНК не только двуцепочечных кластеров, которые работают в клетках зародышевого пути, но и одноцепочечных кластеров, которые, как считается, работают исключительно в соматических тканях гонад.
МАТЕРИАЛЫ И МЕТОДЫ
Линии Drosophila melanogaster и условия культивирования
Для нокдауна гена rhino использовали систему UAS-GAL4, основанную на скрещивании драйверной и трейсерной линий D. melanogaster, несущих соответственно конструкцию с геном, который кодирует белок GAL4 и находится под тканеспецифичным промотором, и конструкцию со шпилькой для siРНК-интерференции под промотором UAS. Для создания гибридов с нокдауном иcследуемого гена использовали самок линии rhi (Vienna Drosophila Resource Center (VDRC) ID 101620) и самцов линии tubP-Gal4 (y1w*; P{tubP-GAL4}LL7/TM3,Sb1Ser1; Bloomington Drosophila Stock Center BL5138; запускает нокдаун во всех тканях). В качестве контроля использовали гибрид самок линии w[1118] (на ее основе была получена линия rhi) и самцов tubP-Gal4. Гибриды от скрещивания драйверной и трейсерной линий культивировали при температуре 27°C. Данная температура была оптимальной для работы драйверной конструкции. Эксперимент проводили в трех повторах.
Выделение РНК, обратная транскрипция и ПЦР в реальном времени
Выделение РНК семидневных самок с нокдауном гена rhino и самок контрольных гибридов проводили из тканей яичников, головы и корпуса с помощью реактива ExtractRNA (Евроген) после вскрытия мух в буфере PBS. Перед постановкой реакции обратной транскрипции образцы РНК обрабатывали ДНКазой I (Thermo Fisher Scientific). Для обратной транскрипции использовали набор MMLV-RT Kit (Евроген). Обратную транскрипцию ставили со случайным праймером (так как экспрессия большинства кластеров в нашем опыте не превышает экспрессию МГЭ). ПЦР проводили в присутствии SYBR Green I (Евроген) в амплификаторе MiniOpticon Real-Time PCR System (Bio-Rad). В опыте анализировали относительную экспрессию ретротранспозонов gypsy, Tirant, roo, copia, blood, HeT-A, TART-A, TART-B, TART-C и кластеров 38C, 20A, 42AB, flamenco, нормированную на экспрессию генов αTub84D, Rpl40 и EloB. Для контроля нокдауна гена rhino использовали праймеры к соответствующему гену (табл. 1). Статистическую обработку результатов ПЦР проводили с помощью критерия Манна–Уитни в среде программирования R. В каждой выборке использовали от пяти до 14 образцов. Праймеры для кластера 42AB были взяты из работы Zh. Zhang и соавт. [6], праймеры для кластеров 38С и 20А были взяты из работы J. Hur и соавт. [16], праймеры для кластера flamenco были подобраны нами к сплайсированной форме транскрипта согласно сиквенсу из базы данных NCBI (Gene ID: 26067356) [17]. Все праймеры подобраны к специфичным частям кластеров и не отжигаются на копиях мобильных элементов, находящихся в других точках генома.
Таблица 1.
Праймеры для обратной транскрипции (ОТ) и ПЦР
| Название | Прямой праймер | Обратный праймер |
|---|---|---|
| Гены | ||
| αTub84D | 5'-GTGCATGTTGTCCAACACCAC-3' | 5'-AGAACTCTCCCTCCTCCATA-3' |
| EloB | 5'-GCACAAACATACACACTCACG-3' | 5'-TTTCCTACTTCGCTTGCACC-3' |
| RpL40 | 5'-CTGCGTGGTGGTATCATTG-3' | 5'-CAGGTTGTTGGTGTGTCC-3' |
| rhino | 5'-CTTCGAGTCGGAGGTCTTCA-3' | 5'-CTGCACCGACTTGGAATGTT-3' |
| Кластеры | ||
| flamenco-spliced | 5'-CATCAGCTCAGCAGCAGTGTA-3' | 5'-AGTCCAATGCTCGCTTGAAA-3' |
| flamenco-unspliced | 5'-GACTTAACACTTACCGCTTGAAA-3' | |
| 42AB-spliced | 5'-GCAGTTGCCGTCTCTCCTT-3' | 5'-TGGGTCAAAGTGCAGCAGTT-3' |
| 42AB-unspliced | 5'-TACGGGAATATAATCGCAGCAGTT-3' | |
| 38C 1 | 5'-AATGGCTAGTTCGCTACCAGACAG-3' | 5'-CGGGTCTTCTCTCAAACGCAATC-3' |
| 38C 2 | 5'-CACAAAATGGCCGCTGGAAA-3' | 5'-AGCCAAACCCTGTGTTGTGA-3' |
| 20A | 5'-GCCTACGCAGAGGCCTAAGT-3' | 5'-CAGATGTGGTCCAGTTGTGC-3' |
| Ретротранспозоны | ||
| gypsy | 5'-CTGCTGAAAGACGGCATTATC-3' | 5'-AGAACTTTGCCTTGCCCAGAT-3' |
| Tirant | 5'-AACGCTATTCATTCTGCAAC-3' | 5'-AGGGTTCTCCTAACTACGTC-3' |
| copia | 5'-CTTCAGTGATGGACAACTG-3' | 5'-CAGTGTAATCTCATGGTCAT-3' |
| roo | 5'-ACAGATTCCCACCTTCTTTG-3' | 5'-AGCCGAGGAGTAAATGAGTA-3' |
| blood | 5'-ATGCGACAACGATACAGG-3' | 5'-TTTGTCAGCATTATCGGCG-3' |
| HeT-A | 5'-TCAGGATACAGGCAAAGGAC-3' | 5'-CGACTATCCACGAGCATGG-3' |
| TART-A | 5'-GCAGCAGTTTCCATTTGAC-3' | 5'-AAGAACACACCATCGCTTG-3' |
| TART-B | 5'-ATAGGGTATGGCACGAAGG-3' | 5'-GGGCAGACCAAGTTTATCC-3' |
| TART-C | 5'-ATAGGGTATGGCACGAAGG-3' | 5'-AAGTAGTGGTCCGAGATTGC-3' |
| Шпилечные праймеры для ОТ | ||
| U6 | 5'-CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAAAAATGTGG-3' | |
| 11620 | 5'-CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGATGGCA-3' | |
| Праймеры для микроРНК | ||
| Универсальный праймер для шпильки | 5'-ACTGGTGTCGTGGAGTC-3' | |
| U6 | 5'-GCTGGGATACAGAGAAGATTAG-3' | |
| 11620 | 5'-GCTGGGTCAACTAGTATTTCTG-3' | |
Для оценки экспрессии piРНК картировали piРНК из базы данных (https://www.pirnadb.org/) на аннотированную последовательность кластера flamenco и выбрали piРНК 11620, так как она оказалась картирована в интрон кластера. В качестве референсной РНК использовали miРНК U6. Для выделения микроРНК использовали набор для выделения суммарной РНК и микроРНК из реагента “Лира” (Biolabmix). Выборки содержали до шести образцов. Обратную транскрипцию проводили с помощью специфичного шпилечного праймера. Для количественной ПЦР использовали праймеры к шпильке и к целевой РНК (табл. 1). ПЦР проводили в присутствии SYBR Green I (Евроген) в амплификаторе MiniOpticon Real-Time PCR System (Bio-Rad). В качестве референсной РНК использовали miРНК U6.
РЕЗУЛЬТАТЫ
Зависимость сплайсинга кластеров piРНК от уровня экспрессии гена rhino
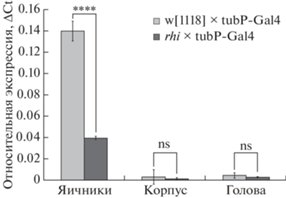
Для подтверждения роли гена rhino в контроле активности ретротранспозонов разных семейств мы получили гибриды с его нокдауном от скрещивания линий D. melanogaster rhi и tubP-Gal4 и проанализировали экспрессию этого гена в тканях яичников, головы и корпуса в сравнении с теми же тканями у контрольных гибридов (w[1118] × tubP-Gal4) (рис. 1). Как и ожидалось, в контрольных гибридах экспрессия гена rhino была наиболее высокой в тканях яичников, где система piРНК-интерференции работает наиболее активно. Примечательно, что мы также обнаружили экспрессию гена rhino в небольшом количестве в тканях корпуса и головы. У гибридов с нокдауном гена rhino мы увидели подавление экспрессии этого гена во всех тканях, однако статистически значимым оно было только в яичниках. Мы предполагаем, что это могло быть вызвано низкой экспрессией rhino в тканях корпуса и головы, а также различиями в контроле. Так как в контроле видим стабильно низкую экспрессию rhino считаем, что в этих тканях нокдаун этого гена был осуществлен успешно.
Рис. 1.
Относительная экспрессия гена rhino в тканях яичников, корпуса и головы у семидневных самок гибридов с нокдауном гена rhino и контрольных гибридов (**** – p < 0.0001 по критерию Манна–Уитни; ns, not significant – статистически незначимое изменение).
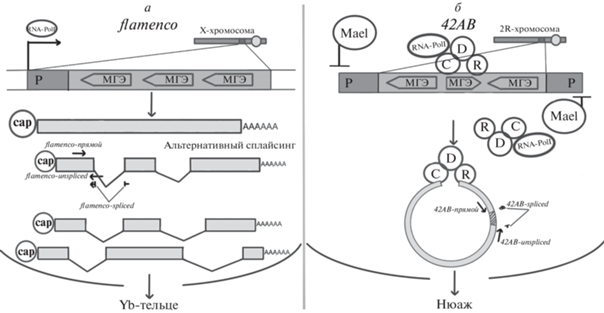
Далее мы провели сравнительный анализ экспрессии сплайсированных и несплайсированных форм предшественников piРНК, транскрибирующихся с одно- и двуцепочечных кластеров и участвующих в сайленсинге МГЭ. Для анализа мы использовали наиболее изученные кластеры piРНК (flamenco (одноцепочечный) и 42AB (двуцепочечный)) [18, 19] (рис. 2,а, б).
Рис. 2.
Регуляции одноцепочечных и двуцепочечных кластеров flamenco и 42AB и схемы локализации праймеров для выявления сплайсированных и несплайсированных форм транскриптов. a – особенности транскрипции одноцепочечных кластеров: транскрипция идет с канонического промотора в одном направлении, транскрипты сплайсируются (праймеры для отличия сплайсированных и несплайсированных форм: flamenco-прямой (общий праймер), flamenco-spliced (для сплайсированной формы), flamenco-unspliced (для несплайсированной формы)); б – особенности транскрипции двуцепочечных кластеров: транскрипция идет с неканонических промоторов в обоих направлениях за счет комплекса RDC и белка Mael, транскрипты не сплайсируются, так как RDC блокирует кэпирование и полиаденилирование транскриптов (праймеры для отличия сплайсированных и несплайсированных форм: 42AB-прямой (общий праймер), 42AB-spliced (для сплайсированной формы), 42AB-unspliced (для несплайсированной формы)). Р – промотор.
Для плохо изученных кластеров 20A (одноцепочечный) и 38С (двуцепочечный) мы провели анализ только несплайсированных форм, поскольку альтернативный сплайсинг для них пока не показан. Анализ для кластера 38С проводили в двух точках, чтобы определить, равнозначно ли отразится на экспрессии разных частей кластера снижение экспрессии rhino.
Измерив относительную экспрессию несплайсированных форм кластеров 20A, 38С, 42AB и flamenco между тканями контрольных гибридов и гибридов с нокдауном rhino, мы в большинстве случаев не обнаружили значимого изменения экспрессии этих кластеров при снижении экспрессии rhino (рис. 3,а). Исключение составило изменение экспрессии кластеров 42AB, 38С и 20А в тканях корпуса. Возможно, это связано с особенностями регуляции кластеров в этих тканях. Примечательно, что в яичниках, основном месте работы кластеров piРНК, при нокдауне rhino повышается экспрессия только сплайсированных форм кластеров flamenco и 42AB. Это наводит на мысль, что rhino может регулировать и сплайсинг flamenco.
Рис. 3.
Относительный уровень транскрипции различных кластеров piРНК в тканях яичников, головы и корпуса. a – относительный уровень транскрипции сплайсированных и несплайсированных форм одноцепочечных и двуцепочечных кластеров; б – соотношение уровней транскрипции сплайсированых и несплайсированных форм транскриптов кластеров flamenco (одноцепочечный) и 42AB (двуцепочечный); в – относительный уровень транскрипции piРНК 11620 (кластер flamenco) в яичниках. Для рис. 3, 4: Я – яичники, К – корпус, Г – голова (* – p < 0.5, ** – p < 0.01, *** – p < 0.001, **** – p < 0.0001 по критерию Манна–Уитни; ns, not significant – статистически незначимое изменение).
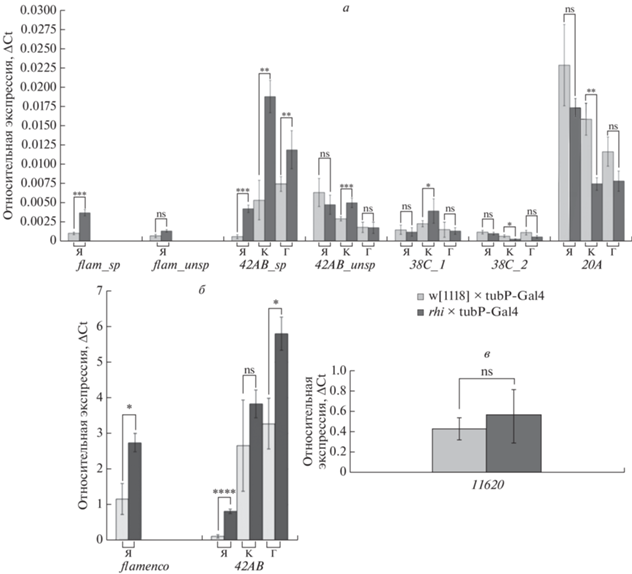
При сравнении относительной экспрессии сплайсированных и несплайсированных форм кластера 42AB, которые являются мишенью Rhi, мы увидели значительное повышение количества сплайсированных форм во всех типах тканей гибридов с нокдауном rhino относительно контроля (рис. 3,б). Более того, значительное повышение экспрессии сплайсированных форм по отношению к экспрессии несплайсированных мы также обнаружили для кластера flamenco, который, в отличие от остальных кластеров, у взрослых самок экспрессируется только в яичниках. Однако при подсчете отношения количества сплайсированных форм к несплайссированным мы обнаружили, что при нокдауне rhino это соотношение повышается не только для кластера 42AB в яичниках и голове, но и для кластера flamenco в яичниках, хотя уровень значимости в случае последнего ниже.
Мы также оценили экспрессию piРНК 11620, которая картирована в интроне кластера flamenco, однако не нашли различий между контролем и мутантом с нокдауном rhino (рис. 3,в).
Таким образом, роль гена rhino не ограничивается только генеративными и соматическими тканями яичников, но и распространяется на другие соматические ткани, где rhino так же, как и в тканях яичников, регулирует сплайсинг кластеров piРНК. Помимо этого мы не исключаем, что процессинг транскриптов кластера flamenco может зависеть от rhino так же, как процессинг транскриптов кластера 42AB. Однако на уровень экспрессии piРНК из этого кластера повышенная частота сплайсинга, вероятно, не влияет.
Зависимость уровня экспрессии ретротранспозонов от уровня экспрессии гена rhino
Далее мы провели исследование транскрипции МГЭ в тканях нокдаун-мутанта по гену rhino и проанализировали взаимосвязь транскрипции МГЭ с уровнем сплайсированных/несплайсированных форм исследуемых кластеров. Для анализа были выбраны ДКП-ретротранспозоны, которые ранее демонстрировали повышение экспрессии в ответ на стресс либо повышали экспрессию в тканях головы при мутациях центральных участников цикла “пинг-понг”, aub либо ago3, по данным P. Perrat и соавт. [14]: gypsy, roo, copia, Tirant и blood. Также мы взяли несколько теломерных LINE-элементов, которые, как показано ранее, по-разному реагируют на снижение экспрессии rhino: HeT-A, TART-А, TART-B и TART-C [20].
С помощью базы данных FlyBase (http://flybase.org) [21] мы проверили наличие копий изучаемых нами ретротранспозонов в исследуемых кластерах (flamenco, 42AB, 20A и 38C) и обнаружили, что в этих кластерах присутствуют только копии ДКП-ретротранспозонов, в то время как теломерные LINE-элементы в них не представлены. Кластер flamenco содержит ДКП-ретротранспозон gypsy, в кластере 20A есть копии roo, copia и blood, кластер 42AB содержит gypsy, roo и Tirant, а в кластере 38С находятся gypsy, copia и roo.
ДКП-ретротранспозоны и теломерные LINE-элементы осуществляют транспозицию путем репликативного синтеза, однако в отличие от ДКП-ретротранспозонов элементы HeT-A, TART-A, TART-B и TART-C встраиваются после районов TAS в теломеры, замещая таким образом функцию теломеразы. LINE-элемент HeT-A в ходе эволюции утерял рамку считывания, кодирующую эндонуклеазу и обратную транскриптазу, поэтому способен осуществлять транспозицию только за счет белков, кодируемых родственными LINE-элементами. В настоящее время считается, что rhino запускает транскрипцию не только с кластеров, но и с одиночных копий как ДКП-ретротранспозонов, так и LINE-элементов, однако его роль в транскрипции одноцепочечных кластеров и в соматических тканях за пределами гонад остается неизученной [22].
Мы обнаружили, что в контрольных гибридах ретротранспозоны по-разному экспрессируются в тканях яичников, корпуса и головы. Большинство исследованных МГЭ закономерно экспрессируются на более низком уровне в яичниках по сравнению с другими тканями. Однако исключение составил МГЭ Tirant, который практически не работает в тканях корпуса и головы, в отличие от яичников. При этом он, скорее всего, содержится в двуцепочечном кластере 42АВ. Также следует отметить, что большинство ретротранспозонов имеют различный уровень транскрипции в голове и тканях корпуса, что может указывать на различный характер регуляции транскрипции МГЭ в разных соматических тканях.
При нокдауне rhino ДКП-ретротранспозоны gypsy, copia и теломерные LINE-элементы HeT-A и TART-C значительно повышают уровень экспрессии в тканях яичников и корпуса. Более того, теломерный LINE-элемент TART-B значительно повышает свою экспрессию не только в тканях яичников, но и во всех соматических тканях, в то время как другой теломерный ретротранспозон, TART-A, только в тканях головы (рис. 4,a, б). Для ДКП-ретротранспозона blood и Tirаnt мы обнаружили падение экспрессии только в одной из исследуемых тканей. Для ДКП-ретротранспозона roo мы обнаружили падение экспрессии в тканях корпуса, в то время как в тканях головы изменений не было.
Рис. 4.
Относительная экспрессия различных ретротранспозонов в тканях яичников, головы и корпуса. а – относительная экспрессия ДКП-ретротранспозонов; б – относительная экспрессия теломерных LINE-элементов.
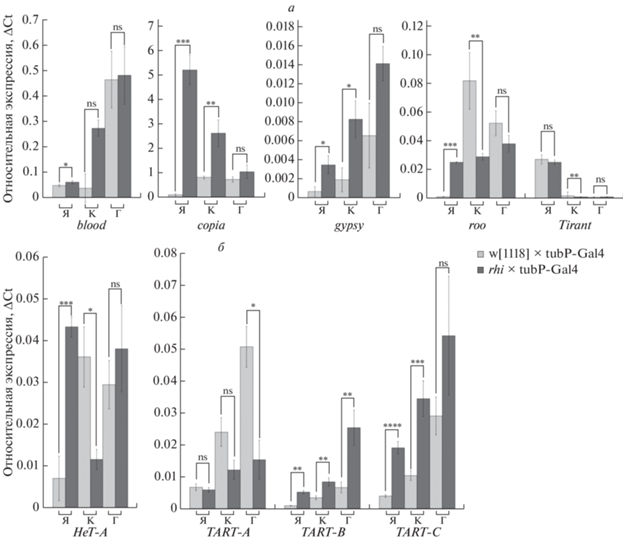
Таким образом, изменения в считывании кластеров могут приводить к повышению уровня транскрипции одних ретротранспозонов и к его снижению у других. Возможно, что при снижении экспрессии rhino транскрипция кластеров изменяется и либо сплайсированные транскрипты способны быть источником piРНК против некоторых ретротранспозонов, либо эти МГЭ активно подавляются системой siРНК-интерференции, куда могут направляться транскрипты кластеров после сплайсинга. Более того, мы увидели повышение транскрипции одних участков кластеров и снижение других, что может быть причиной усиления образования пре-si и piРНК против roo, но падения количества антисмысловых транскриптов против gypsy, copia, TART-B, TART-C и HeT-A.
ОБСУЖДЕНИЕ
Белок Rhi входит в состав комплекса RDC, взаимодействуя гетерохроматиновыми метками H3K9Me3, маркирующими кластеры piРНК [10]. С помощью хромотеневого (chromoshadow) домена Rhi взаимодействует с белком Del, который, в свою очередь, связывается с белком Cuff. Роль Сuff заключается в конкурировании за кэп на $5{\kern 1pt} '$-конце и препятствовании полиаденилированию транскрипта piРНК. Кроме того, Cuff взаимодействует с комплексом TREX, который отвечает за транспорт пре-piРНК из ядра. Таким образом, Rhi является фундаментом для построения комплекса RDC, который необходим для запуска транскрипции двуцепочечных кластеров с неканонических сайтов, участвуя в транскрипции piРНК как с двуцепочечных кластеров, так и с единичных вставок ретротранспозонов. Новые ретротранспозоны становятся кластерами после присоединения меток H3K9me3 благодаря привлечению гетерохроматиновых белков с помощью PIWI и препятствию началу транскрипции с канонических сайтов белком Mael [9, 19, 23–25].
В нашей работе мы продемонстрировали, что функция гена rhino необходима для функционирования как двуцепочечных, так и одноцепочечных кластеров. Мы обнаружили, что снижение экспрессии rhino приводит к активному сплайсингу транскриптов одноцепочечного кластера flamenco так же, как и двуцепочечного кластера 42AB (при этом экспрессия несплайсированных форм кластеров в яичниках остается на прежнем уровне), что указывает на возможную зависимость процессинга транскриптов flamenco от комплекса RDC, фундаментом которого является rhino. Однако изменений в экспрессии piРНК этого кластера мы не наблюдаем. Подобные эффекты наблюдали и другие исследователи, изучая вопрос взаимодействия rhino c кластером flamenco с помощью Chip-seq [6, 26]. В данных работах взаимодействие flamenco и rhino не было достоверно установлено, однако в нашем случае мы видим слабое влияние нокдауна rhino, оказываемое на сплайсинг. Так как транскрипция кластеров – это сложный процесс, который не до конца изучен, мы не исключаем, что кластер flamenco может обладать минорными механизмами регуляции. Например, кластер 42AB транскрибируется с неканонических сайтов только в одном направлении и требует присутствия комплекса RDC только для транскрипции одной из цепей [27]. Таким образом, в нормальных условиях присутствует некоторое количество сплайсированых и несплайсированных форм кластеров. При нокдауне rhino баланс сдвигается в сторону увеличения количества сплайсированных форм, и вероятно происходит вырезание участков, которые в норме используются для процессинга piРНК. Повышение количества сплайсированных форм кластера 42AB в тканях яичников и головы в ответ на понижение экспрессии rhino подтверждает наше предположение, что кластеры могут выполнять роль источника piРНК и в соматических тканях. В работах других исследователей было показано, что rhino экспрессируется в соматических клетках и в клетках зародышевого пути яичников дрозофилы [28]. Считается, что наличие сплайсинга приводит к отправке транскриптов по пути siРНК, что было показано для кластеров 42AB и flamenco в голове у дрозофилы [29]. Возможно, при отсутствии rhino система siРНК-интерференции частично замещает piРНК-интерференцию.
Показано, что одиночные копии I-элемента, относящегося к классу LINE-элементов, могут играть роль источников piРНК, причем эти кластеры делятся на сильные и слабые в зависимости от обогащения данных участков генома белком Rhi. Более того, старые копии I-элемента в линии R (чувствительна к I-элемент-индуцируемому гибридному дисгенезу) могут являться источниками первых piРНК для установки меток H3K9me3 на новых инсерциях [22]. Также источниками piРНК может служить сателлитная ДНК с метками H3K9me3; транскрипты с таких участков присутствуют не только в гонадах, но и различных тканях тела [18]. Также известно, что у дрозофилы некоторые кластеры транскрибируются полоспецифично [30].
В настоящей работе мы увидели, что снижение экспрессии rhino сказывается неоднозначно на экспрессии различных ретротранспозонов в соматических тканях. Большинство ретротранспозонов отреагировали на снижение экспрессии данного гена только в яичниках. Ретротранспозоны copia, gypsy, TART-C и HeT-A повысили уровень экспрессии только в гонадах и корпусе, а ретротранспозон TART-B начал активно экспрессироваться во всех тканях.
Известно, что теломерные сателлитные районы (TAS), а также теломерные транспозоны HeT-A, TAHRE и TART являются rhino-зависимыми источниками piРНК и транскрибируются с двух цепей [22]. Более того, гетерохроматиновые метки H3K9me3 обнаруживаются у дрозофилы и на теломерах в соматических тканях [20]. Повышение активности теломерных транспозонов не обязательно приводит к удлинению теломер за счет активной транспозиции. Так, мутация гена hiphop (привлекает белок Hp1a для метилирования участков, содержащих МГЭ) приводит к значительному увеличению экспрессии теломерных ретротранспозонов без повышения транспозиционной активности [22]. Показано, что мутации генов spnE и enok системы piРНК-интерференции приводят к повышению экспрессии различных семейств ретротранспозонов, включая теломерные [8].
Регуляция различных ретротранспозонов может быть тканеспецифичной, что мы наблюдаем в случае ретротранспозонов copia, TART-C, HeT-A, gypsy и TART-B, первые четыре из которых регулируются компонентами системы piРНК только в яичниках и корпусе, а последний – во всем теле. Мы предполагаем, что различия в экспрессии ретротранспозонов в разных тканях единого организма могут зависеть как от работы кластеров в этих тканях, так и от дополнительных факторов транскрипции.
Авторы выражают благодарность старшему научному сотруднику Института молекулярной генетики РАН М.С. Кленову за предоставление линии rhi и старшему научному сотруднику Института биологии развития им. Н.К. Кольцова РАН Ю.Е. Воронцовой за предоставление линии tubP-Gal4.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Park E.G., Ha H., Lee D.H. et al. // Genomic analyses of non-coding RNAs overlapping transposable elements and its implication to human diseases // Intern. J. Mol. Sciences. 2022. V. 23. P. 8950. https://doi.org/10.3390/ijms23168950
Schwarz D.S., Hátvágner G., Haley B., Zamore P.D. Evidence that siRNAs function as guides, not primers, in the drosophila and human RNAi pathways // Mol. Cell. 2002. V. 10. P. 537–548. https://doi.org/10.1016/S1097-2765(02)00651-2
Guerreiro G.M.P. What makes transposable elements move in the Drosophila genome // Heredity. 2012. V. 108. P. 461–468. https://doi.org/10.1038/hdy.2011.89
Carthew R.W., Sontheimer E.J. Origins and mechanisms of miRNAs and siRNAs // Cell. 2009. V. 136. P. 642–655. https://doi.org/10.1016/j.cell.2009.01.035
Yamanaka S., Siomi M.C., Siomi H. PiRNA clusters and open chromatin structure // Mobile DNA. 2014. V. 5. P. 22. https://doi.org/10.1186/1759-8753-5-22
Zhang Zh., Wang J., Schultz N. et al. The HP1 homolog Rhino anchors a nuclear complex that suppresses piRNA precursor splicing // Cell. 2014. V. 157. № 6. P. 1353–1363. https://doi.org/10.1016/j.cell.2014.04.030
Claycomb J.M. Emerging from the Clouds: Vasa helicase sheds light on piRNA amplification // Dev. Cell. 2014. V. 29. P. 632–634. https://doi.org/10.1016/j.devcel.2014.06.009
Tsai S.-Y., Huang F. Acetyltransferase Enok regulates transposon silencing and piRNA cluster transcription // PLoS Genetics. 2021. V. 17. № 2. https://doi.org/10.1371/journal.pgen.1009349
Yu B., Lin Y.A., Parhad S.S. et al. Structural insights into Rhino–Deadlock complex for germline piRNA cluster specification // EMBO Reports. 2018. V. 19. https://doi.org/10.15252/embr.201745418
Théron E., Dennis C., Brasset E., Vaury C. Distinct features of the piRNA pathway in somatic and germ cells: From piRNA cluster transcription to piRNA processing and amplification // Mobile DNA. 2014. V. 5. № 28. https://doi.org/10.1186/s13100-014-0028-y
Kim K.W., Tang N.H., Andrusiak M.G. et al. A neuronal piRNA pathway inhibits axon regeneration in C. elegans // Neuron. 2018. V. 97(3). P. 511–519. https://doi.org/10.1016/j.neuron.2018.01.014
Wakisaka K.T., Tanaka R., Hirashima T. et al. Novel roles of drosophila FUS and Aub responsible for piRNA biogenesis in neuronal disorders // Brain Research. 2019. V. 1708. P. 207–219. https://doi.org/10.1016/j.brainres.2018.12.028
Kim K.W. PIWI proteins and piRNAs in the nervous system // Mol. Cells. 2019. V. 42(12). P. 828–835. https://doi.org/10.1016/j.neuron.2018.01.014
Perrat P.N., DasGupta Sh., Wang J. et al. Transposition driven genomic heterogeneity in the drosophila brain // Science. 2013. V. 340(6128). P. 9195. https://doi.org/10.1126/science.1231965
Лавренов А.Р., Нефедова Л.Н., Романова Н.И., Ким А.И. Экспрессия генов семейства HP1 и их возможная роль в формировании фенотипа flamenco у D. melanogaster // Биохимия. 2014. Т. 79. № 11. С. 1554–1560.
Hur J.K., Luo Y., Moon S. et al. Splicing-independent loading of TREX on nascent RNA is required for efficient expression of dual-strand piRNA clusters in drosophila // Genes & Development. 2016. V. 30. P. 840–855. https://doi.org/10.1101/gad.276030.115
Sayers E.W., Bolton E.E., Brister J.R. et al. Database resources of the national center for biotechnology information // Nucl. Ac. Res. 2022. V. 50. (D1):D20–D26. https://doi.org/10.1093/nar/gkab1112
Wei X., Eickbush D.G., Speece I., Larracuente A.M. He-terochromatin-dependent transcription of satellite DNAs in the Drosophila melanogaster female germline // eLife. 2021. V. 10. https://doi.org/10.7554/eLife.62375
Chang T.H., Mattei E., Gainetdinov I. et al. Maelstrom represses canonical polymerase II transcription within bi-directional piRNA clusters in Drosophila melanogaster // Mol. Cell. 2019. V. 73. P. 291–303. https://doi.org/10.1016/j.molcel.2018.10.038
Radion E., Morgunova V., Ryazansky S. et al. Key role of piRNAs in telomeric chromatin maintenance and telomere nuclear positioning in drosophila germline // Epigenetics & Chromatin. 2018. V. 11:40. https://doi.org/10.1186/s13072-018-0210-4
Gramates L.S., Agapite J., Attrill H. et al. FlyBase: A guided tour of highlighted features // Genetics. 2022. V. 220. № 4. https://doi.org/10.1093/genetics/iyac035
Cui M., Bai Y., Li K., Rong Y.S. Taming active transposons at drosophila telomeres: The interconnection between HipHop’s roles in capping and transcriptional silencing // PLoS Genetics. 2021. V. 17(11). https://doi.org/10.1371/journal.pgen.1009925
Sato K., Siomi M.C. Two distinct transcriptional controls triggered by nuclear Piwi-piRISCs in the drosophila piRNA pathway // Curr. Op. in Structural Biology. 2018. V. 53. P. 69–76. https://doi.org/10.1016/j.sbi.2018.06.005
Zhang G., Tu S., Yu T. et al. Co-dependent assembly of drosophila piRNA precursor complexes and piRNA cluster heterochromatin // Cell Reports. 2018. V. 24. P. 3413–3422. https://doi.org/10.1016/j.celrep.2018.08.081
Akulenko N., Ryazansky S., Morgunova V. et al. Transcriptional and chromatin changes accompanying de novo formation of transgenic piRNA clusters // RNA. 2018. V. 24. № 4. P. 574–584. https://doi.org/10.1261/rna.062851.117
Klattenhoff C., Xi H., Li C. et al. The drosophila HP1 homolog Rhino is required for transposon silencing and piRNA production by dual-strand clusters // Cell. 2009. V. 138. № 6. P. 1137–1149. https://doi.org/10.1016/j.cell.2009.07.014
Parhad S.S., Yu T., Zhang G. et al. Adaptive evolution targets a piRNA precursor transcription network // Cell Reports. 2020. V. 30. № 8. P. 2672–2685. https://doi.org/10.1016/j.celrep.2020.01.109
Volpe A.M., Horowitz H., Grafer C.M. et al. Drosophila rhino encodes a female-specific chromo-domain protein that affects chromosome structure and egg polarity // Genetics. 2001. V. 159. P. 1117–1134. https://doi.org/10.1093/genetics/159.3.1117
Chung W.-J., Okamura K., Martin R., Lai E.C. Endogenous RNA interference provides a somatic defense against drosophila transposons // Curr. Biology. 2008. V. 18. P. 795–802. https://doi.org/10.1016/j.cub.2008.05.006
Chen P., Luo Y., Aravin A.A. RDC complex executes a dynamic piRNA program during drosophila spermatogenesis to safeguard male fertility // PLoS Genetics. 2021. V. 17(9). https://doi.org/10.1371/journal.pgen.1009591
Дополнительные материалы отсутствуют.


