Генетика, 2023, T. 59, № 2, стр. 157-169
Полиморфизм и дифференциация трех популяций эворонской полевки по данным изменчивости контрольного региона митохондриальной ДНК
И. Н. Шереметьева 1, *, И. В. Картавцева 1, Л. В. Фрисман 2
1 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии Дальневосточного отделения
Российской академии наук
690022 Владивосток, Россия
2 Институт комплексного анализа региональных проблем Дальневосточного отделения
Российской академии наук
679016 Биробиджан, Россия
* E-mail: sheremet76@yandex.ru
Поступила в редакцию 11.03.2022
После доработки 27.04.2022
Принята к публикации 11.05.2022
- EDN: EGKVYG
- DOI: 10.31857/S0016675823010101
Аннотация
В результате анализа контрольного региона митохондриальной ДНК для эворонской полевки было обнаружено высокое гаплотипическое (97%) и нуклеотидное (1.53%) разнообразие. Полученные данные свидетельствуют о низком уровне генетической дифференциации популяций. При этом популяции, географически расположенные ближе, подразделены сильнее, чем удаленные. В целом наблюдается согласованность между данными изменчивости контрольного региона митохондриальной ДНК и хромосомными расами. Однако если данные хромосомного анализа указывают на сохранение более древних вариантов кариотипа хромосомной расы “эворон” в Эвороно-Чукчагирской низменности, то данные молекулярно-генетического анализа свидетельствуют в пользу сохранения наиболее древнего мт гаплотипа (106-19) в популяции Верхнебуреинской впадины (хромосомная раса “арги”).
Alexandromys evoronensis (Kovalskaya et Sokolov, 1980) – узкоареальный эндемик Дальнего Востока России [1–4]. В настоящее время некоторые исследователи рассматривают эворонскую полевку как полувид надвида “maximowiczii”. В данный надвид кроме эворонской полевки еще входят полевка Максимовича (Аlexandromys maximowiczii Schrenck, 1858) и муйская полевка (Аlexandromys mujanensis Orlov et Kowalskaia, 1978) [5]. Между представителями надвида “maximowiczii” выявлен низкий, сопоставимый с внутривидовым, уровень дифференциации по морфологическим, аллозимным и молекулярно-генетическим данным [6–12]. Несмотря на это, данные кариологического анализа и экспериментальной гибридизации свидетельствуют в пользу самостоятельности всех трех видов [6, 13–16]. До последнего времени принято было считать, что эворонская полевка обитает только в пределах Эвороно-Чукчагирской низменности [3, 6, 14, 17, 18] и ее проникновение на запад затруднено горными хребтами Буреинским, Дуссе-Алинь и Ям-Алинь [6]. Однако за последние 10 лет для эворонской полевки были обнаружены две новые популяции за пределами Эвороно-Чукчагирской низменности: в Верхнебуреинской впадине в долине р. Ургал [19] и на северо-востоке Верхнезейской равнины в долине р. Арги [20]. Кариологический анализ показал наличие двух хромосомных рас у A. evoronensis: “эворон” (2n = 38–41, NF = 54–59) и “арги” (2n = 34, 36, 37, NF = 51–56) [15, 16]. К первой расе относятся полевки Эвороно-Чукчагирской низменности, ко второй – особи Верхнебуреинской впадины и Верхнезейской равнины.
Цель настоящей работы – оценить генетическое разнообразие и уровень дифференциации трех популяций эворонской полевки по данным изменчивости контрольного региона мтДНК и сопоставить полученные результаты с данными кариологического анализа.
МАТЕРИАЛЫ И МЕТОДЫ
В работе использованы образцы тканей от 43 эворонских полевок, отловленных в трех популяциях: Эвороно-Чукчагирская низменность (n = 12), Верхнебуреинская впадина (n = 17) и Верхнезейская равнина (n = 14) (табл. 1). Часть из этих полевок ранее были кариотипированы [15, 16]. Образцы хранятся в УНУ “Биоресурсная коллекция ФНЦ Биоразнообразия ДВО РАН” (г. Владивосток). Также в анализ включены пять нуклеотидных последовательностей контрольного региона мтДНК эворонской полевки из Эвороно-Чукчагирской низменности, полученных нами ранее [8] и помещенных в GenBank (NCBI) под номерами HM135858–HM135862.
Таблица 1.
Материал
| Код популяции | Локалитет/год/координаты | № хром. препарата | Хром. раса#/вариант кариотипа | Номер образца | Номер GenBank |
|---|---|---|---|---|---|
| Evoron | Окрест. с. Харпичан/2006/51°19′ с.ш., 136°35′ в.д. | 2050 | I/2n = 40а, NF = 57* | 81-06 | HM135862*** |
| Слияние рек Нимелен и Амгунь/2008/ 52°26′ с.ш., 136°34′ в.д. | 2355 | I/2n = 41a, NF = 56* | 50-08 | HM135861*** | |
| – | 51-08 | HM135860*** | |||
| – | 59-08 | OM966944 | |||
| – | 60-08 | OM966945 | |||
| 2369 | I/2n = 41a, NF = 56* | 87-08 | HM135858*** | ||
| 2370 | I/2n = 41a, NF = 56* | 88-08 | HM135859*** | ||
| Окрест. с. Полина Осипенко/2016/ 52°25.42′ с.ш., 136°29.67′ в.д. | – | 11-16 | OM966947 | ||
| – | 23-16 | OM966948 | |||
| – | 34-16 | OM966949 | |||
| 0060 | I/2n = 41a, NF = 56* | 58-16 | OM966950 | ||
| 0064 | I/2n = 40f, NF = 55* | 91-16 | OM966951 | ||
| 0063 | I/2n = 41a, NF = 56* | 92-16 | OM966952 | ||
| 0062 | I/2n = 40e, NF = 54* | 93-16 | OM966946 | ||
| – | 94-16 | OM966953 | |||
| 0066 | I/2n = 41a, NF = 56* | 95-16 | OM966954 | ||
| – | 141-16 | OM966955 | |||
| Argi | Слияние р. Арги и Зейской протоки/ 2015/54°40′ с.ш., 129°06′ в.д. | – | 2-15 | OM966968 | |
| – | 3-15 | OM966959 | |||
| 3950 | II/2n = 36a, NF = 54** | 4-15 | OM966969 | ||
| – | 15-15 | OM966956 | |||
| – | 20-15 | OM966960 | |||
| 3991 | II/2n = 36a, NF = 54** | 21-15 | OM966961 | ||
| 3992 | II/2n = 36a, NF = 54** | 22-15 | OM966962 | ||
| 3993 | II/2n = 34b, NF = 52** | 23-15 | OM966963 | ||
| 3994 | II/2n = 36c, NF = 55** | 24-15 | OM966964 | ||
| 3995 | II/2n = 36c, NF = 55** | 25-15 | OM966965 | ||
| 3996 | II/2n = 36d, NF = 55** | 26-15 | OM966958 | ||
| 3997 | II/2n = 36c, NF = 55** | 27-15 | OM966966 | ||
| – | 28-15 | OM966967 | |||
| 3999 | II/2n = 36c, NF = 55** | 29-15 | OM966957 | ||
| Urgal | Окрест. пгт. Чегдомын/2014/ 51°05′ с.ш., 132°33′ в.д. | – | 139-14 | OM966985 | |
| – | 146-14 | OM966986 | |||
| Окрест. пгт. Чегдомын/2016/ 51°05′ с.ш., 132°33′ в.д. | – | 36-16 | OM966983 | ||
| – | 90-16 | OM966984 | |||
| Окрест. пос. Ургал/2019/ 51°07′ с.ш., 132°31′ в.д. | – | 36-19 | OM966972 | ||
| – | 37-19 | OM966973 | |||
| – | 70-19 | OM966975 | |||
| – | 72-19 | OM966974 | |||
| – | 73-19 | OM966977 | |||
| – | 74-19 | OM966970 | |||
| – | 75-19 | OM966971 | |||
| – | 80-19 | OM966976 | |||
| – | 82-19 | OM966978 | |||
| 4549 | II/2n = 36c, NF = 55** | 101-19 | OM966979 | ||
| 4554 | II/2n = 37a, NF = 55** | 106-19 | OM966982 | ||
| 4556 | II/2n = 37b, NF = 55** | 107-19 | OM966980 | ||
| 4557 | II/2n = 36b, NF = 56** | 108-19 | OM966981 |
Выделение ДНК осуществляли методом солевой экстракции [21] из фиксированных в спирте тканей мышц. Подробная методика по приготовлению реакционной смеси, схема ПЦР-реакции для получения фрагмента и подготовки проб к секвенированию были описаны ранее [22]. Нуклеотидную последовательность прочитывали на автоматическом секвенаторе ABI Prizm 3130 на базе центра коллективного пользования “Биотехнология и генетическая инженерия” ФНЦ Биоразнообразия ДВО РАН (г. Владивосток). Редактирование и выравнивание полученных последовательностей проводили с использованием программы BioEdit 7.0.9.0 [23]. Построение филогенетических деревьев выполнено в программе MEGA X [24] методом максимального правдоподобия (Maximum Likelihood), достоверность кластеризации оценивали с помощью бутстрэп-анализа (1000 повторностей). Анализ максимального правдоподобия проводили с использованием эволюционной модели Тамура–Нея93 (TN93) [28] со значением эволюционной нейтральности сайтов (+I) равного 0.87 и максимальным логарифмом правдоподобия – 1970.162. Выбор эволюционной модели ML-анализа определяли по результатам модель-теста в программе MEGA X [24], по которым для модели TN93+I были получены минимальные значения байесового информационного критерия (BIC = 5001.735). Сети гаплотипов построены при помощи программы Network 10.0.0.0 с использованием метода “median joining” [25]. Показатели генетического разнообразия и дифференциации популяций рассчитаны с помощью DnaSP 6.00 [26]. Расчет статистик нейтральности (Tajima’s D и Fu’s Fs), анализ демографической и пространственной экспансии проводили в программе Arlequin 3.5 [27].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Для всех 43 новых образцов эворонской полевки из трех популяций были получены последовательности контрольного региона мтДНК длиной 952 пн, все они внесены в GenBank (NCBI) под номерами OM966944–OM966984. Уровень изменчивости митохондриальных фрагментов контрольного региона эворонской полевки (число нуклеотидных замен) был достаточно высоким, число нуклеотидных различий между отдельными гаплотипами варьировало от 0 до 28 (0.0 до 2.9%) (табл. 2) и в среднем было равно 14.42. Частоты нуклеотидов составляли A = 29.17%, T = 32.72%, C = 25.55% и G = 12.56%. Все нуклеотидные последовательности вида (48 экз.) содержали 76 вариабельных сайтов (включая пять делеций), среди которых 48 были парсимониально информативны. Частота транзиций составила 0.0659, а трансверсий – 0.0095. В целом для вида обнаружено 38 гаплотипов, из них пять были описаны ранее [8]. Из обнаруженных гаплотипов один был встречен у шести особей, один – у трех, три – у двух, остальные 33 гаплотипа (86.84%) были уникальными (встречены только у одной особи). Гаплотипическое разнообразие для вида в целом было – 0.97 ± 0.014, а нуклеотидное – 0.0153 ± 0.0005.
Таблица 2.
Полиморфные сайты контрольного региона мтДНК эворонской полевки
| Urgal | Evoron | Argi | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 106-19 | 74-19 | 36-19 | 70-19 | 73-19 | 82-19 | 108-19 | 36-16 | 90-16 | 87-08 | 88-08 | 51-08 | 50-08 | 81-06 | 59-08 | 60-08 | 93-16 | 11-16 | 23-16 | 34-16 | 58-16 | 91-16 | 92-16 | 94-16 | 95-16 | 141-16 | 15-15 | 29-15 | 3-15 | 20-15 | 21-15 | 22-15 | 23-15 | 25-15 | 27-15 | 28-15 | 2-15 | 4-15 | |
| 37* | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 38* | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . |
| 40* | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 41 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | G | . | . |
| 73 | A | . | . | . | . | G | . | . | . | . | . | . | . | . | G | G | . | . | . | . | G | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . |
| 75 | C | . | . | . | . | . | . | . | . | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T |
| 80* | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | T | T | T | T | T | T | T | T | T | T | T |
| 109 | G | . | . | . | . | . | . | . | . | A | A | A | A | . | A | . | . | . | . | A | A | A | A | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . |
| 123 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 125 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 142 | G | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | A | A | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 148 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | C | C | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . |
| 155 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 156 | A | G | G | G | G | G | G | G | G | . | . | . | . | . | . | G | . | . | . | . | . | G | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 163* | A | G | G | T | T | G | G | G | G | G | G | G | G | G | G | . | G | G | G | G | G | G | G | G | G | G | . | . | . | G | . | . | . | G | . | G | G | . |
| 164 | A | G | . | . | . | G | G | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 171 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 193 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | T | . | T | T | . |
| 195 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | G | . | . | . | . | G | . | . | . | . | . |
| 196* | T | . | . | . | . | . | . | . | . | A | A | A | A | . | A | C | . | . | A | A | A | A | A | . | A | . | . | . | C | . | C | C | . | . | C | . | . | . |
| 209 | A | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 213 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 245 | T | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 272 | T | . | . | . | . | . | . | . | C | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | C | C | C | C | C | C | C | C | C | C | C | C |
| 273 | C | . | . | . | . | . | . | . | T | T | T | T | T | T | T | . | . | . | T | T | T | T | T | T | T | T | . | . | . | . | . | . | . | . | . | . | . | . |
| 274 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | C | C | . | C | C | C | . | C | . | . | C |
| 278 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . |
| 286 | C | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T |
| 288 | G | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | A | A | A | . | . | . | . | A | A | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 370 | C | . | . | T | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 411 | T | C | C | . | . | C | C | C | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 432 | T | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 617 | T | C | C | . | . | C | C | C | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 628 | T | . | . | C | C | . | . | . | . | C | . | . | . | . | C | . | . | . | . | C | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 630 | G | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | C | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A | A |
| 631* | C | T | T | . | . | T | T | T | T | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 632 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 633 | G | A | A | . | . | A | A | A | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 652 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 653 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . |
| 655 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 656 | C | . | . | . | . | . | . | . | . | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | . | . | . | . | . | . | . | . | . | . | . | . |
| 657 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | C | C | C | C | C | C | C | C | C | C | C |
| 669 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 670 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 677 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . |
| 685 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | A | . | . | . | . |
| 700 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 706 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | A | . | A | A | . |
| 711 | T | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 723 | G | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 728 | A | . | . | . | . | . | . | . | . | G | G | . | G | . | . | . | G | G | . | . | . | G | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 737 | T | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | C | C | C | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 741 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | G | . |
| 742 | G | . | . | . | . | . | . | . | . | A | A | A | A | . | A | . | . | . | . | . | A | A | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 743 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | C | . | . | . | . |
| 748 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 751 | T | . | . | . | . | . | . | . | . | C | C | C | C | . | C | . | . | . | . | C | C | C | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 754 | C | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 759 | C | T | T | T | T | T | T | T | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 760 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 761 | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 762* | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | A | A | . | . | A | . | . | . |
| 768* | T | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 769# | A | . | . | . | . | . | . | . | . | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | . | . | . | G | . | . | . | G | . | G | G | . |
| 770 | T | . | . | . | . | . | . | . | . | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | . | . | . | . | . | . | . | . | . | . | . | . |
| 771# | - | - | - | - | - | - | - | - | - | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | - | - | - | - | - | - | - | - | - | C |
| 772# | - | - | - | - | - | - | - | - | - | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | T | - | - | - | - | - | - | - | - | - | T |
| 774# | T | . | C | C | C | . | . | C | . | - | - | - | - | - | - | - | - | . | . | . | . | . | . | . | . | . | . | . | . | . | C | C | C | . | C | . | . | . |
| 781 | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | A | A | A | A | A | A | A | A | A | A | A |
| 784 | C | T | T | T | T | T | T | T | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 787 | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | T | . | . | . | . | . |
| 789# | - | - | - | - | - | - | - | - | - | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C | C |
| 790 | A | G | G | G | G | G | G | G | G | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | G | G | . |
| 793 | C | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| 796 | C | T | T | T | T | T | T | T | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | T | T | T | T | T | T | T | T | T | T | T |
В выборке эворонской полевки из Эвороно-Чукчагирской низменности нуклеотидные последовательности содержали 43 вариабельных сайта, среди которых 16 были парсимониально информативны (табл. 3). Для этой выборки характерны самые высокие показатели как гаплотипического (100%), так и нуклеотидного (1.1%) разнообразия, обнаруженные в пределах данного вида. Все отловленные в этой низменности особи имели уникальные гаплотипы, различающиеся 1–21 нуклеотидной заменой. Среднее число попарных нуклеотидных различий между гаплотипами внутри выборки составило 10.26.
Таблица 3.
Показатели генетического разнообразия трех популяций эворонской полевки
| Параметр | Evoron | Argi | Urgal |
|---|---|---|---|
| n | 17 | 14 | 17 |
| N | 17 | 12 | 9 |
| Vs | 43 | 24 | 25 |
| k ± S.E. | 10.26 ± 1.924 | 8.41 ± 1.710 | 8.68 ± 1.823 |
| h ± S.D. | 1.000 ± 0.0202 | 0.9782 ± 0.027 | 0.8603 ± 0.02 |
| π ± S.D. | 0.01058 ± 0.0057 | 0.0086 ± 0.0047 | 0.0068 ± 0.0038 |
| Tajima’s D (P) | –0.99 (0.165) | 0.36 (0.679) | –0.51 (0.319) |
| Fu’s Fs (P) | –9.27 (0.00) | –7.64 (0.00) | –12.60 (0.00) |
| Показатели пространственной экспансии | |||
| τ | 8.8 | 7.9 | 8.2 |
| θ | 2.529 | 3.188 | 2.986 |
| М | 249.86 | 31.24 | 1.73 |
| SSD (P) | 0.007 (0.40) | 0.014 (0.45) | 0.028 (0.80) |
| Показатели демографической экспансии | |||
| τ | 11.2 | 11.1 | 11.2 |
| θ0 | 0.007 | 0.002 | 0.000 |
| θ1 | 44.093 | 27.148 | 10.238 |
| SSD (P) | 0.005 (0.90) | 0.008 (0.95) | 0.040 (0.45) |
Примечание. n – объем выборки; N – число гаплотипов; Vs – число вариабельных сайтов; k – среднее число попарных нуклеотидных различий; h – гаплотипическое разнообразие; π – нуклеотидное разнообразие; Tajima’s D – коэффициент теста Таджимы; Fu’s Fs – коэффициент теста Фу; τ – время экспансии в мутационных единицах; θ – мутационные параметры на начальном и конечном этапах роста численности; М – межпопуляционный обмен мигрантами; SSD – сумма квадратов отклонений между наблюдаемым и ожидаемым распределением попарных нуклеотидных различий; S.D. – стандартное отклонение; S.E. – стандартная ошибка; P – p-значение.
Генетическое разнообразие выборки из Верхнезейской равнины было ниже, чем для Эвороно-Чукчагирской низменности. Гаплотипическое разнообразие составило здесь 97.8%, а нуклеотидное – 0.86% (табл. 3). В выборке было обнаружено 12 гаплотипов, только два из которых были встречены у двух особей. В целом нуклеотидные последовательности этой выборки имели минимальное число вариабельных (24) и парсимониально информативных (16) сайтов. Минимальное среднее число попарных нуклеотидных различий равно 8.41, при этом число нуклеотидных различий между отдельными гаплотипами варьировало от 1 до 14.
Самые низкие значения гаплотипического (86%) и нуклеотидного (0.68%) разнообразия были отмечены в выборке Верхнебуреинской впадины (табл. 3). Так же для этой выборки характерно минимальное число парсимониально информативных (10) сайтов. При этом общее число вариабельных сайтов было 25. Всего для выборки Верхнебуреинской впадины обнаружено 9 гаплотипов на 17 особей. Треть этих гаплотипов была обнаружена больше чем у одной особи. Отдельные гаплотипы отличались друг от друга 1–19 нуклеотидной заменой, при этом среднее число попарных нуклеотидных различий между гаплотипами внутри выборки было не намного выше, чем в выборке Верхнезейской равнины, и составило 8.68.
Значения D Tajima для всех популяций эворонской полевки были незначимыми, но только для Верхнебуреинской впадины и Эвороно-Чукчагирской низменности они были отрицательными. Результаты теста Фу (Fu’s Fs) на селективную нейтральность для всех популяций были не только отрицательными, но и высоко достоверно отличались от нуля, что может указывать на рост численности в прошлом (табл. 3).
Распределение попарных нуклеотидных различий между гаплотипами особей во всех популяциях было полимодальным (рис. 1). При этом максимальная частота для сравнений внутри популяций Эвороно-Чукчагирской низменности и Верхнезейской равнины располагается в области 9–10 (значения парных различий), а внутри популяции Верхнебуреинской впадины – в области 16–18.
Рис. 1.
Частота (ось ординат) попарных нуклеотидных различий (ось абсцисс) в трех популяциях эворонской полевки. Наблюдаемая частота обозначена сплошной линией, ожидаемое распределение – пунктирной линией.
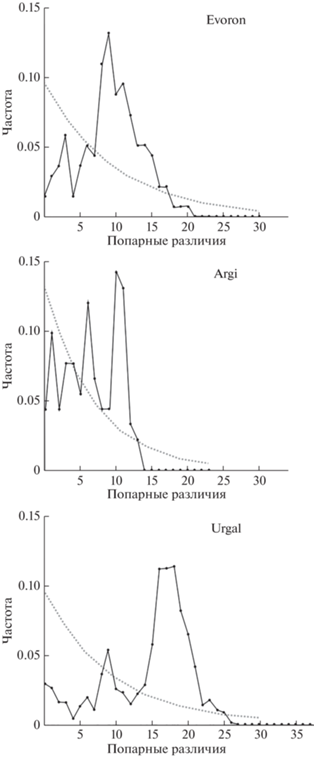
Невысокие значения условного времени τ, а также низкие и статистически недостоверные значения суммы отклонений SSD свидетельствуют в пользу гипотезы роста численности с последующей пространственной экспансией для всех исследуемых популяций в недавнем прошлом приблизительно в одно и то же время (табл. 3).
ML-анализ нуклеотидных последовательностей позволил построить дендрограмму и объединить последовательности в группы, характеризующие близкие гаплотипы эворонской полевки (рис. 2,а). На дендрограмме можно выделить клады, которые соответствуют трем популяциям: Эвороно-Чукчагирской низменности (Evoron), Верхнебуреинской впадины (Urgal) и Верхнезейской равнины (Argi). Исключением является гаплотип одной особи из популяции Urgal (106-19U). Этот гаплотип на дендрограмме имеет базальное расположение в единой для гаплотипов популяции Urgal и Argi кладе. С целью проведения более точной кластеризации гаплотипов эворонской полевки была построена медианная сеть (рис. 1). В целом сеть подтверждает данные, полученные при ML-анализе, показывая разделение всех гаплотипов на три группы согласно их географической приуроченности. Общих гаплотипов для двух и более популяций не обнаружено. Добавление в гаплотипическую сеть в качестве внешней группы гаплотипов характерных для полевки Максимовича позволило выявить, что гаплотип 106-19 имеет базальное расположение не только внутри единой клады “Urgal” и “Argi”, но и для вида в целом. Следует сказать, что ранее при проведении кариологического анализа у данной особи (№ 4554) была обнаружена неидентифицированная перестройка [16].
Рис. 2.
ML-дендрограмма (а) и филогенетическая сеть гаплотипов мтДНК (б) эворонской полевки из трех популяций. Размеры кружков пропорциональны количеству образцов с данным гаплотипом. Образцы обозначены в соответствии с табл. 1. Цифры на ветвях сети соответствуют числу нуклеотидных замен больше 2.

Средний коэффициент генной дифференциации (GST) для выборок всех популяций составил 0.0635, что указывает на то, что только 6.35% генетической изменчивости приходится на вариацию между популяциями (табл. 4). Значение Nm (потока генов) при этом составило 3.69, что указывает на высокий уровень потока генов и низкую или среднюю генетическую дифференциацию между популяциями. Стандартный тест на гомогенность при попарном сравнении выборок из разных популяций выявил достоверный (p < 0.05) уровень подразделенности только внутри пар Urgal–Argi (χ2 = 31.00, P = 0.040) и Urgal–Evoron (χ2 = 34.00, P = 0.049) (табл. 4). При этом значение коэффициента GST для этих пар выборок было одинаковым и составило 0.048 и 0.052 соответственно. А значение Nm при этом не превышало 4.94. Стандартный тест на гомогенность при попарном сравнении выборок из популяций Эвороно-Чукчагирской низменности (Evoron) и Верхнезейской равнины (Argi) не выявил достоверный уровень подразделенности (χ2 = 31.00, P = 0.154). При этом только 2.34% генетической изменчивости приходится на вариацию между популяциями, а поток генов был в два раза выше (Nm = 10.41). При этом следует отметить, что популяция Верхнезейской равнины географически удалена от популяций Эвороно-Чукчагирской низменности больше чем популяции Верхнебуреинской впадины (табл. 4).
Таблица 4.
Показатели генетической подразделенности трех популяций эворонской полевки
| Параметр | Evoron/Urgal | Evoron/Argi | Urgal/Argi |
|---|---|---|---|
| Среднее число нуклеотидных замен между популяциями | 18.799 | 17.592 | 17.878 |
| GST | 0.0522 | 0.0234 | 0.0482 |
| Nm | 4.54 | 10.41 | 4.94 |
| χ2 (P) | 34.00 (0.049) | 31.00 (0.154) | 31.00 (0.040) |
| Расстояние (в км) по прямой между выборками | 306 | 536 | 460 |
В результате анализа контрольного региона митохондриальной ДНК для эворонской полевки было показано высокое генетическое разнообразие для вида (гаплотипическое разнообразие равно 97%, нуклеотидное – 1.53%), несмотря на его небольшой ареал. В целом эти показатели оказались значительно выше, чем аналогичные для муйской полевки (92 и 1.2%) [29], и сопоставимы с генетическим разнообразием для таких широкоареальных видов рода как полевка Максимовича (99 и 1.41%) [22] и дальневосточная полевка (94 и 1.63%) [30]. Полученные нами данные свидетельствуют о низком уровне генетической дифференциации трех популяций. Самый большой поток генов обнаружен между популяциями полевок Эвороно-Чукчагирской низменности и Верхнезейской равнины, которые относятся к двум разным хромосомным расам – “эворон” и “арги” соответственно. Проведенный нами анализ показал, что географически удаленные популяции имеют больше сходства (табл. 4). Две популяции эворонской полевки, относящиеся к одной хромосомной расе “арги”, на филогенетических реконструкциях – сестринские. Наибольшим генетическим разнообразием отличались полевки, отловленные в Эвороно-Чукчагирской низменности, относящиеся к хромосомной расе “эворон”. Самые низкие показатели генетического разнообразия выявлены для особей из Верхнебуреинской впадины, относящиеся к хромосомной расе “арги”, при этом именно в этой популяции обнаружен гаплотип 106-19, который имеет базальное расположение на филогенетических реконструкциях и может считаться наиболее древним для вида.
Согласно данным хромосомного анализа можно считать, что в популяции Эвороно-Чукчагирской низменности сохранились наиболее древние варианты кариотипа (с наибольшим числом акроцентрических пар хромосом – 26 и 2n = 41), поскольку анцестральный для этого вида кариотип имел 2n = 42 [16]. Кроме того, учитывая, что показатели генетического разнообразия выше в центральных популяциях вида, можно было бы считать популяцию Эвороно-Чукчагирской низменности исходной для вида. Однако обнаружение наиболее древнего гаплотипа (106-19) в популяции Верхнебуреинской впадины ставит это предположение под сомнение и нуждается в дальнейшем детальном исследовании.
Работа выполнена при частичной поддержке гранта РФФИ 15-04-03871, в рамках Государственного задания Министерства науки и высшего образования Российской Федерации (тема № 121031500274-4 “Эволюционные аспекты формирования наземной биоты востока Азии”).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Gerrie R., Kennerley R. Microtus evoronensis (errata version published in 2017) // The IUCN Red List of Threatened Species. 2016. e.T13429A115112487. https://doi.org/10.2305/IUCN.UK.2016-3.RLTS.T1-3429A22350421
Shenbrot G.I., Krasnov B.R. An Atlas of the Geographic Distribution of the Arvicoline Rodents of the World (Rodentia, Muridae: Arvicolinae). Sofia: Pensoft, 2005. 336 p.
Громов И.М., Ербаева М.А. Млекопитающие фауны России и сопредельных территорий. Зайцеобразные и грызуны. СПб.: Изд. Зоол. ин-та РАН, 1995. 520 с.
Костенко В.А. Грызуны (Rodentia) Дальнего Востока России. Владивосток: Дальнаука, 2000. 209 с.
Абрамсон Н.А., Лисовский А.А. Род Alexandromys Ognev, 1914 // Млекопитающие России: ситематико-географический справочник (Сб. трудов Зоологического музея МГУ. Т. 52) / Под ред. Павлинова И.Я., Лисовского А.А. М.: Т-во науч. изд. КМК, 2012. С. 258–365.
Мейер М.Н., Голенищев Ф.Н., Раджабли С.И., Саблина О.Л. Серые полевки фауны России и сопредельных территорий СПб.: Зоол. ин-т РАН, 1996. 320 с.
Bannikova A.A., Lebedev V.S., Lissovsky A.A. et al. Molecular phylogeny and evolution of the Asian lineage of vole genus Microtus (Arvicolinae, Rodentia) inferred from mitochondrial cytochrome b sequence // Biological J. Linnean Society. 2010. V. 99. P. 595–613. https://doi.org/10.1111/j.1095-8312.2009.01378.x
Haring E., Sheremetyeva I., Kryukov A. Phylogeny of Palearctic vole species (genus Microtus, Rodentia) based on mitochondrial sequences // Mammalian Biology. 2011. V. 76. P. 258–267. https://doi.org/10.1016/j.mambio.2010.04.006
Лисовский А.А., Оболенская Е.В. Структура краниометрического разнообразия серых полевок Microtus подрода Alexandromys // Тр. Зоол. ин-та РАН. 2011. Т. 315. № 4. С. 461–477. https://www.zin.ru/journals/trudyzin/doc/vol_315_4/ tz_315_4_lisovsky.pdf
Фрисман Л.В., Картавцева И.В., Шереметьева И.Н. и др. Аллозимная дифференциация и кариотипические особенности восточноазиатских полевок Дальнего Востока России // Вecтник СВНЦ ДВО РАН. 2016. № 3. С. 93–103.
Lissovsky A.A., Petrova T.V., Yatsentyuk S.P. et al. Multilocus phylogeny and taxonomy of East Asian voles Alexandromys (Rodentia, Arvicolinae) // Zool. Scripta. 2018. V. 47. № 1. P. 9–20. https://doi.org/10.1111/zsc.12261
Лисовский А.А., Кадетова А.А., Оболенская Е.В. Морфологическое определение видов восточноазиатских серых полевок Alexandromys (Rodentia, Cricetidae) России и сопредельных территорий // Зоол. журн. 2018. Т. 97. № 1. С. 101–113. https://doi.org/10.7868/S0044513418010099
Ковальская Ю.М., Соколов В.Е. Новый вид полевок (Rodentia, Cricetidae, Microtus) из нижнего Приамурья // Зоол. журн. 1980. Т. 59. № 9. С. 1409–1416.
Голенищев Ф.Н., Раджабли С.И. Новый вид серой полевки с берегов озера Эворон // Докл. Акад. наук СССР. 1981. Т. 257. № 1. С. 248–250.
Kartavtseva I.V., Sheremetyeva I.N., Pavlenko M.V. Intraspecific multiple chromosomal variations including rare tandem fusion in the russian far eastern endemic evoron vole Alexandromys evoronensis (Rodentia, Arvicolinae) // Comparative Cytogenetics. 2021. V. 15. № 4. P. 393–411. https://doi.org/10.3897/compcytogen.v15.i4.67112
Картавцева И.В., Шереметьева И.Н., Павленко М.В. Множественный хромосомный полиморфизм, хромосомной расы “эворон” эворонской полевки (Rodentia, Arvicolinae) // Генетика. 2021. Т. 57. № 1. С. 82–94. https://doi.org/10.31857/S0016675821010082
Картавцева И.В., Шереметьева И.Н., Немкова Г.А., Лазурченко Е.В. Хромосомные исследования полевки Максимовича Microtus maximowiczii Schrenk, 1858 в Норском заповеднике Амурской области и эворонской Microtus evoronensis Kovalsk. et Socolov, 1980 окрестностей озера Эворон Хабаровского края // Териофауна России и сопредельных территорий. М.: Тов-во науч. изд. КМК, 2007. С. 188.
Шереметьева И.Н., Картавцева И.В., Войта Л.Л., Тиунов M.П. Новые данные по распространению серых полевок рода Microtus (Rodentia: Cricetidae) на Дальнем Востоке России // Зоол. журн. 2010. Т. 89. № 10. С. 1273–1276.
Шереметьева И.Н., Картавцева И.В., Васильева Т.В., Фрисман Л.В. Серые полевки рода Alexandromys из Верхнебуреинской впадины // Зоол. журн. 2016. Т. 95. № 5. С. 597–603. https://doi.org/10.7868/S0044513416050111
Шереметьева И.Н., Картавцева И.В., Васильева Т.В. Обитает ли эворонская полевка (Alexandromys evoronensis) на северо-востоке Верхнезейской равнины? // Зоол. журн. 2017. Т. 96. № 4. С. 477–474. https://doi.org/10.7868/S0044513417020076
Aljanabi S.M., Martinez I. Universal and rapid salt extraction of high quality genomic DNA for PCR-based techniques // Nucl. Acids Res. 1997. V. 25. № 22. P. 4692–4693. https://doi.org/10.1093/nar/25.22.4692
Шереметьева И.Н., Картавцева И.В., Фрисман Л.В. и др. Полиморфизм и генетическая структура полевки Максимовича Microtus maximowiczii (Schrenck, 1858) (Rodentia, Cricetidae) Среднего Приамурья по данным секвенирования контрольного региона мтДНК // Генетика. 2015. Т. 51. № 10. С. 1154–1162. https://doi.org/10.7868/S0016675815100161
Hall T.A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98 // Nucl. Acids Symposium Series. 1999. V. 41. № 41. P. 95–98. https://doi.org/10.1021/bk-1999-0734.ch008
Kumar S., Stecher G., Li M. et al. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms // Mol. Biol. Evol. 2018. V. 35. № 6. P. 1547–1549. https://doi.org/10.1093/molbev/msy096
Bandelt H.J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C. et al. DnaSP 6: DNA sequence polymorphism Analysis of large datasets // Mol. Biol. Evol. 2017. V. 34. P. 3299–3302. https://doi.org/10.1093/molbev/msx248
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resources. 2010. № 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Tamura K., Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees // Mol. Biology and Evolution. 1993. V. 10. P. 512–526. https://doi.org/10.1093/oxfordjournals.molbev.a040023
Картавцева И.В., Васильева Т.В., Шереметьева И.Н. и др. Генетическая изменчивость трех изолированных популяций муйской полевки Alexandromys mujanensis Orlov et Kovalskaja, 1978 (Rodentia, Arvicolinae) // Генетика. 2019. Т. 55. № 8. С. 920–935. https://doi.org/10.1134/S0016675819080071
Шереметьева И.Н., Картавцева И.В., Павленко М.В. и др. Формирование генетической структуры популяций дальневосточной полевки Alexandromys fortis (Rodentia, Arvicolinae) северной периферии ареала // Зоол. журн. 2022. Т. 101. № 10. С. 1173–1189.
Дополнительные материалы отсутствуют.


