Генетика, 2023, T. 59, № 2, стр. 147-156
Структура и разнообразие ДНК-транспозонов Tc1/mariner в геноме ушастой медузы Aurelia aurita
Ю. Н. Улупова 1, Л. В. Пузакова 1, М. В. Пузаков 1, *
1 Федеральный исследовательский центр “Институт биологии южных морей
им. А.О. Ковалевского” Российской академии наук
299011 Севастополь, Россия
* E-mail: puzakov@ngs.ru
Поступила в редакцию 30.03.2022
После доработки 08.08.2022
Принята к публикации 30.08.2022
- EDN: KYLGLQ
- DOI: 10.31857/S0016675823020133
Аннотация
Мобильные генетические элементы – ДНК-транспозоны и ретротранспозоны – это последовательности ДНК, способные к перемещениям внутри генома. Предполагается, что они играют одну их ключевых ролей в адаптивных и эволюционных процессах. Одной из наиболее изученных групп ДНК-транспозонов является инфракласс ITm и, в частности, суперсемейство Tc1/mariner. В настоящей работе мы рассмотрели представленность, структуру и эволюцию ДНК-транспозонов Tc1/mariner ушастой медузы Aurelia aurita. Было установлено, что преобладающая доля элементов Tc1/mariner медузы представлена семейством TLE. Выявлено новое подсемейство элементов TLE, названное Aurum. Кроме того, в семействе Visitor обнаружены две группы элементов VS-aura и VS-beplau, которые, вероятно, также являются отдельными подсемействами. Анализ структуры и разнообразия элементов Tc1/mariner показал, что в настоящий момент транспозоны Tc1/mariner в геноме медузы находятся на стадии деградации и элиминации. Практически все элементы делетированы или имеют структурные изменения и соответственно не имеют потенциально функциональных копий.
Мобильные генетические элементы (МГЭ) – это последовательности ДНК, способные к самостоятельным перемещениям внутри генома. Впервые мобильные генетические элементы были описаны в 1940-х гг. Барбарой Макклинток [1]. В настоящее время известно, что они встречаются практически во всех организмах и суммарно составляют большой процент генома у высших э-укариот [2, 3]. Известно, что последовательности МГЭ могут быть основой для возникновения новых генов или регуляторных последовательностей [4]. Кроме того, МГЭ являются высокомутабильными структурами геномов организмов, а также вносят существенный вклад в спонтанный мутагенез, формируя хромосомные перестройки (делеции, инверсии, дупликации), тем самым вызывая как вредные, так и полезные мутации для организма и популяции в целом. Предполагается, что они играют одну их ключевых ролей в адаптивных и эволюционных процессах [5].
МГЭ эукариот можно разделить на два класса – ретротранспозоны и ДНК-транспозоны [4, 6]. Ретротранспозоны – элементы I класса, кодируют обратную транскриптазу и для своего перемещения используют в качестве посредника молекулу РНК. Такой механизм называется “копирование–вставка”. С помощью этого механизма ретротранспозонам удается быстро увеличивать число копий. ДНК-транспозоны – элементы II класса, кодируют транспозазу и перемещаются по геному без копирования путем вырезания собственной копии и ее последующей вставки в какой-либо другой участок генома. Такой механизм называется “вырезание–вставка” [4, 6].
Одной из наиболее изученных групп ДНК-транспозонов является ITm-элементы. На данный момент элементы ITm обнаружены у большинства живых организмов [7, 8]. В геномах можно обнаружить несколько сотен и даже тысяч копий этой группы [9–11]. Протяженность ITm-элементов составляет от 1 до 3 тыс. пар нуклеотидов. Транспозоны ITm имеют концевые инвертированные повторы (КИП), длина которых составляет от 20 до 1900 пн [12, 13]. У некоторых транспозонов есть и субконцевые инвертированные повторы (СИП), длиной от 175 до 1403 пн [13]. В центральной части транспозона находится, как правило, единая открытая рамка считывания (ОРС), кодирующая фермент транспозазу, длина которого варьирует от 350 до 650 а.о. Для транспозазы элементов ITm свойственно наличие ДНК-связывающего домена (PAIRED) и каталитического домена (DDE/D). ДНК-связывающий домен находится в N-концевой части транспозазы и состоит из шести α-спиралей. Первые три α-спирали получили название PAI-субдомен, вторые три α-спирали – RED-субдомен. Между этими доменами находится GRPR-подобный мотив, который обеспечивает связывание PAIRED с ДНК-мишенью [14]. Каталитический домен находится на С-концевой части транспозазы. DDE/D-домен обладает эндонуклеазной и лигирующей активностью, обеспечивая вырезание и вставку МГЭ. Также транспозаза ITm может включать NLS-мотив (сигнал ядерной локализации). NLS-мотив предположительно обеспечивает транспорт транспозазы из цитоплазмы в ядро [15, 16].
На данный момент ITm-транспозоны классифицируют как инфракласс, так как эта группа включает несколько суперсемейств: Tc1/mariner, pogo, IS630, Sailor, Gambol [8, 17, 18]. Суперсемейство Tc1/mariner включает в себя несколько семейств: Tc1-подобные элементы (TLE/DD34-46E), mariner-подобные элементы (MLE/DD34D), Visitor (DD41D), maT (DD37D), Guest (DD39D), mosquito (DD37E), L18 (DD37E), L31 (DD37E) [11, 13, 17, 19–21].
Здесь мы рассматриваем представленность, структуру и эволюцию ДНК-транспозонов ITm суперсемейства Tc1/mariner медузы Aurelia aurita. Ушастая медуза Aurelia aurita (также называемая обыкновенной медузой или лунной медузой) является разновидностью рода Aurelia (тип Cnidaria, класс Scyphozoa). Вид A. aurita встречается в Северном, Черном, Балтийском и Каспийском морях, северо-восточной Атлантике, Гренландии, северо-востоке США и Канаде, северо-западной части Тихого океана и Южной Америке [22, 23]. Ушастые медузы являются одним из важных компонентов многих экосистем, кроме того обладают удивительной способностью выдерживать значительные колебания температур и солености воды [24]. У медузы A. aurita, а также у других представителей Scyphozoa структура, эволюция и представленность элементов Tc1/mariner практически не изучены.
МАТЕРИАЛЫ И МЕТОДЫ
Поиск транспозонов
Для поиска ДНК-транспозонов суперсемейства Tc1/mariner был использован BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). В качестве матрицы использовались аминокислотные последовательности транспозаз Tc1/mariner, таких семейств как TLE (Passport (CAB51371), Mariner-5_CGi (Repbase), DrTRT [25], IN_Machilis_hrabei [26], TR-Habu [27], IT_Cf [28], MLE (Heteropoda venatoria (BAP05478), Mamestra brassicae (AAL69970), Adineta vaga (AAZ67099), Girardia tigrina (CAA50801), Bytmar1 (CAD45367), Bombyx mori (ABF51498), Tvmar1 (AAP45328)); Visitor (Crmar2.5 (AAK61417)), maT (Bmmar1 (U47917)), Guest (Soymar1 (AF078934)), mosquito (Ae-atropalpus1 (AF377999)). Полногеномные последовательности ДНК медузы A. aurita (GCA_004194415) были взяты из базы данных NCBI. Для того чтобы выяснить являются ли нуклеотидные последовательности МГЭ полными, мы брали гомологичные транспозазе последовательности с наивысшей идентичностью к матрице из соответствующих скаффолдов. У каждой извлеченной последовательности был совершен поиск инвертированных повторов (КИП и СИП) с помощью BLASTn [29]. Полная последовательность каждого нового обнаруженного элемента использовалась для уточнения границ и определения количества копий, присутствующих в геноме. Копии с протяженностью от 10 до 100% от длины полноразмерного МГЭ подсчитывались как общее количество копий. Копии менее 10% от длины полноразмерного МГЭ при подсчете не учитывались. Копии, кoторые составляли >95% от длины полноразмерного МГЭ, имели КИП и полную транспозазу (~300 а.о.), считались полноразмерными. Потенциально функциональными копиями мы считали такие элементы, у которых были интактная транспозаза (отсутствие стоп-кодонов и сдвигов ОРС), КИП, ДНК-связывающий домен (PAIRED), GRPR-мотив и каталитический домен (DDE/D).
Анализ последовательностей
Границы предполагаемых ОРС определяли с помощью ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/) и далее уточняли визуально. Последовательность сигнала ядерной локализации (NLS) выявляли с помощью ScanProsite [30]. ДНК-связывающий мотив PAIRED определяли, используя PSIPRED v3.3 [31]. Мотив GRPR-типа, а также DDE/D-домен идентифицировали визуально.
Филогенетический анализ
Для филогенетического анализа были взяты аминокислотные последовательности транспозаз, относящиеся к различным группам суперсемейства ITm-транспозонов (рис. 1). Множественное выравнивание аминокислотных последовательностей было выполнено с помощью MUSCLE [32] с использованием стандартных настроек. Поиск наилучшей модели для филогенетического анализа и филогенетический анализ проводились с использованием MEGA7.0 [33] по методу максимального правдоподобия.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
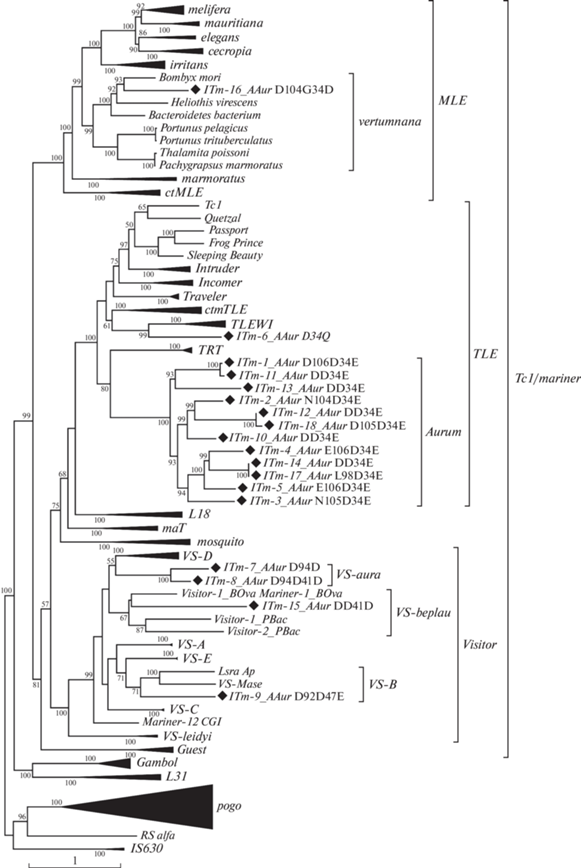
Проанализировав геном медузы A. aurita, нами было обнаружено 18 МГЭ (табл. 1). Для того чтобы соотнести к каким группам суперсемейства Tc1/mariner они относятся, мы провели филогенетический анализ. Для филогенетического анализа были использованы все известные семейства группы Tc1/mariner. В качестве внешней группы были задействованы аминокислотные последовательности элементов суперсемейств IS630 и pogo. В результате филогенетического анализа было установлено, что 13 элементов (ITm-1_AAur, ITm-2_AAur, ITm-3_AAur, ITm-4_AAur, ITm-5_AAur, ITm-6_AAur, ITm-10_AAur, ITm-11_AAur, ITm-12_AAur, ITm-13_AAur, ITm-14_AAur, ITm-17_AAur, ITm-18_AAur) входят в семейство TLE, четыре элемента (ITm-7_AAur, ITm-8_AAur, ITm-9_AAur, ITm-15_AAur) относятся к семейству Visitor и еще один элемент (ITm-16_AAur) принадлежит к семейству MLE. МГЭ, относящиеся к семействам maT, mosquito, Guest, L18, обнаружить не удалось (рис. 1). Отсутствие у ушастой медузы элементов L18 также было показано ранее [34].
Таблица 1.
ДНК-транспозоны суперсемейства Tc1/mariner в геноме медузы A. aurita
| Элемент | Семейство/ подсемейство |
Длина, пн | КИП, пн |
СИП, пн |
ТП, а.о. | Общее число копий | Число ПР |
Число ПФ |
Каталитический домен* |
|---|---|---|---|---|---|---|---|---|---|
| ITm-1_AAur | TLE/Aurum | 4322 | 90/90 | – | 359 | 9 | 1 | 1 | D106D34E, DNDPSQ |
| ITm-2_AAur | TLE/Aurum | 2152 | 26/26 | – | 358 | 31 | 5 | 2 | N104D34E, DGDPSQ |
| ITm-3_AAur | TLE/Aurum | 4614 | 229/230 | – | 370 | 415 | 1 | 0 | N105D34E, DGDPSQ |
| ITm-4_AAur | TLE/Aurum | 963 | – | – | 320 | 8 | 0 | 0 | E106D34E, DGDPSQ |
| ITm-5_AAur | TLE/Aurum | 960 | – | – | 319 | 9 | 0 | 0 | E106D34E, DGDPSQ |
| ITm-6_AAur | TLE | 267 | – | – | 89 | 1 | 0 | 0 | ?D34Q, DNNPKH |
| ITm-7_AAur | Visitor/VS-aura | 813 | – | – | 270 | 23 | 0 | 0 | D94D?, DGGSAH |
| ITm-8_AAur | Visitor/VS-aura | 1044 | – | – | 347 | 29 | 0 | 0 | D94D41D, DGAPAH |
| ITm-9_AAur | Visitor/VS-B | 699 | – | – | 232 | 1 | 0 | 0 | D92D47E, DGAPPH |
| ITm-10_AAur | TLE/Aurum | 381 | – | – | 127 | 6 | 0 | 0 | D92D47E, DGDPSQ |
| ITm-11_AAur | TLE/Aurum | 249 | – | – | 89 | 5 | 0 | 0 | ?D34E, DNDPSQ |
| ITm-12_AAur | TLE/Aurum | 249 | – | – | 84 | 8 | 0 | 0 | ?D34E, DGDPSQ |
| ITm-13_AAur | TLE/Aurum | 348 | – | – | 116 | 5 | 0 | 0 | ?D34E, DKDPSQ |
| ITm-14_AAur | TLE/Aurum | 321 | – | – | 106 | 6 | 0 | 0 | ?D34E, DGCPRQ |
| ITm-15_AAur | Visitor/VS-beplau | 1847 | 87/87 | – | 107 | 129 | 0 | 0 | ?D41D, DDAPVH |
| ITm-16_AAur | MLE/vertumnana | 1325 | 50/52 | – | 347 | 49 | 22 | 0 | D104G34D, GHASAH |
| ITm-17_AAur | TLE/Aurum | 3770 | 34/35 | 40/41 | 303 | 28 | 3 | 0 | L98D34E, DGCPRQ |
| ITm-18_AAur | TLE/Aurum | 732 | – | – | 244 | 5 | 0 | 0 | D105D34E, DGDPSQ |
Примечание. пн – пары нуклеотидов, а.о. – аминокислотные основания, КИП – концевые инвертированные повторы, СИП – субконцевые инвертированные повторы, ТП – транспозаза, ПР – полноразмерные копии, ПФ – потенциально-функциональные копии, * – указан паттерн DDE/D-домена, включающий маркерные аминокислотные основания и протяженность между ними в а.о., а также консервативную область из шести а.о. в районе второго маркерного аминокислотного основания, “?” – неидентифицированный элемент каталитического домена.
Visitor/DD41D
На данный момент элементы Visitor/DD41D классифицируют как отдельное семейство [11, 13]. Это семейство было разделено на шесть подсемейств (VS-A, VS-B, VS-C, VS-D, VS-E, Leidyi) [35]. В настоящем исследовании в геноме медузы мы обнаружили 4 МГЭ, относящихся к группе Visitor (табл. 1). Элементы ITm-15_AAur, Visitor-1_PBac, Visitor-2_PBac, Visitor-1_BOva сформировали группу с достаточно высоким значением бутстрепа (67%), названную нами VS-beplau. Элементы ITm-7_AAur, ITm-8_AAur сформировали группу (бутстреп 100%), которую мы назвали VS-aura. Можно предположить, что элементы ITm-7_AAur, ITm-8_AAur являются частью группы VS-D с низким значением бутстрепа (55%). Элемент ITm-9_AAur вошел в группу VS-B (рис. 1).
Для МГЭ семейства Visitor общая длина транспозонов составляет примерно 1300–4400 пн. Длина КИП варьирует от 15 до 1900 пн [11, 13, 36]. У ITm-8_AAur КИП обнаружить не удалось, следовательно элемент не способен к перемещениям, однако ОРС транспозазы ITm-8_AAur не имеет повреждений. Это может свидетельствовать о том, что транспозаза сохраняет свою ферментативную активность. У элемента ITm-15_AAur общий размер транспозона был типичен для этого семейства (1847 пн), а также были обнаружены КИП типичного размера (87 пн). Однако транспозаза элемента оказалась делетированной (107 а.о.). Элементы ITm-7_AAur, ITm-9_AAur представляют собой укороченные последовательности транспозазы без КИП (табл. 1).
У элементов ITm-7_AAur, ITm-8_AAur общее число копий составило от 23 до 29. Элемент ITm-9_AAur присутствует в виде единственной копии. ITm-15_AAur был представлен 129 копиями. При этом у четырех обнаруженных МГЭ полноразмерных копий не выявлено (табл. 1).
Как известно, потенциальную активность mariner-подобных транспозаз можно оценить по наличию ДНК-связывающего домена PAIRED, GRPR-мотива в головной части белка и каталитического домена (DDE/D) в хвостовой части. Также у некоторых mariner-подобных транспозаз определяется NLS – последовательность сигнала ядерной локализации.
Анализ транспозаз обнаруженных нами элементов показал, что PAI-субдомен присутствует только в транспозазах элементов ITm-7_AAur и ITm-8_AAur – у транспозазы элемента ITm-7_AAur в наличии только одна альфа-спираль, а у транспозазы элемента ITm-8_AAur две спирали вместо трех. Вторые три альфа-спирали RED-субдомена обнаружились у транспозазы элемента ITm-8_AAur, но третья альфа-спираль очень укорочена, что скорее всего свидетельствует о ее нефункциональности.
GRPR-мотив найден в транспозазах элементов ITm-7_AAur и ITm-8_AAur. Он расположен типично, в головной части белка, между PAI и RED субдоменами. NLS у элементов данного семейства не найден.
Каталитическому домену для сохранения своей ферментативной активности необходимо наличие всех трех маркерных аминокислотных остатков (двух аспартатов (D) и третьего глутамата (Е) или аспартата – DDE/D-домен), поэтому данная область обладает наибольшей консервативностью по сравнению с другими частями транспозазы [37] (рис. 1). В большинстве случаев для семейства Visitor характерен каталитический домен DD40-41D [38]. ITm-8_AAur имеет классический домен, свойственный транспозонам этой группы. У ITm-15_AAur сохранилась только вторая часть каталитической триады D41D. У двух других элементов – ITm-7_AAur и ITm-9_AAur в домене имелись видимые отличия, а именно у элемента ITm-7_AAur третий D (аспартат) в каталитической триаде отсутствовал, а у элемента ITm-9_AAur он был замещен на Е (глутамат), также количество аминокислотных остатков между аспартатом и глутаматом составило 47 вместо типичных 40–41 (рис. 2). Данные изменения в консервативном домене вероятно могут негативно повлиять на ферментативные свойства транспозазы.
Рис. 2.
Множественное выравнивание аминокислотных последовательностей транспозаз A. aurita. Серым выделены шесть α-спиралей, формирующих PAIRED-домен; жирным курсивом обозначен гипотетический сигнал ядерной локализации (NLS); выделен жирным и подчеркиванием GRPR-подобный мотив; черным показаны маркерные локусы каталитического домена.

Обобщая результаты анализа доменной структуры элементов Visitor ушастой медузы, мы предполагаем, что семейство Visitor представлено в геноме только нефункциональными копиями, поскольку каждый элемент имеет какое-либо видимое повреждение.
MLE/DD34D
MLE является хорошо изученным семейством группы Tc1/mariner [17]. Это семейство считается одной из основных групп ДНК-транспозонов и широко представлено во всех организмах. Транспозоны MLE особенно часто встречаются у высших животных, где они способны к горизонтальному переносу между видами [39]. Протяженность транспозонов MLE составляет около 1300 пн. Размеры КИП варьируют от 20 до 40 пн и транспозаза состоит из 340 а.о. [40]. В результате филогенетического анализа было установлено, что один из обнаруженных нами элементов (ITm-16_AAur) относится к этому семейству (рис. 1). Общая длина транспозона и самой транспозазы, а также КИП была типична для семейства MLE. ОРС элемента ITm-16_AAur содержит множество стоп-кодонов.
Семейство MLE имеет характерный каталитический домен DD34D [40]. У ITm-16_AAur каталитический домен претерпел изменения: в каталитической триаде второй D (аспартат) был замещен на (G) глицин (рис. 2).
ДНК-связывающий мотив у транспозазы элемента ITm-16_AAur также оказался неполноценен: из трех альфа-спиралей в PAI-субдомене присутствуют только две. GRPR-мотив и NLS не обнаружены.
В результате анализа, проведенного с помощью программы BLASTn, было обнаружено 49 копий элемента ITm-16_AAur. При этом количество полноразмерных копий составило 22 (табл. 1). Однако анализ доменной структуры транспозазы показал, что все обнаруженные элементы в настоящее время являются нефункциональными.
TLE/DD34–38E
Семейство TLE – также одно из самых распространенных семейств группы Tc1/mariner. Первый представитель семейства TLE, транспозон Tc1, имеющий активную транспозазу, был обнаружен в геноме нематоды Caenorhabditis elegans [41, 42]. Транспозоны этой группы также широко распространены в геномах позвоночных, включая телеостных рыб [43]. Семейство TLE включает элементы протяженностью 1200–2000 пн [25–28, 35, 43, 44]. Для транспозонов TLE характерен домен DD34E [45]. Тем не менее за последние годы были обнаружены элементы TLE с нехарактерным доменом для этого семейства. Таким образом это семейство разделилось на несколько подсемейств: TRT/DD37E [25], Incomer/DD36E [26], Intruder/DD38E [28], TLEWI/DD36E [44], Traveler/DD35E [27] и ctmTLE/DD34E [35].
В геноме A. aurita мы обнаружили 13 элементов, относящихся к группе TLE (табл. 1). В результате филогенетического анализа 12 элементов сформировали отдельную группу Aurum с высоким значением бутстрепа (100%) (рис. 1). На филогенетическом дереве видно, что элементы группы Aurum близко расположены к подсемейству TRT. Таким образом можно предположить, что отделившаяся группа Aurum представляет новое подсемейство TLE. Здесь же следует отметить, что элемент ITm-6_AAur не вошел в эту группу, а также ни в одно известное подсемейство TLE. Однако на основании проведенного филогенетического анализа можно сказать, что он также относится к семейству TLE (рис. 1).
Анализ числа копий транспозонов, относящихся к семейству TLE, показал, что у десяти элементов (ITm-1_AAur, ITm-4_AAur, ITm-5_AAur, ITm-6_AAur, ITm-10_AAur, ITm-11_AAur, ITm-12_AAur, ITm-13_AAur, ITm-14_AAur, ITm-18_AAur) низкое количество копий – от 1 до 9. Два элемента (ITm-17_AAur, ITm-2_AAur) имеют от 28 до 31 копии. И только один элемент (ITm-3_AAur) имеет очень высокое (415) число копий (табл. 1). Из 13 МГЭ этой группы полноразмерные копии обнаружены только у четырех элементов (ITm-1_AAur, ITm-2_AAur, ITm-3_AAur, ITm-17_AAur). У транспозонов ITm-1_AAur, ITm-3_AAur полноразмерные копии присутствовали в единственном числе. У ITm-2_AAur и ITm-17_AAur обнаружено пять и три полноразмерных копий соответственно.
Длина полноразмерных транспозонов группы Aurum варьировала от 2152 до 4614 пн, тогда как характерная длина транспозонов для семейства TLE составляет 1200–2000 пн. Увеличение длины могло произойти из-за вставок в некодирующую последовательность. Длина полноразмерных транспозаз варьировала от 303–370 а.о., что является типичным для транспозаз этого семейства. Также у найденных нами полноразмерных МГЭ были обнаружены КИП, длина которых колебалась от 26 до 230 пн, что также характерно для семейства TLE. У ITm-17_AAur были обнаружены СИП, длина которых составила 40/41 пн. Однако у остальных полноразмерных транспозонов СИП обнаружены не были. У других элементов (ITm-4_AAur, ITm-5_AAur, ITm-6_AAur, ITm-10_AAur, ITm-11_AAur, ITm-12_AAur, ITm-13_AAur, ITm-14_AAur, ITm-18_AAur) длина транспозона оказалась значительно ниже, чем у TLE, и составила от 249 до 963 пн. Длина транспозазы варьировала от 84 до 320 а.о. КИП обнаружены не были (табл. 1).
Необходимо добавить, что при сравнении обнаруженных консервативных доменов с характерным паттерном для семейства TLE (DD34E) нами были выявлены некоторые различия. Два элемента (ITm-1_AAur, ITm-18_AAur) имели классический домен DD34E. У других десяти элементов были отличия в каталитической триаде, а именно у элементов ITm-2_AAur, ITm-3_AAur первый аспартат (D) заменился на аспарагин (N), у ITm-4_AAur, ITm-5_AAur первый аспартат (D) – на глутамат (E), а у ITm-17_AAur первый аспартат (D) – на лейцин (L). У элементов ITm-11_AAur, ITm-12_AAur, ITm-13_AAur, ITm-14_AAur первый аспартат (D) отсутствовал, зато вторая часть каталитической триады была сохранена D34E. Также следует отметить, что у элемента ITm-10_AAur в каталитическом домене DD47E наблюдались различия в протяженности между вторым аспартатом (D) и третьим глутаматом (E) (рис. 2). Однако на филогенетическом дереве видно, что этот транспозон все же относится к семейству TLE (рис. 1). При сравнении каталитического домена у транспозона ITm-6_AAur с классическим доменом семейства TLE (DD34E) были выявлены отличия, а именно в каталитической триаде первый аспартат (D) отсутствовал, а глутамат (E) был замещен на глутамин (Q) (рис. 2). В результате филогенетического анализа выявлено, что транспозон ITm-6_AAur относится к семейству TLE.
Анализ транспозаз обнаруженных нами элементов показал, что первые три альфа-спирали PAI-субдомена присутствуют только в транспозазах элементов ITm-1_AAur, ITm-2_AAur и ITm-3_AAur, у транспозаз остальных элементов они укорочены или отсутствуют. Вторые три альфа-спирали RED-субдомена обнаружились у транспозаз элементов ITm-1_AAur, ITm-2_AAur, ITm-4_AAur, ITm-5_AAur и ITm-17_AAur (рис. 2). Однако у элементов ITm-1_AAur и ITm-5_AAur альфа-спирали фрагментированы, что может свидетельствовать о их нефункциональности.
GRPR-мотив присутствует в транспозазах элементов ITm-1_AAur, ITm-2_AAur, ITm-3_AAur, ITm-4_AAur, ITm-5_AAur и ITm-17_AAur.
NLS у элементов ITm-4_AAur и ITm-17_AAur локализован в двух различных районах транспозазы. В транспозазах элементов ITm-1_AAur и ITm-5_AAur найдено по одной последовательности NLS, однако только у элемента ITm-1_AAur NLS расположен типично, между ДНК-связывающим и каталитическим доменами. Таким образом, потенциально функциональным условно можно считать только элемент ITm-1_AAur, поскольку он имеет черты функционального элемента (неповрежденную ОРС, КИП, GRPR-мотив, классический каталитический домен DD34E и NLS), однако из-за фрагментации одной альфа-спирали в ДНК-связывающем мотиве он может быть нефункционален, хотя насколько это сказывается на его активности не известно. Элемент ITm-2_AAur, вероятнее всего, также нефункционален, так как наличие мутации первого аминокислотного остатка в каталитическом домене, который является наиболее консервативной областью, может нарушать работу транспозазы.
Преобладающая доля элементов Tc1/mariner медузы представлена элементами семейства TLE. В этой группе обнаружено новое подсемейство, названное Aurum. В семействе Visitor обнаружены две группы элементов VS-aura и VS-beplau, которые, возможно, являются новыми подсемействами. Анализ элементов Tc1/mariner в геноме медузы показал, что в настоящий момент изучаемые транспозоны находятся на стадии деградации и элиминации. Практически все элементы делетированы или имеют структурные изменения и соответственно не имеют потенциально функциональных копий. Элемент ITm-3_AAur был, по всей видимости, очень активен в прошлом (415 копий), возможно из-за особенностей его структуры или по другим причинам, однако на момент исследования этот элемент так же, как и другие, находится на завершающей стадии жизненного цикла.
Работа проведена в рамках Государственного задания ФГБУН ИМБИ “Функциональные, метаболические и токсикологические аспекты существования гидробионтов и их популяций в биотопах с различным физико-химическим режимом”, номер гос. регистрации 121041400077-1.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
McClintock B. Chromosome organization and genetic expression // Cold Spring Harbor Symp. Quant. Biol. 1951. V. 16. P. 13–47. https://doi.org/10.1101/sqb.1951.016.01.004
Guo B., Zou M., Gan X., He S. Genome size evolution in pufferfish: an insight from BAC clone-based Diodon holocanthus genome sequencing // BMC Genomics. 2010. V. 11. P. 396. https://doi.org/10.1186/1471-2164-11-396
Feschotte C., Pritham E.J. DNA transposons and the evolution of eukaryotic genomes // Annu. Rev. Genet. 2007. V. 41. P. 331–368. https://doi.org/10.1146/annurev.genet.40.110405.090448
Bourque G., Burns K.H., Gehring M. et al. Ten things you should know about transposable elements // Genome Biology. 2018. V. 19. № 1. P. 199. https://doi.org/10.1186/s13059-018-1577-z
Piacentini L., Fanti L., Specchia V. et al. Transposons, environmental changes, and heritable induced phenotypic variability // Chromosoma. 2014. V. 123. № 4. P. 345–354. https://doi.org/10.1007/s00412-014-0464-y
Kojima K.K. Structural and sequence diversity of e-ukaryotic transposable elements // Genes Genet. Syst. 2020. V. 94. P. 233–252. Epub. 2018. Nov. 9.https://doi.org/10.1266/ggs.18-00024
Dupeyron M., Baril T., Bass C., Hayward A. Phylogenetic analysis of the Tc1/mariner superfamily reveals the unexplored diversity of pogo-like elements // Mobile DNA. 2020. V. 11. P. 21. https://doi.org/10.1186/s13100-020-00212-0
Gao B., Wang Y., Diaby M. et al. Evolution of pogo, a separate superfamily of IS630-Tc1-mariner ransposons, revealing recurrent domestication events in vertebrates // Mobile DNA. 2020. V. 11. P. 25. https://doi.org/10.1186/s13100-020-00220-0
Lee C.C., Wang J. Rapid expansion of a highly germline-expressed Mariner element acquired by horizontal transfer in the fire ant genome // Genome Biol. Evol. 2018. V. 10. № 12. P. 3262–3278. https://doi.org/10.1093/gbe/evy220
Xie L.Q., Wang P.L., Jiang S.H. et al. Genome-wide identification and evolution of TC1/Mariner in the silkworm (Bombyx mori) genome // Genes Genomics. 2018. V. 40. № 5. P. 485–495. https://doi.org/10.1007/s13258-018-0648-6
Shen D., Gao B., Miskey C. et al. Multiple invasions of visitor, a DD41D family of Tc1/mariner transposons, throughout the evolution of vertebrates // Genome Biol. Evol. 2020. V. 12. № 7. P. 1060–1073. https://doi.org/10.1093/gbe/evaa135
Claudianos C., Brownlie J., Russell R. et al. maT – a clade of transposons intermediate between mariner and Tc1 // Mol. Biol. Evol. 2002. V. 19. № 12. P. 2101–2109. https://doi.org/10.1093/oxfordjournals.molbev.a004035
Zhang H.H., Shen Y.H., Xiong X.M. et al. Identification and evolutionary history of the DD41D transposons in insects // Genes Genomics. 2016. V. 38. P. 109–117. https://doi.org/10.1007/s13258-015-0356-4
Ivics Z., Izsvák Z. Sleeping Beauty transposition // Microbiol. Spectrum. 2015. V. 3. № 2. MDNA3-0042-2-14. https://doi.org/10.1128/microbiolspec.MDNA3-0042-2014
Ivics Z., Hackett P.B., Plasterk R.H., Izsvák Z. Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells // Cell. 1997. V. 91. № 4. P. 501–510. https://doi.org/10.1016/s0092-8674(00)80436-5
Plasterk R.H., Izsvák Z., Ivics Z. Resident aliens: the Tc1/mariner superfamily of transposable elements // Trends in Genet. 1999. V. 15. № 8. P. 326–332. https://doi.org/10.1016/s0168-9525(99)01777-1
Tellier M., Bouuaert C.C., Chalmers R. Mariner and the ITm superfamily of transposons // Microbiol. Spectrum. 2015. V 3. № 2. MDNA3-0033-2014. https://doi.org/10.1128/microbiolspec.MDNA3-0033-2014
Shi S., Puzakov M., Guan Z. et al. Prokaryotic and eukaryotic horizontal transfer of Sailor (DD82E), a new superfamily of IS630-Tc1-mariner DNA transposons // Biology (Basel). 2021. V. 10. № 10. P. 1005. https://doi.org/10.3390/biology10101005
Wang S., Diaby M., Puzakov M. et al. Divergent evolution profiles of DD37D and DD39D families of Tc1/mariner transposons in eukaryotes // Mol. Phylogenet. Evol. 2021. V. 161. P. 107143. https://doi.org/10.1016/j.ympev.2021.107143
Shao H., Tu Z. Expanding the diversity of the IS630-Tc1-mariner superfamily: Discovery of a unique DD37E transposon and reclassification of the DD37D and DD39D transposons // Genetics. 2001. V. 159. № 3. P. 1103–1115. https://doi.org/10.1093/genetics/159.3.1103
Puzakov M.V., Puzakova L.V., Cheresiz S.V. An analysis of IS630/Tc1/mariner transposons in the genome of a Pacific oyster, Crassostrea gigas // J. Mol. Evol. 2018. V. 86. № 8. P. 566–580. https://doi.org/10.1007/s00239-018-9868-2
Lawley J.W., Gamero-Mora E., Maronna M.M. et al. The importance of molecular characters when morphological variability hinders diagnosability: Systematics of the moon jellyfish genus Aurelia (Cnidaria: Scyphozoa) // Peer J. 2021. V. 9. e11954. https://doi.org/10.7717/peerj.11954
Dawson M.N., Sen Gupta A., England M.H. Coupled biophysical global ocean model and molecular genetic analyses identify multiple introductions of cryptogenic species // Proc. Natl Acad. Sci. USA. 2005. V. 102. P. 11968–11973. https://doi.org/10.1073/pnas.0503811102
Луппова Н.Е. Динамика численности и биомассы популяций черноморского макрозоопланктона // Бюл. науки и практики. 2020. Т. 6. № 5. С. 74–82. https://doi.org/10.33619/2414-2948/54/09
Zhang H.H., Li G.Y., Xiong X.M. et al. TRT, a vertebrate and protozoan Tc1-like transposon: Current activity and horizontal transfer // Genome Biol. Evol. 2016. V. 8. № 9. P. 2994–3005. https://doi.org/10.1093/gbe/evw213
Sang Y., Gao B., Diaby M. et al. Incomer, a DD36E family of Tc1/mariner transposons newly discovered in animals // Mobile DNA. 2019. V. 10. P. 45. https://doi.org/10.1186/s13100-019-0188-x
Zong W., Gao B., Diaby M. et al. Traveler, a new DD35E family of Tc1/mariner transposons, invaded vertebrates very recently // Genome Biol. Evol. 2020. V. 12. № 3. P. 66–76. https://doi.org/10.1093/gbe/evaa034
Gao B., Zong W., Miskey C. et al. Intruder (DD38E), a recently evolved sibling family of DD34E/Tc1 transposons in animals // Mobile DNA. 2020. V. 11. № 1. P. 32. https://doi.org/10.1186/s13100-020-00227-7
Zhang Z., Schwartz S., Wagner L.W. et al. A greedy algorithm for aligning DNA sequences // J. Computational Biol. 2000. V. 7. № 1–2. P. 203–214. https://doi.org/10.1089/10665270050081478
De Castro E., Sigrist C.J., Gattiker A. et al. ScanProsite: Detection of PROSITE signature matches and ProRule-associated functional and structural residues in proteins // Nucl. Acids Res. 2006. V. 34. P. W362–W365. https://doi.org/10.1093/nar/gkl124
Buchan D.W., Minneci F., Nugent T.C. et al. Scalable web services for the PSIPRED Protein Analysis Workbench // Nucl. Acids Res. 2013. V. 41 (W1). P. W349–W357. https://doi.org/10.1093/nar/gkt381
Edgar R.C. MUSCLE: A multiple sequence alignment method with reduced time and space complexity // Nucl. Acids Res. 2004. V. 32. P. 1792–1797. https://doi.org/10.1093/nar/gkh340
Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. № 7. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Пузаков М.В., Пузакова Л.В. Распространенность, разнообразие и эволюция ДНК-транспозонов L18 (DD37E) в геномах стрекающих (Cnidaria) // Мол. биология. 2022. Т. 56. № 3. С. 1–15 https://doi.org/10.31857/S0026898422030120
Puzakov M.V., Puzakova L.V., Cheresiz S.V., Sang Y. The IS630/Tc1/mariner transposons in three ctenophore genomes // Mol. Phylogenet. Evol. 2021. V. 163. P. 107231. https://doi.org/10.1016/j.ympev.2021.107231
Bouallègue M., Filée J., Kharrat I. et al. Diversity and evolution of mariner-like elements in aphid genomes // BMC Genomics. 2017. V. 18. № 1. P. 494. https://doi.org/10.1186/s12864-017-3856-6
Benjamin B., Yves B., Corinne A.G. Assembly of the Tc1 and mariner transposition initiation complexes depends on the origins of their transposase DNA binding domains // Genetica. 2007. V. 130. № 2. P. 105–120. https://doi.org/10.1007/s10709-006-0025-2
Gomulski L.M., Torti C., Bonizzoni M. et al. A new basal subfamily of mariner elements in Ceratitis rosa and other tephritid flies // J. Mol. Evol. 2001. V. 53. № 6. P. 597–606. https://doi.org/10.1007/s002390010246
Robertson H.M., Lampe D.J. Distribution of transposable elements in arthropods // Annual Rev. Entomol. 1995. V. 40. P. 333–357. https://doi.org/10.1146/annurev.en.40.010195.002001
Jacobson J.W., Medhora M.M., Hartl D.L. Molecular structure of a somatically unstable transposable element in Drosophila // Proc. Natl Acad. Sci. USA. 1986. V. 83. № 22. P. 8684–8688. https://doi.org/10.1073/pnas.83.22.8684
Emmons S.W., Yesner L. High-frequency excision of transposable element Tc1 in the nematode Caenorhabditis elegans is limited to somatic cells // Cell. 1984. V. 36. № 3. P. 599–605. https://doi.org/10.1016/0092-8674(84)90339-8
Eide D., Anderson P. Transposition of Tc1 in the nematode Caenorhabditis elegans // Proc. Natl Acad. Sci. USA. 1985. V. 82. № 6. P. 1756–1760. https://doi.org/10.1073/pnas.82.6.1756
Guo X.M., Zhang Q.Q., Sun Y.W. et al. Tc1-like transposase Thm3 of silver carp (Hypophthalmichthys molitrix) can mediate gene transposition in the genome of blunt snout bream (Megalobrama amblycephala) // G3: Genes, Genomes, Genetics. 2015. V. 5. № 12. P. 2601–2610. https://doi.org/10.1534/g3.115.020933
Puzakov M.V., Puzakova L.V., Cheresiz S.V. The Tc1-like elements with the spliceosomal introns in mollusk genomes // Mol. Genet. Genomics. 2020. V. 295. № 3. P. 621–633. https://doi.org/10.1007/s00438-020-01645-1
Capy P., Vitalis R., Langin T. et al. Relationships between transposable elements based upon the integrase-transposase domains: is there a common ancestor? // J. Mol. Evol. 1996. V. 42. № 3. P. 359–368. https://doi.org/10.1007/BF02337546
Дополнительные материалы отсутствуют.