Генетика, 2023, T. 59, № 5, стр. 544-561
Предполагаемый локус, вовлеченный в изменчивость размеров черепа лисиц (Vulpes vulpes)
А. В. Харламова 1, *, С. Г. Шихевич 1, А. В. Владимирова 1, А. В. Кукекова 2, В. М. Ефимов 1, 3, 4
1 Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
2 Университет штата Иллинойс в Урбана-Шампейн
61801 Иллинойс, США
3 Новосибирский государственный университет
630090 Новосибирск, Россия
4 Томский государственный университет
634050 Томск, Россия
* E-mail: kharlam@bionet.nsc.ru
Поступила в редакцию 21.11.2022
После доработки 24.11.2022
Принята к публикации 29.11.2022
- EDN: FNUESY
- DOI: 10.31857/S0016675823050089
Аннотация
Представлены результаты анализа промеров черепа лисиц (Vulpes vulpes) из популяций, прошедших длительный отбор на дружелюбное (ручное, или доместикационное) и агрессивное поведение по отношению к человеку, а также не подвергавшихся направленному отбору по свойствам поведения. Краниометрические показатели сняты с двух выборок лисиц: 1) со 140 лисиц, потомков возвратного скрещивания гибридов первого поколения между представителями дружелюбной и агрессивной линий (F1), c представителями дружелюбной линии, 2) с объединенной выборки из 50 дружелюбных, 50 агрессивных и 50 неселекционированных по поведению (контрольных) особей. Потомки возвратного скрещивания ранее были генотипированы по 350 микросателлитным маркерам. Эти микросателлиты были использованы в настоящей работе для проведения совместного статистического анализа методом 2B-PLS генетических и краниометрических данных лисиц из первой выборки. Выявлен регион на 10-й хромосоме лисицы, маркированный тремя микросателлитами (FH2535, RVC1, REN193M22), контролирующий изменчивость размеров черепа. Вторая выборка была генотипирована только по трем маркерам, которые показали ассоциацию с размерами черепа у потомков возвратного скрещивания. Аналогичный анализ методом 2B-PLS этой выборки подтвердил участие региона на 10-й хромосоме лисицы в контроле размеров черепа, причем эта связь оказалась более выраженной у самцов, чем у самок. В идентифицированном геномном участке расположен ген IGF1, который обусловливает 15% изменчивости размеров тела у собак. Полученные в настоящей работе результаты дают основание предполагать участие гена IGF1 в контроле размеров черепа лисиц.
Процесс доместикации характеризуется комплексными генетическими преобразованиями животных [1–3]. Одомашненные животные, как правило, отличаются от диких видов-предшественников пониженной агрессивностью по отношению к человеку и устойчивостью к стрессирующим факторам [4]. Хотя изменения в поведении являются ключевыми характеристиками доместикации, одомашненные животные также отличаются от диких предков и по другим признакам, в частности морфологическим параметрам [5]. Видом с наиболее ярким морфологическим разнообразием является домашняя собака (Canis familiaris). Доместикация собаки от волка началась не менее 15 тыс. лет назад [6–9]. Первые признаки использования собак, в соответствии с их морфологическими характеристиками, были обнаружены при анализе черепов собак, живших около 9 тыс. лет назад [10]. В популяции собак на о-ве Жохов прослеживается четкое разделение между особями среднего размера, которых использовали как ездовых собак, и крупными собаками, которых использовали для охоты. Хотя основные морфологические типы уже обнаруживаются у собак, живших несколько тысяч лет назад [11–16], наиболее интенсивная селекция собак по морфологическим признакам началась с образованием пород. Регистрация стандартов пород собак в XIX в. и последующее их разведение как изолированных генетических линий способствовали увеличению морфологических различий. Например, чихуахуа и дог различаются по весу более чем в 20 раз. Породное разведение также привело к значительным различиям в пропорциях черепа: брахицефальные породы собак характеризуются большой черепной коробкой и короткой мордой, а долихоцефальные породы – узким и удлиненным черепом. В настоящее время зарегистрировано около 400 пород собак, стандарт каждой из которых описывает породные характеристики поведения и морфологии.
Многообразие пород создало беспрецедентную возможность для изучения генетики морфологических признаков собак [17, 18]. В отличие от генетики человека, где такие количественные признаки, как рост, регулируются малыми вкладами большого числа генов [19], исследования морфологических признаков собак и сельскохозяйственных животных показывают, что количественные признаки у одомашненных животных могут контролироваться небольшим числом генов, имеющих значительный эффект [20–23]. Так, у собак около 65% вариации в размере тела контролируются шестью генами: GHR, HMGA2, IGF1, IGF1R, SMAD2 и STC2 [24]. Генетический анализ размеров и формы черепа собак также обнаружил небольшое число локусов, вносящих значительный вклад в формирование межпородных различий [25–28]. Относительно простая генетическая архитектура морфологических признаков собак объясняется историей породного разведения. Хотя исследования на исторически одомашненных видах внесли фундаментальный вклад в изучение генетического регулирования признаков поведения и морфологии [29, 30], длительный искусственный отбор, который прошли все современные домашние животные, создает трудности в поиске генов, вклад которых был важен на ранних этапах одомашнивания. Гены, влияющие на генетическое преобразование животных на начальных этапах одомашнивания, можно попытаться найти в анализе экспериментально одомашненных животных.
Эксперимент по одомашниванию лисицы, проводимый более 60 лет в новосибирском Институте цитологии и генетики (ИЦиГ СО РАН), привел к созданию популяции ручных лисиц, напоминающих по поведению домашнюю собаку [3]. В отличие от исторически одомашненных животных, история которых включает в себя селекцию по самым разнообразным признакам, экспериментальная доместикация лисиц велась путем интенсивного отбора по единственному признаку – на дружелюбное отношение к человеку. Кроме того, в ИЦиГ СО РАН более сорока лет ведется отбор совхозных лисиц на агрессивное поведение по отношению к человеку [3, 31]. Агрессивная популяция характеризуется более негативным отношением к человеку, чем совхозные лисицы, разводимые в клеточных условиях, но не подвергающиеся прямому отбору по поведению. Экспериментальная популяция дружелюбных лисиц может быть использована как аналог ранних этапов одомашнивания при сравнении ее с совхозными лисицами. Как и исторически одомашненные виды, лисицы ИЦиГ СО РАН демонстрируют многие признаки, связанные с изменением поведения [3, 31]. Интересным представляется исследование связи отбора с изменением пропорций черепа, поскольку показано, что краниальные признаки в процессе доместикации млекопитающих претерпевают значительные изменения [32, 33]. Ранее у дружелюбных лисиц были показаны изменения пропорций черепа [34, 35], а также увеличение объема серого вещества по сравнению с совхозными неселекционируемыми по поведению лисицами [36].
В настоящей работе мы провели генетический анализ краниометрических данных дружелюбных, агрессивных и неселекционированных по поведению (контрольных) лисиц. Первый этап анализа был проведен с использованием беккроссов, являющихся потомками возвратного скрещивания гибридов первого поколения между представителями дружелюбной и агрессивной линий (F1), c представителями дружелюбной линии. Поскольку ранее эти животные были генотипированы по 350 микросателлитным маркерам равномерно распределенными по геному лисицы, а также протестированы по их реакции на человека [37], мы поставили задачу провести поиск участков генома лисицы, вовлеченных в вариацию краниологических признаков, и попытаться выявить локусы, общие для вариации этих признаков и поведения. В результате генетического анализа были обнаружены участки в геноме лисицы, показавшие связь с краниометрическими признаками независимо от поведения. Один из обнаруженных локусов содержит ген IGF1, который отвечает за ~15% вариации в размере собак [26]. Анализ данного локуса в независимых выборках из ручной, агрессивной и совхозной популяций подтвердил связь выявленного участка генома с краниометрическими признаками лисиц.
МАТЕРИАЛЫ И МЕТОДЫ
Экспериментальные животные
В исследовании использованы лисицы из экспериментальных популяций новосибирского ИЦиГ СО РАН, прошедшие многолетний отбор по свойствам поведения – на доместикационное (доместицируемые, или ручные лисицы) и агрессивное поведение по отношению к человеку, а также лисицы ИЦиГ СО РАН, не подвергающиеся специальному отбору по свойствам поведения. На первом этапе в анализ были включены 140 потомков возвратного скрещивания гибридов первого поколения (F1) между доместицируемыми и агрессивными особями с представителями исходной популяции доместицируемых лисиц. Возраст животных на момент исследования составлял 12 мес.
На втором этапе использована вторая выборка – из ручных, агрессивных и не подвергавшихся отбору лисиц ИЦиГ СО РАН (по 50 животных из каждой популяции, в равном количестве самцов и самок, всего 150 животных). На данном этапе исследования животные из трех указанных популяций были объединены вместе. В анализ включали объединенную группу, состоящую из ручных, агрессивных и неселекционируемых по поведению лисиц. Возраст животных на момент исследования – 8 мес. В этом возрасте молодые лисицы близки по размеру и пропорциям тела к взрослым особям.
Правила работы с животными в ИЦиГ СО РАН соответствуют международным нормам охраны животных, используемых в научных целях (Директива 2010/63/EU Европейского Парламента и Совета Европейского Союза). Условия содержания и ухода идентичны для всех лисиц, содержащихся на экспериментальной ферме ИЦиГ СО РАН.
Фенотипирование по краниометрическим признакам
Измерение краниальных признаков лисиц-потомков возвратного скрещивания (n = 140) проводили на цифровых рентгенограммах с использованием программы Looker (разработчик – Институт прикладной физики СО РАН, Новосибирск). Для этого животных рентгеноскопировали в трех проекциях (фронтальной, левой и правой) в возрасте одного года. Каждое животное было охарактеризовано по 21 метрическому признаку (список промеров приведен в табл. 1). Схема измерений была опубликована ранее [38]. Для минимизации возможных ошибок все измерения были проведены дважды одним исследователем. Для каждого из промеров были вычислены среднегрупповые значения и среднеквадратическое отклонение (сигма). После этого искали значения, уклоняющиеся от среднегруппового за пределы трех сигм. Такие измерения считались выполненными некорректно, или “выбросами”, и были исключены. Такие промеры были повторены третий раз. В анализ всегда включали среднее значение из двух корректных измерений.
Таблица 1.
Корреляции морфологических признаков с первой парой бикомпонент (генетической и морфологической). 2B-PLS-анализ потомков лисиц от возвратного скрещивания гибридов первого поколения между представителями дружелюбной и агрессивной линий (F1) c представителями дружелюбной линии
| Признак | Генетическая ось_01 | Морфологическая ось_01 | Генетическая ось_02 | Морфологическая ось_02 |
|---|---|---|---|---|
| Длина черепа (фронт.) | 0.663 | 0.927 | –0.149 | –0.236 |
| Длина черепа (лев.) | 0.591 | 0.910 | –0.156 | –0.252 |
| Длина черепа (прав.) | 0.655 | 0.909 | –0.168 | –0.275 |
| Скуловая ширина | 0.685 | 0.879 | 0.063 | 0.097 |
| Ширина атланта | 0.619 | 0.865 | –0.016 | –0.125 |
| Длина нижней челюсти (прав.) | 0.560 | 0.847 | –0.192 | –0.311 |
| Ширина моляр | 0.560 | 0.813 | –0.025 | 0.004 |
| Ширина нижней челюсти (лев.) | 0.613 | 0.805 | 0.087 | 0.059 |
| Длина носовой кости | 0.480 | 0.790 | –0.275 | –0.391 |
| Ширина сосцевидного отростка | 0.559 | 0.775 | 0.158 | 0.131 |
| Ширина мозговой капсулы | 0.521 | 0.675 | 0.033 | 0.129 |
| Длина шеи | 0.460 | 0.640 | 0.068 | 0.012 |
| Высота носовой кости (прав.) | 0.402 | 0.549 | 0.06 | 0.095 |
| Высота мозговой капсулы | 0.330 | 0.465 | 0.454 | 0.629 |
| Высота носовой кости (лев.) | 0.319 | 0.464 | 0.024 | 0.072 |
| Ширина между клыками | 0.215 | 0.402 | 0.112 | 0.168 |
| Длина нижней челюсти (лев.) | 0.236 | 0.321 | –0.204 | –0.331 |
| Высота нижней челюсти (прав.) | 0.232 | 0.300 | 0.331 | 0.379 |
| Высота мозговой капсулы без слуховых барабанов | 0.151 | 0.219 | 0.415 | 0.59 |
| Ширина плеч | 0.138 | 0.184 | 0.311 | 0.356 |
| Высота нижней челюсти (лев.) | –0.015 | 0.005 | 0.188 | 0.362 |
Краниометрические признаки животных из второй выборки (исходных родительских популяций ручных, агрессивных и неселекционированных лисиц) были измерены на очищенных черепах, полученных путем вываривания после планового забоя. Измерения сделаны с использованием электронного цифрового штангенциркуля (цена деления 0.01 мм). Каждое животное охарактеризовано 23 краниометрическими признаками. Один и тот же исследователь проводил измерения дважды. Как и в первой выборке, значения, уклоняющиеся от среднегруппового за пределы трех сигм, считались некорректными, исключались и измерялись заново, как описано выше для беккроссов. В анализ всегда включали среднее значение из двух корректных измерений. Перечень промеров и схема измерения из второй выборки приведены на рис. 1. Средние значения промеров для самцов и самок приведены в табл. 2.
Рис. 1.
Схема промеров черепа лисиц из выборки ручных, агрессивных и неселекционированных по поведению. а – фронтальная проекция, вид сверху; б – фронтальная проекция, вид снизу; в – боковая проекция. 1 – общая длина черепа; 2 – ширина на уровне орбит; 3 – скуловая ширина; 4 – длина лицевой части черепа; 5 – базальная длина черепа; 6 – ширина на уровне клыков; 7 – ширина на уровне премоляр; 8 – ширина на уровне моляр; 9 – длина нёбной кости; 10 – ширина мозгового черепа; 11 – ширина на уровне сосцевидного отростка (мастоида); 12 – высота мозговой капсулы без слуховых барабанов; 13 – максимальная высота мозговой капсулы; 14 – заглазничная ширина; 15 – межглазничная ширина; 16 – высота нижней челюсти (лев.); 17 – длина нижней челюсти (лев.); 18 – высота нижней челюсти (прав.); 19 – длина нижней челюсти (прав.); 20 – нёбный отросток преднёбной верхней челюсти; 21 – длина верхнего зубного ряда (прав.); 22 – длина верхнего зубного ряда (лев.); 23 – длина твердого нёба.
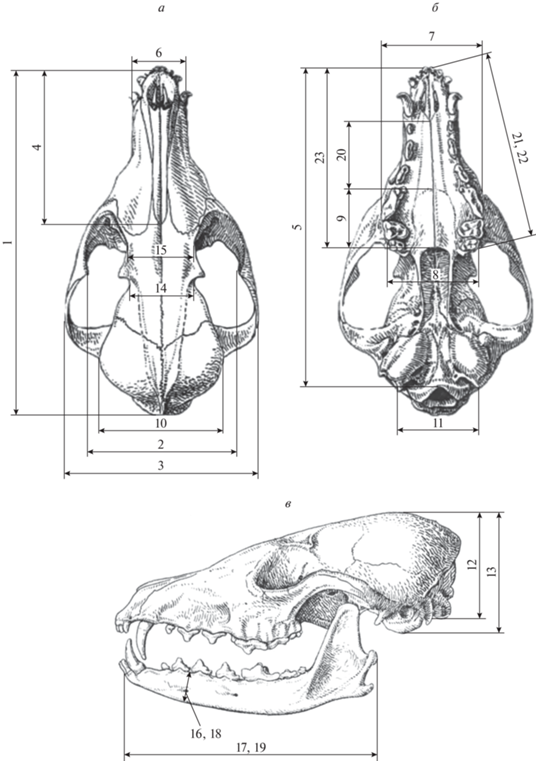
Таблица 2.
Средние значения (мм) промеров ручных, агрессивных и неселекционируемых по поведению самцов и самок лисиц
| Номер промера | Ручные самцы (n = 25) | Ручные самки (n = 23) | Агрессивные самцы (n = 25) | Агрессивные самки (n = 25) | Контрольные самцы (n = 25) | Контрольные самки (n = 26) |
|---|---|---|---|---|---|---|
| 1 | 148.0 ± 0.88 | 139.7 ± 0.44 | 147.4 ± 0.62 | 140.6 ± 0.57 | 146.2 ± 0.68 | 141.1 ± 0.80 |
| 2 | 63.9 ± 0.54 | 61.0 ± 0.30 | 64.7 ± 0.33 | 62.5 ± 0.31 | 63.4 ± 0.38 | 62.1 ± 0.54 |
| 3 | 75.0 ± 0.40 | 71.1 ± 0.30 | 76.0 ± 0.34 | 72.5 ± 0.30 | 73.4 ± 0.35 | 72.3 ± 0.45 |
| 4 | 55.2 ± 0.62 | 51.8 ± 0.35 | 54.7 ± 0.35 | 51.7 ± 0.46 | 55.4 ± 0.49 | 52.3 ± 0.39 |
| 5 | 134.9 ± 0.74 | 127.5 ± 0.44 | 133.3 ± 0.61 | 127.7 ± 0.64 | 132.4 ± 0.69 | 128.4 ± 0.68 |
| 6 | 24.3 ± 0.18 | 22.6 ± 0.16 | 25.1 ± 0.39 | 23.7 ± 0.19 | 24.8 ± 0.22 | 23.6 ± 0.22 |
| 7 | 41.6 ± 0.24 | 39.5 ± 0.22 | 41.5 ± 0.23 | 40.1 ± 0.21 | 41.1 ± 0.25 | 40.2 ± 0.26 |
| 8 | 37.4 ± 0.22 | 35.6 ± 0.20 | 37.6 ± 0.18 | 36.3 ± 0.20 | 37.2 ± 0.28 | 36.3 ± 0.25 |
| 9 | 21.7 ± 0.24 | 20.7 ± 0.19 | 21.5 ± 0.26 | 20.8 ± 0.26 | 20.9 ± 0.23 | 20.7 ± 0.19 |
| 10 | 48.3 ± 0.18 | 47.0 ± 0.23 | 48.3 ± 0.21 | 47.2 ± 0.26 | 48.1 ± 0.26 | 47.6 ± 0.26 |
| 11 | 35.9 ± 0.20 | 34.4 ± 0.19 | 35.5 ± 0.15 | 34.3 ± 0.19 | 35.3 ± 0.15 | 34.4 ± 0.1 |
| 12 | 41.8 ± 0.18 | 40.5 ± 0.15 | 41.5 ± 0.15 | 40.7 ± 0.23 | 41.9 ± 0.23 | 41.1 ± 0.24 |
| 13 | 49.6 ± 0.24 | 48.2 ± 0.15 | 49.4 ± 0.18 | 48.5 ± 0.27 | 50.1 ± 0.22 | 49.2 ± 0.30 |
| 14 | 24.6 ± 0.17 | 24.9 ± 0.30 | 25.1 ± 0.21 | 25.2 ± 0.25 | 24.5 ± 0.26 | 24.3 ± 0.31 |
| 15 | 26.4 ± 0.23 | 24.8 ± 0.21 | 28.1 ± 0.20 | 27.0 ± 0.19 | 26.9 ± 0.24 | 25.9 ± 0.21 |
| 16 | 10.3 ± 0.11 | 9.4 ± 0.11 | 10.0 ± 0.09 | 9.6 ± 0.09 | 10.4 ± 0.10 | 9.9 ± 0.11 |
| 17 | 109.4 ± 0.49 | 102.3 ± 0.38 | 108.3 ± 0.45 | 103.5 ± 0.41 | 107.6 ± 0.56 | 102.9 ± 0.56 |
| 18 | 10.5 ± 0.10 | 9.6 ± 0.10 | 10.2 ± 0.09 | 9.9 ± 0.10 | 10.6 ± 0.10 | 10.1 ± 0.14 |
| 19 | 109.1 ± 0.47 | 101.8 ± 0.42 | 108.4 ± 0.47 | 103.3 ± 0.40 | 107.3 ± 0.57 | 102.5 ± 0.54 |
| 20 | 33.5 ± 0.37 | 31.9 ± 0.32 | 32.5 ± 0.33 | 31.4 ± 0.28 | 32.6 ± 0.34 | 31.4 ± 0.37 |
| 21 | 77.9 ± 0.47 | 74.0 ± 0.34 | 77.3 ± 0.39 | 74.0 ± 0.44 | 77.4 ± 0.47 | 74.7 ± 0.47 |
| 22 | 77.9 ± 0.48 | 74.0 ± 0.32 | 77.2 ± 0.39 | 73.9 ± 0.44 | 77.2 ± 0.45 | 74.9 ± 0.48 |
| 23 | 77.0 ± 0.54 | 73.2 ± 0.35 | 75.9 ± 0.41 | 72.8 ± 0.44 | 75.1 ± 0.46 | 72.6 ± 0.50 |
Примечание. Обозначения и схема промеров приведены на рис. 1. Значения приведены в формате M ± SEM.
Анализ исходных признаков второй выборки проводили методом главных компонент [39] в пакете Statistica10. По значению первой из главных компонент (определяющей общий размер) были выделены особи, характеризующиеся наиболее крупным и наиболее мелким черепом, которые затем анализировались с помощью программы 2B-PLS, как и потомки возвратного скрещивания. Две выборки (потомков возвратного скрещивания и исходных родительских линий) никогда не смешивались и всегда анализировались раздельно. Для того чтобы исключить влияние пола на размерные признаки, исходные данные были выровнены по полу (для этого из исходных значений у самок было вычтено среднее значение по самкам, у самцов – среднее значение по самцам). После этой процедуры проводили анализ главных компонент. Анализ морфологической изменчивости лисиц и собак, проведенный нами ранее, показал высокую степень их гомологии по главным компонентам – интегральным показателям, выявленным для скелетных признаков, а также высокую наследуемость компонент [40, 41].
Генотипирование по микросателлитным маркерам
Потомки от возвратного скрещивания были ранее генотипированы по 350 микросателлитным маркерам (SSR, simple sequence repeat) для поиска локусов, отвечающих за доместикационное и агрессивное поведение, процедура генотипирования и подсчета аллелей для маркеров описана ранее [37]. Список маркеров см. [Supplementary, Table 3, 37].
Выделение ДНК и генотипирование животных из второй выборки (представителей ручных, агрессивных и неселекционированных лисиц) проводили в ходе настоящей работы в ИЦиГ СО РАН. Образцы крови были собраны из vena safena. ДНК выделяли из образцов крови методом фенольной экстракции [42]. Животные из второй выборки были генотипированы по трем микросателитным маркерам (RVC1, REN193M22 и FH2535) – тем, которые показали ассоциацию с размером черепа беккроссов.
Генотипы ручных, агрессивных и неселекционированных лисиц по микросателлитам RVC1, REN193M22 и FH2535 определяли с помощью ПЦР. Праймеры и условия амплификации описаны ранее [43, 44]. Прямой праймер каждого маркера был помечен флуоресцентным красителем FAM. Последующий фрагментный анализ проводили в ЦКП “Геномика” ИЦиГ СО РАН на приборе Applied Biosystems 3130xl в присутствии Gene-Scan-500 LIZ Size Standard. Для анализа результатов использовали программное обеспечение “Peak Scanner” (Appleid Biosystem, США). Результаты ПЦР анализировали индивидуально для каждого маркера, аллели идентифицировали исходя из длины фрагментов микросателлитной последовательности. Информация о генотипах животных из второй выборки по каждому из аллелей микросателлитных маркеров лисицы для включения ее в анализ была записана в следующем виде: название маркера_длина микросателлитной последовательности_количество копий конкретного аллеля у конкретного животного, т.е. “0” – отсутствие данного аллеля, “1” – гетерозигота и “2” – гомозигота по данному аллелю.
2B-PLS-анализ
Первая и вторая выборки анализировались раздельно с помощью метода 2B-PLS (two-block partial least-squares analysis) [45] в пакетах программ PAST и Jacobi4 [46]. Данные для анализа каждого животного из первой выборки (поколение беккроссов) включали информацию о генотипах по 350 микросателлитным маркерам, записанную в виде длин двух аллелей (далее они определяются как “генетические” признаки), и 21 промере черепа (далее – “морфологические” признаки). Данные для анализа второй выборки (ручные, агрессивные и неселекционированные лисицы) включали информацию о генотипах по трем маркерам (RVC1, REN193M22 и FH2535), локализованным на 10-й хромосоме, и 23 краниометрическим признакам. Метод 2B-PLS базируется на возможности геометрического представления объектов в виде точек евклидова пространства невысокой размерности для любых типов описаний. Для каждого типа данных строится своя матрица расстояний между объектами. По всем матрицам расстояний между объектами вычисляются представляющие их евклидовы пространства. Системы координат в этих пространствах вращаются до получения максимального соответствия между конфигурациями объектов. Далее конфигурации объектов исследуются средствами многомерного анализа. В методе 2B-PLS вычисляются линейные комбинации (латентные векторы, или компоненты) между переменными двух блоков, которые максимально учитывают ковариацию между двумя исходными наборами данных, хотя существуют расширения PLS для моделирования отношений между большим количеством наборов [45, 47, 48]. Метод 2B-PLS успешно используется в мировой практике для анализа сложных количественных признаков [49–56], в том числе при анализе соответствия морфологических и молекулярно-генетических данных [29, 57–63]. В обоих рассматриваемых нами случаях для морфологических и генетических признаков лисиц были вычислены конгруэнтные друг другу компоненты краниометрической и генетической изменчивости (бикомпоненты – “морфологические” и “генетические”). Набор корреляций исходных морфологических признаков с “морфологическими” компонентами позволяет дать им фенотипическую интерпретацию. Корреляции микросателлитных маркеров с генетическими компонентами могут дать информацию о локализации в геноме лисицы участков, вовлеченных в контроль изучаемых признаков [57, 58].
Пороговые значения уровня значимости для корреляции фенотипических и генетических признаков с бикомпонентами были рассчитаны с использованием критерия false discovery rate (FDR) [64]. Модуль порогового значения корреляции признаков с генетической и морфологической компонентами для уровня статистической значимости Р < 0.01 составил 0.348, для уровня P < 0.05–0.280.
Для обработки данных использовались пакеты программ PAST (Paleontological statistics software package) [65], а также Jacobi4, разрабатываемый в ИЦиГ СО РАН [46]. Jacobi4 специально разработан для анализа и комбинирования данных различных типов, в том числе количественных и качественных признаков.
РЕЗУЛЬТАТЫ
2B-PLS анализ первой выборки (поколение бекроссов)
На основе данных о морфологии (краниометрических промерах) и генотипах (данные по 350 микросателлитным маркерам для каждой особи) лисиц поколения беккроссов были вычислены пары бикомпонент с помощью метода 2B-PLS. Первая из них описывает максимальную долю общей ковариации морфологических и генетических признаков (в данном анализе – 66%) (табл. 3). Биологическая интерпретация компонент определяется величиной и знаком корреляции исходных признаков с каждой из компонент. По значениям корреляции краниометрических признаков с первой “морфологической” компонентой (табл. 1) ее следует интерпретировать как размерную, так как все признаки показывают с ней высокую положительную корреляцию. Следовательно, более крупные особи будут иметь большие значения первой компоненты. Последующие компоненты вносят меньший вклад в изменчивость изучаемых признаков (табл. 3). Они имеют различный знак и разный уровень корреляции с признаками, по которым эти компоненты могут быть интерпретированы как определяющие пропорции краниальных признаков. Для примера в табл. 1 представлены корреляции признаков со второй парой генетической и морфологической бикомпонент. В настоящей работе мы рассматриваем первую пару бикомпонент. Далее мы будем называть первую морфологическую компоненту “размерной”.
Таблица 3.
Доля ковариации генетической и морфологической изменчивости, описываемой бикомпонентами, рассчитанная для потомков лисиц от возвратного скрещивания (беккроссов) на ручного родителя
| Бикомпоненты (пары осей) | Собственное значение | Процент общей ковариации | Кумулятивный % общей ковариации |
|---|---|---|---|
| Оси_01 | 0.646 | 66.431 | 66.431 |
| Оси_02 | 0.068 | 7.034 | 73.465 |
| Оси_03 | 0.044 | 4.569 | 78.034 |
| Оси_04 | 0.038 | 3.953 | 81.988 |
| Оси_05 | 0.032 | 3.249 | 85.237 |
| Оси_06 | 0.027 | 2.745 | 87.981 |
| Оси_07 | 0.022 | 2.283 | 90.265 |
| Оси_08 | 0.016 | 1.685 | 91.950 |
| Оси_09 | 0.014 | 1.458 | 93.408 |
| Оси_10 | 0.012 | 1.253 | 94.662 |
| Оси_11 | 0.011 | 1.145 | 95.807 |
| Оси_12 | 0.009 | 0.894 | 96.701 |
| Оси_13 | 0.008 | 0.801 | 97.501 |
| Оси_14 | 0.006 | 0.662 | 98.163 |
| Оси_15 | 0.006 | 0.572 | 98.734 |
| Оси_16 | 0.004 | 0.438 | 99.172 |
| Оси_17 | 0.003 | 0.330 | 99.502 |
| Оси_18 | 0.002 | 0.188 | 99.690 |
| Оси_19 | 0.001 | 0.148 | 99.838 |
| Оси_20 | 0.001 | 0.117 | 99.956 |
| Оси_21 | 0.000 | 0.044 | 100.000 |
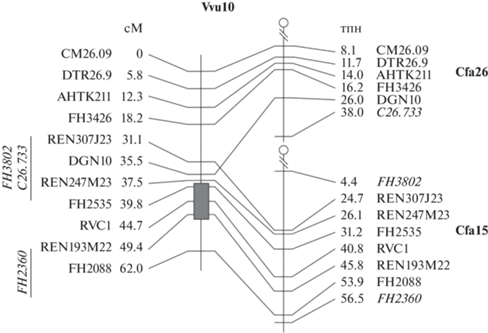
Корреляции же генетического признака с обеими бикомпонентами могут являться показателем участия данного локуса в формировании изучаемых фенотипических признаков. Так, статистически значимая корреляция с первой парой бикомпонент была обнаружена для 55 аллелей 48 микросателлитных маркеров, локализованных почти на каждой из хромосом (за исключением 3-й и 13-й) у лисиц-потомков возвратного скрещивания (Дополнительный материал, табл. 1). Выявленные локусы были сопоставлены нами с известными из литературы данными по генетическому контролю скелетных признаков у собак. Одним из наиболее интересных локусов является участок на 10‑й хромосоме лисицы протяженностью менее 10 сМ. Три полиморфных маркера, локализованных в данном участке, – RVC1, REN193M22 и FH2535 показали в настоящей работе статистически значимую связь с размерной осью (табл. 4, 5). Выявленный регион синтенен одному из ключевых регионов в геноме собак, вовлеченному в контроль изменчивости размеров тела [17, 26, 66]. Взаимное расположение этих микросателлитов на 10-й хромосоме лисицы, а также соответствующие им синтенные участки 15-й хромосомы собаки показаны на рис. 2. На 15-й хромосоме собаки в районе 43.5 тпн, т.е. в участке между двумя микросателлитами, использованными в настоящей работе (RVC1_151 и REN193M22), локализован ген IGF-1, являющийся одним из ключевых в контроле размеров тела собак [21].
Таблица 4.
Корреляции генетических признаков (микросателлитных маркеров) с первой парой бикомпонент (генетической и морфологической осями) на 10-й хромосоме лисицы. 2B-PLS-анализ, беккроссы
| Генотип | Генетическая ось_01 | Морфологическая ось_01 | Частота носителей генотипа, % |
|---|---|---|---|
| Ch10_RVC1_151_0 | 0.333 | 0.333 | 60.0 |
| Ch10_RVC1_151_1 | –0.313 | –0.321 | 32.3 |
| Ch10_REN193M22_188_2 | 0.407 | 0.310 | 67.1 |
| Ch10_REN193M22_191_0 | 0.407 | 0.310 | 67.1 |
| Ch10_FH2535_186_0 | 0.154 | 0.289 | 65.0 |
| Ch10_FH2535_186_1 | –0.141 | –0.282 | 26.0 |
Таблица 5.
Генетические варианты, выявленные у лисиц для каждого из маркеров Ch10_RVC1, Ch10_REN193M22, Ch10_FH2535
| Название маркера | Хромосома лисицы | Хромосома собаки | Число аллелей маркера у лисицы | Перечень аллелей (длина в пн) |
|---|---|---|---|---|
| FH2535 | 10 | 15 | 5 | 174, 182, 186, 178, 196 |
| RVC1 | 10 | 15 | 4 | 149, 151, 153, 155 |
| REN193M22 | 10 | 15 | 2 | 188, 191 |
Рис. 2.
Сравнительная карта 10-й хромосомы лисицы (Vvu10) с гомологичными участками генома собаки (Cfa15 и Cfa26 – 15-я и 26-я хромосомы собаки) (из: Kukekova et al., 2007, Genome Research [44]).
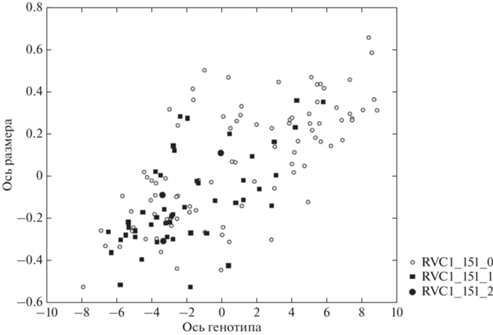
Распределение особей по генотипам одного из трех использованных в работе микросателлитов представлено графически на плоскости первых бикомпонент (рис. 3). Как было сказано выше, отрицательные значения оси соответствуют наиболее мелким, положительные – более крупным особям. Ось генотипов показывает количество копий соответствующего аллеля у каждой из особей (0, 1 или 2). Как следует из рис. 3, особи, несущие по крайней мере одну копию аллеля RVC1_151, по размеру черепа мельче особей, не имеющих данного аллеля. Неслучайное распределение особей разного генотипа в пространстве бикомпонент указывает на влияние аллеля RVC1_151 на размер черепа лисиц (значение корреляций микросателлитных маркеров с первой морфологической осью приведены в табл. 4).
Рис. 3.
Распределение генотипов маркера RVC1 на плоскости бикомпонент у потомков возвратного скрещивания. Оси размера и генотипа – первые, т.е. максимальные по вкладу в изменчивость морфологическая (“размерная”) и генетическая оси, рассчитанные методом 2B-PLS для оценки ковариации генетических и морфологических признаков лисиц. RVC1_151 – один из аллелей маркера RVC1 длиной 151 пн. Полые кружки RVC1_151_0 – особи, не имеющие данного аллеля; закрашенные кружки RVC1_151_2 – особи, гомозиготные по RVC1_151; закрашенные квадраты RVC1_151_1 – особи, гетерозиготные по данному аллелю.
Анализ “размерного” локуса на десятой хромосоме лисиц из второй выборки (ручные, агрессивные и неселекционированные лисицы)
На втором этапе был проведен отдельный независимый анализ промеров черепа лисиц из второй выборки (по 25 самцов и 25 самок из ручной, агрессивной и неселекционируемой популяций – всего 150 особей). Эти животные представляют собой репрезентативную выборку из экспериментальных популяций, содержащихся на данном этапе на лисьей ферме ИЦиГ СО РАН. Очищенные черепа были измерены, полученные промеры проанализированы методом главных компонент. Как ранее было показано на собаках, главные компоненты (ГК) представляют собой интегральные признаки, которые могут охарактеризовать особь как по размерам, так и по пропорциям скелетных признаков [17]. Первая из компонент как у собак, так и в нашем исследовании объясняет наибольший процент изменчивости и описывает вариации размеров. Корреляции исходных признаков с ГК1 имеют очень высокие значения (например, с длиной черепа 0.94, со скуловой шириной 0.78). С учетом результатов анализа главных компонент для последующего генотипирования были отобраны 34 особи с мелкими размерами черепов (из области отрицательных значений ГК1), а также с крупными черепами (из области положительных значений ГК1), наиболее контрастные как по абсолютным размерам черепа, так и по первой главной компоненте.
Генотипы определяли по трем описанным выше микросателлитным маркерам (RVC1, REN193M22 и FH2535) для подтверждения вклада этих маркеров в вариацию размеров черепа лисиц. Перечень контрастных животных, их принадлежность к той или иной популяции, краниометрические данные и генотипы представлены в табл. 6. Информация о генотипе каждого животного, как и ранее для беккроссов, была записана в виде дозы (числа копий 0, 1 или 2) каждого аллеля каждого из трех маркеров и проанализирована в пакете программ PAST для оценки степени их совместного влияния на краниологический фенотип – были вычислены PLS-оси (бикомпоненты) совместной генетической и морфологической изменчивости. В результате была выявлена статистически значимая связь между генетической и морфологической осями (коэффициент корреляции между осями r = –0.4137, P = 0.015; n = 34, самцы и самки вместе, рис. 4).
Таблица 6.
Генотипы (пн) по трем маркерам и значение “размерной” компоненты (ГК1) для 34 лисиц, наиболее контрастных по размеру черепа
| Номер | ГК1 | Популяция | Возраст | REN193M22 | REN193M22 | FH2535 | FH2535 | RVC1 | RVC1 |
|---|---|---|---|---|---|---|---|---|---|
| 7424-1м | –7.64 | А | 243 | 188 | 188 | 178 | 196 | 149 | 151 |
| Кафе-1м | –7.32 | Р | 231 | 188 | 191 | 178 | 196 | 153 | 151 |
| 466-1м | –6.59 | Н | 243 | 188 | 188 | 178 | 186 | 155 | 155 |
| 7456-1м | –5.84 | А | 246 | 188 | 191 | 174 | 178 | 153 | 155 |
| 7824-1м | –5.74 | А | 246 | 188 | 188 | 178 | 182 | 153 | 155 |
| 292-1м | –5.50 | Н | 231 | 188 | 191 | 174 | 186 | 153 | 155 |
| Джей-1м | –4.89 | Р | 227 | 191 | 191 | 178 | 178 | 149 | 151 |
| 254-1м | –4.74 | Н | 248 | 188 | 188 | 182 | 186 | 155 | 155 |
| 232-1м | –4.65 | Н | 231 | 188 | 188 | 182 | 186 | 153 | 155 |
| Мура-1м | –4.55 | Р | 235 | 188 | 191 | 182 | 186 | 149 | 151 |
| 508-1ж | –3.96 | Н | 232 | 188 | 188 | 174 | 186 | 153 | 155 |
| 7936-1м | –2.72 | А | 243 | 188 | 188 | 174 | 178 | ||
| 256-1ж | –2.45 | Н | 232 | 188 | 188 | 182 | 190 | 153 | 151 |
| 314-1ж | –2.29 | Н | 239 | 188 | 188 | 174 | 178 | 153 | 155 |
| Аспи-1м | –1.72 | Р | 204 | 188 | 188 | 182 | 182 | 153 | 151 |
| 7972-1ж | –0.84 | А | 255 | 188 | 188 | 196 | 186 | 155 | 155 |
| 7838-1ж | –0.66 | А | 254 | 188 | 188 | 182 | 196 | 153 | 155 |
| 7848-1ж | –0.53 | А | 247 | 188 | 188 | 178 | 186 | 155 | 151 |
| 7768-1ж | –0.24 | А | 238 | 188 | 188 | 178 | 196 | 153 | 155 |
| 618-1м | 0.01 | Н | 235 | 188 | 188 | 174 | 178 | 153 | 155 |
| 612-1ж | 0.20 | Н | 259 | 188 | 188 | 182 | 182 | 153 | 151 |
| 230-1м | 0.34 | Н | 188 | 191 | 182 | 196 | 153 | 155 | |
| 264-1м | 2.45 | Н | 231 | 188 | 188 | 182 | 182 | 153 | |
| Дева-1м | 3.10 | Р | 250 | 188 | 188 | 182 | 182 | 153 | 153 |
| 452-1м | 3.96 | Н | 232 | 188 | 188 | 174 | 182 | 153 | 153 |
| 612-2ж | 4.06 | Н | 259 | 188 | 188 | 174 | 182 | 153 | 151 |
| 602-1ж | 4.62 | Н | 247 | 188 | 188 | 178 | 182 | 149 | 153 |
| Адап-1м | 4.65 | Р | 247 | 188 | 191 | 182 | 186 | 151 | 151 |
| 7244-1ж | 5.00 | А | 248 | 188 | 188 | 196 | 153 | 155 | |
| 7528-1ж | 5.19 | А | 252 | 188 | 188 | 196 | 196 | 153 | 155 |
| 960-1ж | 5.42 | Н | 248 | 188 | 188 | 178 | 196 | ||
| Экст-1ж | 5.55 | Р | 244 | 188 | 188 | 182 | 178 | 153 | 151 |
| 7972-2ж | 5.87 | А | 255 | 188 | 188 | 196 | 186 | 155 | 155 |
| 7754-1ж | 6.27 | А | 250 | 188 | 188 | 178 | 178 | 153 | 155 |
Рис. 4.
Распределение в пространстве первой пары (размерной и генетической) осей особей ручных, агрессивных и неселекционированных лисиц. Ось генотипа рассчитана на основе информации по микросателлитным маркерам Ch10_RVC1, Ch10_REN193M22 и Ch10_FH2535 для n = 34 контрастных по размеру черепа лисиц (в том числе 18 самцов и 16 самок). Линия показывает регрессию генотипической оси на размерную ось.
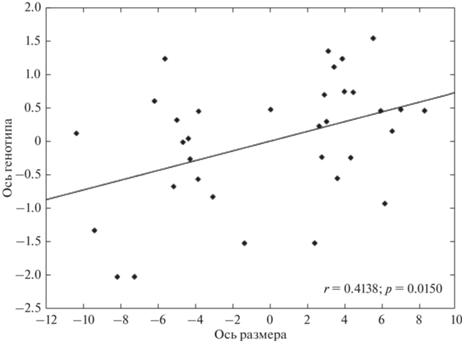
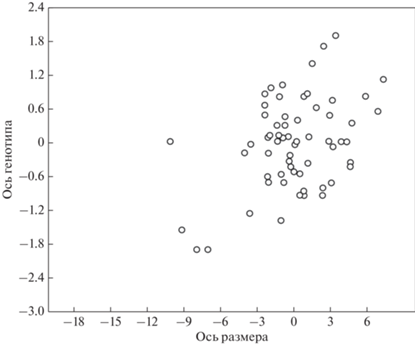
После этого мы определили генотипы по тем же трем микросателлитам у оставшихся животных из второй выборки, добавили их данные к контрастным по размерам животным и провели аналогичный анализ. Компонентный анализ показал, что на долю первой компоненты (ГК1), описывающей вариацию размеров черепа этой выборки, приходится 49.9% общей изменчивости. Далее значение ГК1 было взято в качестве размерного фенотипа, и в пакете PAST был вычислен коэффициент корреляции ГК1 с генотипом по трем перечисленным выше микросателлитам, локализованным на 10-й хромосоме лисицы, для объединенной выборки измеренных лисиц (также без учета их поведения): r = 0.240, P = 0.007 (n = 127). Анализ, проведенный отдельно для самцов и самок, показал статистически достоверную корреляцию у самцов (r = 0.282, P = 0.026, n = 62; рис. 5). У самок не обнаружено статистически значимой корреляции (r = 0.202, P = 0.106, n = 65).
Рис. 5.
Распределение в пространстве первой пары (размерной и генетической) осей самцов ручных, агрессивных и неселекционированных лисиц (корреляция размерной оси с осью генотипа: r = 0.282, P = 0.026; n = 62). Ось генотипа рассчитана на основе информации по трем микросателлитным маркерам: Ch10_RVC1, Ch10_REN193M22 и Ch10_FH2535.
ОБСУЖДЕНИЕ
Несмотря на то что размер тела является одним из классических примеров полигенных признаков, в последние годы у разных видов животных выявляется ключевое влияние отдельных генов на этот признак. В особенности это касается исследований на одомашненных видах, претерпевших сильное давление искусственного отбора по различным признакам, в том числе по размеру тела. Например, показано влияние на размер тела генов из семейства Insulin growth factor у собак [21, 66]. Одним из генов-кандидатов у свиней является IGFBP-6 – ген insulin-like growth factor binding protein-6 (IGF-связывающий белок) [67]. Описаны четыре ключевых гена, участвующих в регуляции размеров тела лошадей [68]. Влияние гена IGF-1 на размерные признаки показано также для мыши [69, 70] и человека [70].
Анализ промеров черепа потомков возвратного скрещивания методом 2B-PLS
Анализ обнаружил значимую ковариацию морфологических признаков с многочисленными микросателлитами, локализованными на различных хромосомах лисицы (см. Дополнительные материалы, табл. 1). В настоящей работе более детальному анализу был подвергнут один из локусов, маркированный тремя микросателлитами, локализованными на участке протяженностью менее 10 сМ на 10-й хромосоме лисицы. Каждый из этих микросателлитов показал статистически значимую связь с наиболее значимой по вкладу в изменчивость, описывающей вариации размеров компонентой. Этот участок генома лисицы синтенен участку 15-й хромосомы собаки, в котором ранее был обнаружен локус, сцепленный с размером тела у самок португальской водной собаки [71]. В нем локализован ген IGF1, обусловливающий 15% вариабельности размеров у собак [26, 66]. Ранее показано, что один из трех наиболее часто встречающихся аллельных вариантов IGF1 является главным фактором, определяющим мелкий размер у собак [21]. Как было сказано выше, участие гена IGF1 в контроле размеров тела показано для разных видов млекопитающих [21, 26, 67–71], причем у псовых размер контролируется наличием разных мутаций [72]. В недавней работе у собак и волков выявлена новая однонуклеотидная замена (SNP) в локусе IGF1, связанная с крупным размером тела [72]. Авторами собран большой материал в поддержку того, что за крупный размер у собак и волков отвечает аллель Т этого SNP. Однако у других псовых, в том числе и у лисиц, аллеля Т обнаружено не было. Как мы показали в настоящей работе, различия по размеру черепа у лисиц также картируются в геномный участок, содержащий ген IGF1. Можно предположить, что если у лисиц размер тоже регулируется действием IGF1, то за это у них отвечает не та же, что у собак и волков, а другая мутация в этом гене. Это предполагает, что механизм вариации размера у псовых (возможно, и в целом у млекопитающих) довольно консервативен, в него вовлечен IGF1, но даже у таких относительно близкородственных видов как лиса и собака мутации, влияющие на размер, могут быть разные. Нельзя исключить, однако, что за различия в размере у лисиц может отвечать и другой ген в этом локусе.
Анализ “размерного” локуса на десятой хромосоме у потомков ручных, агрессивных и неселекционированных лисиц
Анализ подтвердил показанную на беккроссах связь размерной компоненты с участком на 10-й хромосоме лисицы. Такая связь была значимой для наиболее контрастных по абсолютным размерам тела лисиц (n = 34, P = 0.015). При анализе же выборки, в которую были включены как селекционированные по поведению, так и контрольные животные, данная связь оказалась более выраженной у самцов лисиц. У самок не обнаружено достоверной корреляции между размерной и генетической осями. Ранее при анализе ручной и агрессивной популяций лисиц влияние отбора по поведению выявлялось для отдельных скелетных признаков [34, 41]. Отбор на ручное поведение изменяет пропорции черепа лисиц в том же направлении, в каком они изменяются у исторически одомашненных видов, и этот эффект был также более выражен у самцов [34, 35]. Мы видим возможное объяснение в том, что искусственный отбор по любым признакам среди самцов является более интенсивным, поскольку в промышленных популяциях лисиц, как и в нашей экспериментальной, соотношение самцов и самок поддерживается на уровне 1 : 3. То есть количество самцов, отбираемых для размножения, как правило в 3 раза меньше количества отбираемых на племя самок. Поэтому можно ожидать, что любые коррелированные изменения в процессе отбора в первую очередь коснутся самцов. На собаках, однако, было показано, что синтенный выявленному нами локус, маркированный микросателлитом FH2295 на 15-й хромосоме португальских водных собак, сцеплен с размерными признаками самок [71].
На современном этапе отбора было выявлено увеличение объема серого вещества мозга у ручных лисиц по сравнению с неселекционируемыми по поведению, причем одни и те же области увеличены в объеме при отборе как на ручное, так и на агрессивное поведение [36]. Этот факт хотя и противоречит общепринятым представлениям об уменьшении объема мозга при доместикации [73], он свидетельствует, что отбор по поведению в противоположных направлениях (на дружелюбие и агрессивность) может аналогичным образом влиять на морфологию мозга и расширяет наши представления о возможных эффектах такого отбора [36]. Кроме того, интересным является то, что аллельный вариант IGF1, показывающий связь с мелким размером собак, в то же время демонстрирует связь с признаками поведения [74]. В настоящей работе также показано, что некоторые из микросателлитных маркеров (на 8-й, 12-й, 14-й и 15-й хромосомах лисицы – см. Дополнительные материалы, табл. 1), обнаруживших значимую ассоциацию с размерами черепа лисиц, расположены в геномных районах, отвечающих за различия в поведении между ручными и агрессивными лисицами [3, 75]. Мы предполагаем провести дальнейший детальный анализ этих районов, чтобы оценить их потенциальную связь с морфологией и поведением лисиц.
Выражаем искреннюю благодарность Л.Н. Трут, Ю.Э. Гербеку и Р.Г. Гулевич за участие в обсуждении результатов, ЦКП Геномных исследований ИЦиГ СО РАН и Г.В. Васильеву за проведенный фрагментный анализ ДНК лисиц, а также Marshfield Laboratories Mammalian Genotyping Service и сотруднице Корнелльского университета (Итака, США) Светлане Темных за генотипирование образцов лисиц-потомков возвратного скрещивания с помощью набора микросателлитных маркеров собак.
Содержание и разведение экспериментальных животных в ЦКП “Генофонды пушных и сельскохозяйственных животных” ИЦиГ СО РАН поддерживались бюджетным проектом № FWNR-2022-0019. Экспериментальная работа частично поддержана проектом РНФ № 21-44-04405.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Belyaev D.K. Destabilizing selection as a factor of domestication // J. Hered. 1979. V. 70. P. 301–308.
Coppinger R., Coppinger L. Dogs: A Startling New Understanding of Canine Origin, Behavior & Evolution. N.Y.: Scribner, 2001. 356 p.
Trut L., Oskina I., Kharlamova A. Animal evolution during domestication: the domesticated fox as a model // BioEssays. 2009. V. 31. № 3. P. 349–360. https://doi.org/10.1002/bies.200800070
Price E.O. Animal Domestication and Behavior. Wallingford, United Kingdom: CABI Publ., 2002. 297 p.
Clutton-Brock J. Origins of the dog: the archaeological evidence // The Domestic Dog: Its Evolution, Behavior and Interactions with People. Second ed. Cambridge: Cambr. Univ. Press, 2017. P. 7–22.
Lindblad-Toh K., Wade C.M., Mikkelsen T.S. et al. Genome sequence, comparative analysis and haplotype structure of the domestic dog // Nature. 2005 V. 8. № 438(7069). P. 803–819. https://doi.org/10.1038/nature04338
Frantz L.A., Mullin V.E., Pionnier-Capitan M. et al. Genomic and archaeological evidence suggest a dual origin of domestic dogs // Science. 2016. V. 3. № 352(6290). P. 1228–1231. https://doi.org/10.1126/science.aaf3161
Germonpré M., Lázničková-Galetová M., Sablin M.V., Bocherens H. Self-domestication or human control? The Upper Palaeolithic domestication of the wolf // Hybrid Communities. London: Routledge, 2018. 324 p.
Bergström A., Frantz L., Schmidt R. et al. Origins and genetic legacy of prehistoric dogs // Science. 2020. V. 30. № 370(6516). P. 557–564. https://doi.org/10.1126/science.aba9572
Pitulko V.V., Kasparov A.K. Archaeological dogs from the Early Holocene Zhokhov site in the Eastern Siberian Arctic // J. Archaeol. Sci.: Reports 13. 2017. P. 491–515. https://doi.org/10.1016/j.jasrep.2017.04.003
Zeuner F.E. A History of Domesticated Animals. London: Hutchinson & Co. (Publishers) Ltd., 1963. 560 p.
Harcourt R.A. The dog in prehistoric and early historic Britain // J. Archaeol. Sci. 1974. V. 1. P. 151–175.
Clutton-Brock J. Domesticated Animals from Early Times. London: British Museum (Natural History) and William Heinemann Ltd., 1981. 208 p.
Clutton-Brock J. Origins of the dog: domestication and early history // The Domestic Dog: Its Evolution, Behaviour, and Interactions with People. Cambridge: Cambr. Univ. Press, 1995. P. 7–20.
Zedda M., Manca P., Chisu V. et al. Ancient pompeian dogs – morphological and morphometric evidence for different canine populations // Anat. Histol. Embryol. 2006. V. 35. № 5. P. 319–324. https://doi.org/10.1111/j.1439-0264.2006.00687.x
Pionnier-Capitan M., Bemilli C., Bodu P. et al. New evidence for Upper Palaeolithic small domestic dogs in South-Western Europe // J. Archaeol. Sci. 2011. V. 38. № 9. P. 2123–2140. https://doi.org/10.1016/j.jas.2011.02.028
Chase K., Carrier D.R., Adler F.R. et al. Genetic basis for systems of skeletal quantitative traits: Principal component analysis of the canid skeleton // PNAS 2002. V. 99. № 15. P. 9930–9935. https://doi.org/10.1073/pnas.152333099
Parker H.G., Shearin A.L., Ostrander E.A. Man’s best friend becomes biology’s best in show: genome analyses in the domestic dog // Annu. Rev. Genet. 2010. V. 44. P. 309–336. https://doi.org/10.1146/annurev-genet-102808-115200
Yengo L., Sidorenko J., Kemper K.E. et al. GIANT Consortium. Meta-analysis of genome-wide association studies for height and body mass index in ∼700 000 individuals of European ancestry // Hum. Mol. Genet. 2018. V. 27. № 20. P. 3641–3649. https://doi.org/10.1093/hmg/ddy271
Rimbault M., Beale H.C., Schoenebeck J.J. et al. Derived variants at six genes explain nearly half of size reduction in dog breeds // Genome Research. 2013. № 23. P. 1985–1995. https://doi.org/10.1101/gr.157339.113
Sutter N.B., Bustamante C.B., Chase K. et al. A single IGF1 allele is a major determinant of small size in dogs // Science. 2007. № 316. P. 112–115. https://doi.org/10.1126/science.1137045
Fang X.B., Liu S.C., Wu Q.Y. et al. Linkage analysis of SNPs in IGFBP-6 and its relation with the body sizes of pig // Genet. Mol. Res. 2015. V. 14. № 4. P. 17273–17280. https://doi.org/10.4238/2015
Makvandi-Nejad S., Hoffman G.E., Allen J.J. et al. Four loci explain 83% of size variation in the horse // PLoS One. 2012. V. 7. № 7. P. e39929. https://doi.org/10.1371/journal.pone.0039929
Plassais J., Kim J., Davis B.W. et al. Whole genome sequencing of canids reveals genomic regions under selection and variants influencing morphology // Nat. Commun. 2019. V. 10. № 1. P. 1489. https://doi.org/10.1038/s41467-019-09373-w
Bannasch D., Young A., Myers J. et al. Localization of canine brachycephaly using an across breed mapping approach // PLoS One. 2010. V. 5. № 3. P. e9632. https://doi.org/10.1371/journal.pone.0009632
Boyko A.R., Quignon P., Li L., Schoenebeck J.J. et al. A simple genetic architecture underlies morphological variation in dogs // PLoS Biol. 2010. № 8. P. e1000451. https://doi.org/10.1371/journal.pbio.1000451
Schoenebeck J.J., Hutchinson S.A., Byers A. et al. Variation of BMP3 contributes to dog breed skull diversity // PLoS Genet. 2012. V. 8. № 8. P. e1002849. https://doi.org/10.1371/journal.pgen.1002849
Schoenebeck J.J., Ostrander E.A. Insights into morphology and disease from the dog genome project // Annu. Rev. Cell Dev. Biol. 2014. V. 30. P. 535–560. https://doi.org/10.1146/annurev-cellbio-100913-012927
Wilson L.A.B., Balcarcel A., Geiger M. et al. Modularity patterns in mammalian domestication: Assessing developmental hypotheses for diversification // Evol. Lett. 2021. https://doi.org/10.1002/evl3.231
Wright D., Henriksen R., Johnsson M. Defining the Domestication Syndrome: Comment on Lord et al. 2020 // Trends Ecol. Evol. 2020. V. 35. № 12. P. 1059–1060. https://doi.org/10.1016/j.tree.2020.08.009
Trut L.N. Early Canid Domestication: The Farm-Fox Experiment: Foxes bred for tamability in a 40-year experiment exhibit remarkable transformations that suggest an interplay between behavioral genetics and development // Am. Sci. 1999. V. 87. № 2. P. 160–169.
Wayne R.K. Consequences of domestication: Morphological diversity of the dog // The Genetics of the Dog. N.Y.: CABI Publ., 2001. P. 43–60.
Evin A., Dobney K., Schafberg R. et al. Phenotype and animal domestication: A study of dental variation between domestic, wild, captive, hybrid and insular Sus scrofa // BMC Evol Biol. 2015. V. 15. P. 6. https://doi.org/10.1186/s12862-014-0269-x
Трут Л.Н., Дзержинский Ф.Я., Никольский В.С. Компонентный анализ краниологических признаков серебристо-черных лисиц (Vulpes vulpes Desm) и их изменений, возникающих при доместикации // Генетика. 1991. Т. 27. № 8. С. 1440–1449.
Трут Л.Н., Дзержинский Ф.Я., Никольский В.С. Внутричерепная аллометрия и краниологические изменения при доместикации серебристо-черных лисиц // Генетика. 1991. Т. 27. № 9. С. 1605–1612.
Hecht E.E., Kukekova A.V., Gutman D.A. et al. Neuromorphological changes following selection for tameness and aggression in the Russian farm-fox experiment // J. Neurosci. 2021. V. 41. № 28. P. 6144–6156. https://doi.org/10.1523/JNEUROSCI.3114-20.2021
Kukekova A.V., Trut L.N., Chase K. et al. Mapping loci for fox domestication: deconstruction/reconstruction of a behavioral phenotype // J. Behav. Genet. 2011. V.41. № 4. P. 593–606. https://doi.org/10.1007/s10519-010-9418-1
Trut L.N., Kharlamova A.V., Kukekova A.V. et al. Morphology and behavior: Are they coupled at the genome level? // The Dog and Its Genome. Woodbury N.Y.: Cold Spring Harbor Lab. Press, 2006. P. 515–538.
Jackson J.E. A User’s Guide to Principal Components. N.Y.: A Wiley Interscience Publ., 1991. 563 p.
Kharlamova A.V., Trut L.N., Carrier D.R. et al. Genetic regulation of canine skeletal traits: Trade-offs between the hind limbs and forelimbs in the fox and dog // Integr. Comp. Biol. 2007. V. 47. P. 373–381. https://doi.org/10.1093/icb/icm023
Харламова А.В., Чейз К., Ларк К.Г. и др. Сопоставление вариации параметров скелетной системы лисиц (Vulpes vulpes), отбираемых по поведению, и собак (Canis familiaris) // Вестник ВОГиС. 2008. Т. 12. № 1/2. С. 32–38.
Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии. Молекулярное клонирование. М.: Мир, 1984. 545 с.
Kukekova A.V., Trut L.N., Oskina I.N. et al. A marker set for construction of a genetic map of the silver fox (Vulpes vulpes) // J. Hered. 2004. V. 95. № 3. P. 185–194. https://doi.org/10.1093/jhered/esh033
Kukekova A.V., Trut L.N., Oskina I.N. et al. A meiotic linkage map of the silver fox, aligned and compared to the canine genome // Genome Res. 2007. V. 17. P. 387–399. https://doi.org/10.1101/gr.5893307
Rohlf F.J., Corti M. Use of two-block partial least squares to study covariation in shape // System. Biology. 2000. V. 49. P. 740–753. https://doi.org/10.1080/106351500750049806
Polunin D., Shtaiger I., Efimov V. JACOBI4 software for multivariate analysis of biological data // bioRxiv. 2019. P. 803684. https://doi.org/10.1101/803684
Wold H. Path models with latent variables: The NIPALS approach // Quantitative Sociology: International Perspectives on Mathematical and Statistical Model Building. N.Y.: Acad. Press, 1975. P. 307–357.
Rosipal R., Krämer N. Overview and recent advances in partial least squares // International Statistical and Optimization Perspectives Workshop “Subspace, Latent Structure and Feature Selection”. Berlin; Heidelberg: Springer, 2005. P. 34–51.
Baab K.L., Freidline S.E., Wang S.L., Hanson T. Relationship of cranial robusticity to cranial form, geography and climate in Homo sapiens // Am. J. Phys. Anthropol. 2010. V. 141. № 1. P. 97–115. https://doi.org/10.1002/ajpa.21120
Goswami A., Polly P.D. Methods for studying morphological integration, modularity and covariance evolution // Quantitative Methods in Paleobiology. Ithaca; N.Y.: Paleontol. Society Papers Series, 2010. V. 16. P. 213–243. https://doi.org/10.1017/S1089332600001881
Goswami A., Polly P.D. The influence of modularity on cranial morphological disparity in carnivora and primates (Mammalia) // PLoS One. 2010. V. 5. № 3. P. e9517. https://doi.org/10.1371/journal.pone.0009517
Goswami A., Smaers J.B., Soligo C., Polly P.D. The macroevolutionary consequences of phenotypic integration: From development to deep time // Phil. Trans. R. Soc. B. 2014. V. 369. № 1649. P. 20130254. https://doi.org/10.1098/rstb.2013.0254
Álvarez A., Perez S.I., Verzi D.H. The role of evolutionary integration in the morphological evolution of the skull of caviomorph rodents (Rodentia: Hystricomorpha) // Evol. Biol. 2015. V. 42. № 3. P. 312–327. https://doi.org/10.1111/j.1420-9101.2011.02395.x
Goswami A., Watanabe A., Felice R.N. et al. High-density morphometric analysis of shape and integration: the good, the bad, and the not-really-a-problem // Integr. Comp. Biol. 2019. V. 59. № 3. P. 669–683. https://doi.org/10.1093/icb/icz120
Alhajeri B.H. Cranial variation in geographically widespread dwarf gerbil Gerbillus nanus (Gerbillinae, Rodentia) populations: Isolation by distance versus adaptation to local environments // J. Zool. Systematics and Evol. Res. 2019. V. 57. № 1. P. 191–203. https://doi.org/10.1111/jzs.12247
Brassard C., Merlin M., Guintard C. et al. Interrelations between the cranium, the mandible and muscle architecture in modern domestic dogs // Evol. Biol. 2020. V. 47. № 4. P. 308–324. https://doi.org/10.1007/s11692-020-09515-9
Ковалева В.Ю., Абрамов С.А., Дупал Т.А., Ефимов В.М. Анализ соответствия и комбинирование молекулярно-генетических и морфологических данных в зоологической систематике // Изв. РАН. Серия биол. наук. 2012. № 4. С. 404–414.
Ковалева В.Ю., Литвинов Ю.Н., Ефимов В.М. Землеройки (Soricidae, Eulipotyphla) Сибири и Дальнего Востока: комбинирование и поиск конгруэнтности молекулярно-генетических и морфологических данных // Зоол. журн. 2013. Т. 92. № 11. С. 1383–1398. https://doi.org/10.7868/S0044513413110081
Klingenberg C.P., Spence J.R., Mirth C.K. Introgressive hybridization between two species of waterstriders (Hemiptera: Gerridae: Limnoporus): Geographical structure and temporal change of a hybrid zone // J. Evol. Biol. 2000. V. 13. № 5. P. 756–765.
Myers E.M., Janzen F.J., Adams D.C., Tucker J.K. Quantitative genetics of plastron shape in slider turtles (Trachemys scripta) // Evolution. 2006. V. 60. № 3. P. 563–572. https://doi.org/10.1111/j.0014-3820.2006.tb01137.x
Claverie T., Patek S.N. Modularity and rates of evolutionary change in a power-amplified prey capture system // Evolution. 2013. V. 67. № 11. P. 3191–3207. https://doi.org/10.1111/evo.12185
Hanot P., Herrel A., Guintard C., Cornette R. Unravelling the hybrid vigor in domestic equids: the effect of hybridization on bone shape variation and covariation // BMC Evol. Biol. 2019. V. 19. № 1. P. 1–13. https://doi.org/10.1186/s12862-019-1520-2
Hanot P., Bayarsaikhan J., Guintard C. et al. Cranial shape diversification in horses: Variation and covariation patterns under the impact of artificial selection // BMC Ecol. Evol. 2021. V. 21. № 1. P. 1–19. https://doi.org/10.1186/s12862-021-01907-5
Benjamini Y., Hochberg Y. Controlling the false discovery rate: A practical and powerful approach to multiple testing // J. Royal Stat. Society. Series B. 1995. № 57. P. 289–300. https://doi.org/10.1111/j.2517-6161.1995.tb02031.x
Hammer Ø., Harper D.A.T., Ryan P.D. PAST: Paleontological statistics software package for education and data analysis // Palaeontol. Electronica. 2001. V. 4. № 1. P. 9.
Rimbault M., Beale H.C., Schoenebeck J.J. et al. Derived variants at six genes explain nearly half of size reduction in dog breeds // Gen. Res. 2013. № 23. P. 1985–1995. 113https://doi.org/10.1101/gr.15733
Fang X.B., Liu S.C., Wu Q.Y. et al. Linkage analysis of SNPs in IGFBP-6 and its relation with the body sizes of pig // Genet. Mol. Res. 2015. V. 14. № 4. P. 17273–17280. https://doi.org/10.4238/2015
Makvandi-Nejad S., Hoffman G.E., Allen J.J. et al. Four loci explain 83% of size variation in the horse // PLoS One. 2012. V. 7. № 7. P. e39929. https://doi.org/10.1371/journal.pone.0039929
Baker J., Liu J.P., Robertson E.J., Efstratiadis A. Role of insulin-like growth factors in embryonic and postnatal growth // Cell. 1993. V. 75. P. 73–82. https://doi.org/10.1016/S0092-8674(05)80085-6
Kawai M., Rosen C.J. Insulin-like growth factor-I and bone: lessons from mice and men // Pediatr. Nephrol. 2009. V. 24. P. 1277–1285. https://doi.org/10.1007/s00467-008-1040-6
Bérubé S.C., Johnsson P.R., Bunimov N. et al. Two length variants of the microsatellite FH2295 as markers for body size of female Portuguese water dogs // J. Appl. Genetics. 2012. V. 53. P. 121–123. https://doi.org/10.1007/s13353-011-0076-7
Plassais J., vonHoldt B.M., Parker H.G. et al. Natural and human-driven selection of a single noncoding body size variant in ancient and modern canids // Current Biol. 2022. V. 32. P. 1–9. https://doi.org/10.1016/j.cub.2021.12.036
Sánchez-Villagra M.R., Geiger M., Schneider R.A. The taming of the neural crest: A developmental perspective on the origins of morphological covariation in domesticated mammals // Royal Soc. Open Sci. 2016. V. 3. № 6. P. 160107. https://doi.org/10.1098/rsos.160107
Zapata I., Lilly M.L., Herron M.E. et al. Genetic testing of dogs predicts problem behaviors in clinical and nonclinical samples // BMC Genomics. 2022. V. 23. P. 1–19. https://doi.org/10.1186/s12864-022-08351-9
Nelson R.M., Temnykh S.V., Johnson J.L. et al. Genetics of interactive behavior in silver foxes (Vulpes vulpes) // Behavior Genet. 2017. V. 47. № 1. P. 88–101.
Дополнительные материалы отсутствуют.


