Генетика, 2023, T. 59, № 5, стр. 562-572
Полногеномный поиск новых генов-кандидатов мясной продуктивности у овец северокавказской мясо-шерстной породы
А. Ю. Криворучко 1, 2, *, Р. В. Зуев 2, А. И. Суров 1, А. В. Скокова 1, А. А. Каниболоцкая 1, А. А. Лиховид 2, О. А. Яцык 1
1 Северо-Кавказский Федеральный научный аграрный центр
356241 Ставропольский край, Михайловск, Россия
2 Северо-Кавказский Федеральный университет
355017 Ставрополь, Россия
* E-mail: rcvm@yandex.ru
Поступила в редакцию 10.08.2022
После доработки 29.09.2022
Принята к публикации 25.10.2022
- EDN: FNVKYA
- DOI: 10.31857/S0016675823050090
Аннотация
Поиск новых генов, влияющих на рост и развитие мышечной ткани, ассоциированных с признаками мясной продуктивности овец, является актуальной задачей, выполнение которой неразрывно связано с перспективами развития маркер-ориентированной и геномной селекции. В статье приведены данные по использованию нового подхода к обнаружению генов-кандидатов мясной продуктивности овец. Генотипирование животных выполняли с использованием ДНК-биочипов Ovine Infinium HD BeadChip 600K, поиск ассоциаций проводили с использованием программного обеспечения PLINK V.1.07. В ходе проделанной работы выявлены SNP, достоверно ассоциированные с фенотипическими признаками, характеризующими мясную продуктивность у овец северокавказской мясо-шерстной породы: высотой в холке, живой массой при рождении, толщиной жировой ткани, шириной “мышечного глаза”. Для поиска генов-кандидатов было отобрано 19 однонуклеотидных замен с наибольшей достоверностью ассоциаций. Определение местоположения анализируемых SNP позволило установить 16 новых генов-кандидатов, ассоциированных с прижизненными показателями мясной продуктивности овец северокавказской мясо-шерстной породы: CILK1, ENO4, CTPS2, GABRB2, SLC44A1, C12ORF45, SLC41A2, CASD1, SINE2, SLC35F1, RP, SH3KBP1, LRRIQ1, SWAP70, THBS1 и FSIP. Генотипирование животных по этим генам следует использовать в маркер-ассоциированной селекции для улучшения показателей мясной продуктивности овец и изучения их влияния на фенотип.
Интенсивное развитие мясного овцеводства, связанное с возрастающим спросом на высококачественную баранину, обусловливает необходимость совершенствования существующих пород с целью повышения их продуктивных качеств. Одним из наиболее перспективных подходов для решения этой задачи является использование методов маркер-ассоциированной селекции по аллелям генов, влияющих на мясную продуктивность [1, 2].
В результате ранее проведенных научных исследований выявлен ряд генов, связанных с количественными и качественными показателями мясной продуктивности овец. Одним из наиболее изученных маркерных генов является ген MSTN (myostatin). Белковый продукт этого гена ограничивает рост мышечной ткани у млекопитающих. Присутствие аллелей гена, ассоциированных со снижением функциональной активности миостатина, приводит к развитию фенотипа двойной мускулатуры [3, 4]. В качестве маркерных также были предложены гены, тем или иным образом влияющие на работу миостатина. Так, ген MYOD1 (myogenic differentiation 1) влияет на параметры мясной продуктивности овец путем регуляции экспрессии MSTN [5]. Ген FST (follistatin) рассматривается как перспективный маркер мясной продуктивности в связи с тем, что мышечные изоформы белка фоллистатина обладают ингибирующей способностью в отношении миостатина [6]. Гормон роста обладает мощным анаболическим и антикатаболическим действием в различных типах клеток. В наибольшей степени эффект гормона проявляется в мышечной и хрящевой тканях [7]. Выявлена связь полиморфизма гена GH (growth hormone) с живой массой и показателями роста овец [8, 9]. Возникновение мутации каллипиги в межгенной области DLK1 (Delta Like Non-Canonical Notch Ligand 1) – GTL2 (WAS/WASL-interacting protein family member) вызывает эктопическую экспрессию кластера импринтированных генов в скелетных мышцах, что приводит к гипертрофии отдельных групп мышц бедер овец и снижению количество жира [10]. Также ранее было показано, что полиморфизм генов REM1 (RRAD and GEM like GTPase 1), MEF2B (myocyte enhancer factor 2B) и FABP4 (fatty acid binding protein 4) связан с рядом прижизненных и убойных показателей продуктивности у овец [11–13].
Несмотря на наличие достаточно большого количества информации о генах, влияющих на показатели мясной продуктивности у овец, их список далеко не полон. Это связано с участием множества белков и ферментов в формировании мышечных волокон, каждый из которых зависит от структуры и функций различного числа генов [14]. Также сложности при поиске новых генов мясной продуктивности возникают в связи с тем, что их эффект может быть скрыт наличием совокупности аллельных вариантов уже известных генов с доказанным влиянием на развитие мышц. Вместе с тем поиск новых генов, влияющих на рост и развитие мышечной ткани, связанных с фенотипическими признаками овец, актуален для дальнейшего развития селекционной работы. В связи с этим целесообразно, по нашему мнению, проводить поиск новых генов-кандидатов мясной продуктивности в группе животных, имеющих сходный генотип по набору полиморфизма известных маркерных генов. Вероятно, разнообразие параметров мясной продуктивности в такой группе будет обусловлено не полиморфизмом уже изученных маркерных генов, а аллельными вариантами ранее не рассмотренных генов.
Стабилизацию влияния одних генов и проявление функции других генов можно наблюдать среди пород, подвергающихся селекции для получения максимальной продуктивности в определенных экологических нишах, различающихся по климату, составу кормов и воды. К таким породам овец относится северокавказская мясо-шерстная, хорошо адаптированная для разведения в условиях засушливых степей Юга России. Она отличается достаточно высокой мясной продуктивностью для своего класса, бараны-производители имеют среднюю массу более 100 кг, а ярки – 60 кг. При этом дисперсия фенотипа мясных форм в породе указывает на определенное разнообразие генетического потенциала и возможность дальнейшего ее совершенствования [15, 16].
Цель нашего исследования – поиск новых генов-кандидатов мясной продуктивности у овец северокавказской мясо-шерстной породы в группе животных с учетом генотипов по полиморфизму известных генов, влияющих на развитие мышечной ткани.
МАТЕРИАЛЫ И МЕТОДЫ
Исследования проводились на базе лабораторий Всероссийского научно-исследовательского института овцеводства и козоводства (ВНИИОК) – филиала ФГБНУ “Северо-Кавказский Федеральный научный аграрный центр”, Сколковского института науки и технологий “Сколтех”.
Объект исследования – бараны северокавказской мясо-шерстной породы в возрасте 12 мес., принадлежащие к племенному ядру. На первом этапе было генотипировано 275 баранов. Среди них были отобраны 50 баранов с наиболее близкими генотипами по полиморфизму известных генов мясной продуктивности: MSTN (oar3_ OAR2_118149265; oar3_OAR2_118150665), MEF2B (oar3_OAR5_3860373; oar3_OAR5_3867887), FABP4 (oar3_OAR9_57537070), GH (oar3_OAR11_47529756; oar3_OAR11_47545769), REM1 (oar3_OAR13_ 60384593; oar3_ OAR13_60385591), MYOD1 (oar3_ OAR15_3434222; oar3_OAR15_3441596), FST (OAR16_ 27849538.1; oar3_OAR16_25632659; oar3_OAR16_ 25632701; oar3_OAR16_25633632$; oar3_OAR16_ 25638968), DLK1 (oar3_OAR18_64313560; oar3_ OAR18_64314938; oar3_OAR18_64341672). Выбранные бараны принадлежали к трем неродственным селекционным линиям, внутри линий степень родства не превышала 50% по отцу. Животные содержались в одинаковых оптимальных условиях в племзаводе “Восток” Ставропольского края. Так как мясная продуктивность породы оценивается по баранам, ярки в группу исследованных животных не входили. У отобранных баранов определяли 15 прижизненных показателей мясной продуктивности. Живую массу при рождении и в 12 мес. измеряли с помощью весов МП 300 ВЕДА Ф-1 (МИДЛиК, Россия), по этим параметрам рассчитывали ежедневный суточный прирост. С использованием линейки и измерительной ленты измеряли высоту в холке и крестце, ширину груди и спины, глубину груди, обхват плеча, предплечья и бедра. С помощью переносного УЗИ-сканера DUS 60 VET (EDAN, Китай), используя линейный датчик с частотой 5.0 МГц, определяли параметры толщины и ширины “мышечного глазка”, толщину бедренной мышцы и жировой ткани в поясничной области. Все животные были клинически здоровы и получали полноценный смешанный рацион.
Генотипирование
Геномную ДНК выделяли из образцов цельной крови, взятой в асептических условиях из яремной вены, с помощью набора Pure Link Genomic DNA MiniKit (Invitrogen Life Technologies, США) в соответствии с протоколом производителя. Генотипирование животных проводили с использованием Ovine Infinium HD BeadChip 600K (Illumina Inc. CA, США) в соответствии с протоколом производителя. Первичную обработку результатов генотипирования проводили с помощью программы Genome Studio 2.0 (Illumina Inc. CA, США).
Контроль качества генотипирования
Контроль качества генотипирования проводили с использованием программного обеспечения PLINK v.1.07 [17]. В обработку данных были включены образцы с показателем количества выявленных однонуклеотидных замен больше 0.95 (Call Rate). Из анализа были исключены SNP (single nucleotide polymorphism), не имеющие хромосомной или физической локализации, с частотой минорных аллелей (MAF – Minor Allele Frequency) меньше 0.01, частотой потерянных генотипов (missing genotype) больше 0.1. В качестве порогового значения по критерию Харди–Вайнберга (Hardy–Weinberg equilibrium) использовалось значение p = 0.0001. С положительным результатом контроль качества генотипирования прошли 50 образцов. Из 606 006 SNP для дальнейшего анализа были использованы 562 549.
Генетический и статистический анализ
Полногеномный анализ ассоциаций (ПГАА) выполняли с использованием программного обеспечения PLINK v1.07 [17] на основе оценки значимости влияния SNP на количественные прижизненные признаки мясной продуктивности овец. Для множественных сравнений вводили поправку Бонферрони, устанавливающую показатель уровня значимости р = 0.8 × 10–6. Визуализацию и построение графиков проводили с применением пакета “QQman” на языке программирования R. Поиск генов-кандидатов осуществляли среди ближайших генов, расположенных на расстоянии, не превышающем 250 000 пн (половина сантиморганиды) от SNP, показавших достоверную ассоциацию с признаками мясной продуктивности. Анализируемые полиморфизмы представлены биаллельными SNP, аллелям присвоены обозначения А и В, в соответствии с наименованием на чипе Ovine Infinium HD BeadChip 600K (Illumina Inc. CA, США). Для картирования SNP использовали сборку генома Ovis_Aries_3.1. Аннотирование генов выполняли с использованием геномных браузеров UCSC (www.genome.ucsc.edu) и Ensembl (www.ensembl.org).
РЕЗУЛЬТАТЫ
Проведенный поиск ассоциаций полиморфизма известных маркерных генов с некоторыми прижизненными параметрами продуктивности показал отсутствие достоверных ассоциаций у обследуемой группы овец северокавказской мясо-шерстной породы (табл. 1). Достоверность ассоциаций для четырех SNP, расположенных в генах MSTN, MEF2B и FABP4, не была рассчитана, так как у изучаемых животных они присутствовали только в виде мономорфных гомозиготных вариантов. Для остальных генетических вариантов показатель достоверности был значительно выше порогового значения p с учетом поправки Бонферрони.
Таблица 1.
Связь полиморфизма маркерных генов с прижизненными показателями мясной продуктивности овец северокавказской мясо-шерстной породы
| Ген | SNP | Хромосома | Позиция | ЖМ, p | ТМГ, p |
|---|---|---|---|---|---|
| MSTN | oar3_OAR2_118149265 | 2 | 118 149 265 | N/A | N/A |
| MSTN | oar3_OAR2_118150665 | 2 | 118 150 665 | N/A | N/A |
| MEF2B | oar3_OAR5_3860373 | 5 | 3 860 373 | 0.93 | 0.98 |
| MEF2B | oar3_OAR5_3867887 | 5 | 3 867 887 | N/A | N/A |
| FABP4 | oar3_OAR9_57537070 | 9 | 57 537 070 | N/A | N/A |
| GH | oar3_OAR11_47529756 | 11 | 47 529 756 | 0.40 | 0.23 |
| GH | oar3_OAR11_47545769 | 11 | 47 545 769 | 0.04 | 0.78 |
| REM1 | oar3_OAR13_60384593 | 13 | 60 384 593 | 0.15 | 0.87 |
| REM1 | oar3_OAR13_60385591 | 13 | 60 385 591 | 0.15 | 0.87 |
| MYOD1 | oar3_OAR15_3434222 | 15 | 3 434 222 | 0.49 | 0.53 |
| MYOD1 | oar3_OAR15_3441596 | 15 | 3 441 596 | 0.61 | 0.19 |
| FST | OAR16_27849538.1 | 16 | 25 631 318 | 0.11 | 0.34 |
| FST | oar3_OAR16_25632659 | 16 | 25 632 659 | 0.56 | 0.83 |
| FST | oar3_OAR16_25632701 | 16 | 25 632 701 | 0.86 | 0.66 |
| FST | oar3_OAR16_25633632 | 16 | 25 633 632 | 0.56 | 0.83 |
| FST | oar3_OAR16_25638968 | 16 | 25 638 968 | 0.03 | 0.54 |
| DLK1 | oar3_OAR18_64313560 | 18 | 64 313 560 | 0.77 | 0.67 |
| DLK1 | oar3_OAR18_64314938 | 18 | 64 314 938 | 0.14 | 0.79 |
| DLK1 | oar3_OAR18_64341672 | 18 | 64 341 672 | 0.99 | 0.55 |
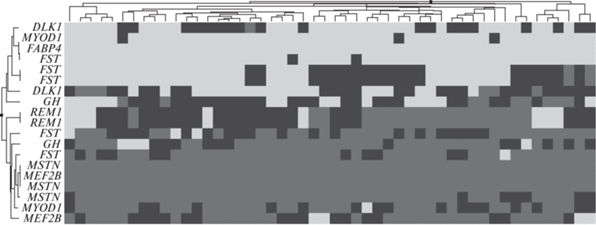
Тепловая карта распределения аллелей SNP маркерных генов (рис. 1) мясной продуктивности демонстрирует отсутствие выраженной кластеризации исследуемых животных по выявленным генотипам. Не сформировано каких-либо достаточно крупных кластеров до 3–4 порядка, распределение генотипов носит случайный характер. Несколько SNP присутствовали в виде мономорфных аллелей (в генах MSTN, MEF2B и FABP4), часть замен в генах FST и DKL1 имела единичные гетерозиготные варианты. Таким образом, в подобранной группе животных полиморфизм известных генов продуктивности, вероятно, не оказывал достоверного влияния на параметры фенотипа овец северокавказской мясо-шерстной породы.
Рис. 1.
Тепловая карта распределения аллелей SNP маркерных генов, достоверно не связанных с мясной продуктивностью, у овец северокавказской мясо-шерстной породы. Кластеризация по генотипам. Светло-серый цвет – гомозигота А, серый цвет – гетерозигота АВ, темно-серый цвет – гомозигота В.
В результате полногеномного поиска ассоциаций выявлено около 20 SNP, достоверно связанных с прижизненными показателями мясной продуктивности у овец северокавказской мясо-шерстной породы (рис. 2).
Рис. 2.
Манхэттенский график результатов ПГАА с набором значений –log10(p) для исследуемых SNP по прижизненным показателям мясной продуктивности: высота в холке (а), живая масса при рождении (б), толщина жировой ткани (в), ширина “мышечного глазка” (г), обхват предплечья (д). Синяя линия обозначает порог ожидаемой достоверности различий при значении –log10(p) = 5.
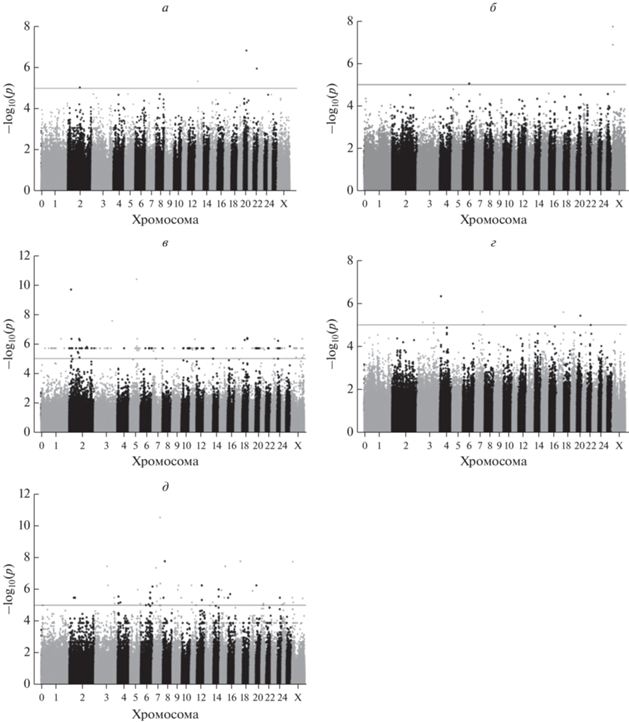
Для высоты в холке максимальный показатель достоверности был у двух SNP, расположенных на хромосомах 20 и 22 (рис. 2,а). Одна из них находилась в интроне гена, другая – в межгенном пространстве. При оценке ассоциаций с живой массой при рождении (рис. 2,б) три замены с высокой достоверностью ассоциаций располагались на хромосоме X. Все они находились очень близко друг от друга рядом с геном CTPS2, что позволяет предположить их совместное наследование.
Для параметра “толщина жировой ткани” было выявлено три SNP, преодолевших порог достоверности с учетом поправки Бонферрони (рис. 2,в). Наибольшую достоверность связи имели замены на хромосомах 2, 3 и 5. Две из них находились в межгенном пространстве, а одна – в интроне гена.
Достоверные связи с параметром “ширина мышечного глазка” были выявлены для нескольких SNP, при этом наибольшую достоверность имели ассоциации SNP, расположенные на хромосоме 4 (рис. 2,г). Они находились в разных интронах одного и того же гена.
В результате ПГАА выявлено несколько SNP, ассоциированных с обхватом предплечья (рис. 2,д), для дальнейшего анализа было отобрано девять замен. Замены с достоверным уровнем ассоциации на хромосомах 8 и 17 расположены в межгенном пространстве, замены на хромосомах 3, 7, 15 и X обнаружены в интронах генов. При этом одна из замен на хромосоме 7 картировалась в интроны сразу двух генов.
Для ряда других прижизненных показателей мясной продуктивности овец северокавказской мясо-шерстной породы, включающего в себя живую массу в годовалом возрасте, среднесуточный прирост, высоту в крестце, ширину и глубину груди, ширину спины, обхваты плеча и бедра, толщину “мышечного глазка” и толщину бедренной мышцы, достоверных ассоциаций с SNP обнаружено не было.
Для дальнейшего изучения и аннотирования близлежащих генов-кандидатов было отобрано 19 SNP, имеющих наибольший уровень достоверности ассоциаций (табл. 2). Из них девять были расположены в интронах генов, остальные 10 – на разном удалении от генов. Ближе всех находилась замена rs411918327 на хромосоме 17 – 17 782 пн. Наиболее отдаленное расположение относительно ближайшего гена-кандидата имела замена rs411177492 на хромосоме 2. Она составила 112 524 пн. Для большинства других замен дистанция не превышала 40 тпн. Это указывает на очень высокую вероятность сцепленного наследования обнаруженных SNP с приведенными в табл. 2 генами-кандидатами продуктивности.
Таблица 2.
Связь SNP с прижизненными показателями мясной продуктивности и новые гены-кандидаты овец северокавказской мясо-шерстной породы
| Признак | Хромросома | SNP | Позиция | p | Ген/расстояние до гена, пн |
|---|---|---|---|---|---|
| ВВХ | 20 | rs405580662 | 25 023 121 | 1.01e-07 | CILK1/intron 11-12 |
| 22 | rs404887224 | 36 141 759 | 1.07e-06 | ENO4/61224 | |
| ЖМР | X | rs410901922 | 14 048 240 | 1.83e-08 | CTPS2/29384 |
| X | rs410836916 | 14 054 083 | 1.83e-08 | CTPS2/23541 | |
| X | rs416819309 | 14 045 890 | 1.30e-07 | CTPS2/31734 | |
| ТЖТ | 5 | rs410336272 | 70 279 825 | 4.07e-11 | GABRB2/intron 9-10 |
| 2 | rs411177492 | 17 793 666 | 2.06e-10 | SLC44A1/112524 | |
| 3 | rs406382505 | 173 590 152 | 2.74e-08 | C12ORF45/26001; SLC41A2/30547 | |
| ШМГ | 4 | rs420242290 | 11 782 536 | 4.64e-07 | CASD1/intron 3-4 |
| 4 | rs427172181 | 11 801 072 | 4.64e-07 | CASD1/intron 10-11 | |
| ОПП | 7 | rs422152157 | 73 053 165 | 2.84e-11 | SINE2/intron 2-3 |
| 8 | rs398907349 | 19 601 156 | 1.66e-08 | SLC35F1/62507 | |
| 17 | rs399242667 | 49 481 697 | 1.66e-08 | RP/34798 | |
| 17 | rs430083361 | 49 493 261 | 1.66e-08 | RP/23234 | |
| 17 | rs411918327 | 49 498 713 | 1.66e-08 | RP/17782 | |
| X | rs428366450 | 17 003 427 | 1.79e-08 | SH3KBP1/intron 13-14 | |
| 3 | rs429375653 | 121 233 501 | 3.43e-08 | LRRIQ1/intron 13-14 | |
| 15 | rs413432564 | 42 537 319 | 3.43e-08 | SWAP70/intron 1-2 | |
| 7 | rs422549589 | 32 364 039 | 4.36e-08 | THBS1/intron 22-23; FSIP1/intron 9-10 |
Ассоциированная с высотой в холке замена rs405580662 располагается в интроне гена CILK1. Ген ENO4 (enolase 4) расположен в 60 тпн от замены rs404887224. Три обнаруженные нами на хромосоме X замены находятся в непосредственной близости (от 23 до 32 тпн) от гена CTPS2 (Cytidine 5'-Triphosphate Synthetase 2). В интроне гена GABRB2 (gamma-aminobutyric acid type A receptor subunit beta2), расположенного на хромосоме 5, находится замена с очень высокими показателями достоверности ассоциаций с толщиной жира в поясничной области. Еще один ассоциированный с толщиной жира SNP расположен в области гена SLC44A1 (solute carrier family 44 member 1). Между геном SLC41A2 (solute carrier family 41 member 2) и геном C12ORF45 (chromosome 12 open reading frame 45) (30 и 26 тпн соответственно) также находится SNP, связанный с толщиной жира. Две замены на хромосоме 4, ассоциированные с шириной “мышечного глазка”, располагаются в интронах гена CASD1 (CAS1 domain containing 1). Ген SINE2 (spectrin repeat containing nuclear envelope protein 2) содержит в своем интроне SNP с очень высокими показателями ассоциации с обхватом предплечья. Еще одна замена, связанная с обхватом предплечья, находится в 60 тпн от гена SLC35F1 (solute carrier family 35 member F1). Сразу три SNP, ассоциированных с обхватом предплечья, находятся в области гена RP (Ribosomal protein) на хромосоме 17, имея расстояние от 17 до 35 тпн до гена. На хромосоме Х находится ген SH3KBP1 (SH3 domain containing kinase binding protein 1), содержащий в интроне SNP с высокой ассоциацией с обхватом предплечья. Также связанная с обхватом предплечья SNP локализуется в интроне гена LRRIQ1 (leucine rich repeats and IQ motif containing 1). Замена rs413432564 обнаружена в интроне гена SWAP70 (switching B cell complex subunit). Замена rs422549589 располагается в локусе, попадающем в интроны сразу двух генов – THBS1 (thrombospondin 1) и FSIP1 (fibrous sheath interacting protein 1).
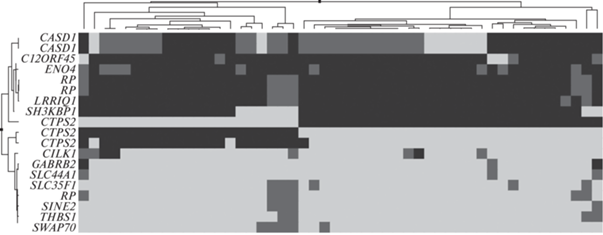
Тепловая карта генотипов животных по обнаруженным нами SNP (рис. 3) показывает совершенно другое распределение гомо- и гетерозиготных вариантов по сравнению с тепловой картой для известных генов мясной продуктивности (рис. 1). Ни одна из замен, формирующих генотипы, не представлена у исследуемых животных в мономорфном варианте, при этом преобладают гомозиготные варианты. Гетерозиготные варианты представлены у достаточно малого числа животных. Кластеризация генотипов позволила выделить две большие группы, существенно различающиеся по аллелям SNP, расположенных преимущественно в двух генах – CTPS2 (CTP synthase 2) и CASD1 (ciliogenesis associated kinase 1).
Рис. 3.
Тепловая карта распределения аллелей SNP, достоверно ассоциированных с прижизненными показателями мясной продуктивностью у овец северокавказской мясо-шерстной породы. Кластеризация по генотипам. Светло-серый цвет – гомозигота А, серый цвет – гетерозигота АВ, темно-серый цвет – гомозигота В.
Таким образом, использованный нами способ выбора животных с минимальным влиянием известных генов продуктивности на фенотип позволил обнаружить новые локусы генома, имеющие достоверную связь с прижизненными показателями мясной продуктивности, и предложить ряд новых генов-кандидатов.
ОБСУЖДЕНИЕ
Проведенный анализ связи полиморфизма известных генов продуктивности с параметрами роста и развития овец показал, что у некоторых представителей овец северокавказской мясо-шерстной породы в результате проводимой селекционной работы эти гены утратили свою роль в обеспечении дисперсии фенотипа. Это связано с тем, что ген миостатина, наиболее сильного регулятора развития мышечной ткани, у обследуемых животных имеет только один аллельный вариант. У них встречается тот же вариант SNP, что и у австралийских мериносовых овец, которые являются одними из предков северокавказской мясо-шерстной породы [18]. На этом фоне влияние остальных известных генов на разнообразие фенотипа существенно снижено еще и по той причине, что замены в них сгруппированы в достаточно случайном порядке, варианты генотипов плохо кластеризуются. Все это указывает на то, что выбранные нами овцы северокавказской мясо-шерстной породы хорошо подходят для поиска новых генов-кандидатов мясной продуктивности, так как животные имеют достаточную вариабельность в параметрах экстерьера и ультразвуковых измерениях. Соответственно обнаруженная дисперсия фенотипа должна иметь под собой основу в виде особенностей генотипа.
Полногеномный поиск ассоциаций SNP с прижизненными параметрами мясной продуктивности выявил несколько замен с достаточно высокими показателями достоверности связи. Для некоторых из них уровень значимости был меньше 0.8 × 10–6. Это указывает на необходимость пристального изучения особенностей строения обнаруженных локусов генома.
Кластеризация и построение тепловой карты для генотипов по SNP, ассоциированным с показателями продуктивности, показали четкое разделение обследованных животных как минимум на две группы. И связано оно в основном с полиморфизмом в области генов CTPS2 и CASD1, влияние которого на параметры фенотипа ранее вообще не рассматривалось. По нашему мнению, именно на них стоит обратить особое внимание при дальнейшем изучении механизма влияния генов продуктивности на параметры роста и развития животных.
Для большинства предлагаемых нами новых генов-кандидатов исследований связи с продуктивными качествами животных ранее не проводилось. Ген CILK1 (Ciliogenesis Associated Kinase 1) кодирует киназу 1, связанную с цилиогенезом, и экспрессируется во многих тканях организма. Мутации в гене CILK1 приводят у человека к наследственным заболеваниям с нарушением развития костно-мышечной системы, легких, головного мозга и других органов. У мышей мутации сопровождаются замедлением роста и патологиями позвоночника [19]. Исходя из этого, ген CILK1 может быть связан с изменениями размера тела у овец и требует дальнейшего изучения.
Продуктом гена ENO4 (enolase 4) является фермент, участвующий в одном из этапов синтеза пирувата в процессе гликолиза. Высокая активность гена выявлена в спермиях человека, барана и мыши. Полиморфизм гена ENO4 у мышей связывают с нарушениями подвижности сперматозоидов и развитием бесплодия [20]. Важность гена ENO4 для энергетического обмена позволяет предположить его влияние на рост и развитие животных.
Ген CTPS2 (Cytidine 5'-Triphosphate Synthetase 2) кодирует одну из изоформ фермента, катализирующего последнюю стадию синтеза пиримидина, входящего в состав молекул урацила, тимина и цитозина. Фермент крайне важен во все периоды развития организма, гомозиготы с нокаутированным геном CTPS2 погибают еще на этапе эмбрионального развития [21]. Учитывая функции гена и особенности распределения генотипов, выявленных в нашем исследовании, следует считать его возможным кандидатом, связанным с продуктивностью изучаемых животных.
Влияние гена GABRB2 (gamma-aminobutyric acid type A receptor subunit beta2) на фенотип животных не изучалось, у человека его полиморфизм связывают с развитием наследственных форм эпилепсии и эпилептической энцефалопатии [22]. Это указывает на его важность для передачи импульсов нейронами и позволяет считать косвенно связанным с регуляцией роста и развития организма.
Кодируемый геном SLC44A1 (solute carrier family 44 member 1) митохондриальный белок отвечает за трансмембранный транспорт холина и регулирует его метаболизм. У мышей высокая активность гена была выявлена в гепатоцитах и мышечной ткани [23]. В связи с этим ген SLC44A1 может рассматриваться в качества кандидата, связанного с продуктивностью у овец.
Ген C12ORF45 (chromosome 12 open reading frame 45) имеет открытую рамку считывания. Несмотря на то что обнаружено достаточно много участков генома, представляющих собой открытые рамки считывания без стоп-кодонов, их функция не ясна. Существует мнение, что они кодируют регуляторные аминокислотные последовательности для различных биологических процессов [24]. Ген SLC41A2 относится к группе, кодирующей белки Mg2+-каналов у бактерий и у высших животных [25]. Оба описанных гена могут влиять на обменные процессы в организме и рассматриваются нами как кандидаты, связанные с мясной продуктивностью.
Ген CASD1 (CAS1 domain containing 1) содержит домен белка, катализирующего превращение сиаловой кислоты. Сиаловая кислота участвует в регуляции межклеточных взаимоотношений, передаче сигналов, распознавании клеток [26]. Мы считаем необходимым дальнейшее изучение роли гена CASD1 в реализации продуктивных качеств у овец.
Есть сообщения о связи мутаций в гене SINE2 (spectrin repeat containing nuclear envelope protein 2) с развитием нейро-мышечной дистрофии и поражением миокарда у человека [27]. Это указывает на возможность связи полиморфизма гена SINE2 с развитием мышечной ткани у животных.
Ген SLC35F1 (solute carrier family 35 member F1) кодирует белок из семейства трансмембранных транспортеров, особенно активно функционирующих в нервной системе при реализации функций гемато-энцефалического барьера [28]. По нашему мнению, ген RP (Ribosomal protein) заслуживает внимания для дальнейшего изучения связи с мясной продуктивностью. Было показано, что рибосомальные протеины не только связаны с функцией рибосом, но и участвуют в процессах канцерогенеза, передаче сигналов в иммунной системе, а также отвечают за нормальное развитие организма [29].
Ген SH3KBP1 (SH3 domain containing kinase binding protein 1) отличается способностью к продукции ряда изоформ белкового продукта за счет альтернативного сплайсинга и принимает участие в перестройке цитоскелета, клеточной адгезии, процессах апоптоза, миграции и инвазии клеток [30]. Такое многообразие функций делает его перспективным геном-кандидатом продуктивных свойств овец.
Функцию генов семейства, к которому относится LRRIQ1 (leucine rich repeats and IQ motif containing 1), связывают с реализацией неспецифического иммунного ответа [31]. Ген SWAP70 (switching B cell complex subunit) участвует в миграции клеток, фагоцитозе, процессах деструкции костной ткани [32]. Оба этих гена представляют интерес для изучения связи их полиморфизма с фенотипическими параметрами овец.
Ген THBS1 (thrombospondin 1) принимает участие в большом количестве обменных процессов, включая межклеточные взаимодействия, регуляцию утилизации глюкозы, метаболизм адипоцитов [33]. Функция гена FSIP1 (fibrous sheath interacting protein 1) до конца не изучена. Имеются сведения об участии гена FSIP1 во взаимодействии ряда белковых молекул, а также о связи его полиморфизма с развитием слизистой меланомы [34] и рака мочевого пузыря [35]. Исходя из структурно-функциональных особенностей локуса, ассоциированного с показателем продуктивности, мы считаем необходимым дальнейшее изучение обоих этих генов на предмет участия в реализации фенотипа животных.
Таким образом, использован новый подход к обнаружению генов-кандидатов продуктивности, основанный на проведении ПГАА в группе животных с учетом генотипа по полиморфизму известных генов, влияющему на развитие мышечной ткани. В результате проведенного ПГАА у овец северокавказской мясо-шерстной породы было выявлено 19 SNP, связанных с прижизненными показателями мясной продуктивности. Определение местоположения анализируемых SNP позволило установить 16 новых генов-кандидатов, ассоциированных с показателями мясной продуктивности овец северокавказской мясо-шерстной породы. Обнаруженные замены располагались в интронах или фланкирующих областях генов: CILK1, ENO4, CTPS2, GABRB2, SLC44A1, C12ORF45, SLC41A2, CASD1, SINE2, SLC35F1, RP, SH3KBP1, LRRIQ1, SWAP70, THBS1 и FSIP1. Предложенные гены-кандидаты участвуют в метаболизме белков и нуклеиновых кислот, обеспечении межклеточных взаимодействий и передачи нервного импульса, в регуляции развития мышечной ткани. Дальнейшие исследования должны быть направлены на подтверждение влияния предложенных генов-кандидатов на фенотип овец, а также на изучение структуры этих генов для выявления конкретных полиморфизмов, вызывающих изменения фенотипа.
Финансирование работы проводилось за счет средств Министерства науки и высшего образования Российской Федерации, грант № 075-15-2021-1037 (внутр. № 15.БРК.21.0001).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Sahu A.R., Nayak N., Panigrahi M., Kumar S. Advances in genomic strategies to improve growth and meat production traits in sheep: An overview // Ind. J. Small Ruminants. 2017. V. 23. № 2. P. 139. https://doi.org/10.5958/0973-9718.2017.00052.6
Trukhachev V.I., Selionova M.I., Krivoruchko A.Y., Aibasov A.M.M. Genetic markers of meat productivity of sheep (Ovis aries L.). I. Myostatin, calpain, calpastatin // Sel’skokhozyaĭstvennaya Biologiya. 2018. V. 53. № 6. P. 1107–1119. https://doi.org/10.15389/agrobiology.2018.6.1107eng
Aiello D., Patel K., Lasagna E. The myostatin gene: An overview of mechanisms of action and its relevance to livestock animals // Animal Genet. 2018. V. 49. № 6. P. 505–519. https://doi.org/10.1111/age.12696
Osman N.M., Shafey H.I., Abdelhafez M.A. et al. Genetic variations in the Myostatin gene affecting growth traits in sheep // Veterinary World. 2021. V. 14. № 2. P. 475. https://doi.org/10.14202/vetworld.2021.475-482
Sousa-Junior L.P.B., Meira A.N., Azevedo H.C. et al. Variants in myostatin and MyoD family genes are associated with meat quality traits in Santa Inês sheep // Animal Biotechnol. 2022. V. 33. № 2. P. 201–213. https://doi.org/10.1080/10495398.2020.1781651
Nissinen T.A., Hentilä J., Fachada V. et al. Muscle follistatin gene delivery increases muscle protein synthesis independent of periodical physical inactivity and fasting // The FASEB J. 2021. V. 35. № 3. P. e21387. https://doi.org/10.1096/fj.202002008R
Sharma R., Kopchick J.J., Puri V., Sharma V.M. Effect of growth hormone on insulin signaling // Mol. Cell. Endocrinol. 2020. V. 518. P. 111038. https://doi.org/10.1016/j.mce.2020.111038
Abdelmoneim T.S., Brooks P.H., Afifi M., Swelum A.A.A. Sequencing of growth hormone gene for detection of polymorphisms and their relationship with body weight in Harri sheep // Ind. J. Animal Res. 2017. V. 51. № 2. P. 205–211. https://doi.org/10.18805/ijar.11457
Gorlov I.F., Kolosov Y.A., Shirokova N.V. et al. Association of the growth hormone gene polymorphism with growth traits in Salsk sheep breed // Small Ruminant Res. 2017. V. 150. P. 11–14. https://doi.org/10.1016/j.smallrumres.2017.02.019
Takeda H., Caiment F., Smit M. et al. The callipyge mutation enhances bidirectional long-range DLK1-GTL2 intergenic transcription in cis // Proc. Natl Acad. Sci. USA. 2006. V. 103. № 21. P. 8119–8124. https://doi.org/10.1073/pnas.0602844103
Cheng J., Zhang X., Li F. et al. Detecting single nucleotide polymorphisms in MEF2B and UCP3 and elucidating their association with sheep growth traits // DNA Cell Biol. 2021. V. 40. № 12. P. 1554–1562. https://doi.org/10.1089/dna.2021.0782
Gavran M., Antunović Z., Gantner V. Candidate genes associated with economically important traits of sheep-a review // Agriculturae Conspectus Scientificus. 2021. V. 86. № 3. P. 195–201.
Trukhachev V.I., Skripkin V.S., Yatsyk O. et al. The polymorphism of REM-1 gene in sheep genome and its influence on some parameters of meat productivity // Research J. Pharmaceutical, Biol. Chem. Sciences. 2016. V. 7. № 3. P. 2351–2357.
Nesvadbova M., Borilova G. Molecular regulation of skeletal muscle tissue formation and development // Veterinarni Med. (Praha). 2018. V. 63. № 11. P. 500–512. https://doi.org/10.17221/7/2018-VETMED
Омаров А.А., Гайдашов С.И. Продуктивные показатели овец северокавказской мясо-шерстной породы и их взаимосвязь с основными селекционируемыми признаками // Вестн. Алтайского гос. аграрного ун-та. 2021. № 2(196). С. 66–72.
Селькин И.И. Породе 50 лет // Сб. науч. трудов Ставропольского научно-исслед. института животноводства и кормопроизводства. 2010. № 13. С. 258–283.
Purcell S., Neale B., Todd-Brown K. et al. PLINK: A tool set for whole-genome association and population-based linkage analyses // The Am. J. Human Genet. 2007. V. 81. № 3. P. 559–575. https://doi.org/10.1086/519795
Kijas J.W., McCulloch R., Edwards J. et al. Evidence for multiple alleles effecting muscling and fatness at the Ovine GDF8 locus // J. Animal Breed. Genet. 2013. V. 130. № 6. P. 468–475. https://doi.org/10.1186/1471-2156-8-80
Fu Z., Gailey C.D., Wang E.J., Brautigan D.L. Ciliogenesis associated kinase 1: Targets and functions in various organ systems // FEBS Letters. 2019. V. 593. № 21. P. 2990–3002. https://doi.org/10.1002/1873-3468.13600
Nakamura N., Dai Q., Williams J. et al. Disruption of a spermatogenic cell-specific mouse enolase 4 (eno4) gene causes sperm structural defects and male infertility // Biol. Reproduction. 2013. V. 88. № 4. P. 90. https://doi.org/10.1095/biolreprod.112.107128
Hickl D., Scheuring D., Möhlmann T. CTP Synthase 2 From Arabidopsis thaliana is required for complete embryo development // Frontiers Plant Sci. 2021. V. 12. P. 652434. https://doi.org/10.3389/fpls.2021.652434
El Achkar C.M., Harrer M., Smith L. et al. Characterization of the GABRB2-Associated Neurodevelopmental Disorders // Annals Neurology. 2021. V. 89. № 3. P. 573–586. https://doi.org/10.1002/ana.25985
Michel V., Bakovic M. The solute carrier 44A1 is a mitochondrial protein and mediates choline transport // The FASEB J. 2009. V. 23. № 8. P. 2749–2758. https://doi.org/10.1096/fj.08-121491
Ladoukakis E., Pereira V., Magny E.G. et al. Hundreds of putatively functional small open reading frames in Drosophila // Genome Biol. 2011. V. 12. № 11. P. 1–17. https://doi.org/10.1186/gb-2011-12-11-r118
Fleig A., Schweigel-Röntgen M., Kolisek M. Solute carrier family SLC41: what do we really know about it? // Wiley Interdisciplinary Reviews: Membrane Transport and Signaling. 2013. V. 2. № 6. P. 227–239. https://doi.org/10.1002/wmts.95
Baumann A.-M.T., Bakkers M.J.G., Buettner F.F.R. et al. 9-O-Acetylation of sialic acids is catalysed by CASD1 via a covalent acetyl-enzyme intermediate // Nat. Communications. 2015. V. 6. P. 7673. https://doi.org/10.1038/ncomms8673
Bamaga A., Vajsar J. Spectrin repeat-containing nuclear envelope protein 2: SYNE2 presenting as a congenital myopathy: A case report // Neuromuscul Disord. 2016. V. 26. P. 139. https://doi.org/10.1016/j.nmd.2016.06.195
Mochizuki T., Mizuno T., Kurosawa T. et al. Functional investigation of solute carrier family 35, member F2, in three cellular models of the primate blood-brain barrier // Drug Metabolism Disposition. 2021. V. 49(1). P. 3–11. https://doi.org/10.1124/dmd.120.000115
Zhou X., Liao W.-J., Liao J.-M. et al. Ribosomal proteins: Functions beyond the ribosome // J. Mol. Cell Biol. 2015. V. 7. № 2. P. 92–104. https://doi.org/10.1093/jmcb/mjv014
Hua X.-Y., Bie X.-X., Cheng X., Zhang S.-G. High expression of CIN85 promotes proliferation and invasion of human esophageal squamous cell carcinoma // Mol. Med. Reports. 2021. V. 23. № 1. P. 12. https://doi.org/10.3892/mmr.2020.11650
Ng A., Xavier R.J. Leucine-rich repeat (LRR) proteins: Integrators of pattern recognition and signaling in immunity // Autophagy. 2011. V. 7. № 9. P. 1082–1084. https://doi.org/10.4161/auto.7.9.16464
Kriplani N., Duncan R.R., Leslie N.R. SWAP70 undergoes dynamic conformational regulation at the leading edge of migrating cells // FEBS Letters. 2019. V. 593. № 4. P. 395–405. https://doi.org/10.1002/1873-3468.13326
Gutierrez L.S., Gutierrez J. Thrombospondin 1 in metabolic diseases // Frontiers Endocrinol. 2021. V. 12. P. 638536. https://doi.org/10.3389/fendo.2021.638536
Chen M., Wu Y., Li W. et al. Loss-of-function variants in FSIP1 identified by targeted sequencing are associated with one particular subtype of mucosal melanoma // Gene. 2020. V. 759. P. 144964. https://doi.org/10.1016/j.gene.2020.144964
Sun M., Zhao W., Zeng Y. et al. Fibrous sheath interacting protein 1 overexpression is associated with unfavorable prognosis in bladder cancer: A potential therapeutic target // OncoTargets Therapy. 2017. V. 10. P. 3949. https://doi.org/10.2147/OTT.S143491
Дополнительные материалы отсутствуют.


