Генетика, 2023, T. 59, № 8, стр. 954-963
Полногеномное ассоциативное исследование риска развития шизофрении в Республике Башкортостан
1 Институт биохимии и генетики Уфимского федерального исследовательского центра
Российской академии наук
450054 Уфа, Россия
2 Башкирский государственный медицинский университет
450008 Уфа, Россия
* E-mail: annagareeva@yandex.ru
Поступила в редакцию 10.02.2023
После доработки 13.03.2023
Принята к публикации 16.03.2023
- EDN: XTJAXD
- DOI: 10.31857/S0016675823080076
Аннотация
Полногеномные ассоциативные исследования оказались мощным подходом к открытию генов подверженности к шизофрении; их выводы имеют важное значение не только для нашего понимания генетической архитектуры данного заболевания, но и для потенциальных применений в области персонализированной медицины. Цель настоящего исследования – изучение генетических факторов риска развития шизофрении при проведении полногеномного анализа ассоциации в Республике Башкортостан.
В мире проведено более 143 полногеномных ассоциативных исследований (GWAS) с риском развития шизофрении. Результаты ряда исследований GWAS показали наличие существенной генетической дифференциации популяций по полиморфным вариантам генов, ассоциированных с данным заболеванием [1, 2]. Самый масштабный на сегодняшний день полногеномный анализ ассоциаций в рамках международного консорциума по психиатрической генетике PGC, с участием 76 755 больных шизофренией и 243 649 здоровых индивидов, выявил 120 генов, участвующих в таких фундаментальных процессах как организация синапсов, дифференцировка нейронов и нейрональная трансмиссия. Среди них были обнаружены ген субъединицы рецептора глутамата GRIN2A, фактор транскрипции SP4, ген конститутивного коактиватора PPAR-гамма-подобного белка 1 FAM120A, ген субъединицы когезина SA-1 (SA1) STAG1, а также ряд других редких разрушительных вариантов генов у больных шизофренией [3].
Недавно проведенное крупнейшее полногеномное секвенирование экзома (WES), включавшее 24 248 больных шизофренией и 97 322 здоровых индивидов, идентифицировало ультраредкие мутации, приводящие к появлению укороченных форм белка в 32 генах, большинство из которых вовлечены в формирование, структуру и функции синапсов и ассоциированы с высоким риском развития шизофрении [4]. Это открытие указывает на синаптическую дисфункцию как на возможную причину развития шизофрении. А идентификация ультраредких вариантов генов субъединицы рецептора NMDA GRIN2A и GRIA3 предполагает нарушение регуляции глутаматергической системы и образование синапсов интернейронов [4]. Важно отметить, что в результате последнего GWAS также были идентифицированы гены STAG1, FAM120A, GRIN2A, SP4, содержащие редкие варианты [4].
Таким образом, конвергенция частых и редких вариантов генов, ассоциированных с шизофренией, поддерживается тем фактом, что недавние крупнейшие GWAS и WES выявили группу генов, участвующих в сходных биологических процессах, таких как пре- и постсинаптические процессы в возбуждающих и тормозных нейронах.
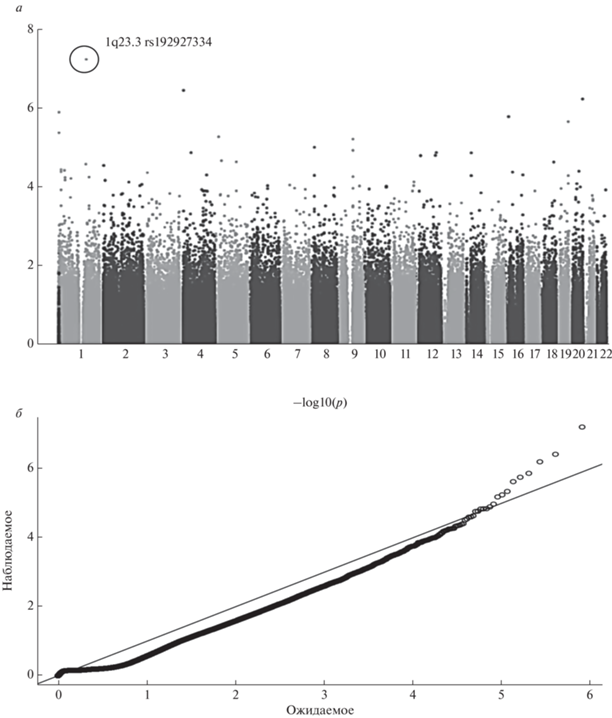
Цель настоящего исследования – изучение генетических факторов риска развития шизофрении при проведении полногеномного анализа ассоциации в Республике Башкортостан (рис. 1).
Рис. 1.
Графическое изображение результатов полногеномного анализа ассоциации 395832 ОНП с параноидной шизофренией (Manhattan plot). По оси X – хромосомная локализация ОНП, по оси Y – значения отрицательного десятичного логарифма уровня значимости P-value (а); б – квентиль-квентиль график (плот) Q-Q plot. Иллюстрация оценки наличия популяционной стратификации.
МАТЕРИАЛЫ И МЕТОДЫ
Объект исследования – 437 мужчин, 379 женщин (из них 320 русских, 357 татар, 139 башкир) с диагнозом параноидная шизофрения (ПШ) F20.xx согласно международной классификации болезней десятого пересмотра (МКБ-10), находящихся на лечении в Республиканской клинической психиатрической больнице № 1 Министерства здравоохранения Республики Башкортостан. Средний возраст больных составил 24.9 ± 8.9 лет. Средний возраст начала заболевания составил 22.4 ± 7.3 лет. Информацию по этнической принадлежности до третьего поколения получали путем опроса.
Контрольная группа состояла из 402 русских, 383 татар, 204 башкир той же возрастной группы, не состоявших на учете у психиатра и нарколога и отрицавших у себя отягощенную наследственность по психическим заболеваниям. Средний возраст здоровых доноров составил 32.4 ± 12.4 года.
Независимая выборка больных состояла из 190 индивидов (68 русской, 61 татарской и 61 башкирской этнической принадлежности).
Независимая выборка контроля состояла из 238 здоровых индивидов: 95 русских, 83 татар и 60 башкир.
ДНК выделяли из периферической крови стандартным методом фенольно-хлороформной экстракции [5].
Полногеномное генотипирование образцов ДНК было проведено на биочипе Illumina Human 610-Quad PsychChip, включавшем 610 000 однонуклеотидных полиморфных вариантов (ОНП).
Полногеномный анализ ассоциации однонуклеотидных полиморфных локусов выполнен с помощью пакета программ PLINK 2.0 [6] в Институте Брода при Гарвардском университете в рамках МКПГ [3].
Проверка качества образцов ДНК и прогенотипированных ОНП подразумевала исключение из дальнейшего анализа образцов ДНК с выявленным несоответствием между обозначенным и установленным при генотипировании полом, а также образцов ДНК, в которых более чем у 2% маркеров не прошло генотипирование. Дуплицированные образцы ДНК и образцы ДНК возможных близких родственников были выявлены и исключены на основе анализа доли идентичных аллелей у различных индивидов и доли аллелей с вероятным общим происхождением. Были исключены ОНП, по которым не прошло генотипирование более чем у 5% индивидов, ОНП с частотой редкого аллеля менее 0.01 и ОНП со статистически значимым отклонением (p = 1.0E-06) от равновесия Харди–Вайнберга. В результате проведения всех этапов контроля качества и корректировки генетической стратификации 395 832 однонуклеотидных полиморфных вариантов были включены в дальнейших анализ. Полногеномный уровень значимости для данного исследования составил p = 1.26E-07.
Для снижения ошибки 1-рода была применена поправка FDR-BH (False Discovery Rate Bengamini-Hochberg) на число множественных сравнений [7].
Выборка больных и контроля, изучаемая в настоящей работе, является генетически гетерогенной, поскольку в нее входят представители различных этнических групп (русских, татар и башкир), которые сформировались на основе различных популяций западно-евразийского и восточно-евразийского происхождения. Нами была применена поправка на этническую гетерогенность групп больных и контроля методом EIGENSTRAT [8], так как смешанное происхождение выборки, отличия по частотам аллелей полиморфных маркеров между этническими группами и различная представленность индивидов из разных этносов в выборках больных и контроля могут привести к случайной ассоциации маркеров с заболеванием.
В основе данного метода лежит вычисление главных компонент генетической изменчивости в исследуемых выборках. Установив оси генетической изменчивости выборки, обусловленные популяционной структурой, но не связанные с заболеванием, метод позволяет для каждого маркера оценить его вес в определении той или иной оси и провести тем самым индивидуальную поправку для каждого кандидатного маркера. Это минимизирует появление ложноположительных ассоциаций в силу генетической гетерогенности выборки и одновременно увеличивает вероятность определения достоверных ассоциаций.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Результаты полногеномного анализа ассоциации параноидной шизофрении представлены на рис. 1.
Наиболее высокий уровень ассоциации параноидной шизофрении был обнаружен с полиморфным вариантом rs192927334 (p = 5.99E-08; pfdr = 2.11E-03), локализованным в межгенном пространстве хромосомной области 1q23.3. В данном регионе расположен ген PBX1 на расстоянии 448 316 тпн от полиморфного локуса rs192927334 (рис. 1).
Ген PBX1 кодирует гомеодомен-содержащий белок, максимально экспрессируется в почках и головном мозге плода [9]. Известно, что белки PBX1 способны взаимодействовать с HOX-белками и рассматриваются как важные HOX кофакторы, участвующие в регуляции генов онтогенеза [10–12]. В частности, белки Prep1 и PBX1 образуют с фактором Hoxb1 тройной комплекс, регулирующий экспрессию генов в эмбриогенезе [10–13]. Было показано, что белки PBX совместно с HOX индуцируют транскрипцию гена SHH. Известно, что белок SHH существенен для развития различных тканей во время эмбриогенеза. Изучение функции SHH во время развития нервной трубки и сомитов было сфокусировано на его роли в спецификации дорсо-вентральной полярности этих структур, однако получены доказательства, что SHH выполняет дополнительные функции по выживанию и пролиферации клеток. Нарушения передачи сигналов SHH после ранней дорсовентральной спецификации краниальной части нервной трубки ведут к усилению клеточной гибели как в нервной трубке, так и нейральном гребне. Это указывает на то, что SHH постоянно необходим как трофический и митогенетический фактор во время развития мозга [14]. Нокаут как Prep1, так и PBX1 приводит к гибели мышиных эмбрионов на ранних стадиях развития [15, 16]. Снижение экспрессии PBX1 в выделенных из жировой ткани мезенхимальных стромальных клетках приводит к значительному усилению способности к дифференцировке [17].
Анализ распределения частот генотипов полиморфного локуса rs192927334 показал, что генотип rs192927334*C/C у больных ПШ встречается с более высокой частотой (98.78%), чем у индивидов контрольной группы (92.74%) (p = 8.3E-09; OR = 6.32; CI95% 3.24–12.33) (табл. 1, 2). При введении поправки на множественное сравнение для оценки доли ложноположительных результатов, проведенной с помощью метода FDR (False Discovery Rate), уровень значимости p остался статистически достоверным (pfdr = 4.68E-04) (табл. 2). Генотип rs192927334*A/C, напротив, чаще встречается в группе контроля – в 7.26%, по сравнению с 1.22% у больных. Показатель отношения шансов (ОШ) для генотипа rs192927334*A/C составил 0.16 (CI95% 0.08–0.31), p = 8.3E-09; pfdr = 5.85E-04 (табл. 2). Частота гомозиготного генотипа rs192927334*A/A составила 0.00% как у больных, так и у здоровых.
Таблица 1.
Однонуклеотидные полиморфные варианты, локализованные в области 1q23.3 и ассоциированные с параноидной шизофренией
| Ген | № rs | ОНП | Аллель 1 | Частота аллеля 1 – больные, % | Частота аллеля 1 – контроль, % | Аллель 2 | p | pfdr |
|---|---|---|---|---|---|---|---|---|
| – | rs192927334 | g.164146979C>A | A | 0.0061 | 0.0363 | C | 5.99E-08 | 2.11E-03 |
| PBX1 | rs61803803 | g.90024C>A | A | 0.0196 | 0.0363 | C | 3.03E-03 | 0.884 |
| – | rs10918018 | g.164505021T>C | C | 0.3556 | 0.3152 | T | 0.011 | 0.914 |
| – | rs10753623 | g.163744981T>C | T | 0.4492 | 0.4099 | C | 0.014 | 0.924 |
| – | rs4085003 | g.164076924C>A | C | 0.286 | 0.2518 | A | 0.019 | 0.930 |
| – | rs7530102 | g.163791020T>A | A | 0.4221 | 0.4597 | T | 0.02 | 0.929 |
| – | rs10753629 | g.163769110T>G | T | 0.4027 | 0.3666 | G | 0.021 | 0.929 |
| PBX1 | rs6672521 | g.59759A>G | G | 0.0863 | 0.1083 | A | 0.032 | 0.948 |
| – | rs6656557 | g.164209417G>A | G | 0.4725 | 0.4386 | A | 0.035 | 0.957 |
| – | rs10917897 | g.164031874G>A | G | 0.3756 | 0.4088 | A | 0.035 | 0.955 |
| – | rs1745611 | g.163686336C>T | C | 0.451 | 0.4848 | T | 0.041 | 0.960 |
| PBX1 | rs1618566 | g.83750G>A | A | 0.2506 | 0.2805 | G | 0.047 | 0.962 |
| – | rs1416261 | g.164478592C>T | T | 0.4578 | 0.424 | C | 0.047 | 0.962 |
Таблица 2.
Распределение частот генотипов и аллелей полиморфного варианта rs192927334 в выборках больных параноидной шизофренией и в контрольных группах различной этнической принадлежности
| Генотип/ аллель | Больные | Контроль | p | pfdr | OR (CI95%) | ||
|---|---|---|---|---|---|---|---|
| ni | pi ± sp, CI95% | ni | pi ± sp, CI95% | ||||
| В целом | |||||||
| A/A | 0 | – | 0 | – | – | – | – |
| A/C | 10 | 1.22 ± 0.38 0.59–2.24 |
72 | 7.26 ± 0.82 5.72–9.05 |
8.3E-09 | 5.85E-04 | 0.16 (0.08–0.31) |
| C/C | 807 | 98.78 ± 0.38 97.76–99.41 |
920 | 92.74 ± 0.82 90.95–94.28 |
8.3E-09 | 4.68E-04 | 6.32 (3.24–12.33) |
| A | 10 | 0.61 ± 0.19 0.29–1.12 |
72 | 3.63 ± 0.42 2.85–4.55 |
5.99E-8 | 2.11E-03 | 0.16 (0.08–0.31) |
| C | 1624 | 99.39 ± 0.19 98.88–99.71 |
1912 | 96.37 ± 0.42 95.45–97.15 |
5.99E-8 | 2.11E-03 | 6.12 (3.15–11.9) |
| Русские | |||||||
| A/A | 0 | – | 0 | – | – | – | – |
| A/C | 2 | 0.62 ± 0.44 0.08–2.24 |
34 | 8.46 ± 1.39 5.93–11.62 |
3.6E-06 | 0.999 | 0.07 ( 0.02–0.29) |
| C/C | 318 | 99.38 ± 0.44 97.76–99.92 |
368 | 91.54 ± 1.39 88.38–94.07 |
3.6E-06 | 0.508 | 14.69 ( 3.5–61.63) |
| A | 2 | 0.31 ± 0.22 0.04–1.12 |
34 | 4.23 ± 0.71 2.95–5.86 |
2.4E-04 | 0.999 | 0.07 ( 0.02–0.29) |
| C | 638 | 99.69 ± 0.22 98.88–99.96 |
770 | 95.77 ± 0.71 94.14–97.05 |
2.4E-04 | 0.999 | 14.09 (3.37–58.88) |
| Татары | |||||||
| A/A | 0 | – | 0 | – | – | – | – |
| A/C | 4 | 1.12 ± 0.56 0.31–2.84 |
27 | 7.05 ± 1.31 4.7–10.09 |
5.7E-05 | 0.999 | OR = 0.15 (0.05–0.43) |
| C/C | 353 | 98.88 ± 0.56 97.16–99.69 |
356 | 92.95 ± 1.31 89.91–95.3 |
5.7E-05 | 0.947 | OR = 6.69 (2.32–19.32) |
| A | 4 | 0.56 ± 0.28 0.15–1.43 |
27 | 3.52 ± 0.67 2.34–5.09 |
4.4E-04 | 0.999 | OR = 0.15 (0.05–0.43) |
| C | 710 | 99.44 ± 0.28 98.57–99.85 |
739 | 96.48 ± 0.67 94.91–97.66 |
4.4E-04 | 0.999 | OR = 6.49 (2.26–8.64) |
| Башкиры | |||||||
| A/A | 0 | – | 0 | – | – | – | – |
| A/C | 4 | 2.88 ± 1.42 0.79–7.2 |
11 | 5.42 ± 1.59 2.74–9.49 |
0.260 | 0.966 | – |
| C/C | 135 | 97.12 ± 1.42 92.8–99.21 |
192 | 94.58 ± 1.59 90.51–97.26 |
0.260 | 0.966 | – |
| A | 4 | 1.44 ± 0.71 0.39–3.64 |
11 | 2.71 ± 0.81 1.36–4.8 |
0.267 | 0.968 | – |
| C | 274 | 98.56 ± 0.71 96.36–99.61 |
395 | 97.29 ± 0.81 95.2–98.64 |
0.267 | 0.968 | – |
Примечание (для табл. 2, 4). ni – численность групп; pi – частота аллеля (генотипа).
Частота встречаемости аллеля rs192927334*A у больных ПШ была значительно ниже (0.61%), чем в контрольной группе индивидов, – 3.63% (p = 5.99E-08; pfdr = 2.11E-03). Показатель ОШ развития ПШ для аллеля rs192927334*A составил 0.16 (CI95% 0.08–0.31), для аллеля rs192927334*C – 6.12 (CI95% 3.15–11.9) (табл. 2).
Распространенность аллеля rs192927334*A у здоровых индивидов (3.63%) была сходной с таковой у индивидов европейского происхождения: финнов (3.0%), англичан (1.6%) (табл. 3).
Таблица 3.
Распределение частот аллелей полиморфного варианта rs192927334 в различных популяциях по данным проекта “1000 Геномов”
| Популяция | Аббревиатура | Частота аллеля A, % | Частота аллеля C, % |
|---|---|---|---|
| Китайцы | CDX/CHB | 0.0000 | 100.0 |
| Европейцы (север/запад) | CEU | 1.52 | 98.48 |
| Финны | FIN | 3.03 | 96.97 |
| Англичане | GBR | 1.65 | 98.35 |
| Мексиканцы | MXL | 1.56 | 98.44 |
| Африканцы | ACB | 0.0000 | 100.0 |
| Японцы | JPT | 0.0000 | 100.0 |
Учитывая этническую гетерогенность исследуемых нами выборок больных и контроля, мы также провели анализ ассоциации полиморфного локуса rs192927334, локализованного в области 1q23.3, с ПШ с учетом этнической принадлежности индивидов для оценки эффективности и достоверности проведения полногеномного анализа ассоциации в объединенной группе больных ПШ и здоровых индивидов с коррекцией на популяционную гетерогенность.
Наиболее выраженная ассоциация ПШ с ОНП rs192927334, локализованным в области 1q23.3, была выявлена у русских. Как и при анализе ассоциации объединенной группы больных и контроля, с самым высоким уровнем значимости был ассоциирован ОНП rs192927334 (табл. 2). Частота аллеля rs192927334*A у русских больных ПШ (0.31%) была значительно ниже, чем у здоровых (4.23%) (p = 2.4E-04; OR = 0.07; CI95% 0.02–0.29), однако после введения поправки FDR различия оказались статистически недостоверными (pfdr = 0.999) (табл. 2).
Анализируя ассоциацию ОНП rs192927334 с ПШ у татар, мы также обнаружили статистически значимые различия между группами больных и контроля (табл. 2). Полиморфный локус rs192927334 был ассоциирован с уровнем значимости p = 4.4E-04. Показатель ОШ для аллеля rs192927334*A, определенного с частотой 0.56% у больных и 3.52% в контроле, составил 0.15 (CI95% 0.05–0.43), однако после введения поправки FDR различия оказались статистически недостоверными (pfdr = 0.999) (табл. 2).
Аллель rs192927334*A у больных ПШ башкирской этнической принадлежности также встречался реже, чем в контрольной группе (1.44% vs 2.71%), но различия оказались не достоверны (p = 0.267; pfdr = = 0.968) (табл. 2).
В рамках проекта “1000 Геномов” было проведено генотипирование полиморфного локуса rs192927334 в ряде популяций мира (табл. 3). Частоты аллелей полиморфного локуса rs192927334 в популяциях Волго-Уральского региона схожи с таковыми у финнов (табл. 2, 3).
Tаким образом, при анализе ассоциации ОНП rs192927334 (1q23.3) с учетом этнической принадлежности индивидов было показано, что ассоциация, установленная нами с полногеномным уровнем значимости в объединенной группе больных и контроля, наблюдается с разной степенью выраженности и при анализе ассоциации в отдельных этнических группах – русских, татар и башкир, что соответствует данным других исследований, согласно которым данная хромосомная область ассоциирована с шизофренией в популяциях европеоидного и азиатского происхождения [18–25].
Для подтверждения результатов полногеномного анализа был проведен репликативный анализ ассоциации в независимой выборке (табл. 4).
Таблица 4.
Распределение частот генотипов и аллелей полиморфного варианта rs192927334 в независимой выборке больных параноидной шизофренией и в контрольных группах различной этнической принадлежности
| Генотип/аллель | Больные | Контроль | p | OR (CI95%) | ||
|---|---|---|---|---|---|---|
| ni | pi ± sp, CI95% | ni | pi ± sp, CI95% | |||
| В целом | ||||||
| A/A | 0 | – | 0 | – | – | – |
| A/C | 3 | 1.6 ± 0.92 0.33–4.62 |
15 | 6.36 ± 1.59 3.6–10.27 |
0.016 | 0.24 (0.07–0.84) |
| C/C | 184 | 98.4 ± 0.92 95.38–99.67 |
221 | 93.64 ± 1.59 89.73–96.4 |
0.016 | 4.16 (1.19–14.59) |
| A | 3 | 0.8 ± 0.46 0.17–2.33 |
15 | 3.18 ± 0.81 1.79–5.19 |
0.017 | 0.25 (0.07–0.87) |
| C | 371 | 99.2 ± 0.46 97.67–99.83 |
457 | 96.82 ± 0.81 94.81–98.21 |
0.017 | 4.06 (1.17–14.13) |
| H-W | 0.229 (0.632) | 0.254 (0.614) | ||||
| Русские | ||||||
| A/A | 0 | – | 0 | – | – | |
| A/C | 1 | 1.49 ± 1.48 0.04–8.04 |
5 | 5.32 ± 2.31 1.75–11.98 |
0.402 | |
| C/C | 66 | 98.51 ± 1.48 91.96–99.96 |
89 | 94.68 ± 2.31 88.02–98.25 |
0.402 | |
| A | 1 | 0.75 ± 0.75 0.02–4.09 |
5 | 2.66 ± 1.17 0.87–6.1 |
0.407 | |
| C | 133 | 99.25 ± 0.75 95.91–99.98 |
183 | 97.34 ± 1.17 93.9–99.13 |
0.407 | |
| Татары | ||||||
| A/A | 0 | – | 0 | – | – | |
| A/C | 1 | 1.69 ± 1.68 0.04–9.09 |
6 | 7.23 ± 2.84 2.7–15.07 |
0.239 | |
| C/C | 58 | 98.31 ± 1.68 90.91–99.96 |
77 | 92.77 ± 2.84 84.93–97.3 |
0.239 | |
| A | 1 | 0.85 ± 0.85 0.02–4.63 |
6 | 3.61 ± 1.45 1.34–7.7 |
0.245 | |
| C | 117 | 99.15 ± 0.85 95.37–99.98 |
160 | 96.39 ± 1.45 92.3–98.66 |
0.245 | |
| Башкиры | ||||||
| A/A | 0 | – | 0 | – | – | |
| A/C | 1 | 1.64 ± 1.63 0.04–8.8 |
4 | 6.78 ± 3.27 1.88–16.46 |
0.203 | |
| C/C | 60 | 98.36 ± 1.63 91.2–99.96 |
55 | 93.22 ± 3.27 83.54–98.12 |
0.203 | |
| A | 1 | 0.82 ± 0.82 0.02–4.48 |
4 | 3.39 ± 1.67 0.93–8.45 |
0.207 | |
| C | 121 | 99.18 ± 0.82 95.52–99.98 |
114 | 96.61 ± 1.67 91.55–99.07 |
0.207 | |
Распределение частот генотипов ОНП rs192927334 в объединенной независимой выборке больных и контроля различной этнической принадлежности соответствовало распределению Харди–Вайнберга (табл. 4). Частоты аллелей и генотипов ОНП rs192927334 в данной независимой выборке больных ПШ и контроля оказались сходными с таковыми в первоначально исследованных группах. Аллель rs192927334*C встречался с более высокой частотой у больных ПШ – 99.2% по сравнению с 96.82% в контроле (p = 0.017; OR = = 4.06 (CI95% 1.17–14.13) (табл. 4).
Распределение частот генотипов и аллелей полиморфного варианта rs192927334 в отдельных этнических группах русских, татар и башкир независимой выборки было схожим с таковым в первоначально исследованных группах. Однако ассоциации полиморфного локуса rs192927334 с ПШ в этнических группах русских, татар и башкир выявлено не было (табл. 4).
Таким образом, результаты репликативного исследования подтверждают данные, полученные в ходе полногеномного анализа, об ассоциации ОНП rs192927334, локализованного в хромосомной области 1q23.3, с развитием параноидной шизофрении у русских, татар и башкир.
Литературных данных, посвященных изучению ассоциации ОНП rs192927334 с параноидной шизофренией, психическими заболеваниями и другими многофакторными заболеваниями, не найдено.
Тем не менее результаты целого ряда исследований демонстрируют вовлеченность полиморфных вариантов генов данной хромосомной области в развитие шизофрении (RGS4 [20], UHMK1 [21], NOSIAP1 [26]), других психических заболеваний, нарушений нейронального развития. Так, была подтверждена вовлеченность ОНП хромосомной области 1q23-25 с развитием шизофрении у 1236 китайцев [23]. Другое полногеномное исследование выявило ассоциацию двух ОНП rs10218843 (p = 3.04E-07), rs11265461 (p = 1.94E-07) гена, кодирующего семейство белков, передающих сигнал об активации лимфоцитарной молекулы член 1 (SLAMF1), расположенных в хромосомной области 1q23.3 с резистентной к терапии шизофрении у 795 больных и 806 здоровых китайцев [24]. Кроме того, GWAS выявил ассоциацию ОНП rs1289726 (p = 2.0E-04), локализованного на расстоянии 297 тпн от гена PBX1 (1q23.3), с шизофренией у европейцев [22]. Сцепление хромосомной области 1q23 с шизофренией в семьях англичан и исландцев было продемонстрировано в ходе GWAS [18].
Была установлена ассоциация аллеля rs2275558*A гена PBX1 подверженности к обсессивно-компульсивному расстройству как в общей выборке бразильцев, так и в выборке мужчин [27].
GWAS в европейских и афро-американских популяциях подтвердил ассоциацию ОНП rs4657247 гена RGS5, лежащего в области 1q23.3, с развитием биполярного расстройства [28]. GWAS-исследование J. Namkung с соавт. [29] подтвердило ранее полученные результаты работы K. Chowdari, показавшие сцепление хромосомной области 1q23.3 с риском развития шизофрении у индусов и индивидов европейского происхождения [19], выявив ассоциацию полиморфного маркера tsc1457991-tsc1254625 гена PBX1 c алкоголизмом у 668 больных алкоголизмом и 285 здоровых индивидов, корейцев по этнической принадлежности [29]. Делеция хромосомной области 1q23.3 (1.871 Mb) приводит к синдрому врожденной аномалии почек и мочевыводящих путей CACUT, для которого характерны проявляющиеся в более позднем возрасте аутизм, шизофрения, эпилепсия, нарушения интеллекта [30–32].
Таким образом, в результате настоящего исследования нами была обнаружена ассоциация однонуклеотидного полиморфного варианта rs192927334, находящегося на расстоянии 448 316 тпн от гена PBX1 в хромосомной области 1q23.3, сцепленной с риском развития шизофрении и других психических заболеваний по данным целого ряда исследований с развитием параноидной шизофрении в трех этнических группах – русских, татар и башкир, проживающих в Республике Башкортостан. Это может свидетельствовать о вероятной вовлеченности гена PBX1 в патогенезе развития шизофрении. Особенно учитывая, что данный ген кодирует транскрипционный фактор, способствующий межбелковому взаимодействию и играющему решающую роль в целом ряде процессов развития, включая формирование структур головного мозга.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Автор выражает огромную благодарность сотрудникам департамента Психиатрической медицины и клинических нейронаук Кардиффского Университета (г. Кардифф, Великобритания) M. O’Donovan, V. Escott-Price, M. Owen, G. Leonenko за советы по генерации и анализу данных и участие в проекте, а также директору ИБГ УФИЦ РАН проф. Э.К. Хуснутдиновой за научное консультирование, экс-Главному врачу РКПБ № 1 Р.Г. Валинурову за помощь в организации забора материала в 2008–2012 гг.
Список литературы
Lam M., Chen C.Y., Li Z. et al. Comparative genetic architectures of schizophrenia in East Asian and European populations // Nat. Genet. 2019. V. 51. № 12. P. 1670–1678. https://doi.org/10.1038/s41588-019-0512-x
Bigdeli T.B., Genovese G., Georgakopoulos P. et al. Contributions of common genetic variants to risk of schizophrenia among individuals of African and Latino ancestry // Mol. Psychiatry. 2020. V. 10. № 10. P. 2455–2467. https://doi.org/1038/s41380-019-0517-y
Trubetskoy V., Pardiñas A.F., Qi T. et al. Mapping genomic loci implicates genes and synaptic biology in schizophrenia // Nature. 2022. V. 604. № 7906. P. 502–508. https://doi.org/10.1038/s41586-022-04434-5
Singh T., Poterba T., Curtis D. et al. Rare coding variants in ten genes confer substantial risk for schizophrenia // Nature. 2022. V. 604. P. 509–516. https://doi.org/10.1038/s41586-022-04556-w
Mathew C.C. The isolation of high molecular weight eucariotic DNA // Methods in Molecular Biology / Ed. Walker J.M. N.Y.: Haman Press, 1984. V. 2. P. 31–34.
Purcell S., Neale B., Todd-Brown K. et al. PLINK: A toolset for whole-genome association and population-based linkage analysis // Am. J. Hum. Genet. 2007. V. 81. № 3. P. 559–575. https://doi.org/10.1086/519795
Benjamini Y., Drai D., Elmer G. et al. Controlling the false discovery rate in behavior genetics research // Behav. Brain Res. 2001. V. 125. № 1–2. P. 279–284. https://doi.org/10.1016/s0166-4328(01)00297-2
Price A.L., Patterson N.J., Plenge R.M. et al. Principal components analysis corrects for stratification in genome-wide association studies // Nat. Genet. 2006. V. 38. № 8. P. 904–909. https://doi.org/10.1038/ng1847
Le Tanno P., Breton J., Bidart M. et al. PBX1 haploinsufficiency leads to syndromic congenital anomalies of the kidney and urinary tract (CAKUT) in humans // J. Med. Genet. 2017. V. 54. № 7. P. 502–510. https://doi.org/10.1136/jmedgenet-2016-104435
Mann R.S., Affolter M. Hox proteins meet more partners // Curr. Opin. Genet. Dev. 1998. V. 8. № 4. P. 423–429. https://doi.org/10.1016/s0959-437x(98)80113
Moens C.B., Selleri L. Hox cofactors in vertebrate development // Dev. Biol. 2006. V. 291. № 2. P. 193–206. https://doi.org/10.1016/j.ydbio.2005.10.032
Luo M., Gu X., Zhou T., Chen C. Prenatal diagnosis and molecular cytogenetic analyses of a paternal inherited deletion of 1q23.3 encompassing PBX1 gene // Mol. Cytogenet. 2022. V. 15. № 1. P. 53. https://doi.org/10.1186/s13039-022-00632-y
Ferretti E., Cambronero F., Tümpel S. et al. Hoxb1 enhancer and control of rhombomere 4 expression: Complex interplay between PREP1–PBX1–HOXB1 binding sites // Mol. Cell. Biol. 2005. V. 25. № 19. P. 8541–8552. https://doi.org/10.1128/MCB.25.19.8541-8552.2005
Takács-Vellai K., Vellai T., Chen E.B. et al. Transcriptional control of Notch signaling by a HOX and a PBX/EXD protein during vulval development in C. elegans // Dev. Biol. 2007. V. 302. № 2. P. 661–669. https://doi.org/10.1242/dev.050567
Selleri L., Depew M.J., Jacobs Y. et al. Requirement for Pbx1 in skeletal patterning and programming chondrocyte proliferation and differentiation // Development. 2001. V. 128. № 18. P. 3543–3557. https://doi.org/10.1242/dev.128.18.3543
Fernandez-Diaz L.C., Laurent A., Girasoli S. et al. The absence of Prep1 causes p53–dependent apoptosis of mouse pluripotent epiblast cells // Development. 2010. V. 137. № 20. P. 3393–3403. https://doi.org/10.1242/dev.050567
Monteiro M.C., Sanyal M., Cleary M.L. et al. PBX1: A novel stage-specific regulator of adipocyte development // Stem. Cells. 2011. V. 29. № 11. P. 1837–1848. https://doi.org/10.1002/stem.737
Gurling H.M., Kalsi G., Brynjolfson J. et al. Genome-wide genetic linkage analysis confirms the presence of susceptibility loci for schizophrenia, on chromosomes 1q32.2, 5q33.2, and 8p21–22 and provides support for linkage to schizophrenia, on chromosomes 11q23.3–24 and 20q12.1–11.23 // Am. J.Hum. Genet. 2001. V. 68. № 3. P. 661–673. https://doi.org/10.1002/stem.737
Chowdari K.V., Mirnics K., Semwal P. et al. Association and linkage analyses of RGS4 polymorphisms in schizophrenia // Hum. Mol. Genet. 2002. V. 11. № 12. P. 1373–1380. https://doi.org/10.1093/hmg/11.12.1373
Chowdari K.V., Bamne M., Wood J. et al. Linkage disequilibrium patterns and functional analysis of RGS4 polymorphisms in relation to schizophrenia // Schizophr. Bull. 2008. V. 34. № 1. P. 118–126. https://doi.org/10.1093/schbul/sbm042
Puri V., McQuillin A., Datta S. et al. Confirmation of the genetic association between the U2AF homology motif (UHM) kinase 1 (UHMK1) gene and schizophrenia on chromosome 1q23.3 // Eur. J. Hum. Genet. 2008. V. 16. № 10. P. 1275–1282. https://doi.org/10.1038/ejhg.2008.76
Need A.C., Ge D., Weale M.E. et al. A genome wide investigation of SNPs and CNVs in schizophrenia // PLoS Genet.2009. V. 5. № 2. P. e1000373. https://doi.org/10.1371/journal.pgen.1000373
Holliday E.G., McLean D.E., Nyholt D.R., Mowry B.J. Susceptibility locus on chromosome 1q23–25 for a schizophrenia subtype resembling deficit schizophrenia identified by latent class analysis // Arch. Gen. Psychiatry. 2009. V. 66. № 10. P. 1058–1067. https://doi.org/10.1001/archgenpsychiatry.2009.136
Liou Y.J., Wang H.H., Lee M.T. et al. Genome-wide association study of treatm.nt refractory schizophrenia in Han Chinese // PLoS One. 2012. V. 7. № 3. P. e33598. https://doi.org/10.1371/journal.pone.0033598
Shriebman Y., Yitzhaky A., Kosloff M., Hertzberg L. Gene expression meta-analysis in patients with schizophrenia reveals up-regulation of RGS2 and RGS16 in Brodmann Area 10 // Eur. J. Neurosci. 2023. V. 57. № 2. P. 360–372. https://doi.org/10.1111/ejn.15876
Cheah S.Y., Lawford B.R., Young R.M. et al. Association of NOS1AP variants and depression phenotypes in schizophrenia // J. Affect. Disord. 2015. V. 188. P. 263–269. https://doi.org/10.1016/j.jad.2015.08.069
Melo-Felippe F.B., Fontenelle L.F., Kohlrausch F.B. Gene variations in PBX1, LMX1A and SLITRK1 are associated with obsessive-compulsive disorder and its clinical features // J. Clin. Neurosci. 2019. V. 61. P. 180–185. https://doi.org/10.1016/j.jocn.2018.10.042
Smith E.N., Bloss C.S., Badner J.A. et al. Genome-wide association study of bipolar disorder in European American and African American individuals // Mol. Psychiatry. 2009. V. 14. № 8. P. 755–763. https://doi.org/10.1038/ejhg.2008.76
Namkung J., Kim Y., Park T. Whole-genome association studies of alcoholism with loci linked to schizophrenia susceptibility // BMC Genet. 2005. V. 6. Suppl. 1. P. S9.
Sun M., Lou J., Li Q. et al. Prenatal findings and molecular cytogenetic analyses of a de novo interstitial deletion of 1q23.3 encompassing PBX1 gene // Taiwan J. Obstet. Gynecol. 2019. V. 58. № 2. P. 292–295. https://doi.org/10.1016/j.tjog.2019.01.022
Luo M., Gu X., Zhou T., Chen C. Prenatal diagnosis and molecular cytogenetic analyses of a paternal inherited deletion of 1q23.3 encompassing PBX1 gene // Mol. Cytogenet. 2022. V. 15. № 1. P. 53. https://doi.org/10.1186/s13039-022-00632-y
Hoshina T., Seto T., Shimono T. et al. Narrowing down the region responsible for 1q23.3q24.1 microdeletion by identifying the smallest deletion // Hum. Genome Var. 2019. V. 6. P. 47. https://doi.org/10.1038/s41439-019-0079-1
Дополнительные материалы отсутствуют.


