Химическая физика, 2020, T. 39, № 6, стр. 13-17
Интермедиаты автокаталитической реакции формирования хромофора зеленого флуоресцентного белка
Б. Л. Григоренко 1, 2, М. Г. Хренова 2, 3, А. М. Кулакова 2, А. В. Немухин 1, 2, *
1 Институт биохимической физики им. Н.М. Эмануэля Российской академии наук
Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
Москва, Россия
3 Институт биохимии им. А.Н. Баха Федерального исследовательского центра “Фундаментальные основы биотехнологии” Российской академии наук
Москва, Россия
* E-mail: anemukhin@yahoo.com
Поступила в редакцию 05.06.2019
После доработки 05.06.2019
Принята к публикации 20.06.2019
Аннотация
Автокаталитическое формирование хромофора в зеленом флуоресцентном белке, активно используемом в качестве маркера для визуализации процессов в живых системах, является важным примером многостадийных реакций в белковых макромолекулах. Изучению интермедиатов элементарных стадий реакции, которые группируются в блоки циклизации и окисления, посвящены кинетические, спектральные и рентгеноструктурные экспериментальные работы и суперкомпьютерные расчеты методами молекулярного моделирования. В статье анализируются совместимые с экспериментальными данными результаты строения интермедиатов реакций формирования хромофора для нативной формы белка, полученные по данным компьютерного моделирования, с целью включения этой информации в базы данных реакционных интермедиатов ферментативных реакций.
ВВЕДЕНИЕ
Реакции химических превращений субстратов в белковых макромолекулах, в частности, реакции ферментативного катализа, как правило, проходят через последовательность элементарных стадий с накоплением и расходованием интермедиатов. Знание механизмов сложных многостадийных реакций в белках, включающее данные об интермедиатах, необходимо для управления соответствующими молекулярными процессами в биотехнологических и биомедицинских приложениях. Информацию о строении и свойствах реакционных интермедиатов получают спектральными методами и методами рентгеноструктурного анализа, хотя для этого приходится применять специальные приемы, например, внося изменения в состав изучаемого белка или субстрата. Молекулярное моделирование, основанное на компьютерных расчетах энергетических профилей реакций, дает прямую информацию об интермедиатах как о конфигурациях системы в точках минимумов на поверхности энергии. К настоящему времени объем данных об интермедиатах реакций в белках таков, что делает актуальной задачу создания соответствующих баз данных, аналогичных базам данных белковых структур (Protein Data Bank) PDB [1].
Автокаталитическое формирование хромофора в зеленом флуоресцентном белке и его производных, активно используемых в качестве маркеров для визуализации процессов в живых системах, – важный пример многостадийных химических реакций в белковых макромолекулах. По результатам кинетических, спектральных, рентгеноструктурных исследований известно, что в присутствии молекулярного кислорода фрагмент полипептидной цепи, состоящий из трех аминокислотных остатков Ser65-Tyr66-Gly67 (для нативного варианта белка – предшественника флуоресцентной формы), преобразуется в органический хромофор 4‑(п-гидроксибензилиден)-5-имидазолидон, ковалентно пришитый к белковой матрице [2]. Химические реакции, отвечающие за этот процесс, группируются в стадии циклизации и окисления (обычно из стадии циклизации еще выделяется процесс дегидратации, но для целей этой работы подобная детализация несущественна). Результаты экспериментальных работ позволяют предложить возможные структуры некоторых интермедиатов сложных молекулярных процессов формирования флуоресцентной формы белка, но не характеризовать детально эти промежуточные частицы. Более подробно весь цикл реакций формирования хромофора описан по результатам моделирования комбинированными методами квантовой и молекулярной механики (КМ/ММ) [3].
По результатам расчетов на пути реакции от исходной системы с трипептидом Ser65-Tyr66-Gly67 (называемой далее REAG) до продукта реакции – белковой молекулы с хромофором Chro (называемого далее PROD) можно выделить еще восемь интермедиатов (INT1–INT8). Анализ полученных структур модельных молекулярных систем позволяет сформулировать механизм химических превращений в хромофорсодержащей области белка, но сами эти структуры не целесообразно рассматривать как элементы базы данных интермедиатов реакций в белках. Дорогостоящие суперкомпьютерные расчеты энергетических профилей и точек локальных минимумов на поверхности энергии методами КМ/ММ требуют упрощения системы; в частности, в работах [3, 4] по моделированию строения и свойств зеленого флуоресцентного белка методами КМ/ММ только центральная часть белковой макромолекулы вокруг хромофорсодержащей области составляла модельную систему. Цель настоящей работы – построение структур REAG–INT1–INT8–PROD на пути многостадийной реакции автокаталитического формирования хромофора нативной формы зеленого флуоресцентного белка в формате, совместимом с правилами PDB, и анализ полученных структур с помощью доступных биоинформатических серверов.
МОДЕЛИ И МЕТОДЫ
Исходные координаты всех атомов (включая атомы водорода) для 10 структур, отвечающих точкам локальных минимумов на поверхности потенциальной энергии, были рассчитаны методом КМ/ММ в работе [3]. Полученные в этих расчетах области, содержащие хромофор в конечной системе PROD или соответствующие молекулярные группы в системах REAG, INT1–INT8, достроены до полной последовательности аминокислотных остатков белка от Ser2 до Ile229 (включая трипептид Ser65-Tyr66-Gly67 в начальной системе REAG). Для генерации характерного “бочонка” из β-листов зеленого флуоресцентного белка использовали программы Discovery Studio [5], основанные на методах молекулярной механики. В качестве шаблона была выбрана кристаллическая структура PDB с кодом 1EMA [6] мутанта Ser65Thr зеленого флуоресцентного белка. При каждом шаге добавления недостающих аминокислотных остатков использовали опции Discovery Studio для получения равновесных геометрических параметров. Все 10 файлов представлены в формате pdb.
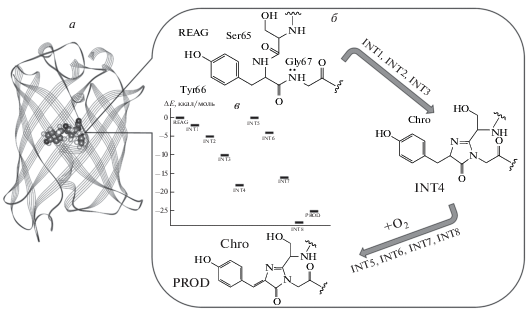
На рис. 1а изображена структура белка, а на вставке (б) – химические формулы исходного трипептида Ser65-Tyr66-Gly67 в структуре REAG, хромофора Chro в структуре PROD и промежуточного соединения в структуре одного из интермедиатов INT4, соответствующего циклизованной (и дегидратированной) форме реакционных частиц. Переход от INT4 к PROD (стадия окисления) требует добавления в систему молекулярного кислорода и проходит еще через четыре интермедиата. На центральной вставке рис. 1в показана энергетическая диаграмма всех интермедиатов, на которой за начало отсчета принят уровень REAG для циклизации или уровень INT4 для окисления.
Рис. 1.
Общий вид зеленого флуоресцентного белка (а), реакция формирования хромофора Chro из трипептида Ser65-Tyr66-Gly67 (б), положения уровней интермедиатов на энергетической шкале (в).
Формат pdb воспринимается большинством современных программных средств анализа белковых структур, в частности, доступными серверами биоинформатики. В этой работе мы используем два подобных сервера, предназначенных для визуализации структур комплексов белков с лигандами, ProteinsPlus (опция PoseView [7]) и LigPlot+ [8]. В нашем случае в качестве лиганда выступает сам хромофор или его предшественники в реакции формирования хромофора. Достоинством подобных программных средств, важным для создания баз данных, является возможность представить довольно обширные области взаимодействия лигандов с белком в виде двумерных диаграмм, построенных по трехмерным структурам. Спроектировать пространственную трехмерную структуру большой области белка, наблюдаемую, например, в программах визуализации Discovery Studio [5], на двумерную плоскость для представления в базу данных весьма непросто из-за сложности картинки.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Прежде всего представляется необходимым доказать, что результаты моделирования согласуются с экспериментом. В работе [3] показано, что энергетическая диаграмма (рис. 1в), дополненная потенциальными барьерами, разделяющими локальные минимумы, полностью соответствует кинетическим данным по константам скорости реакций циклизации и окисления [2]. Также хорошо согласуются вычисленные энергии вертикальных переходов между основным и первым возбужденным состояниями системы [2, 3] со сформированным хромофором и измеренные положения полос в оптических спектрах.
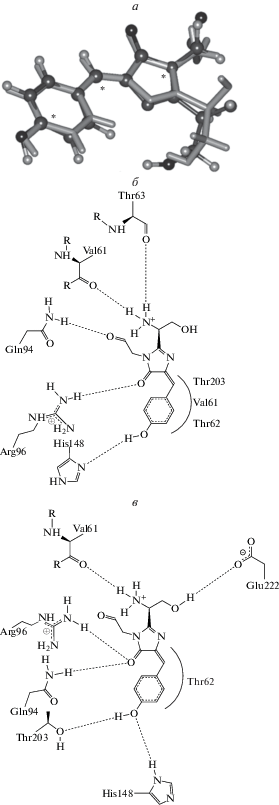
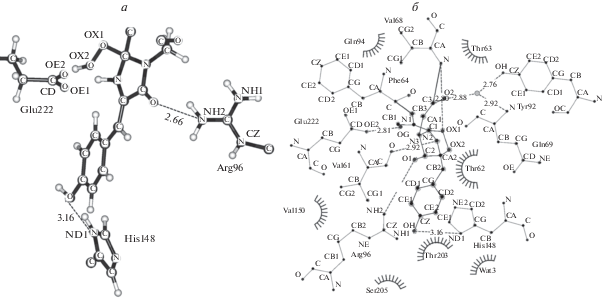
В данной работе мы приводим дополнительные аргументы в пользу результатов компьютерного моделирования. Теоретическая структура зеленого флуоресцентного белка со сформированным хромофором (PROD) может быть сопоставлена с кристаллографической структурой высокого разрешения PDB с кодом 2WUR [9]. Важно отметить, что эти экспериментальные данные не были использованы в качестве шаблона при построении теоретических структур интермедиатов. На рис. 2а показана суперпозиция структур хромофора (стержни – экспериментальные данные, шары и стержни – результаты расчетов), демонстрирующая идентичные геометрические параметры. На рис. 2б и 2в показаны двумерные диаграммы контактов хромофора с соседними аминокислотными остатками, рассчитанные программой PoseView [7], для структур PROD и PDB с кодом 2WUR соответственно. Практически одни и те же аминокислотные остатки (Val61, Thr62, Thr63, Gln94, Arg96, His148, Thr203, Glu222) формируют хромофорсодержащий “карман” в обеих структурах.
Рис. 2.
Сопоставление теоретической структуры зеленого флуоресцентного белка со сформированным хромофором (PROD) и кристаллической структуры PDB с кодом 2WUR: а – суперпозиция структур хромофора (стержни – экспериментальные данные, шары и стержни – результаты расчетов, звездочками отмечены атомы, по которым проводилось выравнивание); б и в – двумерные диаграммы контактов хромофора с соседними аминокислотными остатками, рассчитанные программой PoseView, для структур PROD и PDB с кодом 2WUR соответственно.
Двумерные карты контактов хромофоробразующих групп с ближайшими аминокислотными остатками могут меняться вдоль реакционного пути. На рис. 3 показаны подобные изменения для трех интермедиатов REAG, INT4, INT7. Эти результаты существенно дополняют данные о химических превращениях реакционных частиц.
Рис. 3.
Двумерные диаграммы контактов молекулярных групп – предшественников хромофора, с соседними аминокислотными остатками, рассчитанные программой PoseView для трех характерных структур интермедиатов на пути реакции.
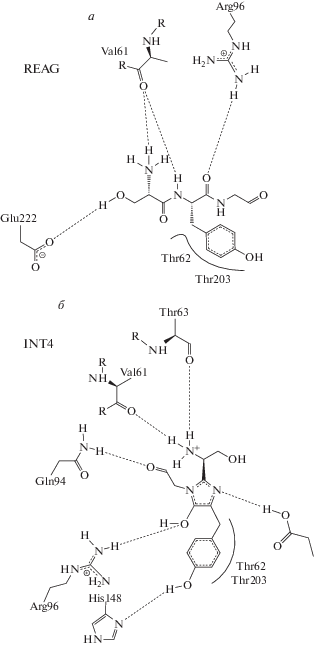
Рис. 3.
Окончание
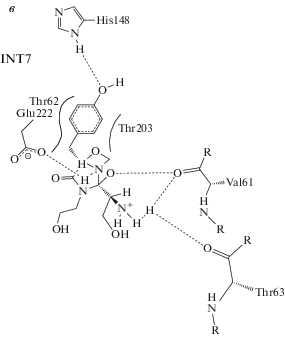
Рисунок 4 демонстрирует дополнительные возможности двумерных карт контактов лигандов с белком, рассчитываемых биоинформатическими программами. Фрагмент трехмерной структуры интермедиата INT7, доступный для изображения (рис. 4а) программой Discovery Studio [5], включает только группы формирующегося хромофора и фрагменты аминокислотных остатков Arg96, His148, Glu222. Добавление других молекулярных групп сделает рисунок слишком запутанным. Применение программы LigPlot+ [8] для этой же структуры INT7 позволяет автоматически генерировать изображение достаточно обширного фрагмента структуры лиганд-связывающей области.
Рис. 4.
а – фрагмент трехмерной структуры интермедиата INT7, доступный для изображения программой Discovery Studio, б – построенная автоматически по трехмерной структуре INT7 двумерная карта контактов лиганда в программе LigPlot+.
Таким образом, для конкретной многостадийной ферментативной реакции – реакции формирования хромофора в зеленом флуоресцентном белке, показано, каким образом можно построить набор структур интермедиатов, полученных в расчетах методами КМ/ММ, в представлении, совместимом со стандартными правилами для белковых структур, анализировать эти структуры современными биоинформатическими серверами и помещать их в базы данных.
ЗАКЛЮЧЕНИЕ
Данные о структурах интермедиатов реакции автокаталитического формирования хромофора зеленого флуоресцентного белка, полученные по результатам молекулярного моделирования, представлены в формате, совместимом с базой данных белковых структур PDB [1], что позволяет использовать для их визуализации и анализа современные серверы биоинформатики. Показано, что результаты компьютерного моделирования структуры белка с хромофором, полученным после реакций циклизации и окисления трипептида Ser-Tyr-Gly, хорошо согласуются с кристаллографической структурой высокого разрешения PDB с кодом 2WUR [9]. Двумерные проекции хромофорсодержащей области, которые построены по координатам полноатомных трехмерных структур интермедиатов, дают возможность представить детальную картину молекулярного механизма химических реакций при многостадийном формировании хромофора, а также использовать это представление в базах данных интермедиатов реакций ферментативного катализа. Другое направление, которое также планируется развивать в рамках создания базы данных – депонирование рассчитанных белковых структур, содержащих точечные мутации [10, 11].
Работа выполнена при финансовой поддержке Российским фондом фундаментальных исследований (проект № 18-29-13006).
Список литературы
Berman H.M., Westbrook J., Feng Z. et al. // Nucleic Acids Res. 2000. V. 28. P. 235; https://doi.org/10.1093/nar/28.1.235
Zimmer M. // Chem. Rev. 2002. V. 102. P. 759; https://doi.org/10.1021/cr010142r
Grigorenko B.L., Krylov A.I., Nemukhin A.V. // J. Amer. Chem. Soc. 2017. V. 139. P. 10239; https://doi.org/10.1021/jacs.7b00676
Grigorenko B.L., Nemukhin A.V., Polyakov I.V. et al. // Ibid. 2013. V. 135. P. 11541; https://doi.org/10.1021/ja402472y
Discovery Studio Visualizer v17.2.0.16349, Accelrys Software Inc.
Ormö M., Cubitt A.B., Kallio K. et al. //Science. 1996. V. 273. P. 1392; https://doi.org/10.1126/science.273.5280.1392
Stierand K., Maaß P., Rarey M. // Bioinformatics. 2006. V. 22. P. 1710; https://doi.org/10.1093/bioinformatics/btl150
Laskowski R.A, Swindells M.B. // J. Chem. Inf. Model. 2011. V. 51. P. 2778; https://doi.org/10.1021/ci200227u
Shinobu A., Palm G.J., Schierbeek A.J. et al. // J. Amer. Chem. Soc. 2010. V. 132. P. 11093; https://doi.org/10.1021/ja1010652
Метелешко Ю.И., Немухин А.В., Хренова М.Г. // Хим. физика. 2019. Т. 38. № 6. С. 1; https://doi.org/10.1039/c8pp00361k
Разникова М.О., Разников В.В. // Хим. физика. 2018. Т. 37. № 4. С. 59; https://doi.org/10.1134/S1990793118020252
Дополнительные материалы отсутствуют.
Инструменты
Химическая физика


