Кристаллография, 2021, T. 66, № 5, стр. 789-792
Компьютерное моделирование структур обратимо переключаемых флуоресцентных белков с LOV-доменами
Ю. И. Метелешко 1, М. Г. Хренова 1, 2, *, А. В. Немухин 1, 3
1 Московский государственный университет им. М.В. Ломоносова
Москва, Россия
2 Институт биохимии им. А.Н. Баха, Федеральный исследовательский центр “Фундаментальные основы биотехнологии” РАН
Москва, Россия
3 Институт биохимической физики им. Н.М. Эмануэля РАН
Москва, Россия
* E-mail: wasabiko@lcc.chem.msu.ru
Поступила в редакцию 06.08.2020
После доработки 14.09.2020
Принята к публикации 16.09.2020
Аннотация
Методами молекулярного моделирования впервые построены полноатомные трехмерные структуры новых обратимо переключаемых флавинсодержащих белков с LOV-доменами (Light, Oxygen, Voltage), используемых в микроскопии сверхвысокого разрешения. Для вариантов белков rsLOV1 и rsLOV2 известны первичные последовательности, но не пространственные модели. При построении моделей rsLOV1 и rsLOV2 за основу взята структура PDB ID: 2MWG фоторецепторного белка YtvA из Bacillus subtilis и выполнены необходимые точечные замены аминокислотных остатков. Построенные модели заключены в водные оболочки, для которых выполнены расчеты молекулярно-динамических траекторий и проведен динамический сетевой анализ. Показано, что динамические свойства нативной и мутантных структур различаются прежде всего в отношении Jα-спирали, ответственной за передачу сигнала при фотовозбуждении.
ВВЕДЕНИЕ
Интерес к флавинсодержащим белкам с LOV-доменами (Light, Oxygen, Voltage) объясняется их применением в качестве флуоресцентных маркеров в живых системах, а в последнее время – в составе оптогенетических конструкций [1, 2]. По сравнению с известными маркерами семейства зеленого флуоресцентного белка флуоресцентные белки на основе LOV-доменов обладают рядом преимуществ: небольшой размер, применимость в анаэробных условиях, возможность применения в широком диапазоне pH (от 4 до 11) [3]. В качестве хромофора большинство LOV-доменов связывает флавинмононуклеотид, широко распространенный в клетках.
Фотопереключение белков с LOV-доменом основано на реакциях образования и разрыва ковалентной связи между атомом серы консервативного аминокислотного остатка цистеина и C4a-атомом флавина. В активном, флуоресцентном состоянии “ON” связи между цистеином и флавином нет, но при освещении синим цветом (488 нм) формируется связь (Cys)S–C4a, и белок переходит в нефлуоресцентное состояние “OFF”. В природных объектах образование ковалентно-связанного аддукта запускает ряд структурных изменений белка, при которых сигнал передается белку-эффектору. При освещении УФ-светом (408 нм) аддукт распадается, и восстанавливается состояние “ON”.
Первые попытки создать обратимо фотопереключаемый маркер были предприняты для белка YtvA-LOV из Bacillus subtilis [4]. Однако слабая флуоресценция и низкая эффективность переключения во флуоресцентное состояние под действием УФ-света не способствовали практическому применению природного белка. В [5] были получены новые варианты обратимо фотопереключаемых белков, названных rsLOV1 и rsLOV2, демонстрирующие повышенную яркость и улучшенную кинетику переключения. Данные белки были синтезированы олигонуклеотид-направленным мутагенезом на основе YtvA-LOV.
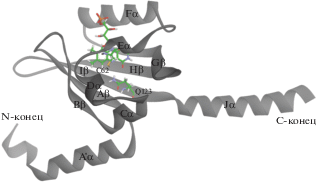
В отличие от исходного природного белка YtvA-LOV, для которого известна пространственная структура (рис. 1), для rsLOV1 и rsLOV2 ранее была известна только первичная последовательность. Знание пространственной структуры необходимо для анализа путей передачи сигнала при фотоиндуцированном возбуждении.
Рис. 1.
Структура домена YtvA-LOV, построенная на основе PDB ID: 2MWG. Стержневой моделью показаны хромофор и аминокислотные остатки Cys62 и Gln123.
Для решения задачи построения трехмерных полноатомных структур rsLOV1 и rsLOV2 в данной работе использованы современные методы молекулярного моделирования. Анализ литературы показывает, что подобные подходы могут оказаться успешными при изучении структурных изменений, происходящих в LOV-доменах [6].
МАТЕРИАЛЫ И МЕТОДЫ
Полноатомные молекулярные модели rsLOV1 и rsLOV2, мутантов YtvA-LOV, получены на основе известной структуры YtvA Bacillus subtilis (PDB ID: 2MWG). Модели построены с тем же количеством аминокислотных остатков, что и в [5]: 1-146 для YtvA-LOV и 1-142 для rsLOV1 и rsLOV2. Для получения структур rsLOV1 и rsLOV2 проведены точечные замены в соответствии с известными первичными последовательностями (рис. 2).
Рис. 2.
Выравненные аминокислотные последовательности YtvA-LOV, rsLOV1 и rsLOV2. Цветом выделены различия.

Боковые цепи аминокислот лизина и аргинина предполагались положительно заряженными, глутаминовой и аспарагиновой кислот – отрицательно заряженными. Модельные системы были сольватированы в водных оболочках размером 94 × 74 × 76 Å3 для YtvA-LOV и 91 × 74 × 76 Å3 для rsLOV1 и rsLOV2. Для нейтрализации зарядов систем были добавлены ионы натрия и хлора таким образом, чтобы ионная сила раствора составляла 0.15 М.
Молекулярно-динамические (МД) расчеты проводили с использованием программного пакета NAMD [7]. Для каждой системы рассчитано по три МД-траектории длительностью 100 нс (шаг интегрирования 1 фс). Динамику Ланжевена использовали для сохранения температуры 300 K и давления 1 атм в NPT-ансамбле. Для электростатических взаимодействий использовали алгоритм PME [8]. Энергии и силы вычисляли с силовым полем CHARMM36 [9] для белковой макромолекулы, TIP3P для молекул воды [10]. Для хромофора использовали параметры и типы атомов CGenFF [11], оптимизированные в [12]. Сетевой динамический анализ выполняли с использованием программ VMD [13] и Carma [14, 15].
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
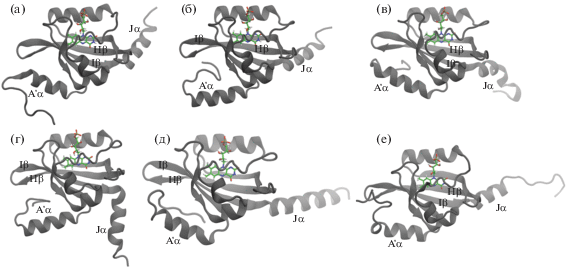
Анализ полученных МД-траекторий показывает, что самое активное движение наблюдается для А'α- и Jα-спиралей (рис. 1), на рис. 3 представлены все варианты их положения. Как видно из рисунка, А'α-спираль либо контактирует с петлей Hβ-Iβ (при этом N-концевая часть находится или под спиралью (рис. 3а), или за ней (рис. 3в)), либо находится ниже N-концевой части белка (рис. 3б, 3г, 3е). Последние два завитка Jα-спирали обладают повышенной подвижностью, причем в некоторых траекториях они стремятся потерять свою структуру (рис. 3в), а в одной траектории, для YtvA-LOV, половина Jα-спирали полностью развернулась (рис. 3е). Для rsLOV2 также наблюдалось необычное движение А'α- и Jα-спиралей навстречу друг другу (на рис. 3г показано их максимальное сближение), после чего они вернулись в обычные позиции. Отметим, что для YtvA-LOV во всех траекториях А'α-спираль располагается ниже N-концевой части белка (рис. 3д, 3е).
Рис. 3.
Характерные конфигурации из молекулярно-динамических траекторий: а, б, г – rsLOV2, в – rsLOV1, д, е – YtvA-LOV.
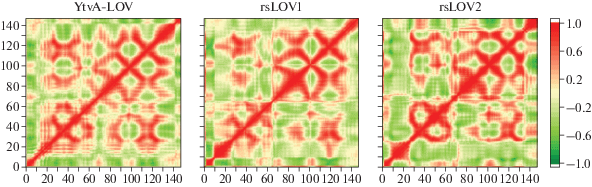
Дополнительную информацию о сходстве и различии структур исследуемых систем предоставляет сетевой динамический анализ. На рис. 4 представлены дисперсионно-ковариационные матрицы. Области, отвечающие скоррелированному движению аминокислотных остатков, отмечены более темными цветами; у rsLOV1 и rsLOV2 они шире, чем у YtvA-LOV. Существенное отличие наблюдается для rsLOV2 в области между 30-м и 62-м остатками, что, вероятно, связано с тем, что там находятся дополнительные мутации, отсутствующие у rsLOV1 (I39V, Q44N, N61S).
Рис. 4.
Дисперсионно-ковариационные матрицы. По осям отложены номера узлов сети. Узлы 0 и 1 отвечают флавину (изоалоксазиновому кольцу и рибитиловому хвосту соответственно), узлы от 2 до 148 соответствуют аминокислотам 1-146.
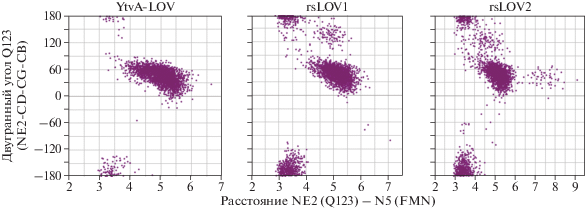
Для исследуемых систем также рассмотрены изменения, происходящие в хромофорсодержащем кармане. Самой подвижной боковой цепью в этой области обладает остаток Gln123 (рис. 1). Известно, что аналогичный остаток глутамина сохраняется во всех LOV-доменах и играет важную роль в передаче сигнала [6]. Чтобы проиллюстрировать поведение боковой цепи Gln123, были рассмотрены такие параметры, как двугранный угол NE2–CD–CG–CB и расстояние между атомом NE2 глутамина и N5 флавина (рис. 5). Из графиков видно, что для природного белка YtvA-LOV характерны расстояния от 4 до 5.5 Å и величины угла от 10° до 70° (рис. 6а). Для белков rsLOV1 и rsLOV2 наблюдается большая заселенность состояний в области около 3–3.5 Å и от 150° до –120° (рис. 6б). Также для rsLOV2 наблюдается слабо заселенное состояние с расстояниями больше 6 Å (рис. 6в), где боковая цепь Gln123 направлена в сторону Jα-спирали.
Для численного сравнения подвижности боковой цепи глутамина для каждой системы была подсчитана доля кадров в МД-траекториях, в которых расстояние NE–N5 не превышало 3.5 Å: 2.6% для YtvA-LOV, 15.9% для rsLOV1 и 13.7% для rsLOV2.
Таким образом, впервые исследованы структуры новых обратимо переключаемых белков rsLOV1 и rsLOV2 и проведено их сравнение со структурой YtvA-LOV. Выявлены отличия в поведении Gln123, находящегося в хромофорсвязывающем кармане. В белках rsLOV1 и rsLOV2 боковая цепь Gln123 обладает большей подвижностью и гибкостью, чем в YtvA-LOV.
Работа выполнена при поддержке Российского научного фонда (проект № 17-13-01051) с использованием оборудования Центра коллективного пользования сверхвысокопроизводительными вычислительными ресурсами МГУ им. М.В. Ломоносова.
Список литературы
Shcherbakova D.M., Shemetov A.A., Kaberniuk A.A. et al. // Annu. Rev. Biochem. 2015. V. 84. P. 519. https://doi.org/10.1146/annurev-biochem-060614-034411
Losi A., Gardner K.H., Möglich A. // Chem. Rev. 2018. V. 118. P. 10659. https://doi.org/10.1021/acs.chemrev.8b00163
Mukherjee A., Walker J., Weyant K.B. et al. // PLoS ONE. 2013. V. 8. P. e64753. https://doi.org/10.1371/journal.pone.0064753
Losi A., Gärtner W., Raffelberg S. et al. // Photochem. Photobiol. Sci. 2013. V. 12. P. 231. https://doi.org/10.1039/C2PP25254F
Gregor C., Sidenstein S.C., Andresen M. et al. // Sci. Rep. 2018. V. 8. P. 1. https://doi.org/10.1038/s41598-018-19947-1
Freddolino P.L., Gardner K.H., Schulten K. // Photochem. Photobiol. Sci. 2013. V. 12. P. 1158. https://doi.org/10.1039/c3pp25400c
Phillips J.C., Braun R., Wang W. et al. // J. Comput. Chem. 2005. V. 26. P. 1781. https://doi.org/10.1002/jcc.20289
Darden T., York D., Pedersen L. // J. Chem. Phys. 1993. V. 98. P. 10089. https://doi.org/10.1063/1.464397
Best R.B., Zhu X., Shim J. et al. // J. Chem. Theory Comput. 2012. V. 8. P. 3257. https://doi.org/10.1021/ct300400x
Jorgensen W.L., Madura J.D. // J. Am. Chem. Soc. 1983. V. 105. P. 1407. https://doi.org/10.1021/ja00344a001
Vanommeslaeghe K., Hatcher E., Acharya C. et al. // J. Comput. Chem. 2010. V. 31. P. 671. https://doi.org/10.1002/jcc.21367
Khrenova M.G., Meteleshko Y.I., Nemukhin A.V. // J. Phys. Chem. B. 2017. V. 121. P. 10018. https://doi.org/10.1021/acs.jpcb.7b07533
Humphrey W., Dalke A., Schulten K. // J. Mol. Graph. 1996. V. 14. P. 33. https://doi.org/10.1016/0263-7855(96)00018-5
Glykos N.M. // J. Comput. Chem. 2006. V. 27. P. 1765. https://doi.org/10.1002/jcc.20482
Koukos P.I., Glykos N.M. // J. Comput. Chem. 2013. V. 34. P. 2310. https://doi.org/10.1002/jcc.23381
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография