Кристаллография, 2021, T. 66, № 5, стр. 697-707
Структурные исследования РНК-белковых комплексов в пострибосомную эру
С. В. Тищенко 1, А. О. Михайлина 1, Н. В. Леконцева 1, Е. А. Столбоушкина 1, Е. Ю. Никонова 1, О. С. Никонов 1, А. Д. Никулин 1, *
1 Институт белка РАН
Пущино, Россия
* E-mail: nikulin@vega.protres.ru
Поступила в редакцию 15.09.2020
После доработки 02.10.2020
Принята к публикации 02.10.2020
Аннотация
Структурные исследования РНК-белковых комплексов являются важным этапом в понимании многих молекулярных механизмов в клетке, например, таких как регуляция белкового синтеза и РНК-шаперонная активность белков. В обзоре рассмотрены различные объекты, исследующиеся в Институте белка РАН. На основе анализа структур комплексов рибосомного белка L1 со специфическими участками на мРНК и рРНК представлены принципы, по которым осуществляется регуляция белком трансляции мРНК своего оперона. Описаны исследования гетеротримерного фактора инициации трансляции IF2 архей и эукариот, приведены данные о взаимодействии глицил-тРНК-синтетазы с IRES вирусов. Приведены результаты исследований взаимодействия молекул РНК с одним из функционально важных участков белка Hfq и выявлены различия в РНК-связывающих свойствах Hfq и архейных белков семейства Lsm.
ОГЛАВЛЕНИЕ
Введение
1. Регуляторные комплексы рибосомного белка L1 с мРНК
2. Определение структуры и исследования РНК-связывающих свойств глобального регулятора трансляции – белка Hfq
3. Исследования РНК-связывающих свойств архейных Lsm белков
4. Исследование функции регуляторов транскрипции UxuR и ExuR
5. Структурные и функциональные исследования глицил-тРНК-синтетазы человека
6. Структурные исследования гетеротримерного фактора инициации трансляции 2
Заключение
ВВЕДЕНИЕ
Определение структур бактериальных рибосом с помощью методов рентгеноструктурного анализа и криоэлектронной микроскопии на рубеже ХХ и ХХI веков стало прорывным событием в исследованиях процесса биосинтеза белка в клетках. Важной стартовой частью этого проекта стало определение структур отдельных компонентов рибосомы, а именно, рибосомных белков и их комплексов с фрагментами рибосомной РНК. Они позволили ускорить интерпретацию получаемых огромных карт электронной плотности рибосом, а также описать подвижные участки рибосомных субчастиц, которые не видны на структурах целых рибосом. В Институте белка РАН (ИБ РАН) найдены условия кристаллизации 70S рибосом и 30S рибосомных субчастиц Thermus thermophilus, определены структуры более пятнадцати рибосомных белков и ряда рРНК-белковых комплексов, что внесло существенный вклад в реализацию масштабного “рибосомного проекта”. Разработан комплексный подход к определению структур РНК-белковых комплексов, включающий в себя рациональный дизайн фрагментов РНК, образующих наиболее стабильные комплексы с белками, кристаллизацию этих комплексов, получение дифракционных данных на лабораторной установке Proteum (Bruker-AXS) и на источниках синхротронного излучения BESSY, DESY, ESRF, MAX IV с последующим определением структур методами многоволновой аномальной дифракции и мультидоменного молекулярного замещения. Накопленный опыт по структурно-функциональным исследованиям рРНК-белковых комплексов позволил перейти к решению новых задач, связанных с изучением целого ряда регуляторных комплексов. Исследования регуляции трансляции и транскрипции в клетках на атомарном уровне позволяют находить ответы на новые вопросы, стоящие перед научным сообществом. В предлагаемом обзоре представлены краткие результаты наиболее интересных работ сотрудников ИБ РАН в этом направлении.
1. РЕГУЛЯТОРНЫЕ КОМПЛЕКСЫ РИБОСОМНОГО БЕЛКА L1 С МРНК
Рибосомный двухдоменный белок L1 является составной частью L1-выступа большой субчастицы рибосомы; у прокариот этот белок также регулирует трансляцию белков своего оперона по принципу обратной связи. В E. coli белок L1 связывается с участком мРНК перед первым геном L11 оперона [1], в экстремально-термофильных бактериях Thermus thermophilus и Thermotoga maritima имеются два участка связывания на мРНК [2], в археях Methanococcus vannielii и Methanococcus jannaschii регуляторный участок на мРНК располагается в начале кодирующей последовательности первого гена L1 оперона [3, 4]. Исследование регуляторных свойств рибосомного белка L1 явилось естественным продолжением работ по изучению структурной основы биосинтеза белка.
Белок L1 – единственный рибосомный белок, регуляторные свойства которого детально исследованы на основе сравнительного анализа структур комплексов L1-рРНК и L1-мРНК. Для получения кристаллизуемых препаратов комплекса L1-мРНК применена стратегия, разработанная в ИБ РАН при исследовании структуры комплекса рибосомного белка S8 со специфическим фрагментом 16S рРНК [5]. Были получены 10 фрагментов мРНК длиной от 36 до 49 нуклеотидов, специфически связывающихся с белком L1. Менялась длина и нуклеотидный состав двух спиралей, фланкирующих ассиметричную петлю, содержащую консервативные нуклеотиды. Верхняя полноразмерная спирально-петельная часть фрагментов мРНК, не содержащая консервативных нуклеотидов, в укороченных вариантах была заменена четырехнуклеотидной петлей UUCG [6]. Семь фрагментов представляли собой различные вариации L1-связывающего фрагмента мРНК M. vannielii, три фрагмента являлись вариациями специфического участка мРНК M. jannaschii. Высокую мозаичность кристаллов удалось понизить добавлением солей ртути в кристаллизационную каплю. Были определены структуры трех комплексов: комплекса L1-мРНК (49 нт) из M. jannaschii (разрешение 3.4 Å [7]) и двух гетерологичных комплексов, образованных белком L1 T. thermophilus и короткими фрагментами мРНК M. vannielii, длиной 38 (разрешение 2.6 Å [8]) и 36 нт (разрешение 2.1 Å [9]).
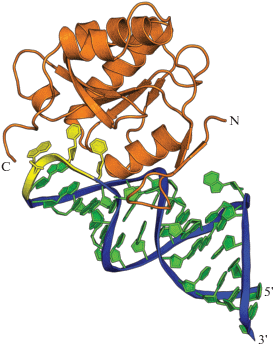
Белок L1 в перечисленных комплексах контактировал преимущественно с атомами сахарофосфатного остова мРНК. Сравнение структур рибосомного [10, 11] и полученных регуляторных комплексов белка L1 показало, что белок образует сеть недоступных растворителю водородных связей с мотивом, содержащим строго консервативные нуклеотиды и расположенным в месте сочленения двух спиралей, структурно инвариантном как на мРНК, так и на рРНК. Со стороны белка L1 в этом взаимодействии участвуют расположенные рядом петли домена I (рис. 1).
Рис. 1.
Наложение структур комплексов рибосомного белка L1 с фрагментами мРНК длиной 49 нуклеотидов (серый, PDB ID 1U63), 38 нуклеотидов (белый, PDB ID 1ZHO) и 36 нуклеотидов (черный, PDB ID 2HW8). Указаны 5´- и 3´-концы РНК, домены I и II белка L1.
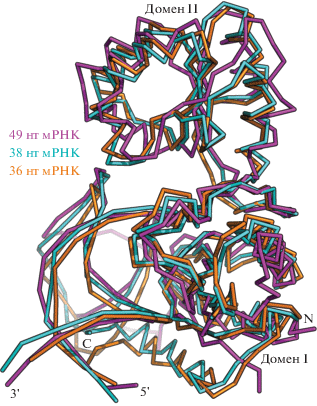
Для исследования особенностей взаимодействия белка L1 со специфическими фрагментами мРНК и рРНК применяли комбинированный структурно-функциональный подход, добавив к структурному анализу метод поверхностного плазмонного резонанса, который позволяет наблюдать кинетику взаимодействия молекул в реальном времени [12]. Специфический фрагмент мРНК отличается от L1-связывающего фрагмента рРНК сильно укороченными участками, соответствующими спирали 78 и петле Б рибосомной РНК (рис. 2). Это приводит к понижению сродства белка L1 к мРНК на несколько порядков по сравнению с комплексом L1-рРНК.
Рис. 2.
Сравнение структур комплексов рибосомного белка L1 с фрагментом 23S рРНК (PDB ID 1MZP) (а) и белка L1 с мРНК (PDB ID 2HW8) (б). Указаны элементы структур РНК и белка.
Основная консервативная область контакта белка L1 расположена на его домене I и может быть разделена на два участка: один из них включает в себя консервативную триаду Thr-Met-Gly, образующую закрытые от растворителя водородные связи с нуклеотидами спирали H77 рРНК [13], другой – N-концевую часть спирали α1, взаимодействующую с нуклеотидами спирали H78 [14]. В комплексах белка L1 с мРНК и рРНК оба участка взаимодействия на домене I имеют одинаковые консервативные водородные связи, однако количество неконсервативных контактов в комплексе с мРНК уменьшается, что значительно снижает сродство белка к матричной РНК. Был получен домен I белка L1 в изолированном виде и показано, что он достаточен для взаимодействия как с рРНК, так и с мРНК [15, 16], а также обладает регуляторными свойствами целого белка [17]. Аминокислотные остатки домена II белка в рибосомном комплексе образуют дополнительные контакты с рРНК, что еще больше увеличивает стабильность рибосомного комплекса. Такое комплексное исследование комплексов белка L1 с мРНК и рРНК выявило причину разницы в сродстве белка к двум разным мишеням и позволило раскрыть основу регуляции трансляции рибосомным белком L1 по механизму обратной связи.
2. ОПРЕДЕЛЕНИЕ СТРУКТУРЫ И ИССЛЕДОВАНИЯ РНК-СВЯЗЫВАЮЩИХ СВОЙСТВ ГЛОБАЛЬНОГО РЕГУЛЯТОРА ТРАНСЛЯЦИИ – БЕЛКА HFQ
РНК-связывающий бактериальный белок Hfq принадлежит к обширному семейству Sm/Lsm белков и в бактериях выполняет роль многофункционального белка-регулятора трансляции [18]. Это небольшой белок размером ∼10 кДа, формирующий в растворе четвертичную структуру в виде гексамера [18]. В 1979 г. белок Hfq был идентифицирован как хозяйский фактор, необходимый для репликации (+)-цепи РНК бактериофага Qβ [19]. В дальнейшем было выявлено, что делеция гена hfq в клетках Escherichia coli приводит к изменению продукции более чем 30 различных белков, включая белки репарации ДНК [20], вовлеченные в метаболизм железа [21] и синтез σs-субъединицы бактериальной РНК-полимеразы [22]. В большинстве случаев регуляция трансляции белком Hfq реализуется посредством транс-кодируемых малых регуляторных РНК (мрРНК; в английской литературе используют аббревиатуру sRNA). Белок Hfq входит в когорту так называемых “РНК-шаперонов”, включающих в себя такие бактериальные регуляторные белки, как CsrA, ProQ, KhpA, KhpB, SpoVG, способствующие взаимодействию мрРНК со своими мишенями на мРНК [23]. Этот способ регуляции позволяет контролировать адаптацию бактерий к изменению внешней среды за короткое время. Hfq – наиболее универсальный белок среди РНК-шаперонов и обнаружен в большинстве бактерий. Он стимулирует взаимодействие мрРНК с комплементарными участками на мРНК с последующей репрессией или стимуляцией трансляции, а также регулирует время жизни мРНК за счет взаимодействия с их поли(А)3'-концевыми участками.
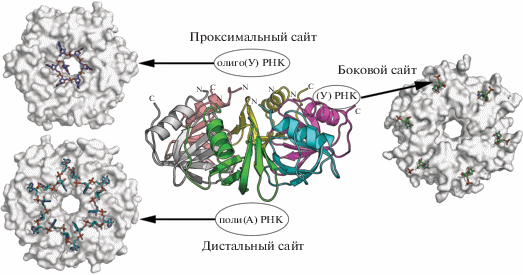
В начале 2000 г. были получены первые структуры белка из Staphylococcus aureus [24], укороченной формы белка из E. coli [25] и полноразмерного белка из Pseudomonas aeruginosa [26], что позволило предложить ряд гипотез о том, как белок способствует взаимодействию двух разных РНК. Были идентифицированы два основных участка связывания РНК на поверхности гексамера белка: уридин-специфический участок на “проксимальной” поверхности белка (со стороны N-концов мономеров белка) и аденин-специфический на противоположной, “дистальной”, поверхности (рис. 3).
Рис. 3.
Расположение участков взаимодействия белка Hfq с РНК. В центре – вид на гексамер белка с боковой стороны. Указаны N- и С-концы мономеров белка. Выделены области связывания РНК: уридин-специфический проксимальный сайт, аденин-специфический дистальный сайт и один из шести уридин-специфических боковых сайтов. Слева вверху показана структура комплекса белка Hfq с 5'-GUUUUUA-3' РНК [25] (вид с проксимальной стороны гексамера; PDB ID 1KQ2), слева внизу – структура комплекса Hfq с олиго(А) РНК [68] (вид со стороны дистального сайта; PDB ID 3GIB), справа посередине – структура комплекса Hfq с УМФ [28] (вид со стороны проксимального сайта; PDB ID 4J6X). Молекулы РНК и нуклеотидов показаны шаростержневой моделью на поверхности гексамеров белка Hfq.
Структуры комплексов Hfq с короткими фрагментами РНК показали детали узнавания оснований РНК аминокислотными остатками белка и объяснили его высокую специфичность к определенным последовательностям РНК. Интересно, что для разрушения вторичной структуры РНК белком Hfq не требуется дополнительной энергии в виде расщепления АТФ или ГТФ, однако ряд биохимических данных позволял предположить наличие АТФазной активности белка [27]. Мы получили структуру комплекса белка Hfq с АТФ и, исходя из особенностей контактов АТФ с аминокислотными остатками белка, сделали вывод, что вероятность специфической АТФазной активности Hfq крайне мала. Параллельно проверили возможность других одиночных нуклеотидов связываться с поверхностью белка и оказалось, что Hfq обладает высоким сродством к УТФ и ЦТФ, но не связывает ГТФ. Положение связанных нуклеотидов и ориентация их оснований полностью соответствовали таковым в структурах комплексов Hfq с олигонуклеотидами [28]. На основе этих наблюдений была предложена методика картирования участков связывания одноцепочечных РНК на поверхности РНК-связывающих белков [29, 30], которая в дальнейшем использовалась при исследовании гомологичных архейных Lsm белков. Более того, удалось идентифицировать третий, “латеральный” участок взаимодействия белка Hfq с РНК (рис. 3). Он расположен на боковой поверхности гексамера белка в области N-концевой спирали мономера и, как оказалось, является местом встречи и взаимодействия двух РНК – мрРНК и мРНК. Данное открытие было сделано одновременно с идентификацией этого участка биохимическими методами, что подтвердило достоверность наших результатов [31–34]. Таким образом, мы впервые показали на структурном уровне, как происходит фиксация цепи РНК с последующим ее взаимодействием с белковым партнером. Эти данные внесли существенный вклад в дальнейшее развитие модели взаимодействия мрРНК с мРНК с участием белка Hfq и процессов регуляции трансляции, опосредованное белками РНК-шаперонами.
3. ИССЛЕДОВАНИЯ РНК-СВЯЗЫВАЮЩИХ СВОЙСТВ АРХЕЙНЫХ LSM БЕЛКОВ
Архейные Lsm белки (или SmAP – Sm Archaeal Proteins), как и бактериальные белки Hfq, принадлежат к семейству Sm/Lsm и имеют аналогичную третичную структуру из пяти-тяжевого β-листа с N-концевой α-спиралью [35]. Геномы различных видов архей могут кодировать от одного до трех паралогов SmAP белков, классифицируемых как SmAP1, SmAP2 и SmAP3. Интересно, что SmAP1 и SmAP2 из клеток одного вида имеют меньшую степень гомологии, чем SmAP1 (или SmAP2) из клеток разных организмов. Однако все они имеют строгий консенсус по аминокислотной последовательности RGXX (где Х – положительно заряженный аминокислотный остаток) в отличие от консенсуса YKHАI бактериальных белков Hfq (рис. 4а). Кроме этого, в отличие от большинства бактериальных белков Hfq архейные SmAP белки имеют удлиненную петлю L4, соединяющую тяжи β3 и β4, и лишены протяженной неупорядоченной С-концевой части. Наиболее заметное отличие SmAP от бактериальных гомологов – формирование ими четвертичной структуры в виде гомогептамеров (за редким исключением), а не гомогексамеров (рис. 4б). Этим свойством они ближе к своим эукариотическим гомологам – белкам Sm/Lsm, которые участвуют в процессинге РНК в клетках и, как правило, входят в состав различных РНП, таких как сплайсосома. В отличие от бактериальных и эукариотических гомологов функции SmAP в архейных клетках не установлены. Имеются данные, что они связываются с рядом “незрелых” РНК, а также с белками, участвующими в процессинге РНК в клетках [36–39], что указывает на их возможную вовлеченность в созревание РНК, однако прямых доказательств функциональной роли SmAP белков пока не имеется.
Рис. 4.
Выравнивание аминокислотных последовательностей представителей белков Hfq (Hfq_Eco – E. coli, Hfq_Pae – P. aeruginosa, Hfq_Sty – Salmonella typhimurium, Hfq_Bsu – Bacillus subtilis), SmAP (SmAP1_Mva – Methanococcus vannielii, SmAP1_Sso – Sulfolobus solfataricus, SmAP1-Afu – Archeoglobus fulgidus) и эукариотических Lsm из Saccharomyces cerevisiae. Внизу показаны элементы вторичной структуры, соответствующие аминокислотным последовательностям белков. Выравнивание сделано в программе T-Coffee с учетом информации по 3D-структурам белков (Espresso) [69] (а). Схематическое представление структур гексамерного бактериального белка Hfq из P. aeruginosa (слева, PDB ID 1U1S) и гептамерного архейного белка SmAP из M. vannielii (справа, PDB ID 5MKI). Вид со стороны α-спиралей мономеров белков (проксимальной стороны) (б).
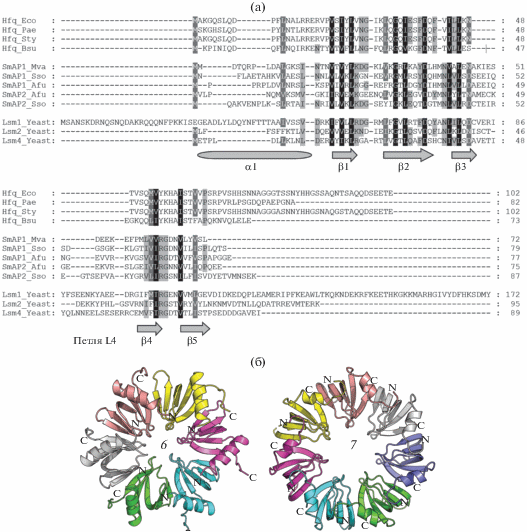
Задачей проводимых исследований стало определение РНК-связывающих и РНК-шаперонных свойств белков SmAP с целью определения возможной функции этих белков в археях. Для этого применили описанную выше технику идентификации РНК-связывающих участков на поверхности SmAP белков, определив ряд структур комплексов белков с рибонуклеотидами. Полученные данные показали, что SmAP белки имеют только один, уридин-специфический РНК-связывающий сайт на проксимальной стороне гомогептамера [40, 41]. Таким образом, сложно предположить, что SmAP белки могут связывать две разные молекулы РНК и участвовать в регуляции трансляции посредством мрРНК. Однако, как было показано ранее, SmAP белки могут разрушать структуру целевой РНК без участия второй, комплементарной, РНК, что потенциально позволяет этим белкам взаимодействовать с мРНК, изменять ее пространственную структуру и таким образом влиять на процесс трансляции. Следовательно, SmAP белки нельзя исключать из числа возможных регуляторов трансляции в археях, но для полного выяснения их функции требуются дополнительные исследования.
4. ИССЛЕДОВАНИЕ ФУНКЦИИ РЕГУЛЯТОРОВ ТРАНСКРИПЦИИ UXUR И EXUR
Экспрессия генов регулируется не только на уровне трансляции, но и на уровне транскрипции. Одним из примеров этого является регуляция метаболического процесса усвоения D-гексуроновых кислот, таких как D-глюкуронат и D-галактуронат (метаболический путь Эшвелла) [42, 43]. В бактериях этот путь является альтернативой наиболее известному гликолитическому пути метаболизма и играет заметную роль для выживания в стрессовых условиях. Процессы утилизации гексуронатов находятся под контролем двух тесно взаимосвязанных регуляторов – UxuR и ExuR [44, 45]. Эти белки являются паралогами, так как ExuR, по-видимому, появился в результате дупликации гена uxuR [46], и относятся к семейству регуляторов GntR. К настоящему времени получены пространственные структуры нескольких белков этого семейства [46, 47], однако степень их гомологии с UxuR и ExuR составляет не более 25% [48]. Целью данной работы стало определение структур этих двух белков и их комплексов с регуляторными областями ДНК, в том числе в присутствии лигандов – глюкороната и галакторуната. Это позволило бы на молекулярном уровне описать регуляцию экспрессии генов, вовлеченных в метаболизм гексуронатов.
Получение белков в препаративных количествах было осложнено высокой склонностью белков UxuR и ExuR к агрегации и их токсичностью для клеток-продуцентов, поэтому пришлось тщательно подбирать условия их получения [49]. Эксперименты по определению сродства белков к целевым участкам ДНК подтвердили наличие двух различающихся участков связывания UxuR и ExuR, причем присутствие сахаров меняло сродство белков к ДНК. Нестабильность белков в условиях кристаллизации не позволила получить кристаллы белков в свободном состоянии и в комплексах с лигандами и/или фрагментами ДНК, поэтому структуры таких комплексов промоделированы с помощью методов гибкого докинга на основе структур гомологичных белков [50]. Это позволило локализовать предположительный участок связывания сахаров в области С-концевых доменов UxuR, совпадающий с местом, предсказанным ранее по данным мутагенеза.
Применение современных методов молекулярного моделирования и молекулярной динамики позволяет получить интересные данные по взаимодействию молекул, однако только определение структур комплексов может подтвердить достоверность расчетных моделей.
5. СТРУКТУРНЫЕ И ФУНКЦИОНАЛЬНЫЕ ИССЛЕДОВАНИЯ ГЛИЦИЛ-ТРНК-СИНТЕТАЗЫ ЧЕЛОВЕКА
Энтеровирусы, а также некоторые родственные им вирусы инициируют синтез своих белков в зараженной эукариотической клетке посредством кэп-независимого механизма. В этом случае специфическая вторичная структура определенного участка вирусной мРНК (IRES) делает возможной сборку трансляционного аппарата непосредственно в месте, близком к старт-кодону. В отличие от кэп-зависимой инициации трансляции механизмы IRES-зависимой инициации весьма разнообразны. IRES из различных источников могут быть абсолютно не похожи друг на друга: не найдено элемента структуры, общего для всех IRES, или какой-либо заметной гомологии, общей для всех их последовательностей. Такое разнообразие вызвало определенные трудности классификации IRES c момента их открытия. Однако интенсивные исследования вирусов, их мРНК и механизмов ее трансляции позволили к настоящему моменту выделить ряд общих черт, позволяющих вполне определенно отличать одни типы IRES от других.
Большинство вирусных IRES обладают стабильной вторичной и третичной структурами, способствующими их эффективному связыванию с 40S субчастицей [51]. Инициация трансляции на всех исследованных на сегодняшний день IRES типа I зависит от наличия ITAF (англ: IRES trans-acting factors, представляют собой специальные факторы трансляции), список которых до сих пор полностью не определен. Одним из ITAFs, необходимых для эффективной инициации трансляции полиовирусной мРНК, является глицил-тРНК-синтетаза человека (GlyRS). Этот белок взаимодействует с апикальной частью домена V IRES полиовируса, которая мимикрирует под антикодоновую шпильку глициновой тРНК и содержит глициновый антикодон. GlyRS имеет трехдоменную структуру, при этом за узнавание глицинового антикодона отвечает отдельный домен, называемый ABD (Anticodon Binding Domein). К настоящему моменту определены структуры GlyRS как в свободной форме, так и в комплексе с глициновой тРНК, что позволило детально изучить механизм узнавания белком антикодоновой петли тРНК [52, 53], однако структура комплекса GlyRS с вирусной мРНК пока не определена.
Ранее были получены фрагменты домена V IRES первого типа (IRES I) и определен минимальный фрагмент, взаимодействующий с GlyRS [54]. Также было показано, что глицил-тРНК-синтетаза человека специфически связывает фрагменты IRES I вирусов, таксономически удаленных друг от друга, с близкими константами. Это указывает на универсальность механизма регуляции инициации трансляции при помощи глицил-тРНК-синтетазы для таких вирусов и на схожесть пространственных структур соответствующих участков их мРНК [55].
Сродство глицил-тРНК-синтетазы человека к фрагментам IRES I примерно в 3 раза превышает сродство к родственной тРНК [55], что дает основания предполагать схожий механизм взаимодействия белка и РНК в обоих случаях. В отличие от тРНК «антикодоновая петля» в составе апикальной части пятого домена IRES содержит не пять, а шесть нуклеотидов. Пространственная структура V домена IRES I до сих пор неизвестна. Однако удалось построить теоретическую модель этого фрагмента и его комплекса с ABD доменом глицил-тРНК-синтетазы человека (рис. 5), основываясь на имеющейся структурной информации [56]. Модель имеет повышенное (по сравнению с гомологичной частью tRNAGly) число водородных связей с белком, что позволяет объяснить полученные нами биохимические данные.
6. СТРУКТУРНЫЕ ИССЛЕДОВАНИЯ ГЕТЕРОТРИМЕРНОГО ФАКТОРА ИНИЦИАЦИИ ТРАНСЛЯЦИИ 2
У бактерий фактор инициации трансляции 2 (IF2) является шестидоменным мономерным белком, который способствует связыванию инициаторной аминоацил-тРНК с малой рибосомной субчастицей. У архей и эукариот фактор инициации трансляции 2 (e/aIF2) состоит из трех различных субъединиц: α, β, γ. Функционально он аналогичен бактериальному IF2, однако ни одна из субъединиц e/aIF2 не имеет гомологии с бактериальным IF2. Субъединицы эукариотического eIF2 по размеру больше, чем гомологичные им субъединицы архейного aIF2. В присутствии ГТФ гетеротримерный фактор e/aIF2 специфически узнает инициаторную Met-тРНКi и образует с ней тройной комплекс e/aIF2–GTP–Met-тРНКi. В такой форме e/aIF2 доставляет Met-тРНКi в P-участок малой рибосомной субчастицы, где за счет кодон-антикодонового взаимодействия происходит точное узнавание инициирующего кодона на мРНК с последующим гидролизом ГТФ. В комплексе с ГДФ фактор инициации трансляции IF2 покидает рибосому, после чего происходит обмен связанного ГДФ на ГТФ, и фактор снова способен образовывать прочный комплекс с Met-тРНКi. Таким образом, фактор инициации трансляции IF2 выполняет очень важную функцию в клетке: запускает синтез белка на рибосоме.
Впервые структура гетеротримерного фактора инициации трансляции IF2 была определена в лаборатории ИБ РАН [57] и до настоящего времени она остается единственной кристаллической структурой полноразмерного фактора инициации трансляции eIF2 эукариотического типа. Структура aIF2 была определена из термофильной археи Sulfolobus solfataricus и в дальнейшем использовалась для построения модели β-субъединицы эукариотического фактора eIF2 в составе пространственной структуры дрожжевого 48S преинициаторного комплекса (48S PIC), которая исследована методом криоэлектронной микроскопии [58]. Общими очертаниями гетеротримерный фактор инициации трансляции 2 напоминает английскую букву L и состоит из конформационно-стабильной центральной части и двух подвижных “крыльев” [57]. Минимальной и достаточной для связывания инициаторной Met-тРНКi является конформационно-стабильная центральная часть aIF2 – γ-субъединица с третьим доменом α-субъединицы aIF2αD3γ.
Практически одновременно были расшифрованы две кристаллические структуры тройного комплекса aIF2–ГТФ–Met-тРНКi с использованием архейного фактора инициации трансляции IF2. В 2012 г. французская группа ученых получила кристаллы тройного комплекса, содержащего полноразмерный гетеротримерный фактор aIF2αβγ, однако построить и уточнить модель для подвижной β-субъединицы не удалось в силу недостаточно ясной электронной плотности [59]. В 2013 г. в лаборатории ИБ РАН были получены кристаллы тройного комплекса, содержащего коровую конформационно-стабильную часть фактора aIF2αD3γ [60]. Обе структуры тройного комплекса дали понимание того, как фактор aIF2 отличает инициаторную тРНК от элонгаторной тРНК. Инициаторная тРНК обращена к белку aIF2 своей D-стороной, тогда как фактор элонгации EF-Tu, структурно схожий с aIF2γ, взаимодействует с элонгаторной тРНК по Т-шпильке [61]. Таким образом, различия в нуклеотидной последовательности T-шпильки инициаторной и элонгаторных тРНК могут предотвращать связывание инициаторной тРНК с EF-Tu, а различия в D-шпильке могут предотвращать связывание элонгаторных тРНК с aIF2.
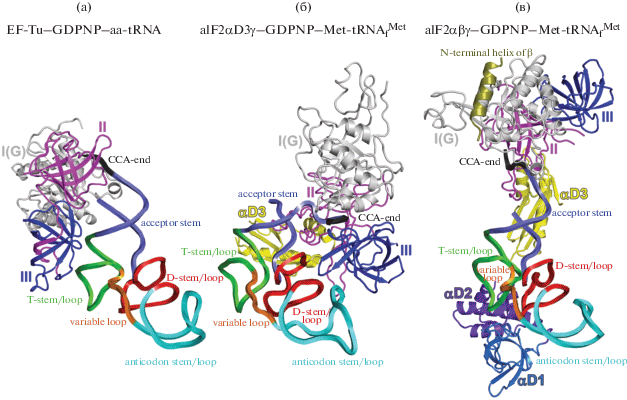
При сравнении двух структур тройного комплекса aIF2–ГТФ–Met-тРНКi было обнаружено, что несмотря на то, что в обеих структурах тРНК обращена к белку своей D-стороной, взаимодействует она с aIF2 несколько по-разному (рис. 6). В структуре тройного комплекса, полученной нашей группой, акцепторный стебель тРНК образует контакты со всеми тремя доменами γ-субъединицы и aIF2αD3: CCA-конец тРНК локализован в расщелине между G(I)- и III-доменами γ-субъединицы, II-домен γ-субъединицы aIF2 взаимодействует с 5′-концом Met-тРНКi, а белок aIF2αD3 контактирует с локтем тРНК [60]. В модели aIF2–ГТФ–Met-тРНКi, полученной группой французских коллег [59], акцепторный стержень тРНК не взаимодействует с III-доменом γ-субъединицы, а локоть тРНК контактирует с aIF2αD1 и aIF2αD2, которые согласно биохимическим данным проявляют неспецифическую РНК-связывающую активность [62, 63].
Рис. 6.
Сравнение структур комплексов тРНК с EF-Tu (PDB ID 1TTT, слева), aIF2αD3γ (PDB ID 3QSY, в центре) и aIF2αβγ (PDB ID 3V11, справа).
В сотрудничестве с лабораторией И. Шатского (НИИ ФХБ МГУ) в экспериментах in vitro показано, что архейный фактор aIF2 способен поддерживать рибосомное сканирование [64]. С помощью метода тоупринтинга выявили, что если добавить архейный фактор aIF2 в систему инициации трансляции млекопитающих, в которой отсутствует его эукариотический гомолог, то 48S инициаторный комплекс образуется, и идет сканирование мРНК в поисках инициирующего AUG-кодона. Эта совместная работа раскрыла особенность второй стадии инициации трансляции – присоединение большой субчастицы к 48S инициаторному комплексу. На этом этапе в работу включаются дополнительные факторы: eIF5 и eIF5B (промотируют присоединение большой субчастицы). У архей aIF5 отсутствует, но мы добавляли eIF5 в систему трансляции с архейным вторым фактором, и оказалось, что если заменить eIF2 на aIF2, то 80S рибосомный комплекс не образуется. Этот результат демонстрирует, что в археях способ стимуляции ГТФазной активности aIF2 отличается от того, который реализуется в эукариотах.
Дальнейшие наши исследования в области архейного фактора aIF2 привели к тому, что был определен ряд пространственных структур γ-субъединицы aIF2, представляющих разные функциональные состояния этой рибосомной ГТФазы: от активного (ON) ГТФ-связанного до неактивного (OFF) ГДФ-связанного состояния, проходя через промежуточные формы, когда белок может быть связан одновременно с ГДФ и неорганическим фосфатом Pi или не содержать нуклеотида вовсе [65]. Сравнение структур впервые продемонстрировало смещение доменов II и III относительно домена G(I), хотя и не столь значительное как в его структурном гомологе, факторе элонгации трансляции EF-Tu. Скоординированные перестройки происходят и в петлях-переключателях Switch 1 и 2. Петля Switch 1 играет роль “застежки” для нуклеотид-связывающего кармана. Обнаружено, что конформация этого переключателя не позволяет полностью закрыть карман в aIF2γ в отличие от EF-Tu. Он остается открытым на протяжении всего цикла работы aIF2γ, и γ-фосфату ничто не препятствует покинуть белок после гидролиза ГТФ. Это в свою очередь указывает на возможность спонтанного гидролиза ГТФ у фактора aIF2 на рибосоме и объясняет отсутствие у архей фактора aIF5, стимулирующего ГТФазную активность aIF2.
При анализе структуры aIF2γ, нуклеотид-связывающий карман которой находится в состоянии интермедиата при переходе из ГТФ- в ГДФ-связанную форму, были обнаружены три кластера молекул воды, образующие водные мостики между γ-фосфатом ГТФ и двумя консервативными остатками Asp19 и Asp93 [66]. Эти водные мостики могут быть использованы для переноса протона, образующегося при каталитической реакции – гидролизе ГТФ.
Кроме этого, выявлена дополнительная петля (третья) у γ-субъединицы aIF2, которая может работать как переключатель во время гидролиза ГТФ [67]. В результате изменения конформации этой петли растворитель (молекулы воды) получает доступ к γ-фосфату ГТФ. Главным привратником на пути к γ-фосфату ГТФ является Met45. Интересно, что у эукариотической γ-субъединицы eIF2 вместо Met45 расположена более крупная аминокислота – изолейцин, и для открытия “ворот” требуется приложить усилия, чтобы не встретить стерические препятствия, что приводит к необходимости использовать белковый фактор eIF5, индуцирующий гидролиз ГТФ. А у фактора элонгации трансляции EF-Tu и вовсе отсутствует эта петля. В совокупности эти и изложенные выше данные лишний раз указывают на вероятность отсутствия ГТФаза-активирующего фактора у архей и спонтанного гидролиза ГТФ у фактора aIF2.
ЗАКЛЮЧЕНИЕ
Накопленный в Институте белка РАН опыт структурно-функциональных исследований РНК-белковых комплексов в рамках “рибосомного проекта” успешно использован при выполнении ряда исследований принципов регуляции трансляции и транскрипции функционально важными объектами. Среди них исследование авторегуляции трансляции универсально-консервативного рибосомного белка L1 в бактериях и археях, регулятора трансляции Hfq и его архейных гомологов, регуляторов транскрипции UxuR и ExuR, исследование дополнительной функции глицил-тРНК-синтетазы человека, изучение гетеротримерного фактора инициации трансляции IF2.
В обзоре кратко представлены результаты проведенных исследований. На молекулярном уровне показана природа различного сродства рибосомного белка L1 к специфически узнаваемым участкам мРНК и рРНК, что позволяет регулировать трансляцию своего оперона по принципу обратной связи. Для белка Hfq выявлены структурные основы узнавания молекул РНК в функционально важном боковом РНК-связывающем участке белка. Исследованы РНК-связывающие свойства ряда архейных белков семейства Lsm и показаны существенные отличия их РНК-связывающих свойств по сравнению с бактериальным гомологом – белком Hfq. Получены структурные данные о взаимодействии глицил-тРНК-синтетазы с IRES вирусов. Проведены обширные комплексные исследования гетеротримерного фактора инициации трансляции IF2 архей и эукариот.
Особую благодарность авторы выражают родоначальникам структурно-функциональных исследований в Институте белка РАН – Марии Борисовне Гарбер, Станиславу Владимировичу Никонову и Наталии Александровне Невской.
Работа выполнена при поддержке следующих фондов: проект “Регуляторные комплексы рибосомного белка L1 с мРНК” – Российский фонд фундаментальных исследований (РФФИ) (гранты № 03-04-48327, 04-04-49634, 05-04-48338, INTAS YSF № 04-83-3842); проект “Исследования РНК-связывающих свойств белка Hfq” – РФФИ (гранты № 04-04-48556, 07-04-00296, 13-04-00783); проект “Исследования РНК-связывающих свойств архейных Lsm белков” – Российский научный фонд (РНФ) (грант № 14-14-00496), РФФИ (грант № 18-04-00222); проект “Исследование функции регуляторов транскрипции UxuR и ExuR” – РНФ (грант № 18-14-00322); проект “Структурные и функциональные исследования глицил-тРНК-синтетазы человека” – РНФ (грант № 15-14-00028), РФФИ (грант № 19-34-90135); проект “Структурные исследования гетеротримерного фактора инициации трансляции IF2” – РФФИ (грант № 18-04-01331).
Список литературы
Gourse R.L., de Boer H.A., Nomura M. // Cell. 1986. V. 44. № 1. P. 197. https://doi.org/10.1016/0092-8674(86)90498-8
Михайлина А.О., Костарева О.С., Никонова Е.Ю. и др. // Молекулярная биология. 2018. Т. 52. № 1. С. 98. https://doi.org/10.7868/S0026898418010135
Mayer C., Köhrer C., Gröbner P., Piendl W. // Mol. Microbiol. 1998. V. 27. № 2. P. 455. https://doi.org/10.1046/j.1365-2958.1998.00693.x
Hanner M., Mayer C., Köhrer C. et al. // J. Bacteriol. 1994. V. 176. № 2. P. 409. https://doi.org/10.1128/jb.176.2.409-418.1994
Tishchenko S.V., Vassilieva J.M., Platonova O.B. et al. // Biochem. 2001. V. 66. № 9. P. 948. https://doi.org/10.1023/a:1012353122174
Ennifar E., Nikulin A., Tishchenko S. et al. // J. Mol. Biol. 2000. V. 304. № 1. P. 35. https://doi.org/10.1006/jmbi.2000.4204
Nevskaya N., Tishchenko S., Gabdulkhakov A. et al. // Nucl. Acids Res. 2005. V. 33. № 2. P. 478. https://doi.org/10.1093/nar/gki194
Nevskaya N., Tishchenko S., Volchkov S. et al. // J. Mol. Biol. 2006. V. 355. № 4. P. 747. https://doi.org/10.1016/j.jmb.2005.10.084
Tishchenko S., Nikonova E., Nikulin A. et al. // Acta Cryst. D. 2006. V. 62. (Pt. 12). P. 1545. https://doi.org/10.1107/S0907444906041655
Nikulin A., Eliseikina I., Tishchenko S. et al. // Nat. Struct. Biol. 2003. V. 10. № 2. P. 104. https://doi.org/10.1038/nsb886
Tishchenko S., Gabdulkhakov A., Nevskaya N. et al. // Acta Cryst. D. 2012. V. 68. (Pt. 8). P. 1051. https://doi.org/10.1107/S0907444912020136
Kostareva O., Tishchenko S., Nikonova E. et al. // J. Mol. Recognit. 2011. V. 24. № 4. P. 524. https://doi.org/10.1002/jmr.1063
Tishchenko S., Kostareva O., Gabdulkhakov A. et al. // Acta Cryst. D. 2015. V. 71. (Pt. 2). P. 376. https://doi.org/10.1107/S1399004714026248
Костарева О.С., Невская Н.А., Тищенко С.В. и др. // Молекулярная биология. 2018. Т. 52. № 1. С. 106. https://doi.org/10.7868/S0026898418010147
Tishchenko S., Nikonova E., Kljashtorny V. et al. // Nucl. Acids Res. 2007. V. 35. № 21. P. 7389. https://doi.org/10.1093/nar/gkm898
Tishchenko S., Kljashtorny V., Kostareva O. et al. // J. Mol. Biol. 2008. V. 383. № 2. P. 301. https://doi.org/10.1016/j.jmb.2008.08.058
Korepanov A.P., Kostareva O.S., Bazhenova M.V. et al. // Protein J. 2015. V. 34. № 2. P. 103. https://doi.org/10.1007/s10930-015-9602-5
Updegrove T.B., Zhang A., Storz G. // Curr. Opin. Microbiol. 2016. V. 30. P. 133. https://doi.org/10.1016/j.mib.2016.02.003
de Fernandes M.T.F., Hayward W.S., August T.J. // J. Biol. Chem. 1972. V. 247. № 3. P. 824.
Schuppli D., Miranda G., Tsui H.C. et al. // Proc. Natl. Acad. Sci. U. S. A. 1997. V. 94. № 19. P. 10239. https://doi.org/10.1073/pnas.94.19.10239
Vecerek B., Moll I., Afonyushkin T. et al. // Mol. Microbiol. 2003. V. 50. № 3. P. 897. https://doi.org/10.1046/j.1365-2958.2003.03727.x
Muffler A., Traulsen D.D., Fischer D. et al. // J. Bacteriol. 1997. V. 179. № 1. P. 297. https://doi.org/10.1128/JB.179.1.297-300.1997
Jørgensen M.G., Pettersen J.S., Kallipolitis B.H. // Biochim. Biophys. Acta – Gene Regul. Mech. 2020. V. 1863. № 5. P. 194504. https://doi.org/10.1016/j.bbagrm.2020.194504
Schumacher M.A., Pearson R.F., Møller T. et al. // EMBO J. 2002. V. 21. № 13. P. 3546. https://doi.org/10.1093/emboj/cdf322
Sauter C., Basquin J., Suck D. // Nucl. Acids Res. 2003. V. 31. № 14. P. 4091. https://doi.org/10.1093/nar/gkg480
Nikulin A., Stolboushkina E., Perederina A. et al. // Acta Cryst. D. 2005. V. 61. (Pt. 2). P. 141. https://doi.org/10.1107/S0907444904030008
Arluison V., Mutyam S.K., Mura C. et al. // Protein Sci. 2007. V. 16. № 9. P. 1830. https://doi.org/10.1110/ps.072883707
Murina V., Lekontseva N., Nikulin A. // Acta Cryst. D. 2013. V. 69. (Pt. 8). P. 1504. https://doi.org/10.1107/S090744491301010X
Nemchinova M., Balobanov V., Nikonova E. et al. // Protein J. 2017. V. 36. № 3. P. 157. https://doi.org/10.1007/s10930-017-9709-y
Balobanov V., Lekontseva N., Mikhaylina A., Nikulin A. // Methods Mol. Biol. 2020. V. 2113. P. 251. https://doi.org/10.1007/978-1-0716-0278-2_17
Chen J., Morita T., Gottesman S. // Front. Cell. Infect. Microbiol. 2019. V. 9. № 6. P. 201. https://doi.org/10.3389/fcimb.2019.00201
Sauer E., Schmidt S., Weichenrieder O. // Proc. Natl. Acad. Sci. 2012. V. 109. № 24. P. 9396. https://doi.org/10.1073/pnas.1202521109
Robinson K.E., Orans J., Kovach A.R. et al. // Nucl. Acids Res. 2013. V. 42. № 4. P. 2376. https://doi.org/10.1093/nar/gkt1171
Panja S., Schu D.J., Woodson S. // Nucl. Acids Res. 2013. V. 41. № 15. P. 7536. https://doi.org/10.1093/nar/gkt521
Мурина В.Н., Никулин А.Д. // Успехи биологической химии. 2011. Т. 51. С. 133.
Fischer S., Benz J., Spath B. et al. // J. Biol. Chem. 2010. V. 285. № 45. P. 34429. https://doi.org/10.1074/jbc.M110.118950
Maier L.K., Benz J., Fischer S. et al. // Biochimie. 2015. V. 117. P. 129. https://doi.org/10.1016/j.biochi.2015.02.023
Märtens B., Bezerra G., Kreuter M. et al. // Life. 2015. V. 5. № 2. P. 1264. https://doi.org/10.3390/life5021264
Märtens B., Hou L., Amman F. et al. // Nucl. Acids Res. 2017. V. 45. № 13. P. 7938. https://doi.org/10.1093/nar/gkx437
Nikulin A., Mikhailina A., Lekontseva N. et al. // J. Biomol. Struct. Dyn. 2017. V. 35. № 8. P. 1615. https://doi.org/10.1080/07391102.2016.1189849
Lekontseva N., Mikhailina A., Fando M. et al. // Biochimie. 2020. V. 175. P. 1. https://doi.org/10.1016/j.biochi.2020.05.001
Ashwell G. // Methods Enzymol. 1962. V. 5. P. 190. https://doi.org/10.1016/S0076-6879(62)05205-2
Peekhaus N., Conway T. // J. Bacteriol. 1998. V. 180. № 14. P. 3495. https://doi.org/10.1128/JB.180.14.3495-3502.1998
Ritzenthaler P., Blanco C., Mata-Gilsinger M. // Mol. Gen. Genet. 1985. V. 199. № 3. P. 507. https://doi.org/10.1007/BF00330766
Utz C.B., Nguyen A.B., Smalley D.J. et al. // J. Bacteriol. 2004. V. 186. № 22. P. 7690. https://doi.org/10.1128/JB.186.22.7690-7696.2004
Suvorova I.A., Tutukina M.N., Ravcheev D.A. et al. // J. Bacteriol. 2011. V. 193. № 15. P. 3956. https://doi.org/10.1128/JB.00277-11
Fillenberg S.B., Friess M.D., Korner S. et al. // PLoS One 2016. V. 11. № 6. P. e0157691. https://doi.org/10.1371/journal.pone.0157691
Tutukina M.N., Potapova A.V., Vlasov P.K. et al. // J. Biomol. Struct. Dyn. 2016. V. 34. № 10. P. 2296. https://doi.org/10.1080/07391102.2015.1115779
Bessonova T.A., Lekontseva N.V., Shvyreva U.S. et al. // Protein Expr. Purif. 2019. V. 161. P. 70. https://doi.org/10.1016/j.pep.2019.05.001
Пуртов Ю.А., Тутукина М.Н., Никулин А.Д., Озолинь О.Н. // Биофизика. 2019. Т. 64. № 1. С. 061. https://doi.org/10.1134/S0006350919010160
Nikonov O.S., Chernykh E.S., Garber M.B., Nikonova E.Y. // Biochemistry (Moscow). 2017. V. 82. № 13. P. 1615. https://doi.org/10.1134/S0006297917130041
Qin X., Hao Z., Tian Q. et al. // J. Biol. Chem. 2014. V. 289. № 29. P. 20359. https://doi.org/10.1074/jbc.M114.557249
Qin X., Deng X., Chen L., Xie W. // J. Mol. Biol. 2016. V. 428. № 18. P. 3603. https://doi.org/10.1016/j.jmb.2016.05.018
Никонова Е.Ю., Михайлина А.О., Леконцева Н.В. и др. // Биофизика. 2016. Т. 61. № 2. С. 277. https://doi.org/10.1134/S0006350916020135
Никонова Е.Ю., Михайлина А.О., Немчинова М.С. и др. // Молекулярная биология. 2018. Т. 52. № 1. С. 10. https://doi.org/10.7868/S0026898418010020
Никонов О.С., Немчинова М.С., Кляшторный В.Г. и др. // Молекулярная биология. 2018. Т. 52. № 1. С. 112. https://doi.org/10.7868/S0026898418010159
Stolboushkina E., Nikonov S., Nikulin A. et al. // J. Mol. Biol. 2008. V. 382. № 3. P. 680. https://doi.org/10.1016/j.jmb.2008.07.039
Llácer J.L., Hussain T., Marler L. et al. // Mol. Cell. 2015. V. 59. № 3. P. 399. https://doi.org/10.1016/j.molcel.2015.06.033
Schmitt E., Panvert M., Lazennec-Schurdevin C. et al. // Nat. Struct. Mol. Biol. 2012. V. 19. № 4. P. 450. https://doi.org/10.1038/nsmb.2259
Stolboushkina E., Nikonov S., Zelinskaya N. et al. // J. Mol. Biol. 2013. V. 425. № 6. P. 989. https://doi.org/10.1016/j.jmb.2012.12.023
Nissen P., Kjeldgaard M., Thirup S.r. et al. // Science. 1995. V. 270. № 5241. P. 1464. https://doi.org/10.1126/science.270.5241.1464
Yatime L., Schmitt E., Blanquet S., Mechulam Y. // J. Biol. Chem. 2004. V. 279. № 16. P. 15984. https://doi.org/10.1074/jbc.M311561200
Pedulla N. // Nucl. Acids Res. 2005. V. 33. № 6. P. 1804. https://doi.org/10.1093/nar/gki321
Dmitriev S.E., Stolboushkina E.A., Terenin I.M. et al. // J. Mol. Biol. 2011. V. 413. № 1. P. 106. https://doi.org/10.1016/j.jmb.2011.08.026
Nikonov O., Stolboushkina E., Arkhipova V. et al. // Acta Cryst. D. 2014. V. 70. (Pt. 3). P. 658. https://doi.org/10.1107/S1399004713032240
Nikonov O., Kravchenko O., Arkhipova V. et al. // Biochimie. 2016. V. 121. P. 197. https://doi.org/10.1016/j.biochi.2015.11.029
Nikonov O., Kravchenko O., Nevskaya N. et al. // Acta Cryst. D. 2019. V. 75. (Pt. 4). P. 392. https://doi.org/10.1107/S2059798319002304
Link T.M., Valentin-Hansen P., Brennan R.G. // Proc. Natl. Acad. Sci. USA. 2009. V. 106. № 46. P. 19292. https://doi.org/10.1073/pnas.0908744106
Notredame C., Higgins D.G., Heringa J. // J. Mol. Biol. 2000. V. 302. № 1. P. 205. https://doi.org/10.1006/jmbi.2000.4042
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография