Кристаллография, 2022, T. 67, № 2, стр. 266-268
Влияние мутаций V23Q и K24T на подвижность атомов молекулы L-аспарагиназы Wolinella succinogenes
В. И. Тимофеев 1, 2, Н. Е. Жухлистова 1, И. П. Куранова 1, 2, *
1 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
2 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
* E-mail: inna@crys.ras.ru
Поступила в редакцию 30.12.2020
После доработки 26.01.2021
Принята к публикации 26.01.2021
- EDN: FGYCOC
- DOI: 10.31857/S0023476122020266
Аннотация
Методом молекулярной динамики исследована динамика молекулы L-аспарагиназы Wolinella succinogenes дикого типа и ее мутантной формы (замены V23Q и K24T). Проведен анализ полученных молекулярно-динамических траекторий. Показано, что подвижность атомов мутантной формы белка существенно выше подвижности атомов белка дикого типа. Выявлены элементы структуры белка, подвижность которых наиболее сильно меняется при введении мутаций. С помощью полученных данных можно объяснить изменение субстратной специфичности мутантной формы L-аспарагиназы Wolinella succinogenes.
ВВЕДЕНИЕ
Аспарагиназы весьма распространены среди микроорганизмов и катализируют гидролиз аспарагина до аспарагиновой кислоты и аммиака [1]. Кроме того, аспарагиназы способны катализировать гидролиз глутамина [2]. Известно, что аспарагиназы обладают противоопухолевой активностью, так как опухолевые клетки не способны синтезировать аспарагин, необходимый для их жизнедеятельности [3]. В связи с глутаминазной активностью противоопухолевый эффект аспарагиназ сопровождается побочными эффектами, кроме того, аспарагиназы относительно нестабильны, что затрудняет их применение в качестве лекарственных средств [4, 5].
Аспарагиназа из Wolinella succinogenes интересна тем, что отличается относительно низкой глутаминазной активностью и поэтому наиболее пригодна для создания медицинского препарата [6]. На основе аспарагиназы из Wolinella succinogenes получена мутантная форма фермента, обладающая на порядок более низкой глутаминазной активностью, а также устойчивостью к действию трипсина [7]. Достичь этого удалось путем введения мутаций V23Q, K24T в N-концевую подвижную петлю молекулы аспарагиназы Wolinella succinogenes дикого типа. Чтобы исследовать влияние данных замен на свойства фермента, были установлены кристаллические структуры апо-формы мутантной формы фермента и его комплексов с аспарагиновой и глутаминовой кислотами [8]. Однако анализ полученных структур показал, что часть аминокислотных остатков подвижной петли, в том числе подвергшиеся заменам V23Q, K24T, не локализованы на картах электронной плотности. Поэтому было сделано предположение, что данные замены увеличивают подвижность N-концевой петли в мутантной форме фермента, что затрудняет образование каталитически активной закрытой конформации активного центра в присутствии глутамина, субстрата большего объема [8].
В настоящей работе методом молекулярной динамики показано, что введение замен V23Q, K24T действительно существенным образом повышает подвижность N-концевой петли, а также ряда других участков молекулы мутантной формы аспарагиназы Wolinella succinogenes.
МАТЕРИАЛЫ И МЕТОДЫ
Для построения первоначальной модели использовали кристаллическую структуру апо-формы аспарагиназы Wolinella succinogenes (PDB ID: 5K4H) [9]. Из структуры были удалены молекулы лигандов и молекулы воды. Для получения модели мутантной формы фермента использовали программу PyMol [10]. Моделирование молекулярной динамики проведено с использованием программного комплекса GROMACS 2020.2 [11]. В качестве силового поля использовано поле CHARMM27 [12]. Белок каждой исследуемой формы помещали в центр прямоугольной ячейки, весь объем ячейки заполняли водой; была использована трехатомная модель воды – TIP3P. На первой стадии проводили минимизацию энергии систем. Далее провели уравновешивание в NVT- и NPT-ансамблях 100 нс в каждом для обеих систем. Величина температуры выбрана равной 300 К, величина давления – 1 атм. Использовали следующие алгоритмы поддержания температуры и давления: термостат V-rescale [13] и баростат Parrinello-Rahman [14] соответственно. Системы промоделированы на временном интервале общей продолжительностью 20 нс каждая с шагом 2 фс.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
По результатам моделирования белков методом молекулярной динамики после применения структурного выравнивания траекторий к исходному положению для дикого типа аспарагиназы Wolinella succinogenes и ее мутантной формы рассчитаны RMSF (Root Mean Square Fluctuation) атомов, т.е. флуктуации координат атомов, усредненные по всему времени моделирования (20 нс), которые удобно использовать для оценки подвижности атомов белка в различных условиях. Известно, что молекула аспарагиназы Wolinella succinogenes в растворе существует в виде гомотетрамера, причем это относится как к дикой, так и мутантной форме фермента [8, 9]. Таким образом, для получения статистически достоверных значений для анализа использовали усредненные по четырем мономерам значения RMSF. Кроме того, рассчитаны средние значения RMSF для Cα-атомов дикого типа белка и его мутантной формы. Для дикой формы это значение составило 0.0616 нм, а для мутантной – 0.0649 нм, что означает увеличение подвижности атомов мутантной формы аспарагиназы более чем на 5%. На рис. 1 изображены RMSF для каждого из Cα-атомов молекулы аспарагиназы дикого типа и мутантной формы. Значения усреднены по четырем величинам, соответствующим RMSF для каждой из четырех аминокислотных цепей.
Рис. 1.
RMSF Сα-атомов дикой формы аспарагиназы Wolinella succinogenes (темная линия) и ее мутантной формы (светлая линия). Значения усреднены по четырем величинам, соответствующим RMSF для каждой из четырех аминокислотных цепей.
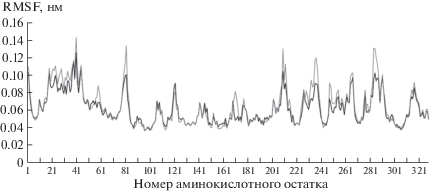
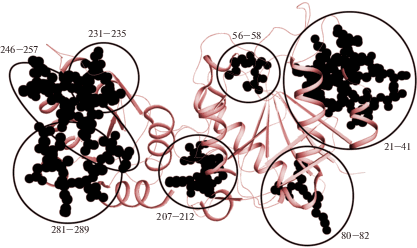
На рис. 1 видно, что участок с 20-го по 40-й аминокислотный остаток существенно более подвижен в случае мутантной формы белка, а именно этот участок и представляет собой фрагмент N-концевой петли. Отметим, что существенная разница в подвижности Cα-атомов наблюдается для аминокислотной последовательности 56–61, причем для 58-го остатка RMSF для дикой формы составляет 0.0691 нм, в то время как для мутантной формы – 0.0877 нм. Следующий существенно различающийся участок – аминокислотные остатки 80–82, где RMSF для 81-го остатка дикой формы составляет 0.1004 нм, а для мутантной формы – 0.1336 нм. Существенная разница наблюдается и для фрагмента, включающего остатки 207–212, причем для 209-го остатка дикой формы белка значение RMSF составляет 0.1048 нм, а для мутантной формы – 0.1298 нм. Кроме того, существенная разница в подвижности атомов выявлена на участках аминокислотной последовательности 231–235, 246–257 и 281–289. Взаимное расположение указанных фрагментов в субъединице белка представлено на рис. 2. Из рисунка видно, что разница в подвижности аминокислотных остатков в составе молекул аспарагиназы дикой и мутантной формы наблюдается не только для N-концевой петли и непосредственно прилегающих к ней участков, но и для удаленных от последней участков молекулы белка.
Рис. 2.
Субъединица молекулы аспарагиназы Wolinella succinogenes. Сферами показаны атомы аминокислотных остатков, наиболее различающихся по подвижности для дикой формы и для мутантной формы фермента. Кластеры аминокислотных остатков обведены черными кругами. Диапазоны аминокислотных остатков, входящих в кластер, обозначены цифрами.
ЗАКЛЮЧЕНИЕ
В результате моделирования молекулы аспарагиназы методом молекулярной динамики показано, что замены V23Q, K24T влияют на подвижность атомов белка не только непосредственно на участке, где проведены мутации, но и на удаленных участках молекулы. Таким образом, подтверждается высказанное ранее предположение [8] об увеличении подвижности N-концевой петли в мутантной форме фермента. Кроме того, обнаружен ряд других дополнительных фрагментов молекулы белка, для которых подвижность атомов в мутантной форме также существенно увеличилась. Одной из причин может быть изменение электростатического потенциала поверхности в результате введения замен аминокислотных остатков [8]. Полученные результаты показывают, что введенные замены V23Q, K24T могут оказывать влияние и на поведение удаленных друг от друга участков молекулы аспарагиназы.
Работа выполнена при поддержке Российского фонда фундаментальных исследований (проект № 19-04-01148) в части расчета молекулярной динамики и при поддержке Министерства науки и высшего образования РФ в рамках Государственного задания ФНИЦ “Кристаллография и фотоника” РАН в части анализа результатов моделирования.
Список литературы
Howard J.B., Carpenter F.J. // J. Biol. Chem. 1972. V. 217. P. 1020. https://doi.org/10.1016/S0021-9258(19)45610-X
Lubkowski J., Wlodawer A., Ammon H.L. et al. // Biochemistry. 1994. V. 33. P. 10257. https://doi.org/10.1021/bi00200a005
Broom J.D. // J. Exp. Med. 1968. V. 127. P. 1055. https://doi.org/10.1084/jem.127.6.1055
Warrell R.P., Chou T.C., Gordon C. et al. // Cancer Res. 1980. V. 40. P. 4546.
Parmentier J.H., Maggi M., Tarasco E. et al. // Leuk. Res. 2015. V. 39. P. 757. https://doi.org/10.1016/j.leukres.2015.04.008
Durden D.L., Distasio J.A. // Cancer Res. 1980. V. 40. P. 1125.
Sannikova E.P., Bulushova N.V., Cheperegin S.E. et al. // Mol. Biotechnol. 2016. V. 58. P. 528. https://doi.org/10.1007/s12033-016-9950-1
Тимофеев В.И., Жухлистова Н.Е., Куранова И.П. // Биоорг. химия. 2020 Т. 46. № 2. С. 140. https://doi.org/10.31857/S0132342320020165
Nguyen H.A., Durden D.L., Lavie A. // Sci. Rep. 2017. V. 7. P. 41643. https://doi.org/10.1038/srep41643
Schrodinger L.L.C. // The PyMOL Molecular Graphics System. Version 1.8. 2015.
Abraham M.J., Murtola T., Schulz R. et al. // SoftwareX. 2015. V. 1–2. P. 19. https://doi.org/10.1016/j.softx.2015.06.001
MacKerell Jr. A.D., Feig M., Brooks III C.L. // J. Comput. Chem. 2004. V. 25 № 11. P. 1400. https://doi.org/10.1002/jcc.20065
Berendsen H.J.C., Postma J.P.M., van Gunsteren W.F. et al. // J. Chem. Phys. 1984. V. 81. № 8. P. 3684. https://doi.org/10.1063/1.448118
Parrinello M., Rahman A. // J. Chem. Phys. 1982. V. 76. № 5. P. 2662. https://doi.org/10.1063/1.443248
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


