Кристаллография, 2022, T. 67, № 4, стр. 632-635
Моделирование взаимодействия цитохрома С с липидным бислоем
П. Д. Короткова 1, *, А. А. Юрченко 2, В. И. Тимофеев 3, 4, Д. Д. Подшивалов 1, 4, А. Р. Гусельникова 2, А. Б. Шумм 1, 5, Ю. А. Владимиров 1, 2, 3
1 Московский государственный университет им. М.В. Ломоносова
Москва, Россия
2 Российский национальный исследовательский медицинский университет им. Н.И. Пирогова Министерства здравоохранения Российской Федерации
Москва, Россия
3 Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
Москва, Россия
4 КК НБИКС-природоподобных технологий, Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
5 Физический институт им. П.Н. Лебедева РАН
Москва, Россия
* E-mail: korotkovapol@gmail.com
Поступила в редакцию 22.08.2021
После доработки 17.09.2021
Принята к публикации 17.09.2021
- EDN: VRNFNY
- DOI: 10.31857/S0023476122030110
Аннотация
Методом молекулярной динамики промоделировано взаимодействие цитохрома С (Цит c) из сердца лошади с липидным бислоем, состоящим из молекул тетраолеилкардиолипина. Показано, что молекула Цит c способна проникать внутрь такого липидного бислоя. Описаны стадии данного процесса, а также конформационные изменения, которые претерпевает молекула Цит c при взаимодействии с бислоем. Полученные данные помогут понять молекулярный механизм апоптоза.
ВВЕДЕНИЕ
Структура комплекса цитохрома С (Цит c) с липидным бислоем изучается давно вследствие ее важности для понимания механизмов апоптоза. Первая гипотеза о структуре такого комплекса была высказана в 1977 г. [1] и заключалась в том, что комплекс представляет собой молекулу Цит c и прилегающий участок липидного бислоя мембраны. Эта гипотеза в дальнейшем развивалась и использовалась в ряде работ [2–5]. Вначале Цит c присоединяется к отрицательным фосфатным группам на поверхности мембраны, содержащей кардиолипин, благодаря электростатическому взаимодействию с лизиновыми аминокислотными остатками [1, 5–8]. Существуют три сайта на поверхности Цит c, в основном представленные аминокислотными остатками лизина [8–11]. Эти сайты получили названия A, С и L. Сайт A включает в себя остатки Lys72, Lys73, Lys86 и Lys87, сайт C состоит из одного остатка аминокислоты – Asn52, а сайт L включает в себя аминокислотные остатки Lys22, Lys25, Lys27, His26 и His33. Сайты А и L взаимодействуют с липидными структурами в основном через электростатические взаимодействия, в то время как сайт C делает это через водородные связи [2].
До последнего времени многие авторы, рассматривающие свойства комплекса Цит c с кардиолипином, считали, что белок не проникает в глубину липидного бислоя мембран, а в лучшем случае лишь прогибает мембрану, а прочность связи обусловлена [1] “заякореванием” белка путем проникновения в его толщу одной или двух жирнокислотных цепей. При этом изменения конформации все же должны происходить, так как иначе не появится ферментативная активность. Согласно [12] изменения конформации значительнее: Цит c распластывается на поверхности липидного слоя мембраны.
В [13] на основании данных, полученных с помощью исследования методом малоуглового рассеяния осадка, состоящего из комплекса Цит c с кардиолипином, предложили совершенно другую схему строения комплекса, в соответствии с которой молекула Цит c в состоянии расплавленной глобулы находится в центре наносферы и окружена монослоем из молекул кардиолипина. Предложенная структура наносферы подразумевает возможность ее проникновения в липидный бислой клеточных и митохондриальных мембран благодаря своей гидрофобной поверхности, образуя “вздутие” на мембране [14]. Предположение сделано на основании данных, полученных на модельных системах кристаллических осадков и ленгмюровских монослоев комплекса Цит c и кардиолипина [14].
В настоящей работе методом молекулярной динамики (МД) получена модель процесса формирования наносферы на молекулярном уровне.
МАТЕРИАЛЫ И МЕТОДЫ
Построение начальной системы. Начальная модель системы, включающая в себя липидный би-слой и молекулу Цит с, была построена с использованием программных пакетов PackMol [15] и AmberTools19 [16], после чего преобразована в формат GROMACS [17] с использованием программы ACPYPE [18]. Для построения топологии гема применяли параметры, полученные в [19]. Для моделирования использовали силовое поле amber14sb [20]. В качестве молекул липидов были взяты 256 молекул тетраолеилкардиолипина.
МД-моделирование. Моделирование проводили с помощью программы GROMACS 2020.3 [17]. В качестве силового поля выбрано поле ff14SB [20], так как в нем существенно улучшена точность моделирования боковых цепей аминокислотных остатков по сравнению с ff99SB [20]. В качестве модели воды выбрана модель TIP3P, как наиболее подходящая для использования с силовым полем ff14SB. Ячейку заполняли молекулами воды. Кроме того, в систему добавляли 0.15 М KCl, а также несколько ионов К для уравновешивания заряда системы. На первом этапе проводили минимизацию энергии с шагом 1 фс до максимальной силы 1000.0 кДж/моль–1 нм–1. Далее система была уравновешена при температуре 310 K и давлении 1 атм путем моделирования в NVT и NTP (100 пс в каждом) соответственно. Температуру и давление в системе контролировали с использованием модифицированного термостата Берендсена [21] и баростата Паринелло–Рахмана [22] с временными константами tau_t = 0.1 пс и tau_p = = 2 пс соответственно. Расчет продуктивной МД проводили в изотермо-изобарическом ансамбле с шагом в 2 фс. Алгоритм LINCS [23] использовали для ограничения связей с участием атомов водорода. Дальнодействующие электростатические взаимодействия рассчитывали с использованием схемы суммирования по Эвальду [24]. Радиусы действия сил Ван-дер-Ваальса и кулоновских взаимодействий были усечены до 1.4 нм, что является наиболее оптимальным для используемого силового поля [20].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
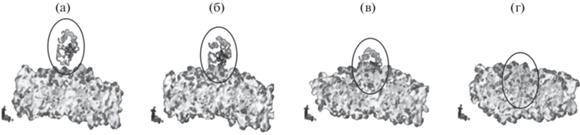
Получена динамическая модель взаимодействия Цит c из сердца лошади и бислоя, состоящего из молекул тетраолеилкардиолипина (рис. 1). Первая стадия (рис. 1а) заключается в приближении глобулы Цит c к данному бислою. Вторая стадия (рис. 1б) представляет собой присоединение посредством электростатических взаимодействий одного из лизиновых кластеров Цит c к бислою. Далее (рис. 1в) происходит погружение молекулы Цит c в бислой из тетраолеилкардиолипина, что сопровождается конформационными изменениями как в молекуле Цит c, так и в би-слое. На рис. 1г изображен комплекс Цит c с тетраолеилкардиолипином в равновесном состоянии. Отметим, что после приближения молекулы Цит c к бислою на расстояние ∼4 Å весь дальнейший процесс погружения происходит относительно быстро и занимает ∼11 нс.
Рис. 1.
Стадии погружения молекулы Цит c из сердца лошади в бислой, состоящий из тетраолеилкардиолипина. Над бислоем изображена вторичная структура молекулы Цит с, она выделена овалом.
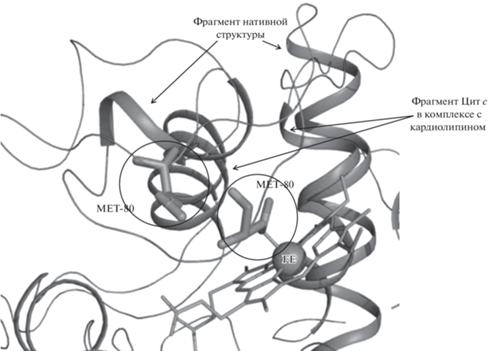
Молекула Цит c при взаимодействии с бислоем тетраолеилкардиолипина претерпевает конформационные изменения: происходит разрыв координационной связи между метионином-80 и железом гема (рис. 2). По-видимому, произошел переход молекулы Цит c в состояние “расплавленной глобулы” [25].
Рис. 2.
Наложение кристаллической структуры Цит c из сердца лошади (PDB ID: 1hrc) на структуру Цит c в комплексе с монослоем тетраолеилкардиолипина, полученную методом молекулярной динамики. На рисунке показаны участки альфа-спиралей интактного Цит с, а также Цит с в комплексе с бислоем тетраолеилкардиолипина. Гем изображен тонкими линиями и его координаты совпадают для двух наложенных структур Цит с. Железо гема отмечено буквами FE. Метионины в 80-м положении (MET-80) полипептидной цепи Цит с изображены палочками, правый из них относится к интактному цит с, а левый – к структуре цит с в комлексе с бислоем тетраолеилкардиолипина. Наложение проводилось программой PyMol по C-alpha атомам Цит c.
Полученные результаты позволяют окончательно завершить затянувшуюся дискуссию о структуре данного комплекса, является он мембрано-связанным Цит c или наносферой, встроенной в липидный бислой, в пользу последнего предположения [13, 25–27]. Однако оно было существенно конкретизировано и в известной степени изменено. В данной гипотезе предполагалось, что заранее сформированная наносфера Цит c–кардиолипин не совсем понятным образом оказывается внутри жидкого и гидрофобного липидного слоя мембраны, так что на обеих поверхностях мембраны образуется пузырь (так называемый bubble). Сам липидный бислой при этом как бы расщепляется на два [26]. Оказалось, что образование наносферы не предшествовало ее внедрению в липидный бислой, как предполагалось ранее, а произошло в момент самого внедрения благодаря тому, что фрагменты молекулы белка диффундировали сквозь цепочки фосфолипидных молекул. Вращательная подвижность участков полипептидной цепи и боковых групп аминокислотных остатков Цит c придает белковой глобуле свойства, характерные не для твердого тела, а для капли жидкости. Эта жидкая капля, не смешиваясь с липидным бислоем, тоже жидким, внедряется внутрь участка мембраны и остается там после установления окончательной стабильной структуры комплекса Цит c-кардиолипин. Таким образом, с использованием метода МД подтверждена сформулированная ранее гипотеза наносферы, а также построена модель такой наносферы на молекулярном уровне.
Работа выполнена при поддержке Российского научного фонда (грант № 19-14-00244) в части моделирования молекулярной динамики и при поддержке Министерства науки и высшего образования в рамках выполнения работ по Государственному заданию ФНИЦ “Кристаллография и фотоника” РАН в части анализа результатов молекулярного моделирования.
Список литературы
Brown L.R., Wuthrich K. // Biochim. Biophys. Acta Biomembr. 1977. V. 468. P. 389. https://doi.org/10.1016/0005-2736(77)90290-5
Kagan V.E., Bayır H.A., Belikova N.A. et al. // Free Radical Biol. Med. 2009. V. 46. P. 1439. https://doi.org/10.1016/j.freeradbiomed.2009.03.004
Sinibaldi F., Howes B.D., Piro M.C. et al. // J. Biol. Inorg. Chem. 2010. V. 15. P. 689. https://doi.org/10.1007/s00775-010-0636-z
Mandal A., Hoop CL., DeLucia M. et al. // Biophys. J. 2015. V. 109. P. 1873. https://doi.org/10.1016/j.bpj.2015.09.016
Hanske J., Toffey J.R., Morenz A.M. et al. // PNAS. 2012. V. 109. P. 125. https://doi.org/10.1073/pnas.1112312108
Quinn P.J., Dawson R.M.C. // Biochem. J. 1969. V. 115. P. 65. https://doi.org/10.1042/bj1150065
Sinibaldi F., Fiorucci L., Patriarca A. et al. // Biochemistry. 2008. V. 47. P. 6928. https://doi.org/10.1021/bi800048v
Sinibaldi F., Howes B.D., Droghetti E. et al. // Biochemistry. 2013. V. 52. P. 4578. https://doi.org/10.1021/bi400324c
Rytömaa M., Kinnunen P.K. // J. Biol. Chem. 1994. V. 269. P. 1770. https://doi.org/10.1016/S0021-9258(17)42094-1
Rytömaa M., Kinnunen P.K. // J. Biol. Chem. 1995. V. 270. P. 3197. https://doi.org/10.1074/jbc.270.7.3197
Kawai C., Prado F.M., Nunes G.L. et al. // J. Biol. Chem. 2005. V. 280. P. 34709. https://doi.org/10.1074/jbc.M412532200
Belikova N.A., Vladimirov Y.A., Osipov A.N. et al. // Biochemistry. 2006. V. 45. P. 4998. https://doi.org/10.1021/bi0525573
Владимиров Ю.А., Ноль Ю.Ц., Волков В.В. // Кристаллография. 2011. Т. 56. № 4. С. 596.
Marchenkova M.A., Dyakova Y.A., Tereschenko E.Y. et al. // Langmuir. 2015. V. 31. P. 12426. https://doi.org/10.1021/acs.langmuir.5b03155
Martínez L., Andrade R.E., Birgin G. et al. // J. Comput. Chem. 2009. V. 30. P. 2157. https://doi.org/10.1002/jcc.21224
Case D.A., Cheatham T.E., 3rd, Darden T. et al. // J. Comput. Chem. 2005. V. 26. P. 1668. https://doi.org/10.1002/jcc.20290
Abraham M.J., Murtolad T., Schulz R. et al. // SoftwareX. 2015. V. 1. P. 19. https://doi.org/10.1016/j.softx.2015.06.001
Da Silva A.W.S., Vranken W.F. // BMC Res. Notes. 2012. V. 5. P. 1. https://doi.org/10.1186/1756-0500-5-367
Giammona D.A. “An examination of conformational flexibility in porphyrins and bulky-ligand binding in myoglobin”. Diss. University of California, Davis, 1984.
Maier J.A., Martinez C., Kasavajhala K. et al. // J. Chem. Theory Comput. 2015. V. 11. P. 3696. https://doi.org/10.1021/acs.jctc.5b00255
Berendsen H.J.C., Postma J.P.M., van Gunsteren W.F. et al. // J. Chem. Phys. 1984. V. 181. P. 3684. https://doi.org/10.1063/1.448118
Parrinello M., Rahman A. // J. Chem. Phys. 1982. V. 76. P. 2662. https://doi.org/10.1063/1.443248
Hess B., Bekker H., Berendsen H.J.C., Fraaije J.G.E.M. // J. Comput. Chem. 1997. V. 18. P. 1463. https://doi.org/10.1002/(SICI)1096-987X(199709)18:12< 1463::AID-JCC4>3.0.CO;2-H
Darden T., York D., Pedersen L. // J. Chem. Phys. 1993. V. 98. P. 10089. https://doi.org/10.1063/1.464397
Hirota S., Ueda M., Hayashi Y. et al. // J. Biochem. 2012. V. 152. P. 521. https://doi.org/10.1093/jb/mvs098
Vladimirov Y.A., Proskurnina E.V., Alekseev A.V. // Biochemistry (Moscow). 2013. V. 78. P. 1086. https://doi.org/10.1134/S0006297913100027
Vladimirov G.K., Vikulina A.S., Volodkin D. et al. // Chem. Phys. Lipids. 2018. V. 214. P. 35. https://doi.org/10.1016/j.chemphyslip.2018.05.007
Дополнительные материалы отсутствуют.
Инструменты
Кристаллография


