Микробиология, 2020, T. 89, № 1, стр. 3-16
Эволюционная география клубеньковых бактерий: видообразование, направляемое растениями-хозяевами
Н. А. Проворов a, *, Е. Е. Андронов a, b, c, А. К. Кимеклис a, Е. Р. Чирак a, Е. С. Карасев a, Т. С. Аксенова a, В. В. Копать a
a ВНИИ сельскохозяйственной микробиологии
196608 Санкт-Петербург, Россия
b Санкт-Петербургский госуниверситет
199034 Санкт-Петербург, Россия
c Почвенный институт им. В.В. Докучаева
119017 Москва, Россия
* E-mail: provorovnik@yandex.ru
Поступила в редакцию 13.05.2019
После доработки 04.07.2019
Принята к публикации 29.09.2019
Аннотация
Для бактерий-симбионтов растений характерны повышенные, по сравнению со свободноживущими сородичами, темпы диверсификации популяций (микроэволюции), а также образования новых видов и надвидовых таксонов (макроэволюции). На примере клубеньковых бактерий (ризобий) – N2-фиксирующих симбионтов бобовых показано, что оба направления эволюции определяются растениями-хозяевами, которые оказывают на микроорганизмы мутагенное и рекомбиногенное воздействие, а также активируют в их популяциях естественный отбор, определяющий судьбу вновь возникших генотипов. Важную роль в эволюции ризобий играет индуцируемая растениями миграция, включая: 1) совместное продвижение симбионтов и их хозяев в новые экологические зоны; 2) локальную циркуляцию симбионтов в системах “растение‒почва”. В первом случае у ризобий происходит аллопатрическое видообразование, определяемое переносом sym-генов между штаммами-интродуцентами и местными бактериями, что приводит к появлению новых симбионтов. Во втором случае происходит симпатрическая дивергенция бактериальных популяций и образование внутривидовых таксонов (биотипов, симбиотипов), различающихся по хозяйской специфичности. У эволюционно молодых видов Rhizobium leguminosarum и Neorhizobium galegae, вступающих в симбиоз с бобовыми растениями галегоидного комплекса (трибы Fabeae, Galegae, Trifolieae), эти процессы связаны с дивергенцией sym-генов, которая происходит под давлением дизруптивного отбора. Он активируется в локальных популяциях ризобий при взаимодействии с хозяевами, обладающими различными симбиотическими свойствами. Параллельно с этим в популяциях ризобий наблюдают образование криптических видов, определяемое дивергенцией коровых частей генома. Она может быть результатом как генетического дрейфа, так и отбора на устойчивость к локальным эдафическим стрессам, который в сочетании с дизруптивным отбором повышает изменчивость ризобий и изменяет структуру их пангеномов.
Бактерии – симбионты эукариотических организмов представляют собой удобные модели для изучения многих проблем эволюционной генетики, включая механизмы видообразования у прокариот, а также их трансформации в клеточные органеллы эукариот. Известно, что у симбиотических микроорганизмов процессы дивергентной эволюции ускоряются под влиянием растений- или животных-хозяев. Так, патогенные формы Salmonella и Shigella генетически более разнообразны, чем комменсальные формы Escherichia (Ronald, 2003; Williams et al., 2010). Еще большим разнообразием обладают клубеньковые бактерии (ризобии), которые включают свыше 10 семейств α-протеобактерий (пор. Rhizobiales) и β-протеобактерий (Berrada, Fikri-Benbrahim, 2014). Эти бактерии разделяют на: а) “первичные” ризобии (Bradyrhizobium), которые возникли из свободноживущих N2-фиксаторов, родственных Rhodopseudomonas; б) “вторичные” ризобии (Rhizobium, Sinorhizobium, Neorhizobium, Mesorhizobium), которые приобрели способность к симбиозу благодаря переносу симбиотически специализированных (sym) генов в различные почвенные бактерии (Проворов, Андронов, 2016).
В ходе коэволюции с бобовыми растениями у ризобий произошло усложнение геномов, связанное с расширением их акцессорных частей, которые содержат sym-гены, кодирующие образование клубеньков (nod) и фиксацию N2 (nif, fix). Это усложнение определяется повышением пластичности бактериальных геномов, которое обусловлено их внутренними перестройками и горизонтальным переносом генов (ГПГ), вызывая существенные изменения пангеномов (Проворов, Андронов, 2016; Andrews et al., 2018). Показано, что ведущими факторами этой эволюции являются растения-хозяева, однако механизмы их влияния на диверсификацию микробных популяций и образование новых видов остаются мало изученными.
Для эволюционно молодых быстрорастущих ризобий, которые обладают многокомпонентными геномами (сем. Rhizobiaceae), характерна относительно независимая эволюция их коровых и акцессорных частей. Например, в популяции R. leguminosarum, состоящей из контрастных по хозяйской специфичности биотипов (bv. viciae – ризобии вики, гороха, чины и чечевицы; bv. trifolii – ризобии клевера), был выявлен ряд криптических видов, которые различаются по структуре коровых частей генома, не принимающих непосредственного участия в симбиозе (Kumar et al., 2015).
Цель нашей статьи заключается в анализе процессов внутри- и межвидовой дивергенции ризобий, которая может принимать как симпатрический, так и аллопатрический характер. На геномном уровне эти процессы вызывают прогрессивную эволюцию (усложнение организации индивидуальных геномов и видовых пангеномов) и адаптивную эволюцию (изменения sym-генов, входящих в акцессорные части генома). Будет показано, что, несмотря на общность молекулярных механизмов макро- и микро-эволюции, их популяционные механизмы существенно различаются. Микроэволюция ризобий обусловлена дизруптивным отбором, происходящим при их взаимодействии с хозяевами, которые обладают разными симбиотическими свойствами. Макроэволюция ризобий может быть связана как с дрейфом генов, контролирующих функции домашнего хозяйства, так и с отбором на устойчивость к локальным эдафическим факторам, либо с частотно-зависимым отбором, обеспечивающим закрепление в популяциях редких штаммов, обладающих измененной организацией генома.
ВОЗРАСТАНИЕ ГЕНЕТИЧЕСКОЙ ИЗМЕНЧИВОСТИ
Изучение механизмов коэволюции организмов, находящихся на разных уровнях организации, показало, что эукариоты-хозяева играют ключевую роль в формировании пула наследственной изменчивости своих прокариотических симбионтов. Доказательства этой роли, полученные более 50 лет назад при сравнении штаммов Sinorhizobium meliloti, выделенных из клубеньков люцерны и выращенных на лабораторных средах (Красильников, Мелкумова, 1963), были позднее дополнены выявлением индуцированных хозяевами геномных перестроек (Румянцева и соавт., 2004) и ГПГ (Чернова и соавт., 1986; Broughton et al., 1987; Pretorius-Güth et al., 1990).
Рядом исследователей показано действие в популяциях микросимбионтов естественного отбора, вызываемого хозяевами. У патогенных бактерий он связан с отношениями “ген-на-ген” и обычно принимает форму отрицательного частотно-зависимого отбора (ЧЗО), поскольку у хозяев отсутствует устойчивость к редким генам вирулентности паразитов (Thompson, Burdon, 1992; Jones, Dangl, 2006). У ризобий с помощью методов математического моделирования удалось разграничить ЧЗО, действующий при конкуренции за инфицирование растений, и дарвиновский отбор, который определяется дифференциальным размножением (отмиранием) бактерий в симбиотических и почвенных нишах (Provorov, Vorobyov, 2006). Роль ЧЗО в эволюции ризобий может быть связана с закреплением в популяциях редких штаммов, в том числе обладающих измененной геномной организацией и повышенной симбиотической эффективностью (Воробьев, Проворов, 2015). Иную роль в эволюции ризобий играет дарвиновский отбор, который поддерживает выживаемость в почве штаммов, устойчивых к эдафическим стрессам, но часто обладающих низкой активностью N2-фиксации (Проворов и соавт., 2014).
Сравнительный анализ популяционного полиморфизма у генетически родственных ризобий вики и клевера (R. leguminosarum bv. viciae и bv. trifolii), проведенный с помощью высокопроизводительного секвенирования, позволил показать, что эффекты указанных форм отбора зависят от уровня, на котором изучают структуру популяции – гаплотипического (индивидуальные последовательности sym-генов), либо генотипического (операционные таксономические единицы, которые объединяют последовательности с уровнем сходства 97‒98%) (Андронов и соавт., 2015). Например, при переходе ризобий из прикорневой зоны бобовых в клубеньки изменчивость по гену nodA (кодирует присоединение жирнокислотных остатков к олигохитиновым цепям сигнальных Nod-факторов) снижается на генотипическом уровне, поскольку в клубеньки проникают лишь высоковирулентные и конкурентоспособные штаммы. Однако на гаплотипическом уровне изменчивость возрастает благодаря отрицательному ЧЗО, который действует в пользу редких аллелей nodA, возникающих у вирулентных бактерий.
Изучение молекулярных механизмов симбиоза показало, что в основе коэволюции ризобий и их хозяев лежат процессы взаимного узнавания партнеров, определяемого действием сигнальных Nod-факторов на растительные LysM-содержащие рецепторы LRR-типа (Wang et al., 2012). Метагеномный анализ позволил выявить у ризобий движущий (позитивный) отбор, увеличивающий число несинонимических замен в функционально значимых участках nod-генов (Андронов и соавт., 2015). При взаимодействии с несколькими хозяевами, обладающими разными симбиотическими предпочтениями, в популяции бактерий происходит дизруптивный отбор, который мы рассмотрим ниже.
Большую роль в эволюции ризобий играют вызываемые хозяевами генетико-автоматические (“стохастические”) процессы, включая генетический дрейф, который связан с клональным инфицированием корней (Bassam et al., 1987) и, в сочетании с отрицательным ЧЗО, значительно повышает популяционную изменчивость бактерий. Еще большую роль в эволюции ризобий играет миграция, включая: а) совместное с растениями продвижение в новые экологические зоны, которое вызывает у бактерий аллопатрическое видообразование; б) их локальную циркуляцию в системах “растение‒почва”, которая вызывает симпатрическое видообразование.
МИГРАЦИЯ И АЛЛОПАТРИЧЕСКОЕ ВИДООБРАЗОВАНИЕ
Эволюционные процессы, приводящие к образованию у ризобий видов и надвидовых таксонов, могут быть индуцированы при совместном продвижении бактерий и растений в новые экологические зоны: адаптация хозяев к “непривычным” условиям наиболее эффективна в том случае, если их сопровождают симбионты-мутуалисты (Richardson et al., 2000; Bena et al., 2005; van der Putten et al., 2007; Litchman, 2010). Совместная миграция партнеров N2-фиксирующего симбиоза в новые местообитания многократно происходила в эволюции бобовых, которые возникли в тропиках и распространились во все климатические зоны Земли (Sprent, 2001), а также при интродукции растений из центров происхождения в районы возделывания (Allen, Allen, 1981). Для изучения происходящих при этом эволюционных процессов были предложены модели, описывающие интродукцию Исходных Симбионтов (ИС) в новые экосистемы, которая вызывает: а) микроэволюцию, связанную с изменением разнообразия ИС, включая их переходы на новых хозяев; б) макроэволюцию, определяемую переносом sym-генов в местные, в том числе и неродственные ИС бактерии, который приводит к образованию Новых Симбионтов (НС), включая расширение видового разнообразия бактерий.
Наиболее простой сценарий этой эволюции реализуется том случае, если она ограничена адаптацией ИС к новым экологическим условиям. Так, при миграции медленнорастущих ризобий люпина и сераделлы (Bradyrhizobium spp.) в Южную Африку и Западную Австралию бактерии сохранили многие генетические маркеры, характерные для исходных европейских популяций (Stepkowski et al., 2005). Однако при воздействии стрессовых факторов среды разнообразие ИС может снижаться, что было показано для штаммов R. leguminosarum bv. trifolii, интродуцированных совместно с клевером переменчивым, Trifolium ambiguum, в Северную Америку из кавказского центра происхождения (Seguin et al., 2001).
Сильное сужение спектра симбионтов выявлено у сои, интродуцированной в Северную Америку из китайского центра происхождения. Произрастающие в нем формы Glycine max и G. soja высоко полиморфны по Rj-генам, контролирующим специфичность к различным штаммам ризобий, причем широкое распространение получила аллель Rfg1, ответственная за симбиоз с быстрорастущим видом Sinorhizobium fredii (Triplett, Sadowsky, 1992). Поэтому в популяциях ризобий сои из Китая выявляют не только различные виды Bradyrhizobium, но и S. fredii, тогда как в США обнаружены лишь некоторые генотипы B. japonicum, что связано с низким полиморфизмом растений по Rj-генам (Devine, Kuykendall, 1996).
Более сложный сценарий, при котором у ризобий происходит аллопатрическое видообразование, осуществляется в том случае, если их продвижение в новые местообитания вызывает перенос sym-генов из штаммов-интродуцентов в местные бактерии (табл. 1). Наиболее изучен этот сценарий для ризобий фасоли, которая в южно- и центрально-американских центрах происхождения вступает в симбиоз с R. tropici и R. etli (Eardly et al., 1995; Bernal, Graham, 2001; Aguilar et al., 2004). В Европе, куда фасоль была интродуцирована около 500 лет назад, выявляется широкий спектр ее новых симбионтов, включая R. gallicum, R. giardinii и R. leguminosarum bv. phaseoli (Amarger et al., 1994; Laguerre et al., 2001). Несмотря на полифилетическое происхождение, эти ризобии содержат универсальный набор nod-генов, которые характерны для R. tropici и R. etli, определяя структурные особенности синтезируемых Nod-фактор-ов.
Таблица 1.
Аллопатрическое образование новых симбионтов путем переноса sym-генов из ризобий-интродуцентов в местные бактерии
| Ризобии-интродуценты | Переносимые элементы генома | Вновь возникшие симбионты | Растения – ко-интродуценты бактерий | Направление и возраст интродукции | Авторы |
|---|---|---|---|---|---|
| Mesorhizobium loti, Mesorhizobium spp. | Хромосомные острова | Mesorhizobium spp. | Lotus corniculatus | Европа → → Новая Зеландия (7 лет) | Sullivan et al., 1995, 2002 |
| Biserrula pelecinus | Средиземноморье → → Австралия (12 лет) | Nandasena et al., 2006 | |||
| Mesorhizobium spp., Rhizobium spp. | Robinia pseudoacacia | Сев. Америка → → Европа (300 лет) | Ulrich, Zaspel, 2000 | ||
| Amorpha fruticosa | Сев. Америка → → Китай (50 лет) | Wang et al., 1999 | |||
| Rhizobium tropici, R. etli | Sym-плазмиды | R. leguminosarum bv. phaseoli, R. gallicum, R. giardinii | Phaseolus vulgaris | Юж. и Центр. Америка → → Европа (500 лет) | Amarger et al., 1994; Laguerre et al., 1993, 2001; Brom et al., 2002 |
| Bradyrhizobium japonicum | Временно существующие репликоны (?) | Bradyrhizobium spp., Rhizobium spp. | Glycine max | Сев. Америка → → Центр. Америка (80 лет) | Chen et al., 2000 |
Быстрое возникновение НС на основе гибридизации ИС с местными бактериями показано для штаммов Mesorhizobium (сем. Phyllobacteriaceae), несущих nod- и nif-гены в составе хромосомных островов, которые могут переноситься как конъюгативные транспозоны через системы секреции 4-го типа (Sullivan et al., 2002). Этот перенос выявлен у ризобий лядвенца рогатого (Lotus corniculatus), интродуцированных из Европы в Новую Зеландию, где в течение 7 лет сформировались стабильные популяции НС. У них обнаруживаются генетические маркеры местных представителей сем. Phyllobacteriaceae в сочетании с sym-генами (“симбиотическими островами”) штаммов ИС, использованных для инокуляции лядвенца в новозеландских экосистемах (Sullivan et al., 1995, 2002). Сходные процессы сопровождали совместное продвижение Mesorhizobium с Robinia pseudoacacia из Северной Америки в Европу, а также с Amorpha fruticosa из Северной Америки в Китай и с Biserrula pelecinus из Европы в Австралию (табл. 1). Одновременный перенос nod- и nif-генов в составе “симбиотических островов” определяет конгруэнтность филогений этих генов, выявленную при анализе полиморфизма M. loti (Qian, Parker, 2002).
Быструю эволюцию НС наблюдали также при интродукции сои из Северной Америки в Парагвай, которая происходила на фоне инокуляции коммерческими штаммами B. japonicum. Благодаря переносу их sym-генов в местные ризобии, в течение 80 лет сформировались обширные популяции НС, включая Bradyrhizobium, Rhizobium и Agrobacterium (Chen et al., 2000). Закрепление НС в почвах Латинской Америки произошло несмотря на то, что большинство штаммов B. japonicum – вероятных доноров sym-генов лишено симбиотических плазмид или островов (Okazaki et al., 2015). Их роль могли выполнять содержащие sym-гены нестабильные элементы генома, образуемые благодаря наличию у B. japonicum множества повторяющихся и инсерционных последовательностей ДНК (Minamisawa et al., 2002).
Особый интерес представляет реконструкция генетических процессов, сопровождавших продвижение бобово-ризобиальных систем из тропиков, где сем. Fabaceae возникло около 60 млн лет назад, в умеренные широты, где произрастает лишь 10% бобовых – травянистых представителей подсемейства Papilionoideae, включая трибы Fabeae, Galegae, Trifolieae, входящие в состав галегоидного комплекса. Удивительным является то, что у бобовых умеренных широт, несмотря на сниженную потребность в азоте, определяемую произрастанием на стабильных, содержащих значительные количества органики почвах, произошло существенное усложнение структуры и повышение N2-фиксирующей активности клубеньков по сравнению с тропическими бобовыми, которые растут на крайне бедных, нестабильных почвах (Sprent, 2001, 2005).
Использование математических моделей эволюции симбиоза показало (Проворов, Воробьев, 2012), что повышение его N2-фиксирующей активности при переходе бобовых из тропиков в умеренные широты могло быть связано как с дивергенцией ризобий по признаку хозяйской специфичности, так и с синхронизацией реакций хозяев на изменения внешних условиях, определяемой снижением филогенетического разнообразия растений. В результате этих процессов узко специфичные симбионты вытесняли из популяций широко специфичных симбионтов, а также не фиксирующих N2 “симбионтов-обманщиков” (Воробьев, Проворов, 2013; табл. 2). Такое изменение популяционной структуры ризобий вызывает отбор в пользу растений, обладающих высокой активностью симбиотрофного питания азотом в сочетании с узкой симбиотической специализацией.
Таблица 2.
Изменения популяционной структуры ризобий при переходе из тропиков в умеренные широты (по данным математического моделирования: Воробьев, Проворов, 2013)
| Микросимбионты | Изменения параметров микробной популяции | |
|---|---|---|
| генотипические частоты микросимбионтов* | дисперсии генотипических частот (×106)** | |
| М1 | 0.16 → 0.31 | 10000 → 80 |
| М2 | 0.16 → 0.31 | 10000 → 80 |
| М3 | 0.16 → 0.07 | 0.2 → 400 |
| Р | 0.52 → 0.31 | 2 → 3 |
Примечание. М1, М2, М3 – симбионты-мутуалисты, которые образуют N2-фиксирующие клубеньки с одним или с обоими доступными генотипами растений (Г1, Г2); Р – симбионт-обманщик, образует только не фиксирующие N2 клубеньки. * Изменения параметров микробных популяций регистрировали при дивергенции ризобий по признаку хозяйской специфичности, при которой М1 образует N2-фиксирующие клубеньки только с Г1, М2 – только с Г2, а М3 – с обоими генотипами растений (в исходной системе специфичность отсутствовала). ** Изменения параметров микробных популяций регистрировали при синхронизации реакций растительных генотипов на быстрые стохастические колебания параметров внешней среды (отражает снижение филогенетического разнообразия бобовых, которое произошло при их переходе из тропиков в умеренные широты).
ДИЗРУПТИВНЫЙ ОТБОР И СИМПАТРИЧЕСКОЕ ВИДООБРАЗОВАНИЕ
Удобной моделью для изучения симпатрического видообразования является вид R. leguminosarum bv. viciae – симбионт бобовых растений трибы Fabeae. От общего предка этой трибы, близким родичем которого считается произрастающая на Кавказе Vavilovia formosa, произошли роды Pisum (при продвижении растений на Ближний Восток), а возможно также Vicia и Lathyrus (при их продвижении в северные регионы Евразии) (Mikič et al., 2013). Одним из маркеров происходившей при этом эволюции симбионтов является ген nodX – фактор широкой хозяйской специфичности, которая представляет собой анцестральный признак ризобий (Lie et al., 1987). Со стороны растений эту специфичность контролирует ген Sym2, рецессивная аллель которого (sym2A), характерная для дикорастущих “афганских” форм P. sativum (sspp. abyssinicum, asiaticum), определяет устойчивость к инокуляции штаммами R. leguminosarum bv. viciae, лишенными nodX. У культурного “европейского” гороха (P. sativum ssp. sativum) аллель sym2A заменена на sym2E, благодаря чему растения перешли к симбиозу с бактериями, лишенными гена nodX, включая симбионтов Vicia и Lathyrus (Mutch, Young, 2004) (рис. 1).
Рис. 1.
Дивергенция вида Rhizobium leguminosarum, связанная с формированием различающихся по хозяйской специфичности биотипов (bv. viciae, bv. trifolii). Общий предок (ОП) вида R. leguminosarum возник до его разделения на биотипы viciae и trifolii, причем все штаммы bv. trifolii сохранили анцестральный маркер – ген nodX. У R. l. bv. viciae nodX сохранился лишь на начальных стадиях эволюции – у симбионтов Vavilovia и дикорастущих “афганских” форм гороха (Pisum ‒ Afghan), однако был утрачен на поздних стадиях эволюции, связанных с возникновением культурных “европейских” форм гороха (Pisum – Euro), а также родов Vicia и Lathyrus. В белом овале представлены гипотетические предковые формы ризобий, в светло-сером овале – примитивные формы с геном nodX, в темно-сером овале – эволюционно продвинутые формы без nodX.
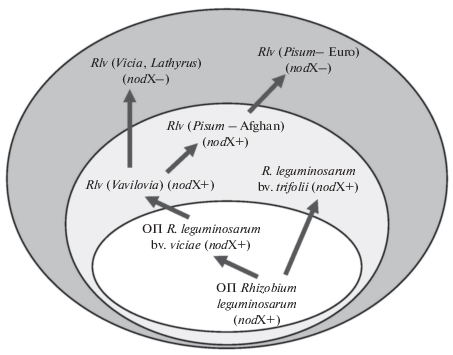
Таким образом, интродукция гороха из ближневосточного генцентра в Европу, которая произошла около 2000 лет назад, привела к тому, что растения, утратив анцестральных симбионтов, вступили в контакт с местными штаммами R. leguminosarum bv. viciae. Это провело к сужению разнообразия симбионтов гороха, способного использовать лишь небольшую часть генофонда европейских ризобий, что было показано путем генотипирования штаммов из клубеньков гороха и вики, собранных в Англии (Mutch, Young, 2004) и Франции (Laguerre et al., 2003). Важно отметить, что анцестральные формы R. leguminosarum bv. viciae (симбиотип vaviloviae) характеризуются сильной дивергенцией по nod-генам, хотя обособления этого симбиотипа от других штаммов bv. viciae по маркерам коровой части генома, а также по специфичности к разным видам растений-хозяев выявить не удалось (Кимеклис и соавт., 2018).
Особый интерес представляет соотношение R. leguminosarum bv. viciae с R. leguminosarum bv. trifolii – симбионтами бобовых рода клевер (Trifolium) из трибы Trifolieae. Эта триба включает также роды люцерна (Medicago), донник (Melilotus) и пажитник (Trigonella), ризобии которых (Sinorhizobium meliloti, S. medicae) неродственны ризобиям клевера. Однако на основе анализа пластидного гена matK род Trifolium был сближен с Vicia, Pisum, Lathyrus (Wojciechowski et al., 2004), что может отражать сходство различных типов внутриклеточных симбионтов растений – древних пластид и эволюционно молодых ризобий. В связи с этим большой интерес представляет выявление у ризобий клевера гена nodX, который оказался обязательным компонентом их геномов. У этих ризобий ген nodX характеризуется повышенной, по сравнению с nodA и nodE, консервативностью и, в отличие от этих генов, подвержен действию индуцированного хозяевами позитивного (движущего) отбора (Аксенова и соавт., 2019), что указывает на высокую симбиотическую значимость nodX.
К числу других nod-генов, по которым показана дивергенция вида R. leguminosarum, относится nodA, обычно включаемый в число “общих” nod-генов (Wang et al., 2012). Однако nodA принимает участие и в контроле хозяйской специфичности, так как кодирует присоединение к Nod-фактору определяющего этот признак жирнокислотного остатка (Moulin et al., 2004). Дивергенция nodA, выявленная при сравнении ризосферных и клубеньковых субпопуляций каждого из хозяев, оказалась меньшей, чем при сравнении ризосфер разных хозяев (рис. 2) (Андронов и соавт., 2015). При этом в ризосферах каждого из растений выявляется 17‒18% “чужеродных” nodA-гаплотипов, тогда как в клубеньках их частота снижена до 0.1‒1.3% (табл. 3). Эти величины, по-видимому, отражают изредка происходящую рекомбинацию генов nodA с nodEF, которые кодируют синтез жирнокислотного остатка, определяющего хозяйскую специфичность ризобий.
Рис. 2.
Дивергенция гена nodA, определяемая миграцией штаммов Rhizobium leguminosarum между ризосферными (Р) и клубеньковыми (К) нишами бобовых растений. Рядом со стрелками, указывающими направления миграции, приведены относительные генетические расстояния (Андронов и соавт., 2015), которые отражают дивергенцию nod-генов ризобий при взаимодействии с растениями из разных групп перекрестной инокуляции: 1 – группа гороха, вики и чины, 2 – группа клевера.
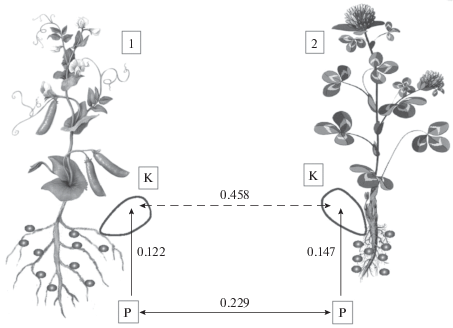
Таблица 3.
Частоты (%) последовательностей гена nodA, характерных для разных биотипов Rhizobium leguminosarum, в субпопуляциях из ризосферы (Р) и клубеньков (К) различных растений-хозяев (по данным Андронов и соавт., 2015)
| Биотипы ризобий | Происхождение субпопуляций ризобий | |||
|---|---|---|---|---|
| Vicia sativa, Lathyrus pratensis | Trifolium hybridum | |||
| Р | К | Р | К | |
| trifolii | 17.3 ± 0.42 | 1.3 ± 0.12 | 81.8 ± 0.63 | 99.9 ± 0.05 |
| viciae | 82.7 ± 0.43 | 98.7 ± 0.13 | 18.2 ± 0.63 | 0.1 ± 0.05 |
Логично предположить, что эволюция гена nodA в метапопуляциях R. leguminosarum (bv. viciae + + bv. trifolii), формируемых при совместном произрастании бобовых трибы Fabeae и рода Trifolium, связана с дизруптивным отбором, возникающим при взаимодействии бактерий с различными хозяевами (рис. 2). Однако его эффект нивелируется переносом nod-генов между разными биотипами R. leguminosarum, что повышает рекомбинационную изменчивость акцессорного генома. При переходе ризобий из ризосферы в клубеньки действует отрицательный ЧЗО, который, как и дизруптивный отбор, повышает разнообразие микробной популяции (Андронов и соавт., 2015).
Сходный эволюционный сценарий выявлен у вида Neorhizobium galegae, разделяемого на биотипы orientalis и officinalis, которые вступают в симбиоз с козлятником восточным (Galega orientalis) и лекарственным (G. officinalis) (Mousavi et al., 2014). Каждый из этих биотипов фиксирует N2 при взаимодействии только со “своим” хозяином, тогда как перекрестная инокуляция между разными видами козлятника приводит к образованию не фиксирующих N2, хотя и нормальных по структуре клубеньков. Наиболее сильное расхождение bv. orientalis и bv. officinalis наблюдают по генам N2-фиксации (nif, fix), проявление которой у N. galegae определяется растениями-хозяевами (Карасев и соавт., 2019).
Важно отметить, что выявленный у R. leguminosarum и N. galegae сценарий дивергентной эволюции, приводящей к сужению хозяйской специфичности, не является единственно возможным. Полногеномный анализ 72 штаммов из локальной популяции R. leguminosarum (Йорк, Англия), позволил выявить 5 криптических видов, которые различаются по структуре коровой части генома, однако содержат штаммы разных биотипов – vic-iae и trifolii (Kumar et al., 2015). Изучение дивергенции этих криптических видов показало, что при построении филогении ризобий может быть использован традиционный для высших организмов критерий видовой идентификации, основанный на генетической изоляции (Майр, 1974), которая у бактерий ограничена коровой частью генома. Аналогичным образом могла происходить и симпатрическая дивергенция распространенных в Китае видов Bradyrhizobium, которые не обнаруживают существенного расхождения по хозяйской специфичности: большинство штаммов B. japonicum и B. elkanii формирует N2-фиксирующие клубеньки с соей, вигной и другими представителями симбиотически неспециализированной трибы Phaseoleae (Berrada, Fikri-Benbrahim, 2014). Расхождение сестринских видов Sinorhizobium meliloti и S. medicae по структуре корового генома происходило при сохранении общего ареала, а также специфичности по отношению к растениям родов Medicago, Melilotus и Trigonella. Эти виды ризобий различаются по способности к симбиозу лишь с некоторыми однолетними видами люцерны, например, с Med. polymorpha (Rome et al., 1996; Bailly et al., 2006).
Таким образом, для ризобий характерны два направления дивергентной эволюции, которые связаны с изменениями либо коровой части генома, кодирующей функции домашнего хозяйства, либо его акцессорной части, кодирующей функции симбиоза. Дивергенция акцессорного генома определяется дизруптивным отбором, который возникает при взаимодействии бактерий с различными растениями-хозяевами и может иметь как симпатрический, так и аллопатрический характер (Андронов и соавт., 2015). В результате этой эволюции возникают внутривидовые таксономические единицы (биотипы, симбиотипы), сохраняющие мало измененной коровую часть генома. Ее дивергенция также возможна, однако она не связана с симбиозом, имеет симпатрический характер и происходит в локальных популяциях вне связи с хозяйской специфичностью. Благодаря этой дивергенции в биотипах N. galegae изменчивость сильнее выражена для функционально разнородных элементов корового генома, чем для акцессорных sym-генов (Карасев и соавт., 2019).
Соотношение этих процессов, которые могут быть обозначены как макроэволюция (дивергенция корового генома) и микроэволюция (дивергенция акцессорного генома) бактерий представляет большой интерес. Особого внимания заслуживают вопросы о том: а) может ли дивергенция симбиотических генов, индуцируемая хозяевами, рассматриваться в качестве начального этапа видообразования, определяемого изоляцией бактерий, взаимодействующих с различными хозяевами; б) как влияет коэволюция с растениями на структуру пангеномов бактерий? Ранее резкое расширение пангенома было выявлено у “первичных” ризобий (Bradyrhizobium), у которых его акцессорная часть увеличена в несколько раз по сравнению со свободноживущими аналогами – диазотрофами Rhodopseudomonas (Oda et al., 2008; Mornico et al., 2012; Tian et al., 2012). Основным механизмом увеличения акцессорной части пангенома ризобий является горизонтальный перенос sym-генов, который наиболее активен у быстрорастущих “вторичных” форм благодаря внехромосомной локализации этих генов.
ЗАКЛЮЧЕНИЕ
Ризобии – удобная модель для сравнительного анализа процессов микроэволюции, видообразования и макроэволюции, осуществляемых бактериями в составе надорганизменных комплексов. Изучение молекулярно-генетических и популяционных механизмов этой эволюции показало, что она включает относительно независимые процессы дивергенции, которая затрагивает: а) содержащие sym-гены акцессорные части генома, приводя к образованию различающихся по хозяйской специфичности внутривидовых таксонов – биотипов и симбиотипов (микроэволюция); б) содержащие гены “домашнего хозяйства” коровые части, приводя к возникновению новых видов и надвидовых таксонов (макроэволюция).
Интенсивность этих процессов определяется высокой миграционной активностью бактерий, проявляемой благодаря совместному с растениями продвижению в новые местообитания, а также локальной циркуляции между альтернативными нишами в системах “хозяин‒среда”. В обоих случаях ключевым фактором эволюции симбионтов является индуцируемый хозяевами отбор, однако его популяционные механизмы, определяющие макро- и микроэволюцию, различаются.
Микроэволюция ризобий определяется дизруптивным отбором, который происходит при их миграции между совместно произрастающими растениями, различающимися по симбиотической специфичности, и вызывает дивергенцию генов, контролирующих этот процесс (рис. 2). Важным результатом микроэволюции ризобий является повышение симбиотической эффективности, которое часто сопровождает сужение хозяйской специфичности (Проворов, Воробьев, 2012).
В то же время, макроэволюционные процессы могут быть связаны как с “дрейфом” генов домашнего хозяйства, так и с различными типами отбора, действующего в локальных популяциях и влияющего на эти гены. Одним из них может быть дизруптивный отбор, связанный с адаптацией к различными микронишам, занимаемым бактериями в гетерогенной почвенной среде. Другим типом локально действующего отбора может быть отрицательный ЧЗО, который активируется при переходе бактерий из прикорневой зоны в клубеньки и поддерживает редкие формы, в том числе и обладающие измененной организацией генома.
Важно отметить, что эволюцию симбиотической системы до сих пор изучали на примере генов сигнально-рецепторного комплекса, по которым обычно наблюдают четкую филогенетическую конгруэнтность партнеров. В то же время, эволюция путей их совместного азотно-углеродного обмена остается мало изученной: например, неясно, как происходит коэволюция симбионтов и хозяев по генам, контролирующим процессы фиксации N2 и ассимиляции ее продуктов. Удобную модель для анализа этой эволюции представляют ризобии козлятника (Neorhizobium galegae): для них характерно расхождение биотипов orientalis и officinalis по признаку хозяин-специфичной нитрогеназной активности, которое сопровождается глубокой дивергенцией nif- и fix-генов (Карасев и соавт., 2019).
Представленные данные показывают перспективность использования бактериальных моделей для оценки соотношения процессов микро- и макроэволюции, которое остается дискуссионным для высших организмов. По мнению ряда авторов (Майр, 1974; Тимофеев-Ресовский и соавт., 1977), у растений и животных макро- и микроэволюция – это тесно связанные стадии единого процесса, контролируемого отбором. По мнению других авторов (Filipcenko, 1927), макро- и микроэволюция определяются разными механизмами, и роль отбора в возникновении надвидовых таксонов неочевидна.
Рассмотренные в нашей статье результаты эволюционно-генетического анализа ризобий согласуются со второй точкой зрения. Действительно, их микроэволюция определяется аллопатрической и симпатрической дивергенцией sym-генов, которая происходит под влиянием миграции и дизруптивного отбора, вызываемых растениями. Симпатрическая дивергенция характерна и для коровых частей бактериального генома, которые гораздо более консервативны, чем акцессорные части, и, возможно, эволюционируют под действием факторов несимбиотической природы, например, локальных эдафических стрессов. Кроме того, возникновение у ризобий надвидовых таксонов (макроэволюция) является результатом их совместной с хозяевами миграции в новые экологические зоны. Она активирует перенос sym-генов из интродуцированных ризобий-доноров в местные, в том числе и в неродственные донорам бактерии, а также отбор, который закрепляет в популяциях вновь возникших рекомбинантов.
Дальнейшее развитие эволюционной генетики симбиоза будет в значительной степени связано с созданием моделей на основе реликтовых бобовых, анализ которых позволяет дать сравнительную характеристику прогрессивной и адаптивной эволюции симбиотических комплексов. У многих из этих бобовых выявлены анцестральные черты организации клубеньков, включая примитивные мультибактериальные симбиосомы, в которых бактероиды характеризуются низким уровнем структурно-функциональной дифференцировки. Такие симбиосомы характерны для Vavilovia formosa – вида растений, близкого к общему предку трибы Fabeae, тогда как у других представителей этой трибы (Pisum, Vicia) выявлены специализированные монобактериальные симбиосомы с глубоко дифференцированными бактероидами (Цыганова и соавт., 2018). Интересно отметить, что у штаммов R. leguminosarum bv. viciae, выделенных из V. formosa, найден ген fixW, кодирующий разрыв дисульфидных связей. Они характерны для богатых цистеином NCR-белков, определяющих глубокую дифференцировку бактероидов, характерную для штаммов из Pisum и Vicia, у которых этот ген отсутствует. У симбионтов вавиловии выявлены и другие черты примитивной организации – крупный размер внехромосомного симбиотического кластера, либо его разделение между двумя плазмидами, а также присутствие гена nodX, определяющего широкую хозяйскую специфичность – анцестральный признак ризобий (Чирак и соавт., 2016).
Не менее значимой для эволюции симбиоза представляется организация микробных сообществ, обитающих в реликтовых бобовых. Например, в клубеньках Vavilovia наряду с типичными для трибы Fabeae симбионтами R. leguminosarum bv. viciae выявляются Bosea и Tardiphaga, которые относятся к сем. Bradyrhizobiaceae, включающему наиболее древние медленно растущие ризобии (Safronova et al., 2014, 2015). Эти штаммы не имеют полного набора sym-генов, однако у Tardiphaga найдена типичная для R. leguminosarum bv. viciae система синтеза цитохромоксидазы FixNOQP, способной функционировать в микроаэрофильных условиях клубенька, благодаря чрезвычайно высокому сродству к О2 (Копать и соавт., 2017). Выявление подобных штаммов позволяет реконструировать процессы сборки систем sym-генов, происходящей на основе внутригеномных перестроек, а также переноса генов в микробных сообществах.
К числу актуальных, но пока еще мало изученных вопросов эволюционной генетики ризобий относится анализ динамики их пангеномов. Показано, что у “первичных” ризобий (Bradyrhizobium) пангеном существенно увеличен по сравнению с вероятными предками (Rhodopseudomonas) в основном за счет расширения акцессорных частей (Oda et al., 2008; Mornico et al., 2012; Tian et al., 2012). У “вторичных” ризобий (Rhizobiaceae) происходит не только приобретение новых генов, но и утрата негативных регуляторов симбиоза, в связи с чем доля акцессорной части в пангеноме может еще более возрастать. Этому способствует и активно действующий на ризобии дизруптивный отбор: расхождение биотипов R. leguminosarum или N. galegae по специфичности к разным хозяевам может увеличивать число генов, кодирующих этот признак.
Реконструкция органеллогенеза имеет не только фундаментальное, но и важное прикладное значение. Выявление механизмов перехода от анцестральных к эволюционно продвинутым формам симбиоза может рассматриваться как модель для разработки методов “симбиотической инженерии”, которая уже делает свои первые шаги (Проворов, Онищук, 2018) и, несомненно, получит активное развитие в ближайшем будущем.
Список литературы
Аксенова Т.С., Чирак Е.Р., Онищук О.П., Курчак О.Н., Иголкина А.А., Пинаев А.Г., Андронов Е.Е., Проворов Н.А. Популяционный полиморфизм генов синтеза Nod-фактора у ризобий клевера (Rhizobium leguminosarum bv. trifolii) в экосистемах, контрастно различающихся по климатическим условиям и доступности растений-хозяев // Экологическая генетика. 2019 (в печати).
Aksenova T.S., Chirak E.R., Onishchuk O.P., Kurchak O.N., Igolkina A.A., Pinaev A.G., Andronov E.E., Provorov N.A. Population polymorphism of Nod-factor synthesis genes in clover rhizobia (Rhizobium leguminosarum bv. trifolii) from ecosystems contrasting in climatic conditions and accessibility of host plants // Rus. J. Genetics: Applied Research. 2019 (in press).
Андронов Е.Е., Иголкина А.А., Кимеклис А.К., Воробьев Н.И., Проворов Н.А. Характеристика естественного отбора в популяциях клубеньковых бактерий (Rhizobium leguminosarum), взаимодействующих с различными видами растений-хозяев // Генетика. 2015. Т. 51. С. 1108–1116.
Andronov E.E., Igolkina A.A., Kimeklis A.K., Vorobyov N.I., Provorov N.A. Characteristics of natural selection in populations of nodule bacteria (Rhizobium leguminosarum) interacting with different host plants // Rus. J. Genetics. 2015. V. 51. P. 949–956.
Воробьев Н.И., Проворов Н.А. Моделирование эволюции бобово-ризобиального симбиоза в условиях экологической нестабильности // Экологическая генетика. 2013. Т. 11. С. 73‒85.
Vorobyov N.I., Provorov N.A. Simulation of the evolution of the legume-rhizobia symbiosis under the conditions of ecological instability // Rus. J. Genetics: Applied Research. 2015. V. 5. P. 91–101.
Воробьев Н.И., Проворов Н.А. Quorum Sensing и нодуляционная конкурентоспособность ризобий при инфицировании бобовых растений // Сельскохозяйственная биология. 2015. Т. 50. С. 298‒304.
Vorobyov N.I., Provorov N.A. The Quorum Sensing and the nodulation competitiveness of rhizobia during infection of leguminous plants // Agricultural Biology. 2015. V. 50. P. 298‒304.
Карасев Е.С., Андронов Е.Е., Аксенова, Т.С., Чижевская Е.П., Тупикин А.Е., Проворов Н.А. Эволюция ризобий козлятника (Neorhizobium galegae): анализ полиморфизма генов фиксации азота и развития клубеньков // Генетика. 2019. Т. 55. С. 234‒238.
Karasev E.S., Andronov E.E., Aksenova T.S., Chizhevskaya E.P., Tupikin A.E., Provorov N.A. Evolution of goat’s rue rhizobia (Neorhizobium galegae): analysis of polymorphism of the nitrogen fixation and nodule formation genes // Rus. J. Genetics. 2019. V. 55. P. 263‒266.
Кимеклис А.К., Кузнецова И.Г., Сазанова А.Л., Сафронова В.И., Белимов А.А., Онищук О.П., Курчак О.Н., Аксёнова Т.С., Пинаев А.Г., Мусаев А.М., Андронов Е.Е., Проворов Н.А. Дивергентная эволюция симбиотических бактерий: ризобии реликтового бобового Vavilovia formosa (Stev.) Fed. формируют обособленную группу в пределах вида Rhizobium leguminosarum bv. viciae // Генетика. 2018. Т. 54. С. 851‒855.
Kimeklis A.K., Kuznetsova I.G., Sazanova A.L., Safronova V.I., Belimov A.A., Onishchuk O.P., Kurchak O.N., Aksenova T.S., Pinaev A.G., Musaev A.M., Andronov E.E., Provorov N.A. The divergent evolution of symbiotic bacteria: the rhizobia of the relic legume Vavilovia formosa form an isolated group within the species Rhizobium leguminosarum bv. viciae // Rus. J. Genetics. 2018. V. 54. P. 866‒870.
Копать В.В., Чирак Е.Р., Кимеклис А.К., Сафронова В.И., Белимов А.А., Кабилов М.Р., Андронов Е.Е., Проворов Н.А. Эволюция генов fixNOQP, кодирующих цитохромоксидазу с высоким сродством к кислороду, у ризобий и родственных им бактерий // Генетика. 2017. 2017. Т. 53. С. 795‒804.
Kopat V.V., Chirak E.R., Kimeklis A.K., Safronova V.I., Belimov A.A., Kabilov M.R., Andronov E.E., Provorov N.A. Evolution of fixNOQP genes encoding cytochrome oxidase with high affinity to oxygen in rhizobia and related bacteria // Rus. J. Genetics. 2017. V. 53. P. 766–774.
Красильников Н.А., Мелкумова Т.А. Изменчивость клубеньковых бактерий внутри клубеньков бобовых растений // Изв. АН СССР. Сер. биол. 1963. № 5. С. 693‒706.
Майр Э. Популяции, виды и эволюция. М.: Мир, 1974. 141 с.
Проворов Н.А., Андронов Е.Е. Эволюция клубеньковых бактерий: реконструкция процессов видообразования, обусловленных перестройками генома в системе симбиоза // Микробиология. 2016. Т. 85. С. 195‒206.
Provorov N.A., Andronov E.E. Evolution of root nodule bacteria: reconstruction of the speciation processes resulting from genomic rearrangements in a symbiotic system // Microbiology (Moscow). 2016. V. 85. P. 131–139.
Проворов Н.А., Воробьев Н.И. Генетические основы эволюции растительно-микробного симбиоза. С.-Петербург: Информ-Навигатор, 2012. 400 с.
Проворов Н.А., Онищук О.П. Эволюционно-генетические основы симбиотической инженерии растений: мини-обзор // Сельскохозяйственная биология. 2018. Т. 53. С. 464‒474.
Provorov N.A., Onishchuk O.P. Evolutionary-genetic bases for symbiotic engineering in plants // Agric. Biol. 2018. V. 53. P. 464‒474.
Проворов Н.А., Онищук О.П., Юргель С.Н., Курчак О.Н., Чижевская Е.П., Воробьев Н.И., Затовская Т.В., Симаров Б.В. Конструирование высокоэффективных симбиотических штаммов бактерий: эволюционные модели и генетические подходы // Генетика. 2014. Т. 50. С. 1273‒1285.
Provorov N.A., Onishchuk O.P., Yurgel S.N., Kurchak O.N., Chizhevaskaya E.P., Vorobyov N.A., Zatovskaya T.V., Simarov B.V. Construction of highly effective symbiotic bacteria: evolutionary models and genetic approaches // Rus. J. Genetics. 2014. V. 50. P. 1125–1136.
Румянцева М.Л., Андронов Е.Е., Сагуленко В.В., Онищук О.П., Проворов Н.А., Симаров Б.В. Нестабильность криптических плазмид у штамма Sinorhizobium meliloti P108 в процессе симбиоза его с люцерной Medicago sativa // Генетика. 2004. Т. 40. С. 454‒461.
Roumiantseva M.L., Andronov E.E., Sagulenko V.V., Onishchuk O.P., Provorov N.A., Simarov B.V. Instability of cryptic plasmids in Sinorhizobium meliloti strain P108 in the course of symbiosis with alfalfa Medicago sativa // Rus. J. Genetics. 2004. V. 40. P. 356–362.
Тимофеев-Ресовский Н.В., Воронцов Н.Н., Яблоков А.В. Краткий очерк теории эволюции. М.: Наука, 1977. 297 с.
Цыганова А.В., Селиверстова Е.В., Онищук О.П., Курчак О.Н., Кимеклис А.К., Сазанова А.Л., Кузнецова И.Г., Сафронова В.И., Белимов А.А., Андронов Е.Е., Цыганов В.Е. Ультраструктурные особенности симбиотических клубеньков реликтовых бобовых // Растения и микроорганизмы: биотехнология будущего / Под ред. Тихоновича И.А. Уфа: АНО Центр поддержки академических инициатив, 2018. С. 246.
Чернова Т.А., Аронштам А.А., Симаров Б.В. Изучение генетической природы невирулентных мутантов СХМ1-125 и СХМ1-126 Rhizobium meliloti // Генетика. 1986. Т. 22. № 8. С. 2066‒2073.
Чирак Е.Р. Копать В.В., Кимеклис А.К., Сафронова, В.И., Белимов А.А., Чирак Е.Л., Тупикин А.Е., Андронов Е.Е., Проворов Н.А. Структурно-функциональная организация плазмидных регионов симбиотических генов у Rhizobium leguminosarum // Микробиология. 2016. Т. 85. С. 693‒702.
Chirak E.R., Kopat V.V., Kimeklis A.K., Safronova V.I., Belimov A.A., Chirak E.L., Tupikin A.E., Andronov E.E., Provorov N.A. Structural and functional organization of the plasmid regulons of Rhizobium leguminosarum symbiotic genes // Microbiology (Moscow). 2016. V. 85. P. 708–716.
Aguilar O.M., Riva O., Peltzer E. Analysis of Rhizobium etli and its symbiosis with Phaseolus vulgaris supports coevolution in centers of host diversification // Proc. Natl. Acad. Sci. USA. 2004. V. 101. P. 13548‒13553.
Allen O.N., Allen E.K. The Leguminosae. A Source Book of Characteristics, Uses and Nodulation. Madison: Univ. Wisconsin Press, 1981. 800 p.
Amarger N., Bours M., Revoy F., Allard M.R., Laguerre G. Rhizobium tropici nodulates field-growing Phaseolus vulgaris in France // Plant Soil. 1994. V. 161. P. 147‒156.
Andrews M., De Meyer S., James E.K., Stępkowski T., Hodge S. Horizontal transfer of symbiosis genes within and between rhizobial genera: occurrence and importance // Genes (Basel). 2018. V. 9. 7. P. 321‒331.
Bailly X., Olivieri I., De Mita S., Cleyet-Marel J.-C., Bena G. Recombination and selection shape the molecular diversity pattern of nitrogen-fixing Sinorhizobium sp. associated to Medicago // Mol. Ecol. 2006. V. 15. P. 2719–2734.
Bassam B.J., Mahanty H.K., Gresshoff P.M. Symbiotic interaction of auxotrophic mutants of Rhizobium trifolii with white clover (Trifolium repens) // Endocyt. C. Res. 1987. V. 4. P. 331‒347.
Bena G., Lyet A., Huguet T., Olivieri I. Medicago – Sinorhizobium symbiotic specificity evolution and the geographic expansion of Medicago // J. Evol. Biol. 2005. V. 18. P. 1547‒1558.
Bernal G., Graham P.H. Diversity on the rhizobia associated with Phaseolus vulraris L. in Ecuador, the comparison with Mexican bean rhizobia // Can. J. Microbiol. 2001. V. 47. P. 526‒534.
Berrada H., Fikri-Benbrahim K. Taxonomy of the rhizobia: current perspectives // British Microbiol. Res. J. 2014. V. 4. P. 616‒639.
Brom S., Girard L., Garcia-de los Santos A., Sanjuan-Pinilla J.M., Olivares J., Sanjuan J. Conservation of plasmid-encoded traits among bean-nodulating Rhizobium species // Appl. Environ. Microbiol. 2002. V. 68. P. 2555‒2561.
Broughton W.J., Samrey U., Stanley J. Ecological genetics of Rhizobium meliloti: symbiotic plasmid transfer in the Medicago sativa rhizosphere // FEMS Microbiol. Lett. 1987. V. 40. P. 251‒255.
Chen L.A., Figueredo A., Pedrosa F.O., Hungria M. Genetic characterization of soybean rhizobia in Paraguay // Appl. Environ. Microbiol. 2000. V. 66. P. 5099‒5103.
Devine T.E., Kuykendall L.D. Host genetic control of symbiosis in soybean (Glycine max L.) // Plant Soil. 1996. V. 186. P. 173‒187.
Eardly B.D., Wang F.-S., Whittam T.S., Selander R.K. Species limits in Rhizobium populations that nodulate the common bean (Phaseolus vulgaris) // Appl. Environ. Microbiol. 1995. V. 61. P. 507‒512.
Filipcenko J. Variabilität und Variation. Berlin: Bornträger, 1927. 215 s.
Jones J.D., Dangl J.L. The plant immune system // Nature. 2006. V. 444. P. 323‒329.
Kumar N., Lad G., Giuntini E., Kaye M.E., Udomwong P., Shamsani N.J., Young J.P.W., Bailly X. Bacterial genospecies that are not ecologically coherent: population genomics of Rhizobium leguminosarum // Open Biol. 2015. 5:140133. https://doi.org/10.1098/rsob.140133
Laguerre G., Geniaux E., Mazurier S.I., Rodrigues C.R., Amarger N. Conformity and diversity among field isolates of Rhizobium leguminosarum bv. viciae, bv. trifolii and bv. phaseoli revealed by DNA hybridization using chromosome and plasmid probes // Can. J. Microbiol. 1993. V. 39. P. 412‒419.
Laguerre G., Lauvrier P., Allard M.R., Amarger N. Compatibility of rhizobial genotypes within natural populations of Rhizobium leguminosarum biovar viciae for nodulation of host legumes // Appl. Environ. Microbiol. 2003. V. 69. P. 2276‒2283.
Laguerre G., Nour S.M., Macheret V., Sanjuan J., Drouin P., Amarger N. Classification of rhizobia based on nodC and nifH gene analysis reveals a close phylogenetic relationship among Phaseolus vulgaris symbionts // Microbiology (SGM). 2001. V. 147. P. 981‒993.
Lie T.A., Göktan D., Engin M., Pijnenborg J., Anlarsal E. Co-evolution of the legume-Rhizobium association // Plant Soil. 1987. V. 100. P. 171‒181.
Litchman E. Invasive invaders: non-pathogenic invasive microbes in aquatic and terrestrial ecosystems // Ecology Lett. 2010. V. 13. P. 1560‒1572.
Mikič A., Smýkal P., Kenicer G., Vishnyakova M., Sarukhanyan N., Akopian J., Vanyan A., Gabrielyan I., Smýkalová I., Sherbakova E. The bicentenary of the research on ‘beautiful’ vavilovia (Vavilovia formosa), a legume crop wild relative with taxonomic and agronomic potential // Bot. J. Linn. Soc. 2013. V. 172. P. 524–531.
Minamisawa K., Itakura M., Suzuki M., Ichige K., Isawa T., Yuhashi K., Mitsui H. Horizontal transfer of nodulation genes in soils and microcosms from Bradyrhizobium japonicum to B. elkanii // Microbes Environ. 2002. V. 17. P. 82‒90.
Mornico D., Miché L., Béna G., Nouwen N., Verméglio A., Vallenet D., Smith A.T., Giraud E., Médigue C., Moulin L. Comparative genomics of Aeschynomene symbionts: insights into the ecological lifestyle of nod-independent photosynthetic bradyrhizobia // Genes. 2012. V. 3. P. 35‒61.
Moulin L., Béna G., Boivin-Masson C., Stepkowski T. Phylogenetic analyses of symbiotic nodulation genes support vertical and lateral gene co-transfer within the Bradyrhizobium genus // Mol. Phylogenet. Evol. 2004. V. 30. P. 720‒732.
Mousavi S.A., Österman J., Wahlberg N., Nesme X., Lavire C., Vial L., Paulin L., de Lajudie P., Lindström K. Phylogeny of the Rhizobium–Allorhizobium–Agrobacterium clade supports the delineation of Neorhizobium gen. nov. // Syst. Appl. Microbiol. 2014. V. 37. P. 208–215.
Mutch L.A., Young J.P.W. Diversity and specificity of Rhizobium leguminosarum biovar viciae on wild and cultivated legumes // Mol. Ecol. 2004. V. 13. P. 2435‒2444.
Nandasena K.G., O’Hara G.W., Tiwari R.P., Howieson J.G. Rapid in situ evolution of nodulating strains for Biserrula pelecinus L. through lateral transfer of a symbiosis island from the original mesorhizobial inoculant // Appl. Environ. Microbiol. 2006. V. 72. P. 7365‒7367.
Oda Y., Larimer F.W., Chain P.S., Malfatti S., Shin M.V., Vergez L.M., Hauser L., Land M.L., Braatsch S., Beatty J.T., Pelletier D.A., Schaefer A.L., Harwood C.S. Multiple genome sequences reveal adaptations of a phototrophic bacterium to sediment microenvironments // Proc. Natl. Acad. Sci. USA. 2008. V. 105. P. 18543‒18548.
Okazaki S., Noisangiam R., Okubo T., Kaneko T., Oshima K., Hattori M., Teamtisong K., Songwattana P., Tittabutr P., Boonkerd N., Saeki K., Sato S., Uchiumi T., Minamisawa K., Teaumroong N. Genome analysis of a novel Bradyrhizobium sp. DOA9 carrying a symbiotic plasmid // PLoS One. 2015. V. 10. e0117392. https://doi.org/10.1371/journal.pone.0117392
Pretorius-Güth I.M., Pühler A., Simon R. Conjugal transfer of megaplasmid 2 between Rhizobium meliloti strains in alfalfa nodules // Appl. Environ. Microbiol. 1990. V. 56. P. 2354‒2359.
Provorov N.A., Vorobyov N.I. Interplay of Darwinian and frequency-dependent selection in the host-associated microbial populations // Theor. Population Biol. 2006. V. 70. P. 262‒272.
Qian J., Parker M.P. Contrasting nifD and ribosomal gene relationships among Mesorhizobium from Lotus oroboides in Northern Mexico // Syst. Appl. Microbiol. 2002. V. 25. P. 68‒73.
Richardson D.M., Allsopp N., d’Antonio C.M., Milton S.J., Rejmanek M. Plant invasions – the role of mutualisms // Biol. Rev. 2000. V. 75. P. 65‒93.
Rome S., Fernandez M.P., Brunel M.P., Normand P., Cleyet-Marel J.C. Evidence that two genomic species of Rhizobium are associated with Medicago truncatula // Int. J. System. Bacteriol. 1996. V. 46. P. 972‒980.
Ronald A. The etiology of urinary tract infection: traditional and emerging pathogens // Dis. Mon. 2003. V. 49. P. 71–82. https://doi.org/10.1067/mda.2003.8
Safronova V.I., Kimeklis A.K., Chizhevskaya E.P., Belimov A.A., Andronov E.E., Pinaev A.G., Pukhaev A.R., Popov K.P., Tikhonovich I.A. Genetic diversity of rhizobia isolated from nodules of the relic species Vavilovia formosa (Stev.) Fed. // Antonie Van Leeuwenhoek. 2014. V. 105. P. 389–399.
Safronova V.I., Kuznetsova I.G., Sazanova A.L., Kimeklis A.K., Belimov A.A., Andronov E.E., Pinaev A.G., Pukhaev A.R., Popov K.P., Akopian J.A., Willems A., Tikhonovich I.A. Extra-slow-growing Tardiphaga strains isolated from nodules of Vavilovia formosa (Stev.) Fed. // Arch. Microbiol. 2015. V. 197. P. 889‒898.
Seguin P., Graham P.H., Sheaffer C.C., Ehlke N.J., Russelle M.P. Genetic diversity of rhizobia nodulating Trifolium ambiguum in North America // Can. J. Microbiol. 2001. V. 47. P. 81‒85.
Sprent J.I. Nodulation in Legumes. Kew Royal Botanical Gardens: Cromwell Press Ltd., 2001. 102 p.
Sprent J.I. West African legumes: the role of nodulation and nitrogen fixation // New Phytologist. 2005. V. 167. P. 326‒330.
Stepkowski T., Moulin L., Krzyianska A., McInnes A., Law I.J., Howieson J. European origin of Bradyrhizobium populations infecting lupins and seradella in soils of Western Australia and South Africa // Appl. Environ. Microbiol. 2005. V. 71. P. 7041‒7052.
Sullivan J.T., Patrick H.N., Lowther W.L., Scott D.B., Ronson C.W. Nodulating strains of Rhizobium loti arise through chromosomal symbiotic gene transfer in the environment // Proc. Natl. Acad. Sci. USA. 1995. V. 92. P. 8985‒8989.
Sullivan J.T., Trzebiatowski J.R., Cruickshank R.W., Gouzy J., Brown S.D., Elliot R.M., Fleetwood D.J., McCalum N.G., Rossbach U., Stuart G.S., Weaver J.E., Webby R.J., de Bruijn F., Ronson C. Comparative sequence analysis of the symbiosis island of Mesorhizobium loti strain R7A // J. Bacteriol. 2002. V. 184. P. 3086‒3095.
Thompson J.N., Burdon J. Gene-for-gene co-evolution between plants and parasites // Nature. 1992. V. 360. P. 121‒125.
Tian C.F., Zhou Y.L., Zhang Y.M., Li Q.Q., Zhang Y.Z., Li D.F., Wang S., Wang J., Gilbert L.B., Li Y.L., Chen W.X. Comparative genomics of rhizobia nodulating soybeans suggests extensive recruitment of lineage-specific genes in adaptations // Proc. Natl. Acad. Sci. USA. 2012. V. 109. P. 8629‒8634.
Triplett E.V., Sadowsky M.E. Genetics of competition for nodulation of legumes // Annu. Rev. Microbiol. 1992. V. 46. P. 399‒428.
Ulrich A., Zaspel I. Phylogenetic diversity of rhizobial strains nodulating Robinia pseudoacacia L. // Microbiology (SGM). 2000. V. 146. P. 2997‒3005.
van der Putten W.H., Klironomos J.N., Wardle D.A. Microbial ecology of biological invasions // IMSE J. 2007. V. 1. P. 28‒37.
Wang D., Yang S., Tang F., Zhu H. Symbiosis specificity in the legume – rhizobial mutualism // Cell Microbiol. 2012. V. 14. P. 334–342.
Wang E.T., van Berkum P., Sui X.H., Beyene D., Chen W., Martinez-Romero E. Diversity of rhizobia associated with Amorpha fruticosa isolated from Chinese soils and description of Mesorhizobium amorphae sp. nov. // Int. J. Syst. Bacteriol. 1999. V. 49. P. 51‒65.
Williams K.P., Gillespie J.J., Sobral B.W.S., Nordberg E.K., Snyder E.E., Shallom J.M., Dickerman A.W. Phylogeny of Gammaproteobacteria // J. Bacteriol. 2010. V. 192. P. 2305‒2314.
Wojciechowski M.F., Lavin M., Sanderson M.J. A phylogeny of legumes (Leguminosae) based on analysis of the plastid matK gene resolves many well-supported subclades within the family // Amer. J. Bot. 2004. V. 91. P. 1846‒1862. https://doi.org/10.3732/ajb.91.11.1846
Дополнительные материалы отсутствуют.
Инструменты
Микробиология


