Микробиология, 2020, T. 89, № 1, стр. 17-33
Современные подходы к дифференциации живых и мертвых бактерий с помощью избирательной амплификации нуклеиновых кислот
Ан. Х. Баймиев a, *, Ал. Х. Баймиев a, Б. Р. Кулуев a, К. Ю. Швец a, d, Р. С. Ямиданов a, Р. Т. Матниязов a, Д. А. Чемерис a, В. В. Зубов b, Я. И. Алексеев c, А. Р. Мавзютов d, Я. А. Иваненков a, e, f, А. В. Чемерис a
a Институт биохимии и генетики УФИЦ РАН
450054 Уфа, Россия
b Институт теоретической и экспериментальной биофизики РАН
142290 Пущино, Россия
c ООО “Синтол”
127550 Москва, Россия
d Башкирский государственный медицинский университет
450000 Уфа, Россия
e Московский физико-технический институт (государственный университет)
117303 Москва, Россия
f Московский государственный университет имени М.В. Ломоносова
119991 Москва, Россия
* E-mail: baymiev@anrb.ru
Поступила в редакцию 07.05.2019
После доработки 01.07.2019
Принята к публикации 29.09.2019
Аннотация
Специфическая амплификация нуклеиновых кислот является удобной и быстрой альтернативой культуральному методу выявления бактериальных клеток. Однако обычная ПЦР и прочие реакции амплификации не в состоянии дифференцировать живые бактерии от мертвых или покоящихся их форм, поскольку способны амплифицировать ДНК, сохраняющуюся довольно длительное время и в бесклеточном состоянии. Для установления жизнеспособности микроорганизмов с помощью амплификации специфичных последовательностей нуклеиновых кислот разработано несколько методов, как регулируемых сменой температур, так и протекающих изотермически. Для части из них мишенью служит модифицированная фенантридиновыми красителями ДНК, причем показано, что предпочтительным является одновременное использование моноазидов этидия и пропидия. Для других методов мишенями выступают непосредственно молекулы РНК или их кДНК копии. Весьма перспективной представляется детекция пре-рРНК, ввиду присутствия этих типов РНК исключительно в живых клетках.
Одной из актуальных проблем прикладной микробиологии остается необходимость надежной, высокочувствительной и специфичной детекции жизнеспособных микроорганизмов в различных средах, проводимой как для диагностики инфекционных заболеваний, так и для оценки санитарного состояния объектов внешней среды (вода, воздух, пищевые продукты и др.). “Золотым стандартом” в настоящее время является культуральный метод, который, однако, имеет ряд существенных недостатков, обусловленных, в частности, значительной продолжительностью исследования. Кроме того, значительная часть микроорганизмов, имеющих медицинское и/или ветеринарное значение, не культивируются в обычных условиях. Таковыми являются практически все известные в настоящее время облигатные и факультативные внутриклеточные паразиты или анаэробные бактерии. Поэтому после появления полимеразной цепной реакции (ПЦР) данный метод стал активно использоваться и в микробиологии для обнаружения конкретных микроорганизмов в анализируемом материале путем амплификации специфичных последовательностей ДНК.
Начало современным подходам к дифференциации живых и мертвых бактериальных клеток с помощью амплификации было положено в середине 1990-х гг., когда в качестве мишеней стали использовать граничные области рРНК и пре-рРНК. Новый импульс таким исследованиям в начале 2000-х гг. придало применение этидия моноазида (Ethidium monoazide, EMA), способного проникать только в мертвые бактерии и интеркалироваться в цепочки ДНК, приводя к невозможности ее амплификации с помощью ПЦР (Nogva et al., 2003). За прошедшие полтора десятилетия с момента разработки такого подхода для дифференциации жизнеспособных и погибших бактерий с помощью ПЦР с фенантридиновыми красителями (EMA, а также PMA – Propidium monoazide или пропидий моноазид) накоплен определенный опыт, что стало основанием для его обобщения и анализа в данном обзоре, в дополнение к которому рассмотрены также и другие способы амплификации нуклеиновых кислот для дифференциации живых и мертвых бактерий. Однако прежде чем приступить к их описанию, необходимо кратко остановиться на статусах жизнеспособности бактерий.
В настоящее время выделяют несколько основных форм жизненного статуса бактерий, среди которых, помимо спор, признается, по крайней мере, еще три: живые (live), мертвые (dead) и покоящиеся бактерии. Последний вариант в литературе принято обозначать как VBNC – viable but non-culturable. Подобный VBNC статус бактериальные клетки могут приобретать, в том числе, под действием бактериостатиков или сублетальных концентраций антибиотиков или прочих бактерицидных препаратов, либо дезинфектантов. В одной из работ (Davey, 2011) можно встретить и большее число стадий между живыми и мертвыми клетками. Так, живая клетка с активным метаболизмом (1) может превратиться сначала в живую клетку со сниженной метаболической активностью (2), затем в клетку с уменьшенным количеством РНК (3), далее она может стать интактной клеткой без признаков метаболической активности (4). Следующие стадии жизненного статуса бактериальных клеток выражаются уже в нарушении целостности мембран (5), затем в деградации ДНК (6) и, наконец, в образовании обломков клеток (7). Причем отмечается, что так называемая “точка невозврата”, после преодоления которой восстановление жизнеспособности бактерий, испытывающих стрессовые воздействия, уже невозможно, неизвестна, как и аналогичная точка гибели бактерий (Davey, 2011). При этом они могут различаться в зависимости от видов бактерий и окружающих условий. Подробнее о жизненных состояниях бактерий, включая покоящиеся клетки, можно прочесть в ряде обзоров (Kell et al., 1998; Barer, Harwood, 1999; Oliver, 2010; Pinto et al., 2013).
СПОСОБЫ ДИФФЕРЕНЦИАЦИИ ЖИВЫХ И МЕРТВЫХ БАКТЕРИЙ ПУТЕМ АМПЛИФИКАЦИИ ФРАГМЕНТОВ РНК ИЛИ ДНК БЕЗ ПРЕДВАРИТЕЛЬНОЙ МОДИФИКАЦИИ ПОСЛЕДНИХ
После появления ПЦР этот метод стал активно использоваться для детекции бактерий в различных средах. Первая такая работа, направленная на детекцию токсигенных Escherichia coli (Olive et al., 1988), была выполнена в Кувейтском университете еще с использованием первого малоэффективного варианта ПЦР с Кленовским фрагментом ДНК полимеразы I E. coli. Чувствительность метода составила не более 1000 бактерий, которые авторы обнаружили через 24 цикла ПЦР и последовавшей гибридизации ампликона размером 299 п.н. с меченым ДНК-зондом. Позже метод был усовершенствован, что позволило обнаруживать E. coli после 30 циклов амплификации с использованием уже термостабильной ДНК полимеразы непосредственно гель-электрофорезом (Olive, 1989). Тогда же была показана возможность практического применения ПЦР для детекции патогенных бактерий (Bernet et al., 1989; Dutilh et al., 1989; Rosa, Schwan, 1989). Особый интерес был проявлен к возможности выявления бактерий, вызывающих серьезные заболевания, но не поддающихся культивированию in vitro. Так, сообщалось о детекции с помощью ПЦР возбудителя проказы ‒ Mycobacterium leprae (Hartskeerl et al., 1989). Спустя несколько лет была опубликована еще одна статья (Jamil et al., 1993), где уже при выявлении жизнеспособных M. leprae вели амплификацию фрагментов ДНК с заметно отличающимися размерами ‒ 571 и 285 п.н.
Довольно быстро пришло понимание того, что молекулы ДНК могут сохраняться в мертвых бактериях и даже в окружающей среде в свободном состоянии, из-за чего их выявление с помощью ПЦР не давало главного ответа о наличии в исследуемых образцах именно жизнеспособных микроорганизмов, способных оказывать воздействие на другие живые организмы, включая человека, или на окружающую среду. В этой связи более пригодными для детекции с помощью ПЦР, сопряженной с обратной транскрипцией (ОТ-ПЦР), стали рассматривать молекулы РНК, поскольку их время жизни значительно меньше, чем ДНК. Так, было показано, что путем амплификации с помощью ПЦР фрагмента кДНК размером 650 п.н., кодирующего mip РНК Legionella pneumonia, возможно дифференцировать не только мертвые и живые бактерии, но и считающиеся VBNC (Bej et al., 1991). Несколько позже этими авторами данный подход был применен и для дифференциации живых и мертвых бактерий Vibrio cholerae (Bej et al., 1996). Применение ОТ-ПЦР, в которой мишенями для амплификации служили несколько видов мРНК, обеспечившие разную степень чувствительности, оказалось успешным при выявлении живых бактерий Listeria monocytogenes в мясных продуктах (Klein, Juneja, 1997). Сообщалось о пригодности метода ОТ-ПЦР с использованием в качестве мишеней мРНК для дифференциации нескольких видов живых бактерий (Sheridan et al., 1998; Lleò et al., 2000). Причем в одной из этих статей (Sheridan et al., 1998) отмечалось также, что использование в качестве мишеней молекул 16S рРНК при детекции с помощью ОТ-ПЦР не позволяет направленно дифференцировать жизнеспособные бактерии ввиду значительного количества в клетках зрелых рРНК, что приводит к искажению результатов. Однако имеются сообщения и об успешном использовании рРНК для дифференциации живых и мертвых бактерий E. coli и Staphylococcus aureus (McKillip et al., 1998). Для ликвидации ложно-позитивных результатов при выявлении жизнеспособных микроорганизмов путем амплификации геномной ДНК вместо мРНК, было предложено использовать повторные обработки препарата РНК ДНКазой до полного исчезновения целевых ампликонов в обычной ПЦР и только после этого проводить ОТ-ПЦР (Kobayashi et al., 2009).
Однако ОТ-ПЦР является не самым идеальным методом обнаружения жизнеспособных бактерий по целому ряду причин. Среди них ‒ необходимость удаления ДНК из исследуемых образцов с помощью ДНКазы, что само по себе непросто ввиду наличия в образце остаточных количеств нуклеиновых кислот этого типа. Возможны загрязнения ДНКазы РНКазой, что может частично или даже полностью разрушить искомую РНК. Также необходимо учитывать, что, ввиду сверхвысокой чувствительности ПЦР при проведении таких экспериментов по обнаружению бактериальных клеток, крайне важно контролировать появление как ложно-позитивных результатов, вызванных загрязнением рабочей зоны или иными причинами, так и ложно-негативных результатов, к которым могли приводить, в том числе, присутствующие в опытных образцах ингибиторы ПЦР различной природы (Чемерис и соавт., 2011, 2012а, 2012б).
Помимо ОТ-ПЦР для детекции бактериальной РНК с целью установления жизненного статуса микроорганизмов было предложено использовать не вовлекающий исходные двуцепочечные молекулы ДНК в этот процесс изотермический метод амплификации, получивший название NASBA (Nucleic Acid Sequence Based Amplification), и на некоторых видах бактерий это было продемонстрировано (Vliet et al., 1994; Simpkins et al., 2000), в том числе с регистрацией результатов амплификации в режиме реального времени (Fykse et al., 2007). Сравнение методов ПЦР, ОТ-ПЦР и NASBA для дифференциации живых и мертвых бактерий показало, что последний отличает наивысшая чувствительность (Birch et al., 2001). Помимо NASBA для детекции мРНК в живых бактериях Mycobacterium tuberculosis использовался совмещенный с обратной транскрипцией метод RT-SDA (Reverse Transcriptase Strand Displacement Amplification), также позволяющий вести амплификацию в изотермических условиях (Hellyer et al., 1999).
Было показано, что использование в ОТ-ПЦР в качестве мишени участка интактной пре-рРНК позволяет отделять устойчивые к действию антибиотиков бактерии M. tuberculosis от неустойчивых (Cangelosi et al., 1996). Причем подбор мест отжига одного из праймеров на участке зрелой 16S рРНК, а другого – на участке, удаляемом во время процессинга, дал возможность детектировать только что образовавшиеся во время транскрипции молекулы пре-рРНК. Однако лишь спустя полтора десятилетия был предложен фактически новый метод дифференциации живых и мертвых бактерий, получивший в итоге название Molecular Viability Testing (MVT) (Cangelosi et al., 2010; Weigel et al., 2017). Такое молекулярное тестирование жизнеспособности бактериальных клеток основано на амплификации с помощью ОТ-ПЦР фрагмента исключительно интактной пре-рРНК благодаря подбору мест отжига праймеров для синтеза кДНК и амплификации на участке как зрелой рРНК, так и на существующем короткое время участке пре-рРНК. Авторы метода утверждают, что он обеспечивает наиболее точное выявление жизненного статуса микроорганизмов, поскольку известно, что в покоящихся клетках новые пре-РНК не образуются, а синтезировавшиеся ранее не сохраняются, тогда как при добавлении питательных веществ покоящиеся клетки могут очень быстро переходить в статус метаболически активных живых клеток, что сопровождается появлением новых пре-рРНК. Причем, применимость MVT на основе детекции с помощью ОТ-ПЦР интактной пре-рРНК для дифференциации жизненных состояний микроорганизмов была продемонстрирована для ряда медленно- и быстрорастущих видов бактерий из разных филогенетических групп M. tuberculosis, Acinetobacter baumanii, Pseudomonas aeruginosa, Staphylococcus aureus. Отмечается, что метод MVT дает более точную оценку жизненного статуса бактериальных клеток, по сравнению с широко используемыми методами, основанными на детекции ДНК бактерий, после воздействия на них фенантридиновыми красителями (Cangelosi, Meschke, 2014), к рассмотрению которых мы, тем не менее, сейчас перейдем.
СПОСОБЫ ДИФФЕРЕНЦИАЦИИ ЖИВЫХ И МЕРТВЫХ БАКТЕРИЙ ПУТЕМ АМПЛИФИКАЦИИ С ПОМОЩЬЮ ВИТАЛЬНОЙ ПЦР ФРАГМЕНТОВ ДНК ИЛИ РНК, ОСНОВАННЫЕ НА ПРИМЕНЕНИИ ФЕНАНТРИДИНОВЫХ КРАСИТЕЛЕЙ
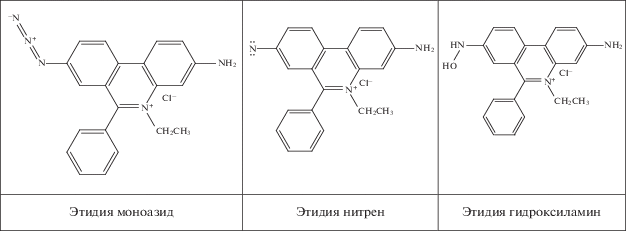
Использование модификации ДНК фенантридиновыми красителями было основано на знании того, что этидий моноазид (3-амино-8-азидо-5-этил-6-фенилфенантридиний хлорид) проникает через поврежденную мембрану мертвых бактериальных клеток и связывается с ДНК, вызывая флуоресценцию, которую можно детектировать с помощью проточного цитофлуориметра, подсчитывая, таким образом, неживые микроорганизмы (Riedy et al., 1991). Важно то, что после освещения видимым светом комплекс ДНК/этидий моноазид, как считалось, становится необратимым и при этом некоторые свойства ДНК меняются. К тому же из предшествующих работ было известно, что под действием света из этидия моноазида, интеркалирующего в цепочки ДНК, образуется крайне реакционноспособный нитрен (Knowles, 1972), который и взаимодействует с ДНК, а свободный этидий моноазид в результате фотолиза в присутствии воды превращается в относительно инертный гидроксиламин этидия (Graves et al., 1981), структурные формулы которых представлены на рис. 1.
Идея метода состояла в том, что проникший через поврежденную мембрану внутрь мертвых бактерий в темноте этидий моноазид после освещения видимым светом необратимо свяжется с клеточной ДНК, после чего такая ДНК в ПЦР вовлекаться не будет. Для количественной оценки живых/мертвых бактерий необходимо проводить ПЦР в режиме реального времени после разделения анализируемого образца на две части, одна из которых обрабатывается фенантридиновым красителем, а вторая не подвергается никаким воздействиям. После этого из обоих препаратов производится выделение ДНК подходящим методом и проводится амплификация нуклеиновых кислот, по соотношению продуктов которых можно судить о пропорциях живых и мертвых бактерий в анализируемом образце. Причем этидий моноазид, разрушившийся за счет фотолиза, уже не в состоянии взаимодействовать в растворе с выделенной нативной ДНК из живых клеток.
Использование в ПЦР этидия моноазида (ЭМА-ПЦР)
Первая экспериментальная статья, в которой было сообщено об успешном применении в ПЦР этидия моноазида для дифференциации мертвых и живых бактерий, была опубликована в 2003 году (Nogva et al., 2003). В этой работе в качестве объектов были взяты следующие микроорганизмы: ряд штаммов токсикогенного варианта E. coli O157:H7, Listeria monocytogenes и Salmonella sp., которые убивали высокой температурой и химическими дезинфектантми. Были исследованы различные концентрации этидия моноазида вкупе с варьированием времени обработки, включая продолжительность и мощность освещения. Для сопоставления результатов определения жизненного статуса образцов использовалась стандартная микроскопия с коммерческим набором LIVE/DEAD BacLight (“Molecular Probes, Inc.”, сейчас “ThermoFisher Scientific”) с двумя красителями SYTO 9 и Propidium iodide, подтвердившая пригодность теста на жизнеспособность испытуемых микроорганизмов с помощью количественной ПЦР с этидием моноазидом. На примере изолятов бактерии Campylobacter jejuni из различных пищевых продуктов было показано превосходство метода ЭМА-ПЦР над микроскопической детекцией, в том числе благодаря возможности игнорирования посторонней микрофлоры (Rudi et al., 2005). Метод ЭМА-ПЦР был адаптирован для дифференциации живых и мертвых бактерий в почве (Pisz et al., 2007), в воде (Inoue et al., 2008). Было показано селективное удаление ДНК из мертвых клеток в бактериальной смеси после воздействия этидием моноазидом (Nocker, Camper, 2006), что даже вызвало определенную дискуссию (Hein et al., 2006). Оказалось, что в данном случае имеет место не необратимое связывание этидия моноазида с ДНК, а фрагментация последней (Soejima et al., 2007). Так, обратив внимание, что количество выделенной ДНК из неживых бактерий после обработки таких поврежденных клеток этидием моноазидом заметно уменьшается, в ходе этой работы было предпринято специальное исследование, чтобы выяснить, что происходит с ДНК в действительности, тем более, что подобный эффект наблюдали и ранее (Nocker, Camper, 2006). Анализ образующихся in vitro продуктов с помощью некоторых вариантов гель-электрофореза, а также электронной микроскопии показал, что в двуцепочечной ДНК, остающейся при сопряженной с освещением обработке этидием моноазидом, возникают одноцепочечные разрывы, и когда их за счет увеличения концентрации в реакционной смеси этидия моноазида становится довольно много, то это приводит фактически и к двуцепочечным разрывам (Soejima et al., 2007). Полученные результаты позволили авторам высказать некоторые предположения относительно механизма разрушения цепей ДНК и заставили по-новому взглянуть на проведение ЭМА-ПЦР для дифференциации мертвых и живых бактерий. Так понимая, что в результате проникновения этидия моноазида в поврежденные бактериальные клетки вследствие его взаимодействия с внутриклеточной ДНК и после воздействия светом, происходит разрушение целостности бактериальной ДНК, оценивалась вероятность наработки ампликонов разного размера у Listeria monocytogenes (Soejima et al., 2008). Оказалось, что в этих образцах ампликон размером 894 п.н. не амплифицировался, тогда как более короткий, размером 113 п.н., все же детектировался, что дополнительно подтверждает происходящую фрагментацию ДНК за счет возникновения разрывов цепей ДНК.
Следует отметить, что в одной из работ на примере двух видов бактерий Campylobacter jejuni и Listeria monocytogenes сообщалось, что ЭМА-ПЦР в реальном времени не обеспечивала должной дифференциации между живыми и мертвыми клетками (Flekna et al., 2007). Тем не менее, после внедрения в практику таких анализов более подходящего пропидия моноазида, этидий моноазид использоваться не перестал (Fukuzawa et al., 2019), и одной из причин является более низкая стоимость последнего. Для повышения достоверности таких исследований на примере Enterobacter sakazakii былa предложена повторная обработка анализируемых образцов низкими концентрациями этидия моноазида (Minami et al., 2010).
Использование в ПЦР пропидия моноазида
Исходя из того, что для дифференциации живых и мертвых бактерий с помощью микроскопии и проточной цитофлуорометрии давно использовался пропидий йодид, проникающий только через мембраны потерявших жизнеспособность бактериальных клеток (Nebe-von Caron et al., 1998), было предложено для витальной ПЦР (виПЦР) использовать, изображенный на рис. 2, его аналог ‒ пропидий моноазид (3-амино-8-азидо-5-{3-[диэтил(метил)аммонио]пропил}-6-фенилфенантридиний дихлорид), который впервые применен в 2006 г. (Nocker et al., 2006). Позднее был выполнен большой цикл экспериментальных работ, где на широком спектре видов микроорганизмов с использованием количественной ПЦР, ДНК-чиповой технологии, пиросеквенирования (Nocker et al., 2007, 2009, 2010; Contreras et al., 2011; Yáñez et al., 2011; Schnetzinger et al., 2013) продемонстрировано успешное применение пропидия моноазида для дискриминации живых и мертвых бактерий.
Значительное число статей посвящено вопросам количественной детекции с помощью ПЦР в реальном времени живых и мертвых бактерий, в том числе, после различных дезинфекционных обработок с целью слежения за численностью бактерий в анализируемых образцах (Bae, Wuertz, 2009; Wahman et al., 2009; Yokomachi, Yaguchi, 2012; Kaushik, Balasubramanian, 2013; Xing-Long et al., 2013; Lee et al., 2015; Kibbee, Örmeci, 2017; Zhou et al., 2017). Для определения численности живых бактерий в сообществе микроорганизмов из сточных вод до и после проведенной дезинфекции, а также в фекальных образцах использовали цифровую монокапельную ПЦР вкупе с пропидием моноазидом (Gobert et al., 2018).
Витальная количественная ПЦР в реальном времени с пропидием моноазидом была применена для выявления живых и мертвых бактерий Chlamydia trachomatis, и авторами было отмечено, что это первая работа по виПЦР для контроля численности возбудителей для заболеваний, передающих половым путем (Janssen et al., 2016). Причем в этой статье, помимо анализа клинических образцов, проводились модельные эксперименты для валидации данного метода, подтвердившие его пригодность при антибиотикотерапии хламидиоза. Продемонстрировано, что использование ПЦР после обработки микрофлоры пропидием моноазидом имеет определенный потенциал для контроля за процессом лечения туберкулезных больных (Nikolayevskyy et al., 2015). Недавно показано, что наряду с пропидием моноазидом, для оценки действия антибиотиков на Pseudomonas aeroginosa может использоваться и подход с детекцией пре-рРНК также с помощью ПЦР (Lee, Bae, 2018a).
Сообщено о проведении мультиплексной виПЦР в режиме реального времени после обработки образцов воды пропидием моноазидом, в которой одновременно детектировались живые микроорганизмы трех видов – Legionella pneumophyla, Salmonella typhimurium и Staphylococcus aureus (Li et al., 2015). Ранее с использованием магнитной иммуносепарации мультиплексная виПЦР с пропидием моноазидом была применена для одновременной детекции в пищевых продуктах жизнеспособных бактерий Legionella monocytogenes, Salmonella typhimurium и E. coli O157:H7 (Yang et al., 2013).
Однако следует заметить, что в литературе встречаются публикации, в которых сообщается о завышении количества живых бактерий в анализируемых образцах при проведении виПЦР с пропидием моноазидом (Lovdal et al., 2011; Ditommaso et al., 2014). Для достижения лучшей дискриминации между живыми и мертвыми бактериями путем увеличения проницаемости поврежденных мембран бактериальных клеток для пропидия моноазида в ряде работ использовали такие детергенты как дезоксихолат натрия, саркозил, додецилсульфат натрия, тритон Х-100, а также хелатирующий агент ЭДТА (Wang et al., 2014a, 2014b; Dong et al., 2018; Zi et al., 2018; Kontchou et al., 2019). С этой же целью был использован диметилсульфоксид (Seidel et al., 2017).
При анализе соотношения живых и мертвых бактерий Legionella pneumophila, преднамеренно внесенных в сточные воды, в том числе после проведенных дезинфекций, с помощью виПЦР с пропидием моноазидом на примере данной модельной системы было обнаружено, что нельзя исключать получение как ложно-позитивных, так и ложно-негативных результатов (Fittipaldi et al., 2011). В нескольких статьях сообщается об успешной обработке микроорганизмов пропидием моноазидом, включая воздействие светом, непосредственно на мембранных фильтрах, которые были использованы для концентрирования бактериальных клеток из водных образцов (Slimani et al., 2012; Villarreal et al., 2013). Помимо выявления жизненного статуса бактерий, количественная ПЦР с пропидием моноазидом была успешно применена для установления жизнеспособности спор Bacillus subtilis (Rawsthorne et al., 2009).
Довольно продолжительное время при проведении виПЦР с фенантридиновыми красителями воздействие видимым светом осуществлялось с помощью мощных галогеновых ламп, приводящих к нагреву образцов, из-за чего требовалось их тем или иным образом охлаждать. После того как было предложено использовать светодиодные лампы синего света с длинами волн от 445 до 485 нм (Vesper et al., 2008), проблема перегрева была снята. Хотя до сих пор при проведении виПЦР применяются галогеновые лампы, все же сейчас преимущественно используются светодиодные источники света, и несколькими фирмами производятся подобные осветители. Так, фирмой “Biotium, Inc.” выпускается два таких устройства ‒ PMA-Lite и Glo-Plate, предназначенные для воздействия синим светом на связавшийся с бактериальной ДНК пропидий моноазид в тестовых пробирках или в планшетах. Этой фирмой также производится целая линейка реактивов и готовых наборов для дифференциации живых и мертвых бактерий, включая новый краситель PMAxx, про который сообщается, что это новая улучшенная версия пропидия моноазида. Также следует заметить, что исключающее нагрев образцов осветительное устройство BLU-V System производит известная фирма “Qiagen”.
Пожалуй, здесь нельзя не коснуться двух недавних работ, посвященных дифференциации живых и мертвых бактериальных клеток и выполненных разными группами авторов (Huang et al., 2018; Willers et al., 2017). В одной из них (Huang et al., 2018) для определения жизненного статуса бактерий E. coli проведено сравнение эффективности культурального метода, виПЦР и проточной цитофлуорометрии, основанной на использовании уже упоминавшегося набора LIVE/DEAD BacLight с красителем SYTO 9 (окрашивающим в зеленый цвет все клетки) и йодидом пропидия (окрашивающим в красный цвет только мертвые клетки). В данном исследовании оценивались многочисленные параметры (продолжительность как подготовительных этапов, так и время, затрачиваемое непосредственно на проведение эксперимента, а также период ожидания конечного результата; стоимость анализов, их массовость (параллелизм) и др.). В целом, если учитывать, что с помощью проточной цитофлуорометрии (хотя и занимающей мало времени) невозможно отличить бактерии разных видов, а метод виПЦР позволяет амплифицировать избирательно только нужную мишень, то следует признать его перспективность. В другой работе (Willers et al., 2017) сравнивалась эффективность РМА-ПЦР и проточной цитофлуориметрии, в которой вместо стандартного набора LIVE/DEAD BacLight использовался один краситель – моноазид пропидия. Несмотря на продемонстрированную быстроту подобного анализа, необходимо заметить, что в эксперименте использовались чистые культуры Helicobacter pylori, однако при применении данного подхода для реальных образцов, потенциально содержащих множество, в том числе, неизвестных бактерий, он не выдержит конкуренции с виПЦР, главным образом из-за неспецифичности детекции жизнеспособных бактерий.
Совместное использование в ПЦР этидия и пропидия моноазидов
С того момента, как было предложено для дифференциации живых и мертвых бактерий использовать такой фенантридиновый краситель как пропидий моноазид, сразу же стали проводиться исследования его эффективности по сравнению с применявшимся ранее этидием моноазидом (Nocker et al., 2006). Главным отличием EMA от PMA служит то, что последний краситель не проникает в живую бактериальную клетку, поскольку имеет два положительных заряда, тогда как имеющий один положительный заряд EMA все же способен проникать через неповрежденную мембрану и затем выходить обратно, приводя к образованию некоего равновесного состояния, что схематично изображено на рис. 3. Причем это может происходить двояко – как за счет пассивной диффузии, так и с помощью клеточного насоса, обеспечивающего бактериям, в том числе, антибиотикорезистентность (Willers et al., 2017).
Рис. 3.
Схема взаимодействия этидия моноазида и пропидия моноазида с ДНК живых (а), покоящихся (б) и мертвых бактерий (в).

К дополнительному преимуществу PMA следует отнести его меньшую токсичность для микроорганизмов. Так, было показано, что у PMA нет антибактериальной активности по отношению к листерии Listeria monocytogenes (Pan, Breidt, 2007). В другой работе при определении жизнеспособности Enterobacter sakazakii с помощью виПЦР было обнаружено, что PMA и EMA предотвращали амплификацию ДНК из мертвых бактерий, но при этом проявлялся ингибирующий эффект протекания ПЦР при анализе ДНК, выделенной из живых бактерий, обработанных EMA (Cawthorn, Witthuhn, 2008).
При исследовании жизнеспособности Clostridium perfringens, Listeria monocytogenes и Salmonellae enterica было обнаружено, что ни EMA, ни PMA в полной мере не обеспечивали исключения доступности ДНК из мертвых бактерий для амплификации (Wagner et al., 2008). Есть работы, в которых показано, что обработка смешанных популяций бактерий фенантридиновыми красителями EMA и PMA приводила к увеличению доли ошибочных данных в случае использования последнего (Lee, Levin, 2009). Другими авторами отмечается, что для ингибирования виПЦР с ДНК из Legionella pneumophila требуется использование концентрации PMA, в 4 раза превышающей достаточную концентрацию EMA, исходя из чего авторы делают вывод о большей пригодности EMA (Chang et al., 2010). Видимо, и более низкая стоимость последнего реагента, о чем уже говорилось, играет немаловажную роль. Для дифференциации живых и мертвых бактерий Helicobacter pylori, напротив, показано, что PMA превосходит EMA, поскольку последний проникает и в живые клетки, блокируя амплификацию ДНК из них (Nam et al., 2011). К сходному выводу пришли и другие авторы при анализе живых и мертвых патогенных бактерий ротовой полости Streptococcus mutans, Prevotella intermedia и Aggregatibacter actinomycetemcomitans (Loozen et al., 2011). Относитель-но недавно выполнено довольно масштабное исследование (Reyneke et al., 2017), в ходе которого на пяти модельных объектах ‒ Legionella pneumophila, Pseudomonas aeruginosa, Salmonella enterica (грамотрицательные бактерии), Staphylococcus aureus, Enterococcus faecalis (грамположительные бактерии) установлены оптимальные для дифференциации живых и мертвых бактерий этих видов концентрации EMA и PMA (6 и 50 мкМ соответственно), а также оценена эффективность применения для этой цели ДНКазы. Также в этой работе было показано, что, по крайней мере, для EMA принципиальных отличий между выявлением статуса жизнеспособности у исследованных видов грамотрицательных и грамположительных микроорганизмов не наблюдается. Ранее другими авторами также сообщалось о сходном воздействии EMA на грамотрицательный вид Campylobacter jejuni и грамположительный Listeria monocytogenes (Flekna et al., 2007). Тем не менее, следует заметить, что некоторые различия в прохождении пропидия моноазида через клеточные стенки грамположительных и грамотрицательных микроорганизмов все же имеются, и для последних фирмой “Biotium” выпускается специальный усилитель для проникновения внутрь пропидия моноазида, что позволяет лучше дифференцировать живые и мертвые бактерии.
Нельзя обойти вниманием статью, в которой предложен альтернативный вариант выявления только живых бактерий с помощью ДНКазы I, разрушающей экстраклеточную ДНК и ДНК в мертвых бактериях с поврежденной клеточной стенкой, тогда как выделенная затем стандартным методом ДНК из оставшихся бактериальных клеток (живых) анализировалась обычной ПЦР (Villarreal et al., 2013). При этом для достижения правильных результатов после завершения этапа разрушения ДНК ДНКазой, ее саму до выделения ДНК предлагалось удалять обработкой протеиназой К. В качестве контроля эффективности удаления ненужной ДНК применялась виПЦР с пропидием моноазидом, показавшая сходные результаты при анализе автохтонной микрофлоры в биопленках, образующихся при производстве питьевой воды в рабочих емкостях.
Использование в ПЦР PEMAX
Выше мы уже упомянули работу (Fittipaldi et al., 2011), которая, возможно, послужила для этой группы авторов отправной точкой в совершенствовании виПЦР. В своей обзорной статье (Fittipaldi et al., 2012) они провели детальный анализ преимуществ и недостатков, используемых в виПЦР фенантридиновых красителей, что чуть позже позволило им разработать новый способ дифференциации жизнеспособности бактериальных клеток, после чего последовала серия подобных работ. Так, сначала при анализе бактерий Salmonella enteritidis как модельной системы было продемонстрировано, что сочетание 10 мкМ EMA и 50 мкМ PMA с последующей виПЦР дает более точные результаты и даже позволяет выявлять не только живые и мертвые бактерии, но и отдельно VBNC клетки (Codony et al., 2015). В Испании была образована фирма “GenIUL”, производящая необходимые реагенты и оборудование для проведения такой виПЦР. Для стандартизации проведения важной процедуры в виде воздействия светом этой фирмой производится несколько типов светодиодных устройств PAUL (Photo Activation Universal Light system), PhotoActivation System for Tubes (PhAST), Phast Blue, снимающих с экспериментаторов заботу о выборе источника света, включая необходимость охлаждения образцов из-за опасения их перегрева в случае использования галогеновых ламп. Отдельного внимания заслуживает реактив PEMAX, состав которого не раскрывается, но сообщается, что он представляет собой смесь двух красителей (вероятно EMA и PMA или их аналогов) и обеспечивает улучшенную дифференциацию живых и мертвых бактерий. С этим реагентом PEMAX недавно опубликовано несколько методических статей, посвященных установлению жизненного статуса некоторых видов бактерий, в ходе выполнения которых показано, что проблему ложно-позитивных результатов можно считать решенной (Agusti et al., 2017; Lizana et al., 2017). Так, например, комбинация двойной световой обработки бактерий Salmonella enterica, в том числе искусственно добавленных в пищевые продукты, позволила элиминировать результат амплификации в виде продукта ПЦР, который мог бы принадлежать более чем 10 млн мертвых бактерий, но PEMAX полностью исключил их ДНК как источник мишеней (Dinh Thanh et al., 2017). Причем смена реакционных пробирок и продолжение эксперимента в новых позволила снизить ложно-позитивные результаты, поскольку уже давно известно, что ДНК может сорбироваться на полипропилене (Belotserkovskii et al., 1996).
Размеры ампликонов для детекции жизнеспособных микроорганизмов
При дифференциации живых и мертвых бактерий размеры амплифицируемых фрагментов ДНК или РНК (вне зависимости от используемого подхода) весьма важны для определения жизненного статуса детектируемых бактерий. Так, еще в 1993 г. было проведено специальное исследование, показавшее, что в результате обработки различной продолжительности разными концентрациями хлора бактерии Legionella pneumophila теряли способность расти на агаре, но при этом в ряде случаев амплификация с помощью обычной ПЦР позволяла детектировать фрагмент ДНК размером 168 п.н., тогда как ампликон увеличенного размера в 650 п.н. уже не нарабатывался (McCarty, Atlas, 1993), что свидетельствовало о более сильной деградации ДНК в связи с гибелью клеток и об отсутствии в них крупных фрагментов этой молекулы. Соответственно, размеры ампликонов являются косвенным показателем жизнеспособного статуса исследуемых бактериальных клеток и поэтому неудивительно, что этому вопросу уделяется значительное внимание при проведении виПЦР, поскольку это служит дополнительным критерием живого или мертвого статуса микроорганизмов. Выше уже говорилось, что при использовании этидий моноазида для выявления живых бактерий Listeria monocytogenes амплификация более протяженных фрагментов ДНК дает лучшие результаты (Soejima et al., 2008). Позднее этой же группой авторов данный подход был применен и для других бактерий. Так, при дифференциации живых и мертвых бактерий пяти видов семейства Enterobacteriaceae с использованием этидия моноазида была закономерно обнаружена обратно пропорциональная зависимость эффективности наработки в виПЦР фрагментов ДНК, имеющих размеры 110, 340, 670, 1490 и 2840 п.н. (Soejima et al., 2011b). В другой работе проведена дискриминация мертвых и живых бактерий Enterobacter sakazakii, обработанных этидием моноазидом, для чего была использована вложенная виПЦР, в которой сначала на первом этапе амплифицировался длинный фрагмент размером 1514 п.н., а на втором для повышения чувствительности детекции – более короткий размером 560 п.н. (Soejima et al., 2012). Также крупный фрагмент ДНК размером 2451 п.н., имеющийся у различных колиформных бактерий, детектировался без выделения ДНК непосредственно в молоке, обработанном этидием моноазидом (Soejima et al., 2012). Причем в этой работе авторам пришлось использовать протеиназу К, чтобы избежать ложно-негативных результатов амплификации, поскольку известно, что молоко содержит немало ингибиторов ПЦР. Дополнительной сложностью было то, что пастеризованное молоко содержит большое количество мертвых бактерий, что также могло влиять на итоговый результат.
С использованием пропидия моноазида оценивалась эффективность виПЦР с вложенными праймерами (Contreras et al., 2011; Banihashemi et al., 2012; Schnetzinger et al., 2013; Martin et al., 2013; Ditommaso et al., 2014; Banihashemi et al., 2017). Показано, что при детекции в ветчине живых сальмонелл более достоверные результаты достигаются при амплификации фрагмента ДНК длиной 417 п.н., по сравнению с более короткими размером в 95 и 285 п.н. (Martin et al., 2013). Для оценки скорости гибели патогенных энтеробактерий Yersinia enterocolitica, Salmonea enterica, Campylobacter jejuni и Arcobacter butzleri в речной воде с помощью виПЦР с пропидием моноазидом велась детекция ампликонов еще более крупного размера: 1213, 1614, 1512 и 1415 п.н. соответственно (Banihashemi et al., 2017). Ранее этой группой авторов в модельных экспериментах было обнаружено, что для детекции живых бактерий Salmonea enterica и Campylobacter jejuni после их обработки пропидием моноазидом наилучшие результаты были получены при амплификации фрагментов ДНК размерами 1614 и 1512 п.н. соответственно, тогда как ампликоны размерами 119, 174 и даже 899 п.н. не обеспечивали должный уровень дискриминации (Banihashemi et al., 2012).
Способы дифференциации живых и мертвых бактерий путем изотермической амплификации фрагментов ДНК или РНК, основанные на применении фенантридиновых красителей
Помимо ПЦР для дифференциации живых и мертвых микроорганизмов используются и другие методы амплификации нуклеиновых кислот, протекающие, в частности, в изотермических условиях, обеспечивающих определенные преимущества. Так, в 2009 г. был предложен метод EMA-LAMP (Ethidium MonoAzide ‒ Loop mediated isothermal AMPlification), позволивший дифференцировать мертвые и живые клетки сальмонелл Salmonella enterica (Lu et al., 2009). Позже вместо EMA в такой LAMP амплификации ДНК при детекции как сальмонелл, так и других бактерий (E. coli, Vibrio parahaemolyticus, Staphylococcus aureus) стали использовать более подходящий PMA (Chen et al., 2011; Youn et al., 2017; Li et al., 2017; Yan et al., 2017; Fang et al., 2018). Однако при разработке метода Rti-LAMP на основе EMA с детекцией результатов амплификации в реальном времени было дополнительно сообщено, что использование PMA для дифференциации мертвых и живых бактерий Salmonella enterica оказалось менее эффективным (Wu et al., 2015). Предложены после исключения из реакции ДНК мертвых бактерий с помощью PMA и другие вариации и способы детекции результатов LAMP амплификации ДНК живых бактерий в виде микрофлуидики, или путем ее объединения с биосенсорными технологиями, либо с латеральной хроматографией (Ahmad et al., 2017; Zhao et al., 2017; Zhang et al., 2017). Еще один метод амплификации нуклеиновых кислот в виде рекомбиназной полимеразной амплификации (Recombinase Polymerase Amplification) в чиповом варианте с использованием PMA позволил за короткое время (около одного часа) дискриминировать живые и мертвые бактерии Legionella ssp., вызывающие такое опасное заболевание как легионеллез, золотым стандартом выявления которых служит культуральный метод, требующий, однако, не менее 10 сут, при том, что живые, но некультивируемые бактерии им не выявляются (Kober et al., 2018). В другой работе на дифферециацию живых и мертвых стрептококков этим методом оказалось достаточно всего 20 мин (Chen et al., 2018).
ЗАКЛЮЧЕНИЕ
Актуальность обнаружения жизнеспособных бактерий в различных средах, включая продукты питания, питьевую воду, образцы выделений и тканей человека и животных, объектов внешней среды (вода, почва, воздух), особенно в изолированных помещениях типа космических станций и пр., не вызывает сомнений. Важной является информация не только о наличии микроорганизмов или их количестве, но и их способность размножаться и, соответственно, представлять собой угрозу здоровью другим биологическим объектам. Дифференциация живых и мертвых бактерий путем амплификации нуклеиновых кислот насчитывает уже почти три десятилетия, и за это время отдельные методы, представленные в табл. 1, получили заметное развитие, другие оказались менее эффективными.
Таблица 1.
Способы дифференциации живых и мертвых бактерий с помощью разных методов амплификации нуклеиновых кислот (ДНК или РНК) вкупе с дополнительными ингредиентами (в хронологическом порядке)
| Год | Метод | Мишени/особенности | Ссылка |
|---|---|---|---|
| 1988 | ПЦР | Впервые детектировано наличие бактерий в исследуемом образце с помощью ПЦР без дифференциации на мертвых и живых | Oliver et al., 1988 |
| 1991 | ОТ-ПЦР | Первое использование в качестве мишени мРНК при дифференциации живых и мертвых бактерий | Bej et al., 1991 |
| 1993 | ПЦР | Впервые при дифференциации живых и мертвых бактерий детектировались ампликоны разных размеров | Jamil et al., 1993 |
| 1994 | NASBA | Изотермическая амплификация РНК живых и мертвых бактерий, не вовлекающая ДНК | van der Vliet et al., 1994 |
| 1996 | ОТ-ПЦР | Первое использование в качестве мишени пограничных областей пре-рРНК и рРНК | Candelosi et al., 1996 |
| 1999 | RT-SDA | Изотермическая амплификация РНК (кДНК), требующая удаления ДНК | Hellyer et al., 1999 |
| 2003 | ПЦР | Впервые предложена ПЦР с фенантридиновым красителем (EMA), позволяющая относительно уверенно дифференцировать живые и мертвые бактерии | Nogva et al., 2003 |
| 2006 | ПЦР | Предложен более специфичный фенантридиновый краситель (PMA), позволяющий лучше дифференцировать живые и мертвые бактерии | Nocker et al., 2006 |
| 2008 | ПЦР | Предложено использовать смесь двух фенантридиновых красителей (EMA и PMA), что позволило более достоверно дифференцировать живые и мертвые бактерии | Cawthorn, Witthuhn, 2008 |
| 2009 | EMA-LAMP | Разработана высокочувствительная изотермическая амплификация с фенантридиновым красителем EMA | Lu et al., 2009 |
| 2010 | ПЦР (MVA) | Новое обращение к мишени в виде пограничных областей пре-рРНК и рРНК | Candelosi et al., 2010 |
| 2011 | PMA-LAMP | Разработана высокочувствительная изотермическая амплификация с фенантридиновым красителем PMA | Chen et al., 2011 |
| 2016 | ПЦР | Дифференциация с помощью ПЦР живых и мертвых бактерий на основе ряда соединений платины и палладия | Soejima et al., 2016; Soejima, Iwatsuki, 2016 |
| 2018 | RPA | Предложена для дифференциации живых и мертвых бактерий быстрая и высокочувствительная изотермическая амплификация с фенантридиновым красителем PMA | Kober et al., 2018 |
| 2018 | ПЦР | Дифференциация с помощью ПЦР живых и мертвых бактерий с применением нового перспективного красителя DyeTox13 Green C‑2 Azide | Lee, Bae, 2018b |
Наиболее активно ведется детекция жизнеспособных бактерий с использованием фенантридиновых красителей, благодаря чему достигнут существенный прогресс, что, однако, не исключает необходимости дальнейшей адаптации данного подхода к конкретным видам/родам бактерий. При этом технические стороны использования моноазидов этидия и пропидия для дифференциации живых и мертвых бактерий, включая разброс концентраций этих соединений, временные интервалы и температурные режимы обработок, которые для разных видов бактерий сильно отличаются, в данном обзоре оставлены практически без внимания, но при необходимости с ними можно ознакомиться в оригинальных статьях, представленных в списке цитированной литературы. Весьма перспективной представляется также детекция живых микроорганизмов путем амплификации с помощью ОТ-ПЦР интактной пре-рРНК, не сохраняющейся в бактериальных клетках, потерявших жизнеспособность. Ведется поиск/синтез других новых соединений, способных дифференцировать мертвые и живые бактерии, в том числе с использованием амплификации их нуклеиновых кислот. Примером может служить недавняя работа (Lee, Bae, 2018b) описывающая успешное использование для установления жизненного статуса как грамотрицательных (Pseudomonas aeroginosa), так и грамположительных бактерий (Enterococcus faecalis) производного известного красителя SYTO 13, получившего название DyeTox13 Green C‑2 Azide, показавшее его преимущество над моноазидами этидия и пропидия. Более того, предложено вместо фенантридиновых красителей использовать с той же целью некоторые соединения платины, которые также способны проникать только через поврежденные мембраны неживых бактерий, образуя с пуриновыми основаниями находящейся внутри таких клеток ДНК хелатные комплексы, препятствуя в дальнейшем амплификации (Soejima et al., 2016a). Причем авторы отмечают, что весь процесс осуществляется на свету без опасения преждевременного вступления в реакцию используемых ими соединений платины, которые к тому же в сотни раз дешевле фенантридиновых красителей. Дальнейшее удешевление процесса дифференциации живых и мертвых бактерий продемонстрировано той же группой авторов, использовавшими целый ряд соединений палладия, также проникающих только через поврежденные мембраны бактериальных клеток и позволивших провести анализ пастеризованного молока со множеством убитых микроорганизмов (Soejima, Iwatsuki, 2016).
Для более точного выяснения статуса жизнеспособности бактерий в различных средах представляется оптимальным исходное деление образца на три части (аликвоты) и проведения, соответственно, трех исследований, в ходе которых в первом случае будет амплифицироваться ДНК всех бактерий, во втором – только жизнеспособных и имеющих сохранившуюся клеточную стенку и/или цитоплазматическую мембрану, через которую не будут проникнуть моноазидные производные фенантридиновых красителей (или их аналоги), а в третьем эксперименте необходимо проводить подходящим методом высокочувствительную амплификацию пре-рРНК без ДНКазной обработки в изотермических условиях. Это не допустит, с одной стороны потенциального разрушения молекул РНК, а с другой – исключит амплификацию ДНК, обеспечив наработку ампликонов исключительно с РНК матриц. Важным моментом такого тройного исследования является еще и то, что бактерии, подвергнутые воздействию ультрафиолетового света, сохранив оболочки, непроницаемые для моноазида пропидия, утратят свою жизнеспособность, но информация об этом не будет получена. Несмотря на необходимость проведения трех независимых реакций, такая трата различных ресурсов может быть оправдана в целом ряде случаев дифференциации живых и мертвых бактерий.
Список литературы
Чемерис А.В., Магданов Э.Г., Гарафутдинов Р.Р., Вахитов В.А. Как исключить появление ложно-позитивных результатов при проведении полимеразной цепной реакции // Вестник биотехнологии и физико-химической биологии им. Ю.А. Овчинникова. 2012. Т. 8. № 3. С. 34‒45.
Чемерис Д.А., Магданов Э.Г., Машков О.И., Гарафтдинов Р.Р., Чемерис А.В. ПЦР с отложенным (горячим или задержанным) стартом // Биомика. 2011. Т. 2. № 1. С. 1‒8.
Чемерис А.В., Чемерис Д.А., Магданов Э.Г., Гарафтдинов Р.Р., Нагаев Н.Р., Вахитов В.А. Причины ложно-негативной ПЦР и недопущение некоторых из них // Биомика. 2012б. Т. 4. № 1. С. 31‒47.
Agustí G., Fittipaldi M., Codony F. False-positive viability PCR results: An association with microtubes // Curr. Microbiol. 2017. V. 74. P. 377‒380. https://doi.org/10.1007/s00284-016-1189-3
Ahmad F., Stedtfeld R. D., Waseem H., Williams M.R., Cupples A.M., Tiedje J.M., Hashsham S.A. Most probable number ‒ loop mediated isothermal amplification (MPN-LAMP) for quantifying waterborne pathogens in less than 25 minutes // J. Microbiol. Methods. 2017. V. 132. P. 27–33. https://doi.org/10.1016/j.mimet.2016.11.010
Bae S., Wuertz S. Discrimination of viable and dead fecal Bacteroidales bacteria by quantitative PCR with propidium monoazide // Appl. Environ. Microbiol. 2009. V. 75. P. 2940‒2944. https://doi.org/10.1128/AEM.01333-08
Banihashemi A., Van Dyke M., Huck P.M. Long-amplicon propidium monoazide-PCR enumeration assay to detect viable Campylobacter and Salmonella // J. Appl. Microbiol. 2012. V. 113. P. 863‒873. https://doi.org/10.1111/j.1365-2672.2012.05382.x
Banihashemi A., Van Dyke M.I., Huck P.M. Application of long amplicon propidium monoazide-PCR to assess the effects of temperature and background microbiota on pathogens in river water // J. Water Health. 2017. V. 15. P. 418‒428. https://doi.org/10.2166/wh.2017.161
Barer M.R., Harwood C.R. Bacterial viability and culturability // Adv. Microb. Physiol. 1999. V. 41. P. 93‒137. https://doi.org/10.1016/S0065-2911(08)60166-6
Bej A.K., Mahbubani M.H., Atlas R.M. Detection of viable Legionella pneumophila in water by polymerase chain reaction and gene probe methods // Appl. Environ. Microbiol. 1991. V. 57. P. 597–600.
Bej A.K., Ng W.Y., Morgan S., Jones D.D., Mahbubani M.H. Detection of viable Vibrio cholerae by reverse-transcriptase polymerase chain reaction (RT-PCR) // Mol. Biotechnol. 1996. V. 5. P. 1‒10. https://doi.org/10.1007/BF02762407
Belotserkovskii B.P., Johnston B.H. Polypropylene tube surfaces may induce denaturation and multimerization of DNA // Science. 1996. V. 271. № 5246. P. 222‒223. https://doi.org/10.1126/science.271.5246.222
Bernet C., Garret M., de Barbeyrac B., Bebear C., Bonnet J. Detection of Mycoplasma pneumoniae by using the polymerase chain reaction // J. Clin. Microbiol. 1989. V. 27. P. 2492‒2496.
Birch L., Dawson C.E., Cornett J.H., Keer J.T. A comparison of nucleic acid amplification techniques for the assessment of bacterial viability // Lett. Appl. Microbiol. 2001. V. 33. P. 296‒301. https://doi.org/10.1046/j.1472-765X.2001.00999.x
Cangelosi G.A., Brabant W.H., Britschgi T.B., Wallis C.K. Detection of rifampin- and ciprofloxacin-resistant Mycobacterium tuberculosis by using species-specific assays for precursor rRNA // Antimicrob. Agents. Chemother. 1996. V. 40. P. 1790‒1795. https://doi.org/10.1128/AAC.40.8.1790
Cangelosi G.A., Meschke J.S. Dead or alive: molecular assessment of microbial viability // Appl. Environ. Microbiol. 2014. V. 80. P. 5884‒5891. https://doi.org/10.1128/AEM.01763-14
Cangelosi G.A., Weigel K.M., Lefthand-Begay C., Meschke J.S. Molecular detection of viable bacterial pathogens in water by ratiometric pre-rRNA analysis // Appl. Environ. Microbiol. 2010. V. 76. P. 960‒962. https://doi.org/10.1128/AEM.01810-09
Cawthorn D.M., Witthuhn R.C. Selective PCR detection of viable Enterobacter sakazakii cells utilizing propidium monoazide or ethidium bromide monoazide // J. Appl. Microbiol. 2008. V. 105. P. 1178‒1185. https://doi.org/10.1111/j.1365-2672.2008.03851.x
Chang B., Taguri T., Sugiyama K., Amemura-Maekawa J., Kura F., Watanabe H. Comparison of ethidium monoazide and propidium monoazide for the selective detection of viable Legionella cells // Jpn. J. Infect. Dis. 2010. V. 63. P. 119‒123. https://doi.org/10.4018/978-1-60566-840-6.ch008
Chen J., Wang Y., Liu X., Chen G., Chen X., Chen J., Liu Z., Gong J., Yang G., Lan Q. Development of propidium monoazide-recombinase polymerase amplification (PMA-RPA) assay for rapid detection of Streptococcus pyogenes and Streptococcus agalactiae // Mol. Cell. Probes. 2018. V. 41. P. 32‒38. https://doi.org/10.1016/j.mcp.2018.08.007
Chen S., Wang F., Beaulieu J.C., Stein R.E., Ge B. Rapid detection of viable salmonellae in produce by coupling propidium monoazide with Loop-Mediated Isothermal Amplification // Appl. Environ. Microbiol. 2011. V. 77. P. 4008–4016. https://doi.org/10.1128/AEM.00354-11
Codony F., Agustí G., Allué-Guardia A. Cell membrane integrity and distinguishing between metabolically active and inactive cells as a means of improving viability PCR // Mol. Cell. Probes. 2015. V. 29. P. 190‒192. https://doi.org/10.1016/j.mcp.2015.03.003
Contreras P.J., Urrutia H., Sossa K., Nocker A. Effect of PCR amplicon length on suppressing signals from membrane-compromised cells by propidium monoazide treatment // J. Microbiol. Methods. 2011. V. 7. P. 89‒95. https://doi.org/10.1016/j.mimet.2011.07.016
Davey H.M. Life, death, and in-between: meanings and methods in microbiology // Appl. Environ. Microbiol. 2011. V. 77. P. 5571‒5576. https://doi.org/10.1128/AEM.00744-11
Dinh Thanh M., Agustí G., Mader A., Appel B., Codony F. Improved sample treatment protocol for accurate detection of live Salmonella spp. in food samples by viability PCR // PLoS One. 2017. V. 12. e0189302. https://doi.org/10.1371/journal.pone.0189302
Ditommaso S., Ricciardi E., Giacomuzzi M., Arauco Rivera S.R., Ceccarelli A., Zotti C.M. Overestimation of the Legionella spp. load in environmental samples by quantitative real-time PCR: pretreatment with propidium monoazide as a tool for the assessment of an association between Legionella concentration and sanitary risk // Diagn. Microbiol. Infect. Dis. 2014. V. 80. P. 260‒266. https://doi.org/10.1016/j.diagmicrobio.2014.09.010
Dong L., Liu H., Meng L., Xing M., Wang J., Wang C., Chen H., Zheng N. Quantitative PCR coupled with sodium dodecyl sulfate and propidium monoazide for detection of viable Staphylococcus aureus in milk // J. Dairy Sci. 2018. V. 101. P. 4936‒4943. https://doi.org/10.3168/jds.2017-14087
Dutilh B. Bébéar C. Rodriguez P., Vekris A., Bonnet J., Garret M. Specific amplification of a DNA sequence common to all Chlamydia trachomatis serovars using the polymerase chain reaction // Res. Microbiol. 1989. V. 140. P. 7‒16. https://doi.org/10.1016/0923-2508(89)90053-3
Fang J., Wu Y., Qu D., Ma B., Yu X., Zhang M., Han J. Propidium monoazide real-time loop-mediated isothermal amplification for specific visualization of viable Salmonella in food // Lett. Appl. Microbiol. 2018. V. 67. P. 79‒88. https://doi.org/10.1111/lam.12992
Fittipaldi M, Nocker A, Codony F. Progress in understanding preferential detection of live cells using viability dyes in combination with DNA amplification // J. Microbiol. Methods. 2012. V. 91. P. 276‒289. https://doi.org/10.1016/j.mimet.2012.08.007
Fittipaldi M., Codony F., Adrados B., Camper A.K., Morató J. Viable real-time PCR in environmental samples: can all data be interpreted directly? // Microb. Ecol. 2011. V. 61. P. 7‒12. https://doi.org/10.1007/s00248-010-9719-1
Flekna G., Stefanic P., Wagner M., Smulders F.J., Mozina S.S., Hein I. Insufficient differentiation of live and dead Campylobacter jejuni and Listeria monocytogenes cells by ethidium monoazide (EMA) compromises EMA/real-time PCR // Res. Microbiol. 2007. V. 158. P. 405‒412. https://doi.org/10.1016/j.resmic.2007.02.008
Fukuzawa S., Shiho H., Fujita T. Selective detection of DNA from viable Mycobacterium tuberculosis complex strains using the EMA-PCR method // Jpn. J. Infect. Dis. 2019. V. 72. P. 19‒22. https://doi.org/10.7883/yoken.JJID.2018.111
Fykse E.M., Skogan G., Davies W., Olsen J.S., Blatny J.M. Detection of Vibrio cholerae by real-time nucleic acid sequence-based amplification // Appl. Environ. Microbiol. 2007. V. 73. P. 1457‒1466. https://doi.org/10.1128/AEM.01635-06
Gobert G., Cotillard A., Fourmestraux C., Pruvost L., Miguet J., Boyer M. Droplet digital PCR improves absolute quantification of viable lactic acid bacteria in faecal samples // J. Microbiol. Methods. 2018. V. 148. P. 64‒73. https://doi.org/10.1016/j.mimet.2018.03.004
Graves D.E., Watkins C.L., Yielding L.W. Ethidium bromide and its photoreactive analogues: spectroscopic analysis of deoxyribonucleic acid binding properties // Biochemistry. 1981. V. 20. P. 1887‒1892. https://doi.org/10.1021/bi00510a026
Hartskeerl R.A., de Wit M.Y., Klatser P.R. Polymerase chain reaction for the detection of Mycobacterium leprae // J. Gen. Microbiol. 1989. V. 135. P. 2357‒2364. https://doi.org/10.1099/00221287-135-9-2357
Hein I., Flekna G., Wagner M. Possible errors in the interpretation of ethidium bromide and PicoGreen DNA staining results from ethidium monoazide-treated DNA // Appl. Environ. Microbiol. 2006. V. 72. P. 6860‒6861. https://doi.org/10.1128/AEM.01243-06
Hellyer T.J., DesJardin L.E., Teixeira L., Perkins M.D., Cave M.D., Eisenach K.D. Detection of viable Mycobacterium tuberculosis by reverse transcriptase-strand displacement amplification of mRNA // J. Clin. Microbiol. 1999. V. 37. P. 518‒523.
Huang Z., Zheng J., Shi C., Chen Q. Flow cytometry-based method facilitates optimization of PMA treatment condition for PMA-qPCR method // Mol. Cell. Probes. 2018. V. 40. P. 37‒39. https://doi.org/10.1016/j.mcp.2018.05.002
Inoue D., Tsutsui H., Yamazaki Y., Sei K., Soda S., Fujita M., Ike M. Application of real-time polymerase chain reaction (PCR) coupled with ethidium monoazide treatment for selective quantification of viable bacteria in aquatic environment // Water Sci. Technol. 2008. V. 58. P. 1107‒1112. https://doi.org/10.2166/wst.2008.474
Jamil S., Keer J.T., Lucas S.B., Dockrell H.M., Chiang T.J., Hussain R., Stoker N.G. Use of polymerase chain reaction to assess efficacy of leprosy chemotherapy // Lancet. 1993. V. 342. № 8866. P. 264‒268. https://doi.org/10.1016/0140-6736(93)91816-5
Janssen K.J., Hoebe C.J., Dukers-Muijrers N.H., Eppings L., Lucchesi M., Wolffs P.F. Viability-PCR shows that NAAT detects a high proportion of DNA from non-viable Chlamydia trachomatis // PLoS One. 2016. V. 11. e0165920. https://doi.org/10.1371/journal.pone.0165920
Kaushik R., Balasubramanian R. Discrimination of viable from non-viable gram-negative bacterial pathogens in airborne particles using propidium monoazide-assisted qPCR // Sci. Total Environ. 2013. V. 449. P. 237‒243. https://doi.org/10.1016/j.scitotenv.2013.01.065
Kell D.B., Kaprelyants A.S., Weichart D.H., Harwood C.R., Barer M.R. Viability and activity in readily culturable bacteria: a review and discussion of the practical issues // An-tonie van Leeuwenhoek. 1998. V. 73. P. 169‒187. https://doi.org/10.1023/A:1000664013047
Kibbee R.J., Örmeci B. Development of a sensitive and false-positive free PMA-qPCR viability assay to quantify VBNC Escherichia coli and evaluate disinfection performance in wastewater effluent // J. Microbiol. Methods. 2017. V. 132. P. 139‒147. https://doi.org/10.1016/j.mimet.2016.12.004
Klein P.G., Juneja V.K. Sensitive detection of viable Listeria monocytogenes by reverse transcription-PCR // Appl. Environ. Microbiol. 1997. V. 63. P. 4441‒4448.
Knowles J.R. Photogenerated reagents for biological receptor-site labeling // Acc. Chem. Res. 1972. V. 5. P. 155–160. https://doi.org/10.1021/ar50052a006
Kobayashi H., Oethinger M., Tuohy M.J., Procop G.W., Hall G.S., Bauer T.W. Limiting false-positive polymerase chain reaction results: detection of DNA and mRNA to differentiate viable from dead bacteria // Diagn. Microbiol. Infect. Dis. 2009. V. 64. № 4. P. 445‒447. https://doi.org/10.1016/j.diagmicrobio.2009.04.004
Kober C., Niessner R., Seidel M. Quantification of viable and non-viable Legionella spp. by heterogeneous asymmetric recombinase polymerase amplification (haRPA) on a flow-based chemiluminescence microarray // Biosens. Bioelectron. 2018. V. 100. P. 49‒55. https://doi.org/10.1016/j.bios.2017.08.053
Kontchou J.A., Nocker A. Optimization of viability qPCR for selective detection of membrane-intact Legionella pneumophila // J. Microbiol. Methods. 2019. V. 156. P. 68‒76. https://doi.org/10.1016/j.mimet.2018.12.003
Lee E.S., Lee M.H., Kim B.S. Evaluation of propidium monoazide-quantitative PCR to detect viable Mycobacterium fortuitum after chlorine, ozone, and ultraviolet disinfection // Int. J. Food Microbiol. 2015. V. 210. P. 143‒148. https://doi.org/10.1016/j.ijfoodmicro.2015.06.019
Lee J.L., Levin R.E. A comparative study of the ability of EMA and PMA to distinguish viable from heat killed mixed bacterial flora from fish fillets // J. Microbiol. Methods. 2009. V. 76. P. 93‒96. https://doi.org/10.1016/j.mimet.2008.08.008
Lee S., Bae S. Molecular viability testing of viable but non-culturable bacteria induced by antibiotic exposure // Microb. Biotechnol. 2018a. V. 11. P. 1008‒1016. https://doi.org/10.1111/1751-7915.13039
Lee S., Bae S. Evaluating the newly developed dye, DyeTox13 Green C-2 Azide, and comparing it with existing EMA and PMA for the differentiation of viable and nonviable bacteria // J. Microbiol Methods. 2018b. V. 148. P. 33‒39. https://doi.org/10.1016/j.mimet.2018.03.018
Li H., Xin H., Li S.F. Multiplex PMA-qPCR assay with internal amplification control for simultaneous detection of viable Legionella pneumophila, Salmonella typhimurium, and Staphylococcus aureus in environmental waters // Environ. Sci. Technol. 2015. V. 49. P. 14249‒14256. https://doi.org/10.1021/acs.est.5b03583
Li Y., Yang L., Fu J., Yan M., Chen D., Zhang L. The novel loop-mediated isothermal amplification based confirmation methodology on the bacteria in Viable but Non-Culturable (VBNC) state // Microb. Pathog. 2017. V. 111. P. 280‒284. https://doi.org/10.1016/j.micpath.2017.09.007
Lizana X., López A., Benito S., Agustí G., Ríos M., Piqué N., Marqués A.M., Codony F. Viability qPCR, a new tool for Legionella risk management // Int. J. Hyg. Environ. Health. 2017. V. 220. P. 1318‒1324. https://doi.org/10.1016/j.ijheh.2017.08.007
Lleò M.M., Pierobon S., Tafi M.C., Signoretto C., Canepari P. mRNA detection by reverse transcription-PCR for monitoring viability over time in an Enterococcus faecalis viable but nonculturable population maintained in a laboratory microcosm // Appl. Environ. Microbiol. 2000. V. 66. P. 4564‒4567. https://doi.org/10.1128/AEM.66.10.4564-4567.2000
Loozen G., Boon N., Pauwels M., Quirynen M., Teughels W. Live/dead real-time polymerase chain reaction to assess new therapies against dental plaque-related pathologies // Mol. Oral. Microbiol. 2011. V. 26. P. 253‒261. https://doi.org/10.1111/j.2041-1014.2011.00615.x
Lovdal T., Hovda M.B., Björkblom B., Møller S.G. Propidium monoazide combined with real-time quantitative PCR underestimates heat-killed Listeria innocua // J. Microbiol. Methods. 2011. V. 85. P. 164‒169. https://doi.org/10.1016/j.mimet.2011.01.027
Lu Y., Yang W., Shi L. et al. Specific detection of viable Salmonella cells by an ethidium monoazide-Loop Mediated Isothermal Amplification (EMA-LAMP) method // J. Health Sci. 2009. V. 55. P. 820‒824. https://doi.org/10.1248/jhs.55.820
Martin B., Raurich S., Garriga M., Aymerich T. Effect of amplicon length in propidium monoazide quantitative PCR for the enumeration of viable cells of Salmonella in cooked ham // Food Anal. Methods. 2013. V. 6. P. 683–690. https://doi.org/10.1007/s12161-012-9460-0
McCarty S.C., Atlas R.M. Effect of amplicon size on PCR detection of bacteria exposed to chlorine // PCR Methods Appl. 1993. V. 3. P. 181‒185. https://doi.org/10.1101/gr.3.3.181
McKillip J.L., Jaykus L.A., Drake M. rRNA stability in heat-killed and UV-irradiated enterotoxigenic Staphylococcus aureus and Escherichia coli O157:H7 // Appl. Environ. Microbiol. 1998. V. 64. P. 4264‒4268.
Minami J., Yoshida K., Soejima T., Yaeshima T., Iwatsuki K. New approach to use ethidium bromide monoazide as an analytical tool // J. Appl. Microbiol. 2010. V. 109. P. 900‒909. https://doi.org/10.1111/j.1365-2672.2010.04716.x
Nam S., Kwon S., Kim M.J., Chae J.C., Jae Maeng P., Park J.G., Lee G.C. Selective detection of viable Helicobacter pylori using ethidium monoazide or propidium monoazide in combination with real-time polymerase chain reaction // Microbiol. Immunol. 2011. V. 55. P. 841‒846. https://doi.org/10.1111/j.1348-0421.2011.00388.x
Nebe-von Caron G., Stephens P., Badley R.A. Assessment of bacterial viability status by flow cytometry and single cell sorting // J. Appl. Microbiol. 1998. V. 84. P. 988‒998. https://doi.org/10.1046/j.1365-2672.1998.00436.x
Nikolayevskyy V., Miotto P., Pimkina E., Balabanova Y., Kontsevaya I., Ignatyeva O., Ambrosi A., Skenders G., Ambrozaitis A., Kovalyov A., Sadykhova A., Simak T., Kritsky A., Mironova S., Tikhonova O., Dubrovskaya Y., Rodionova Y., Cirillo D., Drobniewski F. Utility of propidium monoazide viability assay as a biomarker for a tuberculosis disease // Tuberculosis (Edinb.) 2015. V. 95. P. 179‒185. https://doi.org/10.1016/j.tube.2014.11.005
Nocker A., Camper A.K. Selective removal of DNA from dead cells of mixed bacterial communities by use of ethidium monoazide // Appl. Environ. Microbiol. 2006. V. 72. P. 1997‒2004. https://doi.org/10.1128/AEM.72.3.1997-2004.2006
Nocker A., Cheung C.Y., Camper A.K. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells // J. Microbiol. Methods. 2006. V. 67. P. 310‒320. https://doi.org/10.1016/j.mimet.2006.04.015
Nocker A., Mazza A., Masson L., Camper A.K., Brousseau R. Selective detection of live bacteria combining propidium monoazide sample treatment with microarray technology // J. Microbiol. Methods. 2009. V. 76. P. 253‒261. https://doi.org/10.1016/j.mimet.2008.11.004
Nocker A., Richter-Heitmann T., Montijn R. et al. Discrimination between live and dead cells in bacterial communities from environmental water samples analyzed by 454 pyrosequencing // Int. Microbiol. 2010. V. 13. P. 59‒65. https://doi.org/10.2436/20.1501.01.111
Nocker A., Sossa K.E., Camper A.K. Molecular monitoring of disinfection efficacy using propidium monoazide in combination with quantitative PCR // J. Microbiol. Methods. 2007. V. 70. P. 252‒260. https://doi.org/10.1016/j.mimet.2007.04.014
Nogva H.K., Drømtorp S.M., Nissen H., Rudi K. Ethidium monoazide for DNA-based differentiation of viable and dead bacteria by 5'-nuclease PCR // Biotechniques. 2003. V. 34. P. 804‒813. https://doi.org/10.2144/03344rr02
Olive D.M. Detection of enterotoxigenic Escherichia coli after polymerase chain reaction amplification with a thermostable DNA polymerase // J. Clin. Microbiol. 1989. V. 27. № 2. P. 261‒265. https://doi.org/10.1016/S0934-8840(11)80610-9
Olive D.M., Atta A.I., Setti S.K. Detection of toxigenic Escherichia coli using biotin-labelled DNA probes following enzymatic amplification of the heat labile toxin gene // Mol. Cell. Probes. 1988. V. 2. P. 47‒57. https://doi.org/10.1016/0890-8508(88)90043-6
Oliver J.D. Recent findings on the viable but nonculturable state in pathogenic bacteria // FEMS Microbiol. Rev. 2010. V. 34. P. 415‒425. https://doi.org/10.1111/j.1574-6976.2009.00200.x
Pan Y., Breidt F., Jr. Enumeration of viable Listeria monocytogenes cells by real-time PCR with propidium monoazide and ethidium monoazide in the presence of dead cells // Appl. Environ. Microbiol. 2007. V. 73. P. 8028‒8031. https://doi.org/10.1128/AEM.01198-07
Pinto D., Santos M.A., Chambel L. Thirty years of viable but nonculturable state research: unsolved molecular mechanisms // Crit. Rev. Microbiol. 2015. V. 41. P. 61‒76. https://doi.org/10.3109/1040841X.2013.794127
Pisz J.M., Lawrence J.R., Schafer A.N., Siciliano S.D. Differentiation of genes extracted from non-viable versus viable microorganisms in environmental samples using ethidium monoazide bromide // J. Microbiol. Methods. 2007. V. 71. P. 312‒318. https://doi.org/10.1016/j.mimet.2007.09.015
Rawsthorne H., Dock C.N., Jaykus L.A. PCR-based method using propidium monoazide to distinguish viable from nonviable Bacillus subtilis spores // Appl. Environ. Microbiol. 2009. V. 75. № 9. P. 2936‒2939. https://doi.org/10.1128/AEM.02524-08
Reyneke B., Ndlovu T., Khan S., Khan W. Comparison of EMA-, PMA- and DNase qPCR for the determination of microbial cell viability // Appl. Microbiol. Biotechnol. 2017. V. 101. № 19. P. 7371‒7383. https://doi.org/10.1007/s00253-017-8471-6
Riedy M.C., Muirhead K.A., Jensen C.P., Stewart C.C. Use of a photolabeling technique to identify nonviable cells in fixed homologous or heterologous cell populations // Cytometry. 1991. V. 12. P. 133‒139. https://doi.org/10.1002/cyto.990120206
Rosa P.A., Schwan T.G. A specific and sensitive assay for the Lyme disease spirochete Borrelia burgdorferi using the polymerase chain reaction // J. Infect. Dis. 1989. V. 160. 6. P. 1018‒1029. https://doi.org/10.1093/infdis/160.6.1018
Roussel C., Galia W., Leriche F., Chalancon S., Denis S., Van de Wiele T., Blanquet-Diot S. Comparison of conventional plating, PMA-qPCR, and flow cytometry for the determination of viable enterotoxigenic Escherichia coli along a gastrointestinal in vitro model // Appl. Microbiol. Biotechnol. 2018. V. 102. P. 9793‒9802. https://doi.org/10.1007/s00253-018-9380-z
Rudi K., Moen B., Drømtorp S.M., Holck A.L. Polymerase chain reaction for the detection of Mycobacterium leprae // Appl. Environ. Microbiol. 2005. V. 71. P. 1018–1024. https://doi.org/10.1128/AEM.71.2.1018-1024.2005
Schnetzinger F., Pan Y., Nocker A. Use of propidium monoazide and increased amplicon length reduce false-positive signals in quantitative PCR for bioburden analysis // Appl. Microbiol. Biotechnol. 2013. V. 97. P. 2153‒2162. https://doi.org/10.1007/s00253-013-4711-6
Seidel L., Strathmann M., Nocker A. The feasibility of improved live-dead distinction in qPCR-based microbial source tracking // J. Microbiol. Methods. 2017. V. 140. P. 23‒31. https://doi.org/10.1016/j.mimet.2017.06.013
Sheridan G.E., Masters C.I., Shallcross J.A., MacKey B.M. Detection of mRNA by reverse transcription-PCR as an indicator of viability in Escherichia coli cells // Appl. Environ. Microbiol. 1998. V. 64. P. 1313‒1318.
Simpkins S.A., Chan A.B., Hays J., Pöpping B., Cook N. An RNA transcription-based amplification technique (NASBA) for the detection of viable Salmonella enterica // Lett. Appl. Microbiol. 2000. V. 30. P. 75‒79. https://doi.org/10.1046/j.1472-765x.2000.00670.x
Slimani S., Robyns A., Jarraud S., Molmeret M., Dusserre E., Mazure C., Facon J.P, Lina G., Etienne J., Ginevra C. Evaluation of propidium monoazide (PMA) treatment directly on membrane filter for the enumeration of viable but non cultivable Legionella by qPCR // J. Microbiol. Methods. 2012. V. 88. P. 319‒321. https://doi.org/10.1016/j.mimet.2011.12.010
Soejima T., Iida K., Qin T., Taniai H., Seki M., Yoshida S. Method to detect only live bacteria during PCR amplification // J. Clin. Microbiol. 2008. V. 46. P. 2305‒2313. https://doi.org/10.1128/JCM.02171-07
Soejima T., Iida K., Qin T., Taniai H., Seki M., Takade A., Yoshida S. Photoactivated ethidium monoazide directly cleaves bacterial DNA and is applied to PCR for discrimination of live and dead bacteria // Microbiol. Immunol. 2007. V. 51. P. 763‒775. https://doi.org/10.1111/j.1348-0421.2007.tb03966.x
Soejima T., Iwatsuki K.J. Innovative use of palladium compounds to selectively detect live Enterobacteriaceae cells in milk by polymerase chain reaction // Appl. Environ. Microbiol. 2016. V. 82. P. 6930‒6941. https://doi.org/10.1128/AEM.01613-16
Soejima T., Minami J., Yaeshima T., Iwatsuki K. An advanced PCR method for the specific detection of viable total coliform bacteria in pasteurized milk // Appl. Microbiol. Biotechnol. 2012. V. 95. P. 485‒497. https://doi.org/10.1007/s00253-012-4086-0
Soejima T., Minami J., Xiao J.Z., Abe F. Innovative use of platinum compounds to selectively detect live microorganisms by polymerase chain reaction // Biotechnol. Bioeng. 2016a. V. 113. P. 301‒310. https://doi.org/10.1002/bit.25711
Soejima T., Schlitt-Dittrich F., Yoshida S. Rapid detection of viable bacteria by nested polymerase chain reaction via long DNA amplification after ethidium monoazide treatment // Anal. Biochem. 2011a. V. 418. P. 286‒289. https://doi.org/10.1016/j.ab.2011.06.033
Soejima T., Schlitt-Dittrich F., Yoshida S. Polymerase chain reaction amplification length-dependent ethidium // Anal. Biochem. 2011b. V. 418. P. 37‒43. https://doi.org/10.1016/j.ab.2011.06.027
van der Vliet G.M., Schepers P., Schukkink R.A., van Gemen B., Klatser P.R. Assessment of mycobacterial viability by RNA amplification // Antimicrob. Agents. Chemother. 1994. V. 38. P. 1959‒1965. https://doi.org/10.1128/AAC.38.9.1959
Vesper S., McKinstry C., Hartmann C., Neace M., Yoder S., Vesper A. Quantifying fungal viability in air and water samples using quantitative PCR after treatment with propidium monoazide (PMA) // J. Microbiol. Methods. 2008. V. 72. P. 180‒184. https://doi.org/10.1016/j.mimet.2007.11.017
Villarreal J.V., Jungfer C., Obst U., Schwartz T. DNase I and Proteinase K eliminate DNA from injured or dead bacteria but not from living bacteria in microbial reference systems and natural drinking water biofilms for subsequent molecular biology analyses // J. Microbiol. Methods. 2013. V. 94. P. 161‒169. https://doi.org/10.1016/j.mimet.2013.06.009
Wagner A.O., Malin C., Knapp B.A., Illmer P. Removal of free extracellular DNA from environmental samples by ethidium monoazide and propidium monoazide // Appl. Environ. Microbiol. 2008. V. 74. P. 2537‒2539. https://doi.org/10.1128/AEM.02288-07
Wahman D.G., Wulfeck-Kleier K.A., Pressman J.G. Monochloramine disinfection kinetics of Nitrosomonas eu-ropaea by propidium monoazide quantitative PCR and Live/dead BacLight methods // Appl. Environ. Microbiol. 2009. V. 75. P. 5555‒5562. https://doi.org/10.1128/AEM.00407-09
Wang H., Gill C.O., Yang X. Use of sodium lauroyl sarcosinate (sarkosyl) in viable real-time PCR for enumeration of Escherichia coli // J. Microbiol. Methods. 2014a. V. 98. P. 89‒93. https://doi.org/10.1016/j.mimet.2014.01.004
Wang L., Li P., Zhang Z., Chen Q., Aguilar Z.P., Xu H., Yang L., Xu F., Lai W., Xiong Y., Wei H. Rapid and accurate detection of viable Escherichia coli O157:H7 in milk using a combined IMS, sodium deoxycholate, PMA and real-time quantitative PCR process // Food Control. 2014b. V. 36. P. 119‒125. https://doi.org/10.1016/j.foodcont.2013.08.011
Weigel K.M., Nguyen F.K., Kearney M.R., Meschke J.S., Cangelosi G.A. Molecular viability testing of UV-inactivated bacteria // Appl. Environ. Microbiol. 2017. V. 83. pii: e00331-17. https://doi.org/10.1128/AEM.00331-17
Willers C., Wentzel J.F., du Plessis L.H., Gouws C., Hamman J.H. Efflux as a mechanism of antimicrobial drug resistance in clinical relevant microorganisms: the role of efflux inhibitors // Expert. Opin. Ther. Targets. 2017. V. 21. P. 23‒36. https://doi.org/10.1080/14728222.2017.1265105
Wu G.P., Chen S.H., Levin R.E. Application of ethidium bromide monoazide for quantification of viable and dead cells of Salmonella enterica by real-time loop-mediated isothermal amplification // J. Microbiol Methods. 2015. V. 117. P. 41‒48. https://doi.org/10.1016/j.mimet.2015.07.012
Xing-Long X., Cong-Cong L., Yang Q., Yi-Gang Y., Hui W. Molecular monitoring of Escherichia coli O157:H7 sterilization rate using qPCR and propidium monoazide treatment // Lett. Appl. Microbiol. 2013. V. 56. P. 333‒339. https://doi.org/10.1111/lam.12052
Yan M., Xu L., Jiang H., Zhou Z., Zhou S., Zhang L. PMA-LAMP for rapid detection of Escherichia coli and shiga toxins from viable but non-culturable state // Microb. Pathog. 2017. V. 105. P. 245‒250. https://doi.org/10.1016/j.micpath.2017.02.001
Yáñez M.A., Nocker A., Soria-Soria E., Múrtula R., Martínez L., Catalán V. Quantification of viable Legionella pneumophila cells using propidium monoazide combined with quantitative PCR // J. Microbiol. Methods. 2011. V. 85. P. 124‒130. https://doi.org/10.1016/j.mimet.2011.02.004
Yang Y., Xu F., Xu H., Aguilar Z.P., Niu R., Yuan Y., Sun J., You X., Lai W., Xiong Y., Wan C., Wei H. Magnetic nano-beads based separation combined with propidium monoazide treatment and multiplex PCR assay for simultaneous detection of viable Salmonella typhimurium, Escherichia coli O157:H7 and Listeria monocytogenes in food products // Food Microbiol. 2013. V. 34. P. 418‒424. https://doi.org/10.1016/j.fm.2013.01.004
Yokomachi N., Yaguchi J. Enumeration of viable Escherichia coli by real-time PCR with propidium monoazide // Water Sci. Technol. 2012. V. 66. P. 2065‒2073. https://doi.org/10.2166/wst.2012.370
Youn S.Y., Jeong O.M., Choi B.K., Jung S.C., Kang M.S. Application of loop-mediated isothermal amplification with propidium monoazide treatment to detect live Salmonella in chicken carcasses // Poult. Sci. 2017. V. 96. P. 458‒464. https://doi.org/10.3382/ps/pew341
Zhang L., Chen Y., Cheng N., Xu Y., Huang K., Luo Y., Wang P., Duan D., Xu W. Ultrasensitive detection of viable Enterobacter sakazakii by a continual cascade nanozyme biosensor // Anal. Chem. 2017. V. 89. P. 10194‒10200. https://doi.org/10.1021/acs.analchem.7b01266
Zhao Y., Jiang X., Qu Y., Pan R., Pang X., Jiang Y., Man C. Salmonella detection in powdered dairy products using a novel molecular tool // J. Dairy. Sci. 2017. V. 100. P. 3480‒3496. https://doi.org/10.3168/jds.2016-12535
Zhou B., Liang T., Zhan Z., Liu R., Li F., Xu H. Rapid and simultaneous quantification of viable Escherichia coli O157:H7 and Salmonella spp. in milk through multiplex real-time PCR // J. Dairy. Sci. 2017. V. 100. P. 8804‒8813. https://doi.org/10.3168/jds.2017-13362
Zi C., Zeng D., Ling N., Dai J., Xue F., Jiang Y., Li B. An improved assay for rapid detection of viable Staphylococcus aureus cells by incorporating surfactant and PMA treatments in qPCR // BMC Microbiol. 2018. V. 18. P. 132. https://doi.org/10.1186/s12866-018-1273-x
Дополнительные материалы отсутствуют.