Микробиология, 2021, T. 90, № 2, стр. 226-235
Возрастные изменения дрожжевой составляющей микробиома Drosophila melanogaster
А. С. Дмитриева a, *, И. А. Максимова a, А. В. Качалкин a, b, А. В. Марков a, c
a Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
b Институт биохимии и физиологии микроорганизмов им. Г.К. Скрябина РАН
142290 Пущино, Россия
c Палеонтологический институт им. А.А. Борисяка РАН
117997 Москва, Россия
* E-mail: dmnastya89@mail.ru
Поступила в редакцию 16.09.2020
После доработки 03.10.2020
Принята к публикации 15.10.2020
Аннотация
Плодовые мушки Drosophila melanogaster – важный модельный объект для изучения многоплановых взаимодействий между многоклеточным организмом и его микробиомом. Характер этих взаимодействий во многом определяется закономерными изменениями численности и состава компонентов микробиома, происходящими в течение жизни организма-хозяина. Имеющиеся на сегодняшний день данные об изменениях микробиома дрозофил в зависимости от возраста и стадии жизненного цикла насекомого относятся в основном к бактериальной части микробиома, в то время как об аналогичных изменениях не менее важного дрожжевого компонента известно немного. В данной работе изучен количественный и качественный состав дрожжевой составляющей микробиома дрозофил из трех лабораторных линий, различающихся условиями содержания, на четырех возрастных стадиях: у поздних личинок и имаго в возрасте 1, 7 и 14 сут после выхода из куколки. Во всех трех линиях численность дрожжей сходным образом меняется с возрастом насекомых, причем максимальное количество дрожжей характерно для имаго 7-дневного возраста, а минимальное – для имаго 1-дневного возраста. У мух, содержащихся на умеренно неблагоприятных субстратах с добавлением 2 и 4% NaCl, численность и видовое разнообразие дрожжей на всех четырех стадиях выше, чем у мух, содержащихся на стандартном (благоприятном) кормовом субстрате. Результаты свидетельствуют о непостоянстве дрожжевой составляющей микробиома дрозофил и его закономерных изменениях с возрастом насекомого, что необходимо учитывать при изучении взаимоотношений между симбиотическими дрожжами и организмом-хозяином.
Важную роль в жизни большинства многоклеточных организмов играет микробиом – сообщество разнообразных микроорганизмов (прокариот, протистов, грибов), связанных с организмом-хозяином и друг с другом сложной сетью взаимодействий, которые могут варьировать от антагонистических до мутуалистических. Компоненты микробиома могут передаваться от родителей к потомкам и влиять на различные аспекты физиологии, онтогенеза и поведения организмов-хозяев, а также на их адаптацию к меняющимся условиям среды (McFall-Ngai, 2002; Zilber-Rosenberg, 2008; Rosenberg, 2007; Dmitrieva et al., 2019). Поэтому в рамках набирающей популярность “хологеномной теории эволюции” предлагается считать базовой единицей отбора не отдельный организм, а “холобионт” (Margulis, Fester, 1991) или “аутоценоз” (Савинов, 2012), то есть систему, включающую макроорганизм и его микробиом (Zilber-Rosenberg, Rosenberg, 2008; Rosenberg et al., 2009; Bordenstein, Theis, 2015). Впрочем, хологеномная теория подвергается критике со стороны других исследователей, подчеркивающих неустойчивость передачи микробиома в ряду поколений, а также нередко наблюдаемую эфемерность и оппортунистический характер взаимоотношений между организмом-хозяином и ассоциированными с ним микробами (Moran, Sloan, 2015).
Плодовая мушка Drosophila melanogaster является одним из наиболее популярных модельных объектов для изучения этих взаимоотношений в силу хорошей изученности данного вида и относительной простоты его микробиома, сходного по ряду признаков с микробиомом млекопитающих (Erkosar et al., 2013; Erkosar, Leulier, 2014; Newell et al., 2014; Hoang et al., 2015; Trinder et al., 2017; Douglas, 2018).
Ключевыми компонентами микробиома D. melanogaster являются бактерии и дрожжи, переносимые мухами в кишечнике и на поверхности тела. Большинство исследований в настоящее время фокусируется на кишечных бактериях дрозофил (Brummel et al., 2004; Shin et al., 2011; Broderick, Lemaitre, 2012; Erkosar et al., 2013; Yamada et al., 2015; Wong et al., 2016). Между тем дрожжи, по-видимому, составляют не менее важную часть микробиома плодовых мушек (Starmer, 1981; Anagnostou et al., 2010; Becher et al., 2012; Chandler et al., 2012; Hoang et al., 2015). Дрожжи необходимы для нормального развития личинок D. melanogaster на природных субстратах (Becher et al., 2012). Разные виды дрожжей по-разному влияют на выживаемость и скорость развития личинок, а личинки проявляют избирательность, предпочитая питаться теми дрожжами, которые положительно влияют на их рост (Starmer, 1981; Anagnostou et al., 2010). Личинки и имаго дрозофил, со своей стороны, влияют на видовой состав дрожжевых сообществ, развивающихся на кормовых субстратах, способствуя размножению определенных видов дрожжей и препятствуя росту мицелиальных грибов (Stamps et al., 2012). Некоторые виды дрожжей выдерживают прохождение через кишечник дрозофил, что позволяет дрозофилам служить эффективными распространителями дрожжей в природных условиях (Reuter et al., 2007; Coluccio et al., 2008; Stamps et al., 2012; Hoang et al., 2015; Günther, Goddard, 2019). Также известно, что видовой состав дрожжевой составляющей микробиома дрозофилид сильно зависит от диеты мух (Chandler et al., 2012). Ранее нами было показано, что изменения дрожжевой составляющей микробиома вносят существенный вклад в наблюдаемую в эволюционных экспериментах адаптацию лабораторных линий D. melanogaster к кормовым субстратам с повышенным содержанием NaCl (Панченко и соавт., 2017; Ивницкий и соавт., 2018; Dmitrieva et al., 2019).
Исключительно важным, но слабо изученным аспектом взаимоотношений между микробиомом и организмом-хозяином являются закономерные изменения численности и состава микробиома, происходящие в течение жизни макроорганизма (Nicholson et al., 2012). Такие изменения показаны для кишечных бактерий D. melanogaster. Общая численность бактерий в кишечнике личинок постепенно растет, выходя на плато к третьей личиночной стадии, и резко снижается на стадии куколки (Broderick, Lemaitre, 2012). В течение жизни имаго в кишечнике D. melanogaster преобладание Lactobacillus fructivorans и других лактобацилл может постепенно сменяться преобладанием Acetobacter pomorum и других ацетобактерий (Wong et al., 2011; Erkosar et al., 2013). Также показано, что рост численности некоторых групп бактерий, особенно гаммапротеобактерий, происходящий в кишечнике D. melanogaster в течение жизни имаго, может приводить к дисфункции кишечника, ускоряя старение и смерть (Clark et al., 2015). В отличие от бактериальной части микробиома D. melanogaster, о возрастных изменениях его дрожжевой части известно очень немного. По-видимому, живые дрожжевые клетки могут передаваться от личинки к имаго в ходе метаморфоза, но только при наличии в кишечнике личинки определенных кишечных бактерий (Guilhot et al., 2020). Данных об изменениях дрожжевой части микробиома с возрастом имаго в литературе найти не удалось. Отсутствие детальных данных об изменениях дрожжевой составляющей микробиома дрозофил, связанных с возрастом и стадией жизненного цикла, препятствует адекватному пониманию взаимоотношений дрожжей с насекомым-хозяином.
В данном исследовании мы предприняли попытку частично восполнить этот пробел, охарактеризовав численность и видовой состав дрожжевой части микробиома трех лабораторных линий D. melanogaster на четырех возрастных стадиях.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Подопытные линии мух. Изучен состав дрожжевой составляющей микробиома у трех лабораторных линий D. melanogaster (линии Н, 2С, 4С), полученных от одной исходной популяции в октябре 2014 г. и содержащихся на разных кормовых субстратах в рамках эволюционного эксперимента, проводимого на кафедре биологической эволюции МГУ (Марков и соавт., 2015; Belkina et al., 2018; Dmitrieva et al., 2019). Исходная популяция была получена в начале сентября 2014 г. от 30 диких особей, пойманных на юго-западе г. Москвы.
Н – контрольная линия, выращиваемая на стандартной благоприятной кормовой среде (60 г дрожжей Saccharomyces cerevisiae, 35 г манной крупы, 50 г сахара, 45 г измельченного изюма кипятятся в 1 л воды в течение 4 ч; за 15 мин до окончания кипячения добавляется 8 г агара и по окончании кипячения – 2 г пропионовой кислоты).
2С и 4С – линии, выращиваемые на такой же среде с добавлением 2 и 4% NaCl соответственно.
Популяции содержали в цилиндрических стеклянных банках с кормом диаметром 64 мм и высотой 100 мм, закрытых ватными, обернутыми в марлю, пробками, при естественном освещении и температуре 20–25°С. Объем корма в банке составлял 84 мл. Каждая популяция занимала по три банки. Каждые две недели все имаго, находящиеся в сосуде, обездвиживали углекислым газом и извлекали из сосуда; затем из них случайным образом отбирали 10 самцов и 10 самок, которые помещали в банку со свежим кормом. Также в каждой банке находился цилиндрический пластиковый резервуар объемом 1 мл, заполненный мокрой ватой (поилка). До начала июня 2015 г. имаго из всех трех банок, относящихся к данной популяции, перемешивались перед каждой пересадкой в свежие банки. Таким образом, население трех сосудов составляло единую популяцию. Начиная с июня 2015 г. тройки линий, составляющие каждую популяцию, содержали раздельно: из каждой банки мухи пересаживали только в одну банку со свежим кормом.
Ранее нами было показано, что мухи из линий 2С и 4С уже спустя год после начала адаптации к соленому кормовому субстрату размножались на таком субстрате эффективнее (оставляли больше потомков), чем мухи из контрольной линии Н (Дмитриева и соавт., 2016). Это различие сохранилось спустя три года после начала эволюционного эксперимента, и было показано, что дрожжевая часть микробиома вносила существенный вклад в наблюдаемую адаптацию дрозофил к соленому субстрату (Дмитриева и соавт., 2016; Панченко и соавт., 2017; Ивницкий и соавт., 2018).
Стадии жизненного цикла. Для каждой из трех линий состав дрожжевой составляющей микробиома оценивали на четырех стадиях жизненного цикла: личинки последней стадии (непосредственно перед окукливанием), молодые имаго в возрасте 1 сут после выхода их куколки, имаго в возрасте 7 сут и имаго в возрасте 14 сут.
Методика проведения эксперимента. Эксперимент проводили в марте–мае 2019 г. Из каждой линии мух случайным образом выбирали “родителей”: 30 самцов и 30 самок. Это делали в конце двухнедельного цикла (см. выше); родители представляли собой молодых имаго в возрасте не более 2–3 сут с момента выхода из куколки. Родителей из каждой линии рассаживали в три банки со свежим кормом, соответствующим данной линии (стандартный корм для линии Н, корм с 2% NaCl для линии 2С, корм с 4% NaCl для линии 4С), по 10 самцов и 10 самок в каждую банку. Таким образом, всего было девять банок с родителями, по три банки на линию. Через 7 сут родителей удаляли из банок. Еще через 5 сут производили отбор личинок для анализа дрожжевой составляющей микробиома. Отбирались личинки последней стадии накануне окукливания (такие личинки покидают кормовой субстрат и выползают на стенки банки). Из каждой линии отбирали по 10 личинок, из которых готовили гомогенат, который затем высевали на пять чашек Петри (см. ниже). Одновременно с личинками из каждой линии отбирали для приготовления гомогената по 10 молодых имаго, вышедших из куколки в течение предшествующих суток. Остальные имаго, тоже вышедшие из куколки в течение предшествующих суток (“фокальная группа” мух с известным нам возрастом), оставались в своих банках. По достижении этими имаго возраста 7 и 14 сут из них отбирали по 10 особей (в каждой из трех линий) для приготовления гомогената, который высевали на пять чашек Петри. В кормовом субстрате при этом продолжали жить и развиваться личинки, среди которых были как сиблинги, так и потомки мух из “фокальной группы”. Такой дизайн эксперимента был выбран, чтобы сукцессия микроорганизмов в кормовом субстрате развивалась “естественным” для данной линии мух образом. Чтобы к фокальной группе мух известного нам возраста не подмешивались новые мухи, из каждой банки регулярно удаляли появляющиеся на ее стенках куколки.
Таким образом, методика эксперимента была ориентирована на анализ естественной для данной линии мух возрастной динамики дрожжевого микобиома. При этом мы не ставили задачу разделения изменений микробиома, обусловленных возрастными изменениями и сменой стадий жизненного цикла самих мух и естественной сукцессией микроорганизмов в кормовом субстрате (подробнее этот вопрос рассмотрен в разделе “Обсуждение”). Не ставилась также и задача разделения дрожжей, находящихся на кутикуле и в пищеварительном тракте насекомого (для решения этих задач необходимы дополнительные исследования и другие методические подходы).
Методика приготовления гомогенатов личинок и имаго. Всего было приготовлено 12 гомогенатов (по одному для каждой из четырех возрастных стадий каждой из трех линий мух). Для приготовления гомогената 10 особей (личинок или имаго) помещали для обездвиживания в морозильную камеру с температурой –20°С на 3 мин. Затем из этих насекомых и 0.5 мл стерильной водопроводной воды готовили гомогенат. Для этого в стерильные эппендорфы с водой помещали мух, растирали их стерильным силиконовым пестиком, а затем обрабатывали на вортексе Multi Reax (“Heidolph”, Германия) в режиме 1700 об./мин в течение 10 мин.
Методика оценки состава дрожжевой составляющей микробиома. Состав дрожжевой составляющей микробиома изучали с помощью метода микробиологического посева гомогенатов мух из линий Н, 2С, 4С на плотную питательную среду. Для этого к 100 мкл гомогената (см. выше) добавляли 900 мкл стерильной воды и еще раз обрабатывали на вортексе в течение 3 мин. Аликвоту полученной суспензии объемом 50 мкл с помощью дозатора со стерильным наконечником наносили на поверхность агаризованной питательной среды ГПД следующего состава (г/л): глюкоза – 20, пептон – 10, дрожжевой экстракт – 5, агар – 20. Для подавления роста бактерий в среду перед разливом добавляли хлорамфеникол (1 г/л). Каждый из 12 гомогенатов был нами посеян по методу Дригальского в 5-кратной повторности (по 5 чашек Петри на каждую комбинацию линия/возрастная стадия). Чашки с посевами инкубировали в течение 5 сут при комнатной температуре (20–22°С). По прошествии этого времени все выросшие колонии дрожжей были на основании макроморфологических признаков разделены на типы и подсчитаны. В результате для каждого образца была определена общая численность дрожжей в колониеобразующих единицах (КОЕ) в пересчете на одну муху. По два–три штамма из каждого морфотипа колоний было выделено в чистую культуру, а затем на основании культуральных и микроморфологических признаков все полученные культуры были сгруппированы. Видовую идентификацию дрожжевых грибов проводили на основе анализа нуклеотидной последовательности ITS региона рДНК. Выделение ДНК и постановку ПЦР проводили по ранее описанной методике (Глушакова, Качалкин, 2017). Секвенирование ДНК проводили с помощью набора реактивов Big Dye Terminator V3.1 Cycle Sequencing Kit (“Applied Biosystems”, США) с последующим анализом продуктов реакции на секвенаторе Applied Biosystems 3130xl Genetic Analyzer в ЗАО “Евроген” (Москва). Для секвенирования был использован праймер ITS5 (5'-GGA AGT AAA AGT CGT AAC AAG G). Идентификацию дрожжей на основании полученных результатов секвенирования проводили, используя данные генбанка NCBI (www.ncbi.nlm.nih.gov) и базы данных MycoID (www.mycobank.org). Полученные в ходе исследования нуклеотидные последовательности были размещены в генбанке NCBI (MT664162−MT664168).
РЕЗУЛЬТАТЫ
В исследованных гомогенатах обнаружено в общей сложности четыре вида дрожжей (рис. 1 ):
Рис. 1.
Численность и видовой состав дрожжевого микробиома мух Drosophila melanogaster из трех лабораторных линий (a – Н; б – 2С; в – 4С) на четырех возрастных стадиях (личинки и имаго в возрасте 1, 7 и 14 сут). Показана общая численность дрожжей в КОЕ на одну муху, ±стандартная ошибка, шкала логарифмическая. Круговые диаграммы показывают видовой состав дрожжей, обнаруженных в посевах: Ccal – Candida californica, Pocc – Pichia occidentalis, Pmem – Pichia membranifacies, Zbai – Zygosaccharomyces bailii. В двух случаях (круговые диаграммы, выделенные пунктиром) нам не удалось точно определить относительное обилие обнаруженных видов дрожжей; в этих случаях показано их приблизительное соотношение: в линии 2С у имаго в возрасте 7 сут дрожжи P. occidentalis и P. membranifacies представлены примерно в равной пропорции, в линии 4С у имаго того же возраста дрожжи P. occidentalis и C. californica присутствуют в соотношении, близком к 10 : 1.
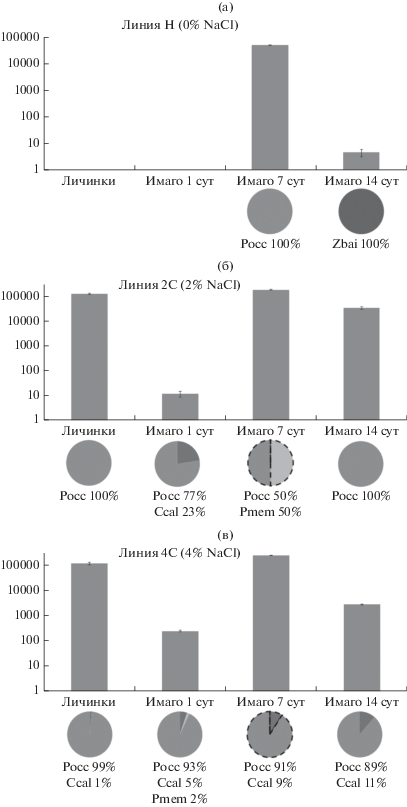
1) Candida californica Mrak & McClung ex (Mrak & McClung ex K.W. Anderson & C.E. Skinner, 2006). Вид обнаружен в линии 2С у имаго в возрасте 1 сут и в линии 4С на всех четырех возрастных стадиях;
2) Pichia membranifaciens (E.C. Hansen, 1904) E.C. Hansen. Вид обнаружен в линии 2С у имаго в возрасте 7 сут и в линии 4С у имаго в возрасте 1 сут;
3) Pichia occidentalis (Kurtzman, Smiley & Johnson, 2008) Kurtzman, Robnett & Basehoar-Powers. Вид обнаружен в линии Н у имаго в возрасте 7 сут, а в двух остальных линиях – на всех четырех возрастных стадиях. В целом можно сказать, что данный вид является доминирующим у изученных линий дрозофил;
4) Zygosaccharomyces bailii (Barnett et al., 1983). Вид обнаружен только в линии Н у имаго в возрасте 14 сут.
Численность и видовой состав дрожжевой составляющей микробиома различались у разных линий мух и на разных возрастных стадиях.
Линия Н. В гомогенатах мух, содержавшихся на корме без добавления соли, обнаружено наименьшее количество дрожжей (рис. 1 а). У личинок и имаго в возрасте 1 сут дрожжи в посевах отсутствуют. При этом в посевах обнаружено большое количество мицелиальных грибов и бактерий, чего не наблюдалось во всех остальных посевах (гомогенатах). Возможно, это говорит об антагонистических отношениях между компонентами микробиома (дрожжи подавляют развитие бактерий и мицелиальных грибов или наоборот).
В посевах гомогената имаго в возрасте 7 сут обнаружено значительно количество дрожжей только одного вида P. occidentalis, средняя численность составила 5 × 104 КОЕ на муху.
В гомогенате имаго в возрасте 14 сут дрожжи тоже присутствовали, но в гораздо меньшем количестве – лишь 4.8 КОЕ на муху. Дрожжи в посевах представлены видом Z. bailii, который встречен только у этой линии и только на этой возрастной стадии.
Линия 2С. В гомогенатах мух, содержавшихся на корме с 2% NaCl, дрожжей обнаружено намного больше, причем на всех четырех возрастных стадиях (рис. 1б). Так, в гомогенате личинок присутствовало много дрожжей вида P. occidentalis (1.4 × 105 КОЕ на муху). У имаго в возрасте 1 сут дрожжей значительно меньше (12.4 КОЕ на муху), причем они представлены сразу двумя видами: P. occidentalis и C. californica. В посевах гомогената имаго в возрасте 7 сут обнаружено значительное количество дрожжей (2.0 × 105 КОЕ на муху) двух видов: P. occidentalis и P. membranifaciens. В гомогенате имаго в возрасте 14 сут дрожжи тоже присутствовали, но в меньшем количестве (3.7 × 104 КОЕ на муху). Они были представлены лишь одним видом P. occidentalis.
Линия 4С. В гомогенатах мух, содержавшихся на корме с 4% NaCl, также обнаружено большое количество дрожжей (рис. 1 в). У личинок присутствовали виды C. californica и P. membranifaciens; общая численность дрожжей – 1.1 × 105 КОЕ на муху. У имаго в возрасте 1 сут дрожжей намного меньше (2.5 × 102 КОЕ на муху), однако они принадлежали сразу к трем разным видам (P. occidentalis, P. membranifaciens, C. californica). В посевах гомогената имаго в возрасте 7 сут обнаружено много (2.4 × 105 КОЕ на муху) дрожжей двух видов (P. occidentalis, C. californica). В гомогенате имаго в возрасте 14 сут дрожжи тоже присутствовали, но в меньшем количестве (2.7 × 103 КОЕ на муху); представлены те же два вида. Видовое разнообразие дрожжей в линии 4С в целом выше, чем в линии 2С: в первой линии на всех четырех возрастных стадиях обнаружено более одного вида дрожжей, тогда как во второй линии на двух из четырех стадий в посевах был обнаружен только один вид дрожжей.
Таким образом, проведенный анализ показал существенные различия по количественному и видовому составу дрожжей в зависимости как от линии мух, так и от возрастной стадии.
ОБСУЖДЕНИЕ
Дрожжевая составляющая микробиома различается у дрозофил, содержащихся на разных кормовых субстратах. Проведенный анализ показал, что в линиях 2С и 4С, содержащихся на кормовом субстрате с добавлением 2 и 4% соли, количество колоний дрожжей в посевах больше и их видовое разнообразие было выше, чем в линии Н, содержащейся на корме без добавления соли. Это справедливо для всех четырех рассмотренных возрастных стадий. Набор обнаруженных видов дрожжей тоже различался: вид P. occidentalis доминировал у всех трех линий, Z. bailii обнаружен только в линии Н, C. californica и P. membranifaciens – только в линиях 2С и 4С. Эти результаты согласуются с нашими прежними выводами о том, что для линий мух, содержащихся на соленом субстрате, характерна повышенная численность и видовое разнообразие дрожжей (Dmitrieva et al., 2019).
Доминирующий у всех трех исследованных линий вид дрожжей Pichia occidentalis характеризуется очень узким ассимиляционным спектром (в качестве единственного источника углерода ассимилирует только глюкозу, этанол, глицерин и молочную кислоту), способен расти на безвитаминных средах, является осмотолерантным видом (выдерживает до 10% NaСl или 5% глюкозы в среде), сбраживает глюкозу, способен к росту при 37°С. Такие физиологические особенности характерны для видов, обитающих в очень специфических локусах, в частности, данный вид, согласно литературным данным, встречается в высокосахаристых плодах и в продуктах их естественного сбраживания, например, винах (Kurtzman et al., 2011). По всей видимости, во все вышеперечисленные субстраты данный вид дрожжей привносится дрозофилами.
Известно, что диета сильно влияет на состав бактериальной составляющей микробиома дрозофил (Broderick, Lemaitre, 2012). В частности, показано, что при переходе мух, содержавшихся на разных диетах, на один и тот же кормовой субстрат, их микробиом становился сходным, а при переносе изогенных популяций с идентичными микробиомами на разные кормовые субстраты состав доминирующих видов бактерий изменяется в зависимости от среды (Chandler et al., 2011). В природных популяциях различных видов дрозофил диета, по-видимому, даже сильнее влияет на состав кишечных бактерий, чем видовая принадлежность мух (Staubach et al., 2013).
О том, как влияет диета на дрожжевую часть микробиома дрозофил, известно немного. По-видимому, как и в случае с бактериями, диета сильнее влияет на состав дрожжей, ассоциированных с дрозофилами, чем видовая принадлежность мух (Chandler et al., 2012). Наши результаты дополняют эти данные, показывая, что повышенное содержание соли в кормовом субстрате, по-видимому, может способствовать росту численности и разнообразия дрожжевого микробома дрозофил. Возможные причины этого эффекта нуждаются в дополнительных исследованиях.
Дрожжевая составляющая микробиома D. melanogaster закономерно меняется в течение жизни насекомого. Во всех трех рассмотренных линиях (Н, 2С, 4С) выявлены сходные тенденции изменения численности дрожжей в зависимости от возрастной стадии. А именно, во всех трех случаях у имаго в возрасте 1 сут наблюдается минимальная численность дрожжей, а у имаго в возрасте 7 сут – максимальная.
Выявленные изменения дрожжевой составляющей микробиома могут быть связаны с двумя факторами. Первый из них – возрастные изменения и смена стадий жизненного цикла самого насекомого. Пищеварительный тракт и поверхность тела личинок и имаго разного возраста могут предоставлять разные условия для выживания и размножения разных видов дрожжей. Поведение мух (в том числе пищевое) тоже может меняться с возрастом, влияя на интенсивность обмена дрожжевыми клетками между субстратом и организмом насекомого. Кроме того, известно, что бактериальная составляющая микробиома дрозофил закономерно меняется с возрастом мухи, а между дрожжами и бактериями, обитающими в кишечнике, вероятно, существует сложная система экологических взаимодействий (Wong et al., 2011; Broderick, Lemaitre, 2012; Clark et al., 2015; Guilhot et al., 2020). Второй фактор – микробиологическая сукцессия на кормовом субстрате, связанная с постепенной утилизацией и переработкой корма личинками и имаго мух и микроорганизмами, с подсыханием корма, с динамикой популяций развивающихся в корме микробов и экологическими взаимодействиями между ними.
В нашем эксперименте кормовой субстрат “старел” вместе с живущими на нем мухами. По-видимому, такая ситуация вполне типична для дрозофил в природных условиях, поскольку дрозофилы, с одной стороны, используют эфемерные кормовые субстраты с быстро меняющимся микробным населением, с другой – склонны проводить большую часть жизни возле кормового субстрата, в котором развиваются их личинки (Broderick, Lemaitre, 2012). Использованный в данной работе подход, таким образом, имитирует реалистичный сценарий взаимодействия между насекомым, его микробиомом и микробным населением кормового субстрата, но при этом затрудняет раздельный анализ влияния двух названных выше факторов. Для проведения такого анализа необходимы дополнительные исследования.
Тем не менее, полученные результаты позволяют утверждать, что, по крайней мере, одна из выявленных тенденций, а именно низкая численность дрожжевой составляющей микробиома у имаго в возрасте 1 сут, по сравнению с поздними личинками, связана именно с возрастной стадией насекомого, а не с сукцессией микроорганизмов в кормовом субстрате (поскольку личинки и однодневные имаго отбирались для анализа одновременно, через 12 сут после начала использования мухами кормового субстрата). Известно, что в процессе метаморфоза у дрозофил происходит резкое обеднение бактериального микробома, что отчасти связано с усилением синтеза антимикробных пептидов на стадии ранней куколки (Tryselius et al., 1992; Broderick, Lemaitre, 2012; Erkosar et al., 2013). С другой стороны, бактерии все же могут передаваться от личинки к имаго, в частности, путем поедания молодыми мухами собственных фекалий (мекония), содержащих остатки личиночной средней кишки (Broderick, Lemaitre, 2012; Téfit et al., 2018; Guilhot et al., 2020). Дрожжевая составляющая микробиома, по-видимому, тоже может быть частично сохранена в процессе метаморфоза (Guilhot et al., 2020). Однако ее численность, судя по нашим данным, сильно сокращается. Об этом свидетельствует резкое (на 3–4 порядка) уменьшение числа дрожжевых клеток в гомогенатах молодых имаго по сравнению с поздними личинками в линиях 2С и 4С (в линии Н дрожжи не были обнаружены ни у молодых имаго, ни у личинок) (рис. 1).
Причины наблюдаемого во всех трех линиях снижения численности дрожжей у имаго в возрасте 14 сут (по сравнению с семидневными мухами) нуждаются в дополнительном изучении. Они могут быть связаны с возрастными изменениями мух, с динамикой бактериального населения их кишечника, а также со “старением” кормовой среды (подсыханием, истощением питательных веществ в поверхностном слое корма из-за деятельности личинок и микроорганимов). Интересно, что наиболее выраженное снижение численности дрожжей у 14-дневных мух по сравнению 7-дневными наблюдается в линии Н (примерно на четыре порядка), тогда как в линиях 2С и 4С численность дрожжей у 14-дневных мух снижается лишь на 1–2 порядка (рис. 1 ). Возможно, это связано с тем, что соль замедляет “старение” кормовой среды, работая как консервант и препятствуя размножению бактерий в большей степени, чем дрожжей.
Особенностью линии Н является отсутствие дрожжей в посевах гомогенатов личинок и молодых имаго (рис. 1а). При этом в обоих случаях в посевах обнаруживается много бактерий (очевидно, устойчивых к присутствующему в среде хлорамфениколу) и мицелиальных грибов, которые в остальных посевах не были зарегистрированы. Возможно, это указывает на конкурентные взаимоотношения между бактериями и мицелиальными грибами с одной стороны и дрожжами – с другой. В связи с этим интересно отметить, что в недавно опубликованной статье, посвященной антагонистической активности некоторых видов дрожжей по отношению к ряду фитопатогенных грибов, было показано, что изоляты дрожжей P. occidentalis (вида, доминирующего в изученных нами линиях дрозофил), способны полностью подавлять рост некоторых микромицетов (Choińska et al., 2020). Это позволяет предположить, что дрожжи P. occidentalis могут вносить вклад в обнаруженную ранее (Stamps et al., 2012) способность дрозофил контролировать рост мицелиальных грибов в своих питательных субстратах.
В целом результаты показывают, что состав и численность дрожжевой составляющей микробиома дрозофил зависят от кормового субстрата, от возраста мух и, возможно, от “возраста” субстрата. Это обстоятельство необходимо учитывать при изучении симбиотических взаимоотношений дрозофил и дрожжей.
Состав дрожжевой составляющей микробиома меняется со временем даже в одной и той же лабораторной линии. Известно, что состав бактериальной составляющей микробиома лабораторных линий D. melanogaster сильно варьирует в разных лабораториях (а иногда и у разных линий в одной и той же лаборатории) даже при использовании одинакового кормового субстрата (Chandler et al., 2011; Broderick, Lemaitre, 2012). По-видимому, это указывает на значительную роль фактора случайности в формировании микробного населения лабораторных линий дрозофил. Наши результаты показывают, что переменчивость свойственна и дрожжевой части микробиома.
Ранее (в ноябре–декабре 2017) мы теми же методами охарактеризовали состав дрожжевой составляющей микробиома у имаго в возрасте 0–5 сут из линий Н и 4С (Dmitrieva et al., 2019). В данной работе мы анализировали те же линии спустя 16–18 месяцев (март–май 2019). Были обнаружены следующие различия. В линии Н в 2017 г. доминировали виды Z. bailii и P. occidentalis, однако кроме них присутствовали также немногочисленные клетки P. membranifaciens. В 2019 г. в этой линии первые два вида по-прежнему присутствовали, однако третий вид не был обнаружен.
В линии 4С в 2017 году были зарегистрированы виды P. occidentalis и C. californica (присутствующие также и в 2019 году), однако доминирующим был вид Starmerella bacillaris (Kroemer, Krumbholz) F.L. Duarte, A. Fonseca, 2012, который в посевах 2019 г. не был обнаружен. Факт необнаружения дрожжей S. bacillaris у мух из линии 4С примечателен, поскольку ранее нами было показано, что эти дрожжи вносят заметный вклад в адаптацию лабораторных линий D. melanogaster к соленому корму, повышая эффективность размножения мух на кормовом субстрате с высоким содержанием NaCl (Ивницкий и соавт., 2018; Dmitrieva et al., 2019). То, что этот важный для жизни на соленом корме вид дрожжей в ходе данного исследования обнаружен не был, говорит о том, насколько неустойчивыми и эфемерными могут быть адаптации, происходящие на уровне “холобионта” путем адаптивной модификации микробиома (Марков, Ивницкий, 2016).
Таким образом, исследование показало, что состав дрожжевой части микробиома лабораторных линий D. melanogaster пластичен и непостоянен. По-видимому, он зависит и от кормового субстрата, и от стадии жизненного цикла насекомого, и от других факторов, включая физические условия и экологические взаимодействия с другими компонентами микробиома (бактериями, грибами). Это непостоянство и зависимость от множества факторов необходимо учитывать при изучении симбиоза дрозофил и дрожжей и его возможной эволюционной роли.
Список литературы
Глушакова А.М., Качалкин А.В. Эндофитные дрожжи в сочных плодах Malus domestica и Pyrus communis в условиях антропогенизации // Микробиология. 2017. Т. 86. С. 114–122.
Glushakova A.M., Kachalkin A.V. Endophytic yeasts in Malus domestica and Pyrus communis fruits under anthropogenic impact // Microbiology (Moscow). 2017. V. 86. P. 128–135.
Дмитриева А.С., Ивницкий С.Б., Марков А.В. Адаптация Drosophila melanogaster к неблагоприятному кормовому субстрату сопровождается расширением трофической ниши // Журн. общей биологии. 2016. Т. 77. С. 249–261.
Dmitrieva A.S., Ivnitsky S.B., Markov A.V. Adaptation of Drosophila melanogaster to unfavorable feed substrate is accompanied by expansion of trophic niche // Biology Bull. Rev. 2017. V. 7. P. 369–379.
Ивницкий С.Б., Максимова И.А., Панченко П.Л., Дмитриева А.С., Качалкин А.В., Корнилова М.Б., Перфильева К.С., Марков А.В. Роль микробиома в адаптации Drosophila melanogaster к кормовому субстрату с повышенной концентрацией NaCl // Журн. общей биологии. 2018. Т. 79. С. 393–403.
Ivnitsky S.B., Maximova I.A., Panchenko P.L., Dmitrieva A.S., Kachalkin A.V., Kornilova M.B., Perfilieva K.S., Markov A.V. Microbiome affects the adaptation of Drosophila melanogaster to a high NaCl concentration // Biology Bull. Rev. 2019. V. 9. P. 465–474.
Марков А.В., Ивницкий С.Б., Корнилова М.Б., Наймарк Е.Б., Широкова Н.Г., Перфильева К.С. Материнский эффект маскирует адаптацию к неблагоприятным условиям и затрудняет дивергенцию у Drosophila melanogaster // Журн. общ. биологии. 2015. Т. 76. С. 429–437.
Markov A.V., Ivnitsky S.B., Kornilova M.B., Naimark E.B., Shirokova N.G., Perfilieva K.S. Maternal effect obscures adaptation to adverse environments and hinders divergence in Drosophila melanogaster // Biology Bull. Rev. 2016. V. 6. P. 429–435.
Марков А.В., Ивницкий С.Б. Эволюционная роль фенотипической пластичности // Вестн. Моск. ун-та. Сер. 16. Биология. 2016. № 4. С. 3–11.
Markov A.V., Ivnitsky S.B. Evolutionary role of phenotypic plasticity // Moscow University Biological Sciences Bulletin. 2016. V. 71. № 4. P. 185–192.
Панченко П.Л., Корнилова М.Б., Перфильева К.С., Марков А.В. Симбиотическая микробиота вносит вклад в адаптацию Drosophila melanogaster к неблагоприятной кормовой среде // Известия РАН. Сер. биол. 2017. Т. 44. С. 341–351.
Panchenko P.L., Kornilova M.B., Perfilieva K.S., Markov A.V. Contribution of symbiotic microbiota to adaptation of Drosophila melanogaster to an unfavorable growth medium // Biology Bulletin. 2017. V. 44. P. 345–354. https://doi.org/10.1134/s1062359017040100
Савинов А.Б., Аутоценоз и демоценоз как симбиотические системы и биологические категории // Журн. общей биологии. 2012. Т. 73. С. 284–301.
Anagnostou C., Dorsch M., Rohlfs M. Influence of dietary yeasts on Drosophila melanogaster life-history traits // Entomol. Exp. Appl. 2010. V. 136. P. 1–11.
Becher P.G., Flick G., Rozpędowska E., Schmidt A., Hagman A., Lebreton S., Larsson M.C., Hansson B.S., Piškur J., Bengtsson M. Yeast, not fruit volatiles mediate Drosophila melanogaster attraction, oviposition and development // Functional Ecology. 2012. V. 26. P. 822–828.
Belkina E.G., Naimark E.B., Gorshkova A.A., Markov A.V. Does adaptation to different diets result in assortative mating? Ambiguous results from experiments on Drosophila // J. Evolution Biol. 2018. V. 31. P. 1803–1814.
Bordenstein S.R., Theis K.R. Host biology in light of the microbiota: ten principles of holobionts and hologenomes // PLoS Biol. 2015. V. 13. e1002226.
Broderick N.A., Lemaitre B. Gut-associated microbes of Drosophila melanogaster // Gut Microbes. 2012. V. 3. P. 307–321.
Brummel T., Ching A., Seroude L., Simon A.F., Benzer S. Drosophila lifespan enhancement by exogenous bacteria // Proc. Natl. Acad. Sci. USA. 2004. V. 101. P. 12974–12979.
Chandler J.A., Eisen J.A., Kopp A. Yeast communities of diverse Drosophila species: comparison of two symbiont groups in the same hosts // Appl. Environ. Microbiol. 2012. V. 78. P. 7327–7336.
Chandler J.A., Lang J.M., Bhatnagar S., Eisen J.A., Kopp A. Bacterial communities of diverse Drosophila species: ecological context of a host–microbe model system // PLoS Genet. 2011. V. 7. P. e1002272.
Choińska R., Piasecka-Jóźwiak K., Chabłowska B., Dumka J., Łukaszewicz A. Biocontrol ability and volatile organic compounds production as a putative mode of action of yeast strains isolated from organic grapes and rye grains // Antonie van Leeuwenhoek. 2020. V. 113. P. 1135–1146.
Clark R.I., Salazar A., Yamada R., Fitz-Gibbon S., Morselli M., Alcaraz J., Rana A., Rera M., Pellegrini M., Ja W.W., Walker D.W. Distinct shifts in microbiota composition during Drosophila aging impair intestinal function and drive mortality // Cell Reports. 2015. V. 12. P. 1656–1667.
Coluccio A.E., Rodriguez R.K., Kernan M.J., Neiman A.M. The yeast spore wall enables spores to survive passage through the digestive tract of Drosophila // PLoS One. 2008. V. 3. e2873.
Dmitrieva A.S., Ivnitsky S.B., Maksimova I.A., Panchenko P.L., Kachalkin A.V., Markov A.V. Symbiotic yeasts affect adaptation of Drosophila melanogaster to food substrate with high NaCl concentration // PLoS One. 2019. V. 14. e0224811.
Douglas A.E. The Drosophila model for microbiome research // Lab. Animal. 2018. V. 47. P. 157–164.
Erkosar B., Leulier F. Transient adult microbiota, gut homeostasis and longevity: novel insights from the Drosophila model // FEBS Lett. 2014. V. 588. P. 4250–4257.
Erkosar B., Storelli G., Defaye A., Leulier F. Host-intestinal microbiota mutualism: “learning on the fly” // Cell Host Microbe. 2013. V. 13. P. 8–14.
Guilhot R., Rombaut A., Xuéreb A., Howell K., Fellous S. Bacterial influence on the maintenance of symbiotic yeast through Drosophila metamorphosis // bioRxiv. Preprint posted June 01, 2020. https://doi.org/10.1101/2020.05.31.126185
Günther C.S., Goddard M.R. Do yeasts and Drosophila interact just by chance? // Fungal Ecology. 2019. V. 38. P. 37–43.
Hoang D., Kopp A., Chandler J.A. Interactions between Drosophila and its natural yeast symbionts – Is Saccharomyces cerevisiae a good model for studying the fly–yeast relationship? // Peer J. 2015. V. 3. e1116.
Kurtzman C.P., Fell J.W., Boekhout T. (eds.) The Yeasts, a Taxonomic Study. 5th edn. Elsevier, 2011. 2080 p.
Margulis L., Fester R. Symbiosis as a Source of Evolutionary Innovation: Speciation and Morphogenesis. Boston: MIT Press, 1991. 470 p.
McFall-Ngai M.J. Unseen forces: the influence of bacteria on animal development // Dev. Biol. 2002. V. 242. P. 1–14.
Moran N.A., Sloan D.B. The hologenome concept: helpful or hollow? // PLoS Biol. 2015. V. 13. e1002311.
Newell P.D., Chaston J.M., Wang Y., Winans N.J., Sannino D.R., Wong A.C.-N., Dobson A.J., Kagle J., Douglas A.E. In vivo function and comparative genomic analyses of the Drosophila gut microbiota identify candidate symbiosis factors // Front. Microbiol. 2014. V. 4. P. 576.
Nicholson J.K., Holmes E., Kinross J., Burcelin R., Gibson G., Jia W., Pettersson S. Host-gut microbiota metabolic interactions // Science. 2012. V. 336. P. 1262–1267.
Reuter M., Bell G., Greig D. Increased outbreeding in yeast in response to dispersal by an insect vector // Curr. Biol. 2007. V. 17. P. R81–R83.
Rosenberg E., Koren O., Reshef L., Efrony R., Zilber-Rosenberg I. The role of microorganisms in coral health, disease and evolution // Nat. Rev. Microbiol. 2007. V. 5. P. 355–362.
Rosenberg E., Sharon G., Zilber-Rosenberg I. The hologenome theory of evolution contains Lamarckian aspects within a Darwinian framework // Environ. Microbiol. 2009. V. 11. P. 2959–2962.
Shin S.C., Kim S.H., You H., Kim B., Kim A.C., Lee K.A., Yoon J.H., Ryu J.H., Lee W.J. Drosophila microbiota modulates host developmental and metabolic homeostasis via insulin signaling // Science. 2011. V. 334. P. 670–674.
Stamps J.A., Yang L.H., Morales V.M., Boundy-Mills K.L. Drosophila regulate yeast density and increase yeast community similarity in a natural substrate // PLoS One. 2012. V. 7. P. e42238.
Starmer W.T. A comparison of Drosophila habitats according to the physiological attributes of the associated yeast communities // Evolution. 1981. V. 35. P. 38–52.
Staubach F., Baines J.F., Künzel S., Bik E.M., Petrov D.A. Host species and environmental effects on bacterial communities associated with Drosophila in the laboratory and in the natural environment // PLoS One. 2013. V. 8. e70749.
Téfit M.A., Gillet B., Joncour P., Hughes S., Leulier F. Stable association of a Drosophila-derived microbiota with its animal partner and the nutritional environment throughout a fly population’s life cycle // J. Insect Physiol. 2018. V. 106. P. 2–12.
Trinder M., Daisley B.A., Dube J.S., Reid G. Drosophila melanogaster as a high-throughput model for host–microbiota interactions // Front. Microbiol. 2017. V. 8. Article 751.
Tryselius Y., Samakovlis C., Kimbrell D.A., Hultmark D. C-ecC, a cecropin gene expressed during metamorphosis in Drosophila pupae // Eur. J. Biochem. 1992. V. 204. P. 395–399.
Wong C.N.A., Ng P., Douglas A.E. Low-diversity bacterial community in the gut of the fruitfly Drosophila melanogaster // Environ. Microbiol. 2011. V. 13. P. 1889−1900.
Wong C.N.A., Vanhove A.S., Watnick P.I. The interplay between intestinal bacteria and host metabolism in health and disease: lessons from Drosophila melanogaster // Dis. Model. Mech. 2016. V. 9. P. 271–281.
Yamada R., Deshpande S.A., Bruce K.D., Mak E.M., Ja W.W. Microbes promote amino acid harvest to rescue undernutrition in Drosophila // Cell Rep. 2015. V. 10. P. 865–872.
Zilber-Rosenberg I., Rosenberg E. Role of microorganisms in the evolution of animals and plants: the hologenome theory of evolution // FEMS Microbiol. Rev. 2008. V. 32. P. 723–735.
Дополнительные материалы отсутствуют.


