Микробиология, 2021, T. 90, № 2, стр. 215-222
Пробиотический штамм Lactobacillus fermentum 39: биохимические свойства, особенности генома, антивирусная активность
И. В. Соловьева a, Н. А. Новикова a, А. Г. Точилина a, *, И. В. Белова a, А. Ю. Кашников a, Т. А. Сашина a, В. А. Жирнов a, С. Б. Молодцова a
a Федеральное бюджетное учреждение науки Нижегородский научно-исследовательский институт эпидемиологии
и микробиологии им. академика И.Н. Блохиной Роспотребнадзора
603950 Нижний Новгород, Россия
* E-mail: lab-lb@yandex.ru
Поступила в редакцию 26.05.2020
После доработки 16.07.2020
Принята к публикации 05.08.2020
Аннотация
Lactobacillus fermentum 39 является известным пробиотическим штаммом и широко используется для производства фармакопейных препаратов-пробиотиков, биологически активных добавок к пище и продуктов, обогащенных пробиотическими микроорганизмами. В ходе работы с использованием трансмиссионной электронной микроскопии изучена морфология клеток, подробно проанализированы биохимические свойства штамма и штаммовые особенности метаболизма сахаров, проведено полногеномное секвенирование, охарактеризован кластер генов, отвечающих за синтез экзополисахарида. Установлено, что клетки Lactobacillus fermentum 39 способны адсорбировать на себе ротавирусные частицы, что позволяет научно обосновать новый аспект применения пробиотиков, включающих данный штамм, при острых гастроэнтеритах, вызванных ротавирусами человека.
В настоящее время пробиотические бактерии родов Lactobacillus и Bifidobacterium признаны в качестве вакцинных адъювантов и терапевтических средств для лечения острого вирусного гастроэнтерита у детей (Majamaa et al., 1995; Isolauri, 2003; Lei et al., 2016). С начала 2000-х годов вопросу лечения и профилактики острых кишечных инфекций (ОКИ) вирусной этиологии с использованием пробиотических бактерий, в частности, бактерий рода Lactobacillus, уделялось много внимания (Ahmadi et al., 2015). Среди основных механизмов ингибирующего действия лактобацилл выделяют иммуностимулирующий эффект, приводящий к усилению иммунного ответа: индукции интерлейкинов, активации макрофагов, натуральных киллеров и Т-хелперов и стимуляции выработки иммуноглобулинов, в частности, иммуноглобулина А (IgA) (Arena et al., 2018). К другому механизму противовирусного действия пробиотиков относят продукцию активных метаболитов – пероксида водорода, лактата, бактериоцинов и бактерицидных субстратов, короткоцепочечных жирных кислот, которые понижают pH среды, предотвращают адгезию вирусных частиц на поверхности энтероцитов за счет изменения свойств их рецепторов и обладают прямым противовирусным действием (Salminen et al., 1998; Ермоленко и соавт., 2003; Botic et al., 2007; Sunmola et al., 2019). Есть данные о том, что выраженным противовирусным действием обладают экзополисахариды лактобацилл, представляющие собой гомо- или гетерополисахариды различного химического состава, имеющие штаммовую специфичность (Kim et al., 2018).
В ряде исследований показан противовирусный эффект пробиотических бактерий, обусловленный механическим связыванием вирусных частиц бактериальными клетками. В работе Н.Г. Шелковой и соавт. описан эффект адсорбции вирионов ротавируса на поверхности клеток и жгутиках E. coli M-17 (Шелковая и соавт., 1991). Изучению механизма адсорбции отводится важная роль в ряде исследований, рассматривающих взаимодействие бактерий рода Lactobacillus с вирусами, принадлежащими к различным семействам. Штаммы видов L. paracasei, L. reuteri, L. plantarum способны адсорбировать частицы вируса везикулярного стоматита (сем. Rabdoviridae), L. reuteri и L. casei – вирус Коксаки А (сем. Picornaviridae) и энтеровирус 71 (сем. Picornaviridae), представители вида L. plantarum – вирус Коксаки В4 (сем. Picornaviridae) и норовирусы человека (сем. Caliciviridae) (Botic et al., 2007; Ang et al., 2016; Lei et al., 2016; Arena et al., 2018). Механизм взаимодействия бактериальных клеток и вирусных частиц большинство исследователей трактует как физическую адсорбцию, которая осуществляется за счет неспецифических ван-дерваальсовых сил. В то же время в отдельных работах показано, что данное явление носит специфический характер, например, отдельные штаммы рода Lactobacillus способны улавливать частицы вируса иммунодефицита человека (сем. Retroviridae) путем распознавания богатых маннозой участков вирусного гликопротеина gp120 (Botic et al., 2007). Во всех исследованиях показано, что способность к адсорбции вирионов является штаммовой характеристикой (Botic et al., 2007; Lei et al., 2016).
В связи с этим исследование подробно охарактеризованных пробиотических штаммов рода Lactobacillus на наличие противовирусных свойств, в частности, изучение их прямого физического взаимодействия с вирусными частицами, представляется актуальной научной и практически значимой задачей.
Цель работы – изучение генетических особенностей, ключевых фенотипических свойств и характера взаимодействия с ротавирусом человека производственного пробиотического штамма Lactobacillus fermentum 39.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объект исследования – штамм Lactobacillus fermentum 39 из Государственной коллекции лактобацилл ФБУН ННИИЭМ им. академика И.Н. Блохиной Роспотребнадзора, зарегистрирован в коллекции Научного центра экспертизы средств медицинского назначения как Lactobacillus fermentum 1-20 (ГКПМ № 790039). Этот штамм давно известен и широко используется в промышленной микробиологии для производства пробиотиков. Lactobacillus fermentum 39 был выделен из фекалий здорового человека на кафедре микробиологии Тартуского государственного университета (Эстония), где в результате изучения биологических свойств была установлена его принадлежность к виду L. fermentum. Далее в 1967 г. штамм был передан в Нижегородский (тогда Горьковский) НИИЭМ, в 1969 году был разработан лабораторный регламент производства сухого пробиотического препарата “Лактобактерин”, где L. fermentum 39 использован в качестве штамма-продуцента. На данный момент L. fermentum 39 входит в перечень штаммов бактерий рода Lactobacillus, используемых в производстве лактосодержащих пробиотиков в Российской Федерации, что регламентировано Общей фармакопейной статьей ОФС.1.7.1.0006.15, где дана подробная характеристика штамма (2015).
Культивирование штамма L. fermentum 39. Для восстановления и рассева штаммов бактерий рода Lactobacillus после вскрытия ампулы лиофильную массу заливали 1 мл среды МРС-1 (Lactobacillus MRS broth, “HiMedia”, Индия), переносили в стерильную пробирку и инкубировали при 37°С в течение 24 ч (I генерация штамма). На вторые сутки 0.5 мл I генерации пересевали на МРС-1 и инкубировали при 37°С 24 ч (II генерация штамма). Далее II генерацию штамма раститровывали в стерильном физиологическом растворе (рН 7.0); с отдельных разведений (10–4–10–7) проводили высевы по 0.05 мл на плотную среду МРС-4 (Lactobacillus MRS agar, “HiMedia”, Индия) и инкубировали 48 ч при 37°С в анаэробных условиях с использованием газогенерирующих пакетов GasPak Anaerobe Gas Generating Pouch System with Indicator (“Becton Dickinson”, США).
Идентификация штамма с использованием MALDI TOF масс-спектрометрии. Идентификацию осуществляли с помощью времяпролетного MALDI масс-спектрометра Autoflex (“Bruker Daltonics”, Германия), оснащенного модифицированным твердотельным лазером. Все измерения осуществляли в линейном режиме, детектируя положительные ионы. Внешнюю калибровку проводили с помощью бактериального тест-стандарта (“Bruker Daltonics”, Германия), в качестве матрицы использовали α-циано-4-гидрокси-коричную кислоту (α-CHCA) (“Bruker Daltonics”, Германия).
Пробоподготовку выполняли методом прямого нанесения по стандартному протоколу, представленному в руководстве пользователя. Идентификацию, запись, обработку и анализ масс-спектров проводили с помощью программно-аппаратного комплекса BioTyper (“Bruker Daltonics”, Германия). О достоверности идентификации судили по значению коэффициента совпадения (Score values): 2.000–3.000 – идентификация до вида, 1.999–1.700 – идентификация до рода, 1.699–0 – идентификация не прошла), а также по значению категорий: А – достоверная идентификация до вида, В – достоверная идентификация до рода, С – недостоверный результат.
Изучение биохимических свойств штамма. Биохимические свойства штамма подтверждали с использованием тест-системы API 50CHL (“BioMerieux”, Франция).
Полногеномное секвенирование и аннотирование генома. Для проведения полногеномного секвенирования геномную ДНК выделяли с использованием коммерческого набора QIAamp DNA Mini Kit (“QIAGEN’, Германия), фрагментацию производили с использованием системы ультразвуковой фрагментации Covaris E210 (“Applied Biosystems”, США) согласно инструкции производителя. Очистку смеси и отбор фрагментов 200–700 п.н. проводили при помощи магнитных частиц Agencourt AMPure beads (“Beckman Coulter”, США) и буфера NEBNext Sizing Buffer (“New England Biolabs”, США). Подготовку библиотек осуществляли с помощью набора TrueSeq (“Illumina Inc.”, США), секвенирование выполняли на платформе MiSeq (“Illumina Inc.”, США). Исходные прочтения были обработаны утилитой Trimmomatic со стандартными параметрами для Illumina. Затем обработанные прочтения использовали для сборки генома de novo при помощи программ Spades, MIRA 4.0, Newbler 2.6.
Аннотацию генома производили с помощью утилиты Prokka v. 1.11 (Seemann, 2014), геномного сервера RAST (Rapid annotations using subsystems technology (http://rast.nmpdr.org) и BASys (Bacterial annotation system (https://www.basys.ca). Изучение CRISPR-кассеты, поиск детерминант антибиотикорезистентности и патогенности проводили с использованием программных продуктов, представленных на сайте Центра геномной эпидемиологии (www.cge.cbs.dk): программ ResFinder 2.0, Pathogen Finder и CrisprFinder (Zankari et al., 2012; Cosentino et al., 2013; Grissa et al., 2017).
Подготовка суспензии вирионов. Для изучения взаимодействия лактобактерий с ротавирусами готовили концентрированную суспензию ротавирионов, полученных из образцов фекалий детей, госпитализированных в Нижегородскую инфекционную больницу с острой кишечной инфекцией. Ротавирусы вида Rotavirus А выявляли с использованием коммерческих ПЦР-тест-систем “АмплиСенс Rotavirus-EPh” и “АмплиСенс Rotavirus/Norovirus/Astrovirus-FL” (ФБУН ЦНИИЭ, Россия), согласно инструкции по применению. Образцы фекалий, положительные на содержание ротавируса, разводили стерильным физиологическим раствором для получения 10%-ной суспензии, которую затем осветляли центрифугированием при 7000 об./мин в течение 30 мин. Для получения концентрированного препарата ротавирионов 0.8 мл осветленной суспензии наслаивали на “подушку” 30%-ной сахарозы в TN-буфере (0.02 М Трис, 1 М NaCl; pH 7.4) объемом 400 мкл, центрифугировали на микро-ультрацентрифуге Sorvall MX150 с ротором S140-AT (“Thermo Scientific”, США) в центрифужных пробирках объемом 2 см3 в течение 90 мин при 43 000 об./мин и 4°С (Баррет и соавт., 1988). После ультрацентрифугирования осадок растворяли в 50 мкл дистиллированной воды. Наличие ротавирусов контролировали методом трансмиссионной электронной микроскопии при увеличении ×20 000. При идентификации ротавирионов основывались на специфической структурной организации вириона размером 75 нм, напоминающего колесо с короткими спицами и хорошо различимым ободом (rota – колесо) (Estes, Kapikian, 2007).
Подготовка бактериальной культуры. Штамм Lactobacillus fermentum 39 выращивали на среде Lactobacillus MRS Broth M369 (“HiMedia’, Индия), в течение 18 ч при 37 ± 1°С, осаждали центрифугированием при 3000 об./мин в течение 10 мин. Осадок ресуспендировали в буфере по Хенелю (рН 6.0–6.1), доводя концентрацию клеток до 109 КОЕ/мл. Содержание и целостность клеток оценивали методом трансмиссионной электронной микроскопии при увеличении ×5000.
Трансмиссионную электронную микроскопию (ТЭМ) осуществляли на электронном микроскопе HT 7700 (“Hitachi’, Япония) при увеличениях 5000–20 000. Препараты готовили стандартным методом “микрокапли” с применением электронно-микроскопических сеток с ячейками 400 меш и парлодиевой пленки-подложки. В качестве контрастера использовали 3% раствор фосфорно-вольфрамовой кислоты (ФВК); рН 6.8.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Идентификация штамма. При рассеве штамма L. fermentum 39 на плотную питательную среду МРС-4 наблюдали белые блестящие выпуклые колонии с ровными краями, от 0.5 до 3 мм в диаметре. При помощи MALDI TOF масс-спектрометрии культура идентифицирована как Lactobacillus fermentum, значения Score values составили от 2.0 до 2.217 (категория А), что говорит о достоверной идентификации.
Биохимические свойства штамма. Установлено, что биохимический профиль L. fermentum 39 соответствует заявленному в ОФС.1.7.1.0006.15. Тест-система API 50CHL содержит расширенный перечень из 49 субстратов, позволивший установить особенности метаболизма штамма L. fermentum 39, способность к гидролизу гентиобиозы, тагатозы, туранозы и арбутина. Для более чем 90% штаммов этого вида такие свойства не характерны, что позволяет считать их отличительной меткой данного штамма.
Результаты полногеномного секвенирования. В ходе проведения полногеномного секвенирования штамма была произведена сборка генома de novo, собрано 55 контигов со средним покрытием 250. Общая длина всех контигов составила 1 829 655 н.о., состав пар Г + Ц в ДНК – 51.6%. В процессе аннотации и анализа генома было определено 1683 последовательности, кодирующие белки, 51 последовательность тРНК, 13 – рРНК и 1 CRISPR-локус. Драфт генома депонирован в международной базе данных GenBank под номером LBDG00000000. Полученные характеристики генома сопоставимы с данными научной литературы, например, геном пробиотического штамма Lactobacillus fermentum 47-7, выделенного из кишечника младенца, имеет размер 1.83 Мб, состоит из 1636 протеин-кодирующих генов, содержит 15 рРНК, 57 тРНК и 4 некодирующих РНК (Konyanee et al., 2019). Полногеномная последовательность штамма была проанализирована на наличие детерминант антибиотикорезистентности, патогенности и вирулентности. По данным сервиса PathogenFinder штамм охарактеризован как непатогенный для человека (score = 0.168), детерминант вирулентности и ан-тибиотикорезистентности выявлено не было.
Способность к утилизации спектра сахаров традиционно лежит в основе фенотипической идентификации лактобацилл, и гены метаболизма сахаров являются одним из наиболее изученных кластеров их генома. В связи с этим был проведен детальный анализ сегмента генома, отвечающего за транспорт и утилизацию сахаров. Установлено, что микроорганизм обладает всем необходимым комплексом ферментов пентозофосфатного пути и ферментами, обеспечивающими транспорт и метаболизм сахаров. В геноме детерминированы опероны метаболизма и транспорта лактозы, арабинозы, сахарозы, галактозы, трегалозы и маннозы.
С использованием инструмента KEGG (Kyoto encyclopedia of genes and genomes) из пакета RAST установлено, что пентозофосфатный путь исследуемого микроорганизма не имеет особенностей. Вариант метаболического кода штамма согласно RAST – 5.1991, что указывает на невозможность осуществления гликолиза за счет отсутствия ключевого фермента этого пути – фосфофруктокиназы. Полученные результаты соответствуют данным научной литературы и отражают особенности метаболизма облигатно гетероферментативных лактобацилл группы С (Hammes, Hertel, 1992).
CRISPR (clustered regularly interspaced short palindromic repeats) – механизм, защищающий клетку от “генетической агрессии” и предотвращающий проникновение в клетку бактериофагов и плазмид (Равин, Шестаков, 2013). С целью поиска штаммоспецифичных особенностей был проанализирован CRISPR-локус штамма, содержащий высоко консервативные белки (Cas-белки) и короткие палиндромные повторы, разделенные спейсерами.
Установлено, что данный оперон находится в пределах 43 контига (NCBI: NZ_LBDG01000018.1), в его структуру входит комплекс cas-белков и 25 повторов, длиной 29 нуклеотидов, которые разделяют 24 уникальных спейсера (рис. 1).
Рис. 1.
Структурная организация CRISPR-локуса L. fermentum 39. Стрелками обозначены соответствующие Cas-белки, ромбами обозначены повторы, прямоугольниками – уникальные спейсеры.

Необходимо отметить, что данные о структуре данного региона и полиморфных спейсеров лежат в основе CRISPR-типирования микроорганизмов (Horvatha et al., 2009; Платонов и соавт., 2013). При анализе организации оперона и представленности Cas-белков (наличие CAS3 белка) выявлено, что система CRISPR/Cas данного штамма относится к I типу (Makarova et al., 2015). При исследовании палиндромного повтора с использованием BLAST установлено, что его последовательность аналогична последовательности повтора CRISPR–Cas системы штаммов L. fermentum CECT 5716 и L. fermentum IFO 3956. В ходе анализа последовательности спейсеров в базе данных GenBank гомологичных последовательностей не было выявлено. Последовательности неидентифицированных спейсеров также можно рассматривать как возможные “метки штамма”.
Трансмиссионная электронная микроскопия. Изучаемый нами штамм таксономически относится к семейству Lactobacteriaceae, роду Lactobacterium, является представителем вида L. fermentum – облигатных гетероферментативных лактобактерий группы С. Морфологически лактобактерии представляют собой грамположительные палочки размером 4–15 × 0.5–0.6 мкм, встречаются изогнутые и булавовидные формы (коринеформы), также короткие коккобактерии. Они, как правило, неподвижны, спор и капсул не образуют (The Firmicutes, 2009).
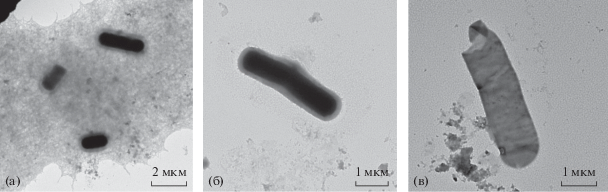
Как видно на электронных микрофотографиях клетки из свежевыращенной культуры (24 ч культивирования) L. fermentum 39 (рис. 2а) представляют собой толстые короткие палочки размером 0.56–0.7 × 1.8–2.0 мкм. Клетки способны производить экзополисахарид, который формирует вокруг тела клетки чехол и экспортируется в окружающую среду (рис. 2б). Со временем клетки увеличиваются, достигая размера 0.8–1.0 × 3.5–4.0 мкм, и принимают форму удлиненных палочек; экзополисахарид уплотняется, формируя слой толщиной 0.2–0.5 мкм. Клетки в “старой” культуре (3–10 сут культивирования) могут достигать размера 1.2 × 4.5 мкм, окружающий клетки массивный слизистый чехол в ряде случаев способен слущиваться с их поверхности (рис. 2в).
Полученные данные подтверждаются данными научной литературы, где указано, что штаммы L. fermentum способны продуцировать экзополисахарид (ЭПС) (Dan et al., 2009; Castro-Bravo et al., 2018; Oleksy, Klewicka, 2018), причем молекулы ЭПС у бактерий данного рода, в отличие от бактерий семейств Streptococcus и Staphylococcus, не имеют ковалентной связи с поверхностью клетки, что обуслoвливает его слущивание (Kang et al., 2015).
Ряд исследователей рассматривает наличие ЭПС как механизм адаптации, позволяющий данному микроорганизму приспосабливаться к изменяющимся условиям окружающей среды, в частности, реализовывать механизм адгезии, эффективно колонизировать и персистировать в ЖКТ (Badel at al., 2011: Хусаинов, 2014). Кроме того, образование экзополисахаридного матрикса позволяет предотвращать инфицирование культуры фагами, ЭПС служит резервным источником углерода при дефиците питательных веществ (Badel at al., 2011).
По мнению ряда исследователей для вида L. fermentum характерен синтез гомополисахаридов, в частности, α-глюкана (декстрана), по другим данным микроорганизмы этого вида продуцируют гетерополисахариды, включающие глюкозу и галактозу и состоящие из остатков α-D-глюкозо-1-Р, β-D-глюкозо-1-Р и α-D-галактозо-1-Р (Dan et al., 2009; Хусаинов, 2014; Castro-Bravo et al., 2018; Oleksy, Klewicka, 2018).
Кластер генов синтеза экзополисахаридов у изучаемого штамма находится в пределах 20 контига (NCBI: LBDG01000035.1) и представлен совокупностью детерминант (рис. 3).
Рис. 3.
Организация кластера синтеза экзополисахарида в геноме штамма Lactobacillus fermentum 39. EpsE – уденилкарпил-фосфат галактосефосфотрансфераза (ЕС 2.7.8.6) Wu68_08600; EpsF – гликозилтрансфераза (ЕС.2.4.1) Wu68_08595; EpsB – марганец-зависимая протеин-тирозин фосфатаза (ЕС 3.1.3.48) Wu68_08610; EpsD – тирозин-протеин киназа (ЕС 2.7.10.2) Wu68_08615; EpsC – тирозин-протеин трансмембранный модулятор Wu68_08620.
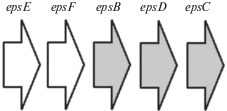
Анализ eps кластера Lactobacillus fermentum 39 позволил предположить, что штамм способен продуцировать гетерополисахарид с помощью реализации внутриклеточного механизма синтеза–перенос фосфатных групп на молекулу глюкозы и сборку “повторяющихся единиц” (repeating units) из глюкозы и галактозы посредством ферментов EpsE и EpsF, формирование из полученных единиц молекулы полисахарида во внеклеточном пространстве и регуляцию длины его цепи с помощью комплекса ферментов – EpsD, EpsC, EpsВ.
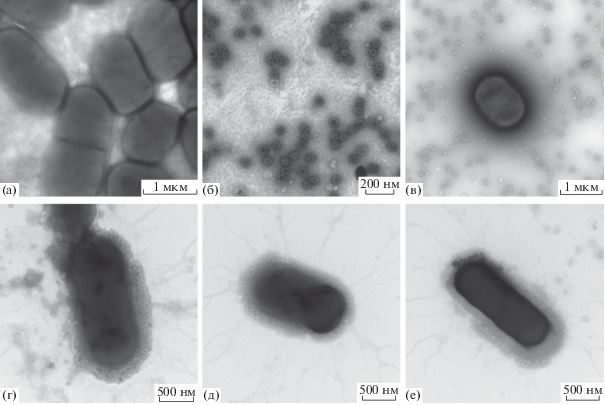
Антивирусная активность штамма. С целью изучения активности штамма Lactobacillus fermentum 39 против ротавируса готовили суспензию клеток в буфере по Хенелю (рН 6.0–6.1) с концентрацией 109 КОЕ/мл. На рис. 4а представлена типичная для лактобактерий морфология клеток Lactobacillus fermentum 39. Также использовали водную суспензию ротавирионов, относительное содержание которых в препарате оценивали методом электронной микроскопии, показавшей наличие 10–15 вирионов на 1 мкм2 пленки-подложки. В препарате были сделаны единичные находки бактериофагов с длинным несократительным хвостовым отростком (Siphoviridae), что, однако, не мешало решению поставленной задачи.
На рис. 4б видны вирионы ротавируса, имеющие характерную структурную организацию – напоминающие колесо с короткими спицами и хорошо различимым ободом (rota – колесо). На фото наблюдается смешанный контраст: контрастер окрашивает не только подложку, на которой расположен вирус, но и проникает внутрь вируса.
Подготовленные препараты клеток Lactobacillus fermentum 39 и ротавирионов смешивали в соотношении 1 : 1, конечный объем 100 мкл (рис. 4в). Затем смесь инкубировали в течение 10 мин при температуре 36.6°С и центрифугировали при 10 000 об./мин в течение 10 мин. Осадок ресуспендировали в дистиллированной воде, с использованием метода “микрокапли” готовили электронно-микроскопические препараты и просматривали в электронный микроскоп при увеличении ×5000 и ×20 000, как осадок, так и супернатант.
В препаратах супернатанта ротавирионы обнаружены не были. Кроме этого, перед экспериментом препарат ротавируса проверяли на способность вирионов к самостоятельному осаждению в условиях центрифугирования при 10 000 об./мин в течение 10 мин. Контроль осадка показал отсутствие ротавирионов.
В препаратах осадков, полученных из смеси лактобактерий и ротавирионов, на поверхности клеток лактобактерий внутри экзополисахрида наблюдались многослойные образования, состоящие из округлых структур разной формы и размера (рис. 4г–4е). При этом основную массу структур составляли округлые частицы размером ~75 нм с четко очерченным краем. Наличие экзополисахарида не позволило установить ультратонкую организацию этих частиц, чтобы убедительно утверждать их принадлежность к ротавирионам. Однако отсутствие ротавиринов в супернатанте центрифугированной смеси, их неспособность самостоятельно осаждаться при 10 000 об./мин и наличие вирусоподобных частиц в составе экзополисахаридного матрикса лактобактерий свидетельствуют о способности штамма Lactobacillus fermentum 39 неспецифически адсорбировать на своей поверхности ротавирионы.
Эффект адгезии вирусных частиц на поверхности бактериальных клеток, поверхности экзополисахарида и жгутиках бактерий – представителей нормофлоры кишечника человека отмечали и ранее (Гостева, 1984; Шелковая и соавт., 1991). Согласно данным научной литературы экзополисахарид бактерий играет важную роль в адгезии вирусов на поверхности бактериальной клетки, обеспечивая ультраструктурные взаимодействия с ротавирусами, хотя точный механизм этого процесса пока неизвестен. Сам эффект адгезии рассматривается как штаммоспецифичный признак и присущ не всем представителям рода Lactobacillus (Botic et al., 2007; Lei et al., 2016).
Эффект адгезии вирионов на поверхности клеток L. fermentum 39 можно расценивать как фактическое подтверждение важной биологической роли пробиотических бактерий в защите кишечника от инфицирования патогенными вирусами, штамм может выступать в роли естественного и высоко эффективного сорбента, связывая большое число вирусных частиц и обеспечивая их исключение из пула свободных вирионов, способных к инфицированию здоровых клеток слизистой кишечника.
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВНастоящая статья не содержит каких-либо исследований с использованием животных в качестве объектов.КОНФЛИКТ ИНТЕРЕСОВВ настоящей статье отсутствует конфликт интересов.
Список литературы
Вирусология. Методы: Пер. с англ. / Баррет Т., Берд. П., Клегг Дж., Гулд Э. и др. Под ред. Мейхи Б. М.: Мир, 1988. 344 с.
Гостева В.В. Взаимодействие бактерий, вирусов и э-укариотических клеток в условиях смешанной инфекции (электронно-микроскопическое исследование). Дис. … канд. биол. н. Москва, 1984. 153 с.
Ермоленко Е.И., Суворов А.Н., Фураева В.А. Противовирусный эффект in vitro метаболитов, выделяемых культурами энтерококка и лактобацилл // Материалы VI Российского съезда врачей инфекционистов, 2003. С. 371.
Общая фармакопейная статья Лактосодержащие пробиотики. ОФС.1.7.1.0006.15. МЗ РФ. 2015. 10 с. [Электронный ресурс]. Режим доступа https://pharmacopoeia.ru/ofs-1-7-1-0006-15-laktosoderzhashhie-probiotiki. Дата обращения 25.04.2019.
Платонов М.Е., Евсеева В.В., Дентовская С.В., Анисимов А.П. Молекулярное типирование Yersinia pestis // Мол. генетика, микробиология и вирусология. 2013. № 2. С. 3–12.
Platonov M.E., Evseeva V.V., Dentovskaya S.V., Anisimov A.P. Molecular typing of Yersinia pestis // Mol. Genet. Microbiol. Virol. 2013. V. 28. P. 41–51.
Равин Н.В., Шестаков С.В. Геном прокариот // Вавиловский журнал генетики и селекции. 2013. Т. 17. С. 972–984.
Ravin N.V., Shestakov S.V. The genome of prokaryotes // Russ. J. Gen.: Applied Research. 2013. V. 17. P. 972–984.
Хусаинов И.А. Современные представления о биосинтезе бактериальных экзополисахаридов // Вестник Казанского технологического университета. 2014. Т. 17. № 5. С. 167–172.
Шелковая Н.Г., Купчинский Л.Г., Знаменский В.А., Бондаренко В.М. Электронно-микроскопическое изучение взаимодействия бактериальной кишечной микрофлоры и вирионов ротавируса // ЖМЭИ. 1991. № 9. С. 18–21.
Ahmadi E., Alizadeh-Navaei R., Rezai M.S. Efficacy of probiotic use in acute rotavirus diarrhea in children: A systematic review and meta-analysis // Caspian J. Intern. Med. 2015. V. 6. P. 187–195.
Ang L.Y., Too H.K., Tan E.L., Chow T.K., Shek L.P., Tham E.H., Alonso S. Antiviral activity of Lactobacillus reuteri protectis against Coxsackievirus A and Enterovirus 71 infection in human skeletal muscle and colon cell lines // Virol. J. 2016. V. 13. P. 111. https://doi.org/10.1186/s12985-016-0567-6
Arena M.P., Elmastour F., Sane F., Drider D., Fiocco D., Spano G., Hober D. Inhibition of Coxsackievirus B4 by Lactobacillus plantarum // Microbiol. Res. 2018. V. 210. P. 59–64. https://doi.org/10.1016/j.micres.2018.03.008
Badel S., Bernardi T., Michaud P. New perspectives for Lactobacilli exopolysaccharides // Biotechnol. Adv. 2011. V. 29. P. 54–66.
Botić T., Klingberg T.D., Weingart H., Cencic A. A novel eu-karyotic cell culture model to study antiviral activity of potential probiotic bacteria // Int. J. Food Microbiol. 2007. V. 115. P. 227–234. https://doi.org/10.1016/j.ijfoodmicro.2006.10.044
Castro-Bravo N., Wells J.M., Margolles A., Ruas-Madiedo P. Interactions of surface exopolysaccharides from Bifidobacterium and Lactobacillus within the intestinal environment // Front. Microbiol. 2018. V. 9. P. 2426. https://doi.org/10.3389/fmicb.2018.02426
Cosentino S., Voldby L.M., Moller A.F., Lund O. Pathogen-Finder – distinguishing friend from foe using bacterial whole genome sequence data // PLoS One. 2013. V. 8. e77302. https://doi.org/10.1371/journal.pone.0077302
Dan T., Fukuda K., Sugai-Bannai M., Takakuwa N., Motoshima H., Urashima T. Characterization and expression analysis of the exopolysacharide gene cluster in Lactobacillus fermentum TDS030603 // Biosci. Biotechnol. Biochem. 2009. V. 73. P. 2656–2664.
Estes M.K., Kapikian A.Z. Rotaviruses // Fields Virology / Eds. Fields B.N., Knipe D.M., Howley P.M. Philadelphia: Wolters Kluwer Health/Lippincott Williams & Wilkins, 2007. P. 1917–1973.
Grissa I., Vergnaud G., Pourcel C. CRISPRFinder: a web tool to identify clustered regularly interspaced short palindromic repeats // Nucl. Acids Res. 2017. V. 35. P. 52–57.
Hammes W.P., Hertel C. The genus of Lactobacillus and Carnobacterium // The Procaryotes / Eds. Balows A., Truper H.G., Dworkin M., Harder W., Schleifer K.-H. Edn. 2. N.Y.: Springer-Verlag, 1992. V. 4.
Horvatha Ph., Coûté-Monvoisina A.C., Romerob D.A., Boyavala P., Fremauxa Ch., Barrangoub R. Comparative analysis of CRISPR loci in lactic acid bacteria genomes // Int. J. Food Microbiol. 2009. V. 131. P. 62–70. https://doi.org/10.1016/j.ijfoodmicro.2008.05.030
Isolauri E. Probiotics for infectious diarrhea // Gut. 2003. V. 52. P. 436–437.
Kang H., Gilbert C., Badeaux F., Atlan D., LaPointe G. A tyrosine phosphorylation switch controls the interaction between the transmembrane modulator protein Wzd and the tyrosine kinase Wze of Lactobacillus rhamnosus // BMC Microbiol. 2015. V. 15. P. 40. https://doi.org/10.1186/s12866-015-0371-2
Kim K., Lee G., Thanh H.D., Kim J.H., Konkit M., Yoon S., Park M., Yang S., Park E., Kim W. Exopolysaccharide from Lactobacillus plantarum LRCC5310 offers protection against rotavirus-induced diarrhea and regulates inflammatory response // J. Dairy Sci. 2018. V. 101. P. 5702–5712. https://doi.org/10.3168/jds.2017-14151
Konyanee A., Yotpanya P., Panya M., Engchanil C., Suebwongsa N., Namwat W., Thaw H., Faksri K., Sankuntaw N., Lulitanond V. Genome sequence of Lactobacillus fermentum 47-7, a good in vitro probiotic strain isolated from a healthy Thai infant // Microbiol. Res. Announc. 2019. V. 8. e01014-19. https://doi.org/10.1128/MRA.01014-19
Lei S., Ramesh A., Twitchell E., Wen K., Bui T., Weiss M., Yang X., Kocher. J., Li G., Giri-Rachman E., Trang N.V., Ji-ang X., Ryan E.P., Yuan L. High protective efficacy of probiotics and rice bran against human norovirus infection and diarrhea in gnotobiotic pigs // Front. Microbiol. 2016. P. 1699. https://doi.org/10.3389/fmicb.2016.01699
Majamaa H., Isolauri E., Saxelin M., Vesikari T. Lactic acid bacteria in the treatment of acute rotavirus gastroenteritis // J. Pediatr. Gastroenterol. Nutr. 1995. V. 3. P. 333–338.
Makarova K.S., Wolf H., Alkhnbashi O.S., Costa F., Shah S.A., Saunders S.J., Barrangou R., Brouns S.J.J., Charpentier E., Haft D.H., Horvath P., Moineau S., Mojica F.J.M., Terns R.M., Terns M.P., White M.F., Yakunin A.F., Garrett R.A., van der Oost J., Backofen R., Koonin E.V. An updated evolutionary classification of CRISPR-Cas systems // Nat. Rev. Microbiol. 2015. P. 722–736. https://doi.org/10.1038/nrmicro3569
Oleksy M., Klewicka E. Exopolysaccharides produced by Lactobacillus sp.: Biosynthesis and applications // Crit. Rev. Food Sci. Nutr. 2018. V. 58. P. 450–462. https://doi.org/10.1080/10408398.2016.1187112
Salminen S., Bouley C., Boutron-Ruault M.C., Cummings J.H., Franck A., Gibson G.R., Isolauri E., Moreau M.C., Roberfroid M., Rowland I. Functional food science and gastrointestinal physiology and function // Br. J. Nutr. 1998. V. 80. P. S147–S171.
Seemann T. Prokka: rapid prokaryotic genome annotation // Bioinformatics. 2014. V. 30. P. 2068–2069. https://doi.org/10.1093/bioinformatics/btu153
Sunmola A.A., Ogbole O.O., Faleye T.O.C. Adetoye A., Adeniji J.A., Ayeni F.A. Antiviral potentials of Lactobacillus plantarum, Lactobacillus amylovorus, and Enterococcus hirae against selected Enterovirus // Folia Microbiol. 2019. V. 64. P. 257–264. https://doi.org/10.1007/s12223-018-0648-6
The Firmicutes // Bergey’s Manual of Systematic Bacteriology / Eds. Vos P., Garrity G., Jones D., Krieg N.R., Ludwig W., Rainey F.A., Schleifer K.-H., Whitman W.B. 2nd edn. New York: Springer, 2009. V. 3. 1450 p.
Zankari E., Hasman H., Cosentino S., Vestergaard M., Rasmussen S., Lund O., Aarestrup F.M., Larsen M.V. Identification of acquired antimicrobial resistance genes // J. Antimicrob. Chemother. 2012. V. 67. P. 2640–2644. https://doi.org/10.1093/jac/dks261
Дополнительные материалы отсутствуют.